
* 通讯作者(Corresponding author): 官春云, E-mail: guancy2011@aliyun.com 第一作者联系方式: E-mail: ttl205@aliyun.com
收稿日期:2015-11-13 接受日期:2016-03-02网络出版日期:2016-03-11基金:
摘要
关键词:甘蓝型油菜; 磷脂二酰甘油酰基转移酶; 基因克隆; 酵母互补实验
Cloning and Characterization of Phospholipids:Diacylglycerol Acyltransferase (BnPDAT1) cDNA from Brassica napus L
TAN Tai-Long, FENG Tao, LUO Hai-Yan, PENG Ye, LIU Rui-Yang, GUAN Chun-Yun

Fund:
Abstract
Keyword: Brassica napus L; PDAT1; Gene clone; Yeast complementary experiment
Show Figures
Show Figures













甘蓝型油菜通过不同途径合成三酰甘油(triacylglycerol, TAG)并形成油体储藏于种子中, 这种储藏油脂是我国最重要的油料来源之一, 对于保证食用油供给十分重要。我国生产的甘蓝型油菜含油量为40%左右, 与加拿大等发达国家尚具较大差距, 提高菜籽含油量可明显提高我国油菜的市场竞争力, 产生可观的社会效益[1]。因此, 研究甘蓝型油菜种子中TAG合成与积累具有重要的意义。
植物油脂代谢具有高度的生物学复杂性, 如拟南芥的油脂代谢网络涉及600多个基因[2], 多个基因影响TAG的合成量。植物TAG合成分为脂肪酸链形成和TAG组装2个过程, 其中TAG组装量直接决定植物种子TAG的总量。植物以三磷酸甘油(sn-glycerol- 3-phosphate, G3P)为骨架, 以脂肪酸(fatty acid, FA)为底物通过一系列酶促反应合成TAG有两种途径。其一, Kennedy途径, 以甘油-3-磷酸酰基转移酶(sn-glycerol-3-phosphate acyltransferase, GPAT)、溶血磷脂酸酰基转移酶(lysophosphatidic acid acyltransferase, LPAAT)、磷脂酸酯酶(phosphatidate phosphatase, PAP)和二脂酰甘油酰基转移酶(diacylglycerol acyltransferase, DGAT)连续催化合成TAG[3]; 其二, 不依赖脂酰-CoA的合成途径(即PDAT途径), 以磷脂二酰甘油酰基转移酶(phospholipids:diacyl- glycerol acyltransferase, PDAT)催化二酰甘油(diacylglycerol, DAG)与磷脂(phospholipids, PC)反应合成TAG与溶血磷脂(lyso-phospholipids, LPC)。两条途径对植物种子油脂累积具有重要的生理作用, 直接影响TAG合成的关键酶分别是DGAT与PDAT。PDAT途径最早发现于酿酒酵母[4], 并证实PDAT对于酵母TAG合成的贡献。以酵母PDAT为探针在拟南芥中获得2个PDAT基因At5g13460 (AtPDAT1)和At3g44830 (AtPDAT2), 对AtPDAT1的功能研究显示了其PDAT酶活性, 能催化不同链长的酰基转移, 尤其是对多双键、环氧化以及羟化酰基具有偏好性, 显示了PDAT在拟南芥TAG合成中的重要作用[5]。PDAT途径在甘蓝型油菜TAG合成中的贡献尚未得到充分研究, 克隆甘蓝型油菜PDAT基因并进行功能解析, 将促进油菜TAG合成路径的完善以及高含油油菜育种的进程。本文通过拟南芥AtPDAT1基因序列设计引物, 从甘蓝型油菜品种湘油15号中克隆获得3个PDAT拷贝, 并对其BnPDAT1进行酵母互补功能验证, 证实BnPDAT1参与TAG合成, 具备PDAT功能活性。
1 材料与方法1.1 试验材料甘蓝型油菜双低品种湘油15号由国家油料改良中心湖南分中心提供。用于异源表达分析的野生酵母INVSc1及用于PDAT1基因功能鉴定的三酰甘油(TAG)合成缺陷酵母菌株H1246由徐荣华博士(中国科学院西双版纳热带植物园)惠赠。
1.2 RNA提取与cDNA合成在22℃、光周期为16 h/8 h条件下培养湘油15号至2片真叶, 取幼苗50~100 mg。加液氮充分研磨后, 使用TRIzol RNA提取试剂盒(TransGen生物技术有限公司)按其说明书方法提取总RNA, 以此RNA为模板合成第1链cDNA, 参见Easy Script First-Strand cDNA Synthesis SuperMix试剂盒(TransGen生物技术有限公司)说明书合成cDNA。
1.3 BnPDAT1基因的克隆及序列分析从拟南芥数据库(http://www.arabidopsis.org/)获取AtPDAT1基因序列。使用Bioinformatics at Geboc数据库(http://www.bioyq.com/blast/blast.php) “ Regular Blast” 进行检索, 获得白菜(AA)和甘蓝(CC)的PDAT1基因序列以及甘蓝型油菜已知的EST序列。用Vector软件拼接获得油菜EST序列, 并与白菜和甘蓝PDAT1基因序列比对。分析比对结果, 在5'端选取一致性较好的区域, 设计含起始密码子的5'端引物, 同法设计一段含有终止密码子的3'端引物, 合成备用。设计的引物序列为BnPDAT1-F: 5'-ATGCCCCTTTTTCAGCGGAAAAAGC-3'; BnPD AT1-R: 5'-TCACAGCTTCAAGTCAATACGCTCA-3'。以1.2所述cDNA为模板进行PCR扩增。PCR体系含50 ng μ L-1模板1 μ L、10 mmol L-1 dNTPs 0.5 μ L、2 μ mol L-1 BnPDAT1-F 0.75 μ L、2 μ mol L-1 BnPDAT1-R 0.75 μ L、10× 反应缓冲液1 μ L、HiFi高保真DNA聚合酶0.5 μ L和ddH2O 15.5 μ L。程序为预变性95℃ 5 min, 95℃ 50 s, 58℃ 45 s, 72℃ 3 min, 35个循环, 72℃后延伸10 min。PCR产物经1.5%琼脂糖凝胶电泳, 用凝胶成像系统(Bio-Rad)检测和记录结果。将PCR产物回收后与pMD18-T载体(TransGen生物技术有限公司)连接, 转化大肠杆菌感受态细胞Trans1-T1 (北京天根生化有限公司), 筛选阳性克隆进行基因测序。通过BRAD数据库(http://brassicadb.org/brad/)对获得的克隆序列进行染色体步移分析。使用DNAman对克隆获得的PDAT1基因全长CDS序列进行序列比对。
1.4 BnPDAT1蛋白生物信息学分析1.4.1 BnPDAT1氨基酸序列特征与二级结构预测
通过ANTHEPROT 6.2软件分析来源于甘蓝型油菜的PDAT1拷贝所编码的蛋白质。统计编码蛋白的氨基酸残基组分、分析编码蛋白的等电点、信号肽序列和亲疏水特征并使用Gariner模型预测二级结构。使用PSORT (http://psort.hgc.jp/form.html)进行亚细胞定位预测。
1.4.2 BnPDAT1蛋白同源结构域分析 使用DNAman对BnPDAT1蛋白进行同源比对分析; 同时使用NCBI在线软件(http://blast.ncbi.nlm.nih.gov/)分别对不同的BnPDAT1蛋白序列进行保守结构域分析; 使用SMART (http://smart.embl-heidelberg.de/)及InterPro (http://www.ebi.ac.uk/interpro/)分析BnPDAT1包含的特殊结构域。
1.4.3 PDAT1蛋白聚类分析 通过NCBI蛋白数据库(http://www.ncbi.nlm.nih.gov/protein)、String数据库(http://string.embl.de/)、PDB数据库(http://www. pdb.org/pdb/home/home.do)检索来源于不同物种的PDAT1相关蛋白序列, 并使用MEGA5.0对来源不同的PDAT1相关蛋白进行聚类分析。
1.5 BnPDAT1基因的酵母互补功能验证1.5.1 酵母表达载体构建、转化与酵母培养 将获得的BnPDAT1基因与HA标签基序融合连接PYES2.0载体, 用氯化锂法转化到酵母菌株H1246 [酵母突变体H1246中编码TAG合成的DGA1 (编码DGAT)和LRO1 (编码PDAT)以及编码固醇脂合成的基因ARE1和ARE2都被敲除了, 因此H1246不能合成中性油脂, 且H1246不能形成油体][6], 同时将对照质粒(未连接BnPDAT1基因的PYES2.0载体)转化入酵母菌株H1246和INVSc1。
参考徐荣华等[7]的方法培养酵母, 将转BnPDAT1基因的酵母(H1246)及转对照质粒的酵母(H1246与INVSc1)在尿嘧啶缺陷的合成完全培养基(synthetic minimal defi ned medium lacking uracil, SC-U)抑制培养基(2%葡萄糖)中220转min-1、30℃过夜培养, 以紫外分光光度计测OD600后, 离心收集菌体, 用SC-U诱导培养基(2%半乳糖)重悬菌体至OD600= 0.4, 在220转min-1、30℃条件下培养, 诱导表达培养36 h, 收集备用。
1.5.2 BnPDAT1基因在酵母中的表达分析 使用TRIzol RNA提取试剂盒(TransGen生物技术有限公司)分别提取转BnPDAT1基因酵母(H1246)及转对照质粒酵母的总RNA, DNaseI消化后以1.5%甲醛变性琼脂糖凝胶电泳检测, 分别根据克隆到的BnPDAT1基因设计特异性引物, 以RT-PCR检测目的基因的表达。
1.5.3 转BnPDAT1基因酵母的蛋白印迹分析
参考Isola等[8]的方法提取转BnPDAT1基因和空载体的酵母总蛋白, 用于蛋白印迹分析。参照鲁少平等[9]的方法进行Western blot。一抗使用HA标签抗体, 从山羊中提取获得, 二抗使用小鼠抗山羊抗体, ECL发光检测。
1.5.4 转BnPDAT1基因酵母的油脂薄层层析(thin- layer chromatography, TLC) 使用液氮反复冻融并研磨破碎酵母细胞, 使用正己烷∶ 异丙醇(3∶ 2)法[10]提取酵母油脂进行TLC分析, 分析不同酵母菌株中三酰甘油(TAG)、游离脂肪酸(FFA)和二酰甘油(DAG)的合成。
1.6 甘蓝型油菜中BnPDAT1基因时空表达分析 以1.2方法提取甘蓝型油菜湘油15号不同时期的种子、根、叶、花和角果皮总RNA, 按照宝生物的PrimeScript RTreagent Kit with gDNA Eraser (Perfect Real-time)试剂盒说明进行反转录, 获得cDNA第1链作为qRT-PCR模板。仔细分析1.3中克隆获得的BnPDAT1片段, 从BRAD数据库中获得其上下游非翻译区(untranslated region, UTR), 根据BnPDAT1各拷贝UTR特点设计分型引物, 以UBC21作为内参基因[11], 用于BnPDAT1的时空表达分析, 每样品进行3次重复。在ABI7500荧光定量PCR系统上运行qRT-PCR。PCR体系包含1 μ L cDNA、10 μ L 2× FastStart Universal SYBR GreenMaster with ROX、10 μ mol L-1正向引物和反向引物各0.5 μ L、8 μ L ddH2O。PCR程序为95℃ 10 min; 95℃ 15 s, 60℃ 15 s, 72℃ 32 s, 35个循环。反应完成后绘制95℃ 20 s, 60℃ 20 s, 95℃ 20 s, 59℃ 20 s熔解曲线, 检测扩增产物的正确性和引物二聚体。
2 结果与分析2.1 BnPDAT1基因全长CDS序列的克隆以湘油15号cDNA为模板, 克隆到3个CDS序列, 长度分别为1998、2002和2005 bp, 分别定位于A02、A10和C09染色体, 分别命名为BnPDAT1- A02、BnPDAT1-A10和BnPDATI-C09。通过BRAD数据库比对, 确认3个CDS序列的完整性, 并以3条CDS的共同序列作为模板对BRAD数据库进行检索, 未发现更多PDAT1同源序列。通过DNAman多序列比对发现, BnPDAT1-A02、BnPDAT1-A10和BnPDATI-C09相似度为95.29%, 显示了所克隆的PDAT1拷贝在进化上的高度同源性, 也体现了甘蓝型油菜基因组异源多倍性的特点。
2.2 BnPDAT1生物信息学分析BnPDAT1-A02、BnPDAT1-A10和BnPDATI-C09基因各自编码长度665、666、667氨基酸残基的蛋白, 分别命名为BnPDAT1-A02、BnPDAT1-A10和BnPDATI-C09。这3个预测的BnPDAT1蛋白的氨基酸组成、摩尔质量、比体积和等电点见表1。
表1
Table 1
表1(Table 1)
| 表1 BnPDAT1蛋白信息汇总表 Table 1 Summary of the deduced BnPDAT1 proteins |
亲疏水性分析显示, BnPDAT1蛋白疏水区域分布比较均衡, 疏水性氨基酸多于亲水性氨基酸, 且N端有明显的亲水区域。未发现BnPDAT1具信号肽序列, 暗示其不存在跨膜转运, 其合成与功能定位处于相同的亚细胞结构域。PSORT亚细胞定位预测显示, BnPDAT1被定位于质膜的可信度为0.790, 显示BnPDAT1是一种膜锚定蛋白。
BnPDAT1-A02、BnPDAT1-A10和BnPDAT1- C09的二级结构预测结果如图1, BnPDAT1-A02具44% α 螺旋, 30% β 折叠, 13% β 转角, 13%无规卷曲; BnPDAT1-A10 相应的比例为33%、21%、24%和22%; BnPDAT1-C09相应的比例为33%、21%、24%和22%。
图1
Fig. 1
| Figure OptionViewDownloadNew Window | |
 | 图1 BnPDAT1蛋白二级结构Fig. 1 Secondary structure of BnPDAT1 |
通过SMART分析发现, BnPDAT1蛋白在结构上都具有相似序列特征和相近排布位置的一个低复杂度区域(LCC)和一个跨膜结构域(TM), 如表2所示。
表2
Table 2
表2(Table 2)
| 表2 BnPDAT1-A02、BnPDAT1-A10和BnPDAT1-C09蛋白特殊结构汇总表 Table 2 Summary of specific region of BnPDAT1-A02, BnPDAT1-A10, and BnPDAT1-C09 |
同源比对(图2)发现, BnPDAT1蛋白存在4段保守的α /β 水解酶折叠区(α /β HF)。通过NCBI数据库检索发现, BnPDAT1蛋白在100 (N末端)~300 (C末端)区间内符合PLN02733超家族特征; 全长序列基本符合PLN02517超家族特征, PLN02733与PLN02517均为磷脂酰胆碱酰基转移酶超家族, 因此预测, BnPDAT1具有酰基转移功能, 这与报道的PDAT1蛋白功能一致。
图2
Fig. 2
| Figure OptionViewDownloadNew Window | |
 | 图2 BnPDAT1氨基酸序列比对Fig. 2 Comparison of the amino acid sequences of BnPDAT1 |
通过不同数据库, 共获得30种来源于不同植物的PDAT1相关蛋白序列, 聚类分析结果如图3。
图3
Fig. 3
| Figure OptionViewDownloadNew Window | |
 | 图3 不同植物PDAT1蛋白的进化树分析Cs: 亚麻芥; At: 拟南芥; Br: 甘蓝; Bn: 甘蓝型油菜; Th: 醉蝶花; Si: 芝麻; Csi: 橙子; Gm: 大豆; Jc: 麻风树; Rc: 蓖麻; Fv: 野草莓; Co: 油茶; Nn: 荷花; St: 马铃薯。Fig. 3 Phylogenetic analysis of PDAT1 in different plant speciesCs: Camelina sativa; At: Arabidopsis thaliana; Br: Brassica rapa; Bn: Brassica nupas; Th, Tarenaya hassleriana; Si: Sesamum indicum; Csi: Citrus sinensis; Gm: Glycine max; Jc: Jatropha curcas; Rc: Ricinus communis; Fv: Fragaria vesca; Co: Camellia oleifera; Nn: Nelumbo nucifera; St: Solanum tumerosum. |
PDAT1相关蛋白聚类上分为单、双子叶PDAT1以及单、双子叶PDAT1类似蛋白4大类, PDAT1蛋白在单双子叶植物之间有较远的遗传距离。BnPDAT1与甘蓝、拟南芥、亚麻芥PDAT1蛋白具有较高的同源性。PDAT1及相关蛋白在进化树中的位置与相对应的物种进化上的位置比较一致, 显示PDAT1蛋白在起源上可能比较古老。
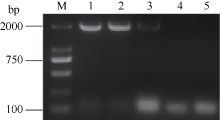
2.3 BnPDAT1基因的酵母互补功能验证2.3.1 BnPDAT1基因在酵母中正常表达 RT-PCR分析结果(图4)显示在转基因酵母(H1246)中, BnPDAT1基因正常编码了mRNA, 而对照组酵母菌株(转空载体的H1246和INVSc1)中无BnPDAT1基因编码的mRNA表达。
图4
Fig. 4
| Figure OptionViewDownloadNew Window | |
 | 图4 转基因酵母菌株的RT-PCR检测M: 2k DNA分子量参照; 1: H1246转PYE+BnPDAT1-A02; 2: H1246转PYE+BnPDAT1-A10; 3: H1246转PYE+BnPDAT1-C09; 4: H1246转PYE; 5: INVSc1转PYE。Fig. 4 RT-PCR analysis of transgenic yeastM: 2k DNA marker; 1: H1246 transformed with PYE+BnPDAT1-A02; 2: H1246 transformed with PYE+BnPDAT1-A10; 3: H1246 transformed with PYE+BnPDAT1-C09; 4: H1246 transformed with PYE; 5: INVSc1 transformed with PYE. |
2.3.2 转BnPDAT1基因酵母合成BnPDAT1蛋白
蛋白印迹(Western blot)结果(图5)显示, 转BnPDAT1基因的酵母中均有BnPDAT1蛋白表达, 转空载体的酵母中未发现BnPDAT1蛋白。
图5
Fig. 5
| Figure OptionViewDownloadNew Window | |
 | 图5 转基因酵母菌株中BnPDAT1蛋白的表达1: H1246转PYE+BnPDAT1-A02; 2: H1246转PYE+BnPDAT1-A10; 3: H1246转PYE+BnPDAT1-C09; 4: H1246转PYE; 5: INVSc1转PYE。Fig. 5 Expression of BnPDAT1 in transgenic yeast1: H1246 transformed with PYE+BnPDAT1-A02; 2: H1246 transformed with PYE+BnPDAT1-A10; 3: H1246 transformed with PYE+BnPDAT1-C09; 4: H1246 transformed with PYE; 5: INVSc1 transformed with PYE. |
2.3.3 BnPDAT1基因恢复酵母突变体(H1246)油脂合成能力 转BnPDAT1基因酵母总油脂TLC分析(图6)发现, BnPDAT1基因恢复酵母H1246的TAG合成能力, 显示BnPDAT1基因在油脂合成中发挥作用。转空载体的H1246菌株相对其他菌株有高得多的DAG含量, 转入BnPDAT1之后DAG含量明显降低, TAG含量明显上升, 显示BnPDAT1能催化DAG合成TAG。
图6
Fig. 6
| Figure OptionViewDownloadNew Window | |
 | 图6 转基因酵母的油脂TLC层析TAG: 三酰甘油; FFA: 自由脂肪酸; DAG: 二脂酰甘油。Fig. 6 TLC analysis of lipid in complementation quadruple mutant strain H1246TAG: triacylglycerol; FFA: free fat acid; DAG: diacylglycerol. |
2.4 甘蓝型油菜中BnPDAT1时空表达特征 为进一步了解BnPDAT1在甘蓝型油菜中的功能, 对甘蓝型油菜不同时期、不同组织中BnPDAT1表达进行了分析, BnPDAT1的分型定量引物为: BnPDAT1-A02-F: 5'-TGAATGGTCTGAGCGTATT-3'; BnPDAT1-A02-R: 5'-CCATGATGATGATCTCTCTTG- 3'; BnPDAT1-A10-F: 5'-GAATGGTCAGAGCGTATT G-3'; BnPDAT1-A10-R: 5'-TGATGATGATGATGATG AGC-3'; BnPDAT1-C09-F: 5'-GAATGGTCAGAGCG TATTG-3'; BnPDAT1-C09-R: 5'-CGTGGAAGCAATA AGTTCA-3'; 内参引物为BnUBC21-F: 5'-CCTCTGC AGCCTCC TCAAGT-3'; BnUBC21-R: 5'-TATCTCCC CTGTCTTGAAATGC-3'。处于种子不同发育时期的湘油15号不同器官组织中BnPDAT1 3个拷贝表达状况如图7所示。
图7
Fig. 7
| Figure OptionViewDownloadNew Window | |
 | 图7 种子不同发育时期种子、叶和花中BnPDAT1的相对表达量Fig. 7 Relative quantity of BnPDAT1 expression in seeds, leaf, and flowerDAP: days after pollination. |
湘油15号种子中BnPDAT1的表达量随种子发育表现先升高后降低的趋势, 在授粉后25~30 d表达量达到最大值; 但BnPDAT1各拷贝的表达变化规律有一定差异, 在授粉25 d后的种子中BnPDAT1-A10和授粉30 d后种子中的BnPDAT1-A02几乎不表达, 同时与之对应的另外2个拷贝高表达, 这暗示BnPDAT1的表达可能具有整体调控的特点, 需要进一步对不同品种(系)的油菜中BnPDAT1表达特征进行分析验证。2个拷贝表达特点在多个不同含油量材料中同样存在(未发表)。湘油15号花、叶片、种子中均有BnPDAT1表达, 但叶片中BnPDAT1-A10表达量极低。BnPDAT1蛋白催化PC与DAG合成TAG, 在油菜发育过程中, 尤其是种子发育与油脂储存的过程中发挥重要功能, BnPDAT1在种子发育过程中的表达先上调后下调, 与种子发育过程中TAG积累模式相吻合, 种子发育中期快速合成和积累TAG, 在种子发育后期TAG存储渐渐饱和, TAG合成量下调。BnPDAT1在花、叶中表达水平低于种子油脂合成期, 与种子发育初期表达水平相当, 也显示了BnPDAT1是一种TAG合成的关键酶, 在花、叶中存在的大量非TAG合成的脂质代谢活动并不能触发BnPDAT1的大量表达。
3 讨论从甘蓝型油菜中克隆获得3个BnPDAT1基因拷贝, 各自编码的BnPDAT1蛋白的生物信息学分析显示其具有PDAT功能结构域, 与甘蓝BrPDAT1蛋白和拟南芥AtPDAT1蛋白均具有较高同源性。通过酵母互补实验证实BnPDAT1基因参与TAG合成过程, 能够恢复中性油脂合成缺陷型酵母的TAG合成与油体形成, 说明所克隆的BnPDAT1基因具有TAG合成功能。
对PDAT及其相似蛋白的聚类分析显示了PDAT蛋白在进化上较高的保守性, 3个BnPDAT1拷贝分别与2个不同的甘蓝BrPDAT1拷贝具有高度同源性, BnPDAT1-A10与BnPDAT1-C09处在进化树同一低阶分支位置, BnPDAT1-A02则处在进化树更高一节点的同一分支位置, 这显示了甘蓝型油菜A、C亚基因组在进化关系上仍然具有较高的同源性, 也间接证实“ 禹氏三角” 中白菜与甘蓝的近缘关系[12]。预测BnPDAT1蛋白被定位于细胞质膜, 这与PDAT1催化PC和DAG反应生产TAG的过程发生于内质网[13]相吻合, 表明油菜中BnPDAT1蛋白合成和修饰过程发生在内质网, 随后锚定到内质网内膜发挥功能。
本文通过酵母互补实验, 验证了BnPDAT1能发挥TAG合成酶功能, 恢复缺陷型酵母的TAG合成能力, 这与酵母内源PDAT蛋白在TAG合成中的作用一致, PDAT途径被认为是酵母TAG合成的辅助途径[14], 而单独转入BnPDAT1即可恢复缺陷酵母TAG合成表型, 显示了BnPDAT1蛋白能独立合成TAG, 也暗示了BnPDAT1可能在逆境状态下合成TAG过程中发挥重要功能。
对植物PDAT蛋白研究显示了其高度的底物偏好性, PDAT对环烷化、羟化以及含多双键底物具有高效催化作用[15]。同样BnPDAT1蛋白的合成与表达可能存在明显的组织特异性, 催化TAG合成中的作用可能因底物类型、丰度和合成部位差异而明显不同, BnPDAT1对TAG甘油骨架中sn-2、sn-3位置的脂肪酸链结构和类型的影响尚待进一步研究, 这对于油菜品质育种将是一个新的方向。
TAG合成是影响甘蓝型油菜产量和菜籽油质量的最关键因素, 研究甘蓝型油菜TAG合成、积累规律, 对于指导油菜育种与生产具有重要意义。而BnPDAT1被证实可参与TAG合成, 可能在油菜TAG合成网络中占有重要地位, 对BnPDAT1基因的深入研究具有重大的科研和社会价值。本文克隆获得的BnPDAT1有3个拷贝, 分别定位在A02、A10、C09染色体上, 进化上分别来源于白菜和甘蓝, 序列相似性超过95%, 这显示PDAT基因起源较早且保守, 在原始的芸苔属植物中即已存在且具有重要功能。但具有不同含油量水平的油菜中BnPDAT1基因的拷贝状态尚需研究, 以进一步证实PDAT途径在油菜TAG合成中的作用。同样湘油15号中克隆所得BnPDAT1的3个拷贝在生长发育过程中的调控模式和各自对TAG合成量的贡献尚需进一步研究。
4 结论从甘蓝型油菜湘油15号cDNA中克隆到3个PDAT1全长编码序列(coding sequence, CDS), 被分别定位于A02、A10、C09染色体, 并命名为BnPDAT1-A02、BnPDAT1-A10和BnPDAT1-C09, 其序列长分别为1998、2002、2005 bp, 各自编码665、666和667个氨基酸残基。该基因编码蛋白与其他植物PDAT1具有较高相似度, 具有典型的PDAT1结构域, 具有PDAT1酶活性。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
参考文献View Option
原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | |
| [3] | |
| [4] | |
| [5] | |
| [6] | |
| [7] | |
| [8] | |
| [9] | |
| [10] | |
| [11] | |
| [12] | |
| [13] | |
| [14] | |
| [15] |
