 , 王新超
, 王新超
* 通讯作者(Corresponding authors): 杨亚军, E-mail: yjyang@mail.tricaas.com, Tel: 0571-86650266; 王新超, E-mail: xcw75@mail.tricaas.com, Tel: 0571-86653162 第一作者联系方式: E-mail: wb32244@tricaas.com, Tel: 18757154898
收稿日期:2015-06-08 接受日期:2015-09-06网络出版日期:2015-10-08基金:
摘要
关键词:茶树; 休眠; 生长素外运载体基因; 表达分析
Cloning and Expression Analysis of Auxin Efflux Carrier Gene CsPIN3 in Tea Plant ( Camellia sinensis)
WANG Bo
 , WANG Xin-Chao
, WANG Xin-Chao
Fund:
Abstract
Keyword:Tea plant ( Camellia sinensis); Dormancy; Auxin efflux carrier; Expression analysis
Show Figures
Show Figures

















茶树[Camellia sinensis(L.) O. Kuntze]是多年生木本植物, 每年秋、冬季, 短日照和低温促使芽进入休眠状态。安全越冬后, 随着气温回升及光照时间的增加, 打破休眠使芽萌发。芽休眠作为茶树长期自然演化获得的一种生物学适应性, 有助于茶树的安全越冬[1]。茶树芽休眠受诸多因素的影响, 其中内源激素生长素是一个重要因素[2], 生长素运输载体蛋白作为生长素转运的载体, 可在植物组织或器官中建立生长素的不对称分布, 生长素通过其运输载体介导的极性运输在植物组织或器官中建立浓度梯度, 进而在植物生长发育过程中表现出广泛的生理效应[3]。因此研究茶树生长素运输载体基因家族与茶树芽休眠和解除的关系, 使之用于茶树种质资源早生品种的筛选鉴定, 以辅助茶树育种工作, 有着重要的理论和实践意义。
PIN蛋白家族是植物生长素运输载体中最为重要的基因家族, 负责向细胞外运输生长素[4], 目前, 已知模式植物拟南芥中PIN蛋白家族有8个成员, 分别被命名为PIN1~PIN8。PIN蛋白家族成员均为膜蛋白, 均由位于两端的疏水区和中间的亲水区构成, 两端的疏水区皆由5个跨膜螺旋构成, 亲水环在细胞内, 在亲水区域存在着糖基化位点和磷酸化位点[5]。PIN1是生长素动态运输的重要组件[6]; PIN2位于根表皮组织基部皮层细胞的顶端, 具有分配与根向地生长有关的生长素功能[7]; PIN4和PIN7主要是非极性定位, PIN4参与根尖分生组织静止中心生长素库的建立[8]; PIN7定位在基部细胞质膜的上表面, 调节胚轴的形成和根中生长素的向顶式运输[9]; 定位于内质网的PIN5、PIN6、PIN8主要参与调节细胞内生长素的动态平衡[8]; Keuskamp等[10]研究表明, PIN3主要定位在茎芽内皮层细胞、重力响应根柱以及中柱鞘细胞质膜上, 能将重力刺激信号转化为生长素的非对称分布, 从而控制植物的向性生长。另外, He等[11]研究表明, 在葡萄越冬芽萌发时, 如果下调PIN3表达会降低生长素从芽内向外运输的能力, 延缓休眠芽从生理休眠向生态休眠的转变, 推迟萌发; Leduc等[12]研究推测, 植物通过调控PIN1的定位, 将光信号转化为生长素信号影响芽的萌发。
PIN蛋白家族在植物生长素运输过程中具有重要功能。目前, 已经从多种植物, 如马铃薯[13]、芒果[14]、狗蔷薇[15]、拟南芥、大米、番茄[16]等中克隆了PIN基因。但茶树中有关PIN蛋白家族的研究还未见报道。本研究利用RACE结合RT-PCR的方法, 以本实验室通过转录组测序获得的茶树中与PIN高度同源的 EST片段为模板, 克隆茶树CsPIN3全长cDNA序列, 分析CsPIN3在茶树不同组织间的表达差异及芽休眠不同阶段的表达模式及其在茶树越冬芽休眠解除中的功能, 为研究生长素在茶树越冬芽休眠与解除中的作用机制提供理论参考。
1 材料与方法1.1 试验材料2013年10月至2015年3月, 在中国农业科学院茶叶研究所内实验基地种植特早生国家级茶树品种龙井43。于茶树盛花期, 取茶树的根、茎、叶、花等组织。从茶树进入休眠期开始, 根据芽的发育情况, 分别于2013— 2014年11月2日、12月1日、1月2日、2月14日、3月3日、3月14日、3月27日, 2014— 2015年11月4日、12月2日、1月5日、2月5日、3月3日、3月18日、3月23日采摘茶树顶芽, 以液氮速冻所有样品并迅速置-80℃冰箱保存待用。
SMART RACE cDNA Synthesis Kit和Advantage 2 PCR Kit、Universal Primer Mix购于Clontech公司(Mountain View, CA, USA); SYBR Premix Ex Taq (Perfect Real Time)试剂盒、Prime Script Reagent试剂盒、克隆载体PCR2.1 vector、IPTG、X-gal、大肠杆菌DH5α 感受态细胞和rTaq酶均为TaKaRa公司产品(大连); 逆转录酶SuperScript III购自Invitrogen公司(Carlsbad, USA); DNA Marker、琼脂糖凝胶回收试剂盒为购自Axygen公司(California, USA)的AxyPrep DNA Gel Extraction Kit。其余试剂为国产分析纯。由杭州擎科生物技术有限公司合成PCR引物, 由上海华津生物技术有限公司测序。
1.2 总RNA的提取、cDNA的合成取0.2~0.5 g不同休眠阶段的茶树样品, 参考Chang等[17]方法提取茶树总RNA。使用NanoDrop ND100微量核酸蛋白测定仪(Pittsburgh, PA, USA)测定吸光值, 确定RNA浓度, 用1.0%变性琼脂糖凝胶电泳检测总RNA质量, 最后调整RNA浓度至1 μ g μ L-1, 于-80℃保存备用。
以1 μ g提取的龙井43顶芽总RNA为模板, 参照SMART RACE cDNA Synthesis Kit操作步骤, 合成5′ 和3′ 的cDNA链, 用于RACE-PCR; 以1 μ g顶芽总RNA为模板, 根据SuperScript III试剂盒的操作说明合成第1链cDNA用于反转录PCR (RT-PCR)来扩增ORF; 用1 μ g不同休眠阶段顶芽提取的总RNA按照Prime Script Reagent试剂盒的操作说明合成cDNA, 稀释10倍后作为模板进行荧光定量PCR (qRT-PCR)检测。
1.3 茶树CsPIN3克隆 本实验室前期对茶树冷驯化样品做了转录组测序(NCBI登录号为: SRA061042)[18], 在此基础上筛选出与生长素外运载体PIN高度同源的EST序列, 以DNAStar软件拼接, 获得一条序列。根据序列设计5′ 和3′ 特异引物(表1), 按照SMART-RACE试剂盒的要求进行5′ -和3′ -RACE-PCR反应, 得到该基因的5′ 和3′ 端扩增序列。RACE-PCR条件为94℃、30 s, 68℃、30 s, 72℃、3 min, 30个循环。RACE-PCR产物经AxyPrep DNA凝胶回收试剂盒纯化回收后, 连接到PCR2.1 vector上, 转化至大肠杆菌DH5α , 蓝白斑筛选挑取白色单菌落鉴定, 对每个连接产物至少挑选3个阳性菌落的菌液送上海华津生物科技有限公司测序。
测序得到的序列去载体、比对拼接, 得到基因的5′ 端和3′ 端序列, 再与原EST序列拼接, 初步得到PIN的cDNA全长序列。在开放阅读框(ORF)上、下游区域设计RT-PCR特异引物(表1), 以PCR扩增得到ORF序列。反应条件为94℃ 4 min, 94℃ 30 s, 55℃ 30 s, 72℃ 2 min, 30个循环; 72℃ 10 min。PCR产物经回收、连接、转化、测序后, 得到茶树完整的PIN全长cDNA序列[19]。
1.4 CsPIN3序列的生物信息学分析试验中所用核酸及氨基酸序列均从NCBI网站下载, 利用NCBI的BlastX工具进行同源性比对分析; 利用DNAStar软件进行序列拼接和开放阅读框查询; 利用ClustalW软件进行多序列对齐和排序, 使用GenDOC和MEGA5.0软件输出同源比对和进化树构建结果[20]; 用SingaIP进行信号肽预测[21]; 用ProtParam计算蛋白质的相对分子质量和理论等电点[22]; 利用ProtScale进行蛋白质疏水性分析; 利用SOPMA进行蛋白质的二级结构分析[23]; DictyOGlyc 1.1 Server预测O-糖基化位点[24]; NetPhos 2.0 Serve软件预测磷酸化位点, PSORT进行蛋白质的亚细胞定位[25]; 利用TMHMM预测蛋白质的跨膜结构域并用TMRPres2D软件作图[26]。
表1
Table 1
表1(Table 1)
| 表1 试验引物及其序列 Table 1 Sequence of primers used in this study |
1.5 CsPIN3基因的表达模式分析以茶树CsPTB基因作为内参基因[27], 根据目的基因序列设计荧光定量PCR引物(表1)。以龙井43不同休眠阶段的顶芽总RNA为材料, 按照Prime Script Reagent试剂盒的操作说明合成cDNA, 稀释10倍作为模板。荧光定量反应体系为SYBR Premix Ex Taq 10 μ L, 上、下游引物(10 μ mol L-1)各0.8 μ L, ROX Dye II 0.4 μ L, cDNA 2 μ L, 加水至终体积20 μ L。反应在ABI PRISM 7500实时定量PCR仪上进行, 95℃变性30 s, 95℃变性5 s, 60℃退火延伸34 s, 40个循环[27]。反应结束后分析荧光值变化曲线和熔解曲线。每个反应重复3次, 采用2-Δ Δ Ct算法[28]分析结果。
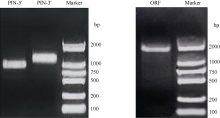
2 结果与分析2.1 CsPIN3cDNA全长序列的的克隆及分析从实验室前期茶树冷驯化转录组测序结果中筛选拼接得到1条PIN的EST序列, 在NCBI中进行BlastX同源性比对, 结果显示该序列与其他植物中的PIN序列高度相似, 但无完整的开放阅读框。根据此中间序列分别设计RACE特异引物, 分别扩增得到约1.2 kb的3′ 端序列和约1 kb的5′ 端序列(图1), 测序后经BlastX显示两端序列均编码PIN, 然后将两序列片段与原EST序列拼接, 能够查找到编码PIN氨基酸序列的完整开放阅读框(ORF), 在ORF两端设计引物, 进行RT-PCR检测并测序验证, 最后获得了长为2654 bp的cDNA序列, 其5′ 端有114 bp的非编码区(5′ -UTR), 3′ 端有624 bp的非编码区(3′ -UTR), 终止子TGA后具有非常保守的多聚腺苷酸加尾结构。依据上述特征, 判断本研究获得了茶树PIN的完整cDNA全长序列。该序列全长2654 bp, 包含1926 bp的ORF, 编码641个氨基酸。对氨基酸序列分析表明, 其含有PIN3蛋白的2个重要保守域, 即NPNXY内在保守结构域和丝氨酸/苏氨酸蛋白激酶(PID/PINOID)磷酸化活性位点— — TPRXS(N/S)结构域, 由此推测克隆到的基因属于PIN3, 命名为CsPIN3, GenBank登录号为KP896474 (图2)。
图1
Fig. 1
| Figure OptionViewDownloadNew Window | |
 | 图1 CsPIN3 5′ 和3′ -RACE(左)及RT-PCR(右)电泳结果Fig. 1 Electrophoresis results of 5′ and 3′ fragments (left) and ORF fragment (right) of CsPIN3 |
图2
Fig. 2
| Figure OptionViewDownloadNew Window | |
 | 图2 茶树生长素外运载体基因CsPIN3全长cDNA的核苷酸序列和推测氨基酸序列Fig. 2 Full length cDNA and putative amino acid sequences of CsPIN3 in tea tree |
2.2 茶树CsPIN3基因系统发育分析 将茶树CsPIN3序列的全长cDNA在NCBI中进行BlastX同源比对, 结果显示它编码的氨基酸序列与其他物种中报道的生长素外运载体PIN蛋白的相似性均在80%左右, 其中与杨树(Populus trichocarpa, XP002308100.1)的相似性最高, 达85%。与葡萄(Vitis vinifera, XP002284302.1)、柑橘(Citrus sinensis, XP006469168.1)、烟草(Nicotiana tomentosiformis, XP 009601548.1)、番茄(Solanum lycopersicum, NP00123 4184.1)、马铃薯(Solanum tuberosum, XP006344239.1)和芝麻(Sesamum indicum, XP011070630.1)等的相似性分别为82%、82%、82%、81%、81%和80%。
图3
Fig. 3
| Figure OptionViewDownloadNew Window | |
 | 图3 茶树CsPIN3与其他植物PIN氨基酸序列比对Fig. 3 Mutiple aligement of deduced amino acid sequence of CsPIN3 with other plant PIN protein |
在NCBI上筛选出与茶树CsPIN3基因氨基酸序列相似性较高的8个不同物种的生长素外运载体PIN蛋白, 并将其与茶树CsPIN3基因预测的氨基酸序列进行多序列比对。结果显示, 茶树CsPIN3与其他PIN氨基酸序列有较高的保守性, 特别是可变结构域、磷酸化位点及糖基化位点等重要功能位点的序列保守型更高。此外, 所有PIN蛋白都存在调控其内吞作用的NPNXY保守内在构型(inner motif, IM)(图3)。在MEGA5.0软件中用邻接相对法绘制CsPIN3与拟南芥及其他物种的18个PIN氨基酸序列之间的进化树(图4), 结果显示茶树的CsPIN3蛋白与茄科植物的亲缘关系最近。此外, 在拟南芥PINs蛋白中, AtPIN3与茶树CsPIN3的亲缘关系较近, 处于不同于其他AtPINs的一个大的进化分支上。
图4
Fig. 4
| Figure OptionViewDownloadNew Window | |
 | 图4 CsPIN3与其他植物PIN氨基酸序列的系统进化分析Fig. 4 Phylogenetic analysis of CsPIN3 and PIN in other species |
2.3 CsPIN3蛋白质特征分析与3D结构预测2.3.1 CsPIN3理化性质
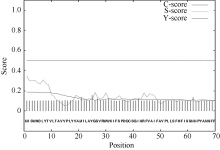
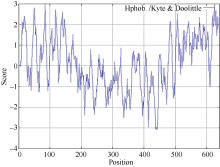
经Protparam程序预测, CsPIN3的理论分子量为70.15 kD, 理论等电点(pI)为8.42。含量最丰富的氨基酸有10.3% Ser、9.0% Leu和7.6% Gly。CsPIN3中的酸性氨基酸(Asp + Glu)有44个, 碱性氨基酸(Arg + Lys)有47个; 利用SignaIP- 4.1分析CsPIN3蛋白信号肽和预测信号肽位点表明, 其平均分小于0.500, 利用最大Y值预测表明CsPIN3蛋白不存在信号肽及切割位点, 属于非分泌蛋白(图5); 作为蛋白质折叠的主要驱动力, 蛋白质的亲水、疏水氨基酸的组成能够影响蛋白质的结构和特性, 经在线软件ProtScale采用Hyhob/Kyte & Doolittle的方法分析显示(图6), 最大正值为2.989, 最小负值为-3.100。CsPIN3蛋白是由N端和C端的疏水区和中间第181氨基酸(N端)至第477氨基酸(C端)的亲水区构成; 蛋白质的功能与亚细胞的定位存在着非常密切的联系, 以往的研究表明, 生长素外运载体PIN家族均为膜蛋白, 利用PSORT分析显示CsPIN3蛋白主要位于质膜上, 内质网上也有少量的分布, 属于典型的膜蛋白。
图5
Fig. 5
| Figure OptionViewDownloadNew Window | |
 | 图5 CsPIN3蛋白的信号肽分析Fig. 5 Signal peptide analysis of CsPIN3 |
图6
Fig. 6
| Figure OptionViewDownloadNew Window | |
 | 图6 CsPIN3亲水性/疏水性预测Fig. 6 Hydrophobicity/hydrophilicity prediction of CsPIN3 |
2.3.2 CsPIN3蛋白的跨膜结构
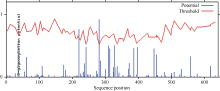
利用在线软件TMpred预测CsPIN3蛋白跨膜结构(图7), 显示该蛋白在N端和C端疏水区各有一个跨膜结构域, 属于跨膜蛋白。其中N端有5个跨膜螺旋, 分别位于第10~29、第42~61、第71~93、第100~122、第132~154位氨基酸之间; C端有4个跨膜螺旋, 分别位于第526~548、第558~580、第585~607、第617~639位氨基酸之间。这表明CsPIN3蛋白是以膜结合蛋白的形式存在的, 这与其作为转运载体的功能是相适应的。
图7
Fig. 7
| Figure OptionViewDownloadNew Window | |
 | 图7 CsPIN3蛋白跨膜区预测Fig. 7 Prediction model of transmenbrane domain of CsPIN3 protein |
2.3.3 CsPIN3蛋白磷酸化和糖基化位点
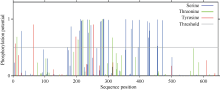
蛋白质的磷酸化和糖基化是对蛋白质的重要修饰, 可以调节蛋白质的活力和功能。分别运用NetPhos 2.0 Serve、DictyOGlyc 1.1 Server在线软件预测CsPIN3基因翻译后磷酸化、O-糖基化修饰情况, 结果显示CsPIN3蛋白有可能发生磷酸化修饰的位点有35个, 其中色氨酸(Ser)、苏氨酸(Thr)、酪氨酸(Tyr)可能发生磷酸化修饰的位点分别有25、6、4个(图8); 有可能发生O-糖基化修饰的位点有2个, 均为苏氨酸(Thr), 分别在肽链的第220、第281位(图9)。糖基化、磷酸化位点均集中分布在中间亲水区。
图8
Fig. 8
| Figure OptionViewDownloadNew Window | |
 | 图8 CsPIN3蛋白氨基酸序列翻译后磷酸化修饰位点预测Fig. 8 Prediction of phosphorylation site modification in amino acid sequence of CsPIN3 protein |
图9
Fig. 9
| Figure OptionViewDownloadNew Window | |
 | 图9 CsPIN3蛋白氨基酸序列翻译后O-糖基化修饰位点预测Fig. 9 Prediction of O-glycosylation site modification in amino acid sequence of CsPIN3 protein |
2.3.4 CsPIN3蛋白结构
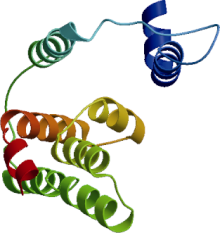
运用SOPAM预测蛋白二级结构显示, CsPIN3蛋白含有23.34%的α -螺旋、15.23%的延伸链和48.52%的随机卷曲。经SMART在线查询后在NCBI上进行保守域分析表明, 茶树CsPIN3蛋白第331~356氨基酸、第431~448氨基酸位之间存在2个可变结构域, 将亲水区分割成3个恒定区。CsPIN3蛋白在第9~636氨基酸位之间是个高度保守的结构功能域— — 生长素外运载体蛋白家族PIN的保守结构域, E值为4.5e-184。此外, 在第495~499氨基酸位置存在CsPIN3蛋白IM保守结构NPNXY, 在第241、第281氨基酸位点还存在着PIN蛋白特有的丝氨酸/苏氨酸蛋白激酶(PID/PINOID)磷酸化活性位点— — TPRXS(N/S)结构域。利用SWISS- MODEL Workspace软件在线预测CsPIN3蛋白的三维结构模型显示(图10), CsPIN3蛋白具有类似运输载体的结构, 充分说明该基因确为茶树生长素外运载体蛋白家族的一员。
图10
Fig. 10
| Figure OptionViewDownloadNew Window | |
 | 图10 CsPIN3蛋白三维结构模型Fig. 10 Tertiary structure homology-modeling of CsPIN3 |
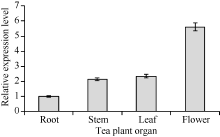
2.4 组织表达特异性分析图11显示, CsPIN3在茶树的根、茎、叶片和花中均有表达, 但表达具有明显的组织差异性; CsPIN3在茶树花中的表达量最高, 在根中的表达量最低, 在茎、叶中的表达量略高于根部, 在花中的表达量是根中的5倍左右。
图11
Fig. 11
| Figure OptionViewDownloadNew Window | |
 | 图11 CsPIN3基因在茶树不同组织中的表达Fig. 11 Expression of CsPIN3 gene in various tea plant organs |
2.5 茶树顶芽发育不同时期CsPIN3基因的表达 利用荧光定量PCR技术分析CsPIN3基因从秋季茶树顶芽开始休眠到春季萌发期间不同时间点的相对表达发现, 该基因在茶树顶芽休眠萌发的不同时期均有表达, 且在萌发阶段的表达量高于休眠阶段(休眠初期到膨大期之间)。与休眠初期相比, 随着休眠程度的加深, 该基因的表达量维持在一个较低的水平, 随着芽发育程度即茶树顶芽由生理休眠向生态休眠继而向一芽一叶转变的过程中, CsPIN3基因的表达量持续上调, 到芽叶期表达量为休眠阶段的3倍以上, 且在茶芽萌动过程中表达上调的速度较明显(图12)。说明该基因与茶树越冬芽的休眠维持与解除关系密切。
图12
Fig. 12
| Figure OptionViewDownloadNew Window | |
 | 图12 顶芽不同阶段CsPIN3基因的相对表达量Fig. 12 Relative expression of CsPIN3 gene at different stages of terminal buds |
3 讨论生长素外运载体蛋白PIN是生长素运输载体中最为重要也是最先发现的一类, 为陆生植物所特有[29], 目前已经从多种植物上分离出PIN基因[13, 14, 15, 16]。其中拟南芥PIN蛋白家族是研究的最好的一类, 根据其亲水环的长度分为两类, 亲水区较长的PIN1、PIN2、PIN3、PIN4、PIN7定位于质膜上, 负责向细胞外运输生长素, 亲水区较短的PIN5、PIN6、PIN8定位于内质网上, 负责胞浆和内质网上的生长素调配[5, 29, 30]。PIN3定位在茎芽内皮层细胞、重力响应根柱以及中柱鞘细胞质膜上, 呈对称分布, 主要通过调节生长素的再分配参与植物的向性生长如向光性、向地性等。本研究从茶树中克隆的CsPIN3基因具有PIN基因的家族典型的序列特征。CsPIN3基因cDNA序列含有1926 bp的完整开放阅读框(ORF), 编码氨基酸数、蛋白质分子量、理论等电点等均与其他植物的PIN相似。CsPIN3蛋白与茄科植物的亲缘关系最近, 与拟南芥AtPIN3从属一个分支。CsPIN3基因编码蛋白不存在信号肽, 属于非分泌的水溶性蛋白。CsPIN3蛋白在第9~636氨基酸位之间是个高度保守的结构功能域— — 生长素外运载体蛋白家族PIN的保守结构域, 该蛋白能够与生长素特定结合, 并能饱和分布于生长素运输细胞的基部[31]。同其他植物的PIN蛋白一样, CsPIN3是由两端的疏水区和中间的亲水区构成, 亲水区较长。CsPIN3 N端疏水区和C端疏水区分别存在5个和4个跨膜螺旋, C端疏水区跨膜螺旋数比拟南芥少1个, 但与水稻结构一致[32], C端疏水区较N端疏水区缺失部分序列, 反映N端和C端的跨膜结构对PIN功能的重要性不同[33]; CsPIN3定位于质膜上, 与AtPIN3不同的是它在内质网上也有少量的分布, 这与以往的一些研究一致[34]。多序列比对显示, CsPIN3编码的氨基酸序列与其他植物的同源性较高, 具有PIN典型的高度保守的NPNXY内在构型, 即IM结构域, 它调控PIN蛋白的内吞作用, 介导小泡分拣过程中膜蛋白与受体细胞的互作[35]。磷酸化和去磷酸化是蛋白质一种重要的转录后修饰, 通过对氨基酸序列的磷酸化位点分析发现, CsPIN3具有PIN蛋白特有的丝氨酸/苏氨酸蛋白激酶(PID/PINOID)磷酸化活性位点TPRXS(N/S)结构域。PID对生长素与质膜、内质网之间的运输有重要作用[36], 通过此位点, PID可以直接磷酸化PIN蛋白的中央亲水区调节PIN蛋白的极性定位进而调控生长素的定向运输[37]。生长素也可以调节PID蛋白的活性, 当PID蛋白磷酸化PIN蛋白引起局部生长素浓度升高时, 胞浆内钙离子增加, 钙离子信号调节的钙结合蛋白(TOUCH3)及PBPI (PINOID Bind Protein1)通过改变PID的活性使得PIN蛋白定位反转, 改变生长素的运输方向[38], 植物的向性生长及生长发育中受拮抗剂、抑制剂的影响机制均是如此[39]。此外CsPIN3还存在着PP2A (Serine/Threo-nine Phosphatase 2A)磷酸酶的活性位点, 它能够调节PIN蛋白的向顶、向基运输, 对PID蛋白诱导的PIN的极性定位有拮抗的作用[36]。依据这些分析结果可以推测本研究克隆的CsPIN3为茶树生长素外运载体基因, 这为进一步研究生长素在茶树芽休眠解除中的作用机制打下了坚实的基础。
通过对CsPIN3在茶树组织中的表达结果分析, 发现在茶树根、茎、叶和花4种组织器官中均能检测到CsPIN3基因的表达, 但表达具有组织特异性。CsPIN3在茶树根中的表达量相对较低, 花中的表达量最高, 在茎、叶中的表达量居中。整体上, CsPIN3基因在茶树中按形态学上端至形态学下端的顺序表达量依次降低, 这与生长素的极性运输大体上是一致的。研究表明, 花的开放受内源激素生长素的调控[40]。尽管内源激素在开花过程中的分配及作用机制还未证实, 但已有的研究表明, 在开花的初始阶段, 生长素内向转运载体Aux/IAA的表达量升高, 并且参与生长素在植物花瓣中细胞间的运输, 促进花的萌发[41]。在盛花期, 花是茶树生长发育比较活跃的组织, 大量的生长素通过生长素外运载体基因运出, 通过位于茎内皮层细胞的载体基因再运输到茶树其他组织中。由此可见, 在茶树生长发育过程中, 生长素外运载体基因参与的生长素极性运输发挥了重要的作用。
在植物的生长发育过程中, 生长素参与其大多数的生命活动, 与植物芽休眠也密切相关, 生长素通过其运输载体介导的极性运输在植物组织或器官中建立生长素的不对称分布, 进而在植物生长发育过程中表现出广泛的生理效应。研究表明, 生长素转运载体参与拟南芥芽萌发早期的生长发育[42]。因此作为生长素外运载体PIN家族的一员, CsPIN3可能在茶树越冬芽的休眠与解除中发挥重要的作用。在2013— 2014和2014— 2015年, 对龙井43茶树芽休眠的CsPIN3不同阶段的表达分析表明, CsPIN3在顶芽萌发阶段的表达量高于休眠阶段(休眠初期到膨大期之间), 休眠初期CsPIN3基因的表达量始终维持在一个较低的水平, 随着芽的萌发CsPIN3基因的表达量持续上调, 到芽叶期表达量为休眠阶段的3倍以上, 且在茶芽萌动过程中表达上调的速度较明显, 与牡丹[43]、葡萄[44]上的研究结果类似。这可能是因为在生理休眠阶段, 芽中各组织活动相对停滞, 生长素内向转运载体的表达量较低, 萌发后芽中各组织细胞恢复生长, 表达量快速升高。Nagar等[45]研究发现, 在茶树休眠芽中游离态的生长素含量较低, 在休眠解除后, 游离态的生长素含量升高, 结合态的生长素则相反。这种结果与本研究所发现的CsPIN3基因表达结果相匹配。Medvedova等[46]、Balla等[47]研究发现, 芽中生长素的外运可以促进芽的萌动。生长素的持续外运诱导PIN基因的表达, 促进PIN蛋白在芽中重新进行极性定位, 新的维管组织将沿着PIN标记的生长素运输路径开始分化, 促进芽的萌发, 因此芽的萌发伴随着PIN的表达, 这与本研究结果一致。
游离态生长素的含量、CsPIN3基因的表达、茶树休眠芽的萌发三者之间存在着密切的联系。芽休眠时, 游离态生长素含量较低, CsPIN3表达量也较低, 萌发后游离态生长素含量增加, CsPIN3表达量也上调。这可能是由于芽休眠时, 游离生长素的含量较低, 无法诱导CsPIN3基因的表达, 但随着生长素内向运输载体在深休眠时表达量的增高, 芽中游离生长素通过不断的累积含量逐渐增加, 通过诱导CsPIN3基因的表达以及诱发钙离子信号调节PIN蛋白的活性提高生长素的外运, 促进萌发。因此, 生长素外运载体CsPIN3可能与茶树越冬芽休眠的维持和解除有关系。
本研究克隆了茶树CsPIN3基因并研究了其在茶树不同组织间的表达差异及芽休眠不同阶段的表达模式, 探讨了生长素外运载体基因CsPIN3与茶树越冬芽休眠解除的关系, 为深入揭示生长素调控越冬芽休眠的分子机制提供了理论依据。但生长素运输载体基因数量众多, 且参与生长素调控越冬芽休眠解除的基因有很多, 如生长素响应因子ARF、生长素受体基因TIR等。因此, 下一步应将PIN与其他基因结合起来, 从基因的功能及信号转导通路方面深入揭示生长素调控茶树越冬芽休眠的作用机制。
4 结论获得茶树生长素外运载体基因CsPIN3的全长cDNA。CsPIN3编码蛋白具有PIN家族的一般特性。CsPIN3编码蛋白是定位于质膜上的非分泌性蛋白, 由两端的疏水区和中间的亲水区构成, 疏水区内有多个跨膜螺旋, 在亲水区存在2个可变结构域, 还存在着糖基化位点和磷酸化位点TPRXS(N/S)结构以及IM保守结构NPNXY, 与茄科植物的PIN3亲缘关系最近。CsPIN3具有组织表达特异性, 在茶树越冬芽萌发阶段的表达量高于休眠阶段(休眠初期到膨大期之间), 且在茶芽萌动过程中表达上调的速度较明显。该基因可能与茶树越冬芽的休眠维持与解除有关。
The authors have declared that no competing interests exist.
参考文献View Option
原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | |
| [3] | |
| [4] | |
| [5] | |
| [6] | |
| [7] | |
| [8] | |
| [9] | |
| [10] | |
| [11] | |
| [12] | |
| [13] | |
| [14] | |
| [15] | |
| [16] | |
| [17] | |
| [18] | |
| [19] | |
| [20] | |
| [21] | |
| [22] | |
| [23] | |
| [24] | |
| [25] | |
| [26] | |
| [27] | |
| [28] | |
| [29] | |
| [30] | |
| [31] | |
| [32] | |
| [33] | |
| [34] | |
| [35] | |
| [36] | |
| [37] | |
| [38] | |
| [39] | |
| [40] | |
| [41] | |
| [42] | |
| [43] | |
| [44] | |
| [45] | |
| [46] | |
| [47] |
