 , 张磊
, 张磊 , 佘茂云
, 佘茂云
*通讯作者(Corresponding authors): 张磊, E-mail: 13955165987@163.com, 佘茂云, E-mail: ahxiaoshe@126.com 第一作者联系方式: E-mail: guixiangyin@126.com
收稿日期:2014-09-30 基金:
摘要
关键词:大豆; TRK-HKT家族; 分子鉴定; 逆境胁迫
Structural Characterization and Abiotic Stress Response of Soybean TRK- HKT Family Genes
YIN Gui-Xiang
 , ZHANG Lei
, ZHANG Lei , SHE Mao-Yun
, SHE Mao-Yun
Abstract
Keyword:Soybean; TRK-HKT family; Molecular characterization; Abiotic stress
Show Figures
Show Figures


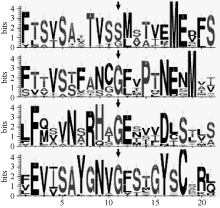


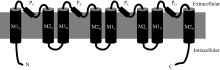




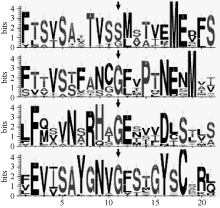






钾元素是高等植物中最丰富的无机矿质元素之一, 参与植物体内诸如光合作用、植株向性生长、体内矿质元素代谢及渗透调控等生命活动过程, 影响作物产量及品质[1]。植物体主要采用高、低两种类型转运蛋白实现对K+运输, 前者包括TRK-HKT家族蛋白、KT/HAK/KUP家族蛋白、KEA转运蛋白及CHX转运蛋白(cation/H+ exchanger, CHX), 后者如shaker类通道蛋白、双孔通道蛋白、环核苷酸门通道蛋白(cyclic nucleotide-gated channels, CNGC)等[2, 3], 其中以植物TRK-HKT蛋白家族研究最为深入[4, 5, 6, 7, 8, 9], 但在大豆(Glycine max L.)中仅发现有限报道[10, 11]。
利用异源表达系统及K+运输缺陷型突变体, 先后在植物中鉴定出许多与K+运输有关的TRK-HKT家族蛋白[12]。小麦(Triticum aestivum L.) TaHKT2; 1是第一个利用此方法鉴定出的植物源HKT蛋白, 表现出K+的高亲和性, 命名为“ 高亲和力K+转运体” (high-affinity potassium transporter, HKT) [13, 14]。然而, 植物TRK-HKT家族蛋白离子运输功能具有双重性, 即低Na+时充当Na+和K+协同转运体, 高Na+时仅运输Na+ [14, 15]。早期研究认为HKT蛋白运输K+的能量不是来源于ATP水解[13], 但随后研究得出相反的结论, 且发现部分蛋白运输能量还依赖Na+偶联产生的电化学梯度, 如TaHKT1[14]。功能研究发现TRK-HKT家族成员在Na+-K+协同运输方式上存在差异, 但目前有关这种转运方式差异的机制并不明确[16]。根据拟南芥(Arabidopsis thaliana L.) AtHKT1和水稻(Oryza sativa L.) OsHKT1蛋白结构对比发现, 在第1个P环(PA)中存在甘氨酸(G)与丝氨酸(S)突变, 认为这可能是导致二者转移K+效率差异的原因[17, 18, 19]。
植物TRK-HKT家族基因广泛参与植物抗逆境胁迫[20, 21, 22]。研究发现, 植物耐盐性与其芽中Na+累积量负相关, 特别在谷类作物如水稻、小麦中尤为明显[23, 24]。拟南芥及水稻的TRK-HKT家族成员对维持细胞内Na+及Na+/K+动态平衡起主要作用, 调控其耐盐性能[25, 26, 27, 28]。OsHKT2; 1功能缺失突变导致水稻在低K+条件下生长缓慢[28]。Chen等[10]研究发现, 外源NaCl处理能诱导大豆根和叶中GmHKT1表达, 且过表达GmHKT1的转基因烟草根和芽中Na+累积减少而K+含量上升, 耐盐性提高, 表明GmHKT1具有促进Na+移出及协同Na+-K+运输的双重作用。此外, 植物体内高浓度的Na+能抑制其对土壤中K+的吸收, 导致植物出现低钾症状, 严重时整个叶片坏死脱落, 植株易倒伏[29, 30]。因此, 提高植物耐盐胁迫能力, 必须降低Na+提升K+含量, 提高K+/Na+比, 而高亲和力的K+转运蛋白(如TRK-HKT家族)在调控植物体内Na+/K+比方面具有明显优势[31]。
大豆属甜土作物, 对高盐等逆境胁迫敏感, 选育耐盐大豆品种是大豆育种的一个重要方向[10]。目前对大豆氮元素代谢研究较多, 而对钾元素研究相对较少, 且发现大豆营养期缺钾容易出现根系老化早衰直至死亡, 严重影响大豆产量[32]。尽管对植物中参与K+运输的TRK-HKT蛋白研究较多, 但对大豆TRK-HKT家族蛋白研究较少, 仅有的几篇报道都集中在对该家族个别成员的功能研究上[10, 11], 缺乏对大豆TRK-HKT家族成员系统的生物学特性分析研究。本研究以大豆TRK-HKT家族为研究对象, 以6个钾利用效率差异大豆品种为试验材料, 以期通过对大豆TRK-HKT家族基因结构及逆境胁迫表达机制分析, 获得对大豆K+主动运输具有重要影响的HKT基因, 为利用转基因技术手段创制耐盐大豆品种提供基因资源。
1 材料与方法1.1 供试材料根据权月伟等[33]、王伟等[34]及本实验室前期研究结果(未发表), 选择吉林省农业科学院大豆研究室馈赠的钾高效大豆品种冀豆8号、中豆32和九农15和河北农业大学张彩英教授课题组馈赠的钾低效大豆品种中黄4号、中黄13、GD8521, 用于TRK-HKT家族基因表达定量分析。
1.2 逆境胁迫处理取饱满、无虫蛀和表皮无破损的大豆种子各10粒, 经70%乙醇和20%次氯酸钠溶液分别浸泡1 min和15 min后, 无菌水洗10次, 置无菌水浸润的滤纸上, 室温暗培养至萌芽。将萌芽后的种子直接播种于Hoagland’ s营养液(四水硝酸钙945 mg L-1、硝酸钾506 mg L-1、硝酸铵80 mg L-1、磷酸二氢钾136 mg L-1、硫酸镁493 mg L-1、铁盐溶液2.5 mL L-1、微量元素液5 mL L-1, pH 6.0, 其中K+浓度约为6 mmol L-1)浸泡过的蛭石上, 期间不断添加新的营养液保持蛭石湿润状态, 27℃, 按16 h光照及8 h黑暗的光周期培养至V2期(vegetative stage 2), 进行各种胁迫处理, 包括低钾胁迫(将Hoagland’ s营养液中K+浓度调整为60 μ mol L-1)、盐胁迫(160 mmol L-1)、ABA (0.1 mmol L-1)、PEG-6000 (100 mmol L-1)、低温(0℃), 除低钾胁迫处理需调整Hoagland’ s溶液中K+含量外, 其余皆直接用Hoagland’ s溶液进行配制。处理时间统一为0、4、8、16和24 h。根据已有文献报道, TRK-HKT家族基因优先在根中表达[14, 15, 16, 17, 18], 故本研究中选择各个处理时间段幼苗的根, 液氮速冻后保存于-80℃冰箱, 用于总RNA提取。
1.3 大豆根总RNA提取与cDNA第1链合成采用TRNzol Kit (北京天根生物技术有限公司), 按照说明书方法从大豆根中提取总RNA。反转录前, 用Dnase I (Sigma D5025)处理, 清除残留的痕量基因组DNA。根据PrimeScript 1st Strand cDNA Synthesis Kit (TaKaRa, 大连)操作说明, 合成cDNA第一链。将合成的cDNA按2× 、10× 、50× 和100× 稀释后扩增大豆actin11及TRK-HKT家族基因, 确定反转录cDNA质量及各引物的扩增特异性。反应体系为20 μ L, 包括1.0 μ L的各种稀释倍数的cDNA模板、10 μ mol L-1各0.8 μ L上下游引物、10.0 μ L 2× PCR mix (北京天根生物技术有限公司)、7.4 μ L ddH2O。反应程序为95℃ 3 min; 95℃ 30 s, 58℃ 30 s, 72℃ 30 s, 30个循环; 最后72℃延伸10 min。2.5%琼脂糖凝胶电泳检测PCR结果。-20℃保存cDNA, 用于后续的大豆TRK-HKT家族基因定量分析。
1.4 大豆TRK-HKT家族基因序列检索及定量分析将拟南芥AtHKT1; 1 (GenBank登录号为NP_ 567354.1)蛋白序列在NCBI (http://www.ncbi. nlm.nih.gov/)非冗余蛋白序列数据库(non-redundant protein sequences)及Phytozome v10 (http://www. phytozome.net/)大豆全基因组序列数据库(G. maxWilliams 82. a2. v1)中进行BlastP检索, 得到4个高度相似的大豆基因, 基因组识别号分别为Glyma01g00530、Glyma06g42340、Glyma07g15590和Glyma12g16040。根据检索结果, 利用IDT-DNA软件(https://www.idtdna.com/PrimerQuest/)设计qRT- PCR (quantitative Real-time PCR)引物, 引物P1 (5′ -ATTCTTGGCTGGTTTGATAG-3′ )和P2 (5′ -ATTT CAACGGCTACCATGCTAG-3′ )用于扩增Glyma01g 00530, 扩增片段大小为195 bp; 引物P3 (5′ -TTTCT GCTTCCACAGTTTC-3′ )和P4 (5′ -ACCACCAACAA ACATGAGAAGG-3′ )用于扩增Glyma06g42340, 扩增片段大小为98 bp; 引物P5 (5′ -TCTACCCTTCCA GCATTT-3′ )和P6 (5′ -TGGTATAACGGGGGTCCTTG G-3′ )用于扩增Glyma07g15590, 扩增片段大小为115 bp; 引物P7 (5′ -CCTGACTCGAACAAGATTAC- 3′ )和P8 (5′ -TTAATCGAGGGCTTCTTTGTGG-3′ )用于扩增Glyma12g16040, 扩增片段大小为102 bp。以大豆内源actin11基因(基因组识别号为Glyma18g 290800)为内标基因, 设计引物P9 (5′ -CTACGAGCA AGAACTGGAGAC-3′ )和P10 (5′ -AGAACCTCTGG ACATCTGAAAC-3′ )扩增该内标基因, 扩增片段大小为117 bp。将在低钾胁迫处理6个大豆品种各个时间段Glyma01g00530的表达量或逆境胁迫处理0 h的Glyma06g42340表达量设为1, 分析大豆TRK-HKT家族成员基因在低钾胁迫条件下6个大豆品种中的相对表达量, 以及Glyma06g42340在4种逆境胁迫条件下6个大豆品种中的表达趋势。利用ABI7500 PCR扩增仪(Applied Biosystems, USA)进行qRT-PCR分析, 反应体系20 μ L, 包含1.0 μ L的cDNA模板、10 μ mol L-1各0.4 μ L上下游引物、10.0 μ L 2× TransStart Green qPCR Supermix UDG (北京全式金生物技术有限公司)、0.4 μ L Passive Reference Dye II, ddH2O补足至20 μ L。反应程序为50℃ 2 min, 95℃ 10 min; 95℃ 5 s, 60℃ 34 s, 共45个循环; 95℃ 15 s, 60℃ 30 s, 95℃ 15 s, 用于熔解曲线绘制。利用2-Δ Δ CT法计算基因的相对表达量, 设每个处理3个重复, 采用DPS 7.05统计分析数据。
1.5 大豆TRK-HKT家族蛋白生化特性分析采用BioEdit软件(http://www.mbio.ncsu.edu/ bioedit/bioedit.html)手工编辑比对大豆TRK-HKT家族蛋白的功能域, 再利用WebLogo (http://weblogo. threeplusone.com/create.cgi)分析残基保守性。蛋白质分子量及等电点预测软件为Compute pI/MW (http:// web.expasy.org/compute_pi/); 蛋白跨膜区域预测软件为TMHMM V2.0 (http://cbs.dtu.dk/services/ TMHMM-2.0/)和SMART (http://smart.emblheidelberg. de/); 蛋白保守域预测软件为Meme V4.9 (http://meme. nbcr.net/meme/cgi-bin/meme.cgi); 蛋白高级结构预测软件为Phyre2(http://www.sbg.bio.ic.ac.uk/phyre2/ html/page.cgi?id=index); 基因启动子元件分析软件为PlantCARE (http://bioinformatics.psb.ugent.be/ webtools/plantcare/html/)。
1.6 植物TRK-HKT家族基因检索及命名根据拟南芥AtHKT1; 1蛋白序列, 采用BlastP方法在NCBI及Phytozome V10中检索已录入或测序物种的HKT编码基因序列。鉴于部分序列测序结果的不完整及可能的编码框预测错误, 本研究对检索到的序列再次利用Softberry网站的FGENESH/ FGENESH+(http://www.softberry.com/)对序列编码区进行2次预测(序列名称及登录号见附表)。另外, 1个来源于大肠杆菌的HKT蛋白(Escherichia coli, 缩写为EcTrkA)用作聚类分析的outgroup。
鉴于TRK-HKT家族基因存在多拷贝现象[19], 为避免命名上混淆, 本研究在Damien Platten等[35]命名规则基础上, 根据检索到的序列实际情况, 将TRK-HKT家族基因命名为xxHKT#1; #2, xx为HKT所属物种的种属名缩写, #1为HKT基因所在进化的一级分支, #2为HKT基因序列分离的时间顺序。其中#2有3种情况: 1)具有原始命名的, 保留原始命名, 如水稻; 2)没有原始命名但物种基因组已测序, 根据其所在染色体的序号依次命名, 如大豆; 3)没有原始命名也无全基因组测序结果, 参考GenBank录入时的名称命名, 如小麦族。
1.7 大豆TRK-HKT家族基因同源比对和聚类分析采用在线ClustalW2 (http://www.ebi.ac.uk/Tools/ clustalw2/index.html)进行序列同源比对, 比对结果经手工调整后, 利用Mega 6.0软件的Neighbor- Joining法构建系统发育树, 并经BootStrap检验, 参数设置为1000个BootStrap, 其他参数为默认。
2 结果与分析2.1 大豆TRK-HKT家族成员低钾胁迫条件响应机制鉴于拟南芥中仅1个HKT编码基因, 本研究以拟南芥的HKT蛋白序列为种子序列, 在NCBI非冗余蛋白序列数据库中检索大豆已录入的HKT蛋白序列, 共得到10个包含全长编码区序列, 经与Phytozome V10数据库中大豆品种Williams 82全基因组序列比对发现, 仅存在4个HKT编码基因, 分别是Glyma01g00530、Glyma06g42340、Glyma07g 15590和Glyma12g16040, 根据方法1.6的命名规则, 依次命名为GmHKT1; 1、GmHKT1; 2、GmHKT1; 3和GmHKT1; 4。进行qRT-PCR分析前, 采用常规PCR进行引物特异性检测及反转录cDNA模板的质控(见附图1), 大豆TRK-HKT家族4个基因及内标基因actin11的PCR扩增产物中无非特异性条带出现, 仅目标带随着模板稀释倍数递增而产率递减, 表明设计的qRT-PCR引物特异性强, 反转录的cDNA完整性好, 能满足qRT-PCR分析要求。
为了解析大豆TRK-HKT家族基因对低钾胁迫的表达响应, 采用qRT-PCR技术, 选用6个大豆品种(钾高效和钾低效品种各3个), 在60 μ mol L-1 K+条件下的大豆TRK-HKT家族基因表达分析表明, 除个别处理时间下个别品种相对表达量有所不同外(如0 h的冀豆8号、8 h的中豆32、16 h的中黄4号和中黄13等), 4个基因在低钾胁迫条件下的总体相对表达量趋势为GmHKT1; 2> GmHKT1; 4> GmHKT1; 1> GmHKT1; 3(图1), 其中, 相差最大的结果出现在8 h的九农15品种中, GmHKT1; 2的表达量约为GmHKT1; 3的3.59倍。比较GmHKT1; 2与其他3个基因的相对表达量表明, 除低钾胁迫8 h中黄13的GmHKT1; 2与其他3个基因相对表达量差异不显著外, 其余均达到显著或极显著水平, 其中, 在低钾处理16 h的GD8521 (钾低效大豆品种)及24 h的中黄13 (钾低效大豆品种)中, GmHKT1; 2仅与GmHKT1; 3的相对表达量差异显著, 而在钾高效品种的部分处理时间段中, GmHKT1; 2与其他3个基因的相对表达量差异均极显著, 如低钾胁迫8 h和16 h的中豆32和九农15等(图1), 表明GmHKT1; 2可能在大豆K+运输中起主要作用。
图1
Fig. 1
| Figure OptionViewDownloadNew Window | |
 | 图1 低钾胁迫条件下不同钾利用效率大豆品种的TRK-HKT家族基因表达谱* 和* * 分别表示显著性水平为0.05和0.01。I~VI: 大豆品种中黄4号、中黄13、GD8521、冀豆8号、中豆32和九农15。Fig. 1 Expression profiling of soybean TRK-HKT family genes in different soybean accessions under low potassium stress* Significant at P< 0.05; * * Significant at P< 0.01. I-VI: G. max accession Zhonghuang 4, Zhonghuang 13, GD8521, Jidou 8, Zhongdou 32, and Jiunong 15, respectively. |
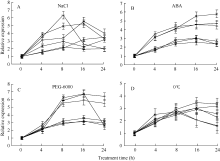
2.2 逆境胁迫下大豆GmHKT1; 2的表达 在培养液中分别添加160 mmol L-1 NaCl、0.1 mmol L-1 ABA、100 mmol L-1 PEG-6000或将植株置于0℃, 模拟自然条件下的逆境胁迫, GmHKT1; 2表达表明, 除低温胁迫对GmHKT1; 2诱导表达在钾利用效率差异品种中不明显外(图2-D), 其余3个胁迫处理在钾高效品种中诱导表达水平明显高于钾低效品种。其中, 冀豆8号在NaCl胁迫处理8 h时, GmHKT1; 2相对表达量最高, 约为处理前的6.3倍(图2-A), 而在ABA处理中, 最高相对表达量出现在中豆32处理24 h, 约为处理前的5.8倍(图2-B)。在模拟旱胁迫(PEG-6000)处理中, 中豆32和九农15处理16 h时表达量最高, 约为处理前的6.8倍(图2-C), 表明这3个处理对钾高效大豆品种的GmHKT1; 2的胁迫诱导明显。在4个处理中, 钾低效品种的GmHKT1; 2的表达量也受到胁迫诱导, 但表达量较低, 表明钾低效品种的GmHKT1; 2对胁迫响应迟缓。
图2
Fig. 2
| Figure OptionViewDownloadNew Window | |
 | 图2 大豆GmHKT1; 2基因在不同品种不同胁迫条件下的表达Fig. 2 Expression profiling of soybean GmHKT1; 2 under different abiotic stresses from different soybean accessions |
2.3 大豆TRK-HKT家族成员基因结构及功能域大豆TRK-HKT家族基因的基因组DNA (genomic DNA, gDNA)全长分析表明, GmHKT1; 1、GmHKT1; 2、GmHKT1; 3和GmHKT1; 4的第1个外显子和内含子分别占各基因gDNA全长的26.89%/ 61.37%、24.08%/59.49%、30.33%/43.43%和26.06%/ 51.18% (见附表), 其中GmHKT1; 2编码蛋白最大(645个氨基酸), 4个编码蛋白的分子量介于56.23~ 73.11 kD, 等电点(pI)相近, 介于9.09~9.23。氨基酸序列两两比对结果表明, GmHKT1; 2和GmHKT1; 4一致性最高(85.7%), 其次是GmHKT1; 1与GmHKT1; 3 (83.3%)。
基于in silico技术, 对大豆TRK-HKT家族4个成员进行氨基酸水平的Blosum62比对, 发现大豆TRK-HKT家族成员全部具有典型的Ser-Gly-Gly- Gly保守motif (每个氨基酸各位于1个P-loop中, 图3箭头)。为了明晰大豆TRK-HKT家族蛋白其他的功能保守域, 利用Meme V4.9共发现3个保守的功能motif (图3中双向箭头)。其中, motif 1位于F223~Y259 (氨基酸位置参考GmHKT1; 1, 下同), 包含K+选择性运输的保守motif中的第2个G, motif 2位于Y409~I442, 而motif 3位于C466~K506, 包含HKT蛋白ATP结合结构域, 序列比对发现, 3个motif的氨基酸组成相似性较高。此外, 利用在线WebLogo对大豆4个功能保守氨基酸残基前后各10个氨基酸分析表明, 在核心功能残基上下游存在许多保守的极性氨基酸, 如丝氨酸(S)、苏氨酸(T)、蛋氨酸(M), 以及带电荷氨基酸, 如天冬氨酸(D)、谷氨酸(E)、天冬酰胺(N)、精氨酸(R)及组氨酸(H)(图4)。
图3
Fig. 3
| Figure OptionViewDownloadNew Window | |
 | 图3 大豆TRK-HKT家族成员序列比对黑色及灰色背景示完全一致和部分一致的序列; 向下箭头表示K+选择性运输保守的氨基酸; 半圆弧表示ATP结合位点; 下画线表示α 螺旋区, 用H#表示; 3个motif和用双向箭头在序列上方表示; 8个跨膜区在序列上方用中括号表示, 并用M1/2A/B/C/D标注(参考文献[17])。Fig. 3 Sequence alignment among the members of soybean TRK-HKT familyAmino acids with high and partial identities are shown in black and grey background, respectively. Downward arrows represent conserved amino acid residues responsible for K+ transportation. ATP binding motif is shown by semicircle. α -helices are underlined sequentially and labeled as H#. Three motifs are shown by double-sided arrows. Eight transmembrane domains are shown as M1/2A/B/C/D in brackets over the sequences according to reference [17]. |
图4
Fig. 4
| Figure OptionViewDownloadNew Window | |
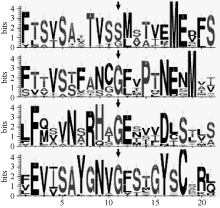 | 图4 大豆TRK-HKT家族蛋白4个P-loop结构域WebLogo分析箭头所示为保守的功能域残基; 自上而下依次为4个功能保守域所在的P-loop; 字母不同高度代表氨基酸残基出现的频度差异程度; 横纵坐标分别表示氨基酸残基数和残基频度。Fig. 4 WebLogo analysis of amino acids of four P-loops in soybean TRK-HKT familyArrows represent the conserved functional motif. Four conserved functional amino acid residues in each P-loop are shown from top to bottom. Height of letter displays relative frequency of each amino acid residue. Abscissa and ordinate demonstrate the number of amino acid residues and relative frequency of each amino acid residue, respectively. |
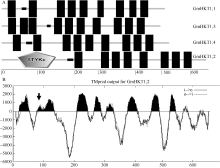
植物TRK-HKT家族蛋白由8个跨膜区和4个P-loop构成, 彼此相间排列(见附图2)。为了解析大豆TRK-HKT家族成员蛋白跨膜区结构, 利用TMHMM V2.0和SMART进行蛋白跨膜区结构预测表明, 除GmHKT1; 2具有8个跨膜结构域外(图5-A), 其余3个成员包含9~10个跨膜区, 其中低复杂区(low complexity region)在4个大豆HKT蛋白中都存在。利用TMHMM V2.0对GmHKT1; 2的跨膜区的2次预测表明GmHKT1; 2具有典型植物HKT蛋白家族的8个跨膜区(图5-B)。
图5
Fig. 5
| Figure OptionViewDownloadNew Window | |
 | 图5 大豆TRK-HKT家族蛋白跨膜结构域分析A: 大豆TRK-HKT家族蛋白跨膜结构域; STYKc: 丝氨酸/苏氨酸/酪氨酸蛋白激酶区; 短粗横线为low complexity region; 柱形为跨膜区; 横坐标表示氨基酸位置; B: GmHKT1; 2蛋白跨膜结构域; 箭头指向第1个P-loop, 横纵坐标分别表示氨基酸位置和预测分值。Fig. 5 Bioinformatic prediction on transmembrane domains of soybean TRK-HKT familyA: Transmembrane domains of soybean TRK-HKT family; STYKc: Ser/Thr/Tyr protein kinase domain; Short horizontal thick lines and columns represent low complexity region and transmembrane domains, respectively; Abscissa demonstrates the position of amino acids. B: Transmembrane domain prediction of GmHKT1; 2; Arrow points to the first P-loop. Abscissa and ordinate demonstrate the position of amino acids and score of prediction, respectively. |
为了进一步解析大豆GmHKT1; 2蛋白空间构象, 利用在线Phyre2对大豆GmHKT1; 2蛋白进行高级结构预测。以枯草杆菌(Bacillus subtilis) Ktrab蛋白(protein data bank, PDB登录号为c4j7ck)的3D结构为模板, 构建大豆GmHKT1; 2蛋白3D模型。该模型覆盖大豆GmHKT1; 2总氨基酸约60%, 模型吻合度为100%。利用PDB中其他3D模板进行大豆GmHKT1; 2模型构建时, 尽管氨基酸覆盖率提高到74%, 但模型吻合度下降为90%, 故选择c4j7ck为模板进行大豆GmHKT1; 2同源建模(图6)。由图6可知, 大豆GmHKT1; 2蛋白结构中无β 折叠存在, 仅有许多α 螺旋和无规则卷曲结构组成, 这与大豆GmHKT1; 2蛋白二级结构预测结果一致(图3)。4个保守氨基酸残基的空间构象形成一个“ 漏斗样” 结构(图6中的黑点), 充当K+/Na+运输通道, 并紧邻ATP结合结构域(图6箭头所示, 保守残基见图3半圆弧所示), 从而方便K+/Na+转运和供能。
图6
Fig. 6
| Figure OptionViewDownloadNew Window | |
 | 图6 大豆GmHKT1; 2蛋白3D模式图A: 正面图; B: 反面图。黑点为4个保守残基; 圆球为转运的离子; 箭头所示为ATP结合位点; α 螺旋和无规则卷曲分别用黑色及灰色表示; N和C分别代表GmHKT1; 2的N端和C端。Fig. 6 3D homologous modeling of GmHKT1; 2A: front view; B: back view. Four conserved residues and transported ions are shown as dots and sphere. Arrows point to ATP binding motif. Random curls and α -helices are shown in grey and black color in ribbon, respectively. N and C: N and C terminus of GmHKT1; 2 separately. |
2.4 大豆TRK-HKT家族成员启动子功能元件HKT蛋白表达调控是启动子上游的顺式作用元件与多种反式作用因子相互作用的结果。对大豆HKT基因起始密码子上游2000 bp区段, 利用PlantCARE数据库, 进行顺式作用元件预测表明这些顺式作用元件主要与激素/环境胁迫响应及组织特异表达有关, 且不同的大豆HKT基因启动子区域所包含的顺式作用元件有所不同, 暗示这些基因表达具有时空差异性(表1)。胁迫相关元件(如ABRE、MBS、TC-rich repeats和5° UTR Py-rich stretch)的存在, 表明部分大豆HKT基因表达受到胁迫调控。大豆HKT基因启动子上游区存在组织/器官特异表达响应元件(如RY-element和Skn-1_motif), 说明这些基因具有组织特异表达特性。这些顺式作用元件或单个或协同发挥作用, 共同调控并实现大豆TRK- HKT家族基因的时空表达。
表1
Table 1
表1(Table 1)
| 表1 大豆TRK-HKT家族基因各种顺式作用元件数目 Table 1 Functional motif numbers of cis-regulatory elements identified in soybean TRK-HKT family genes | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
2.5 大豆TRK-HKT家族成员系统进化为了构建大豆TRK-HKT家族成员的系统发育树, 利用拟南芥AtHKT1; 1蛋白序列, 在NCBI非冗余蛋白序列数据库进行BlastP检索, 共得到111个序列, 覆盖了17个物种。随后, 在Phytozome V10数据库中, 利用BlastP检索全基因组已测序植物HKT蛋白序列, 将2种检索结果比对, 并通过FGENESH/FGENESH+进行序列编码区的2次确认。根据序列检索结果, 发现部分物种TRK-HKT家族基因存在选择性拼接现象, 为了降低序列冗余度, 提高聚类分析的可靠性, 在聚类分析时仅选择最长的序列, 最终得到62个HKT蛋白序列(包括进化树构建中outgroup的大肠杆菌TrkA蛋白)(见附表1)。对基因组已测序物种的TRK-HKT家族成员基因的结构分析表明, 这些基因均包含3个外显子和2个内含子。对各个外显子及内含子所占基因的gDNA全长比例的统计分析发现, 第1个外显子及内含子片段大小占基因总长度约70% (见附表1), 且这2个片段在所有检索到的植物TRK-HKT家族中差异显著(P=0.032和P=0.019)。
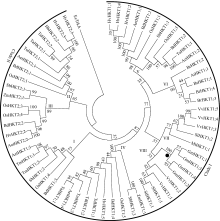
为了解析大豆TRK-HKT家族成员系统进化关系, 对检索到的61个植物源HKT蛋白序列进行聚类分析, 构建无根进化树, 其中来自大肠杆菌的EcTrkA用作outgroup (图7)。结果表明, 植物TRK-HKT家族成员共形成2个主分支, 根据其进化顺序分为8个亚家族, 其中第一分支较大, 包含45个HKT蛋白, 由所有的双子叶植物和部分单子叶植物的HKT蛋白构成, 共分为6个亚家族(I、IV、V、VI、VII和VIII), 而第二分支主要包含单子叶植物的16个HKT蛋白, 组成2个亚家族(II和III)。从图7可看出, 来源于同一物种的不同HKT蛋白家族成员分布在不同的分支或亚家族, 特别是单子叶植物的HKT蛋白, 表明这些蛋白的早期编码基因复制发生在物种分化之前。大豆TRK-HKT家族成员进化上较为保守, 主要位于第一分支的亚家族VIII, 进一步细分为2个小亚群, 即GmHKT1; 1和GmHKT1; 3为一个小亚群, 而GmHKT1; 2和GmHKT1; 4为另一个小亚群, 2个小亚群内部可能存在功能相关性。此外, 大豆TRK-HKT家族基因在进化上与VvHKT1; 1、VvHKT1; 2、SlHKT1; 1及StHKT1; 1进化关系较近, 而与豆科植物苜蓿MtHKT1; 1进化关系较远, 后者与SlHKT1; 2进化关系较近(亚家族VII)。
图7
Fig. 7
| Figure OptionViewDownloadNew Window | |
 | 图7 61个来自不同植物的TRK-HKT家族成员系统发育树分支节点数值表示该进化分支在BootStrap检验后的置信度; 系统发育树有2个分支包含8个亚家族(I~VIII); 来自大肠杆菌的TrkA用作outgroup; 圆点表示大豆TRK-HKT家族的4个成员。各基因命名及登录号见附表。Fig. 7 Phylogenetic tree among the deduced amino acid sequences of 61 members of plant TRK-HKT familyNumbers on the main branches indicate BootStrap percentages for 1000 replicates. Two major clades (1-2) with a black arc consisting of eight subgroups in nodes (I-VIII) identified in the TRK-HKT family are highlighted. TrkA from E. coli was used as outgroup for phylogentic analysis. Four members of soybean TRK-HKT family are shown by dots in node. Refer to supplemental Table for the nomenclature and accession numbers of plant TRK-HKT family genes. |
3 讨论HKTs蛋白属膜整合蛋白(integral membrane proteins, IMPs)的一种, 参与植物对各种阳离子转运, 协调植物对各种逆境胁迫的耐受性[22]。然而, 由于植物TRK-HKT家族成员较多, 其参与植物对逆境胁迫调控可能涉及一个负责的调控网络。因此, 对植物TRK-HKT家族基因功能研究, 首先应对其结构、表达和进化等方面进行深入研究。
3.1 大豆TRK-HKT家族基因结构保守性典型的HKT蛋白由8个跨膜区(transmembrane domain)和4个P-loop构成, 即4个串联的MPM (transmembrane-pore-transmembrane)结构[17]。本研究通过对大豆TRK-HKT家族成员蛋白跨膜区结构预测, 发现仅GmHKT1; 2符合这一典型结构(图5-A和B), 表明GmHKT1; 2可能对大豆K+转运具有更高的亲和性。
对大豆TRK-HKT家族4个P-loop氨基酸组成分析发现, 在4个P-loop中以极性氨基酸居多, 如S和T等, 同时也发现带负电氨基酸残基的存在(如D及E)。根据Mä ser等[19]利用ConSurf软件对这些位点的分析, 认为植物TRK-HKT家族蛋白P-loop区极性氨基酸(如S和T)具有辅助阳离子运输作用, 而紧接着第3个P-loop的G下游保守的E可能起阻止阴离子渗入的作用。进一步对大豆TRK-HKT家族第3个P-loop和motif 3的氨基酸组成分析, 发现在保守G残基上游的第3和第2位为保守的带正电残基RH motif (图3), 在motif 3中有2个保守的GR motif (图3中半圆弧所示)。Kato等[36]及Cao等[37]研究认为, 前者(即RH motif)在离子选择性透过方面有重要作用, 而后者(即GR motif)则充当ATP结合位点, 推测大豆TRK-HKT家族成员可能通过这2个保守功能域协同作用, 参与和维持K+的转运和能量供应。
进化分析表明, 大豆TRK-HKT家族基因十分保守, 全部4个成员同属第一分支的第VIII亚家族(图7), 除具有第1分支保守的4个残基外, 本研究也发现V65T、M73L、F217S、F/Y228L和I/L357C 5个位点与2个进化分支具有较高连锁性(见附图3虚线箭头所示), 表明这5个位点可能也与2个进化分支的功能连锁, 但确切功能未知, 下一步可通过定点突变技术研究这些保守位点功能。相比较而言, 单子叶植物TRK-HKT家族基因进化较为活跃, 分属2个进化分支, 暗示TRK-HKT家族基因复制发生在单子叶和双子叶植物分化之前, 伴随基因复制, 功能也发生改变。进一步分析发现, 来源于同一单子叶植物的HKT基因位于不同的亚群, 表现为同一单子叶植物的HKT彼此分离, 而与其他单子叶植物的同类基因共为一支, 如粗山羊草的AetHKT2; 2与AetHKT2; 1发生进化分离, 而与HvHKT2; 2共为一个分支, 且AetHKT2; 2与AetHKT2; 1分属不同的亚家族, 表明单子叶植物内部的基因复制也发生在单子叶物种分化之前。Waters等[22]研究发现, 尽管OsHKT2; 1具有Ser-Gly-Gly-Gly保守的motif, 但进化上属于第二分支, 可能存在其他保守的氨基酸残基决定了OsHKT2; 1对运输离子的选择性, 暗示后续对上述5个进化保守残基功能研究的必要性。
3.2 大豆TRK-HKT家族基因功能多样性植物TRK-HKT基因进化不同预示着功能差别[38]。研究表明, 第1分支的HKT蛋白成员具有清除芽木质部Na+功能[39], 而大豆TRK-HKT家族4个成员全部聚类在第1分支, 推测其可能在减少大豆芽木质部Na+累积方面具有重要作用。然而, 第一分支的HKT家族成员的离子转运功能可能要更复杂, 如对盐水芹(Thelungiella salsuginea) HKT的2个成员蛋白结构研究发现, 位于PB和M2B的D207和D238对盐水芹HKT的K+转运能力至关重要[40], 在拟南芥及其他植物中, 这2个位点均为天冬酰胺(Asn), 而在大豆中, 则分别被S225及N256 (图3)替换, 表明大豆TRK-HKT家族成员具有更加复杂的离子转运特性。进化上, 大豆TRK-HKT家族成员与SlHKT1; 1进化关系较近(同属第VIII亚家族), 而与SlHKT1; 2关系较远(分属第VII、VIII亚家族, 图7)。对SlHKT1; 2和SlHKT1; 1离子转运功能研究发现, SlHKT1; 1对Na+具有更高的转运能力[41], 推测大豆TRK-HKT家族蛋白对Na+转运可能具有较高亲和力。
真核生物基因功能的发挥通常受其上游启动子元件调控。对大豆TRK-HKT家族基因启动子功能元件预测表明, 大豆TRK-HKT基因对各种激素、逆境胁迫都有响应, 但针对各种响应元件, 不同的成员中差别较大, 表现为有或无、多与少的区别, 表明大豆TRK-HKT各成员对不同信号响应存在差别, 即存在功能上的趋异分化。对番茄栽培种(Solanum lycopersicum)和野生近缘种(S. pennellii)的HKT1; 2功能对比研究发现, SlHKT1; 2具有“ 排Na+” 功能, 耐盐性较强, 而SpHKT1; 2具有“ 容Na+” 功能, 耐盐性较弱[42], 说明同一基因存在进化上的功能分化。大豆与其野生种HKT基因是否也存在这种现象, 值得深入探究。尽管水稻中的HKT基因存在7个同源基因(本研究中blastP结果是9个, 其中有2个选择性拼接的结果), 但只有OsHKT1; 4和OsHKT1; 5参与水稻的耐盐胁迫调控, 且2个基因对清除植株不同部位冗余Na+具有功能分工, 即OsHKT1; 5清除根中大部分冗余Na+, 而OsHKT1; 4则主要清除植株地上部分冗余的Na+, 是对OsHKT1; 5功能的进一步完善和补充[43]。然而大豆TRK-HKT家族基因是否也存在这种功能分工, 目前还没有精确组织定位的实验证据。研究认为, 室内试验中发现的不同HKT蛋白间的功能差别可能是对自然条件下各基因功能差别的一个真实反映[44]。同时, 在对番茄SlHKT1; 1和SlHKT1; 2的功能研究时也发现, 启动子差异是导致二者功能差异的原因所在[41]。因此, 可以构建由大豆TRK-HKT家族各基因启动子与GUS基因的融合载体, 实现融合基因在大豆各组织中表达, 并人工模拟自然界的各种逆境条件处理受体材料, 通过GUS基因的定性和定量表达, 解析大豆TRK-HKT家族各基因功能组织定位, 及对各种胁迫应激响应机制等。
3.3 大豆TRK-HKT家族基因表达与逆境胁迫相关性作物生长过程易受各种自然条件的胁迫影响, 如干旱、低温、高盐等胁迫。通过实验手段, 模拟作物在自然生长条件下经历的各种逆境胁迫, 有助于深入理解作物是如何调节自身的生理及表型变化以适应这些胁迫[12]。本研究利用实验室模拟自然条件下5种胁迫处理, 通过qRT-PCR方法, 初步得出大豆TRK-HKT家族基因表达受到低钾胁迫诱导, 但相比其他3个基因, GmHKT1; 2受低钾胁迫诱导明显, 同时, 在供试的6个大豆品种中, GmHKT1; 2对各种逆境胁迫也表现出明显的诱导表达效应, 推测它可能是大豆各种逆境胁迫条件响应最活跃的一个基因。尽管有研究发现另外的大豆HKT基因对逆境胁迫具有积极的响应表达[11], 推测可能是所选材料的差异以及qRT-PCR分析时所选植株的营养生长阶段(文献[11]所用的是V1期, 而本研究所用的是V2期)的不同所致, 且此现象已在很多物种的基因表达研究中得以证实[45, 46, 47], 同时这也是mRNA差别显示技术(mRNA differential display)筛选植物目标性状关联基因重要依据之一[48]。因此, 本研究中筛选到的大豆逆境胁迫条件下活跃表达的GmHKT1; 2基因, 有可能参与大豆逆境胁迫调控过程, 对该基因的深入研究将有助于进一步揭示大豆耐逆境胁迫分子机制, 为大豆耐逆境胁迫分子育种提供新的基因资源。
4 结论根据NCBI及Phytozome v10数据库, 筛选到4个大豆TRK-HKT家族基因, 分别命名为GmHKT1; 1、GmHKT1; 2、GmHKT1; 3和GmHKT1; 4。该家族基因受低钾胁迫诱导表达, 其中GmHKT1; 2对逆境胁迫如NaCl、ABA和PEG-6000表达响应明显。GmHKT1; 2蛋白存在4个串联的MPM结构和3个motif, 每个P-loop中各包括一个保守的功能氨基酸残基, 且4个保守的残基形成“ 漏斗样” 结构, 并毗邻ATP结合结构域, 共同促进K+/Na+转运。该家族各基因间内含子及外显子大小差异较大, 从而引起各基因gDNA长度不同。该家族成员包含参与组织表达定位和各种应激反应的重要顺式作用元件。该家族基因在进化上十分保守, 主要位于第一进化分支第VIII亚家族, 除4个保守的功能残基外, 还具有5个保守的、与该进化分支密切相关的位点。
Supplemental table 1
附表1(Supplemental table 1)
| 附表1 植物TRK-HKT家族基因结构、命名及登录号 Supplemental table 1 Structure, nomenclature, and accession numbers of plant TRK-HKT family genes |
Supplemental fig. 1
| Figure OptionViewDownloadNew Window | |
 | 附图1 大豆TRK-HKT家族基因qRT-PCR引物特异性检测及合成的cDNA质控分析M: DNA marker I (北京天根生物技术有限公司); 用于每个基因扩增的模板cDNA稀释倍数依次为: 2× 、10× 、50× 、100× (从左至右)。Supplemental fig. 1 Quality check for qRT-PCR primer specificity in soybean TRK-HKT family genes and quality control for transcripted cDNAM: DNA marker I (Beijing Tiangen Co. Ltd.); Diluted cDNAs for each gene amplification are 2× , 10× , 50× , and 100× shown from left to right. |
Supplemental fig. 2
| Figure OptionViewDownloadNew Window | |
 | 附图2 典型的HKT蛋白MPM(transmembrane-pore-transmembrane)结构[17]PA/B/C/D表示4个P-loop; M1/2A/B/C/D表示8个跨膜区。N和C表示蛋白的N和C端。Supplemental fig. 2 Schematic of classic MPM (transmembrane-pore-transmembrane) domain of HKT proteins[17]PA/B/C/Drepresent four P-loop; M1/2A/B/C/Ddenote eight transmembrane domains. N and C: N and C terminus of HKT proteins separately. |
Supplemental fig. 3
| Figure OptionViewDownloadNew Window | |
 | 附图3 植物TRK-HKT家族蛋白P-loop结构比对分析PA、PB、PC和PD表示4个P-loop。实线箭头表示与2个分支密切相关的氨基酸残基, 虚线箭头表示与2个分支可能密切相关的保守残基。* 表示完全一致的氨基酸, : 表示高度相似的氨基酸, · 表示部分相似的氨基酸。横线为2个分支的分界线。大豆TRK-HKT家族成员用粗的进化分支表示。Supplemental fig. 3 Sequence alignment of P-loop of plant TRK-HKT family proteinsPA, PB, PC, and PD represent 4 P-loops. Solid and dotted arrows display confirmed and putative amino acids relevant to functional difference in evolution, respectively. * , :, and . denote those amino acid residues with high identity, high similarity, and partial similarity separately. The two clades were separated by a horizontal line. The bold braches represent the members of soybean TRK-HKT family. |
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
参考文献View Option
原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | |
| [3] | |
| [4] | |
| [5] | |
| [6] | |
| [7] | |
| [8] | |
| [9] | |
| [10] | |
| [11] | |
| [12] | |
| [13] | |
| [14] | |
| [15] | |
| [16] | |
| [17] | |
| [18] | |
| [19] | |
| [20] | |
| [21] | |
| [22] | |
| [23] | |
| [24] | |
| [25] | |
| [26] | |
| [27] | |
| [28] | |
| [29] | |
| [30] | |
| [31] | |
| [32] | |
| [33] | |
| [34] | |
| [35] | |
| [36] | |
| [37] | |
| [38] | |
| [39] | |
| [40] | |
| [41] | |
| [42] | |
| [43] | |
| [44] | |
| [45] | |
| [46] | |
| [47] | |
| [48] |
