

1. 武汉理工大学 化学化工与生命科学学院,湖北 武汉 430070;
2. 武汉理工大学 三亚科教创新园,海南 三亚 572024
收稿日期:2020-07-12;接收日期:2020-09-22;网络出版时间:2020-10-30
基金项目:国家中组部“****”青年项目(No. 40127002),三亚崖州湾科技城管理局研发重点项目(No. SKJC202001004) 资助
摘要:乳酸是一种重要的工业化学品,被广泛应用于各个行业。近年来,随着聚乳酸(PLA) 市场的兴起,乳酸原料的需求也在不断增加。糖基异养生产乳酸所带来的高昂成本与市场需求的矛盾吸引着研究人员积极寻找其他有利的解决方案。蓝细菌光合固碳生产乳酸是一种潜力巨大的新型原料供应策略,基于光合自养的细胞工厂,可以在单一平台上以太阳能为驱动力,从二氧化碳中直接生产出高光学纯度的乳酸。该方法原料廉价易得、过程简单可控、产物明确且易分离,同时达到节能减排和高附加值产品生产的双重效果,具有重要的研究与应用价值。文中回顾了蓝细菌固碳产乳酸技术的发展历程,从代谢基础、代谢工程策略、代谢动力学分析与技术应用等方面,梳理其研究进展和所遇到的技术难点,并对该技术的未来进行展望。
关键词:蓝细菌乳酸代谢工程光合作用共培养动力学分析
Production of lactate from carbon fixation by cyanobacteria: development and prospect
Jianxun Xiao1,2, Pier-Luc Tremblay1, Tian Zhang1,2


1. School of Chemistry, Chemical Engineering and Life Sciences, Wuhan University of Technology, Wuhan 430070, Hubei, China;
2. Sanya Yazhou Bay Science and Technology City, Wuhan University of Technology, Sanya 572024, Hainan, China
Received: July 12, 2020; Accepted: September 22, 2020; Published: October 30, 2020
Supported by: Chinese Thousand Talents Plan Program (No. 40127002), Hainan Yazhou Bay Science and Technology Bureau Grant (No. SKJC-2020-01-004)
Corresponding author: Tian Zhang. Tel: +86-27-87749379; E-mail: tzhang@whut.edu.cn.
Abstract: Lactate is an important industrial chemical and widely used in various industries. In recent years, with the increasing demand for polylactic acid (PLA), the demand for lactate raw materials is also increasing. The contradiction between the high cost and the market demand caused by the heterotrophic production of lactate attracts researchers to seek other favorable solutions. The production of lactate from photosynthetic carbon fixation by cyanobacteria is a potential new raw material supply strategy. Based on the photosynthetic autotrophic cell factory, it can directly produce high optical purity lactate from carbon dioxide on a single platform driven by solar energy. The raw materials are cheap and easy to obtain, the process is simple and controllable, the products are clear and easy to separate, and the double effects of energy saving and emission reduction and production of high value-added products are achieved at the same time, which has important research and application value. This paper reviews the development history of cyanobacteria carbon sequestration to produce lactate, summarizes its research progress and encounters technical difficulties from the aspects of metabolic basis, metabolic engineering strategy, metabolic kinetics analysis and technical application, and prospects the future of this technology.
Keywords: cyanobacterialactatemetabolic engineeringphotosynthesisco-culturekinetic analysis
化石能源的发现点燃了工业革命,然而资源的滥用不仅导致化石能源濒临枯竭,还释放着不计其数的温室气体[1],这种建立在不可再生资源炼制体系基础上的发展模式已经暴露出各种各样的缺点。以生物细胞工厂为核心的技术体系[2],通过温和的条件将可再生资源转化为所需的化学品,逐渐取代石油和石油衍生品,正在逐渐展现其优势。
聚乳酸(Polylactic acid,PLA) 由乳酸聚合而成,是一种有可能取代聚乙烯塑料的可生物降解材料[3]。传统的乳酸生产工艺分为化学法和微生物发酵法。糖类底物是微生物发酵法常用的碳源,以葡萄糖、玉米糖化液或纤维素等糖类为原料生产化学品总是存在一些限制性障碍。为了使聚乳酸的生产更为经济高效,这需要从更便宜的碳源中生产出乳酸,因此寻找一个高效且稳定的乳酸合成平台,拓宽碳源供应来源,对建立可持续、低成本、低能耗、不受地域限制的乳酸制备新路线具有重要意义。
蓝细菌是一类原核光自养微生物,除了可用于研究光合作用外,在光驱动合成生物学和生物技术方面也具有极大的潜力[4]。蓝细菌相对简单的结构,较高的生长速率和易于遗传操作的特点,使其能够通过对蓝细菌天然代谢途径的修饰或引入异源基因来实现细胞内碳汇和能量流向的重新分配[5]。从而可以制造高光学纯度的乳酸,降低下游处理分离难度。相比于传统的生物发酵制备技术,特别是对使用糖类底物进行代谢合成而言,光驱动的蓝细菌细胞工厂生产乳酸,依靠低能耗、低成本的处理就可收获高附加值的化学品,即实现CO2到乳酸的简单而明确的直接转化,这在技术与成本控制上将更加有优势。
本文将从蓝细菌合成乳酸的代谢基础、光合产乳酸的代谢工程开发策略以及光驱共生体系的应用等多方面对蓝细菌光合产乳酸技术进行综述总结,并对未来的发展方向进行展望。
1 蓝细菌合成乳酸的代谢基础蓝细菌可以利用太阳光的能量分解水,产生化学能(ATP) 和还原剂(NADH/NADPH),然后在通过卡尔文循环固定CO2的同时推动乳酸在细胞内的可持续合成[6-8]。乳酸的合成依赖于乳酸脱氢酶(Lactate dehydrogenase,Ldh) 的催化作用[9]。不同构象的乳酸由不同的Ldh进行催化反应[10]。细胞中的丙酮酸通过Ldh的作用直接转化为乳酸,然后被构建的乳酸盐转运蛋白转运至细胞外。
最近有研究团队发现在聚球藻PCC 7942 (Synechococcus elongatus PCC 7942) 中存在另一种不同的合成途径[1]。以卡尔文循环中,通量更大的二羟基丙酮磷酸盐(Dihydroxyacetone phosphate,DHAP) 为底物,由甲基乙二醛合成酶、乙二醛酶Ⅰ、Ⅱ和乳酸盐转运蛋白参与催化,通过异源性甲基乙二醛合成酶的作用将DHAP转化为甲基乙二醛,然后在蓝细菌自有的乙二醛酶作用下经由中间体乳酰谷胱甘肽最终代谢为D-乳酸(图 1)[11]。与丙酮酸一步合成乳酸过程略有不同,从DHAP代谢为乳酸的过程中并不需要NADH等还原力来推动反应的进行。
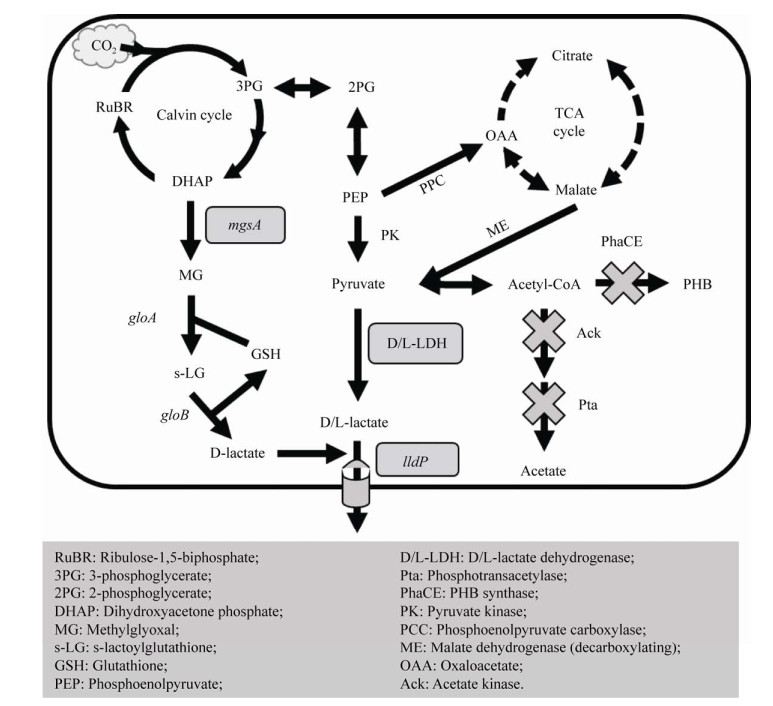 |
| 图 1 蓝细菌乳酸合成和代谢网络 Fig. 1 Cyanobacterial lactate synthesis and metabolic network. |
| 图选项 |
2 蓝细菌合成乳酸的代谢工程开发策略虽然部分蓝细菌自身携带乳酸脱氢酶,但野生型乳酸表达水平几乎检测不到[12-13]。通过代谢工程技术进行改造,构建新的代谢通路,是加强乳酸合成能力、提高光合产乳酸技术应用潜力的重要手段。2010年,第一例表达异源D-乳酸脱氢酶的工作被报道[14],异源L-乳酸脱氢酶表达的工作在2012年被首次发表[15],研究人员在接下来的研究里对蓝细菌的乳酸代谢过程开发做了多方面的努力(表 1)。
表 1 乳酸生物生产研究Table 1 List of lactate bioproduction studies
| Strain | Genetic engineering | Titer (mg/L) | Productivity (mg/L/day) | References | |
| D-lactate | PCC 7942 | +ldhA, +lldP, +udhA | 55 | 13.8 | [14] |
| PCC 7942 | +ldhD, +lldP | 829 | 82.9 | [17] | |
| PCC 7942 | +mgsA, +lldP | 1 230 | 51.3 | [1] | |
| PCC 6803 | +gldA101, +sth | 2 170 | 190.0 | [22] | |
| PCC 6803 | +gldA101, +sth | 1 200 | 60.0 | [31] | |
| PCC 6803 | +LmldhD | 350 | 25.0 | [21] | |
| PCC 6803 | +Dldh; ?pta, ?phaCE | 1060 | 265.0 | [16] | |
| PCC 6803 | ppc-ox; ?ackA | 10 700 | 1 783.3 | [27] | |
| PCC 6803 | me-ox, +ddh; ?ackA | 26 600 | 4 433.0 | [28] | |
| PCC 7002 | ?narB | [26] | |||
| UTEX 2973 | +omcS, +ldh | 300 | 75.0 | [18] | |
| L-lactate | PCC 6803 | +ldh, +sth | 288 | 20.6 | [15] |
| PCC 6803 | +ldh, +lldP | 15 | 0.9 | [13] | |
| PCC 6803 | +ldh | 1 800 | 45.0 | [19] | |
| PCC 6803 | pk-ox, +ldh; △ppc | 837 | 59.8 | [20] | |
| PCC 7002 | +ldh; ?glnA | 795 | 199.0 | [12] |
表选项
2.1 关键基因的修饰乳酸合成途径关键基因的编辑对提升乳酸产量具有重要意义,Niederholtmeyer等首次将异源性的ldh基因引入蓝细菌,合成出较高滴度的乳酸[14],推开了对蓝细菌光合产乳酸研究的大门。常用的Ldh编码基因来源有大肠杆菌[14, 16]、保加利亚乳杆菌[17-18]、枯草芽孢杆菌[15]、乳酸乳球菌[19]等,为探寻不同来源的Ldh对蓝细菌生长和代谢的影响,Joseph等[13]测试了5个不同的Ldh编码基因对蓝细菌的影响,发现大部分基因插入对蓝细菌的生长并无明显影响,作用良好,且不同来源ldh基因的密码子优化还可以在原基础上提高约10%的Ldh表达水平[19]。对工程藻株中ldh的控制系数进行计算,Angermayr等[20]发现ldh活性的增加导致代谢通量的成比例增加,实验表明,含有相同ldh基因的额外拷贝,显示出大约2倍的乳酸生产率,通过乳酸乳杆菌Ldh的过表达可以实现从低表达量到中等量表达时通量的成比例增加,但在最高表达量的突变菌株中,比例相关性会被破坏[21]。Zhou等[16]也发现了保加利亚乳杆菌的DldhL过表达菌株的酶效率是大肠杆菌的DldhE过表达菌株的1.5倍,是基准菌株的2.9倍。具有不同强度的启动子对插入ldh基因的表达也有较大影响,在PrnpB控制下表达乳酸乳杆菌ldh的菌株不显示乳酸产生,由PpsbA2和Ptrc驱动表达乳酸乳杆菌ldh的菌株,在培养的10 d内积累了55 mg/L和126 mg/L的乳酸[19],这表明包括启动子、核糖开关、核糖体结合位点工程等都应该在设计细胞工厂时考虑进去。
近来,有一个研究团队另辟蹊径,通过催化DHAP,建立代谢通路,所得到的工程化菌株的生产速率能够达到51.3 mg/(L·d) (表 1),然而,与野生型相比,通过这种技术改造获得的菌株中都观察到了生长缺陷,这是对乳酸脱氢酶进行改造的菌株中不曾有的[1]。值得注意的是,不管是外源性LDH基因的插入还是构建额外代谢通路,关键功能蛋白质的表达都受到了蓝细菌遗传和代谢背景的影响,这就意味着在未来细胞工厂的构建与开发中,适配性是一个必须考虑的因素。
2.2 辅助因子的改变细胞中低浓度的NADH也是一个主要的限制性因素。光条件下,PCC 7942的NADH与NADPH的比值约为0.15,集胞藻PCC 6803 (Synechocystis sp. PCC 6803) 的胞内NADH/NADPH约为0.14[22-23],而充分供应NADH或NADPH及其高效再生,是通过代谢工程生产化学品的关键[24]。乳酸脱氢酶基因和转氢酶基因的共表达有助于增加细胞内的NADH/NADPH比率,从而使依赖NADH的乳酸脱氢酶效率提升。共表达NADPH/NADH转氢酶编码基因udhA和lldP的基因工程藻株比只表达lldP的藻株乳酸盐产量高了6倍[14]。引入铜绿假单胞菌转氢酶sth可进一步提高乳酸表达水平,并且促使其在生长阶段和非生长阶段都产生乳酸[22]。将PCC 7942中NADH依赖的Ldh通过工程技术手段转变为NADPH依赖,辅因子偏好的改变导致了3.6倍以上的乳酸产量增加[17]。Gordon等也在聚球藻PCC 7002 (Synechococcus sp. PCC 7002) 中验证了技术的可行性[12]。不管是表达转氢酶还是修改乳酸脱氢酶的辅助因子特异性,都会引起藻株的生长受限[14-15, 22],可能的解释就是高速率合成乳酸导致NADPH水平的降低,更多的碳通量从细胞生物质转向合成乳酸,从而扰乱了生长,因此需要协调好各个模块的表达来维持生长和乳酸生产之间的平衡,实现高效、稳定且持续的生产目标。
2.3 乳酸盐转运蛋白的引入限制乳酸合成能力的不仅是代谢过程中合成基因的表达与调控,其关键是如何将胞内分泌的乳酸及时转运至胞外,疏水性细胞膜是基因工程蓝细菌分泌亲水性产物如乳酸盐的主要屏障。乳酸盐转运蛋白是一种非特异性的D-乳酸转运蛋白,通过在大肠杆菌和蓝细菌中使用质子动力来有效地跨膜转运D-乳酸[25]。引入lldP的PCC 6803生长速度加快的同时可以明显观察到上清液中乳酸含量的增加[13]。以异丙基-β-D-硫代半乳糖苷为诱导启动子,在PCC 7942中同时表达大肠杆菌ldhA和lldP基因,细胞向培养基中分泌了超过只表达ldhA基因细胞4倍水平的乳酸产量[14]。
2.4 碳通量的调节蓝细菌在单位时间内捕获与转化的CO2量是固定的,而复杂的生理代谢过程所合成的产物却多种多样,如何限制碳通量向其他产物如糖原、聚-3-羟基丁酸酯(Poly-hydroxybutrate,PHB) 和乙酸等流动,也是当前较为热门的研究方向。在应激条件下,PCC 6803可积累高达其干细胞重量40%的PHB。通过阻断PHB合成途径使D-乳酸产量增加1倍,相同条件下阻断乙酸合成途径可以提高50%的D-乳酸产量,而同时阻断这两条途径可导致D-乳酸产量增加8倍。基因破坏显著改变了碳通量,同时阻断乙酸盐和PHB合成途径对乳酸盐产生具有协同作用[16]。通过下调氮同化关键节点来增加中心碳通量,减缓通过谷氨酰胺合成酶途径的氮同化速率,调节与糖原降解和糖酵解相关的代谢过程,从而提高了固定碳向丙酮酸的流量,将乳酸产率从4.05 mg/(L·h)提高到8.28 mg/(L·h)[12]。用编码氨基糖苷磷酸转移酶Ⅱ(aphAⅡ) 的DNA片段替换PCC 7002中的narB基因,糖原分解代谢中更多的还原当量将转移到还原产物中,结果表明这种方法能提高1倍的D-乳酸产量[26]。Angermayr等发现沉默磷酸烯醇丙酮酸羧化酶基因(ppc) 的表达,可以增加碳向乳酸的分配,但会导致生长缓慢,总固碳率降低[20]。Hasunuma等却发现在Ptrc启动子控制下,过表达内源性磷酸烯醇式丙酮酸羧化酶基因(ppc-ox),以高浓度生物质在厌氧黑暗条件下继续培养可以获得高达1 783 mg/(L·d) 的乳酸产率[27],同样的,Hidese过表达苹果酸脱氢酶(Malic enzyme,ME) 的同时敲除乙酸激酶,黑暗厌氧高生物密度条件下继续培养,可以收获超过26 g/L的乳酸产量[28]。较为理想的实验结果证明了限制和优化碳汇流向来增加乳酸产量的策略在蓝细菌生物炼制系统中具有良好的应用前景。
2.5 基因编辑工具的选择与传统的异养细胞相比,蓝细菌仅可利用有限的遗传工具进行从单基因途径到全基因组水平的操作[29]。在对蓝细菌基因修饰过程中,绝大部分工作者都是通过同源重组对蓝细菌基因组进行编辑[1, 14-15],少数也使用CRISPR干扰下调氮同化关键节点来控制代谢流向[12]。然而蓝细菌基因组多倍体特性导致其通过同源重组的策略获得纯菌株的过程十分烦琐且耗时,而能施加选择性压力的CRISPR技术却能很好地解决这个问题。与同源重组相比,无论是CRISPR/Cas 9还是对蓝细菌更安全的CRISPR/Cpf 1技术都体现出其在无标记敲除、敲除和点突变工程中的实用价值[30]。在未来,有望开发和应用更多、更适配的遗传工具来对工业型菌株进行编辑,而不是仅局限于模式蓝细菌。
3 光限制条件下的乳酸合成的动力学分析与预测细胞阴影的存在,使得辐照度在空间上会随着容器深度和生物密度的变化而变化,而发酵液的充分混合,可以认为分子底物浓度在空间维度上是恒定均一的,因此,将光子作为限制性底物来构建对蓝细菌的生长与产物分泌动力学模型是有利且必要的。Pirt等[32]在其团队之前的工作上进一步地概述了光合作用下生长的热力学与动力学,为光限制条件下的生长建模奠定了理论基础。随后的研究陆续揭示了有关于光限制生长参数的有用信息,如空间平均辐照[33]–总吸光度的测量[34]和连续生长实验中的光分布模型[35]等,但是所涉及的情况为较低密度下的连续培养,难以应用于分批高密度发酵。Clark等构建了一个新的框架,用来量化光限制条件下蓝细菌生长和产物分泌的动力学,并利用了被设计为能合成L-乳酸或L-赖氨酸的PCC 7002与聚球藻UTEX 2973 (Synechococcus elongatus UTEX 2973) 两株工程蓝细菌,详细地推导和证明了在不同实验体系中的蓝细菌生长与产物合成是可以通过使用体积光子传输效率来容易地进行预测与缩放[36]。Clark等的工作对传统的利用比生产率的报告方式进行了修改,提出了以线性产率(Linear production rate,LPR)、线性增长率(Linear growth rate,LGR) 和体积光子传输效率为主体的新型报告方式,从而可以在不同的实验系统之间进行简单的比较,以便在前人基础上进行进一步的研究。虽然简单模型并不能描述产品分泌的全部动态过程,但确实提供了一些关于生产率如何在分批培养过程以及不同实验系统中变化的信息。未来的研究应当偏向于考虑合成乳酸的生物质比率在时间和相关过程条件下的变化,构建统一的动力学框架模型来预测蓝细菌的生长和产物分泌,为工程蓝细菌的代谢动力学提供更多的见解。
4 基于蓝细菌光驱固碳合成乳酸技术的共生体系应用近来,有团队设计并构建了一个双物种微生物共生群落,用于从光产生电能[18]。微生物群落由一株工程蓝细菌和一株工程希瓦氏菌组成,能够快速生长的UTEX 2973充当充电单元,而希瓦氏菌Shewanella oneidensis充当放电单元。整个生物光电系统由工程化UTEX 2973从光和CO2中产生D-乳酸开始,然后工程化希瓦氏菌消耗乳酸对外释放电子。结果显示,D-乳酸盐介导的蓝细菌-希瓦氏菌双物种共生系统能够在40 d以上的时间内从光中产生相对较高功率的电[18]。在共生系统中,乳酸被作为一种高效的能量载体,负责蓝细菌与希瓦氏菌之间的化学通讯与单向能量传递,这种限定微生物种类与约束电子流的策略共同促成了光合-外生电微生物群落的高效与长期功率输出,验证了人工构建光驱固碳共生合成系统理论[37]有效的同时也为乳酸的原位利用提供了实证。
5 总结与展望基于基因、蛋白、生化途径等各个层面上的进步使得乳酸生物合成技术得以逐渐发展起来,定制化的蓝细菌能够突破限制,高效、高通量、定向转化为高纯度的乳酸产品。相较于其他生物制乳酸技术,以太阳能为能源驱动的代谢合成可以同时达到固碳节能和高附加值产品输出的效果。然而,要切实地推动蓝细菌光驱固碳乳酸合成技术的工业化应用,需要解决的问题还有很多。
在2015年发现的UTEX 2973被看作是最佳底盘细胞,短至1.9 h的倍增时间可与酵母相媲美[38],利用更精准的CRISPR技术对UTEX 2973进行代谢工程改造,获得高产且稳定藻株的方法是可行且必要的技术攻关方向。其次,氮磷消耗是需要值得注意的一点,因而以污废水为培养环境,通过消耗废水中的氮磷等元素来维持化学品的生产,这种炼制系统是理想的,也是被证实是具有可行性的[31],这一从废物中进行生物炼制的模式亦可以拓展到其他微生物细胞工厂[39]。蓝细菌的细胞特征,包括氧化还原状态,NADPH/NADH的平衡,以及光合作用-光呼吸的比率,在剪接或微调人工合成途径都应该被考虑进去。值得注意的是,乳酸生产工程藻株受损的细胞强健性和适应性仍然是影响光合细胞工厂大规模应用的潜在因素。同时,为了克服发酵过程中昼夜周期和环境压力带来的障碍,应该通过引入新的保护装置或机制来重新设计细胞工厂的生理功能,以保持甚至加强蓝细菌细胞工厂的效率,并实现稳定且持续的光合生物生产。
参考文献
| [1] | Hirokawa Y, Goto R, Umetani Y, et al. Construction of a novel D-lactate producing pathway from dihydroxyacetone phosphate of the Calvin cycle in cyanobacterium, Synechococcus elongatus PCC 7942. J Biosci Bioeng, 2017, 124(1): 54-61. DOI:10.1016/j.jbiosc.2017.02.016 |
| [2] | Liu ZH, Wang K, Chen Y, et al. Third-generation biorefineries as the means to produce fuels and chemicals from CO2. Nat Catal, 2020, 3(3): 274-288. DOI:10.1038/s41929-019-0421-5 |
| [3] | Panchal SS, Vasava DV. Biodegradable polymeric materials: synthetic approach. ACS Omega, 2020, 5(9): 4370-4379. DOI:10.1021/acsomega.9b04422 |
| [4] | Hitchcock A, Hunter CN, Canniffe DP. Progress and challenges in engineering cyanobacteria as chassis for ligxht-driven biotechnology. Microb Biotechnol, 2020, 13(2): 363-367. DOI:10.1111/1751-7915.13526 |
| [5] | 郗欣彤, 张杉杉, 毛绍名, 等. 蓝细菌光驱固碳合成蔗糖技术的发展与展望. 生物工程学报, 2019, 35(8): 1411-1423. Chi XT, Zhang SS, Mao SM, et al. Cyanobacteria based photosynthetic production of sucrose: development and prospect. Chin J Biotech, 2019, 35(8): 1411-1423 (in Chinese). |
| [6] | Khan S, Fu PC. Biotechnological perspectives on algae: a viable option for next generation biofuels. Curr Opin Biotech, 2020, 62: 146-152. DOI:10.1016/j.copbio.2019.09.020 |
| [7] | Khan AZ, Bilal M, Mehmood S, et al. State-of-the-art genetic modalities to engineer cyanobacteria for sustainable biosynthesis of biofuel and fine chemicals to meet bio-economy challenges. Life (Basel), 2019, 9(3): 54. |
| [8] | Oliver NJ, Rabinovitch-Deere CA, Carroll AL, et al. Cyanobacterial metabolic engineering for biofuel and chemical production. Curr Opin Chem Biol, 2016, 35: 43-50. DOI:10.1016/j.cbpa.2016.08.023 |
| [9] | Ali Abdel-Rahman M, Sonomoto K. Opportunities to overcome the current limitations and challenges for efficient microbial production of optically pure lactic acid. J Biotechnol, 2016, 236: 176-192. DOI:10.1016/j.jbiotec.2016.08.008 |
| [10] | Ito S, Takeya M, Osanai T. Substrate specificity and allosteric regulation of a D-lactate dehydrogenase from a unicellular cyanobacterium are altered by an amino acid substitution. Sci Rep, 2017, 7: 15052. DOI:10.1038/s41598-017-15341-5 |
| [11] | Knoop H, Grundel M, Zilliges Y, et al. Flux balance analysis of cyanobacterial metabolism: the metabolic network of Synechocystis sp. PCC 6803. PLoS Comput Biol, 2013, 9(6): e1003081. DOI:10.1371/journal.pcbi.1003081 |
| [12] | Gordon GC, Korosh TC, Cameron JC, et al. CRISPR interference as a titratable, trans-acting regulatory tool for metabolic engineering in the cyanobacterium Synechococcus sp. strain PCC 7002. Metab Eng, 2016, 38: 170-179. DOI:10.1016/j.ymben.2016.07.007 |
| [13] | Joseph A, Aikawa S, Sasaki K, et al. Utilization of lactic acid bacterial genes in Synechocystis sp. PCC 6803 in the production of lactic acid. Biosci Biotechnol Biochem, 2013, 77(5): 966-970. DOI:10.1271/bbb.120921 |
| [14] | Niederholtmeyer H, Wolfst?dter BT, Savage DF, et al. Engineering cyanobacteria to synthesize and export hydrophilic products. Appl Environ Microbiol, 2010, 76(11): 3462-3466. DOI:10.1128/AEM.00202-10 |
| [15] | Angermayr SA, Paszota M, Hellingwerf KJ. Engineering a cyanobacterial cell factory for production of lactic acid. Appl Environ Microbiol, 2012, 78(19): 7098-7106. DOI:10.1128/AEM.01587-12 |
| [16] | Zhou J, Zhang HF, Meng HK, et al. Production of optically pure D-lactate from CO2 by blocking the PHB and acetate pathways and expressing D-lactate dehydrogenase in cyanobacterium Synechocystis sp. PCC 6803.. Process Biochem, 2014, 49(12): 2071-2077. DOI:10.1016/j.procbio.2014.09.007 |
| [17] | Li C, Tao F, Ni J, et al. Enhancing the light-driven production of D-lactate by engineering cyanobacterium using a combinational strategy. Sci Rep, 2015, 5: 9777. DOI:10.1038/srep09777 |
| [18] | Zhu HW, Meng HK, Zhang W, et al. Development of a longevous two-species biophotovoltaics with constrained electron flow. Nat Commun, 2019, 10: 4282. DOI:10.1038/s41467-019-12190-w |
| [19] | Angermayr SA, Hellingwerf KJ. On the use of metabolic control analysis in the optimization of cyanobacterial biosolar cell factories. J Phys Chem B, 2013, 117(38): 11169-11175. DOI:10.1021/jp4013152 |
| [20] | Angermayr SA, Van Der Woude AD, Correddu D, et al. Exploring metabolic engineering design principles for the photosynthetic production of lactic acid by Synechocystis sp. PCC6803. Biotechnol Biofuels, 2014, 7: 99. DOI:10.1186/1754-6834-7-99 |
| [21] | Angermayr SA, Van der Woude AD, Correddu D, et al. Chirality matters: synthesis and consumption of the D-enantiomer of lactic acid by Synechocystis sp. Strain PCC6803. Appl Environ Microbiol, 2016, 82(4): 1295-1304. DOI:10.1128/AEM.03379-15 |
| [22] | Varman AM, Yu Y, You L, et al. Photoautotrophic production of D-lactic acid in an engineered cyanobacterium. Microb Cell Fact, 2013, 12: 117. DOI:10.1186/1475-2859-12-117 |
| [23] | Takahashi H, Uchimiya H, Hihara Y. Difference in metabolite levels between photoautotrophic and photomixotrophic cultures of Synechocystis sp. PCC 6803 examined by capillary electrophoresis electrospray ionization mass spectrometry. J Exp Bot, 2008, 59(11): 3009-3018. DOI:10.1093/jxb/ern157 |
| [24] | Meng HK, Liu P, Sun HB, et al. Engineering a D-lactate dehydrogenase that can super-efficiently utilize NADPH and NADH as cofactors. Sci Rep, 2016, 6: 24887. DOI:10.1038/srep24887 |
| [25] | Nú?ez MAF, Kwon O, Wilson TH, et al. Transport of L-lactate, D-lactate, and glycolate by the LldP and GlcA membrane carriers of Escherichia coli. Biochem Biophys Res Commun, 2002, 290(2): 824-829. DOI:10.1006/bbrc.2001.6255 |
| [26] | Qian X, Kumaraswamy GK, Zhang SY, et al. Inactivation of nitrate reductase alters metabolic branching of carbohydrate fermentation in the cyanobacterium Synechococcus sp. strain PCC 7002. Biotechnol Bioeng, 2016, 113(5): 979-988. DOI:10.1002/bit.25862 |
| [27] | Hasunuma T, Matsuda M, Kato Y, et al. Temperature enhanced succinate production concurrent with increased central metabolism turnover in the cyanobacterium Synechocystis sp. PCC 6803. Metab Eng, 2018, 48: 109-120. DOI:10.1016/j.ymben.2018.05.013 |
| [28] | Hidese R, Matsuda M, Osanai T, et al. Malic enzyme facilitates D-lactate production through increased pyruvate supply during anoxic dark fermentation in Synechocystis sp. PCC 6803. ACS Synth Biol, 2020, 9(2): 260-268. DOI:10.1021/acssynbio.9b00281 |
| [29] | Sun T, Li SB, Song XY, et al. Toolboxes for cyanobacteria: Recent advances and future direction. Biotechnol Adv, 2018, 36(4): 1293-1307. DOI:10.1016/j.biotechadv.2018.04.007 |
| [30] | Ungerer J, Pakrasi HB. Cpf1 is a versatile tool for CRISPR genome editing across diverse species of cyanobacteria. Sci Rep, 2016, 6: 39681. DOI:10.1038/srep39681 |
| [31] | Hollinshead WD, Varman AM, You L, et al. Boosting D-lactate production in engineered cyanobacteria using sterilized anaerobic digestion effluents. Bioresour Technol, 2014, 169: 462-467. DOI:10.1016/j.biortech.2014.07.003 |
| [32] | Monod J. The growth of bacterial cultures. Annu Rev Microbiol, 1949, 3: 371-394. DOI:10.1146/annurev.mi.03.100149.002103 |
| [33] | Du W, Jongbloets JA, Hernández HP, et al. Photonfluxostat: A method for light-limited batch cultivation of cyanobacteria at different, yet constant, growth rates. Algal Res, 2016, 20: 118-125. DOI:10.1016/j.algal.2016.10.004 |
| [34] | Touloupakis E, Cicchi B, Torzillo G. A bioenergetic assessment of photosynthetic growth of Synechocystis sp. PCC 6803 in continuous cultures. Biotechnol Biofuels, 2015, 8: 133. DOI:10.1186/s13068-015-0319-7 |
| [35] | Perin G, Bernardi A, Bellan A, et al. A mathematical model to guide genetic engineering of photosynthetic metabolism. Metab Eng, 2017, 44: 337-347. DOI:10.1016/j.ymben.2017.11.002 |
| [36] | Clark RL, Mcginley LL, Purdy HM, et al. Light-optimized growth of cyanobacterial cultures: Growth phases and productivity of biomass and secreted molecules in light-limited batch growth. Metab Eng, 2018, 47: 230-242. DOI:10.1016/j.ymben.2018.03.017 |
| [37] | Beliaev AS, Romine MF, Serres M, et al. Inference of interactions in cyanobacterial-heterotrophic co-cultures via transcriptome sequencing. ISME J, 2014, 8(11): 2243-2255. DOI:10.1038/ismej.2014.69 |
| [38] | Yu JJ, Liberton M, Cliften PF, et al. Synechococcus elongatus UTEX 2973, a fast growing cyanobacterial chassis for biosynthesis using light and CO2. Sci Rep, 2015, 5: 8132. DOI:10.1038/srep08132 |
| [39] | 齐允晶, 王家林, 栾国栋, 等. 蓝细菌乙醇光合细胞工厂的发展与展望. 生物工程学报, 2017, 33(6): 891-909. Qi YJ, Wang JL, Luan GD, et al. Cyanobacteria cell factories for ethanol photosynthetic production: development and prospect. Chin J Biotech, 2017, 33(6): 891-909 (in Chinese). |
