孙瑨原1,2, 朱彤1,2, 李涛1,2, 崔颖璐1, 吴边1


1. 中国科学院微生物研究所, 中国科学院微生物生理与代谢工程重点实验室, 微生物资源前期开发国家重点实验室, 北京 100101;
2. 中国科学院大学生命科学学院, 北京 100049
收稿日期:2021-01-19;修回日期:2021-03-11;网络出版日期:2021-08-10
基金项目:中国科学院战略性先导科技专项(XDA24020101);中国科学院战略生物资源计划(KFJ-BRP-009,KFJ-BRP-017-58)
作者简介:吴边,北京大学药学本科,荷兰格罗宁根大学博士毕业,现就职于中国科学院微生物研究所,博士生导师,研究员,获国家自然科学基金委优青资助。主要工作致力于微生物酶相关的机制解析与功能设计。近年来,研究团队将蛋白质计算的前沿技术引入微生物研究领域中,解析了数类微生物碳氮成键酶的进化机制与反应机理,通过人工改造将其应用于生物分子的精准合成与定向修饰,促进了微生物大分子元件设计的发展。相关工作发表在Nature Catalysis、Nature Chemical Biology、National Science Review、ACS Catalysis、Advanced Science等学术刊物上.
*通信作者:吴边, Tel/Fax: +86-10-64806035;E-mail: wub@im.ac.cn.
摘要:可数据化是现代生命科学研究,尤其是合成生物学的一项关键特性。酶作为生命体内催化生化反应的关键分子,其数据化对推动生命科学的基础研究和实际应用都有重要意义。当下商品化的酶大都来源于微生物,建立微生物酶资源数据库不仅可以为酶的分类提供标准参考,还可以指导新型催化元件的挖掘、改造与从头设计。本综述对国际上已有的酶资源数据库建设与发展作了简要介绍,并对基于数据库的微生物酶资源利用作出展望。
关键词:酶数据库大数据微生物资源
Development and applications of enzyme databases
Jinyuan Sun1,2, Tong Zhu1,2, Tao Li1,2, Yinglu Cui1, Bian Wu1


1. CAS Key Laboratory of Microbial Physiological and Metabolic Engineering, State Key Laboratory of Microbial Resources, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
2. College of Life Sciences, University of Chinese Academy of Sciences, Beijing 100049, China
Received: 19 January 2021; Revised: 11 March 2021; Published online: 10 August 2021
*Corresponding author: Bian Wu, Tel/Fax: +86-10-64806035; E-mail: wub@im.ac.cn.
Foundation item: Supported by the Strategic Priority Science and Technology Program from the Chinese Academy of Sciences (XDA24020101) and by the Biological Resources Program from the Chinese Academy of Sciences (KFJ-BRP-009, KFJ-BRP-017-58)
Abstract: Enzymes catalyze most biochemical reactions in living organisms, and the high efficiency, chemo-, regio- and stereoselectivity make enzymes appealing catalysts in many fields. The digitalization of enzyme-related information is of great importance to both academic research and industrial applications. Microbial enzyme resource databases establish foundations for the mining, engineering, and utilization of new biocatalysts. This mini-review will briefly introduce the current development of enzyme databases and discuss the enzyme resources applications facilitated by databases.
Keywords: enzyme databasebig datamicrobial resources
酶作为生命体内化学反应的催化剂,在长期进化或人工选择中获得了高催化活性、高区域与立体选择性等众多特性,在自然科学研究和生物医药、工业、农业等关系到国计民生的重要领域有着广泛应用。微生物是工业酶或商品化酶的最主要来源[1]:聚合酶链式反应(PCR)中使用的Taq酶[2]来源于耐热古菌水生栖热菌(Thermus aquaticus);工业上用于植物纤维材料降解的淀粉酶、纤维素酶和果胶酶等糖苷水解酶通常来自细菌或者真菌[3-4];用于生产药物西格列汀的转氨酶[5]则来源于一株节杆菌(Arthrobacter sp.)。巨大的应用价值促进了酶学研究的快速发展,PubMed中收录的与酶相关的文献已经超过了300万篇,对这些文献报道信息进行系统归纳与整理的数据库也应运而生,这些都为酶的科学研究与应用提供了重要保障。
数据库在现代生物学研究中占有举足轻重的地位。美国国家生物技术信息中心(NCBI)的GenBank数据库是全世界生物学研究人员最常用的生物数据库之一。英国牛津大学出版社每年出版专注于生物信息学数据库研究与发展的《生物数据库和管理杂志》(Database-The Journal of Biological Databases and Curation),同一出版社的著名生物学期刊《核酸研究》(Nucleic Acids Research)同样每年会出版数据库专刊。随着高通量测序和基因合成成本的大幅度降低以及生物信息学工具的发展,新酶的发掘不再依赖特定物种的活菌株,而是通过在数据库中收录的全基因组或宏基因组序列中发掘筛选符合需求的酶序列,然后合成基因并建立重组表达系统,这打破了世界各地生物学研究人员在空间层面上的隔阂。由于能够提供丰富的酶学信息且能辅助新酶的发现,酶资源数据库已经成为酶学研究与应用不可或缺的一部分。通过在PubMed数据库中检索标题或摘要中包含“Enzyme”和“Database”关键词的文献,利用摘要文本制作了词云图(图 1),可以直观地显示酶数据库相关研究的热点。本综述首先介绍酶数据库的构建方式,按照不同的类别介绍常用的微生物相关酶资源数据库,讨论基于微生物酶资源数据库挖掘的应用,最后展望微生物酶资源数据库发展的机遇与挑战。
 |
| 图 1 酶数据库相关文献词云图 Figure 1 Word cloud image created using abstracts of enzyme database publications. |
| 图选项 |
1 酶资源数据库构建方式 酶资源数据库的构建主要使用以下三种方式:(1) 对公共序列/结构数据库如PDB、UniProt中注释信息进行整理与提取;(2) 对文献信息进行人工提取与计算机挖掘;(3) 使用生物信息学方法,如序列同源性比较、系统发育分析等。专注于酶分类与命名的数据库以ExplorEnz为代表,其数据来源于国际酶学委员会(EC)发布的酶分类号(EC number)及其对应的序列。该数据库根据最小酶学分类收集该类酶在文献中常用的名称,并根据文献进行简短的注解。而以BRENDA为代表的数据库则关注酶的科学与应用研究,其数据来源于酶分类与命名数据库、文献和公共序列/结构数据库,构建该类数据库首先要科研人员手动从文献中提取所需信息,以构建包含酶的来源、序列与性质等较为详细注释的核心库。完成核心库的构建后,部分数据库会从人工收集的文献中提取关键词,再使用文本挖掘程序从更大范围的文献中提取相关信息,或利用收集到的序列和结构对公共序列/结构数据库进行搜索、分类,然后整合纳入数据库中。为了方便数据库的后续维护,许多数据库开放了用户上传的权限,允许并鼓励用户上传结构化的实验数据。数据库通常使用SQL构建,其网页服务允许用户浏览或者使用文本搜索来获取所需信息,一些序列数据库会提供序列搜索服务,另有部分大型数据库还会提供简单对象访问协议(SOAP)或者应用程序接口(API),从而允许用户高效快速地访问数据库。构建酶资源数据库的通用流程示意图如图 2所示。
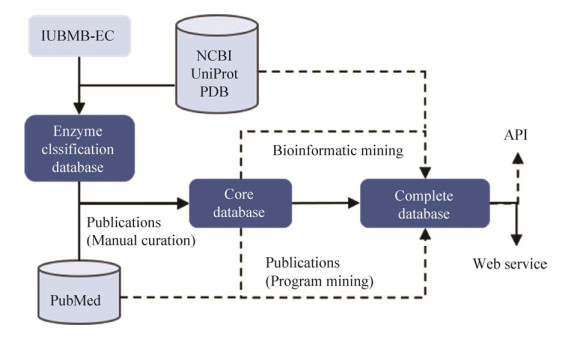 |
| 图 2 构建酶资源数据库的通用流程示意图 Figure 2 General workflow of construction of enzyme database. |
| 图选项 |
总体而言,目前酶数据库的构建对人工依赖程度很高。尽管人工收集文献并组织整理的过程有利于培养科研人员的文献调研能力并拓展他们的视野,但是这种枯燥的劳动密集型工作也会消磨科研人员的兴趣,因此更高程度的自动化仍然是酶资源数据库构建的主要发展方向。挖掘科研论文获得结构化数据的工作主要属于自然语言处理(NLP)的自然语言理解应用范畴,相比于常见的NLP应用场景如机器翻译,科研论文往往具有相对标准的格式和相对集中的单词,现阶段在获得人工标注的核心数据库基础上,NLP的应用将推动酶资源数据库的加速构建。
2 酶资源数据库分类简介 2.1 酶基本信息与机理数据库 国际酶学委员会(EC)由国际生物化学联合会(IUBMB)于1956年成立,该委员会建立了带有系统化和推荐名称的酶代码编号(EC编号)系统[6]。EC号的出现极大地便利了酶的分类,使得国际酶学研究人员交流更加简便。酶学命名与分类数据库方便了科研和工程人员按照EC号查询酶的信息。酶催化机理数据库可以帮助修正公共数据库中的机器注释,也为对酶进一步的改造、设计提供了知识基础。
经过60余年的不断发展,该系统已经确定了7大类共7787种酶(表 1)。目前记录、更新由IUBMB-EC发布的酶分类与定义描述的数据库主要有以下几种:爱尔兰的ExplorEnz数据库记录了所有酶的名称(包括常用名、学名、常用同义名)、反应(包括底物、产物、辅因子),并在网页上提供了对酶的简单注释、连接到其他数据库的链接和文献参考[7];而欧洲生物信息学实验室(EMBL-EBI)的IntEnz数据库[8]和瑞士生物信息学研究所(SIB)的ENZYME数据库[9]中不仅列出了酶催化反应的化学式,还加入了经实验验证后的蛋白质序列信息。
表 1. 数据库中酶数据统计概览(统计至2020年12月) Table 1. Statistics of the enzyme database (status as of December 2020)
| Class | Top level EC number | Subclasses | Determined structure | Annotated sequence |
| Oxidoreductases | 1 | 2420 | 44818 | 3×106 |
| Transferases | 2 | 2107 | 101238 | 7×106 |
| Hydrolases | 3 | 1785 | 103554 | 4×106 |
| Lyases | 4 | 818 | 20517 | 2×106 |
| Isomerases | 5 | 326 | 10570 | 1×106 |
| Ligases | 6 | 241 | 6014 | 2×106 |
| Translocase | 7 | 90 | 10148 | 2×106 |
表选项
解析酶的催化机理是酶学研究的核心内容之一。EMBL-EBI的M-CSA数据库提供了数百种酶催化反应机理的注释[10],每个条目都有序列、晶体结构、InterPro数据库[11]和CATH数据库[12]的链接。这些注释分为两类,其中684条标记为“详细机理”(detailed mechanism)的条目以电子流箭头的形式展示了催化过程中的中间体,另有280条标记为“催化位点”(catalytic site)的条目记录了反应所需的催化残基,但未展示具体反应机理。日本数据库EzCatDB记录了879条酶催化的机理信息[13],和M-CAS直观展示催化机理不同,EzCatDB按催化机理对酶晶体结构进行了归类,并且衍生出了EzMetAct在线服务器,可以预测晶体结构中的催化位点。美国加州大学洛杉矶分校建立维护了SFLD数据库,专注于记录酶的结构-功能关系[14]。酶的分类通常依赖于催化反应的差异或序列相似度的差异,但化学反应/序列相似度的阈值难以界定,因此会导致机器错误注释[15]。SFLD根据相同的化学功能对进化相关的酶进行分类,并将功能映射到保守的活性位点,对每一类酶都给出了逐步反应机制,标注了重要功能残基的代表性序列比对(MSA)、隐谱马尔可夫模型(HMM)、蛋白质结构以及序列相似度网络。
2.2 综合性酶学数据库 生物学界多年的酶学研究积累了大量的酶分类、性质、应用和生理生化信息等数据,以这些数据为基础可构建更为全面的综合性酶学数据库。德国布伦瑞克工业大学创立的BRENDA数据库是目前最为全面的酶资源数据库[16],其核心信息是从原始文献中手动提取的,目前已经收录了超过137000篇文献、83000种酶的约300万条信息(表 2)。除了人工提取信息以外,该机构还在BRENDA的基础上利用文本挖掘程序搜索PubMed数据库中的标题和摘要,建立了AMENDA和FRENDA[17]两个扩展数据库。BRENDA数据库提供了强大的搜索功能,除了支持根据EC号、酶的名称、化合物名称进行搜索外,还支持小分子结构相似度查询,可以通过底物、产物或抑制剂的结构搜索相应的酶。BRENDA数据库提供了SOAP网络服务,允许注册用户使用Python等高级编程语言高效快速地获取大量数据。
表 2. BRENDA数据库部分数据统计(统计截至2020年更新) Table 2. Number of entries in selected data fields of BRENDA database (status as 2020 update)
| Enzyme information | Number of entries |
| Substrates and products | 471088 |
| Inhibitors | 226424 |
| Cofactors | 16954 |
| Metals and ions | 41985 |
| Activating compounds | 29025 |
| Km values | 154081 |
| Ki values | 42468 |
| kcat values | 75441 |
| Specific activity | 50238 |
| IC50 values | 60923 |
| Enzyme names and synonyms | 123077 |
| Citations (manually annotated) | 168008 |
| Isolation and preparation/crystallization | 107365 |
| Enzyme structure | 286714 |
| Mutant enzymes | 93590 |
| Enzyme stability | 52926 |
| Enzyme application | 18798 |
表选项
日本KEGG (Kyoto Encyclopedia of Genes and Genomes)下属的ENZYME数据库记录了各类酶经过人工校对的序列、催化的生化反应和所处代谢通路的数据[18]。中国科学院计算生物学重点实验室在ENZYME数据库[9]的酶学分类基础上增加了酶催化的化学反应的信息,构建了EnzyMine数据库[19],并且对化学反应特征进行了再次挖掘,进一步包括了反应中心、反应规则、“核心到核心”(core-to-core, C2C)分析,其中C2C分析可以显示酶反应中独特而复杂的底物骨架变化。美国的MetaCyc数据库在人工整理了超过58000篇文献的基础上,不仅提供了有关单个酶的大量数据,而且按照代谢通路,提供每一步反应和对应酶的EC号、代谢物结构式以及计算的吉布斯自由能[20]。MetaCyc数据库还衍生出EcoCyc数据库,按照大肠杆菌K-12菌株的代谢途径收录了相关的酶学信息[21]。这些数据库被广泛用于代谢通路的注释、改造和预测工作。
综合性酶学数据库内容非常全面,可以称得上是酶学百科全书,而且查询方便,外部链接丰富。利用这些数据库,科研人员可以快速了解包括某一个酶的特定性质、催化的反应类型以及在代谢通路中的功能等信息,再根据外部链接查找相关文献和其他信息。大量人工标注的数据也为开发基于深度学习的自然语言处理方法、人工智能辅助的酶工程和生物合成/降解路线设计提供了前提条件。
2.3 酶结构-功能家族数据库 由于某些大类的酶在各界生物中分布广泛、研究历史较长或已经产生了较高应用价值,在酶分类与机理数据库和综合数据库的基础上,衍生出了许多酶结构-功能家族数据库,这些数据库通常是按照序列、结构和功能的相似度来收录大型数据库中的部分条目。
法国的CAZy数据库是目前最权威的碳水化合物活性酶类数据库,收录的酶包括糖苷水解酶、糖基转移酶、多糖裂解酶、碳水化合物酯酶和氧化还原酶(辅助分解木质纤维素)[22]。碳水化合物结合模块(carbohydrate binding module,CBM)虽然不参与催化,但是对碳水化合物酶活性非常重要,而且具有自己独特的结构,所以也被CAZy数据库收录并归类。从CAZy数据库还衍生出了dbCAN-seq数据库,专门提供CAZy数据库中各个家族的序列和隐谱马尔可夫模型[23]用于基因组中的碳水化合物活性酶注释(表 3)。英国的MERPOS数据库收录了约4000条蛋白水解酶和近700条蛋白水解酶抑制蛋白信息,除了提供每一类的详细注释和参考文献,该数据库还提供在线的BLASTp搜索和序列比对用于蛋白酶及抑制蛋白的注释[24]。法国的β-内酰胺酶数据库(β-lactamases database)收录了1147个细菌来源的β-内酰胺酶,包含主要氨基酸序列、结构信息和四个类别的系统发育关系[25]。法国的ESTHER数据库根据结构分类收录α/β水解酶,目前包含超过30000种手动整理的蛋白质,分类为148个家族,罗列于数据库总表中,每个家族都有交互式的结构、突变体、底物、抑制剂等数据链接[26]。
表 3. 数据库部分数据统计(统计至2020年12月) Table 3. Statistics of CAZy database (status as December 2020)
| Enzyme class | Number of sequences | Number of families |
| Glycoside hydrolases | 886312 | 168 |
| Glycosyl transferases | 760421 | 111 |
| Polysaccharide lyases | 28387 | 40 |
| Carbohydrate esterases | 88130 | 18 |
| Auxiliary activities | 16200 | 16 |
| Carbohydrate-binding modules | 241705 | 88 |
表选项
这些酶结构-功能家族数据库方便了对特定酶类的搜索,对酶进一步的系统分类和进化研究提供了大量的数据,也使得基因组和宏基因组数据的注释更加快捷、准确,能够利用生物信息学方法快速搜索是否有某家族的同源基因来预测是否存在某种对应的生物学功能,甚至预测更加详细的特征性质。
2.4 酶应用分类数据库 酶已经被广泛用于工业、农业、食品、医疗和科研等领域,一些数据库按照应用的领域对不同功能和结构的酶进行了归纳。美国明尼苏达大学建立了UM-BBD数据库,包含近1200种化合物、超过800种酶、近1300种反应、近500种微生物和近200种途径。该数据库目前由瑞士联邦水科学与技术研究所维护并更名为EAWAG-BBD,提供在线的生物合成与降解途径预测[27]。日本科学家构建了MetaBioME数据库,收集了510种有商业价值的酶(commercially useful enzymes,CUEs),并把CUEs按照农业、生物传感、生物技术、能源、环境、食品与营养、医疗等领域分类,然后对971个完整细菌基因组和10个宏基因组数据进行了CUEs的预测[28]。印度的BioFuelDB数据库是在生物燃料生产中应用的酶数据库,通过文本挖掘,共鉴定了131种与生物燃料生产有关的酶,并将其分为乙醇生产、生物柴油生产、燃料电池和替代生物燃料4个应用类别[29]。基于BioFuelDB开发的预测工具Benz能够从基因组和宏基因组中预测生物燃料酶序列。BRENDA数据库的应用条目中也记录了酶应用相关的文献[17]。
中国科学院微生物研究所微生物资源与大数据中心牵头构建的生物资源衍生库(casbrc.org/ servicedata.jsp)中包含了工业反应酶库(图 3),通过人工收集文献信息结合数据挖掘方法,对目前重要酶资源进行系统性的整理、分析、研究、优化,建立标准数据条目,可以根据酶名称、反应等信息进行快速检索。该数据库对各种工业酶或有潜在应用的酶按照EC号分类,并在每个条目中补充了对酶的重要信息说明,包括酶的中英文名称、所属类别、来源物种、保存方法以及Rhea、KEGG和UniProt数据库的链接等信息。对于可催化多种反应的酶,数据库的条目中对每一个反应的底物、产物、反应式、反应中化学物质的分子式、CAS-ID、PubChemID和CHEBI都进行了详细的记录,并附有参考文献。目前数据库内包含了氧化还原酶、转移酶、水解酶、裂解酶、异构酶五大类的酶共356条数据,以细菌来源的酶占大多数(70.51%)。除了支持搜索功能,该数据库支持按酶的类别查看、按催化反应查看和按反应物查看三种查询方式,方便科研人员从需求出发快速查询数据库。该数据库的信息仍在不断更新中,数据库的新功能也在不断建设,将重点开发并提供不同类型宏基因组数据中基于生物信息学虚拟筛选出的新型工业酶序列信息,为快速筛选出优势生物酶催化剂、缩短新型酶制剂的研发周期、实现绿色高效的产品合成提供良好的信息平台。
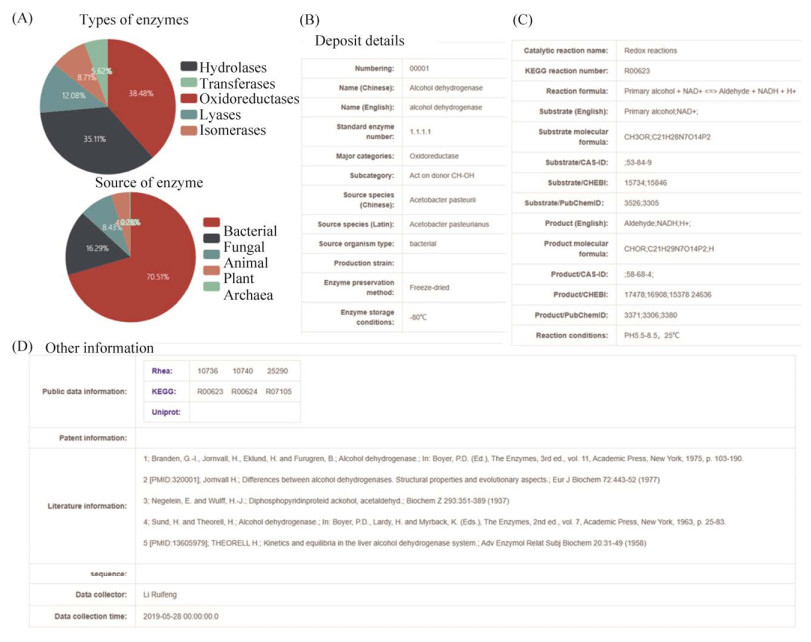 |
| 图 3 工业反应酶库 Figure 3 Web pages of Industrial reaction enzyme database. A: The types of enzymes currently recorded in the database and the proportion of enzyme source species. B: Examples of detailed information about the preservation of enzymes in data records, including number, name, classification, source species, etc. C: Examples of reaction information, including name, KEGG reaction number, reaction formula, etc. D: Examples of other information, including public database information and patent document information. |
| 图选项 |
微生物的次级代谢产物是抗生素和许多抗癌药物的重要来源,而这些复杂的天然产物合成是由复杂的酶系完成的。荷兰的MIBiG数据库通过文本挖掘,人工校正和重新注释整理了2036个次级代谢产物合成基因簇,其中包括了超过10000条参与次级代谢产物生物合成的酶[30]。丹麦的antiSMASH-DB数据库从6200个细菌基因组精细图和18576个细菌基因组草图中预测了152106个生物合成基因簇,包括了超过七十万条参与生物合成的酶[31]。
这些酶应用数据库记录的信息可以让科研人员快速查询到可以满足某些应用需求的酶,并了解相关酶的性质。利用数据库中已知序列信息进行基因组学数据挖掘,可以快速筛选出大量的候选序列,再通过高通量筛选即可获得针对特定应用类型的高质量实体微生物酶资源库。
2.5 酶学性质与改造数据库 在酶学科研工作中,通常需要测试或改变某些酶学性质,从而积累了大量的酶学实验数据,如酶的最适pH、最适温度、选择性、底物谱和动力学参数等。BRENDA数据库作为大型综合数据库,记录了大量的酶突变体的酶学性质,但是实验数据与序列和结构的联系不够紧密,难以直观地进行分析观察。
德国的SABIO-RK数据库是一个基于人工收集的数据库,其中包含有关酶催化的生化反应动力学速率方程以及参数和实验条件的信息[32]。美国的ProtaBank数据库记录了酶设计和改造实验,每一个项目条目内都有实验数据和对应的序列。ProtaBank的网页提供了许多在线交互式的可视化分析工具,帮助科研人员利用数据集理解酶的结构-功能关系。该数据库在开发人员完成了对酶设计改造实验的人工收集整理后向用户开放提交权限,鼓励用户上传酶工程实验数据,这种开源共享的方式大幅提高了数据库收集数据的速度和质量[33]。德国的BioCatNet数据库整合了细胞色素P450酶、糖苷水解酶、油酸水合酶、亚胺还原酶、漆酶、脂肪酶、ω-转氨酶、短链脱氢酶、硫胺素焦磷酸依赖性酶、TEM β-内酰胺酶和三萜环化酶共十一大类酶的序列、结构和酶工程改造信息[34](表 4)。BioCatNet的下属子库分别对酶按照序列、结构和功能关系进行了多等级的分类,便于进行同源性搜索和进化关系分析[35]。奥地利的MuteinDB数据库记录了底物,产物和酶促反应与酶的突变体之间的联系,以P450酶和腈水解酶为主,目前有22个野生型的491种突变,共3037种催化反应[36]。
表 4. BioCatNet下属各数据库序列与结构数量(统计至2020年12月) Table 4. Family-specific enzyme database under BioCatNet infrastructure (status as December 2020)
| Database name | Enzyme class | Number of sequences | Number of structures |
| CYPED | Cytochrome P450 | 52674 | 595 |
| GH19ED | Glycoside hydrolase 19 | 22461 | 27 |
| HYED | Hydratase | 2046 | 3 |
| IRED | Imine reductase | 1409 | 8 |
| LCCED | Laccase and multicopper oxidase | 51058 | 229 |
| LED | Lipase | 280638 | 1557 |
| oTAED | ω-transaminase | 114655 | 234 |
| SDRED | Short-chain dehydrogenase/reductase | 168212 | 688 |
| TEED | Thiamine diphosphate-dependent enzymes | 119567 | 308 |
| TEMLACED | TEM lactamase | 483 | 65 |
| TTCED | TriTerpene cyclase | 2794 | 18 |
表选项
酶学性质研究和酶工程对其应用的开发至关重要,收集整理这些实验数据并将其电子化对推动酶的应用具有重要意义。但是这些数据往往个性极强,现有算法难以对其进行系统的收集和提取,仍大量依赖人工提取与输入形成数据库。发表的数据往往是有较好结果的突变体测得的数据,许多实验数据未被发表,但并不代表没有价值,失败的设计和改造也能让科研人员进一步理解酶的结构-功能关系。ProtaBank和BioCatNet都鼓励用户主动上传实验数据,这种开源合作的方式促进了酶工程化发展,也使得一些阴性对照数据不至于史海钩沉。
3 基于数据库的微生物酶资源利用 伴随着微生物酶资源在电子数据库中的积累和生物信息学技术的发展,基于数据库挖掘的微生物酶资源利用已经初见成效。对于微生物酶数据资源的挖掘与利用主要分为两种模式,一是利用序列的来源、注释和进化关系等信息直接挖掘实体序列资源;二是利用序列资源中包含的氨基酸的保守性和共进化等信息,指导酶的改造与设计。
3.1 直接挖掘序列资源 目前可通过合适的生物信息学方法对序列数据库中的序列进行搜索和分析,直接挖掘实体序列资源,来获得有工业应用潜力的酶。中国科学院微生物研究所吴边课题组通过对GenBank数据库收录的全基因组数据利用序列相似度网络进行挖掘,鉴定了一株耐热、耐盐、耐有机溶剂的纤维素酶[37]。此外,还通过挖掘微生物转氨酶数据库,根据转氨酶底物选择性机理,利用序列中底物结合位点多态性对转氨酶分类后构建实体酶库,实现了天然氨基酸到酮酸的转化[38]。Turner课题组通过对宏基因组数据的高通量处理,获得了302个去冗余的亚胺还原酶序列,进一步加入了涵盖真菌、植物、动物、细菌的90个潜在亚胺还原酶序列。同时开发了高通量的测试方法,针对36种底物使用384孔板筛选,只有2个底物没有筛选到任何可催化的酶。筛选获得了300多种新型的亚胺还原酶,其中有22个具有较高的热稳定性[39]。Baker课题组为了建立新型一碳化合物利用途径,通过将BRENDA数据库中挖掘得到的能高效地将甲酰-CoA还原为甲醛的酰化醛脱氢酶(LmACDH),与大肠杆菌来源的乙酰-CoA合酶(EcACS)串联,打通了甲酸到甲醛的转化,最终实现利用甲酸合成磷酸二羟丙酮[40]。
3.2 利用数据资源指导酶改造与设计 除了对天然微生物酶资源的直接利用,研究人员还可以从数据库中提取信息指导酶的设计(图 4)。2020年,Ranganathan课题组从分支酸变位酶序列数据出发,使用直接耦合分析(direct coupling analysis,DCA)统计模型,该模型考虑了氨基酸位置的保守性以及氨基酸对(pairs)在进化中的相关性,设计了分支酸变位酶的新人工序列。实验证明了人工设计的分支酸变位酶显示出与天然酶相当的催化功能与活性[41]。吴边课题组在对降解PET塑料的IsPETase进行热稳定性计算再设计时,通过与耐热同源序列比对后设计了在耐热同源酶中的平行进化突变,其中最好的突变将蛋白质的表观熔融温度提高了8.5 ℃[42]。2021年,查尔姆斯理工大学的Zelezniak课题组发表了ProteinGAN,一种快速生成蛋白序列的生成式人工智能模型[43]。以目前数据库中的细菌苹果酸脱氢酶作为训练数据集,训练过程中使用了16706条序列,使用NVIDIA Tesla P100训练了9 d。合成的60条序列有19条成功表达,并具有可检测的苹果酸脱氢酶活性。在以AlphaFold2为代表的无模板结构预测方法中,借助积累的序列资源提取蛋白质的共进化信息,利用神经网络学习序列信息与结构信息之间的关系,实现了蛋白质结构的准确预测[44]。
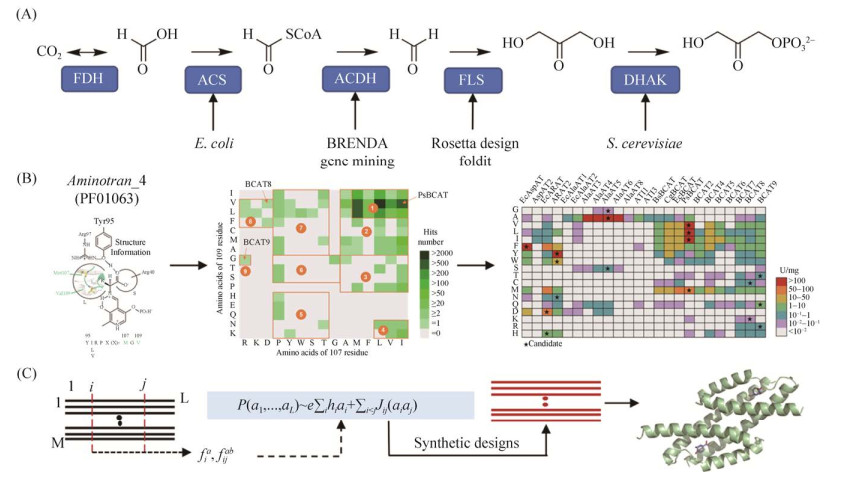 |
| 图 4 基于数据挖掘的微生物酶资源利用 Figure 4 Utilizations of microbial enzyme resources through data mining. A: De novo designed one-carbon assimilation pathway. B: Transaminases toolkit developed from enzyme database analysis. C: Statical approach for enzyme sequence design. |
| 图选项 |
众多的酶资源与利用的成功案例证实微生物酶资源数据库能够为代谢通路重构、新酶挖掘、新酶设计提供丰富的数据资源。如何从海量数据的数据库中精确锁定目标或抽取有用的信息,将序列结构信息和功能表型联系起来,是酶学和信息学未来交叉融合发展的一个重要方向。
4 总结和展望 酶资源是自然界馈赠人类的珍贵宝藏,上百年来数代科学家积累了宝贵的酶学知识和实验数据。自1913年米氏方程[45]被提出以来,酶学的研究已超过百年,奠定了当今众多工业酶在各个领域广泛应用的基础[46]。但时至今日,精确表征一个酶的性质仍然需要耗费大量的实验资源。随着基因测序技术的快速发展以及生物信息学与酶学的交流融合,酶资源数据库得到了快速发展,促进了酶的科学研究和应用,表 5列出了截止到2020年11月时部分酶学数据库的地址与简介。
表 5. 部分微生物酶资源数据库地址与简介 Table 5. Information about microbial enzyme databases
| Region | Database | Address | Theme | Reference |
| USA | ThYme | enzyme.cbirc.iastate.edu | Database for thioester-active enzymes | [47] |
| PlantCAZyme | cys.bios.niu.edu/plantcazyme | Database for plant carbohydrate-active enzymes | [48] | |
| MetaCyc | metacyc.org | Pathway information of enzymes | [20] | |
| EcoCyc | ecocyc.org | Pathway information of enzymes in E. coli K12 | [21] | |
| TECRDB | randr.nist.gov/enzyme/Default.aspx | Thermodynamics of enzyme-catalyzed reactions | [49] | |
| CASTLE | castle.cbe.iastate.edu | Carboxylic ester hydrolases | [50] | |
| REBASE | rebase.neb.com | Database for DNA restriction and modification | [51] | |
| SFLD | sfld.rbvi.ucsf.edu | Sequence-structure-function relation of enzymes | [14] | |
| LAHEDES | homingendonuclease.net | LAGLIDADG homing endonuclease database | [52] | |
| ProtaBank | protabank.org | Enzyme engineering data | [33] | |
| Europe | SDRED | sdred.biocatnet.de | The short-chain dehydrogenase/reductase engineering database | [53] |
| IntEnz | ebi.ac.uk/intenz | Enzyme classification and nomenclature | [8] | |
| ExplorEnz | enzyme-database.org | Enzyme classification and nomenclature | [7] | |
| ENZYME | expasy.org/enzyme | Enzyme classification and nomenclature | [9] | |
| M-CSA | ebi.ac.uk/thornton-srv/m-csa | Enzyme reaction mechanisms and active sites | [10] | |
| BRENDA | brenda-enzymes.org | Comprehensive enzyme information database | [16] | |
| MERPOS | ebi.ac.uk/merops | Database of proteolytic enzymes | [24] | |
| EAWAG-BBD | eawag-bbd.ethz.ch | Biocatalysis/Biodegradation database | [27] | |
| MIBiG 2.0 | mibig.secondarymetabolites.org | Biosynthetic gene clusters of known function | [30] | |
| antiSMASH-DB | antismash-db.secondarymetabolites.org | A comprehensive resource on secondary metabolite biosynthetic gene clusters | [31] | |
| SABIO-RK | sabio.h-its.org | Biochemical reaction kinetics | [32] | |
| BioCatNet | biocatnet.de | A repository of sequence, structure and biocatalytic data on protein families to facilitate protein engineering | [34] | |
| CYPED | cyped.biocatnet.de | Cytochrome P450 monooxygenases | [54] | |
| GH19ED | gh19ed.biocatnet.de | Glycoside hydrolase 19 engineering database | - | |
| HYED | hyed.biocatnet.de | Hydratase engineering database | - | |
| IRED | ired.biocatnet.de | imine reductase database | [55] | |
| LCCED | lcced.biocatnet.de | Laccase and multicopper oxidase engineering database | [56] | |
| LED | led.biocatnet.de | Lipase engineering database | [57] | |
| oTAED | otaed.biocatnet.de | ω-Transaminase engineering database | [58] | |
| TEED | teed.biocatnet.de | Thiamine diphosphate-dependent enzymes engineering database | [59] | |
| TEMLACED | temlaced.biocatnet.de | TEM lactamase database | [60] | |
| TTCED | ttced.biocatnet.de | Triterpene cyclase database | [61] | |
| Cofactor database | ebi.ac.uk/thornton-srv/databases/CoFactor | Organic cofactors in enzyme | [62] | |
| MuteinDB | muteindb.genome.tugraz.at | Substrates, products and enzymatic reactions linked with variants | [36] | |
| CAZy | cazy.org | Carbohydrate-active enzymes database | [22] | |
| beta-lactamase | ifr48.timone.univ-mrs.fr/beta-lactamase/ public | Database of β-lactamase enzymes | [25] | |
| ESTHER | bioweb.supagro.inra.fr/esther | Database of the α/β-hydrolase fold superfamily of proteins | [26] | |
| PLPMDB | studiofmp.com/plpmdb/index | Pyridoxal-5'-phosphate dependent enzymes | [63] | |
| China | UPObase | upobase.bioinformaticsreview.com | Database of unspecific peroxygenases | [64] |
| dbCAN-seq | bcb.unl.edu/dbCAN_seq | Database of carbohydrate-active enzyme (CAZyme) sequence and annotation | [23] | |
| EnzyMine | rxnfinder.org/enzymine | Database for enzyme function annotation with enzymatic reaction chemical feature | [19] | |
| IndRED | casbrc.org/servicedata.jsp | Industrial reaction enzyme database | - | |
| South Korea | FCPD | p450.riceblast.snu.ac.kr | Fungal cytochrome P450 database | [65] |
| India | BioFuelDB | metabiosys.iiserb.ac.in/biofueldb/index.html | Enzymes involved in biofuels production | [29] |
| Japan | KEGG ENZYME | genome.jp/kegg/annotation/enzyme.html | Enzyme and metabolism | [18] |
| EzCatDB | ezcatdb.cbrc.jp | Enzyme reaction database | [13] | |
| MetaBioME | metasystems.riken.jp/metabiome | Commercially useful enzymes in metagenomic data | [28] |
表选项
尽管已经取得相当程度的发展,酶资源数据库的构建仍然大量依赖人工,导致难以及时更新、后期维护成本高昂等问题,自动化的数据库构建维护方法成为了酶资源数据库发展所亟需的前沿技术。近期,英国Cronin课题组构建出人工智能平台,实现了计算机对有机合成文献的有效阅读,并将其转化为可操控机械臂的代码,自动进行化合物的全合成[66]。这一重大突破也为未来酶资源数据库自动构建、维护与应用提供了发展契机。将机器学习、自然语言处理与目前蓬勃发展的酶物理计算设计相互结合,发展新一代酶工程技术,促进微生物酶资源的挖掘与改造利用,将成为现代生物学领域前景广阔的发展方向。
References
| [1] | Kirk O, Borchert TV, Fuglsang CC. Industrial enzyme applications. Current Opinion in Biotechnology, 2002, 13(4): 345-351. DOI:10.1016/S0958-1669(02)00328-2 |
| [2] | Chien A, Edgar DB, Trela JM. Deoxyribonucleic acid polymerase from the extreme thermophile Thermus aquaticus. Journal of Bacteriology, 1976, 127(3): 1550-1557. DOI:10.1128/jb.127.3.1550-1557.1976 |
| [3] | Choi JM, Han SS, Kim HS. Industrial applications of enzyme biocatalysis: Current status and future aspects. Biotechnology Advances, 2015, 33(7): 1443-1454. DOI:10.1016/j.biotechadv.2015.02.014 |
| [4] | Bajaj P, Mahajan R. Cellulase and xylanase synergism in industrial biotechnology. Applied Microbiology and Biotechnology, 2019, 103(21/22): 8711-8724. |
| [5] | Savile CK, Janey JM, Mundorff EC, Moore JC, Tam S, Jarvis WR, Colbeck JC, Krebber A, Fleitz FJ, Brands J, Devine PN, Huisman GW, Hughes GJ. Biocatalytic asymmetric synthesis of chiral amines from ketones applied to sitagliptin manufacture. Science, 2010, 329(5989): 305-309. DOI:10.1126/science.1188934 |
| [6] | Webb EC. Enzyme Nomenclature 1992. Recommendations of the Nomenclature Committee of the International Union of Biochemistry and Molecular Biology on the Nomenclature and Classification of Enzymes. 6. London: Academic Press, 1992. |
| [7] | McDonald AG, Boyce S, Tipton KF. ExplorEnz: the primary source of the IUBMB enzyme list. Nucleic Acids Research, 2009, 37(suppl_1): D593-D597. |
| [8] | Fleischmann A, Darsow M, Degtyarenko K, Fleischmann W, Boyce S, Axelsen KB, Bairoch A, Schomburg D, Tipton KF, Apweiler R. IntEnz, the integrated relational enzyme database. Nucleic Acids Research, 2004, 32(suppl_1): D434-D437. |
| [9] | Bairoch A. The ENZYME database in 2000. Nucleic Acids Research, 2000, 28(1): 304-305. DOI:10.1093/nar/28.1.304 |
| [10] | Ribeiro AJM, Holliday GL, Furnham N, Tyzack JD, Ferris K, Thornton JM. Mechanism and Catalytic Site Atlas (M-CSA): a database of enzyme reaction mechanisms and active sites. Nucleic Acids Research, 2018, 46(D1): D618-D623. DOI:10.1093/nar/gkx1012 |
| [11] | Mitchell AL, Attwood TK, Babbitt PC, Blum M, Bork P, Bridge A, Brown SD, Chang HY, El-Gebali S, Fraser MI, Gough J, Haft DR, Huang HZ, Letunic I, Lopez R, Luciani A, Madeira F, Marchler-Bauer A, Mi HY, Natale DA, Necci M, Nuka G, Orengo C, Pandurangan AP, Paysan-Lafosse T, Pesseat S, Potter SC, Qureshi MA, Rawlings ND, Redaschi N, Richardson LJ, Rivoire C, Salazar GA, Sangrador-Vegas A, Sigrist CJA, Sillitoe I, Sutton GG, Thanki N, Thomas PD, Tosatto SCE, Yong SY, Finn RD. InterPro in 2019:improving coverage, classification and access to protein sequence annotations. Nucleic Acids Research, 2019, 47(D1): D351-D360. DOI:10.1093/nar/gky1100 |
| [12] | Sillitoe I, Dawson N, Lewis TE, Das S, Lees JG, Ashford P, Tolulope A, Scholes HM, Senatorov I, Bujan A, Ceballos Rodriguez-Conde F, Dowling B, Thornton J, Orengo CA. CATH: expanding the horizons of structure-based functional annotations for genome sequences. Nucleic Acids Research, 2019, 47(D1): D280-D284. DOI:10.1093/nar/gky1097 |
| [13] | Nagano N, Nakayama N, Ikeda K, Fukuie M, Yokota K, Doi T, Kato T, Tomii K. EzCatDB: the enzyme reaction database, 2015 update. Nucleic Acids Research, 2015, 43(D1): D453-D458. DOI:10.1093/nar/gku946 |
| [14] | Akiva E, Brown S, Almonacid DE, Barber AE, Custer AF, Hicks MA, Huang CC, Lauck F, Mashiyama ST, Meng EC, Mischel D, Morris JH, Ojha S, Schnoes AM, Stryke D, Yunes JM, Ferrin TE, Holliday GL, Babbitt PC. The structure-function linkage database. Nucleic Acids Research, 2014, 42(Database issue): D521-D530. |
| [15] | Schnoes AM, Brown SD, Dodevski I, Babbitt PC. Annotation error in public databases: misannotation of molecular function in enzyme superfamilies. PLoS Computational Biology, 2009, 5(12): e1000605. DOI:10.1371/journal.pcbi.1000605 |
| [16] | Schomburg I, Jeske L, Ulbrich M, Placzek S, Chang A, Schomburg D. The BRENDA enzyme information system-From a database to an expert system. Journal of Biotechnology, 2017, 261: 194-206. DOI:10.1016/j.jbiotec.2017.04.020 |
| [17] | Chang A, Scheer M, Grote A, Schomburg I, Schomburg D. BRENDA, AMENDA and FRENDA the enzyme information system: new content and tools in 2009. Nucleic Acids Research, 2009, 37(Database): D588-D92. DOI:10.1093/nar/gkn820 |
| [18] | Kanehisa M. Enzyme annotation and metabolic reconstruction using KEGG. Methods in Molecular Biology. New York, NY: Springer New York, 2017: 135-145. |
| [19] | Sun DD, Cheng XX, Tian Y, Ding SZ, Zhang DC, Cai PL, Hu QN. EnzyMine: a comprehensive database for enzyme function annotation with enzymatic reaction chemical feature. Database, 2020: baaa065. DOI:10.1093/database/baaa065 |
| [20] | Caspi R, Foerster H, Fulcher CA, Kaipa P, Krummenacker M, Latendresse M, Paley S, Rhee SY, Shearer AG, Tissier C, Walk TC, Zhang PF, Karp PD. The MetaCyc Database of metabolic pathways and enzymes and the BioCyc collection of Pathway/Genome Databases. Nucleic Acids Research, 2008, 36(Database issue): D623-D631. |
| [21] | Keseler IM, Mackie A, Santos-Zavaleta A, Billington R, Bonavides-Martínez C, Caspi R, Fulcher C, Gama-Castro S, Kothari A, Krummenacker M, Latendresse M, Mu?iz-Rascado L, Ong Q, Paley S, Peralta-Gil M, Subhraveti P, Velázquez-Ramírez DA, Weaver D, Collado-Vides J, Paulsen I, Karp PD. The EcoCyc database: reflecting new knowledge about Escherichia coli K-12. Nucleic Acids Research, 2017, 45(D1): D543-D550. DOI:10.1093/nar/gkw1003 |
| [22] | Lombard V, Golaconda Ramulu H, Drula E, Coutinho PM, Henrissat B. The carbohydrate-active enzymes database (CAZy) in 2013. Nucleic Acids Research, 2014, 42(D1): D490-D494. DOI:10.1093/nar/gkt1178 |
| [23] | Huang L, Zhang H, Wu PZ, Entwistle S, Li XQ, Yohe T, Yi HD, Yang ZL, Yin YB. dbCAN-seq: a database of carbohydrate-active enzyme (CAZyme) sequence and annotation. Nucleic Acids Research, 2018, 46(D1): D516-D521. DOI:10.1093/nar/gkx894 |
| [24] | Rawlings ND, Barrett AJ, Thomas PD, Huang XS, Bateman A, Finn RD. The MEROPS database of proteolytic enzymes, their substrates and inhibitors in 2017 and a comparison with peptidases in the PANTHER database. Nucleic Acids Research, 2018, 46(D1): D624-D632. DOI:10.1093/nar/gkx1134 |
| [25] | Keshri V, Diene SM, Estienne A, Dardaillon J, Chabrol O, Tichit L, Rolain JM, Raoult D, Pontarotti P. An integrative database of β-lactamase enzymes: sequences, structures, functions, and phylogenetic trees. Antimicrobial Agents and Chemotherapy, 2019, 63(5): e02319-18. |
| [26] | Lenfant N, Hotelier T, Velluet E, Bourne Y, Marchot P, Chatonnet A. ESTHER, the database of the α/β-hydrolase fold superfamily of proteins: tools to explore diversity of functions. Nucleic Acids Research, 2012, 41(D1): D423-D429. DOI:10.1093/nar/gks1154 |
| [27] | Gao JF, Ellis LBM, Wackett LP. The University of Minnesota Biocatalysis/Biodegradation Database: improving public access. Nucleic Acids Research, 2010, 38(suppl_1): D488-D491. DOI:10.1093/nar/gkp771 |
| [28] | Sharma VK, Kumar N, Prakash T, Taylor TD. MetaBioME: a database to explore commercially useful enzymes in metagenomic datasets. Nucleic Acids Research, 2010, 38(suppl_1): D468-D472. DOI:10.1093/nar/gkp1001 |
| [29] | Chaudhary N, Gupta A, Gupta S, Sharma VK. BioFuelDB: a database and prediction server of enzymes involved in biofuels production. PeerJ, 2017, 5: e3497. DOI:10.7717/peerj.3497 |
| [30] | Kautsar SA, Blin K, Shaw S, Navarro-Mu?oz JC, Terlouw BR, van der Hooft JJJ, van Santen JA, Tracanna V, Suarez Duran HG, Pascal Andreu V, Selem-Mojica N, Alanjary M, Robinson SL, Lund G, Epstein SC, Sisto AC, Charkoudian LK, Collemare J, Linington RG, Weber T, Medema MH. MIBiG 2.0:a repository for biosynthetic gene clusters of known function. Nucleic Acids Research, 2020, 48(D1): D454-D458. |
| [31] | Blin K, Pascal Andreu V, de los Santos ELC, Del Carratore F, Lee SY, Medema MH, Weber T. The antiSMASH database version 2:a comprehensive resource on secondary metabolite biosynthetic gene clusters. Nucleic Acids Research, 2019, 47(D1): D625-D630. DOI:10.1093/nar/gky1060 |
| [32] | Wittig U, Rey M, Weidemann A, Kania R, Müller W. SABIO-RK: an updated resource for manually curated biochemical reaction kinetics. Nucleic Acids Research, 2018, 46(D1): D656-D660. DOI:10.1093/nar/gkx1065 |
| [33] | Wang CY, Chang PM, Ary ML, Allen BD, Chica RA, Mayo SL, Olafson BD. ProtaBank: a repository for protein design and engineering data. Protein Science, 2018, 27(6): 1113-1124. DOI:10.1002/pro.3406 |
| [34] | Buchholz PCF, Vogel C, Reusch W, Pohl M, Rother D, Spie? AC, Pleiss J. BioCatNet: a database system for the integration of enzyme sequences and biocatalytic experiments. ChemBioChem, 2016, 17(21): 2093-2098. DOI:10.1002/cbic.201600462 |
| [35] | Buchholz PCF, Ohs R, Spiess AC, Pleiss J. Progress curve analysis within BioCatNet: comparing kinetic models for enzyme-catalyzed self-ligation. Biotechnology Journal, 2019, 14(3): 1800183. DOI:10.1002/biot.201800183 |
| [36] | Braun A, Halwachs B, Geier M, Weinhandl K, Guggemos M, Marienhagen J, Ruff AJ, Schwaneberg U, Rabin V, Torres Pazmi?o DE, Thallinger GG, Glieder A. MuteinDB: the mutein database linking substrates, products and enzymatic reactions directly with genetic variants of enzymes. Database, 2012, 2012(10.1093): database. |
| [37] | Zhu T, Li RF, Sun JY, Cui YL, Wu B. Characterization and efficient production of a thermostable, halostable and organic solvent-stable cellulase from an oil reservoir. International Journal of Biological Macromolecules, 2020, 159: 622-629. DOI:10.1016/j.ijbiomac.2020.05.021 |
| [38] | Li T, Cui XX, Cui YL, Sun JY, Chen YC, Zhu T, Li CJ, Li RF, Wu B. Exploration of transaminase diversity for the oxidative conversion of natural amino acids into 2-ketoacids and high-value chemicals. ACS Catalysis, 2020, 10(14): 7950-7957. DOI:10.1021/acscatal.0c01895 |
| [39] | Marshall JR, Yao PY, Montgomery SL, Finnigan JD, Thorpe TW, Palmer RB, Mangas-Sanchez J, Duncan RAM, Heath RS, Graham KM, Cook DJ, Charnock SJ, Turner NJ. Screening and characterization of a diverse panel of metagenomic imine reductases for biocatalytic reductive amination. Nature Chemistry, 2021, 13(2): 140-148. DOI:10.1038/s41557-020-00606-w |
| [40] | Siegel JB, Smith AL, Poust S, Wargacki AJ, Bar-Even A, Louw C, Shen BW, Eiben CB, Tran HM, Noor E, Gallaher JL, Bale J, Yoshikuni Y, Gelb MH, Keasling JD, Stoddard BL, Lidstrom ME, Baker D. Computational protein design enables a novel one-carbon assimilation pathway. Proceedings of the National Academy of the Sciences of the United States of America, 2015, 112(12): 3704-3709. |
| [41] | Russ WP, Figliuzzi M, Stocker C, Barrat-Charlaix P, Socolich M, Kast P, Hilvert D, Monasson R, Cocco S, Weigt M, Ranganathan R. An evolution-based model for designing chorismate mutase enzymes. Science, 2020, 369(6502): 440-445. DOI:10.1126/science.aba3304 |
| [42] | Cui Y, Chen Y, Liu X, Dong S, Tian Ye, Qiao Y, Mitra R, Han J, Li C, Han X, Liu W, Chen Q, Wei W, Wang X, Du W, Tang S, Xiang H, Liu H, Liang Y, Houk KN, Wu B. Computational redesign of a PETase for plastic biodegradation under ambient condition by the GRAPE strategy. ACS Catalysis, 2021, 11(3): 1340-1350. DOI:10.1021/acscatal.0c05126 |
| [43] | Repecka D, Jauniskis V, Karpus L, Rembeza E, Zrimec J, Poviloniene S, Rokaitis I, Laurynenas A, Abuajwa W, Savolainen O, Meskys R, Engqvist MKM, Zelezniak A. Expanding functional protein sequence space using generative adversarial networks. bioRxiv, 2019. DOI:10.1101/789719 |
| [44] | Senior AW, Evans R, Jumper J, Kirkpatrick J, Sifre L, Green T, Qin CL, ?ídek A, Nelson AWR, Bridgland A, Penedones H, Petersen S, Simonyan K, Crossan S, Kohli P, Jones DT, Silver D, Kavukcuoglu K, Hassabis D. Improved protein structure prediction using potentials from deep learning. Nature, 2020, 577(7792): 706-710. DOI:10.1038/s41586-019-1923-7 |
| [45] | Johnson KA, Goody RS. The original Michaelis constant: translation of the 1913 Michaelis-menten paper. Biochemistry, 2011, 50(39): 8264-8269. DOI:10.1021/bi201284u |
| [46] | Bornscheuer UT, Huisman GW, Kazlauskas RJ, Lutz S, Moore JC, Robins K. Engineering the third wave of biocatalysis. Nature, 2012, 485(7397): 185-194. DOI:10.1038/nature11117 |
| [47] | Cantu DC, Chen Y, Lemons ML, Reilly PJ. ThYme: a database for thioester-active enzymes. Nucleic Acids Research, 2011, 39(Database): D342-D346. DOI:10.1093/nar/gkq1072 |
| [48] | Ekstrom A, Taujale R, McGinn N, Yin YB. PlantCAZyme: a database for plant carbohydrate-active enzymes. Database, 2014, 2014(10.1093): database. |
| [49] | Goldberg RN, Tewari YB, Bhat TN. Thermodynamics of enzyme-catalyzed reactions-a database for quantitative biochemistry. Bioinformatics, 2004, 20(16): 2874-2877. DOI:10.1093/bioinformatics/bth314 |
| [50] | Chen YF, Black DS, Reilly PJ. Carboxylic ester hydrolases: Classification and database derived from their primary, secondary, and tertiary structures. Protein Science, 2016, 25(11): 1942-1953. DOI:10.1002/pro.3016 |
| [51] | Roberts RJ, Vincze T, Posfai J, Macelis D. REBASE-a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Research, 2015, 43(D1): D298-D299. DOI:10.1093/nar/gku1046 |
| [52] | Taylor GK, Petrucci LH, Lambert AR, Baxter SK, Jarjour J, Stoddard BL. LAHEDES: the LAGLIDADG homing endonuclease database and engineering server. Nucleic Acids Research, 2012, 40(W1): W110-W116. DOI:10.1093/nar/gks365 |
| [53] | Gr?ff M, Buchholz PCF, Stockinger P, Bommarius B, Bommarius AS, Pleiss J. The Short-chain Dehydrogenase/Reductase Engineering Database (SDRED): a classification and analysis system for a highly diverse enzyme family. Proteins: Structure, Function, and Bioinformatics, 2019, 87(6): 443-451. DOI:10.1002/prot.25666 |
| [54] | Gricman ?, Vogel C, Pleiss J. Identification of universal selectivity-determining positions in cytochrome P450 monooxygenases by systematic sequence-based literature mining. Proteins: Structure, Function, and Bioinformatics, 2015, 83(9): 1593-1603. DOI:10.1002/prot.24840 |
| [55] | Fademrecht S, Scheller PN, Nestl BM, Hauer B, Pleiss J. Identification of imine reductase-specific sequence motifs. Proteins: Structure, Function, and Bioinformatics, 2016, 84(5): 600-610. DOI:10.1002/prot.25008 |
| [56] | Sirim D, Wagner F, Wang L, Schmid RD, Pleiss J. The Laccase Engineering Database: a classification and analysis system for laccases and related multicopper oxidases. Database, 2011, 2011(10.1093): database. |
| [57] | Widmann M, Juhl PB, Pleiss J. Structural classification by the Lipase Engineering Database: a case study of Candida antarctica lipase A. BMC Genomics, 2010, 11: 123. DOI:10.1186/1471-2164-11-123 |
| [58] | Bu? O, Buchholz PCF, Gr?ff M, Klausmann P, Rudat J, Pleiss J. The ω-transaminase engineering database (oTAED): a navigation tool in protein sequence and structure space. Proteins: Structure, Function, and Bioinformatics, 2018, 86(5): 566-580. DOI:10.1002/prot.25477 |
| [59] | Vogel C, Pleiss J. The modular structure of ThDP-dependent enzymes. Proteins: Structure, Function, and Bioinformatics, 2014, 82(10): 2523-2537. DOI:10.1002/prot.24615 |
| [60] | Zeil C, Widmann M, Fademrecht S, Vogel C, Pleiss J. Network analysis of sequence-function relationships and exploration of sequence space of TEM β-lactamases. Antimicrobial Agents and Chemotherapy, 2016, 60(5): 2709-2717. DOI:10.1128/AAC.02930-15 |
| [61] | Racolta S, Juhl PB, Sirim D, Pleiss J. The triterpene cyclase protein family: a systematic analysis. Proteins: Structure, Function, and Bioinformatics, 2012, 80(8): 2009-2019. |
| [62] | Fischer JD, Holliday GL, Thornton JM. The CoFactor database: organic cofactors in enzyme catalysis. Bioinformatics, 2010, 26(19): 2496-2497. DOI:10.1093/bioinformatics/btq442 |
| [63] | Di Giovine P. PLPMDB: Pyridoxal-5'-phosphate dependent enzymes mutants database. Bioinformatics, 2004, 20(18): 3652-3653. DOI:10.1093/bioinformatics/bth399 |
| [64] | Faiza M, Lan DM, Huang SF, Wang YH. UPObase: an online database of unspecific peroxygenases. Database, 2019, 2019(10.1093): database. |
| [65] | Park J, Lee S, Choi J, Ahn K, Park B, Park J, Kang S, Lee YH. Fungal cytochrome P450 database. BMC Genomics, 2008, 9: 402. DOI:10.1186/1471-2164-9-402 |
| [66] | Mehr SHM, Craven M, Leonov AI, Keenan G, Cronin L. A universal system for digitization and automatic execution of the chemical synthesis literature. Science, 2020, 370(6512): 101-108. DOI:10.1126/science.abc2986 |
