徐欣韵1#, 王宁2,3#, 丁佳2, 陈妍3, 田光明1


1. 浙江大学环境与资源学院, 浙江 杭州 310000;
2. 中国农业大学资源与环境学院, 北京 100193;
3. 中国农业大学有机循环研究院(苏州), 江苏 苏州 215000
收稿日期:2021-01-15;修回日期:2021-04-22;网络出版日期:2021-08-05
基金项目:国家重点基础研究发展计划(2016YFD0800605)
*通信作者:田光明, Tel/Fax: +86-571-88982335; E-mail: gmtian@zju.edu.cn.
#共同第一作者
摘要:[目的] 从抑病型番茄根际土壤中筛选青枯病的高效拮抗促生菌,阐明其防病促生机制。[方法] 以番茄青枯雷尔氏菌(Ralstonia solanacearum)为靶病原菌,采用平板抑菌圈法,筛选拮抗菌; 通过BOX-PCR指纹图谱鉴定菌株多样性,以平板透明圈法评价其产酶活性,并针对抑菌能力强、产酶种类多的拮抗菌开展16S rRNA基因系统发育分析; 通过温室试验评价拮抗菌的防病促生能力,并在此基础上通过实时荧光定量PCR研究生防细菌对番茄青枯病的防病促生机制。[结果] 从番茄根际土壤分离获得29株细菌,其中15株对青枯菌具有拮抗功能,进一步通过BOX-PCR指纹图谱、酶活分析获得4株具有潜在防治番茄青枯病、促进生长的功能菌(B2、B5、B20、B23),通过16S rRNA系统发育分析鉴定B2拮抗菌为解淀粉芽孢杆菌(Bacillus amyloliquefaciens),B5和B20拮抗菌为枯草芽孢杆菌(Bacillus subtilis),B23拮抗菌为贝莱斯芽孢杆菌(Bacillus velezensis); 温室试验表明,B2、B5、B20、B23拮抗菌的抑病效果分别为35.59%、8.47%、32.20%、96.61%,并且均能显著增加番茄生物量和生理性状,如地上部鲜重、总叶绿素含量、地下部根尖数等。B2、B5、B23拮抗菌显著促进番茄株高和根长,B2、B20、B23拮抗菌显著增加茎粗; 而B23拮抗菌显著增加根系分叉数; 实时荧光定量分析表明,B2、B20、B23拮抗菌株可促进抗病相关功能基因PR1α、POD1的表达量,B2、B5、B23拮抗菌促进吲哚乙酸(IAA)信号通路应答关键基因ctd1的表达量,B2、B5、B20、B23拮抗菌均降低乙烯(ETH)信号通路应答关键基因ERF2的表达量。[结论] 本研究分离筛选获得4株对番茄青枯病具有显著防治效果以及促进番茄生长的PGPR菌株,可为定向筛选植物促生防病菌提供理论依据。
关键词:番茄青枯雷尔氏菌功能细菌防病促生长
Isolation and identification of antagonistic bacteria against tomato bacterial wilt and the mechanisms in disease prevention and plant growth promotion
Xinyun Xu1#, Ning Wang2,3#, Jia Ding2, Yan Chen3, Guangming Tian1


1. College of Environmental and Resources Sciences, Zhejiang University, Hangzhou 310000, Zhejiang Province, China;
2. College of Resources and Environmental Sciences, China Agricultural University, Beijing 100193, China;
3. Organic Recycling Research Institute(Suzhou) of China Agricultural University, Suzhou 215000, Jiangsu Province, China
Received: 15 January 2021; Revised: 22 April 2021; Published online: 5 August 2021
*Corresponding author: Guangming Tian, Tel/Fax: +86-571-88982335; E-mail: gmtian@zju.edu.cn.
Foundation item: Supported by the National Key Basic Research and Development Program of China (2016YFD0800605)
#These authors contributed equally to this work
Abstract: [Objective] Isolation plant growth promoting rhizobacteria (PGPR) from disease-resistant tomato rhizosphere soil to control bacterial wilt caused by Ralstonia solanacearum and improve tomato growth, and explored the mechanisms. [Methods] PGPR bacteria with antibacterial activity was screened from tomato rhizosphere soil by inhibition zone method using a moderately pathogenic Ralstonia solanacearum HN4 as target; The highly similar strains were identified by BOX-PCR fingerprints, and the enzyme-producing activity of different genotype strains was evaluated by the plate transparent circle method; The 16S rRNA phylogenetic analysis was applied to identify the antagonistic strains with strong antibacterial ability and many kinds of enzyme production; The potential antagonistic bacteria were assessed for plant disease prevention and growth promotion in greenhouse, and the plant disease prevention and growth promotion related genes expression level were tested by real-time PCR. [Results] 29 strains were isolated and purified from tomato rhizosphere soil, of which 15 strains had antagonistic function against Ralstonia solanacearum. Moreover, four strains involved in controlling tomato bacterial wilt and promoting growth, were verified by the analysis of BOX-PCR fingerprinting and enzyme activity. In the result of 16S rRNA phylogenetic analysis, strain B2 was identified as Bacillus amyloliquefaciens. Strain B5 and B20 were identified as Bacillus subtilis, respectively. Strain B23 was identified as Bacillus velezensis. In greenhouse experiment, strain B2, B5, B20 and B23 inhibited Ralstonia solanacearum effectively, with the efficiency of 35.59%, 8.47%, 32.20% and 96.61%, respectively. Furthermore, all of them significantly increased the plant fresh weight, the total chlorophyll content and the number of root tips. The strain B2, B5 and B23 efficiently promoted plant height and root length of tomato. The strain B2, B20 and B23 efficiently promoted stem thickness of tomato. And strain B23 efficiently promoted the number of root forks. In the result of real-time PCR, strain B2, B20 and B23 upregulated the expression of PR1α and POD1 gene. Strain B2, B5 and B23 upregulated the expression of ctd1 gene. And strain B2, B5, B20 and B23 downregulated the expression of ERF2 gene. [Conclusion] In this study, four biocontrol strains were isolated and identified to control tomato bacterial wilt and promote tomato growth, and can provide a theoretical basis for the targeted screening of plant growth-promoting and disease preventing bacteria.
Keywords: tomatoRalstonia solanacearumfunctional bacteriadisease preventionplant-growth promotion
番茄青枯病是由青枯雷尔氏菌(Ralstonia solanacearum)引起的病害,被认为是最具侵染力和破坏力的土传病害之一[1]。据估算,青枯病引起的经济损失高达30万/hm2,我国山东、新疆、内蒙古、河北、河南、云南、广西、江苏等地番茄青枯病发生严重,常常发生毁灭性灾害[2]。青枯雷尔氏菌其菌系复杂[3],寄主种类多样,广泛分布于热带、亚热带和某些温带地区[4],严重制约世界上番茄生产国家的果实产量和品质。目前番茄青枯病的防控策略主要包括:抗性番茄品种筛选,例如FL7514和BHN466品种[5-6],但通常以牺牲果实大小为代价[7],并且抗病育种过程复杂、耗时长,极大消耗时间成本;低毒化学农药喷施,往往造成土壤污染[8];嫁接方式,能够增强植物抗病能力,但是费时费力,我国采用嫁接技术栽培番茄的比例不足2%[9];植物源活性物质的应用,虽然具有易被自然微生物所分解、不污染环境、不易产生抗药性、专一性强、对有益生物群体无毒害作用等优点,但是绝大多数植物源抑菌剂产品处于试验研究阶段,生产工艺不成熟,不能形成规模化推广应用[10]。目前尚未有单一的物理化学方法能够完全有效地抑制番茄青枯病[11]。
关于番茄青枯病生物防治的报道已经有很多,主要集中在根际促生菌(plant growth promoting rhizobacteria,PGPR)作为拮抗微生物抵御病原菌的侵染[4]。其中芽孢杆菌(Bacillus spp.)广泛分布于空气、水、土壤等环境中,容易培养并经常用于植物病害的防治[12]。已知的生防芽孢杆菌包括短小芽孢杆菌(B. pumilus)、巨大芽孢杆菌(B. megaterium)、解淀粉芽孢杆菌(B. amyloliquefaciens)、蜡状芽孢杆菌(B. cereus)、枯草芽孢杆菌(B. subtilis)、多粘芽孢杆菌(B. polymyxa)等[13]。国外已有B. amyloliquefaciens FZB42和B. subtilis GB03、MB1600、QST713、A-13、RN14、NB22等菌株获得商品化生产许可,然而,国内仅有少数菌株获得商品化生产许可,如B. subtilis B916和B3等菌株[14],与国外同类研究相比仍有较大差距。发掘重要的青枯病拮抗促生微生物资源具有重要意义。
根际促生菌(PGPR)对植物抗病和生长能力具有调节作用[15],其抗病机制包括分泌抗菌物质、与病菌竞争生态位和营养物质、诱导抗性等,其中植物防御基因PR1α和抗病相关酶过氧化物酶(POD)的表达与植物信号传导途径的启动和抗病性有关[16]。促生机制包括分泌生长素(IAA)直接调控植物生长,转化土壤中难溶性磷转化为植物可吸收态,固定还原空气中N2形成NH4+,以及间接提高植物内源生长素(IAA)或者降低ETH水平等,从而促进植物生长[17]。目前对功能微生物菌种的筛选非常广泛,但是对其抗病和促生机理的探究不够全面。因此通过进一步检测微生物作用的植物抗病促生相关基因的表达,对明确筛选出的根际微生物菌种的功能是十分必要的。
本研究拟以番茄青枯雷尔氏菌(Ralstonia solanacearum)为靶病原菌,从番茄根围土壤中分离筛选拮抗细菌,测定拮抗菌对青枯菌的抑菌圈范围与其产酶活能力,并通过温室实验评估拮抗细菌对青枯病的防效以及测量番茄地上部、地下部多个生物指标评估其促生长能力,并且在基因层面进一步验证,可为后续定向筛选植物促生防病菌提供一定的研究思路。
1 材料和方法 1.1 供试材料与培养基 供试植株材料:温室盆栽实验番茄品种为上海合作903;供试病原菌Ralstonia solanacearum HN4,由南京农业大学植物保护学院生物防治及绿色植保实验室提供。
供试培养基材料:LB培养基、YGPA培养基参考标准的分子生物学方法配制;蛋白酶检测培养基(g/L):酪蛋白5.0,酵母膏2.5,葡萄糖1.0,脱脂奶粉100.0,琼脂18.0;纤维素酶培养基(g/L):蛋白胨10.0,酵母粉10.0,羧甲基纤维素钠10.0,NaCl 5.0,KH2PO4 1.0,琼脂20.0,pH 7.0;淀粉酶检测培养基(g/L):蛋白胨5.0,牛肉浸膏3.0,可溶性淀粉10.0,琼脂18.0。
细菌基因组DNA提取试剂盒、植物RNA提取试剂盒,采购于上海赛百盛基因技术有限公司;基因PR1α (Gene ID:X71592.1)、POD1 (Gene ID:KT834988)、ctd1 (Gene ID:543750)、ERF2 (Gene ID:543866)、SlActin (Gene ID:EU884309)序列引物由生工生物工程(上海)股份有限公司合成。
1.2 样本采集及菌株分离纯化 苏州市番茄种植面积一直稳定在2000多万平方米,近几年苏州雨水天气逐年增加,番茄青枯病有逐年增加的趋势,本实验采集苏州番茄青枯病发病果园中健康植株根际土壤,装入无菌样品袋,于实验室中4 ℃保存。称取1 g土壤添加到盛有10 mL无菌水的三角瓶中,室温180 r/min均匀振荡3 min后,将三角瓶置于75 ℃恒温水浴15 min,将土壤样品液经梯度稀释涂布分离[18],在30 ℃过夜培养24 h,挑取细菌菌落形态不同的单菌落进行划线纯化。细菌的分离、纯化均在LB培养基上进行。
1.3 拮抗细菌的分离筛选 参考Xue等[19]的研究方法进行番茄青枯菌拮抗细菌初筛,将分离得到的细菌在LB液体培养基中30 ℃、220 r/min过夜培养,获得拮抗菌液(菌含量为108 CFU/mL)。将青枯菌R. solanacearum HN4在YGPA液体培养基中30 ℃、220 r/min过夜培养,获得青枯病菌菌液(OD600=0.5)。将1.0 mL青枯菌HN4菌液与20 mL YGPA固体培养基(45-50 ℃)混合,充分摇匀后倒入直径为8 cm的培养皿中,制成含病原菌的平板,吹干冷却后,吸取5.0 μL拮抗菌液滴加到含病原菌的平板上,以无菌水作为对照,空气干燥10 min,将其培养在30 ℃培养箱中48 h,记录抑菌圈半径,所有处理重复3次。使用以下公式计算抑菌圈半径:抑菌圈半径=(透明抑制区的直径-拮抗板中细菌菌落的直径)/2。
1.4 BOX-PCR指纹图谱分析及聚类分析 采用细菌基因组提取试剂盒提取基因组DNA。以DNA为模板,BOXAIR (5′-CTACGGC AAGGCGACGCTGACG-3′)通用引物进行扩增。扩增体系为(25 μL):rTaq酶Mix 12.5 μL,BOXAIR (10 μmol/L) 1 μL,模板DNA 1 μL,ddH2O 10.5 μL。BOX-BOX-PCR扩增程序为:94 ℃ 7 min;94 ℃ l min,53 ℃ l min,65 ℃ 8 min,35个循环;65 ℃ 16 min,4 ℃维持。获得的PCR产物于-20 ℃保存。取BOX-PCR扩增产物5 μL,用1%的琼脂糖凝胶60 V电泳4 h。利用Quantity One (version 4.6.2)分析软件对电泳图谱进行分析,采用非加权配法(UPGMA)聚类分析并构建遗传分析树状图,取相似度大于等于0.8者为同一亚型,小于0.8者为不同的基因型[20]。
1.5 16S rRNA基因序列测定和分析 根据李生樟等[21]的方法,利用PCR扩增细菌的16S rRNA基因序列,正向引物27F:5′-AGAGTTTGATCATGGCTCAG-3′,反向引物1492R:5′-TACGGTTACCTTGTTACGACTT-3′,PCR反应体系及扩增条件参考傅本重等[22]的方法,将16S rRNA产物用1%的琼脂糖凝胶120 V电泳30 min检测后送往生工生物工程(上海)股份有限公司进行双端测序。将拼接好的16S rRNA基因测序结果在NCBI BLAST上与已知菌株序列进行相似性比对,查找并下载相似性最高的菌株16S rRNA序列,运用MEGA 6.0软件中ClustalW功能,采用Neighbor-Joining法Bootstrap 1000次,构建系统发育树。
1.6 拮抗菌种产酶活性测定 选取具有拮抗功能且不同基因型的细菌进行酶活测定,在LB液体培养基中30 ℃、220 r/min过夜培养(OD600=0.8),参考王超等[23]方法进行蛋白酶活、纤维素酶活、淀粉酶活性测定,30 ℃培养24 h后观察透明圈有无。对具有拮抗功能及产酶能力的菌株采用Venny 2.1在线软件(https://bioinfogp.cnb.csic.es/tools/venny)制作韦恩图,筛选多功能菌株进行后续试验。
1.7 番茄幼苗温室防效促生试验
1.7.1 供试菌株菌悬液及病原菌菌悬液的制备: 取保存于-70 ℃超低温冰箱中的功能菌株及青枯病菌HN4分别划线培养于LB及YGPA平板培养基上。在30 ℃下培养24 h后转接于相应5 mL试管培养液中,30 ℃、220 r/min培养24 h制成种子液。以1%的接种量接种在相应培养液中扩繁。30 ℃、220 r/min扩繁24 h后所得菌液用相应培养液进行稀释,将功能菌株浓度调至5×108 CFU/mL,HN4浓度调至5×107 CFU/mL,备用。
1.7.2 功能菌株对番茄青枯病的温室防治试验: 番茄种子催芽后在育苗盘中生长至四叶期,选取长势一致的番茄幼苗进行试验,每个处理选取24株幼苗并分为3个重复,每个重复8株幼苗,分别移栽至容积约为350 cm3的一次性塑料杯(基质土: 蛭石: 珍珠岩=3:1:1)中,移栽当天预接种功能菌液20 mL/杯,后接种青枯菌HN4菌液20 mL/杯,对照组浇灌等体积无菌水,将处理后的番茄苗置于温度为32 ℃、相对湿度为90%、光周期为16 h/ 8 h的温室中培育,每个处理位置随机摆放。病原菌处理后每天观察其生长状况和发病情况,21 d后统计最终结果,选取每个处理中具有代表性的15株番茄进行拍照统计。参照Wang等[24]的方法统计番茄发病情况,病害严重度分别按照式(1-2)和生物防效分别按照式(1-2)计算。
 | (公式1) |
 | (公式2) |
1.7.3 功能菌株对番茄幼苗促生长试验: 功能菌株处理番茄幼苗方法参考1.7.2,对照组以等体积无菌水处理,将处理后的番茄幼苗置于温度为28 ℃、相对湿度为50%、光周期为16 h/8 h的温室中培育,每天观察其生长情况,28 d后,参考刘陈晨[25]的方法对地上部鲜重、株高、茎粗、总叶绿素含量进行统计,同时用根系扫描仪(Epson Perfection V800 Photo)对根系进行扫描,利用LA-S系列(万深)植物图像分析系统对根系图像进行分析,统计地下部植物根长、根直径、根尖数和分叉数。每个处理重复测量3次。
1.8 实时荧光定量PCR分析番茄抗病防卫基因及生长相关基因的表达 在温室防治番茄青枯病试验及促生长试验第15天用总RNA提取试剂盒分别提取对照组及处理组番茄叶片的总RNA,反转录为cDNA,以适量的cDNA为模板进行荧光定量PCR反应[26]。在荧光定量PCR仪CFX 96上分析测定基因(病原相关蛋白PR1α基因、抗病相关酶过氧化物酶POD基因、吲哚乙酸(IAA)信号通路应答关键基因ctd1、乙烯(ETH)信号通路应答关键基因(ERF2)的表达情况[16-17]。以真核生物内保守表达的SlActin作为对照[27],采用ΔΔCT方法[26]进行试验数据处理,每个处理重复3次。
2 结果和分析 2.1 细菌拮抗活性及BOX-PCR分析 采用稀释涂布法根据形态不同从番茄根围土壤中共分离出29株细菌,以青枯雷尔氏菌HN4为靶病原菌,通过平板抑菌圈法,筛选出15株具有明显拮抗功能的细菌菌株,分别为B1、B2、B5、B6、B7、B10、B12、B15、B17、B18、B20、B23、B24、B25、B27号菌,抑菌圈半径均在0.5 mm以上(图 1-A和B)。采用细菌基因组提取试剂盒分别提取分离得到的29株细菌的基因组DNA,以BOXA1R为引物进行BOX-PCR扩增以反映菌株在染色体水平上的差异,扩增结果见图 1-C。利用Quantity One (version 4.6.2)分析软件中非加权配(UPGMA)法对细菌的BOX-PCR指纹图谱进行聚类分析(图 1-D),在80%的相似系数下,29株细菌可划分为24个组,其中组11包含了3株菌株,组1、7、13包含2株菌株,其他组中只包含1株菌株,具有拮抗功能的菌株主要分布在组1、10、11、13、14、15、16、17、20、21、22,根据拮抗菌株的抑菌圈半径大于0.5 mm并且菌株属于不同分组,选择B15、B18、B17、B6、B25、B24、B23、B20、B2、B5、B1菌株作为候选功能菌株。
 |
| 图 1 29株分离细菌拮抗青枯菌HN4的初筛结果(A-B),BOX-PCR指纹图谱分析(C)及UPGMA聚类分析(D) Figure 1 Antagonistic ability of 29 isolated bacteria against Ralstonia solanacearum HN4 (A), the inhibition zone radius in YGPA medium (B), BOX-PCR fingerprint analysis (C), and UPGMA cluster analysis (D). The vertical line on the scale 0.8 indicates that the strains with a similarity of less than 80% belong to different genera. Black dots indicate that the strain has antagonistic function and red dots indicate that the strains have antagonistic functions and belong to different groups. |
| 图选项 |
2.2 候选功能菌株产酶能力评估 对筛选获得的11株细菌进行蛋白酶、淀粉酶及纤维素酶活测定,以期获得可潜在应用于废弃物转化的候选菌株。结果表明B15号菌不具有产生这3种酶的能力,同时B1、B17和B24号菌不具有产生淀粉酶能力,其中B8号菌产蛋白酶能力最强,B6号菌产淀粉酶能力最强,B23号菌产纤维素酶能力最强(图 2-A-C)。结合11株细菌抑菌圈半径大于1.5 mm及其产酶种类,利用Venny 2.1在线软件制作韦恩图,结果显示菌株编号为B2、B5、B20、B23的细菌均产生这3种酶,并且抑菌圈直径大于1.5 mm (图 2-D),表明其生防潜能及酶活种类在11株候选细菌中相对较强,并将在温室条件下验证其对青枯病的防治能力。
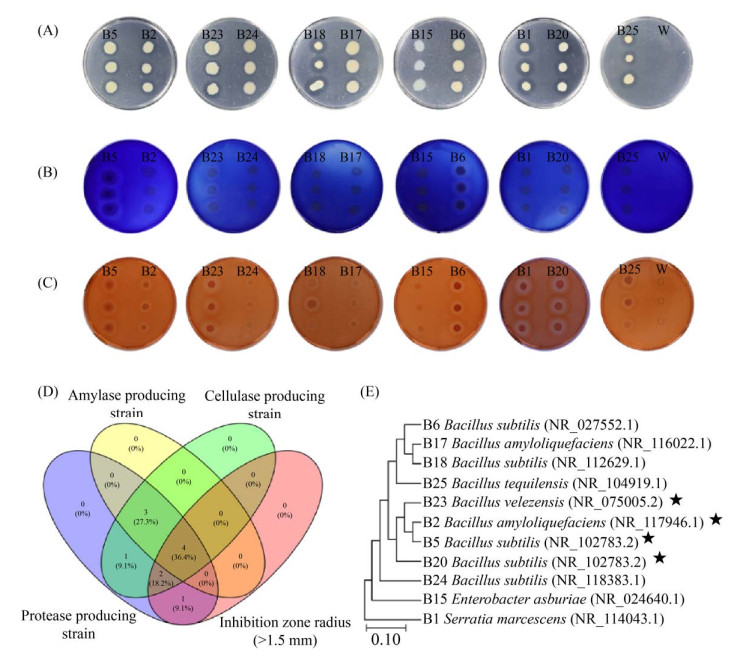 |
| 图 2 细菌产蛋白酶(A)、淀粉酶(B)、纤维素酶(C)活性及产酶种类分析(D),16S rRNA基因序列系统发育树分析(E) Figure 2 Analysis of the activity of protease (A), amylase (B) and cellulase (C) activity and enzyme types produced by selected 11 bacteria (D). And Neighbor-Joining phylogenetic trees based on 16S rRNA gene sequences of strains (E). |
| 图选项 |
2.3 16S rRNA序列的系统发育关系 对筛选获得的11株细菌提取基因组DNA,并将测得的16S rRNA序列提交到NCBI核酸数据库中,进行BLAST在线分析,根据序列相似性确定细菌种属。对11个测序结果采用Neighbor-Joining法Bootstrap 1000次,构建系统发育树(图 2-E)。聚类结果表明11株细菌中有5株为Bacillus subtilis,2株为Bacillus amyloliquefaciens,1株为Bacillus tequilensis,1株为Bacillus velezensis,1株为Enterobacter asburiae,1株为Serratia marcescens。其中B2号菌为Bacillus amyloliquefaciens,B5、B20号菌为Bacillus subtilis,B23号菌为Bacillus velezensis。
2.4 4株功能菌株对番茄青枯病的温室防效 生长至四叶期的番茄接种病原菌HN4后第7天对照组开始出现萎蔫症状,而4株功能菌株(B2、B5、B20、B23号)处理组中均在不同程度上推迟了番茄感病的时间,病原菌HN4接种21 d后进行了病害严重度统计分析,结果表明,4株功能菌株对番茄青枯病均有一定的防治效果(图 3-A-E),B2拮抗菌防效为35.59%,B5拮抗菌的防效为8.47%,B20拮抗菌的防效为32.20%,B23拮抗菌的防效为96.61% (图 3-F-G)。
 |
| 图 3 温室条件下B2、B5、B20、B23拮抗菌对番茄青枯病具有生防功能 Figure 3 Biological control effect of strain B2, B5, B20 and B23 on tomato bacterial wilt in greenhouse. Values with different letters within the same column differ significantly according to Duncan's test (P < 0.05). |
| 图选项 |
2.5 生防潜能较强的4株功能菌株的温室促生效果 筛选获得的4株生防潜能较强的菌株,在番茄地上部以及地下部均表现出一定的促生效果(图 4,图 5),4株菌株均能显著促进番茄地上部鲜重、总叶绿素含量(图 4-F和I)以及地下部根尖数(图 5-H)的增加,其中B2、B5、B23拮抗菌显著促进番茄的株高生长(图 4-G)以及地下部根长(图 5-F)的生长,B2、B20、B23拮抗菌显著促进番茄茎粗(图 4-H)的增加,B23拮抗菌能显著促进地下部根分叉数(图 5-I)的增加,但是4株菌株对根直径(图 5-G)没有显著的促进作用。
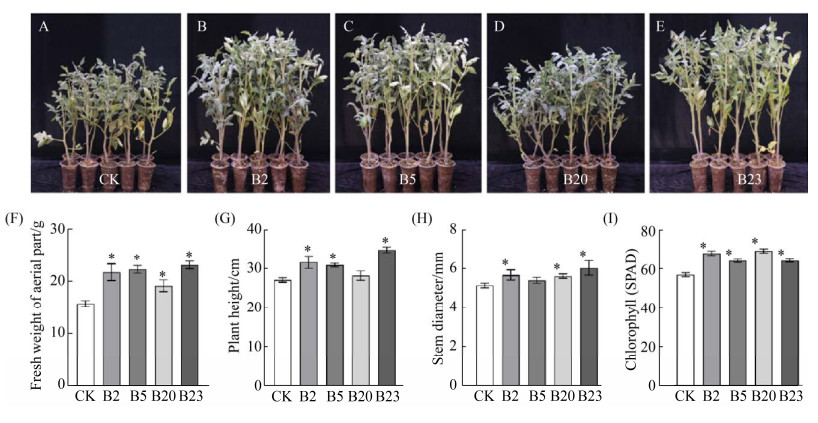 |
| 图 4 温室条件下B2、B5、B20、B23拮抗菌菌株对番茄地上部的促生效果 Figure 4 Plant growth-promoting effect of strain B2, B5, B20 and B23 on the aboveground part of tomato in greenhouse (28 d). Asterisks in (F-I) indicate significance difference between data of treatments and data of 4 strains as determined by the Student's t-test. ?: P < 0.05. |
| 图选项 |
 |
| 图 5 温室条件下B2、B5、B20、B23拮抗菌株对番茄地下部根系生长的影响 Figure 5 Effect of strain B2, B5, B20 and B23 on the root growth of tomato in greenhouse (28 d). Asterisks in (F-I) indicate significance difference between data of treatments and data of 4 strains as determined by the Student's t-test. ?: P < 0.05. |
| 图选项 |
2.6 4株功能菌株对番茄抗病防卫基因及生长相关基因表达的影响 采用实时荧光定量PCR分析4株功能菌株处理后番茄抗病防卫基因(PR1α、POD)及生长相关基因(ctd1、ERF2)的表达。B2、B20、B23拮抗菌处理后二次接种青枯病菌HN4的番茄病原相关蛋白PR1α基因与对照相比表达量分别提高了约2.6倍、5.5倍、3.0倍,抗病相关酶过氧化物酶POD基因与对照相比表达量分别提高了约2.3倍、4.8倍、1.9倍,B5拮抗菌处理后二次接种青枯病菌HN4后番茄的PR1α及POD基因表达量没有显著的变化(图 6-A和B);B2、B5、B23拮抗菌处理后的番茄中吲哚乙酸(IAA)信号通路关键基因ctd1与对照相比表达量分别提高了约3.1倍、3.5倍、3.4倍,B20拮抗菌处理后的番茄中ctd1基因没有表现出显著的变化(图 6-C);但4株功能菌株处理后番茄中乙烯(ethylene,ETH)信号通路关键基因ERF2均出现下调现象(图 6-D)。因此,B2和B23拮抗菌可能同时具有调控植物抗病及促进植物生长的功能;B5拮抗菌主要具有促进植物生长方面的功能;B20拮抗菌以调控植物抗病为主要功能兼具促进植物生长的能力。
 |
| 图 6 番茄抗病防卫基因PR1α (A)、POD (B)及生长相关基因ctd1 (C)、ERF2 (D)的表达情况 Figure 6 Transcript expression analysis of defense-related genes PR1α (A), POD (B) and growth-related genes ctd1 (C), ERF2 (D) in bacteria-treated and control (CK) tomato. Data are presented as mean of three independent biological replicates. Asterisks indicate statistically significant differences between different treatments. *: P < 0.05, Student's t-test. |
| 图选项 |
3 讨论 植物根际土壤中存在着大量的细菌,在提高植物抗病[23]及促进植物生长[17]、维持土壤微生态平衡[28]方面起着重要的作用。研究通过对细菌拮抗青枯菌能力大小、BOX-PCR聚类分析及产酶能力分析,从番茄根围土壤中分离筛选获得29株形态不同的细菌,结合韦恩图精确定位具有多功能的4株PGPR,并进一步通过温室防病促生长实验,阐述拮抗菌调控植物抗病促生长关键基因的表达从而探明番茄抗病和促生长的机理,为防治番茄青枯病筛选候选菌株及为后期应用于转化有机废弃物提供了研究思路和理论基础。
芽孢杆菌对环境友好,并且易于生产,是最具前景的生防资源[29]。有研究表明枯草芽孢杆菌具有很强的抑制青枯菌的能力[30],其防治效果最强可达80%,解淀粉芽孢杆菌QL-18[31]、BS2004[32]、SY290[33]、SQY162[34]对番茄青枯病防治的效果均在70%以上,同时枯草芽孢杆菌和解淀粉芽孢杆菌均能促进番茄植株生长[35-36]。目前贝莱斯芽孢杆菌在NCBI Taxonomy数据库中属于枯草芽胞杆菌群(Bacillus subtilis group)分类单元中解淀粉芽胞杆菌亚群(Bacillus amyloliquefaciens group)[37],主要在防治真菌性病害方面有一些研究报道,如宗英[38]筛选出1株贝莱斯芽孢杆菌JS25R,可高效抑制禾谷镰刀菌;杨胜清等[39]分离纯化了贝莱斯芽孢杆菌S6拮抗物质,能显著抑制番茄早疫病病原菌菌丝生长和孢子萌发;刘雪娇等[40]发现贝莱斯芽孢杆菌3A3-15具有5个抑菌基因和2个促生长基因,对尖孢镰刀菌孢子萌发抑制率达93.2%,并且具有较好的生防和促生潜力。但是贝莱斯芽孢杆菌在防治细菌性病害尤其在番茄青枯病方面未见有报道,本研究筛选获得4株生防菌均能一定程度地防治番茄青枯病,并且促进番茄植株的生长,其中贝莱斯芽孢杆菌(B23)的防青枯病效果尤其显著,防治高达96.61%,弥补了目前国内贝莱斯芽孢杆菌在防治番茄青枯病病害方面的空白。
高效、便捷的筛选体系对功能细菌的筛选是十分重要的,Berg等[41]建立了内生菌筛选评估体系,成功筛选出防治马铃薯病害的根际促生细菌,Yang等[42]利用生防菌筛选体系筛选出了用于高效防治生姜病害的细菌,谢越盛等[17]将赋值法应用于植物促生细菌筛选体系的建立,并在温室条件下得到了验证。本研究以平板拮抗活性初筛,用BOX-PCR指纹图谱区分高度相似菌株,并结合蛋白酶、纤维素酶、淀粉酶等作为降解有机废弃物中有机大分子体外评估指标进行复筛,筛选出4株具有潜在防治番茄青枯病、促进番茄生长且能高效降解有机大分子物质的菌株,通过温室实验以及在基因层面最终验证本研究所采用的筛选体系在防治番茄青枯病方面具有一定的可行性,后期应用于转化有机废弃物需要进一步实验。
前人研究发现植物防御反应病原相关蛋白(PR)基因的表达与调控植物信号传导和植物的抗病性相关[43],过氧化物酶POD酶是植物体内防御活性氧毒性的保护酶,在病害侵入过程中参与植物体内多种防卫反应,与植物的抗病性密切相关[43],其活性的高低可作为植物抗逆性的指标之一[44]。Niu等[45]发现Bacillus cereus AR156可激发拟南芥植株体内PR1、PR2、PR5等防卫相关基因的表达从而提高了植株抵御病原菌入侵的能力;生防芽孢杆菌能诱导植物抗病相关基因PR1、SOD、POD的显著上调表达[16];植物IAA、ETH等激素信号通路也受到PGPR菌的调控作用[46],其中低浓度IAA能促进植物生长发育,而ETH对植物的生长发育有一定的抑制作用[15, 17]。Ryu等[47]的研究表明Bacillus subtilis GB03可以调控植物细胞分裂素促进植物生长,谢越盛等[17]研究发现Bacillus subtilis JC01能够分泌促生效应的挥发性有机化合物(VOC),增强IAA信号水平,同时降低ETH信号水平,促进植物生长。本研究发现筛选获得的4株功能菌株中B1、B20、B23拮抗菌能显著提高防病相关基因的表达,表明其能增强植物的抗病能力;而B2、B5、B23拮抗菌能显著增强植物IAA关键基因ctd1的表达,促进番茄生长;B2、B5、B20、B23拮抗菌显著减弱植物ETH关键基因ERF2的表达,减弱ETH对番茄的抑制作用,同时表现出促进番茄生长。
本研究共筛选出4株具有防病促生长且降解大分子有机物质效果较好的功能菌株,研究同时发现相对平板拮抗活性较低的B23拮抗菌表现出显著防治番茄青枯病的能力。由于青枯病的生物防效与拮抗菌在根际定殖能力成正相关[48],我们推测B23拮抗菌能迅速定殖在植物根部并提高植物抵御青枯菌入侵的能力,并且需要综合更多作用机制来全面评估其作用机制。4株功能菌株应用于转化有机废弃物以及田间的应用效果尚需要进一步的试验验证,并且进一步探究复合菌剂的综合作用,解决目前单一菌种微生物菌剂效果弱、环境适应性差等问题,以期开发出可应用不同有机废弃物原料以及不同作物上均具有较好效果的复合微生物产品。
References
| [1] | Solke H, De B. Book review: Bacterial wilt disease and the Ralstonia solanacearum species complex. European Journal of Plant Pathology, 2006, 114: 227-228. DOI:10.1007/s10658-005-5039-6 |
| [2] | Mansfield J, Genin S, Magori S, Citovsky V, Sriariyanum M, Ronald P, Dow M, Verdier V, Beer SV, Machado MA, Toth I, Salmond G, Foster GD. Top 10 plant pathogenic bacteria in molecular plant pathology. Molecular Plant Pathology, 2012, 13(6): 614-629. DOI:10.1111/j.1364-3703.2012.00804.x |
| [3] | Kwak MJ, Kong HG, Choi K, Kwon SK, Song JY, Lee J, Lee PA, Choi SY, Seo M, Lee HJ, Jung EJ, Park H, Roy N, Kim H, Lee MM, Rubin EM, Lee SW, Kim JF. Rhizosphere microbiome structure alters to enable wilt resistance in tomato. Nature Biotechnology, 2018, 36(11): 1100-1109. DOI:10.1038/nbt.4232 |
| [4] | Chen Y, Yan F, Chai YR, Liu HX, Kolter R, Losick R, Guo JH. Biocontrol of tomato wilt disease by Bacillus subtilis isolates from natural environments depends on conserved genes mediating biofilm formation. Environmental Microbiology, 2013, 15(3): 848-864. DOI:10.1111/j.1462-2920.2012.02860.x |
| [5] | Ji P, Momol MT, Olson SM, Hong J, Pradhanang P, Anith KN, Jones JB. New tactics for bacterial wilt management on tomatoes in the southern us. Acta Horticulturae, 2005(695): 153-160. |
| [6] | Pradhanang PM, Ji P, Momol MT, Olson SM, Mayfield JL, Jones JB. Application of acibenzolar-S-methyl enhances host resistance in tomato against Ralstonia solanacearum. Plant Disease, 2005, 89(9): 989-993. DOI:10.1094/PD-89-0989 |
| [7] | Hanson PM, Licardo O, Hanudin, Wang JF, Chen JT. Diallel analysis of bacterial wilt resistance in tomato derived from different sources. Plant Disease, 1998, 82(1): 74-78. DOI:10.1094/PDIS.1998.82.1.74 |
| [8] | Sarkar L. Use of chemical pesticides and its environmental occurence in the soil, water and on health: a study on hansqua Tea estate area of west Bengal. Indian Journal of Environmental Protection, 2018, 38(10): 806-816. |
| [9] | Zhang YG, Yang XD, Wei JP, Wang LW, Liu SS, Zhou F. Domestic situation and development trend of grafted seedling of tomato. Horticulture & Seed, 2016, 36(9): 5-9. (in Chinese) 张元国, 杨晓东, 魏家鹏, 王林武, 刘树森, 周峰. 国内番茄嫁接育苗现状与发展趋势. 园艺与种苗, 2016, 36(9): 5-9. |
| [10] | Wang J, Long SF, Wang ZW, Chen JW, Jiang FY, Li X. Research progress in controlling tomato bacterial wilt. China Vegetables, 2020(1): 22-30. (in Chinese) 王杰, 龙世芳, 王正文, 谌金吾, 姜发洋, 李星. 番茄青枯病防治研究进展. 中国蔬菜, 2020(1): 22-30. |
| [11] | Yuliar, Nion YA, Toyota K. Recent trends in control methods for bacterial wilt diseases caused by Ralstonia solanacearum. Microbes and Environments, 2015, 30(1): 1-11. DOI:10.1264/jsme2.ME14144 |
| [12] | Cheng L, You CP, Xiao AP. Advance in the study on antagonistic bacteria. Acta Agriculturae Universitis Jiangxiensis, 2003, 25(5): 732-737. (in Chinese) 程亮, 游春平, 肖爱萍. 拮抗细菌的研究进展. 江西农业大学学报, 2003, 25(5): 732-737. DOI:10.3969/j.issn.1000-2286.2003.05.020 |
| [13] | Peng Y, Chen XY, Qiu JY, Yang G, Wang WM, Chen LL. Research progress of biocontrol Bacillus. Shandong Agricultural Sciences, 2013, 45(7): 138-140. (in Chinese) 彭研, 陈相艳, 裘纪莹, 杨革, 王未名, 陈蕾蕾. 生防芽孢杆菌的研究进展. 山东农业科学, 2013, 45(7): 138-140. DOI:10.3969/j.issn.1001-4942.2013.07.042 |
| [14] | Chen ZY, Liu YF, Liu YZ, Zhang RS. Research progress in biocontrol of Bacillus spp. against plant diseases. Jiangsu Journal of Agricultural Sciences, 2012, 28(5): 999-1006. (in Chinese) 陈志谊, 刘永峰, 刘邮洲, 张荣胜. 植物病害生防芽孢杆菌研究进展. 江苏农业学报, 2012, 28(5): 999-1006. |
| [15] | Bal HB, Das S, Dangar TK, Adhya TK. ACC deaminase and IAA producing growth promoting bacteria from the rhizosphere soil of tropical rice plants. Journal of Basic Microbiology, 2013, 53(12): 972-984. DOI:10.1002/jobm.201200445 |
| [16] | Wang LY, Jiang PP, Gan Y, Chen ZY, Liu YF. Increase of defense enzyme activity and up-expression of defense gene in tomato induced by Bacillus amyloliquefaciens B1619. Chinese Journal of Biological Control, 2017, 33(2): 234-240. (in Chinese) 王璐瑶, 蒋盼盼, 甘颖, 陈志谊, 刘永峰. 解淀粉芽胞杆菌B1619诱导番茄防御酶活性提高和抗病相关基因的表达上调. 中国生物防治学报, 2017, 33(2): 234-240. |
| [17] | Xie YS, Xu Q, Wang DC, Wang N, Wang C, Jiang CH, Guo JH. Screening of plant growth-promoting Bacillus subtilis JC01 producing volatile organic compound. Microbiology China, 2016, 43(10): 2187-2196. (in Chinese) 谢越盛, 许泉, 王大成, 王宁, 王超, 蒋春号, 郭坚华. 植物根际促生枯草芽孢杆菌JC01筛选. 微生物学通报, 2016, 43(10): 2187-2196. |
| [18] | Wang CX, Zhang QM, Li GF, Dong XL, Li BH. Identification of the antagonistic bacteria BJ1 and its antifungal activity against Valsa ceratosperma. Acta Phytophylacica Sinica, 2012, 39(5): 431-437. (in Chinese) 王彩霞, 张清明, 李桂舫, 董向丽, 李保华. 苹果树腐烂病拮抗细菌菌株BJ1的鉴定及其抑菌作用. 植物保护学报, 2012, 39(5): 431-437. |
| [19] | Xue QY, Chen Y, Li SM, Chen LF, Ding GC, Guo DW, Guo JH. Evaluation of the strains of Acinetobacter and Enterobacter as potential biocontrol agents against Ralstonia wilt of tomato. Biological Control, 2009, 48(3): 252-258. DOI:10.1016/j.biocontrol.2008.11.004 |
| [20] | Chen Z, Wang QF, Yang H, Lang BT, Yang JS, Li J, Liu Y, Zhang J, Chen CP, Chen Q. Fungal communities diversity in salviae miltiorrhizae planting rhizospheric and non-rhizospheric soil analyzed by PCR-DGGE in Zhongjiang, Sichuan Province. Southwest China Journal of Agricultural Sciences, 2014, 27(5): 2040-2044. (in Chinese) 陈章, 王强锋, 杨红, 郎伯涛, 杨季声, 李洁, 刘燕, 张俊, 陈翠平, 陈强. 四川中江丹参根际和非根际土壤真菌种群多样性PCR-DGGE分析. 西南农业学报, 2014, 27(5): 2040-2044. DOI:10.3969/j.issn.1001-4829.2014.05.046 |
| [21] | Li SZ, Chen Y, Yang RH, Zhang CP, Liu Z, Li YL, Chen T, Chen GY, Zou LF. Isolation and identification of a Bacillus velezensis strain against plant pathogenic Xanthomonas spp... Acta Microbiologica Sinica, 2019, 59(10): 1969-1983. (in Chinese) 李生樟, 陈颖, 杨瑞环, 张翠萍, 刘昭, 李逸朗, 陈涛, 陈功友, 邹丽芳. 一株拮抗黄单胞菌的贝莱斯芽孢杆菌的分离和鉴定. 微生物学报, 2019, 59(10): 1969-1983. |
| [22] | Fu BZ, Chen QQ, Wei M, Zhu JQ, Yang XH, Li GY, Zou LP, Wang DB. Investigation of walnut bacterial blight pathogens based on 16S-rDNA sequences and RFLP. Journal of Agricultural University of Hebei, 2016, 39(5): 63-68. (in Chinese) 傅本重, 陈倩倩, 魏蜜, 朱洁倩, 杨新河, 李国元, 邹礼平, 汪殿蓓. 基于16S rDNA序列的核桃细菌性疫病病原菌遗传多态性分析. 河北农业大学学报, 2016, 39(5): 63-68. |
| [23] | Wang C, Shen CM, Zheng L, Xue QY, Guo JH. Isolation and screening of antagonistic bacterial strains and their biocontrol efficiency against bacterial wilt. Plant Protection, 2014, 40(2): 43-47, 69. (in Chinese) 王超, 申成美, 郑丽, 薛庆云, 郭坚华. 烟草青枯病生防细菌的筛选与生防效果研究. 植物保护, 2014, 40(2): 43-47, 69. DOI:10.3969/j.issn.0529-1542.2014.02.008 |
| [24] | Wang N, Wang LY, Zhu K, Hou SS, Chen L, Mi DD, Gui Y, Qi YJ, Jiang CH, Guo JH. Plant root exudates are involved in Bacillus cereus AR156 mediated biocontrol against Ralstonia solanacearum. Frontiers in Microbiology, 2019, 10: 98. DOI:10.3389/fmicb.2019.00098 |
| [25] | Liu CC, Ren SW, Wang N, Hu ZP. Effect of water-soluble fertilizer on the growth of tomato and the root-knot nematode by adding BRs. Heilongjiang Agricultural Sciences, 2017(9): 55-58. (in Chinese) 刘陈晨, 任士伟, 王娜, 胡兆平. 水溶肥添加油菜素内酯对番茄促生及抗根结线虫病的效果. 黑龙江农业科学, 2017(9): 55-58. |
| [26] | Wang C, Wang C, Gao YL, Wang YP, Guo JH. A consortium of three plant growth-promoting rhizobacterium strains acclimates Lycopersicon esculentum and confers a better tolerance to chilling stress. Journal of Plant Growth Regulation, 2016, 35(1): 54-64. DOI:10.1007/s00344-015-9506-9 |
| [27] | Fuentes A, Ortiz J, Saavedra N, Salazar LA, Meneses C, Arriagada C. Reference gene selection for quantitative real-time PCR in Solanum lycopersicum L. inoculated with the mycorrhizal fungus Rhizophagus irregularis. Plant Physiology and Biochemistry, 2016, 101: 124-131. DOI:10.1016/j.plaphy.2016.01.022 |
| [28] | Gu SH, Wei Z, Shao ZY, Friman VP, Cao KH, Yang TJ, Kramer J, Wang XF, Li M, Mei XL, Xu YC, Shen QR, Kümmerli R, Jousset A. Competition for iron drives phytopathogen control by natural rhizosphere microbiomes. Nature Microbiology, 2020, 5(8): 1002-1010. DOI:10.1038/s41564-020-0719-8 |
| [29] | Liu F, Liao XQ, Zhou RH, Rao B, Chen W, Huang DY, You JM. Optimization of fermentation mediaum and conditions for Bacillus subtilis BZJN1. Hubei Agricultural Sciences, 2019, 58(20): 93-96. (in Chinese) 刘芳, 廖先清, 周荣华, 饶犇, 陈伟, 黄大野, 游景茂. 枯草芽孢杆菌BZJN1发酵培养基和培养条件的优化. 湖北农业科学, 2019, 58(20): 93-96. |
| [30] | Algam S, Xie GL, Li B, Coosemans J, Liu B. Comparative performance of Bacillus spp.in growth promotion and suppression of tomato bacterial wilt caused by Ralstonia solanacearum. Journal of Zhejiang University (Agriculture and Life Sciences), 2004, 30(6): 603-610. |
| [31] | Wei Z, Yang XM, Yin SX, Shen QR, Ran W, Xu YC. Efficacy of Bacillus-fortified organic fertiliser in controlling bacterial wilt of tomato in the field. Applied Soil Ecology, 2011, 48(2): 152-159. DOI:10.1016/j.apsoil.2011.03.013 |
| [32] | Zhou Q, Wang X, Su T, Lu M, Xie GL. Optimization of medium and cultural conditions for biocontrol bacterial strain BS2004 against Ralstonia solanacearum and Plasmodiophora brassicae. Chinese Journal of Biological Control, 2012, 28(4): 537-544. (in Chinese) 周青, 王笑, 苏婷, 路梅, 谢关林. 青枯病和根肿病生防细菌BS2004的发酵培养基和培养条件优化. 中国生物防治学报, 2012, 28(4): 537-544. DOI:10.3969/j.issn.2095-039X.2012.04.015 |
| [33] | Zhang B, Qiao JQ, Liang XJ, Liu YZ, Chen ZY. Evaluation of antagonistic bacteria against Fusarium oxysporum f. sp. lycopersici and Ralstonia solanacearum. Journal of Plant Protection, 2015, 42(3): 353-361. (in Chinese) 张斌, 乔俊卿, 梁雪杰, 刘邮洲, 陈志谊. 番茄枯萎病菌和青枯病菌拮抗细菌的评价. 植物保护学报, 2015, 42(3): 353-361. |
| [34] | Wu K, Su L, Fang ZY, Yuan SF, Wang LL, Shen B, Shen QR. Competitive use of root exudates by Bacillus amyloliquefaciens with Ralstonia solanacearum decreases the pathogenic population density and effectively controls tomato bacterial wilt. Scientia Horticulturae, 2017, 218: 132-138. DOI:10.1016/j.scienta.2017.01.047 |
| [35] | Qiao JQ, Chen ZY, Liang XJ, Liu YF, Liu YZ. Biocontrol efficacy on tomato bacterial wilt by Bacillus subtilis Bs916. Chinese Journal of Biological Control, 2016, 32(2): 229-234. (in Chinese) 乔俊卿, 陈志谊, 梁雪杰, 刘永锋, 刘邮洲. 枯草芽孢杆菌Bs916防治番茄青枯病. 中国生物防治学报, 2016, 32(2): 229-234. |
| [36] | Zhao WS, Guo QG, Yu WQ, Wang PP, Su ZH, Zhang XY, Lu XY, Ma P, Li SZ. Phosphate-solubilizing characteristics of Bacillus amyloliquefaciens PHODB35 and its growth-promoting effect on tomato. Acta Microbiologica Sinica, 2020, 60(7): 1370-1383. (in Chinese) 赵卫松, 郭庆港, 于稳欠, 王培培, 苏振贺, 张晓云, 鹿秀云, 马平, 李社增. 解淀粉芽胞杆菌PHODB35的溶磷特性及其对番茄的促生作用. 微生物学报, 2020, 60(7): 1370-1383. |
| [37] | Zhang CW, Cheng K, Zhang X, Liu B, Du HB, Yao S. Taxonomy and functions of Bacillus velezensis: a review. Food and Fermentation Industries, 2019, 45(17): 258-265. (in Chinese) 张彩文, 程坤, 张欣, 刘波, 都海渤, 姚粟. 贝莱斯芽胞杆菌(Bacillus velezensis)分类学及功能研究进展. 食品与发酵工业, 2019, 45(17): 258-265. |
| [38] | Zong Y, Zhao YJ, Liu Y, Yang QL. Study on the inhibitory effect of Bacillus velezensis on Fusarium graminearum. Journal of Nuclear Agricultural Sciences, 2018, 32(2): 310-317. (in Chinese) 宗英, 赵月菊, 刘阳, 杨庆利. 一株贝莱斯芽孢杆菌抑制禾谷镰刀菌的研究. 核农学报, 2018, 32(2): 310-317. |
| [39] | Yang SQ, Zhang F, Ma GL. Purification and inhibitive mechanism of antagonist substances from Bacillus velezensis S6 strain. Agrochemicals, 2017, 56(9): 645-648, 660. (in Chinese) 杨胜清, 张帆, 马贵龙. 贝莱斯芽孢杆菌S6拮抗物质分离纯化及抑菌机理. 农药, 2017, 56(9): 645-648, 660. |
| [40] | Liu XJ, Li HY, Li SN, Zhu BC, Gao TG. Biocontrol and growth promotion mechanisms of Bacillus velezensis 3A3-15. Journal of Hebei University: Natural Science Edition, 2019, 39(3): 302-310. (in Chinese) 刘雪娇, 李红亚, 李术娜, 朱宝成, 高同国. 贝莱斯芽孢杆菌3A3-15生防和促生机制. 河北大学学报: 自然科学版, 2019, 39(3): 302-310. DOI:10.3969/j.issn.1000-1565.2019.03.012 |
| [41] | Berg G, Krechel A, Ditz M, Sikora RA, Ulrich A, Hallmann J. Endophytic and ectophytic potato-associated bacterial communities differ in structure and antagonistic function against plant pathogenic fungi. FEMS Microbiology Ecology, 2005, 51(2): 215-229. DOI:10.1016/j.femsec.2004.08.006 |
| [42] | Yang JH, Liu HX, Zhu GM, Pan YL, Xu LP, Guo JH. Diversity analysis of antagonists from rice-associated bacteria and their application in biocontrol of rice diseases. Journal of Applied Microbiology, 2008, 104(1): 91-104. |
| [43] | Van Loon LC, Rep M, Pieterse CMJ. Significance of inducible defense-related proteins in infected plants. Annual Review of Phytopathology, 2006, 44: 135-162. DOI:10.1146/annurev.phyto.44.070505.143425 |
| [44] | Thoma I, Loeffler C, Sinha AK, Gupta M, Krischke M, Steffan B, Roitsch T, Mueller MJ. Cyclopentenone isoprostanes induced by reactive oxygen species trigger defense gene activation and phytoalexin accumulation in plants. The Plant Journal, 2003, 34(3): 363-375. DOI:10.1046/j.1365-313X.2003.01730.x |
| [45] | Niu DD, Wang CJ, Guo YH, Jiang CH, Zhang WZ, Wang YP, Guo JH. The plant growth-promoting rhizobacterium Bacillus cereus AR156 induces resistance in tomato with induction and priming of defence response. Biocontrol Science and Technology, 2012, 22(9): 991-1004. DOI:10.1080/09583157.2012.706595 |
| [46] | Huang XF, Zhou D, Guo J, Manter DK, Reardon KF, Vivanco JM. Bacillus spp. from rainforest soil promote plant growth under limited nitrogen conditions. Journal of Applied Microbiology, 2015, 118(3): 672-684. DOI:10.1111/jam.12720 |
| [47] | Ryu CM, Farag, Mohamed A, Hu CH, Reddy, Munagala S, Wei HX, Paré, Paul W, Kloepper. Bacterial volatiles promote growth in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(8): 4927-4932. DOI:10.1073/pnas.0730845100 |
| [48] | Huang JF, Wei Z, Tan SY, Mei XL, Yin SX, Shen QR, Xu YC. The rhizosphere soil of diseased tomato plants as a source for novel microorganisms to control bacterial wilt. Applied Soil Ecology, 2013, 72: 79-84. DOI:10.1016/j.apsoil.2013.05.017 |
