任凯, 郭强, 徐欣


口腔疾病研究国家重点实验室, 国家口腔疾病临床医学研究中心, 四川大学华西口腔医院牙体牙髓病科, 四川 成都 610041
收稿日期:2020-09-16;修回日期:2020-12-07;网络出版日期:2021-01-28
基金项目:国家自然科学基金(81500842)
*通信作者:徐欣, Tel: +86-28-85503462;E-mail: xin.xu@scu.edu.cn.
摘要:链球菌是人类口腔中最为常见的细菌类群之一,在口腔微生态平衡的维持与致病中发挥了重要作用。口腔链球菌中的大多数可以进入感受态,在此生理状态下,细菌可摄取环境中的DNA并整合进入自身基因组从而获得新的遗传表型或特性。大量研究表明,口腔链球菌的感受态调控通路不是孤立的,与生物膜形成、细菌素产生、耐酸、氧应激、细胞自溶和耐药性等多个表型的调控存在紧密关系,研究这些不同表型间的相互影响对理解口腔菌群稳态及防治疾病有重要意义。本文以变异链球菌、格氏链球菌、血链球菌和肺炎链球菌4种典型的口腔链球菌为代表,对感受态与口腔链球菌多种表型间关系的研究进展做一综述。
关键词:口腔链球菌感受态微生态平衡龋病生物膜
Research progress on the relationship between competence and phenotypic traits of oral Streptococci
Kai Ren, Qiang Guo, Xin Xu


State Key Laboratory of Oral Diseases & National Clinical Research Center for Oral Diseases & Deptment of Operative Dentistry and Endodontics, West China Hospital of Stomatology, Sichuan University, Chengdu 610041, Sichuan Province, China
Received: 16 September 2020; Revised: 7 December 2020; Published online: 28 January 2021
*Corresponding author: Tel: +86-28-85503462; E-mail: Xin Xu, xin.xu@scu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China(81500842)
Abstract: Streptococci are the most common oral bacteria that play an important role in oral microbial ecology and pathogenicity. Most of oral streptococci can enter a specific physiological state known as competence, during which the bacteria can take up DNA from the environment and integrate it into their genome to gain new phenotypes. Many studies have shown that the competence regulatory pathways of oral streptococci are closely associated with their phenotypes including biofilm formation, bacteriocin production, acid resistance, oxidative stress, cell autolysis and drug resistance. Studies on the interplay among different phenotypes can not only advance the understanding of microbial interactions in the oral cavity, but also promote the management of oral infectious diseases. This review article summarizes the research progress on the relationship between competence and phenotypic traits of four typical oral streptococci, including Streptococcus mutans, Streptococcus gordonii, Streptococcus sanguis and Streptococcus pneumoniae.
Keywords: oral streptococcicompetencemicroecological balancedental cariesbiofilm
自然的DNA转化是一个水平基因转移(horizontal gene transfer)过程,细菌通过这个过程吸收环境中的裸露DNA并整合进自身基因组,从而获得新的遗传特性。此转化过程需要细菌进入一种特殊生理状态,即感受态(competence)。目前研究普遍认为这种水平的基因转移方式可以提高细菌对环境的适应性和存活能力,因此遗传转化也被认为是细菌进化的主要方式之一[1]。尽管遗传转化让细菌基因组更具可塑性和适应性,也可能给细菌带来不良的影响,甚至影响细菌生长[1-3]。因此,感受态的开启受到细菌自身调控系统的严格控制与外部特异性环境条件的影响[4-7]。研究表明,口腔链球菌感受态的调控通路与其生物膜形成[8-13]、细菌素产生[14-17]、耐酸[18]、氧应激[19-20]、细胞自溶[21-23]及耐药性获得[24-30]等表型的调控通路存在密切的联系。
目前,已确认在人类口腔中有分布的链球菌包括变异链球菌(Streptococcus mutans)、血链球菌(Streptococcus sanguis)、唾液链球菌(Streptococcus salivarius)、格氏链球菌(streptococcus gordonii)、肺炎链球菌(Streptococcus pneumoniae)等共25种[31]。文献回顾发现,有关口腔链球菌感受态与其他表型相互影响的研究以变异链球菌为主,此外还涉及格氏链球菌、血链球菌、肺炎链球菌。综上所述,本文将以变异链球菌、格氏链球菌、血链球菌和肺炎链球菌4种常见的口腔链球菌为对象,回顾目前口腔链球菌感受态与多个表型关系以及相关致病性的研究现状。
1 口腔链球菌感受态调控系统 1.1 核心感受态调控蛋白ComX 链球菌感受态的激活及遗传转化的发生依赖肽类信号分子激活σ-70家族的备择σ因子(alternative sigma factor) ComX (也被称为SigX)[7],从而引导RNA多聚酶转录一组编码DNA结合、输入和重组相关蛋白的晚期感受态基因。相对于这些晚期感受态基因,负责胞外信号感知与comX激活的基因被称为早期感受态基因。在链球菌属中,ComX受到细菌分泌至胞外的信号肽调控,这些信号肽的前体在胞内经特异性的转运蛋白加工成为成熟的信号肽,并转运到胞外。链球菌属的感受态调控受到环境因素的影响,比如抗生素、酸性环境、氧或温度刺激和营养信号等[4, 32]。当胞内ComX蛋白累计到特定浓度后,将激活下游晚期感受态基因的表达[7]。因此,ComX在链球菌感受态的调控网络中居于核心地位。
1.2 感受态与群体感应系统 群体感应(quorum sensing,QS)系统是指细菌的一种细胞密度依赖的细胞间信号传导机制[7]。群体感应的过程就是细菌通过产生、释放和识别自诱导剂(autoinducer)来完成细菌间的交流,并协调整体行为的过程[33]。目前发现了三类调控系统参与口腔链球菌QS系统的调节,即CSP(competence- stimulating peptide)介导的ComDE调控系统、XIP(sigX-inducing peptide)介导的ComRS调控系统和AI-2(autoinducer-2)介导的LuxS调控系统。而参与激活口腔链球菌comX的转录的各种com基因均受到ComDE和ComRS系统的调节,因此可以认为口腔链球菌感受态受到QS系统的调节。事实上,在大量关于口腔链球菌感受态及QS系统的研究中,对于两者调节通路的总结有类似以及交叉的部分[7, 12, 33, 34]。感受调控系统相当复杂,目前还发现了HdrRM、CiaHR、RcrRPQ等多个调控系统参与感受态的调控[18, 35-36],因此也有****认为,感受态的调节通络跟传统的QS系统模式不同,它可响应环境的微调,形成正反馈调节通路[37]。
1.3 ComDE与ComRS调控系统 经典的双组分信号转导系统(two-component signal transduction system) ComDE最早在1996年由Havarstein等在肺炎链球菌中发现[38-39],也是链球菌中研究最多的双组分系统之一。在变异链球菌中,由comC编码的46个氨基酸的CSP前肽由ATP转运蛋白ComAB处理并分泌至胞外,经细胞膜表面SepM蛋白酶加工为18个氨基酸的成熟CSP。CSP在胞外累积到阈值浓度后,与跨膜组氨酸激酶受体ComD胞外区域结合,后者特异的组氨酸残基自发性磷酸化,并将磷酰基转移至胞内同源反应调控子ComE的天冬氨酸残基上,从而激活ComE。这个过程导致ComE蛋白的二聚化并与感受态早期基因的启动子区域结合,并激活它们的转录。肺炎链球菌、格氏链球菌和血链球菌中,感受态信号通路主要由ComDE系统调控,当ComE激活后,可以直接激活comX的转录,从而激活感受态。
缓症链球菌群、咽峡炎链球菌群以外的其他链球菌菌群,在comX上游缺少ComCDE的同源分子和ComE结合序列,提示存在另一种信号传导系统在调节comX的转录[40]。2009年,Gardan及其同事发现在化学培养基(chemically defined medium,CDM)中,嗜热链球菌(Streptococcus thermophilus)可自发出现感受态,并且其DNA的转化依赖Opp寡肽通透酶系统[41]。寡肽通透酶系统与许多革兰阳性菌的胞外信号传导有关,它能转运寡肽信号肽进入胞内引起胞内后续反应。2010年,Fontaine等在唾液链球菌和嗜热链球菌中识别了由Opp寡肽通透酶系统转运至胞内的ComS前肽分子及其同源胞质效应器ComR[34]。随后,Mashburry-Warren及其同事在牛链球菌群、变异链球菌和化脓性链球菌中也证实了ComRS系统的存在[6]。
在变异链球菌中,XIP的前体ComS经过目前尚不明确的转运体分泌至胞外,形成最终成熟形式为7个氨基酸的肽链。分泌后的XIP在胞外累积到阈值浓度后,与Opp寡肽通透酶系统发生结合并被运输至胞内。XIP与转录激活子ComR结合形成ComR-XIP聚合体,此聚合体与位于comX和comS启动子上的ComR-box结合,从而促进他们的转录。通过此过程,ComR-XIP聚合体不仅刺激comS转录,形成一个正反馈回路,还直接促进comX的转录,调控感受态的形成。以变异链球菌为代表的变异链球菌群(mutans group)在感受态调控方面最为特殊,是链球菌属中目前已知的唯一具有ComDE和ComRS这两类感受态调控系统的菌群[5]。在变异链球菌中,ComE激活后无法直接激活comX的转录,而是通过目前尚不清楚的机制激活ComRS通路后激活comX的转录,从而激活感受态。本课题组在影响变异链球菌感受态调控的内外源因素方面的前期研究发现感受态的调控与变异链球菌的生长环境及生长阶段密切相关,变异链球菌感受态的激活受到培养基中肽类成分的强烈影响,CSP只能在富含肽类的天然培养基中诱导感受态,而XIP只能在不含肽类的合成培养基中诱导感受态[32]。在对数生长早期,感受态的诱导水平较高,而对数中期及后期comX对XIP和CSP的响应水平急剧下降。此外,感受态的诱导还与细菌生长环境的pH值有关,酸性环境会抑制感受态的激活[4]。感受态的其他调控通路如HdrRM、CiaHR和RcrRPQ等与表型间的关系还处于研究阶段[18, 35-36],故本文将以典型的ComDE和ComRS感受态调控系统(图 1)进行感受态与口腔链球菌多个表型关系的介绍。
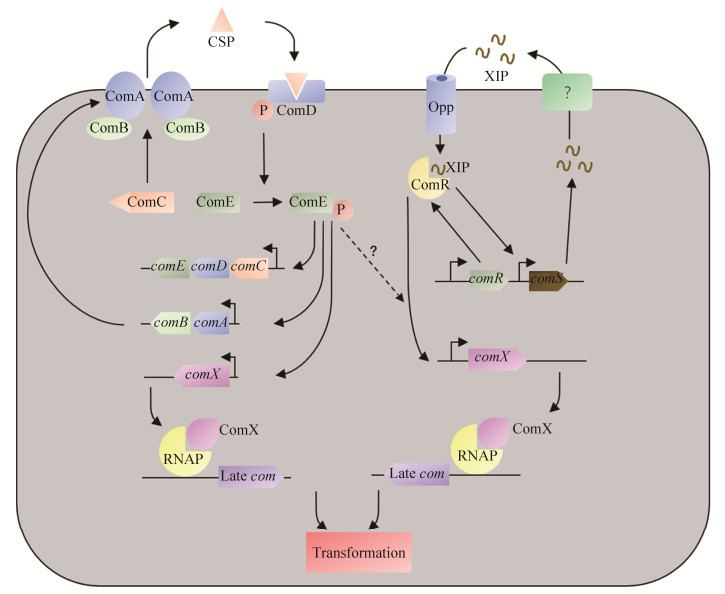 |
| 图 1 口腔链球菌感受态ComDE系统与ComRS系统 Figure 1 The ComDE and ComRS systems of the competence in oral Streptococci. |
| 图选项 |
2 口腔链球菌感受态调控与多种表型关系密切 大量研究发现,感受态的调控通路与口腔链球菌生物膜形成、细菌素产生、细胞自溶、耐酸和氧应激等多种表型存在密切关系,而上述表型的表达与调控对细菌维持自身细胞生理活动、影响口腔微生物稳态,进而影响宿主有重要影响。
2.1 变异链球菌 人类口腔中的多种微生物与龋病发生、发展密切相关,其中变异链球菌尤其引人注目。变异链球菌可以利用蔗糖合成葡聚糖产生大量的粘性胞外基质形成生物膜,介导其在牙齿表面的黏附和共聚;同时,变异链球菌能快速代谢多种糖类产酸,使得牙体组织脱矿形成龋坏;此外,拥有在低pH环境下存活的强耐酸能力。依靠这三大毒力特性,变异链球菌在牙菌斑生物膜致龋过程中发挥了重要作用,因此被认为是人类龋病的主要条件致病菌[42]。多个研究表明,变异链球菌的感受态调控系统与其多个毒力表型的调控通路存在紧密联系。
2.1.1 生物膜形成: 变异链球菌在牙面附着和聚集,并进一步形成牙菌斑生物膜是其致龋的前提和基础[43]。在变异链球菌生物膜形成过程中,适当浓度的感受态信号肽CSP有助于增加生物膜的生物量。Zhang等的研究发现,在加入外源性CSP(终浓度1.0 μg/mL)12 h和24 h后,生物膜中死亡和存活的细菌数量均多于未加CSP的对照组[44]。他们认为由CSP引起的细胞死亡对于生物膜的成熟是有益的。细胞死亡引起胞内基因组DNA的释放,而释放到环境中的胞外DNA(extracellular DNA,eDNA)被认为是主要的胞外黏附性基质之一,在生物膜的细胞之间起到连接、黏附作用[45-46]。
Senpuku H等通过基因芯片技术筛选到74个与变异链球菌生物膜形成有关的基因,研究人员随机选取了其中的21个,并识别出8个由CSP诱导的基因,其中之一是smu.940基因。变异链球菌在失活该基因后,以添加0.25%浓度葡萄糖的TSB(tryptic soy broth)培养基培养时,出现依赖葡聚糖的强烈共聚,生物膜形成能力显著增强。变异链球菌葡聚糖结合蛋白C(glucan-binding protein C,GbpC)参与葡萄糖依赖的共聚作用,进而影响其黏附和聚集,实验发现,SMU.940敲除突变株的gbpC基因表达水平比野生株更高,表明由CSP诱导的smu.940基因通过影响GbpC的表达,参与变异链球菌葡萄糖依赖的共聚过程和生物膜的形成过程[8]。
Senadheera和Hung等[9-10]发现,其他影响葡聚糖合成的基因如VicRK等的上游也存在ComE调节蛋白假定结合位点,暗示对葡聚糖合成的调控过程很可能是通过感受态调控系统ComE的共同作用来实现。Tamura S等将唾液链球菌和变异链球菌共培养时发现后者生物膜形成过程受到了严重抑制,进一步研究发现此抑制作用与唾液链球菌失活变异链球菌CSP信号肽进而影响其感受态信号传导通路有关[11]。
Li等则直接敲除变异链球菌NG8菌株的comC、comD、comE、comX感受态基因来观察生物膜形成的变化,实验发现ComC敲除株形成的生物膜结构异常,而ComD,ComE,ComX敲除株形成的生物膜量减少。外源性加入CSP或通过质粒构建comC基因回复株后,ComC敲除株形成生物膜恢复野生株表型,而ComD,ComE,ComX敲除株没有明显改变。该实验直接证明,与感受态调节相关的ComDE通路异常时,生物膜形成过程也出现异常[12]。
2.1.2 变链素产生: 变链素(mutacin)是指由变异链球菌产生的细菌素,可抑制与变异链球菌竞争同一生态位点的其他细菌,有助于其在牙面的定殖和致龋[47]。如表 1所示,目前共发现了十多种变链素,分为两大类,即抗菌谱较广、对革兰阳性菌及不分泌变链素的变异链球菌均有作用的羊毛硫抗生素(lantibiotics,如变链素Ⅰ、Ⅱ、Ⅲ)和仅对近缘菌种有效的非羊毛硫抗生素(non-lantibiotics,如变链素Ⅳ、Ⅴ、Ⅵ、N)[47]。研究表明,变链素的产生受到ComDE感受态系统的调控。Kreth等发现,在变异链球菌UA140中,外源性加入CSP可以诱导感受态和变链素Ⅳ基因nlmAB的协同表达[14]。而van der Ploeg的实验则进一步揭示了某些变链素基因上游存在ComE的结合位点。DNA测序分析发现,在nlmAB基因的上游存在2段被12个碱基隔开的9个碱基的保守序列,可被磷酸化的ComE蛋白识别,从而实现感受态与变链素产生的协同调控[15]。通过构建融合nlmAB基因启动子的半乳糖苷酶报告菌株,他们发现,在变异链球菌处于高细胞浓度时,变链素合成有关基因的表达上升;同样,在加入0.5 μg/mL CSP的情况下,其表达上调60倍,而敲除comDE基因后表达中止。
表 1. 变异链球菌变链素 Table 1. The mutacins of Streptococcus mutans
| Names | Producer strains | Regulators |
| Lantibiotics | MutR | |
| Mutacin I[48] | UA140,CH43 | MutR |
| Mutacin Ⅱ[49] | T8 | MutR |
| Mutacin Ⅲ[50-51] | UA787,JH1140 | |
| Mutacin B-NY266[52] | NY266 | ComE,HdrR?BrsR? |
| Mutacin Smb[53] | GS5(SmbA,SmbB) | |
| Mutacin K8[54] | K8 | MukR |
| Non-lantibiotics | ||
| Mutacin Ⅳ[55](NlmA,NlmB) | UA140,UA159,L13 | ComE,HdrR,BrsR |
| Mutacin Ⅴ[56](CipB) | UA159,UA140 | ComE,HdrR,BrsR |
| Mutacin Ⅵ[57](NlmD) | UA159 | ComE,HdrR,BrsR |
| Mutacin N[58] | N | ComE? |
表选项
有趣的是,Perry及Dufour等研究发现,变异链球菌中由CSP诱导产生的CipB蛋白(变链素V)在胞内、胞外的作用是不同的。CipB已被证明参与细胞自溶、eDNA释放等细菌生理过程[59]。通过Nlm TE运输至胞外后,CipB主要行使抑制近缘细菌的细菌素作用,而CipB在胞内则可以影响感受态相关基因表达来参与感受态调节[59]。基因芯片表达谱显示,在CSP诱导条件下,胞内的CipB能调节包括comDE、comR和comX在内的约130个基因,从而将ComDE双组分系统以及ComRS感受态调控系统联系起来[59-60]。
除了CSP信号肽以外,XIP信号肽对变链素合成过程也具有调控作用。Wenderska等通过RNAseq的方法研究了XIP对变异链球菌转录组的作用。实验在UA159菌株培养基中加入了1 μmol/L XIP的培养至对数中期,同时为了排除内源性XIP的影响,研究人员构建了敲除comS的ΔSMcomS菌株,并在同样条件下培养。在加入外源性XIP后,UA159和ΔSMcomS菌株中与感受态、变链素相关的基因表达确实得到了上调,证明了感受态ComRS系统与变链素产生存在直接联系[16]。
2.1.3 细胞自溶和eDNA释放: 细菌自溶指的是细菌在不适合生长合成代谢的条件下,在自身水解酶类作用下细胞的消解过程,并伴随着胞内物质如eDNA的释放。Perry及Dufour等也发现胞外CipB蛋白作为细菌素对自身、近亲细胞的攻击、溶解现象。实验发现,尽管敲除一些变链素基因影响了CSP诱导条件下吸收外源DNA的能力,但只有无法产生变链素V的cipB突变株转化效率严重降低。另外,CipB虽然能引起细胞的溶解,但实验发现,细胞溶解后释放的eDNA并没用被转化进入感受态细胞,这可能和前面提到的胞内CipB蛋白对感受态的调节有关[59-60]。这些研究结果表明调节细胞自溶与eDNA释放的作用机制也同时影响了感受态的信号通路。除了CipB引起的细胞自溶外,变异链球菌还存在其他自溶机制。在变异链球菌中,lytF基因编码一种保守的肽聚糖水解酶,而lytF本身受到ComX的调控,属于晚期感受态基因,参与CSP诱导的细胞自溶过程。Dufour等发现当细胞受到热、H2O2和酸等环境因素刺激时,CSP浓度升高,而这种变化将会激活lytF启动子,进而合成水解酶LytF,引起细胞自溶[21]。Ahn等从变异链球菌中分离得到自溶素AltA,他们发现无论是否加入外源性CSP,altA缺陷菌株中能进入感受态的细菌数量都明显的下降,而RT-PCR显示comD、ciaR、comX等感受态相关基因表达有明显下降,这表明变异链球菌感受态至少在转录水平上受到了自溶素基因缺失的影响[22]。
2.1.4 耐酸: 变异链球菌拥有的强耐酸能力有助于其在牙菌斑生物膜致龋过程中发挥重要的作用。2006年Ahn等实验发现,变异链球菌BM71菌株在高细胞浓度时的耐酸性明显高于低细胞浓度。实验所构建的comC、comD、comE单基因缺陷突变株对酸性物质的刺激耐受明显存在缺陷,而在comC缺陷株的培养基中加入外源性CSP后,菌株可恢复一定的耐酸性[18]。Senadheera等在构建VicRK缺陷株时发现缺陷株乳酸产量下降,但在pH < 3.5环境中,缺陷株存活率却明显升高,考虑到VicRK上游存在ComE假定结合位点,这也表明细菌耐酸与感受态表型之间存在交叉调控的可能性。
2.1.5 氧应激: 细菌的氧应激,指的是氧化物胁迫造成细菌胞内氧化破坏,作为一种生理信号激活特定的调控子,诱导合成某些抗氧化物质从而保护自身的现象[61]。为了观察不同氧刺激下变异链球菌感受态核心调控因子的响应情况,De Furio等构建了融合comX启动子的荧光蛋白报告菌株,通过使用百草枯产生超氧负离子、H2O2产生羟基,发现变异链球菌在接触到两种不同的氧刺激后,comX表达的激活程度不同,这表明变异链球菌氧应激可影响感受态信号通路,并且不同氧刺激对感受态核心调控基因的影响程度不同,相应的作用机制可能也存在差异[19]。
2.1.6 糖代谢: 变异链球菌可代谢多种糖底物,其糖代谢不仅能提供细菌各种生理活动所需能量,更是细菌产酸、形成生物膜等毒力表型的基础。变异链球菌感受态相关基因的表达与细菌糖代谢有着密切的关系。Zeng等为了研究变异链球菌糖代谢中缺失糖运输通透酶对感受态的影响,通过实验构建了与葡萄糖代谢相关的manLMN基因和与果糖代谢相关的fruRKI基因的突变株,比较了这些缺陷株的各个毒力表型的改变。实验发现,变异链球菌涉及果糖代谢的部分基因突变后,细菌的生长、毒力相关表型都出现了变化。FruR或者FruK的突变株其感受态相关基因(包括早期基因、comX、晚期基因)的表达以及多种转录调节子、负责能量代谢的酶、生物膜形成、生物合成作用、降解作用和对外界环境刺激因子的耐受能力都受到了影响[62]。
2.2 格氏链球菌 格氏链球菌作为革兰阳性的草绿色链球菌之一,是牙菌斑生物膜早期定殖菌之一,属于人类口腔正常菌群成分之一[63]。目前认为其在口腔中定殖对于维持人类健康个体的牙菌斑微生物稳态发挥重要作用[64]。多项研究表明,格氏链球菌的感受态与其生物膜形成、细菌素产生、细菌自溶及eDNA释放的调控通路关系紧密。
2.2.1 生物膜形成: 与变异链球菌相似,格氏链球菌生物膜形成的过程也与感受态调节通路有着密切的关系。SdbA是格氏链球菌的一种二硫键氧化还原酶,Davey等的研究发现,ΔsdbA突变株无法形成二硫键,造成一些蛋白错误折叠。这些错误折叠的蛋白作为一种应激信号,激活CiaRH双组份系统,进一步激活DegP(HtrA)蛋白酶,降低细胞膜上的异常蛋白。敲除sdbA的效应是多效性的,突变株的感受态、细菌素产生、自溶及eDNA释放等表型均受到抑制,而生物膜形成能力却增强[13]。为了研究生物膜形成增强的现象,实验人员构建了ΔsdbA,ΔciaRH两种单突变株和ΔsdbAΔciaRH双突变株。研究发现,加入外源性CSP并不会影响WT菌株生物膜的形成,同时也不影响ΔciaRH和ΔsdbAΔciaRH双突变株的生物膜形成,但会造成ΔsdbA突变株的生物膜形成能力下降到与WT相当水平。由此推断,格氏链球菌的生物膜表型受到CiaRH系统和ComDE系统相互作用的影响。而这种CiaRH系统与ComDE系统的相互作用在体内外实验中都有表现,体外表现为生物膜形成能力增强,而小鼠体内实验表明ΔsdbA菌株定殖能力增强[13]。
2.2.2 链球菌素产生: 格氏链球菌可以产生被称为链球菌素的细菌素。2006年,Heng等发现在格氏链球菌DL1菌株中,链球菌素/β-溶血素直接受到感受态的调控。格氏链球菌产生链球菌素STH1和STH2需要完整的comX和comAB基因的参与,链球菌素基因位点(sthA,sthB)在感受态晚期得到表达。comAB基因缺陷株在加入外源性CSP的情况下仍不能完成链球菌素的合成,提示两种链球菌素可能均由ComAB处理并外排[17]。Davey等发现格氏链球菌sdbA基因敲除后其链球菌素产生受到抑制[13],进一步研究发现,该现象是CiaRH系统与ComDE系统相互作用的结果,CiaRH系统激活抑制了ComDE感受态调控系统,从而抑制了感受态及链球菌素的产生。当敲除ΔsdbA突变株的ciaRH基因后,产链球菌素的表型得到恢复[65]。
2.2.3 细胞自溶及eDNA释放: 格氏链球菌细胞自溶及eDNA释放与感受态的紧密关系也有文献报道。eDNA的释放是细胞自溶的典型结果,研究发现格氏链球菌的eDNA释放由丙酮酸氧化酶产生的H2O2决定,外源性H2O2会诱导不产生H2O2条件下的细胞释放eDNA[66]。自溶素AtlS和胞壁水解酶LytF均涉及格氏链球菌eDNA释放过程,研究发现,编码其中任意一种或两种酶的基因发生突变都会减少eDNA的释放[23]。对LtyF突变株,即使外源性添加H2O2也无法诱导其释放eDNA,表明格氏链球菌的LytF酶在其eDNA释放中具有非常重要的作用。考虑到编码LytF的基因受到感受态系统的调控,可以认为格氏链球菌自溶及释放eDNA的行为是感受态系统调节下的对H2O2刺激的应激反应。
2.3 血链球菌 血链球菌最早能在萌出的牙齿上检测到,是最早定居于牙面的细菌之一,也是口腔正常菌群的优势菌之一。目前普遍认为,血链球菌是重要的牙周有益菌,通过产血链素和H2O2对大多数牙周可疑致病菌产生拮抗作用[67]。
Cullin等研究发现,当以CSP诱导血链球菌感受态时,在pH 6.8条件下,comD基因表达上调4倍,而comYA和lytF表达上调超过100倍。lytF基因属于血链球菌晚期感受态基因,编码水解酶LytF。转化能力实验发现,与野生株相比,ΔlytF突变株转化能力降低4倍。LytF可以诱导血链球菌细胞自溶及eDNA的释放,且如果LytF的调节出现异常,细胞会出现形态学异常从而进一步表现为生物膜的异常。这表明,血链球菌感受态调节与其生物膜形成、细胞自溶及eDNA释放等表型调控存在紧密关系[68]。
Ting等对血链球菌中SpxA调节子的研究发现了血链球菌感受态和氧应激的关系。SpxA是一组调节子蛋白,通过直接与RNA聚合酶反应来调节转录起始。在枯草芽孢杆菌中,SpxA可以负向调节感受态,在肺炎链球菌中,敲除spxA基因会改变com基因表达水平[69-70]。血链球菌有2个SpxA调节因子,即SpxA1和SpxA2。SpxA1通过激活链球菌中已经明确的氧应激基因(如nox和pox)参与链球菌在氧应激条件下的存活,pox基因(编码丙酮酸盐氧化酶)则参与了有氧环境下内源性H2O2的产生[71]。Li等在研究SpxA对血链球菌H2O2生成和感受态的影响时发现,SpxA2基因能通过抑制pox基因表达减少内源性H2O2的产生量,而内源性H2O2能负向调节感受态而不影响spxA2的表达。与血链球菌野生株相比,ΔspxA2突变株遗传转化效率下降,并且com基因表达也下调2-15倍[20]。
2.4 肺炎链球菌 肺炎链球菌最常见于人类上呼吸道黏膜表面,在口腔牙菌斑生物膜中亦有检出,当人体免疫力降低时,定殖的肺炎链球菌可引起感染。临床上,肺炎链球菌感染可导致肺炎、脑膜炎和败血症等,目前在口腔中的致病情况尚未见报道[72]。肺炎链球菌的致病毒力因子主要有荚膜(capsule,CPS)、肺炎链球菌溶血素(pneumolysin,PLY)、自溶素(LytA),肺炎链球菌表面蛋白(pneumococcal surface protein,Psp)和表面黏附素(pneumococcal surface adhesin A,PsaA)等[73]。肺炎链球菌感受态系统可以直接或间接调节细菌多个毒力因子,影响其在呼吸道的黏附和定殖。此外,直接由感受态调控的细菌溶解也可以调节肺炎链球菌的溶血素和脂磷壁酸的释放[72, 74]。
Kowalko等通过构建小鼠肺炎感染模型,发现肺炎链球菌ComE缺失可提高细菌在呼吸道的定殖能力,影响细菌的毒力和致病性[75]。Zheng等发现ComE缺失可导致细菌荚膜增厚,不利于细菌表面结构(如黏附素等)的暴露,从而影响细菌黏附定殖[76];此外,ComE的缺失消除了ComE对产荚膜的负向调控,增强了细菌的抗吞噬能力从而增强细菌毒力[76]。在急性肺炎感染模型中,感受态的激活将导致LytA、CbpD以及肺炎链球菌溶血素的释放,从而致病。Lau和Tal-Gan等合成了CSP拟似物CSP1-E1A以及CSP2-E1Ad10,发现它们分别对肺炎链球菌D39菌株以及TIGR4菌株溶血素的释放和其导致的溶血现象有竞争性抑制作用。但这两种拟似物因菌株抑制作用过于局限且酶降解稳定性差,均未有显著的临床治疗潜力[77-78]。Yang等通过肽环化设计并合成了一种针对肺炎链球菌CSP的拟似物CSP1-E1A-cyc(Dap6E10),后者可有效抑制D39和TIGR4菌株溶血素的释放与溶血作用[79]。在肺炎链球菌D39和TIGR4小鼠肺炎感染模型中,Dap6E10处理均能降低感染的发生,提高小鼠生存率。Dap6E10作为肺炎链球菌CSP的拟似物,可有效竞争CSP与ComD的结合,导致感受态晚期基因表达受限,表明肺炎链球菌CSP与ComD的正常结合从而激活感受态的过程正向调控了溶血素的产生和释放。
链球菌耐药是一个日益受到关注的现象,其中一个重要的耐药机制是水平基因转移,包括转化、转导与接合[24-25]。转化作为肺炎链球菌感受态系统下游一个重要的水平基因转移方式,与其耐药性的获得有着密不可分的关系[26-28]。转座子Tn2010属于肺炎链球菌接合转座子Tn916家族的一员,携带大环内酯类抗生素抗性基因及大环内酯类外排泵编码基因[29]。Zhou等研究发现,在CSP编码基因comC缺失的情况下,供体细菌Tn2010几乎无法转移到受体肺炎链球菌或粪肠球菌中[30]。阻断肺炎链球菌的感受态激活通路,可直接影响抗性基因及毒力因子相关基因的转化[80]。Domenech等通过高通量筛选获得了一种感受态阻断剂(COM-blockers),可通过抑制质子动力(proton motive force,PMF)阻断感受态通路的激活。COM-blockers可扰乱CSP向胞外外排的过程,阻断多种临床耐药菌株的感受态激活及转化,并且在小鼠感染模型中有效阻断HGT过程[80]。上述研究均表明,当肺炎链球菌的感受态激活过程受阻时,其下游与转化相关的基因表达同样受到抑制,表明至少转化来源的耐药基因转移过程受到了感受态的调控。
3 评语和展望 感受态与口腔链球菌多种表型间存在密切关系,但具体分子机制尚有待进一步深入研究。未来的研究应该更加深入地挖掘口腔链球菌感受态与其他表型之间相互作用的潜在分子机制与关键调控基因。变异链球菌作为人口腔主要致龋菌,其致龋性与口腔微生态环境有不可分割的关系,后续研究应考虑到各菌种间的相互影响,通过疾病模型研究与临床研究,进一步证实其感受态与致龋性的关系。肺炎链球菌作为感受态调控通路研究的模式菌种,其感受态晚期基因中有至少60个并不是转化必须基因[81-83],因此肺炎链球菌感受态直接调控的下游晚期基因与致病毒力表型的相关性仍值得进一步深入研究。在肺炎链球菌感染性疾病中,从鼻咽部位的定殖到侵入感染性疾病的发生,感受态是否参与细菌-宿主反应也应该是后续研究的方向之一[72]。总的来说,口腔链球菌感受态的激活直接或间接调控了细菌的部分遗传表型与致病毒力,尚需要进一步结合细菌本身感受态特点、细菌自身毒力因子以及细菌所处的微生态环境综合考虑进行深入研究。深入研究感受态及口腔链球菌表型间交叉作用的关键机制将使我们对细菌的定殖生长、细菌毒力表型的表达、菌种间的相互促进与拮抗、微生态环境的形成以及致病的发生发展等方面获得一个更清晰的认识,从而有助于推动口腔感染性疾病的生态防治。
References
| [1] | Didelot X, Maiden MCJ. Impact of recombination on bacterial evolution. Trends in Microbiology, 2010, 18(7): 315-322. DOI:10.1016/j.tim.2010.04.002 |
| [2] | Tian XL, Dong GF, Liu TL, Gomez ZA, Wahl A, Hols P, Li YH. MecA protein acts as a negative regulator of genetic competence in Streptococcus mutans. Journal of Bacteriology, 2013, 195(22): 5196-5206. DOI:10.1128/JB.00821-13 |
| [3] | Mirouze N, Bergé MA, Soulet AL, Mortier-Barrière I, Quentin Y, Fichant G, Granadel C, Noirot-Gros MF, Noirot P, Polard P, Martin B, Claverys JP. Direct involvement of DprA, the transformation-dedicated RecA loader, in the shut-off of pneumococcal competence. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(11): E1035-E1044. DOI:10.1073/pnas.1219868110 |
| [4] | Guo Q, Ahn SJ, Kaspar J, Zhou XD, Burne RA. Growth phase and pH influence peptide signaling for competence development in Streptococcus mutans. Journal of Bacteriology, 2014, 196(2): 227-236. DOI:10.1128/JB.00995-13 |
| [5] | H?varstein LS. Increasing competence in the genus Streptococcus. Molecular Microbiology, 2010, 78(3): 541-544. DOI:10.1111/j.1365-2958.2010.07380.x |
| [6] | Mashburn-Warren L, Morrison DA, Federle MJ. A novel double-tryptophan peptide pheromone controls competence in Streptococcus spp. via an Rgg regulator. Molecular Microbiology, 2010, 78(3): 589-606. DOI:10.1111/j.1365-2958.2010.07361.x |
| [7] | Shanker E, Federle MJ. Quorum sensing regulation of competence and bacteriocins in Streptococcus pneumoniae and mutans. Genes (Basel), 2017, 8(1): 15. DOI:10.3390/genes8010015 |
| [8] | Senpuku H, Yonezawa H, Yoneda S, Suzuki I, Nagasawa R, Narisawa N. SMU.940 regulates dextran-dependent aggregation and biofilm formation in Streptococcus mutans. Molecular Oral Microbiology, 2018, 33(1): 47-58. DOI:10.1111/omi.12196 |
| [9] | Senadheera D, Krastel K, Mair R, Persadmehr A, Abranches J, Burne RA, Cvitkovitch DG. Inactivation of VicK affects acid production and acid survival of Streptococcus mutans. Journal of Bacteriology, 2009, 191(20): 6415-6424. DOI:10.1128/JB.00793-09 |
| [10] | Hung DCI, Downey JS, Ayala EA, Kreth J, Mair R, Senadheera DB, Qi FX, Cvitkovitch DG, Shi WY, Goodman SD. Characterization of DNA binding sites of the ComE response regulator from Streptococcus mutans. Journal of Bacteriology, 2011, 193(14): 3642-3652. DOI:10.1128/JB.00155-11 |
| [11] | Tamura S, Yonezawa H, Motegi M, Nakao R, Yoneda S, Watanabe H, Yamazaki T, Senpuku H. Inhibiting effects of Streptococcus salivarius on competence-stimulating peptide-dependent biofilm formation by Streptococcus mutans. Oral Microbiology and Immunology, 2009, 24(2): 152-161. DOI:10.1111/j.1399-302X.2008.00489.x |
| [12] | Li YH, Tang N, Aspiras MB, Lau PCY, Lee JH, Ellen RP, Cvitkovitch DG. A quorum-sensing signaling system essential for genetic competence in Streptococcus mutans is involved in biofilm formation. Journal of Bacteriology, 2002, 184(10): 2699-2708. DOI:10.1128/JB.184.10.2699-2708.2002 |
| [13] | Davey L, Halperin SA, Lee SF. Mutation of the Streptococcus gordonii thiol-disulfide oxidoreductase SdbA leads to enhanced biofilm formation mediated by the CiaRH two-component signaling system. PLoS One, 2016, 11(11): e0166656. DOI:10.1371/journal.pone.0166656 |
| [14] | Kreth J, Merritt J, Shi WY, Qi FX. Co-ordinated bacteriocin production and competence development: a possible mechanism for taking up DNA from neighbouring species. Molecular Microbiology, 2005, 57(2): 392-404. DOI:10.1111/j.1365-2958.2005.04695.x |
| [15] | van der Ploeg JR. Regulation of bacteriocin production in Streptococcus mutans by the quorum-sensing system required for development of genetic competence. Journal of Bacteriology, 2005, 187(12): 3980-3989. DOI:10.1128/JB.187.12.3980-3989.2005 |
| [16] | Wenderska IB, Latos A, Pruitt B, Palmer S, Spatafora G, Senadheera DB, Cvitkovitch DG. Transcriptional profiling of the oral pathogen Streptococcus mutans in response to competence signaling peptide XIP. mSystems, 2017, 2(1): e00102-16. |
| [17] | Heng NCK, Tagg JR, Tompkins GR. Competence-dependent bacteriocin production by Streptococcus gordonii DL1(Challis). Journal of Bacteriology, 2007, 189(4): 1468-1472. DOI:10.1128/JB.01174-06 |
| [18] | Ahn SJ, Wen ZT, Burne RA. Multilevel control of competence development and stress tolerance in Streptococcus mutans UA159. Infection and Immunity, 2006, 74(3): 1631-1642. DOI:10.1128/IAI.74.3.1631-1642.2006 |
| [19] | De Furio M, Ahn SJ, Burne RA, Hagen SJ. Oxidative stressors modify the response of Streptococcus mutans to its competence signal peptides. Applied and Environmental Microbiology, 2017, 83(22): e01345-17. |
| [20] | Li T, Xu MY, Zheng LY. Is SpxA2 involved in hydrogen peroxide production and competence development in Streptococcus sanguinis?. Journal of Medical Microbiology, 2017, 66(7): 981-989. DOI:10.1099/jmm.0.000506 |
| [21] | Dufour D, Lévesque CM. Cell death of Streptococcus mutans induced by a quorum-sensing peptide occurs via a conserved streptococcal autolysin. Journal of Bacteriology, 2013, 195(1): 105-114. DOI:10.1128/JB.00926-12 |
| [22] | Ahn SJ, Burne RA. The atlA operon of Streptococcus mutans: role in autolysin maturation and cell surface biogenesis. Journal of Bacteriology, 2006, 188(19): 6877-6888. DOI:10.1128/JB.00536-06 |
| [23] | Xu YF, Kreth J. Role of LytF and AtlS in eDNA release by Streptococcus gordonii. PLoS One, 2013, 8(4): e62339. DOI:10.1371/journal.pone.0062339 |
| [24] | Bello-López JM, Cabrero-Martínez OA, Ibá?ez-Cervantes G, Hernández-Cortez C, Pelcastre-Rodríguez LI, Gonzalez-Avila LU, Castro-Escarpulli G. Horizontal gene transfer and its association with antibiotic resistance in the genus Aeromonas spp.. Microorganisms, 2019, 7(9): 363. DOI:10.3390/microorganisms7090363 |
| [25] | Lu M, Gong T, Zhang AQ, Tang BY, Chen JM, Zhang Z, Li YQ, Zhou XD. Mobile genetic elements in Streptococci. Current Issues in Molecular Biology, 2019, 32: 123-166. |
| [26] | Croucher NJ, Harris SR, Fraser C, Quail MA, Burton J, van der Linden M, McGee L, von Gottberg A, Song JH, Ko KS, Pichon B, Baker S, Parry CM, Lambertsen LM, Shahinas D, Pillai DR, Mitchell TJ, Dougan G, Tomasz A, Klugman KP, Parkhill J, Hanage WP, Bentley SD. Rapid Pneumococcal evolution in response to clinical interventions. Science, 2011, 331(6016): 430-434. DOI:10.1126/science.1198545 |
| [27] | Chewapreecha C, Harris SR, Croucher NJ, Turner C, Marttinen P, Cheng L, Pessia A, Aanensen DM, Mather AE, Page AJ, Salter SJ, Harris D, Nosten F, Goldblatt D, Corander J, Parkhill J, Turner P, Bentley SD. Dense genomic sampling identifies highways of Pneumococcal recombination. Nature Genetics, 2014, 46(3): 305-309. DOI:10.1038/ng.2895 |
| [28] | Gladstone RA, Lo SW, Lees JA, Croucher NJ, van Tonder AJ, Corander J, Page AJ, Marttinen P, Bentley LJ, Ochoa TJ, Ho PL, du Plessis M, Cornick JE, Kwambana-Adams B, Benisty R, Nzenze SA, Madhi SA, Hawkins PA, Everett DB, Antonio M, Dagan R, Klugman KP, von Gottberg A, McGee L, Breiman RF, Bentley SD. The Global Pneumococcal sequencing consortium. International genomic definition of Pneumococcal lineages, to contextualise disease, antibiotic resistance and vaccine impact. EBioMedicine, 2019, 43: 338-346. DOI:10.1016/j.ebiom.2019.04.021 |
| [29] | Del Grosso M, Northwood JGE, Farrell DJ, Pantosti A. The macrolide resistance genes erm(B) and mef(E) are carried by Tn2010 in dual-gene Streptococcus pneumoniae isolates belonging to clonal complex CC271. Antimicrobial Agents and Chemotherapy, 2007, 51(11): 4184-4186. DOI:10.1128/AAC.00598-07 |
| [30] | Zhou WQ, Yao KH, Zhang G, Yang YH, Li Y, Lv Y, Feng J. Mechanism for transfer of transposon Tn2010 carrying macrolide resistance genes in Streptococcus pneumoniae and its effects on genome evolution. Journal of Antimicrobial Chemotherapy, 2014, 69(6): 1470-1473. DOI:10.1093/jac/dku019 |
| [31] | Nicolas GG, Lavoie MC. Streptococcus mutans and oral Streptococci in dental plaque. Canadian Journal of Microbiology, 2011, 57(1): 1-20. DOI:10.1139/W10-095 |
| [32] | Son M, Ahn SJ, Guo Q, Burne RA, Hagen SJ. Microfluidic study of competence regulation in Streptococcus mutans: environmental inputs modulate bimodal and unimodal expression of comX. Molecular Microbiology, 2012, 86(2): 258-272. DOI:10.1111/j.1365-2958.2012.08187.x |
| [33] | Rocha FR, Regis WFM, Duarte S, Muniz FWMG, Rodrigues LKA. Effect of bioactive compounds on the regulation of quorum sensing network-associated genes and virulence in Streptococcus mutans-a systematic review. Archives of Oral Biology, 2020, 119: 104893. DOI:10.1016/j.archoralbio.2020.104893 |
| [34] | Fontaine L, Boutry C, de Frahan MH, Delplace B, Fremaux C, Horvath P, Boyaval P, Hols P. A novel pheromone quorum-sensing system controls the development of natural competence in Streptococcus thermophilus and Streptococcus salivarius. Journal of Bacteriology, 2010, 192(5): 1444-1454. DOI:10.1128/JB.01251-09 |
| [35] | Okinaga T, Niu GQ, Xie ZJ, Qi FX, Merritt J. The hdrRM operon of Streptococcus mutans encodes a novel regulatory system for coordinated competence development and bacteriocin production. Journal of Bacteriology, 2010, 192(7): 1844-1852. DOI:10.1128/JB.01667-09 |
| [36] | Seaton K, Ahn SJ, Burne RA. Regulation of competence and gene expression in Streptococcus mutans by the RcrR transcriptional regulator. Molecular Oral Microbiology, 2015, 30(2): 147-159. DOI:10.1111/omi.12079 |
| [37] | Hagen SJ, Son M. Origins of heterogeneity in Streptococcus mutans competence: interpreting an environment-sensitive signaling pathway. Physical Biology, 2017, 14(1): 015001. DOI:10.1088/1478-3975/aa546c |
| [38] | H?varstein LS, Coomaraswamy G, Morrison DA. An unmodified heptadecapeptide pheromone induces competence for genetic transformation in Streptococcus pneumoniae. Proceedings of the National Academy of Sciences of the United States of America, 1995, 92(24): 11140-11144. DOI:10.1073/pnas.92.24.11140 |
| [39] | H?varstein LS, Gaustad P, Nes IF, Morrison DA. Identification of the Streptococcal competence-pheromone receptor. Molecular Microbiology, 1996, 21(4): 863-869. DOI:10.1046/j.1365-2958.1996.521416.x |
| [40] | Martin B, Quentin Y, Fichant G, Claverys JP. Independent evolution of competence regulatory cascades in Streptococci?. Trends in Microbiology, 2006, 14(8): 339-345. DOI:10.1016/j.tim.2006.06.007 |
| [41] | Gardan R, Besset C, Guillot A, Gitton C, Monnet V. The oligopeptide transport system is essential for the development of natural competence in Streptococcus thermophilus strain LMD-9. Journal of Bacteriology, 2009, 191(14): 4647-4655. DOI:10.1128/JB.00257-09 |
| [42] | Takahashi N, Nyvad B. The role of bacteria in the caries process: ecological perspectives. Journal of Dental Research, 2011, 90(3): 294-303. DOI:10.1177/0022034510379602 |
| [43] | Rabin N, Zheng Y, Opoku-Temeng C, Du YX, Bonsu E, Sintim HO. Biofilm formation mechanisms and targets for developing antibiofilm agents. Future Medicinal Chemistry, 2015, 7(4): 493-512. DOI:10.4155/fmc.15.6 |
| [44] | Zhang K, Ou MZ, Wang WL, Ling JQ. Effects of quorum sensing on cell viability in Streptococcus mutans biofilm formation. Biochemical and Biophysical Research Communications, 2009, 379(4): 933-938. DOI:10.1016/j.bbrc.2008.12.175 |
| [45] | Bayles KW. The biological role of death and lysis in biofilm development. Nature Reviews Microbiology, 2007, 5(9): 721-726. DOI:10.1038/nrmicro1743 |
| [46] | Petersen FC, Tao L, Scheie AA. DNA binding-uptake system: a link between cell-to-cell communication and biofilm formation. Journal of Bacteriology, 2005, 187(13): 4392-4400. DOI:10.1128/JB.187.13.4392-4400.2005 |
| [47] | Merritt J, Qi F. The mutacins of Streptococcus mutans: regulation and ecology. Molecular Oral Microbiology, 2012, 27(2): 57-69. DOI:10.1111/j.2041-1014.2011.00634.x |
| [48] | Qi FX, Chen P, Caufield PW. Purification and biochemical characterization of mutacin I from the group I strain of Streptococcus mutans, CH43, and genetic analysis of mutacin I biosynthesis genes. Applied and Environmental Microbiology, 2000, 66(8): 3221-3229. DOI:10.1128/AEM.66.8.3221-3229.2000 |
| [49] | Novák J, Caufield PW, Miller EJ. Isolation and biochemical characterization of a novel lantibiotic mutacin from Streptococcus mutans. Journal of Bacteriology, 1994, 176(14): 4316-4320. DOI:10.1128/jb.176.14.4316-4320.1994 |
| [50] | Hillman JD, Novák J, Sagura E, Gutierrez JA, Brooks TA, Crowley PJ, Hess M, Azizi A, Leung K, Cvitkovitch D, Bleiweis AS. Genetic and biochemical analysis of mutacin 1140, a lantibiotic from Streptococcus mutans. Infection and Immunity, 1998, 66(6): 2743-2749. DOI:10.1128/IAI.66.6.2743-2749.1998 |
| [51] | Qi FX, Chen P, Caufield PW. Purification of mutacin Ⅲ from group Ⅲ Streptococcus mutans UA787 and genetic analyses of mutacin Ⅲ biosynthesis genes. Applied and Environmental Microbiology, 1999, 65(9): 3880-3887. DOI:10.1128/AEM.65.9.3880-3887.1999 |
| [52] | Mota-Meira M, Lacroix C, LaPointe G, Lavoie MC. Purification and structure of mutacin B-Ny266:a new lantibiotic produced by Streptococcus mutans. FEBS Letters, 1997, 410(2/3): 275-279. |
| [53] | Yonezawa H, Kuramitsu HK. Genetic analysis of a unique bacteriocin, Smb, produced by Streptococcus mutans GS5. Antimicrobial Agents and Chemotherapy, 2005, 49(2): 541-548. DOI:10.1128/AAC.49.2.541-548.2005 |
| [54] | Robson CL, Wescombe PA, Klesse NA, Tagg JR. Isolation and partial characterization of the Streptococcus mutans type AII lantibiotic mutacin K8. Microbiology (Reading), 2007, 153(Pt 5): 1631-1641. |
| [55] | Qi FX, Chen P, Caufield PW. The group I strain of Streptococcus mutans, UA140, produces both the lantibiotic mutacin I and a nonlantibiotic bacteriocin, mutacin Ⅳ. Applied and Environmental Microbiology, 2001, 67(1): 15-21. DOI:10.1128/AEM.67.1.15-21.2001 |
| [56] | Hale JDF, Ting YT, Jack RW, Tagg JR, Heng NCK. Bacteriocin (mutacin) production by Streptococcus mutans genome sequence reference strain UA159:elucidation of the antimicrobial repertoire by genetic dissection. Applied and Environmental Microbiology, 2005, 71(11): 7613-7617. DOI:10.1128/AEM.71.11.7613-7617.2005 |
| [57] | Xie ZJ, Okinaga T, Niu GQ, Qi FX, Merritt J. Identification of a novel bacteriocin regulatory system in Streptococcus mutans. Molecular Microbiology, 2010, 78(6): 1431-1447. DOI:10.1111/j.1365-2958.2010.07417.x |
| [58] | Balakrishnan M, Simmonds RS, Carne A, Tagg JR. Streptococcus mutans strain N produces a novel low molecular mass non-lantibiotic bacteriocin. FEMS Microbiology Letters, 2000, 183(1): 165-169. DOI:10.1111/j.1574-6968.2000.tb08952.x |
| [59] | Dufour D, Cordova M, Cvitkovitch DG, Lévesque CM. Regulation of the competence pathway as a novel role associated with a Streptococcal bacteriocin. Journal of Bacteriology, 2011, 193(23): 6552-6559. DOI:10.1128/JB.05968-11 |
| [60] | Perry JA, Cvitkovitch DG, Lévesque CM. Cell death in Streptococcus mutans biofilms: a link between CSP and extracellular DNA. FEMS Microbiology Letters, 2009, 299(2): 261-266. DOI:10.1111/j.1574-6968.2009.01758.x |
| [61] | Ezraty B, Gennaris A, Barras F, Collet JF. Oxidative stress, protein damage and repair in bacteria. Nature Reviews Microbiology, 2017, 15(7): 385-396. DOI:10.1038/nrmicro.2017.26 |
| [62] | Zeng L, Chakraborty B, Farivar T, Burne RA. Coordinated regulation of the EIIMan and fruRKI operons of Streptococcus mutans by global and fructose-specific pathways. Applied and Environmental Microbiology, 2017, 83(21): e01403-17. |
| [63] | Jenkinson HF, Lamont RJ. Streptococcal adhesion and colonization. Critical Reviews in Oral Biology & Medicine, 1997, 8(2): 175-200. |
| [64] | Abranches J, Zeng L, Kajfasz JK, Palmer SR, Chakraborty B, Wen ZT, Richards VP, Brady LJ, Lemos JA. Biology of oral Streptococci. Microbiology Spectrum, 2018, 6(5). DOI:10.1128/microbiolspec.GPP3-0042-2018 |
| [65] | Davey L, Halperin SA, Lee SF. Mutation of the thiol-disulfide oxidoreductase SdbA activates the CiaRH two-component system, leading to bacteriocin expression shutdown in Streptococcus gordonii. Journal of Bacteriology, 2016, 198(2): 321-331. DOI:10.1128/JB.00800-15 |
| [66] | Itzek A, Zheng L, Chen Z, Merritt J, Kreth J. Hydrogen peroxide-dependent DNA release and transfer of antibiotic resistance genes in Streptococcus gordonii. Journal of Bacteriology, 2011, 193(24): 6912-6922. DOI:10.1128/JB.05791-11 |
| [67] | Kreth J, Giacaman RA, Raghavan R, Merritt J. The road less traveled-defining molecular commensalism with Streptococcus sanguinis. Molecular Oral Microbiology, 2017, 32(3): 181-196. DOI:10.1111/omi.12170 |
| [68] | Cullin N, Redanz S, Lampi KJ, Merritt J, Kreth J. Murein hydrolase LytF of Streptococcus sanguinis and the ecological consequences of competence development. Applied and Environmental Microbiology, 2017, 83(24): e01709-17. |
| [69] | Nakano MM, Nakano S, Zuber P. Spx (YjbD), a negative effector of competence in Bacillus subtilis, enhances ClpC-MecA-ComK interaction. Molecular Microbiology, 2002, 44(5): 1341-1349. DOI:10.1046/j.1365-2958.2002.02963.x |
| [70] | Turlan C, Prudhomme M, Fichant G, Martin B, Gutierrez C. SpxA1, a novel transcriptional regulator involved in X-state (competence) development in Streptococcus pneumoniae. Molecular Microbiology, 2009, 73(3): 492-506. DOI:10.1111/j.1365-2958.2009.06789.x |
| [71] | Chen L, Ge XC, Wang XJ, Patel JR, Xu P. SpxA1 involved in hydrogen peroxide production, stress tolerance and endocarditis virulence in Streptococcus sanguinis. PLoS One, 2012, 7(6): e40034. DOI:10.1371/journal.pone.0040034 |
| [72] | Kadioglu A, Weiser JN, Paton JC, Andrew PW. The role of Streptococcus pneumoniae virulence factors in host respiratory colonization and disease. Nature Reviews Microbiology, 2008, 6(4): 288-301. DOI:10.1038/nrmicro1871 |
| [73] | Blumental S, Granger-Farbos A, Mo?si JC, Soullié B, Leroy P, Njanpop-Lafourcade BM, Yaro S, Nacro B, Hallin M, Koeck JL. Virulence factors of Streptococcus pneumoniae. comparison between african and french invasive isolates and implication for future vaccines. PLoS One, 2015, 10(7): e0133885. DOI:10.1371/journal.pone.0133885 |
| [74] | Claverys JP, Martin B, H?varstein LS. Competence-induced fratricide in Streptococci. Molecular Microbiology, 2007, 64(6): 1423-1433. DOI:10.1111/j.1365-2958.2007.05757.x |
| [75] | Kowalko JE, Sebert ME. The Streptococcus pneumoniae competence regulatory system influences respiratory tract colonization. Infection and Immunity, 2008, 76(7): 3131-3140. DOI:10.1128/IAI.01696-07 |
| [76] | Zheng YQ, Zhang XM, Wang XF, Wang LB, Zhang JH, Yin YB. ComE, an essential response regulator, negatively regulates the expression of the capsular polysaccharide locus and attenuates the bacterial virulence in Streptococcus pneumoniae. Frontiers in Microbiology, 2017, 8: 277. |
| [77] | Koirala B, Lin JJ, Lau GW, Tal-Gan Y. Development of a dominant negative competence-stimulating peptide (dnCSP) that attenuates Streptococcus pneumoniae infectivity in a mouse model of acute pneumonia. Chembiochem, 2018, 19(22): 2380-2386. DOI:10.1002/cbic.201800505 |
| [78] | Zhu L, Lau GW. Inhibition of competence development, horizontal gene transfer and virulence in Streptococcus pneumoniae by a modified competence stimulating peptide. PLoS Pathogens, 2011, 7(9): e1002241. DOI:10.1371/journal.ppat.1002241 |
| [79] | Yang YF, Lin JJ, Harrington A, Cornilescu G, Lau GW, Tal-Gan Y. Designing cyclic competence-stimulating peptide (CSP) analogs with pan-group quorum-sensing inhibition activity in Streptococcus pneumoniae. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(3): 1689-1699. DOI:10.1073/pnas.1915812117 |
| [80] | Domenech A, Brochado AR, Sender V, Hentrich K, Henriques-Normark B, Typas A, Veening JW. Proton motive force disruptors block bacterial competence and horizontal gene transfer. Cell Host & Microbe, 2020, 27(4): 544-555. e3. |
| [81] | Claverys JP, Prudhomme M, Martin B. Induction of competence regulons as a general response to stress in gram-positive bacteria. Annual Review of Microbiology, 2006, 60: 451-475. DOI:10.1146/annurev.micro.60.080805.142139 |
| [82] | Prudhomme M, Attaiech L, Sanchez G, Martin B, Claverys JP. Antibiotic stress induces genetic transformability in the human pathogen Streptococcus pneumoniae. Science, 2006, 313(5783): 89-92. DOI:10.1126/science.1127912 |
| [83] | Slager J, Kjos M, Attaiech L, Veening JW. Antibiotic-induced replication stress triggers bacterial competence by increasing gene dosage near the origin. Cell, 2014, 157(2): 395-406. DOI:10.1016/j.cell.2014.01.068 |
