路娟娥, 张彪, 邓磊, 刘素可, 武陶

 , 阮海华
, 阮海华

天津商业大学生物技术与食品科学学院, 天津 300134
收稿日期:2020-11-03;修回日期:2021-03-18;网络出版日期:2021-05-18
基金项目:国家自然科学基金(21908168, 31870122);天津市自然科学基金(18JCYBJC96000)
*通信作者:武陶, E-mail: wut126@163.com;
阮海华, E-mail: ruanhaihua@tjcu.edu.cn.
摘要:由食源性致病菌引发的食品安全问题是威胁人类健康的重要因素。因此,研究食源性致病菌的感染机理对于控制病原菌危害具有重要意义。[目的] 以常见的食源性致病菌——鼠伤寒沙门氏菌为研究对象,以其发挥重要致病性的转录调控因子SlyA为靶标,比较胞嘧啶单碱基编辑技术(CRISPR/Cas9-guided-Cytidine Base Editor,CBE)和λ-Red同源重组技术在构建鼠伤寒沙门氏菌SlyA敲除菌株方面的方法差异,为鼠伤寒沙门氏菌的基因编辑技术应用提供数据。同时,也为其他类型病原菌的基因编辑技术开发提供有力参考。[方法] 采用PCR、Golden Gate、Sanger测序等方法完成CBE系统以及λ-Red系统的构建以及敲除结果的验证,采用Editor-R软件分析CBE系统的单碱基编辑效率,采用Western blotting在蛋白表达层面对敲除结果进行验证。此外,本研究还结合了表型鉴定的方法验证了基因敲除结果。[结果] 经PCR产物测序鉴定、Western blotting分析及溶血素活性鉴定等结果表明,本研究成功将CBE系统应用于鼠伤寒沙门氏菌slyA的单碱基编辑中,应用前述两种方法构建了鼠伤寒沙门氏菌SlyA敲除菌株。[结论] CBE系统虽然以其操作的简便性在基因编辑中优势明显,但同λ-Red系统相比,该方法需要设立特定的gRNA及PAM位点,在非模式菌株中的普适性较低,且在进行编辑时,CBE系统存在不稳定的问题。尽管如此,但CBE系统在鼠伤寒沙门氏菌中的成功建立,为进一步拓展与完善该菌的基因编辑系统提供了基础。
关键词:鼠伤寒沙门氏菌SlyA胞嘧啶单碱基编辑系统λ-Red同源重组技术基因编辑
Comparative study on construction of SlyA knockout strain of Salmonella typhimurium using CRISPR/Cas9-guided-Cytidine Base Editor and λ-Red homologous recombination technology
Juane Lu, Biao Zhang, Lei Deng, Suke Liu, Tao Wu

 , Haihua Ruan
, Haihua Ruan

School of Biotechnology and Food Science, Tianjin University of Commerce, Tianjin 300134, China
Received: 3 November 2020; Revised: 18 March 2021; Published online: 18 May 2021
*Corresponding author: Tao Wu, E-mail: wut126@163.com;
Haihua Ruan, E-mail: ruanhaihua@tjcu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (21908168, 31870122) and by the Natural Science Foundation of Tianjin (18JCYBJC96000)
Abstract: Food safety issues caused by food-borne pathogens have become a critical factor threatening human health. Therefore, studying the infection mechanism of food-borne pathogens is of great significance to control the harm caused by pathogens. [Objective] Taking the common food-borne pathogenic bacteria Salmonella typhimurium as the research object, we studied the genetic manipulation methods for knocking out its pathogenicity associated transcriptional regulator coding gene SlyA by systematically compared the technical differences between CRISPR/Cas9-guided-Cytidine Base Editor (CBE) and λ-Red homologous recombination methods. The work present here not only provides detailed data for the application of gene editing technology for S. typhimurium, but also provides a useful reference for the development of gene editing technology for other pathogen species. [Methods] The PCR, Golden Gate, and Sanger sequencing were used to construct CBE and λ-Red gene editing systems and verify the gene edit results. Editor-R was used to analysis base edit efficiency of CBE system. Western blot was used to verify the results of gene editing at the protein expression level. In addition, phenotypic identification was used to verify the knockout results. [Results] The PCR product sequencing, Western blot verification results and hemolysin activity identification showed that the CBE gene editing system has been successfully applied to S. typhimurium. The SlyA null mutant of S. typhimurium was constructed based on the two above-mentioned technology, respectively. [Conclusion] Although the CRISPR/Cas9-guided Cytidine Base Editor has the advantages of simpler operation and more efficient editing than the λ-Red system, the system still has several shortcomings, such as the need to establish a specific gRNA sequence and PAM locus which are not so common in non-model strains. In addition, the CBE system has the problem of instability during gene editing. Nevertheless, CBE was successfully applied to Salmonella typhimurium for the first time, which will provide a basis for optimizing and improving the gene editing of this strain.
Keywords: Salmonella typhimuriumSlyACRISPR/Cas9-guided-Cytidine Base Editorλ-Red homologous recombination technologygene editing
鼠伤寒沙门氏菌(Salmonella typhimurium)是常见的食源性病原菌之一[1]。其主要传染途径与其他沙门氏菌大致相同,都是通过感染食物来间接感染人类或畜禽类,导致肠道感染及腹泻,严重感染可导致宿主死亡[2]。据统计,沙门氏菌病每年可造成9380万例食源性疾病和15.5万人死亡[3]。感染鼠伤寒沙门氏菌的症状主要包括腹痛、呕吐、恶心、发烧、头痛、肠胃炎、败血症等[4-5],由于沙门氏菌病暴发对人类造成的负面影响,研究沙门氏菌的特性并降低其在食品中的存活率对确保食品安全非常重要。
SlyA蛋白,是鼠伤寒沙门氏菌的一种致病性转录调控因子,由slyA基因编码,分属于MarR/SlyA家族[6]。slyA基因是启动鼠伤寒沙门氏菌二型毒力岛相关基因表达的中间环节,其转录调控蛋白SlyA与毒力、抗氧化应激、抗微生物肽和鼠伤寒沙门氏菌在巨噬细胞中的存活等有关[7-8]。研究表明,SlyA突变对沙门氏菌的毒力有显著影响,可导致鼠伤寒沙门氏菌的感染效率减弱,且在小鼠感染模型中无法有效地定殖Peyers斑块、肠系膜淋巴结等[9-11]。上述结果表明,SlyA能够提高鼠伤寒沙门氏菌对宿主的感染效率,这在一定程度上增加了食源性致病菌的危害。
目前,常见的基因编辑系统有CRISPR/Cas9和λ-Red同源重组系统。CRISPR/Cas9是一种存在于大多数细菌和古细菌中的适应性免疫防御系统,主要用来识别并清除二次入侵的病毒或外源DNA[12]。该系统能通过一系列反应实施靶位点特异性切割产生DNA双链断裂,激发细胞非同源末端连接(non-homologous end joining,NHEJ)或者同源介导的双链DNA修复(homology directed repair,HDR)机制,从而实现高效的基因组定点敲除、定点敲入和基因修饰等[13-14]。但此系统在进行敲除时,随机的插入和缺失经常导致移码突变,有时甚至给目标蛋白引入新的功能。在此形势下,单碱基编辑系统(CRISPR/Cas9-guided Base Editor)迅速兴起。单碱基编辑技术分为两类:嘧啶碱基编辑技术(C-G到T-A)和嘌呤碱基编辑技术(A-T到G-C)[15]。其中,胞嘧啶单碱基编辑系统(CRISPR/Cas9-guided- Cytidine Base Editor,CBE)的基本原理为:将Cas9切口酶(nickase Cas9,nCas9)与胞嘧啶脱氨酶融合,通过sgRNA靶向到目标序列,脱氨酶将编辑窗口内单链DNA中的胞嘧啶(cytidine,C)脱氨形成尿嘧啶(uracil,U),通过nCas9在与sgRNA互补DNA链上的切割作用,造成单链断裂,同时激活细胞内的DNA修复机制,促使细胞以被脱氨后的单链为模板进行修复,最后将原有的C-G碱基对替换为T-A。采用该系统进行单碱基编辑,可产生终止密码子(TAA,TGA)从而达到终止目标基因表达的效果,目前备受青睐[16-17]。λ-Red同源重组系统属于λ噬菌体的重组体系,主要编码exo、beta、gam三个基因,可经阿拉伯糖诱导表达Exo、Beta、Gam蛋白[18]。其中,Gam能够抑制宿主细胞中的核酸酶活性,从而保证外源线性DNA片段的稳定性,例如PCR产物;Exo具有5′-3′DNA依赖的核酸外切酶活性,可使长的线性双链DNA产生突出的3′粘性末端;Beta蛋白可结合到线性双链DNA的3′突出的粘性末端上,保护替代基因的DNA双链,并促进替代基因两端的序列和与其同源的靶基因序列间发生重组[19-21]。在此3种蛋白的协同作用下,λ-Red重组系统可以对任意大小和任意位置的DNA分子进行广泛的修饰。
本研究首次将CBE系统应用于鼠伤寒沙门氏菌,分别用CBE系统和λ-Red同源重组系统构建了鼠伤寒沙门氏菌SlyA敲除菌株,并将两种基因编辑技术进行比较分析,以期为鼠伤寒沙门氏菌基因编辑提供参考,同时也为进一步研究鼠伤寒沙门氏菌生物学功能奠定基础。
1 材料和方法 1.1 材料
1.1.1 菌株与质粒: 鼠伤寒沙门氏菌S. typhimurium LT2保藏于中国微生物菌种保藏管理委员会普通微生物中心(CGMCC),保藏编号为No.7020。质粒pKD46、pKD4和pCP20由耶鲁大学大肠杆菌保藏中心(E. coli Genetic Resources,the E. coli Genetic Stock Center at Yale University)馈赠。质粒pBAD24、pBAD24-Myc-SlyA为本实验室保存,质粒pnCas9-AID、p034-apr为中国科学院天津工业生物技术研究所馈赠[15]。
1.1.2 主要试剂与培养基: LB培养基(酵母浸出粉5 g/L、胰蛋白胨10 g/L、NaCl 10 g/L、琼脂粉15 g/L,pH值7.0);血琼脂培养基(血液琼脂基础38 g/L、脱纤维羊血6%,pH值7.0);氨苄青霉素(ampicillin,Amp)、氯霉素(chloramphenicol,Chl)、卡那霉素(kanamycin,Kan)、L-阿拉伯糖、异丙基-β-D-硫代半乳糖苷(isopropyl-β-D- thiogalactopyranoside,IPTG)、硫酸安普霉素(Apramycin Sulfate,Apr)均购自生工生物工程(上海)股份有限公司;KOD DNA聚合酶购自TOBOYO公司;DNA Marker购自天根生物技术有限公司;DNA回收试剂盒、偶联HRP羊抗兔二抗均购自Promega公司;蛋白Marker购自Fermentas公司;SopB多克隆抗体为本实验室保存;SlyA多克隆抗体由本实验室制备[22-23][本课题组以SlyA蛋白为抗原,对成年兔进行皮下注射,获取鼠伤寒沙门氏菌SlyA蛋白的多克隆抗体。多克隆抗体制备技术工艺流程为:制备SlyA蛋白-选择实验动物(成年雄兔)-进行动物免疫-试取血进行测试(免疫双扩散法验证是否成功免疫)-采集全部血清-纯化抗体-鉴定抗体(纯度及特异性)]。
1.1.3 仪器与设备: 紫外分光光度仪购于日本岛津公司;高压蒸汽灭菌锅购于日本SANYO公司;生化培养箱购于上海申贤恒温设备厂;电转化仪购于美国BTX公司;全自动化学发光成像系统购于上海天能科技有限公司;HZ-8811K恒温振荡器购于太仓市科教器材厂;PCR仪、核酸电泳仪、可见光凝胶成像分析系统、SDS-PAGE蛋白电泳仪均购于美国Bio-Rad公司。
1.1.4 引物设计与合成: 本研究所用引物如表 1所示。
表 1. 本研究所用引物 Table 1. Primers used in this study
| Primer | Sequence (5′→3′) |
| For homologous fragments amplification | |
| ??S1 | GGAATTGACGCAGACACATTGGGTCACGTTGCACAATATTCATCAATTGCGTAGGCTGGAGCTGCTTC |
| ??S2 | AATCAGAAGCTCAATCTCCTCTGAAGAAATCCCAGCCAAAATTTCACCGCCATGGTCCATATGAATATCCTCCTT |
| For slyA verification | |
| ??S-F | GTCGCAGATTCAATTGGCTAAAG |
| ??S-R | AATGACCTCTTCCATCTCAGC |
| ??slyA-F | ATGAAATTGGAATCGCCACTAGGTTCT |
| ??slyA-R | TCAATCGTGAGAGTGCAATTCCATAATGT |
| For gRNA fragments amplification | |
| ??gRNA1-F | CCAGGTCTCACGGTTGACCAGTCGCAGATTCAATGTTTTAGAGCTAG |
| ??gRNA2-F | CCAGGTCTCACGGTATTCAATTGGCTAAAGCGATGTTTTAGAGCTAG |
| ??gRNA3-F | CCAGGTCTCACGGTGTTGGATCAACTTGAAGATAGTTTTAGAGCTAG |
| ??gRNA-R | CCAGGTCTCAACCGAGATCTGACTCCATAACAGAGTACTCGC |
| For pgRNA verification | |
| ??99A-apr_F | CGTGAAAGGCGAGATCACC |
| ??99A-apr_R | GTCTGACGCTCAGTGGAAC |
| For slyA mutant verification near N20 | |
| ??N20-YZ-F | GGAATCGCCACTAGGTTCTG |
| ??N20-YZ-R | TCACATGGCCACACGTATGC |
| The underlined sequence is homologous with the sequence of slyA in S. typhimurium LT2. | |
表选项
1.2 CBE法构建SlyA敲除菌株 CBE法构建敲除菌株的示意图如图 1所示。
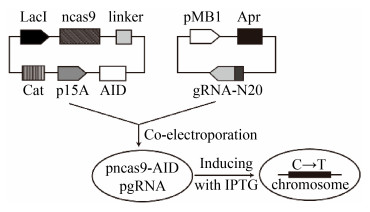 |
| 图 1 CBE法构建敲除菌株模式图 Figure 1 Pattern diagram of knockout strain constructed by CRISPR/Cas9-guided-Cytidine Base Editor. |
| 图选项 |
1.2.1 CBE系统的构建: CBE系统需要nCas9蛋白和gRNA的共同作用才能发挥基因编辑功能,构建gRNA质粒时,需要先设计相应的gRNA。根据GenBank中得到的沙门氏菌S. typhimurium LT2 slyA基因的相关序列信息,以及设计gRNA的基本原则,设计3个gRNA如表 2所示。
表 2. gRNA序列 Table 2. gRNA sequence
| Name | Sequence (5′→3′) |
| gRNA1 | TGACCAGTCGCAGATTCAATTGG |
| gRNA2 | ATTCAATTGGCTAAAGCGATAGG |
| gRNA3 | GTTGGATCAACTTGAAGATAAGG |
| The underlined part is the location of the target codon; the bold part is the PAM site. | |
表选项
3个gRNA的N20中都含有目标密码子CAG或CAA,位于N20 (左端)的第4–8位,目标密码子与PAM间隔12–14 bp,且目标密码子的上游紧邻碱基不为G,通过单碱基编辑实现C到T的转换,形成终止密码子TAA或TAG,可终止该蛋白的表达。以质粒p034-apr为模板,设计相应的扩增引物,通过Golden Gate反应,分别构建3个gRNA质粒。
1.2.2 鼠伤寒沙门氏菌电击转化感受态细胞的制备: 挑S. typhimurium LT2单菌落于5 mL LB液体培养基中,置于37 ℃、200 r/min摇床中过夜培养后,按1%接种量转接,并于37 ℃、200 r/min条件下培养至OD600为0.4–0.6,将菌液置于冰水浴中20 min,然后于4 ℃、4500 r/min条件下离心30 min收集菌体,并用预冷的10%甘油洗3–4次,最后用10%甘油重悬菌体,按100 μL分装至无菌离心管中,液氮速冻后保存于–80 ℃冰箱中待用。
1.2.3 CBE系统的转化及碱基编辑鉴定: 将pnCas9-AID质粒与gRNA质粒共同电击转化(2.0 kV,6 ms)至前述制备的S. typhimurium LT2感受态细胞中,菌液复苏45 min后涂布于添加2%葡萄糖的LB平板并过夜培养。次日挑取单菌落于37 ℃摇床培养,待菌株生长进入对数期时,加入终浓度为0.4 mmol/L的IPTG,过夜诱导nCas9蛋白表达,次日蘸取少量菌液,于LB抗性平板上划线培养,次日挑取单菌落,选用在N20附近设计的鉴定引物进行PCR扩增,并将PCR扩增产物送苏州金唯智生物科技有限公司测序。
1.2.4 SlyA敲除菌株在蛋白水平上的鉴定: 在上述实验的基础上,将测序成功的菌株于37 ℃摇床培养,次日分别取1 mL上述菌液,12000 r/min离心3 min收集菌体进行蛋白免疫印迹(Western blotting,WB)检测。步骤包括15% SDS-PAGE、转膜、封闭、SlyA多克隆抗体为一抗偶联HRP羊抗兔二抗进行蛋白印迹杂交、洗膜、全自动化学发光成像系统成像等。在进行WB实验时,选用SlyA蛋白作为阳性对照,并选用沙门氏菌中的分泌蛋白SopB作为内参,以定量每个泳道的上样量。
1.2.5 SlyA敲除菌株的溶血性能: SlyA最初被鉴定为是一种溶血素,由于抗原(红血球)和抗体(溶血素)可进行特异性的结合,同时吸引补体,使红血球在补体的作用下被溶解,因此可产生溶血现象[24]。基于此特性,本实验通过溶血素活性鉴定实验来验证敲除菌株是否构建成功。分别蘸取上述少量S. typhimurium LT2和SlyA敲除菌株菌液,划线至血琼脂培养基表面,放置于恒温培养箱,37 ℃培养12 h后,观察菌落周边的溶血情况。并在此基础上,将pBAD24-Myc-SlyA质粒转化至上述2个SlyA敲除菌株中,构建回补株C△slyA,并以转化了空载体pBAD24的△slyA菌株作为对照组。转化成功后,蘸取菌液划线至血琼脂培养基表面,37 ℃恒温培养12 h后,观察回补SlyA后菌落的溶血情况。
1.3 λ-Red法构建SlyA敲除菌株 λ-Red法构建敲除菌株的示意图如图 2所示。
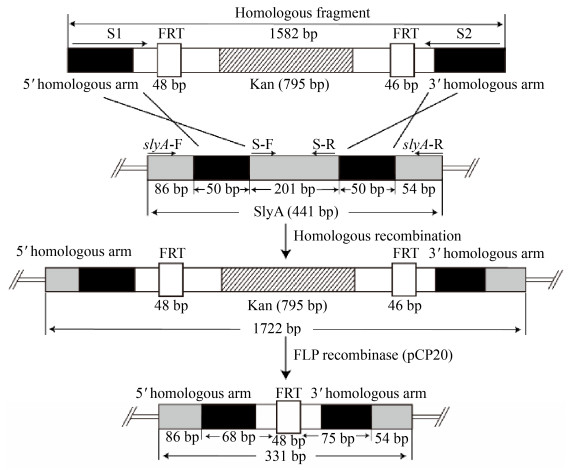 |
| 图 2 λ-Red同源重组技术构建敲除菌株模式图 Figure 2 Pattern diagram of knockout strain constructed by λ-Red homologous recombination technology. |
| 图选项 |
1.3.1 Red重组酶的诱导表达与感受态细胞的制备: 将pKD46质粒电转化(2.0 kV,6 ms)至S. typhimurium LT2感受态细胞中,30 ℃于Amp+ (100 μg/mL)平板上筛选阳性克隆,并将其接入5 mL含Amp+的LB液体培养基中,于30 ℃振荡培养,次日以1%接种量转接,30℃振荡培养至OD600于0.2–0.3,立即加入终浓度为5 mmol/L的L-阿拉伯糖诱导Red重组蛋白的表达;诱导1 h后将培养物置于冰水浴中20 min,4 ℃条件下4500 r/min离心30 min收集菌体,用10%甘油洗3–4次,最后用10%甘油重悬菌体,按100 μL分装至无菌离心管中,液氮速冻后于–80 ℃待用。
1.3.2 同源片段的获得及其转化: 以pKD4质粒为模板,含沙门氏菌slyA基因同源臂序列的引物S1和S2为上下游引物,扩增两端带有目的基因slyA同源序列的KanR片段。反应条件为:95 ℃ 5 min;95 ℃ 30 s,55 ℃ 30 s,68 ℃ 2 min,30个循环;68 ℃ 10 min。扩增结束后,用1%的琼脂糖凝胶电泳对PCR产物进行检测,并用DNA回收试剂盒回收PCR产物。后经Dpn Ⅰ消化(37 ℃酶切30 min),去除模板质粒后,回收同源打靶片段于无菌水中;取5 μL的同源片段至100 μL含有Red重组酶的沙门氏菌感受态细胞进行电击转化(2.0 kV,6 ms),之后于37 ℃条件下振荡培养6 h,涂布于含Kan+的平板上,37 ℃倒置培养。
1.3.3 slyA基因的敲除及鉴定: 随机挑取重组成功的6个单克隆,接种至5 mL LB液体培养基37 ℃过夜振荡培养。之后取2 μL菌液作为模板,利用slyA-F、slyA-R和S-F、S-R两对引物分别进行菌落PCR鉴定,同时以野生型菌株作为对照,PCR反应条件同1.3.2,最后用1%琼脂糖凝胶电泳鉴定slyA基因是否成功敲除,并在上述实验的基础上,按照1.2.4的方法进行WB检测。
1.3.4 SlyA敲除菌株的溶血性能: 与1.2.5方法相同,分别蘸取上述少量S. typhimurium LT2和随机抽取的2个SlyA敲除菌株菌液,划线至血琼脂培养基表面,放置于恒温培养箱,37 ℃培养12 h后,观察菌落周边的溶血情况。此外,将pBAD24-Myc-SlyA质粒转化至上述2个SlyA敲除菌株中,构建回补株C△slyA,并以转化了空载体pBAD24的△slyA菌株作为对照组。转化成功后,蘸取菌液划线至血琼脂培养基表面,37 ℃恒温培养12 h后,观察回补SlyA后菌落的溶血情况。
1.3.5 抗性基因的去除: 将筛选到的6个缺失株于不含抗生素的LB平板上划线,43 ℃培养过夜。次日挑取单菌落,于Amp+的平板上划线,再以牙签的同一部位于含Kan+的平板上划线,37 ℃培养过夜,挑选对Amp+敏感而对Kan+具有抗性的菌株,即为去除pkD46质粒的SlyA敲除菌株;将pCP20质粒用电转化法转化到上述敲除菌株中,转化后先30 ℃培养,消除抗性基因,再42 ℃培养,消除质粒pCP20,随后将缺失株转接至无抗生素的LB液体培养基中37 ℃培养过夜,取过夜培养菌进行PCR验证。
2 结果和分析 2.1 CBE系统SlyA碱基编辑结果
2.1.1 CBE碱基编辑系统的构建: 以p034-apr为质粒骨架,采用gRNA1-F和gRNA-R、gRNA2-F和gRNA-R、gRNA3-F和gRNA-R这3对引物进行PCR扩增,并通过Golden Gate反应完成自连,后经菌落PCR以及测序鉴定,证明3个gRNA质粒均构建成功,将其命名为pgRNA1、pgRNA2、pgRNA3,与之前已构建好的pnCas9-AID质粒构成了碱基编辑系统。CBE法构建敲除菌株模式图如图 1所示。
2.1.2 SlyA碱基编辑的鉴定: 挑取多个平板上过夜培养后获取的单克隆重组转化子,选用引物N20-YZ-F与引物N20-YZ-R进行PCR扩增,并将扩增后的产物送至公司测序,具体测序结果如图 3所示。通过比对测序结果发现,转化pgRNA1质粒的2号菌落以及转化pgRNA2质粒的A菌落均成功编辑,成功实现了C到T的转换,即出现了终止密码子TAG和TAA,而pgRNA3质粒所对应的菌落未能成功编辑,这可能是由于编辑过程中诱导不充分,或是gRNA位置选择不佳所致。
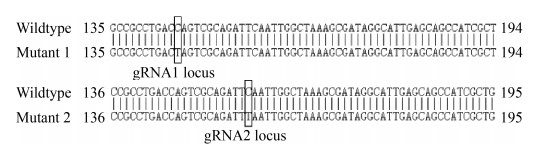 |
| 图 3 测序结果图 Figure 3 Sequencing result diagram. |
| 图选项 |
在测序结果的基础上,为验证单碱基突变后SlyA蛋白在蛋白质水平上是否被成功敲除,进行了WB分析鉴定。将上述筛选到的两株敲除菌株和野生型菌株分别接种于5 mL LB液体培养基中,培养16 h后,收取1 mL菌体,进行WB检测,结果如图 4所示。泳道1、2在16.69 kDa附近,未检测到任何蛋白条带;在泳道3、4中检测到分子量大约为16.69 kDa的蛋白条带,即与野生型沙门氏菌相比,利用CBE系统构建的△slyA菌株失去了表达SlyA蛋白的能力,从而在蛋白水平上证实slyA基因已被成功敲除。且在此实验中,选用沙门氏菌中的分泌蛋白SopB作为内参,以定量每个泳道的上样量。
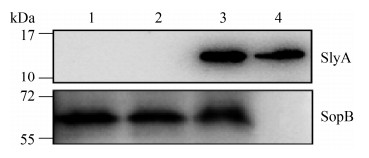 |
| 图 4 CBE法构建SlyA敲除菌株WB验证结果图 Figure 4 Construction of Western blotting verification result map of SlyA knockout strain by CRISPR/Cas9- guided-Cytidine Base Editor. M: Protein Marker; lane 1–2: Western blot result of two △slyA; lane 3: Western blotting result of S. typhimurium LT2; lane 4: Western blotting result of SlyA protein. |
| 图选项 |
2.1.3 编辑效率结果(利用Editor R软件): 为进一步考察slyA碱基编辑的效率,将液体培养诱导后的菌液同样采用引物N20-YZ-F与引物N20-YZ-R进行菌液PCR,并将PCR产物送测序公司测序,通过Editor R软件[25]对测序结果进行分析,分析结果如图 5所示。根据图中的分析结果,可看出pgRNA1与pgRNA2菌液中C转换成T的编辑效率分别为78%与87%,与文献中报道的单碱基编辑效率相比[26],属于较高的编辑效率水平。
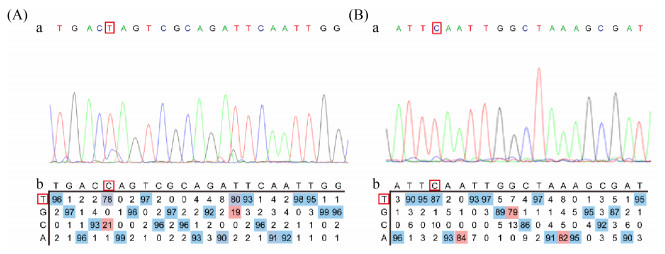 |
| 图 5 pgRNA1和pgRNA2对应菌液编辑效率分析图 Figure 5 Analysis diagram of editing efficiency of pgRNA1 and pgRNA2 corresponding to bacterial solution. A: analysis diagram of editing efficiency of pgRNA1 corresponding to bacterial solution; B: analysis diagram of editing efficiency of pgRNA2 corresponding to bacterial solution. a: peak diagram of sequencing results; b: the conversion ratio between bases. |
| 图选项 |
2.1.4 SlyA敲除菌株的溶血鉴定: 溶血结果如图 6所示,野生型沙门氏菌(A)生长的培养基菌落周围的红血球已完全发生溶解,且有明显可见的透明溶血圈,表现为β-溶血性状;而△slyA菌株(B)生长的平板上则未发现溶血现象,表现为γ-溶血(不溶血)性状,表明slyA基因已被成功敲除,菌株丧失了溶血能力,毒性减弱。在回补实验中,转化了pBAD24的对照组(C)同△slyA菌株性状相一致,表现为γ-溶血性状;而回补株C△slyA(D)的表型与野生型沙门氏菌一致,表现为β-溶血性状,表明沙门氏菌中slyA基因的缺失能够影响菌体的溶血能力。
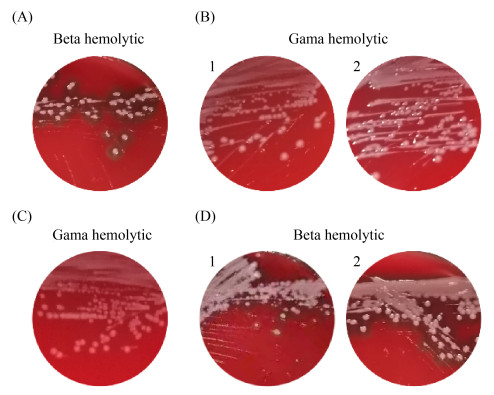 |
| 图 6 slyA基因敲除前、后的菌株及回补株的溶血现象(CBE法) Figure 6 Hemolysis of strains before and after slyA gene knockout and replacement strain (CBE method). A: S. typhimurium LT2; B: 1 and 2 are △slyA strains; C: Transformation of empty vector by △slyA strain; D: 1 and 2 are the replenishment results of the △slyA strains. |
| 图选项 |
2.2 λ-Red法构建SlyA敲除菌株结果
2.2.1 slyA基因敲除菌株的PCR鉴定: 以pKD4质粒为模板,利用S1、S2引物进行PCR扩增出两翼与slyA基因序列同源、中间为KanR的长同源臂DNA片段,片段长度理论值为1582 bp。将该同源打靶片段转化至含有Red重组酶的沙门氏菌感受态细胞后,随机挑取Kan+平板上的6个单克隆,将其溶于20 μL无菌水中,取2 μL菌液加入PCR小管中,再向各PCR管中加入48 μL除模板外的PCR反应体系,选用slyA-F、slyA-R和S-F、S-R两对引物分别进行菌落PCR鉴定。如图 7-A,采用引物slyA-F、slyA-R均扩增出了1722 bp左右的分子条带,且对照组扩增DNA片段长度为441 bp;图 7-B中,所选的6个单克隆通过引物S-F、S-R扩增均无条带显示,对照组则扩增出201 bp左右的分子条带,两组引物相互验证,可充分表明slyA基因中间201 bp大小的片段已被成功敲除。
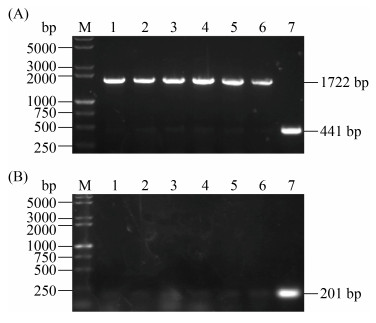 |
| 图 7 λ-Red法SlyA敲除菌株的PCR验证结果图 Figure 7 PCR validation results of SlyA knockout strain by λ-Red technology. A: M: D15000 DNA marker; lane 1–6: PCR result of 6 clones with primers slyA-F and slyA-R; lane 7: PCR result of S. typhimurium LT2. B: M: D15000 DNA marker; lane 1–6: PCR result of 6 selected mutants with primers S-F and S-R; lane 7: PCR result of S. typhimurium LT2. |
| 图选项 |
2.2.2 slyA基因敲除菌株的WB鉴定: 在沙门氏菌生长过程中,转录调控因子SlyA能够在菌体中正常表达。基于此,将上述筛选到的6株△slyA菌株和野生型沙门氏菌接种于5 mL LB液体培养基中,培养16 h后,收取1 mL菌体,进行WB检测。结果如图 8所示,在野生型沙门氏菌和SlyA蛋白中能够检测到分子量在17 kDa以下约为16.69 kDa大小的蛋白条带。与此相反,在筛选到的6株△slyA菌株中则未能检测到任何SlyA蛋白。即与野生型沙门氏菌相比,利用λ-Red重组系统构建的敲除菌株失去了表达SlyA蛋白的能力,从而在蛋白水平上证实slyA基因已被成功敲除。且在此实验中,选用沙门氏菌中的分泌蛋白SopB作为内参,以定量每个泳道的上样量。
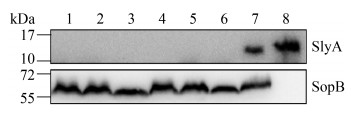 |
| 图 8 λ-Red法构建SlyA敲除菌株的WB验证结果图 Figure 8 Construction of Western blotting verification result map of SlyA knockout strain by λ-Red technology. M: protein marker; lane 1–6: Western blotting result of two △slyA; lane 7: Western blotting result of S. typhimurium LT2; lane 8: Western blotting result of SlyA protein. |
| 图选项 |
2.2.3 抗性基因的去除与验证: 筛选到的6株SlyA敲除菌株含有pKD46质粒(Amp+)和KanR。其中,pKD46质粒在30 ℃培养时可以正常复制,而在43 ℃培养时质粒则完全丢失[27]。如图 9-A所示,经43 ℃培养后敲除菌株均能够在含有Kan+的LB平板上生长,但不能在含Amp+的LB平板上生长。表明敲除菌株中pKD46质粒均被成功去除;针对于SlyA敲除菌株中的KanR,向其中转化能够编码FRT位点的pCP20质粒(含FLP重组酶),从而去除2个FRT位点间的KanR。将去除KanR后的菌株进行PCR验证,验证引物选择slyA基因的上游和下游引物,即slyA-F和slyA-R。若成功去除KanR,理论上,扩增的分子条带大约为331 bp,若未去除,则菌落PCR将扩增出接近1722 bp的DNA条带。根据图 9-B,筛选得到的菌株均得到了分子量大约为331 bp的DNA扩增条带,与KanR去除后的理论计算值相符。这表明成功去除了SlyA敲除菌株中的抗性基因,将新菌种命名为S. typhimurium LT2 △slyA。
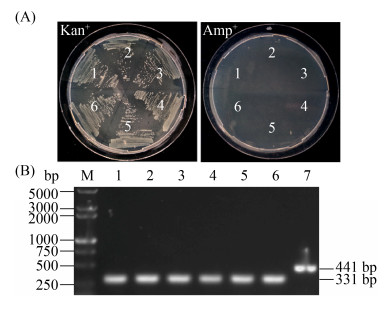 |
| 图 9 λ-Red法抗性基因的去除与验证结果图 Figure 9 Removal and verification of resistance genes by λ-Red technology. A: The left side shows the results of streak culture of 6 positive clones on the plate containing Kan+. On the right side, 6 positive clones were cultured on Amp+ plate. B: M: D15000 DNA marker; lane 1–6: PCR result of 6 clones with primers slyA-F and slyA-R; lane 7: PCR result of S. typhimurium LT2. |
| 图选项 |
2.2.4 SlyA敲除菌株的溶血鉴定: 基于上述实验结果,随机抽取2个SlyA敲除菌株,对其进行溶血活性检测,溶血结果如图 10所示。野生型沙门氏菌(A)生长的培养基菌落周围的红血球已完全发生溶解,且有明显可见的透明溶血圈,表现为β-溶血性状;而随机抽取的2个△slyA菌株(B)所生长的平板无溶血现象,表现为γ-溶血性状,这一现象表明了△slyA菌株中的slyA基因已被成功敲除,菌株丧失了溶血能力,毒性减弱。在回补实验中,转化了pBAD24的对照组(C)同△slyA菌株性状相一致,表现为γ-溶血性状;而回补株C△slyA(D)表现为β-溶血性状,表明沙门氏菌中slyA基因的缺失能够影响菌体的溶血能力。
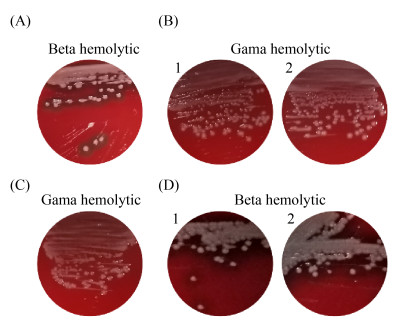 |
| 图 10 slyA基因敲除前、后的菌株及回补株的溶血现象(λ-Red法) Figure 10 Hemolysis of strains before and after slyA gene knockout and replacement strain (λ-Red method). A: S. typhimurium LT2; B: 1 and 2 are ΔslyA strains; C: Transformation of empty vector by △slyA strain; D: 1 and 2 are the replenishment results of the △slyA strains. |
| 图选项 |
3 讨论 鼠伤寒沙门氏菌是一种常见的食源性致病菌,对公众健康有重大威胁。SlyA作为鼠伤寒沙门氏菌的一个致病性转录调控因子,不仅编码溶血素,还参与沙门氏菌的毒力调控,对其进行深入研究有利于揭示鼠伤寒沙门氏菌的致病机理[28]。在鼠伤寒沙门氏菌侵染过程中,SlyA的基因表达调控是启动其二型毒力岛相关基因表达的重要步骤,它与鼠伤寒沙门氏菌其他转录调控因子之间存在着复杂的协同调控关系。这在一定程度上增加了沙门氏菌的危害性。因此,通过构建鼠伤寒沙门氏菌SlyA敲除菌株,可以为进一步了解鼠伤寒沙门氏菌的侵染机制提供帮助,同时也为提供更加有效的预防和控制沙门氏菌疾病措施提供理论支持。
近年来迅速兴起的CRISPR/Cas9系统是一种快速、简单且较为有效的基因编辑方法,能够实现永久性的基因组修饰,其主要通过NHEJ和HDR进行修复。虽然NHEJ和HDR介导的基因敲除较为有效,但已有研究表明CRISPR/Cas9技术在双链DNA断裂过程中可能会产生不良影响[29-32]。首先,系统敲除时,由于插入和缺失经常导致移码突变,可能会引入无法控制的随机变化,并且不是所有的随机插入或缺失都会导致基因敲除,有时候也可能给目标蛋白引入新的功能。其次,更关键的是,原始的CRISPR/Cas9技术对双链DNA的切割已被证明能够导致过度的DNA损伤和细胞死亡,特别是在多拷贝基因组区域。基于此,我们选用在CRISPR/Cas9基础上发展而来的胞嘧啶单碱基编辑技术,并将此技术首次尝试运用于沙门氏菌的基因编辑中,通过设计相应的gRNA,采用CBE系统,成功实现了碱基C到T的转变。上述结果表明,现有的碱基编辑系统可以较好地应用于沙门氏菌的基因编辑中,但其编辑效率还有改进与提高的空间。两个系统的比较分析如表 3所示。
表 3. CBE系统与λ-Red系统比较汇总表 Table 3. Summary of comparison between CBE system and λ-Red system
| Items | CBE system | λ-Red system |
| Operating system | Base editing system composed of pgRNA and pnCas9-AID | Three plasmids cooperation system of pKD4/pKD3- pKD46-pCP20 |
| PAM locus | Exist | Null |
| Universality | Bad | Good |
| Stability | Instability | Stability |
| Simplicity | Simpleness | Troublesome |
| Time | 10 d | 20 d |
表选项
相较于CBE系统,λ-Red同源重组技术则需要提供携带预期DNA变化的供体DNA,通过将两侧含有同源序列的线性抗性基因片段转入感受态细胞,使片段在λ噬菌体重组酶Exo、Beta、Gam的作用下与染色体序列发生双交换,从而实现基因编辑。λ-Red重组体系采用了“pKD4/pKD3-pKD46-pCP20”的3质粒协作系统,其中包括用于扩增抗性基因片段的模板质粒pKD4 (或pKD3)、含有λ-Red重组酶的pKD46质粒以及辅助质粒pCP20[33-34]。λ-Red同源重组是一个精确的过程,能够用抗性基因靶向改变基因组,但在去除抗性基因后,染色体被敲除基因座位处会留有一个FRT位点,故当敲除基因数量较多时,容易造成染色体的不稳定,从而无法实现基因的连续敲除[35]。另一方面,λ-Red重组技术在基因整合时受到很大限制。基因整合需要将目的基因与抗性基因进行拼接,由于λ-Red系统质粒无法进行直接的构建操作,基因整合只能采用如下策略:(1) 改造pKD4模板质粒,但pKD4质粒的拷贝数极低,用传统的构建方式难以实现;(2) 在普通质粒上拼接目的基因与抗性基因,则会导致重组后出现大量假阳性,对后续的筛选不利;(3) 选用不依赖质粒拼接方式的交错PCR技术,则无法获得高浓度的目的基因,导致重组率低下[36]。以上问题导致在使用λ-Red技术进行外源基因整合时,存在较大困难。
本研究成功在鼠伤寒沙门氏菌中建立了碱基编辑系统,并应用CBE系统和λ-Red同源重组两种方法构建鼠伤寒沙门氏菌SlyA敲除菌株。CBE系统以其操作的简便性在基因编辑中优势明显,仅需改变20 bp的gRNA序列,通过改变单核苷酸产生终止密码子即可敲除目标基因,在实验设计及操作上都极为简便且耗时较短,仅需10 d左右即可完成从编辑系统设计到获得编辑菌株的全部过程;λ-Red同源重组需要进行同源打靶片段的扩增,且需要诱导表达重组酶,在敲除成功后还需设计筛选引物,相比之下操作较为复杂。也有研究表明,CRISPR/Cas系统在针对不同的删除目标时作用效果并不稳定,且CBE系统存在针对目标基因的潜在gRNA数量较少,在非模式菌株中的普适性较低等问题;而λ-Red技术的优势在于无PAM位点要求,相对稳定,具有普适性,也适用于没有合适gRNA序列的基因。但尽管如此,CBE系统在沙门氏菌中的成功建立,亦为进一步拓展与完善该菌的基因编辑系统提供了基础。
References
| [1] | Islam MS, Zhou Y, Liang L, Nime I, Liu K, Yan T, Wang XH, Li JQ. Application of a phage cocktail for control of Salmonella in foods and reducing biofilms. Viruses, 2019, 11(9): E841. DOI:10.3390/v11090841 |
| [2] | Rugbjerg H, Wingstrand A, Hald T, Andersen JS, Lo Fo Wong DMA, Korsgaard H. Estimating the number of undetected multi-resistant Salmonella Typhimurium DT104 infected pig herds in Denmark. Preventive Veterinary Medicine, 2004, 65(3/4): 147-171. |
| [3] | Ritter AC, Tondo EC, Siqueira FM, Soggiu A, Varela APM, Mayer FQ, Brandelli A. Genome analysis reveals insights into high-resistance and virulence of Salmonella Enteritidis involved in foodborne outbreaks. International Journal of Food Microbiology, 2019, 306: 108269. DOI:10.1016/j.ijfoodmicro.2019.108269 |
| [4] | Eng SK, Pusparajah P, Ab Mutalib NS, Ser HL, Chan KG, Lee LH. Salmonella: a review on pathogenesis, epidemiology and antibiotic resistance. Frontiers in Life Science, 2015, 8(3): 284-293. DOI:10.1080/21553769.2015.1051243 |
| [5] | Haraga A, Ohlson MB, Miller SI. Salmonellae interplay with host cells. Nature Reviews Microbiology, 2008, 6(1): 53-66. DOI:10.1038/nrmicro1788 |
| [6] | Ellison DW, Miller VL. Regulation of virulence by members of the MarR/SlyA family. Current Opinion in Microbiology, 2006, 9(2): 153-159. DOI:10.1016/j.mib.2006.02.003 |
| [7] | Haider F, Lithgow JK, Stapleton MR, Norte VA, Roberts RE, Green J. DNA recognition by the Salmonella enterica serovar Typhimurium transcription factor SlyA. International Microbiology the Official Journal of the Spanish Society for Microbiology, 2008, 11(4): 245-250. |
| [8] | Kaneko A, Mita M, Sekiya K, Matsui H, Kawahara K, Danbara H. Association of a regulatory gene, slyA with a mouse virulence of Salmonella serovar choleraesuis. Microbiology and Immunology, 2002, 46(2): 109-113. DOI:10.1111/j.1348-0421.2002.tb02666.x |
| [9] | Cabezas CE, Briones AC, Aguirre C, Pardo-Esté C, Castro-Severyn J, Salinas CR, Baquedano MS, Hidalgo AA, Fuentes JA, Morales EH, Meneses CA, Castro-Nallar E, Saavedra CP. The transcription factor SlyA from Salmonella Typhimurium regulates genes in response to hydrogen peroxide and sodium hypochlorite. Research in Microbiology, 2018, 169(6): 263-278. DOI:10.1016/j.resmic.2018.04.003 |
| [10] | Zou LF, Zeng Q, Lin HP, Gyaneshwar P, Chen GY, Yang CH. SlyA regulates type III secretion system (T3SS) genes in parallel with the T3SS master regulator HrpL in Dickeya dadantii 3937. Applied and Environmental Microbiology, 2012, 78(8): 2888-2895. DOI:10.1128/AEM.07021-11 |
| [11] | Haque MM, Kabir MS, Aini LQ, Hirata H, Tsuyumu S. SlyA, a MarR family transcriptional regulator, is essential for virulence in Dickeya dadantii 3937. Journal of Bacteriology, 2009, 191(17): 5409-5418. DOI:10.1128/JB.00240-09 |
| [12] | Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [13] | Chandrasegaran S, Carroll D. Origins of programmable nucleases for genome engineering. Journal of Molecular Biology, 2016, 428(5): 963-989. DOI:10.1016/j.jmb.2015.10.014 |
| [14] | Huo XY, Du YT, Lu J, Guo M, Li ZK, Zhang SY, Li XH, Chen ZW, Du XY. Analysis of microsatellite instability in CRISPR/Cas9 editing mice. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 2017(797/798/799): 1-6. |
| [15] | Zhao DD, Li J, Li SW, Xin XQ, Hu MZ, Price MA, Rosser SJ, Bi CH, Zhang XL. Glycosylase base editors enable C-to-A and C-to-G base changes. Nature Biotechnology, 2021, 39(1): 35-40. DOI:10.1038/s41587-020-0592-2 |
| [16] | Kuscu C, Parlak M, Tufan TR, Yang JK, Szlachta K, Wei XL, Mammadov R, Adli M. CRISPR-STOP: gene silencing through base-editing-induced nonsense mutations. Nature Methods, 2017, 14(7): 710-712. DOI:10.1038/nmeth.4327 |
| [17] | Komor AC, Kim YB, Packer MS, Zuris JA, Liu DR. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016, 533(7603): 420-424. DOI:10.1038/nature17946 |
| [18] | 郭玲玲. λRed重组和CRISPR/Cas9技术在构建禽病原性大肠杆菌ahpF、yfgC基因突变株中的运用. 扬州大学硕士学位论文, 2016. |
| [19] | Karlinsey JE. Λ-red genetic engineering in Salmonella enterica serovar typhimurium. Methods in Enzymology, 2007(421): 199-209. |
| [20] | Zhang XN, Huang Y, Cao W, Hou Y, Zhao WQ, Guan LY, Chen SM, Wu YM. Design and construction of red proteins expressing in mammalian cell nucleolus. Progress in Modern Biomedicine, 2008, 8(10): 1809-1812. (in Chinese) 张晓楠, 黄勇, 曹蔚, 侯颖, 赵玮钦, 关璐媛, 陈苏民, 吴元明. Red系统三种蛋白在哺乳动物细胞核中定位表达的设计和构建. 现代生物医学进展, 2008, 8(10): 1809-1812. |
| [21] | Wang HL, Feng EL, Shi ZX, Yao X, Su GF, Huang LY. Quick knockout of Shigella flexneri asd gene with Red system. Bulletin of the Academy of Military Medical Sciences, 2002, 26(3): 172-175. (in Chinese) 王恒樑, 冯尔玲, 史兆兴, 姚潇, 苏国富, 黄留玉. 用Red系统快速敲除痢疾杆菌asd基因. 军事医学科学院院刊, 2002, 26(3): 172-175. DOI:10.3969/j.issn.1674-9960.2002.03.004 |
| [22] | Zhao YJ, Wang AX, Jia Q, Hua ZC. Expression of tomato senescence-associated protein SENU3 in Escherichia coli and preparation of its polyclonal antibody. Plant Physiology Communications, 2005, 41(2): 208-210. (in Chinese) 赵永娟, 王傲雪, 贾琪, 华子春. 番茄衰老相关蛋白SENU3在大肠杆菌中的表达及其抗体制备. 植物生理学通讯, 2005, 41(2): 208-210. |
| [23] | Zhang SM, Wang Y. The study of the human endothelial cell specific molecule-1 expression in MEK transfected endothelial cells. Acta Universitis Medicinalis Anhui, 2004, 39(1): 73. (in Chinese) 张素梅, 汪渊. MEK转染的内皮细胞表达人内皮细胞特异性分子-1的初步研究. 安徽医科大学学报, 2004, 39(1): 73. DOI:10.3969/j.issn.1000-1492.2004.01.029 |
| [24] | Tan KQ, Ma XY, Cui YY, Tian ZM, Deng D. Construction of enterotoxigenic E. coli LT knockout strain using CRISPR/Cas9 and λ-red cascaded technology. China Animal Husbandry & Veterinary Medicine, 2020, 47(3): 666-675. (in Chinese) 檀克勤, 马现永, 崔艺燕, 田志梅, 邓盾. 利用CRISPR/Cas9和λ-Red级联技术构建产肠毒素大肠杆菌LT敲除菌株. 中国畜牧兽医, 2020, 47(3): 666-675. |
| [25] | Kluesner MG, Nedveck DA, Lahr WS, Garbe JR, Abrahante JE, Webber BR, Moriarity BS. EditR: a method to quantify base editing from Sanger sequencing. The CRISPR Journal, 2018, 1(3): 239-250. DOI:10.1089/crispr.2018.0014 |
| [26] | Zheng K, Wang Y, Li N, Jiang FF, Wu CX, Liu F, Chen HC, Liu ZF. Highly efficient base editing in bacteria using a Cas9-cytidine deaminase fusion. Communications Biology, 2018(1): 32. |
| [27] | Li Y, Zhang XX, Guo MZ, Wang SY, Zhang KS, Ruan HH. Deletion of sopB gene of Salmonella typhimurium LT2 by λRed recombination system. Biotechnology Bulletin, 2014(9): 171-177. (in Chinese) 李晔, 张西轩, 郭梦征, 王素英, 张坤生, 阮海华. 利用λRed重组系统敲除鼠伤寒沙门氏菌sopB基因. 生物技术通报, 2014(9): 171-177. |
| [28] | Tian SC, Li YY, Tang T. Research progress on activation and regulation mechanism of SlyA in Salmonella typhimurium. Modern Preventive Medicine, 2019, 46(21): 3985-3988. (in Chinese) 田思成, 李泳榆, 唐田. 鼠伤寒沙门菌SlyA的激活及调控机制研究进展. 现代预防医学, 2019, 46(21): 3985-3988. |
| [29] | Aguirre AJ, Meyers RM, Weir BA, Vazquez F, Zhang CZ, Ben-David U, Cook A, Ha G, Harrington WF, Doshi MB, Kost-Alimova M, Gill S, Xu H, Ali LD, Jiang GZ, Pantel S, Lee Y, Goodale A, Cherniack AD, Oh C, Kryukov G, Cowley GS, Garraway LA, Stegmaier K, Roberts CW, Golub TR, Meyerson M, Root DE, Tsherniak A, Hahn WC. Genomic copy number dictates a gene-independent cell response to CRISPR/Cas9 targeting. Cancer Discovery, 2016, 6(8): 914-929. DOI:10.1158/2159-8290.CD-16-0154 |
| [30] | Hart T, Chandrashekhar M, Aregger M, Steinhart Z, Brown KR, MacLeod G, Mis M, Zimmermann M, Fradet-Turcotte A, Sun S, Mero P, Dirks P, Sidhu S, Roth FP, Rissland OS, Durocher D, Angers S, Moffat J. High-resolution CRISPR screens reveal fitness genes and genotype-specific cancer liabilities. Cell, 2015, 163(6): 1515-1526. DOI:10.1016/j.cell.2015.11.015 |
| [31] | Wang T, Birsoy K, Hughes NW, Krupczak KM, Post Y, Wei JJ, Lander ES, Sabatini DM. Identification and characterization of essential genes in the human genome. Science, 2015, 350(6264): 1096-1101. DOI:10.1126/science.aac7041 |
| [32] | Wang T, Yu HY, Hughes NW, Liu BX, Kendirli A, Klein K, Chen WW, Lander ES, Sabatini DM. Gene essentiality profiling reveals gene networks and synthetic lethal interactions with oncogenic ras. Cell, 2017, 168(5): 890-903. DOI:10.1016/j.cell.2017.01.013 |
| [33] | Doublet B, Douard G, Targant H, Meunier D, Madec JY, Cloeckaert A. Antibiotic marker modifications of lambda Red and FLP helper plasmids, pKD46 and pCP20, for inactivation of chromosomal genes using PCR products in multidrug-resistant strains. Journal of Microbiological Methods, 2008, 75(2): 359-361. DOI:10.1016/j.mimet.2008.06.010 |
| [34] | Fu XA, Zhang DX, Zhou W, Yu LH, Tian CL, Liu MC. Applications of λ red recombination in bacteria and analysis of influencing factors. Progress in Veterinary Medicine, 2015, 36(1): 91-95. (in Chinese) 付喜爱, 张德显, 周维, 于立辉, 田春莲, 刘明春. 细菌λ Red重组技术的应用及其影响因素. 动物医学进展, 2015, 36(1): 91-95. DOI:10.3969/j.issn.1007-5038.2015.01.022 |
| [35] | Madyagol M, Al-Alami H, Levarski Z, Drahovská H, Turňa J, Stuchlík S. Gene replacement techniques for Escherichia coli genome modification. Folia Microbiologica, 2011, 56(3): 253-263. |
| [36] | Wang Y, Xu Y, Chen N, Xu XY, Liu WF, Tao Y. Novel efficient strategy for λ-Red-mediated gene knock-out/in in Escherichia coli using SCLM system. Microbiology China, 2015, 42(4): 699-711. (in Chinese) 王瑶, 许杨, 陈楠, 徐欣怡, 刘伟丰, 陶勇. 在大肠杆菌中利用SCLM系统进行高效率λ-Red基因敲除/整合的新策略. 微生物学通报, 2015, 42(4): 699-711. |
