赵立斌, 徐魁

 , 连英丽, 颜庆云, 贺志理
, 连英丽, 颜庆云, 贺志理

中山大学环境科学与工程学院环境微生物组学研究中心, 南方海洋科学与工程广东省实验室(珠海), 广东 广州 510006
收稿日期:2020-06-26;修回日期:2020-08-28;网络出版日期:2020-10-22
基金项目:广东省自然科学基金(2018A030313788);国家自然科学基金(91951207,31770539)
*通信作者:Kui Xu, E-mail: xukui@mail.sysu.edu.cn;
Zhili He, E-mail: hezhili@mail.sysu.edu.cn.
摘要:原绿球藻(Prochlorococcus)作为海洋丰度最高的浮游植物,对海洋生态系统的物质循环和能量流动起着重要的驱动作用。原绿球藻生长和光合作用活性容易受到环境胁迫的影响,进而影响整个海洋生态系统的稳定性。因此,研究原绿球藻应对环境胁迫的响应机制具有重要的生态意义。原绿球藻主要通过分化出不同的生态型来适应不同光照和营养盐的海洋环境,但仍然会很难快速适应各种突如其来的海洋环境变化。本文从原绿球藻应对环境胁迫的角度,探讨了其生理和分子响应机制的最新研究进展,包括光系统Ⅰ循环电子传递在光照变化时发挥的重要作用,通过RNA快速响应而调控基因表达应对环境胁迫,以及在辅助异养细菌的保护下应对活性氧的胁迫等。本文也展望了原绿球藻对环境胁迫响应的生理和分子机制的未来研究方向,旨在为原绿球藻抗逆机制的深入研究提供参考。
关键词:原绿球藻(Prochlorococcus)抗逆机制光系统Ⅰ循环电子传递藻菌共生调控RNA
Physiological and molecular mechanisms of Prochlorococcus in response to environmental stresses
Libin Zhao, Kui Xu

 , Yingli Lian, Qingyun Yan, Zhili He
, Yingli Lian, Qingyun Yan, Zhili He

Southern Marine Science and Engineering Guangdong Laboratory(Zhuhai), Environmental Microbiomics Research Center, School of Environmental Science and Engineering, Sun Yat-sen University, Guangzhou 510006, Guangdong Province, China
Received: 26 June 2020; Revised: 28 August 2020; Published online: 22 October 2020
Foundation item: Supported by the National Natural Science Foundation of Guangdong Province of China (2018A030313788) and by the National Natural Science Foundation of China (91951207, 31770539)
Abstract: As the most abundant phytoplankton in the marine environment, Prochlorococcus plays a vital role in driving element cycle and energy flow of marine ecosystem. Numerous studies indicate that the growth and photosynthetic activity of Prochlorococcus are vulnerable to environmental stresses, thus resulting in the damage to marine ecosystem stability. Therefore, it is crucial to study the responses of Prochlorococcus to environmental stresses for maintaining the marine ecological stability. Generally, Prochlorococcus could adapt to different light and nutrient conditions in the ocean by differentiating into different ecotypes. However, they cannot cope with sudden changes in the marine environment. Here, we review the progress on the responses of Prochlorococcus at physiological and molecular level to environmental stresses, which includes the important role of cyclic electron transport around photosystem Ⅰ in light changes, RNA-regulated gene expression, and the protection Prochlorococcus against oxidative stress by heterotrophic bacteria. Moreover, we discuss the future directions for studies on Prochlorococcus responding to environmental stresses, aiming to provide fundamental bases to further explore anti-stress mechanisms of Prochlorococcus.
Keywords: Prochlorococcusresistance mechanismcyclic electron transport around photosystem Ⅰalgae-bacteria symbiosisregulatory RNA
原绿球藻是海洋中直径最小(0.5–0.7 μm)的原核单细胞浮游植物,是地球上丰度最高的光合生物,其叶绿素含量约占整个海洋叶绿素总量的50%[1-2]。据估计原绿球藻每年可固定约40亿t碳[2],相当于全球所有农作物每年净固定的碳总量[3],对全球碳循环起着举足轻重的作用。与此同时,原绿球藻作为全球重要的初级生产者,特别是在寡营养盐海域(水体中硅、磷、氮等营养盐缺乏,南北纬20°以内的太平洋开放海域),通过主动外排有机物[4]或捕食作用等,为该水域其他生物提供碳(C)源和氮(N)源等营养物质,对整个海洋食物网和能量流起着重要的驱动作用[5]。
由于原绿球藻生境相比于其他浮游植物的生境更稳定,原绿球藻通过分化出不同的生态型来适应不同海域的较稳定的光照和营养盐等[6],从而导致其缺乏其他浮游植物抵抗环境胁迫的典型调节机制[7]。原绿球藻受波浪扰动在垂直方向的移动会导致其受到光照变化的胁迫,强光会快速损害原绿球藻的光反应系统或诱导出活性氧导致原绿球藻的失活[8]。同时,随着人类活动对海洋环境的影响加剧,如温室效应和海洋污染等都对不同生态型原绿球藻产生影响,已经有研究表明原绿球藻对痕量有机物[9]、多环芳烃[10]、铜[11]等多种污染物极其敏感,致使原绿球藻所面对的环境胁迫压力也越来越大。有研究发现塑料渗滤液可以导致原绿球藻光合色素生物合成基因的转录降低[12]。而在有机污染物条件下,原绿球藻的光合基因表达量显著下降,表明有机污染物影响了原绿球藻的光合功能[13]。由于原绿球藻缺乏编码过氧化氢酶的基因,海洋中的H2O2也对原绿球藻的生存造成了威胁。本文将从光系统Ⅰ循环电子传递应对光照变化、RNA调控基因表达、藻菌共生应对H2O2胁迫3个方面阐释原绿球藻适应环境胁迫并取得优势的生理和分子响应机制。
1 原绿球藻的分布 原绿球藻是海洋寡营养盐区域进行放氧光合作用且丰度最高的浮游植物[14],在南北纬40°之间营养盐较少的水域占主导地位,在较冷的水域生长不良,如图 1-A[15-16]。原绿球藻在海洋中的垂直分布也十分广泛,如图 1-B,包括从海洋表面到真光层最底部,最深可达200 m[17]。原绿球藻在夏天温度适宜时,在南太平洋环流的细胞丰度可以达到1.6×1013 cells/m3[7],其垂直分布在水深60 m左右的时候丰度最高可以达到3.5×105 cells/m3[18]。原绿球藻广泛分布于海洋热带和亚热带的透光层,是很多水域的绝对优势种,其在我国南海和东海外海也大量分布,同时季节性地分布于黄海东南部和珠江入海口[19-21]。
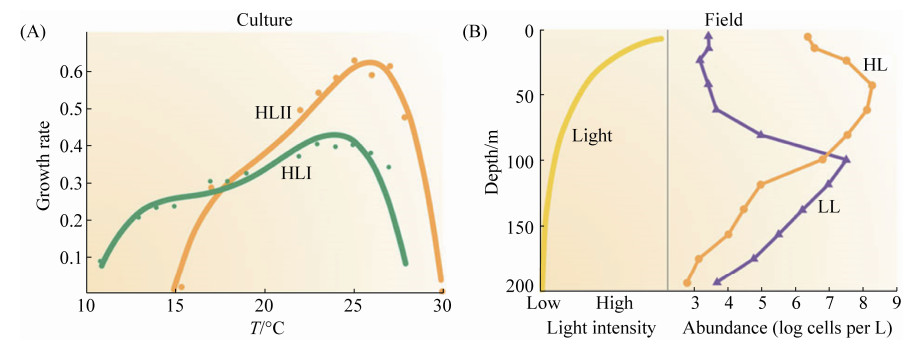 |
| 图 1 原绿球藻的生长速率及深度分布 Figure 1 Growth rate and depth distribution of Prochlorococcus. A: The growth rate of two high-light adapted Prochlorococcus at different temperatures; B: Changes in light intensity and abundance of Prochlorococcus at different depths of the ocean (modified from Biller[5]). HL: High-light adapted ecotype; LL: Low-light adapted ecotype. |
| 图选项 |
为适应不同垂直海域光照,原绿球藻进化出不同的生态型类群(指同一物种由于长期生活在不同生态环境而产生趋异适应和进化的不同类群,尽管它们在生理、生化、生态及遗传上可能会有差异,但仍为同一物种),如高光适应型(high-light adapted ecotype,HL)原绿球藻MED4,低光适应型(low-light adapted ecotype,LL)原绿球藻MIT9313[22]。然而,其他环境因素,如温度、营养物质和竞争对手的丰度,也影响生态型的分布,HL和LL生态型可以进一步分为6个不同的亚类,2个属于高光适应型(HLI和HLII),4个属于低光适应型(LLI–LLIV)[23]。在这个分类中,MED4和MIT9313代表 2个关系最远的原绿球藻分支(HLI与LLIV),虽然它们的16S rRNA有97.5%的同源性,但它们的蛋白编码潜力不同,MED4有1955个注释蛋白编码基因,MIT9313有2843个注释蛋白编码基因,共有的基因只有1447个[24]。原绿球藻6个分支的代表性菌株及其分布如表 1所示。
表 1. 原绿球藻的主要分支及其分布 Table 1. The main branches and distribution of Prochlorococcus
| Clades | Representative strains | Habitats | References |
| HL Ⅰ | MED4, MIT9515 | Distributed in the upper-middle euphotic zone, typically in the subtropical ocean, and the distribution is shifted to higher latitudes. | [25] |
| HL Ⅱ | AS9601, MIT9215, MIT9312, MIT9301 | Often found throughout the euphotic zone, and typically among the most abundant clade in the water column. Especially abundant at lower latitudes consistent with their higher optimum growth temperature relative to HLI cells. | [25-26] |
| LL Ⅰ | NATL1A, NATL2A | Typically most abundant in the middle euphotic zone of stratified waters, and often remain abundant in mixed waters throughout the water column owing to their ability to tolerate light shock. | [23] |
| LL Ⅱ | SS120 | Usually found in the middle-lower euphotic zone. | [23, 27] |
| LL Ⅲ | MIT9211 | Usually found in the middle-lower euphotic zone. | [23, 27] |
| LL Ⅳ | MIT9303, MIT9313 | Typically most abundant near the base of the euphotic zone and highly sensitive to light shock. | [23] |
表选项
2 原绿球藻光合作用特性及其适应光照变化的机制 原绿球藻具有广泛的基因组流线型和非常小的细胞尺寸,其基因组高度缩减,这些适应被认为在营养有限的环境中生存提供了竞争优势[7]。原绿球藻细胞(直径0.5–0.7 μm)和基因组(1.70–2.56 Mb)是所有已知的光合自养生物中最小的[28-29]。与其他蓝藻相比,原绿球藻丢失了编码负责捕光的富氮藻胆体的基因[30]。它具有与低铁诱导的铁胁迫诱导蛋白(ISIA)同源的原叶绿素结合蛋白(Pcb),这些蛋白可以作为原绿球藻中光系统Ⅰ (Photosystem Ⅰ,PS Ⅰ)的捕光蛋白[30]。在捕光色素方面,原绿球藻用二乙烯基叶绿素a和b (DV Chl a, b)代替单乙烯基叶绿素(MV Chl),这也导致了原绿球藻光合作用机制与其他蓝藻有所不同[23, 31]。MV Chl a和DV Chl a的结构式分别如图 2-A和2-B所示,DV Chl a对应MV Chl a的8号碳原子的乙基转变为乙烯基。色素结构的改变导致了光能吸收性质上的差异,如图 2-C所示,DV Chl a在蓝光波段的吸收峰相比于MV Chl a红移了7–10 nm,这使得DV Chl a更有效地吸收蓝光,帮助原绿球藻适应深海只有微弱蓝光的环境。同时,通过对原绿球藻的检测,已经证明DV Chl a和二乙烯基脱镁叶绿素(DV-Phe)分别是原绿球藻PS Ⅰ和PS Ⅱ的初级电子受体[32-33]。
 |
| 图 2 两种叶绿素a的结构式、吸收光谱以及原绿球藻PSII结构 Figure 2 Two kinds of chlorophyll a structural formulas, absorption spectrum and PSII structure of Prochlorococcus. A: MV Chl a structural formula; B: DV Chl a structural formula; C: Comparison of absorption spectra of two kinds of chlorophyll a in diethyl ether, acetone and methanol at room temperature (dashed line is MV Chl a, solid line is DV Chl a, modified from Komatsu[33]); D: Photosystem Ⅱ (PS Ⅱ) structure diagram of Prochlorococcus protothecoides (modified from Biller[5]). Pcb: prochlorophyte chlorophyll-binding proteins; CP43/CP47: antenna protein; OEC: oxygen-evolving complex; Cyt b559: cytochrome b559. |
| 图选项 |
原绿球藻的主要捕光复合体是由天线蛋白复合体Pcb组成,如图 2-D。这种天线复合物收集光能,并将能量传递到PS Ⅱ核心天线蛋白(CP43和CP47),然后进入光系统Ⅱ (Photosystem Ⅱ,PS Ⅱ)反应中心,包括多种蛋白质如D1、D2以及细胞色素b559。据报道,原绿球藻可以根据光线条件调整Pcb单元的数量和类型[34]。原绿球藻光合作用的光反应过程主要包括从水到NADP+的矢量电子转移,涉及类囊体膜中存在的蛋白质复合物,即PS Ⅱ、细胞色素b6f复合物、PS Ⅰ和铁氧还蛋白NADP+还原酶,与可溶性载体如质体醌(Plastoquinone,PQ)、质体蓝蛋白和铁氧还蛋白(Fd)连接。原绿球藻的吸光效率高于其他的海洋浮游植物,这种高的吸光效率不仅与原绿球藻大的比表面积有关,也得益于其独特的光合色素[31]。因此,原绿球藻可以在比其他大多数浮游植物所需的光照强度更低的条件下生存,其在海洋垂直方向的分布基本上达到了海洋光合生物的下限。
原绿球藻的类胡萝卜素(car)在其光合作用中也承担着重要角色,研究表明原绿球藻中的类胡萝卜素主要是叶黄素(lutein),其次是β-胡萝卜素,α-胡萝卜素和玉米黄素(zeaxanthin)等[35-38],它们最关键的功能是参与清除活性氧(ROS)和光保护,例如β-胡萝卜素就可以消除细胞内的单线态氧。叶黄素循环是一种重要的光保护机制,它通过环氧化叶黄素在强光下转化为脱环氧化形式以耗散多余能量,以及脱环氧化叶黄素在弱光或黑暗中的环氧化反应[39],可以避免过量的光导致光系统的光抑制和失活。Kulk等表明当光照强度增加时,原绿球藻中玉米黄素/DV-Chl a的比值明显上升[40],而玉米黄素是叶黄素循环的载体之一,暗示原绿球藻可能通过提高玉米黄素含量来进行光保护。但该玉米黄素具体的作用机制还知之甚少。
低光型原绿球藻相比于高光型原绿球藻具有高的DV-Chl b/Chl a,而生长光强升高时原绿球藻的Car/Chl a也会升高[40-41],这说明细胞中Chl和Car都在原绿球藻适应高光中起着作用。但仅仅靠色素含量的改变难以适应海洋表面的高光强,过高的光照强度会产生光抑制现象,导致反应中心的D1蛋白的氧化损伤,破坏PS Ⅱ反应中心,从而降低量子效率和最大光合速率[42-43]。浮游植物在面对过度光照的胁迫时,会产生一些抗逆机制,如捕光色素相关的非光化学淬灭、状态转换、环式电子传递等[44]。捕光色素相关的非光化学淬灭可以将过剩的光能耗散掉,从而避免其传递给氧产生活性氧而造成对PS Ⅱ的损伤。状态转换是指能量在2个反应中心进行重新分配以达到平衡,被认为在蓝藻适应光变中起着重要作用[45]。但原绿球藻没有藻胆体,没有与藻胆体相关的淬灭机制和状态转换,而目前也没有发现与乙烯基叶绿素蛋白复合体相关的淬灭机制和状态转换[31, 46-47]。Bailey等发现在铁限制的条件下,高光能诱导出原绿球藻明显的非光化学淬灭[48]。而Xu等用蓝光提供光源,通过不断提高光强至原绿球藻PS Ⅱ反应中心逐渐完全关闭,发现没有明显的非光化学淬灭[47]。这表明在正常的生长条件下,原绿球藻的非光化学淬灭似乎非常有限,强光可能很容易破坏原绿球藻的D1蛋白进而破坏PS Ⅱ反应中心。由于PS Ⅰ循环电子传递形成跨膜质子梯度产生的ATP,在PSII受损伤时,维持高的能量产出,不仅为D1蛋白的合成供能,还是其他生理活动的能量来源。因此,PSI循环电子传递在原绿球藻的生理生态过程中承担着不可缺少和至关重要的作用。
蓝细菌中NDH依赖的PS Ⅰ循环电子传递途径是主要途径,其机制如图 3所示。Fd接受PS Ⅰ传递的电子,并将之进一步传递给NDH复合物,并由PQ质体醌将电子转移至细胞色素b6f复合物,与此同时,NDH将质子从细胞质侧泵到类囊体腔侧,形成跨膜质子梯度驱动ATP生成。在目前已测序的所有原绿球藻亚种中,都保留着ndhD1和ndhF1基因[49]。ndhD1和ndhF1基因是蓝藻NDH依赖的PS Ⅰ循环电子传递必不可少的关键基因[49]。这说明原绿球藻中存在着NDH依赖的PS Ⅰ循环电子传递途径。但目前对于原绿球藻PS Ⅰ循环电子传递的研究还很匮乏,其途径和生理意义还不清楚。
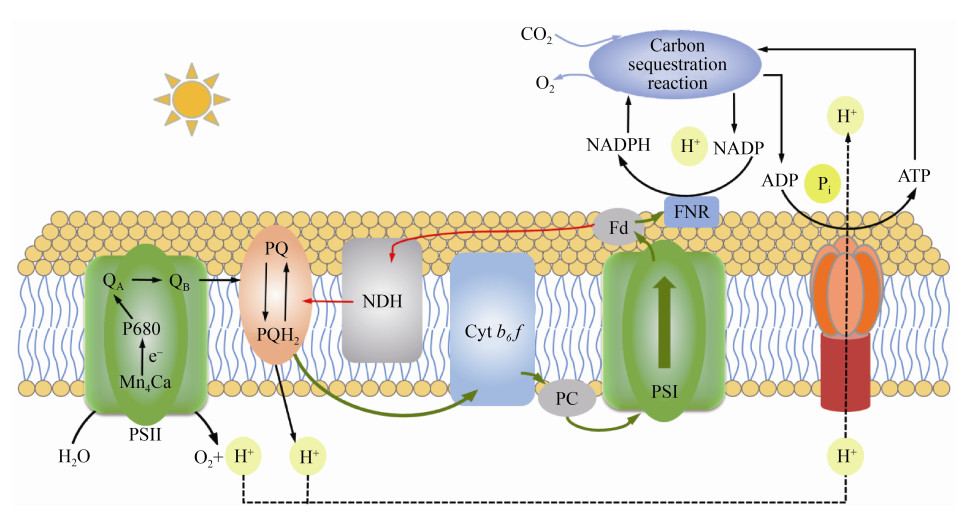 |
| 图 3 蓝细菌光合作用系统电子传递示意图 Figure 3 Schematic diagram of electron transfer in the cyanobacteria photosynthesis system. Black and green arrows indicate linear electron transfer paths, and red arrows indicate circulating electron transfer paths. QA: primary plastoquinone electron acceptor of PS Ⅱ; QB: secondary plastoquinone electron acceptor of PS Ⅱ; PQ: plastoquinone; NDH: NADH dehydrogenase complex; Cyt b6f: cytochrome b6f complex; PC: plastocyanin; Fd: ferredoxin; FNR: ferredoxin-NADP reductase. |
| 图选项 |
Zore等通过测定原绿球藻光合关键蛋白含量,发现原绿球藻MIT9313有较高的PS Ⅰ: PS Ⅱ比值(达到9),推测MIT9313通过诱导PS Ⅰ循环电子传递维持在低光下的快速生长[50]。Bonisteel等发现原绿球藻适应高光的能力与其清除D1蛋白的速率有关[43]。Fridman等用最新一代的叶绿素荧光/吸收仪测定原绿球藻PS Ⅰ氧化还原的数据,该数据直接反映了原绿球藻PS Ⅰ循环电子传递[51],通过测定病毒感染的原绿球藻PS Ⅰ氧化还原速率,发现原绿球藻可诱导PS Ⅰ循环电子传递来应对病毒感染。
PS Ⅰ结构域较大,必须从细胞色素b6f复合物中扩散的还原型质体蓝素分子进一步扩散,才能将电子传递到PS Ⅰ复合物的受体侧,这将改变线性电子流动的速率[52]。铁氧还蛋白以循环电子传输为目的的扩散也是如此。在原绿球藻中,PS Ⅰ结构域的大小有可能优化线性和循环电子流之间的平衡,从而根据生物的代谢需要控制ATP和NADPH的产生。例如,已经证明在植物中,主要包含PS Ⅱ和细胞色素b6f复合物的结构域大小差异可能对循环和线性电子流产生显著影响[53]。但是,在可以得出关于原绿球藻中循环和线性电子流动的结论之前,需要进一步阐明PS Ⅱ和细胞色素b6f复合物的结构域。
除了PS Ⅰ循环电子传递,原绿球藻还具有光合类囊体膜的结构适应性。Macgregor等利用原子力显微镜对MED4、MIT9313和SS120三种不同生态型的原绿球藻的PS Ⅰ膜结构进行了成像,揭示了一系列特殊的PS Ⅰ构型[52]。MED4的PS Ⅰ结构域松散地堆积在类囊体膜中,三聚体PS Ⅰ复合物较少,有更多的单体和二聚体,当将其暴露于低光强下后,三聚体复合物的数量显著增加。低光MIT9313中的PS Ⅰ被组织成紧密堆积的伪六角晶格,增加了PS Ⅰ在类囊体膜中的丰度,从而实现对光的捕获最大化。而SS120有一种不同的策略来应对低光水平,其PS Ⅰ三聚体被Pcb蛋白的18元环包围,增加了三聚体之间的距离,实现较大的捕光效率。这些原绿球藻中PS Ⅰ的结构适应性可以优化能量捕获和电子传输,还可以改变线性电子流动的速率,提高原绿球藻应对光照变化的适应性。
3 原绿球藻的调控RNA 到目前,已经有12株原绿球藻分离株的基因组被完全测序,显示出基因组紧凑和流线型的趋势[23, 46, 54]。这种基因组流线型已被认为是海洋浮游细菌对寡营养盐环境的典型适应[55]。由于基因组的简化,原绿球藻基因组中的鸟嘌呤和胞嘧啶残基被耗尽,密集地填充着小的基因间隔区,并且只包含几个编码参与转录、信号转导和基因表达调控的蛋白基因[23, 46, 54],不存在许多其他细菌广泛分布的双组分系统和DNA结合蛋白。尽管原绿球藻的基因组密集,转录蛋白调节因子的数量相对较少,如MED4和MIT9313分别只有5和8个σ因子,6和7个反应调节因子,以及6和10个组氨酸激酶[29]。但是,原绿球藻仍然能够有效地适应环境胁迫,这可能与原绿球藻中相对大量的非编码RNA (ncRNA)有关,调控RNA可能在原绿球藻基因表达中起重要作用[56]。在一昼夜周期的某个时间点,Waldbauer等检测到了MED4中所有基因中73%的反义转录本,这一百分比大大高于之前报道的任何细菌中与反义RNA (asRNA)分子相关的基因比例,而且小RNA (sRNA)的丰度相对较高,平均为相应mRNA浓度的35%[57]。
已经有研究证明asRNA可以介导mRNA稳定性的调控,直接影响mRNA的半衰期[58]。在噬菌体感染时,asRNA通过形成asRNA-mRNA双链,屏蔽了核糖核酸内切酶E (RNase E)的识别位点,从而实现对mRNA的保护。Voigt等发现了与原绿球藻MED4和MIT9313的asRNA相关的1875个和2613个基因,证实了原绿球藻中潜在的高度反义转录,并分析了它们在不同胁迫条件下的差异表达特征[24]。在氮限制条件下,约300-nt的asRNA诱导表达,这可能在cynS (编码一种氰化酶,可将氰酸盐转化为二氧化碳和氨)的调控中起着重要作用;在铁限制条件下,asRNA可以调节yocE (编码一种Ⅱ型脂肪酸去饱和酶)的表达;由psbY编码的锰结合光系统Ⅱ蛋白Y的mRNA被一个120个核苷酸长的asRNA覆盖,psbY的asRNA可能使psbY的基因表达调控解耦。这些结果表明原绿球藻中存在一个通过asRNA调控的非常复杂的网络,也说明了调控性RNA在原绿球藻基因调控中具有重要作用。
sRNA存在于生命的所有领域,并调节目标基因的表达[59]。与其他基因表达调控因子(如转录因子)相比,sRNA在调控动力学方面表现出一些独特的特征,即sRNA的调控比转录因子的调控更快地导致靶基因蛋白产物水平的下降[60]。对细菌和古菌中的sRNA的研究表明,它们可以通过封闭或暴露核糖体结合位点或核糖核酸酶裂解位点,在转录后水平调节基因的表达[59]。已经有很多研究揭示了不同的sRNA作用机制,它们通常需要RNA伴侣(RNA chaperones),如Hfq或ProQ来实现其完整的功能[59, 61-62]。Hfq或ProQ促进了sRNA与其靶mRNA的相互作用,因此参与了许多重要的调控过程,包括sRNA介导的翻译调控[63]。但在包括MED4在内的12株测序的原绿球藻中,有10株缺失编码Hfq RNA伴侣的基因,这与其他完全测序的蓝藻形成对比,它在原绿球藻进化过程中的丢失可能是该种群中存在RNA-RNA相互作用的新机制。
已经有多项研究提供了有关MED4基因的转录组信息,并在一定程度上提供了MIT9313基因的转录组信息。Axmann等在原绿球藻MED4中首次鉴定出7个sRNA,并被标记为Yfr1–Yfr7[64]。其中Yfr7与6S RNA同源[65],已知后者在大肠杆菌中具有调控功能。Yfr1在其他蓝藻中也有同源物,是在多种胁迫条件下生长所必需的[66],在MED4中具有调控外膜蛋白PMM1119和PMM1121的功能[67]。随后,Steglich等又发现了MED4中14个新的sRNA,并标记为Yfr8–Yfr21[56]。同时,他们发现当原绿球藻从黑暗移至光下时,Yfr11和Yfr19转录表达下调了一倍以上,而Yfr20转录上调表达;当原绿球藻在光照下加入DCMU (一种光合抑制剂,可以抑制从PS Ⅱ上的Q向PQ的电子传递)时,Yfr11和Yfr19发生了上调表达,这表明光合作用电子传递链的氧化还原状态与这些sRNA的表达之间存在联系。用噬菌体对原绿球藻进行感染时,Yfr9和Yfr14在感染初期上调表达,Yfr15在感染中后期上调表达,且上调幅度最高,这提示Yfr15可能在噬菌体与宿主的相互作用过程发挥重要作用。Lambrecht等的研究表明,光照条件的变化会调节Yfr10的表达,但光变条件下参与调控Yfr10基因表达的调控因子尚不清楚[68]。Yfr2可能调控光合作用和呼吸作用相关的基因表达,同时还对其他调控因子如转录调节因子rbcR、传感器组氨酸激酶nblS进行调控,这也表明流线型MED4基因组中剩下的不可或缺的基于蛋白的调节在很大程度上是由sRNA控制的。两个Yfr2 sRNA同源物在转移到高光或缺氮的过程中高度上调,其表达受GntR家族转录调节因子PMM1637的调控[69]。
虽然以上研究均表明了sRNA对于原绿球藻在不同的光照和环境胁迫下的调控中发挥了重要作用,但其具体的作用机制目前尚不清楚。我们可以推测的是,不同的sRNA与它们的目标调控基因之间存在一个复杂的、相互联系的网络。大多数的原绿球藻中没有Hfq,这些sRNA在没有RNA伴侣的支持下发挥作用,或许存在与其他蓝细菌不同的作用机制,亦或许有一些新的RNA伴侣尚未发现,这还有待进一步的研究和探索。
4 异养细菌对原绿球藻的保护作用 原绿球藻的基因组测序分析结果表明,原绿球藻基因组中没有过氧化氢酶编码基因[29, 70],这导致其对H2O2胁迫非常敏感(生长允许浓度 < 200 nmol/L)。而海洋中复杂的环境条件,会通过多种方式产生H2O2 (图 3),例如真核浮游植物可以利用跨膜NADPH氧化酶(NOX)来产生胞外O2–[71],而原核生物可以利用表面相关的动物血红素过氧化物酶(AHP)来实现这一功能[72]。O2–与溶解有机物(DOM)或金属反应产生H2O2。细胞内也有产生H2O2的机制,包括通过超氧化物歧化酶(SOD)歧化O2–,黄素蛋白中还原的黄素的自氧化[73],以及通过细胞表面局部氨基酸脱氨酶(AA-DA)来产生H2O2等[74]。已经有研究表明原绿球藻在面对光照变化和海洋污染等环境胁迫时,其细胞内也会产生H2O2[8, 75]。H2O2可以通过Fenton反应氧化Fe(II)进行非生物分解(图 4),其产物羟基自由基(HO·)是一种氧化电位很高的氧化剂,可以破坏蛋白质、脂质、DNA和大多数其他生物分子。正是由于这个原因,原绿球藻需要与异养细菌合作,借助异氧细菌帮助其清除环境中的H2O2[76]。
 |
| 图 4 海洋环境中异养细菌对原绿球藻的保护示意图 Figure 4 Schematic diagram of the protection of Prochlorococcus by heterotrophic bacteria in the marine environment. H2O2 production (green) and removal (red) (modified from Zinser[75]). AA-DA: amino acid deaminase; AHP: animal haem peroxidase; (C)DOM: (Chromophoric) dissolved organic matter; NOX: NADPH oxidase; FP: flavo-proteins; SOD: superoxide dismutase. |
| 图选项 |
为了解释原绿球藻与异养细菌的这种作用,有研究者提出了黑色皇后假说[77],该假说假定自由生活的微生物群落进化并维持某些基本功能的分工,一些生产型细胞具有生产公共产品的基本功能,使得非生产型细胞能够从这一活动中受益,并避免自己进行这项活动的成本。因为其他细胞有很强的选择性压力来避免H2O2的损害,所以只要原绿球藻能够依靠其他细胞进行保护,即使其自身不产生过氧化氢酶,也可以被附近的异养细菌保护而不受H2O2的影响。
一些研究已经证明几种异养细菌菌株能促进许多生态型原绿球藻的生长,并表明在这种情况下促进的机制是氧化应激的降低[7, 78]。Ma等最近的研究证实异养细菌可以通过减少氧化性物质保护原绿球藻氧化应激,并进一步证明这种相互作用可以促进原绿球藻对温度胁迫的积极响应,这种保护其免受环境胁迫的促进作用可以扩大微生物生态位[76]。另一项共培养转录组研究发现,即使H2O2浓度是亚致死的,异养细菌Alteromonas也可以为原绿球藻提供保护,异养细菌的存在降低了原绿球藻中众多应激反应基因的表达,包括编码高光诱导蛋白、DNA修复酶和烷基过氧化氢还原酶的编码基因[79],此外,在与原绿球藻共培养过程中,异养细菌中过氧化氢酶基因的表达增加,表明在共培养的情况下,这些异养细菌的辅助功能可能会增强。
微生物之间的相互作用影响着生态系统功能,尽管海洋微生物相互作用通常发生在纳米或微米尺度上,但它们最终影响了整个海洋生态系统和全球生物地球化学元素的循环[80]。Morris等发现现有微生物群落的活性可以将H2O2维持在原绿球藻生长允许的浓度小于200 nmol/L[81]。当海水中的微生物群落(包括原绿球藻)被去除,并且这些海水暴露在阳光下时,H2O2的浓度可以上升到较高水平(≥800 nmol/L),其可杀死在混合层中发现的所有生态型的原绿球藻[82]。唯一可以在这种H2O2浓度下生长的生态型MIT9313在混合层中从未发现,这在很大程度上是因为它对高光敏感[25, 83]。这些研究结果有力地支持了这样的观点,即如果没有现存的微生物群落,原绿球藻将不能在混合层中生存。
异养细菌中清除H2O2的酶主要有两类,一个是过氧化氢酶,它将2个H2O2分子转化为1个O2和2个H2O;另一个是过氧化物酶,它通过底物分子的氧化,将H2O2分子转化为H2O (图 4),一些过氧化氢酶(如KatG)也具有过氧化物酶活性,被称为过氧化氢酶-过氧化物酶。一些酶是与细胞表面相关的,例如动物血红素过氧化物酶(AHP),它也可以产生O2–,也可以直接将H2O2分解[72]。过氧化氢酶和过氧化物酶对需氧菌是常见的,因此很多海洋微生物可能会对H2O2的去除起到潜在的作用,例如Morris等的研究确定了33个过氧化氢酶阳性的异养菌株,它们可以帮助原绿球藻在平板上生长[70]。
当然,并不是所有的异养细菌都对原绿球藻的生长有益,Sher等通过将一些异养细菌与原绿球藻进行共培养,发现有些异养细菌对原绿球藻的生长有抑制或中性的影响,而这种影响的具体机制还不清楚,可能是通过分泌的化感物质或者是紧密接触介导的相互作用来实现的[84]。原绿球藻无疑也会以一些方式影响异养细菌,例如,某些菌株产生显著多样性的环肽二级代谢物[85],虽然它们在原绿球藻中的功能尚不清楚,但类似的化合物具有从抗生素到表面活性剂的多种功能,这可能会影响异养细菌群落的分布。此外,原绿球藻不断释放细胞外膜囊泡(~100 nm)[4],其中含有广泛的成分,包括脂质、蛋白质以及DNA和RNA的小片段,虽然这些囊泡的生态功能目前尚不清楚,但它们可能作为碳源在海洋食物网中移动或水平基因转移的载体。
5 结语和展望 原绿球藻具有重要的生态意义,其采用非常经济的生存方式,如很小的细胞、高度缩减的基因以及较少的蛋白调节因子等。较小的细胞体积可以提高其捕光的效率,基因组的缩减以及更多的依赖调控RNA而不是蛋白调节因子进行生理调控可以有效降低其对一些营养盐的需求,从而维持了它在寡营养盐区域的生存优势。也正是由于原绿球藻基因组的高度精简,在面对环境胁迫时表现得非常敏感。虽然已经分化出了不同的生态型来适应不同的光照条件,但这对于复杂的海洋环境和应对各种胁迫来说还远远不够,特别是原绿球藻在夏季通常能广泛的分布于近岸海域,其生存所面临的胁迫将兼有多样化和突发性的特点。这就需要我们从生态型适应机制以外,去探讨原绿球藻适应这些胁迫的各种机制。我们从原绿球藻的抗逆性的角度出发,探讨了其在光合生理以及调控RNA分子上的响应机制,也探讨了异养细菌在原绿球藻应对H2O2胁迫时的协同机制,使我们对原绿球藻应对环境胁迫的最新进展有了更全面的了解,为后续相关研究提供参考。
已有研究使我们对原绿球藻的抗逆性有了一定的认知,但其对环境胁迫的生理和分子响应机制仍需要在以下五方面进行更深入的探究(图 5)。
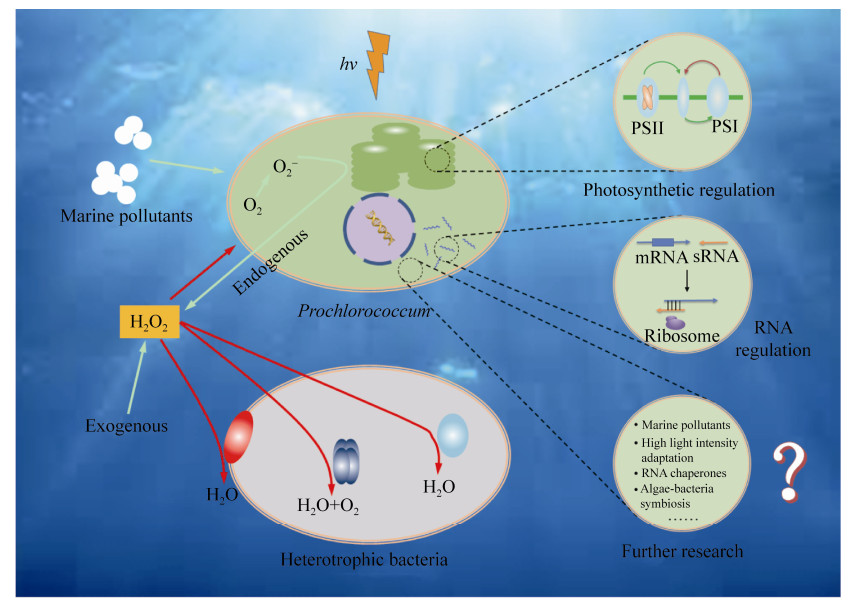 |
| 图 5 原绿球藻应对环境胁迫的生理和分子响应机制简图 Figure 5 A schematic presentation of possible physiological and molecular mechanisms of Prochlorococcum responses to environmental stresses. |
| 图选项 |
(1) 原绿球藻对光强、光照时间和光质的响应及其高光强(如海洋表面)的适应机制。
(2) 原绿球藻在低铁培养条件下,可以诱导出较明显的非光化学淬灭,这种非光化学淬灭与捕光色素和反应中心色素的关系及其诱导机制。
(3) 海洋污染物,特别是新型污染物(如纳米颗粒、微塑料、抗生素等)对原绿球藻生长和光合作用的影响及其响应机制。
(4) 原绿球藻主要依赖sRNA进行生理调节但缺乏RNA伴侣,进一步探究sRNA的作用机制并寻找新型RNA伴侣非常必要。
(5) 利用合成微生物生态学理论和方法,在实验室构建合成微生物群落,解析原绿球藻与其他微生物的互作网络。
References
| [1] | Schattenhofer M, Wulf J, Kostadinov I, Gl?ckner FO, Zubkov MV, Fuchs BM. Phylogenetic characterisation of picoplanktonic populations with high and low nucleic acid content in the North Atlantic Ocean. Systematic and Applied Microbiology, 2011, 34(6): 470-475. DOI:10.1016/j.syapm.2011.01.008 |
| [2] | Flombaum P, Gallegos JL, Gordillo RA, Rincón J, Zabala LL, Jiao NZ, Karl DM, Li WK, Lomas MW, Veneziano D, Vera CS, Vrugt JA, Martiny AC. Present and future global distributions of the marine Cyanobacteria Prochlorococcus and Synechococcus. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(24): 9824-9829. DOI:10.1073/pnas.1307701110 |
| [3] | Huston MA, Wolverton S. The global distribution of net primary production: resolving the paradox. Ecological Monographs, 2009, 79(3): 343-377. DOI:10.1890/08-0588.1 |
| [4] | Biller SJ, Schubotz F, Roggensack SE, Thompson AW, Summons RE, Chisholm SW. Bacterial vesicles in marine ecosystems. Science, 2014, 343(6167): 183-186. DOI:10.1126/science.1243457 |
| [5] | Biller SJ, Berube PM, Lindell D, Chisholm SW. Prochlorococcus: the structure and function of collective diversity. Nature Reviews Microbiology, 2015, 13(1): 13-27. DOI:10.1038/nrmicro3378 |
| [6] | Xiao XL, Zeng QL, Zhang R, Jiao NZ. 2018. Prochlorococcus viruses — from biodiversity to biogeochemical cycles. Science China Earth Sciences, 2018, 48(12): 1589-1599. (in Chinese) 肖喜林, 曾庆璐, 张锐, 焦念志. 原绿球藻病毒研究进展——从多样性到生物地球化学过程. 中国科学: 地球科学, 2018, 48(12): 1589-1599. |
| [7] | Partensky F, Garczarek L. Prochlorococcus: advantages and limits of minimalism. Annual Review of Marine Science, 2009, 2: 305-331. |
| [8] | Murphy CD, Roodvoets MS, Austen EJ, Dolan A, Barnett A, Campbell DA. Photoinactivation of photosystem Ⅱ in Prochlorococcus and Synechococcus. PLoS One, 2017, 12(1): e0168991. DOI:10.1371/journal.pone.0168991 |
| [9] | Echeveste P, Dachs J, Berrojalbiz N, Agustí S. Decrease in the abundance and viability of oceanic phytoplankton due to trace levels of complex mixtures of organic pollutants. Chemosphere, 2010, 81(2): 161-168. DOI:10.1016/j.chemosphere.2010.06.072 |
| [10] | Echeveste P, Agustí S, Dachs J. Cell size dependent toxicity thresholds of polycyclic aromatic hydrocarbons to natural and cultured phytoplankton populations. Environmental Pollution, 2010, 158(1): 299-307. DOI:10.1016/j.envpol.2009.07.006 |
| [11] | Mann EL, Ahlgren N, Moffett JW, Chisholm SW. Copper toxicity and cyanobacteria ecology in the Sargasso Sea. Limnology and Oceanography, 2002, 47(4): 976-988. DOI:10.4319/lo.2002.47.4.0976 |
| [12] | Tetu SG, Sarker I, Schrameyer V, Pickford R, Elbourne LDH, Moore LR, Paulsen IT. Plastic leachates impair growth and oxygen production in Prochlorococcus, the ocean's most abundant photosynthetic bacteria. Communications Biology, 2019, 2: 184. DOI:10.1038/s42003-019-0410-x |
| [13] | Fernández-Pinos MC, Vila-Costa M, Arrieta JM, Morales L, González-Gaya B, Pi?a B, Dachs J. Dysregulation of photosynthetic genes in oceanic Prochlorococcus populations exposed to organic pollutants. Scientific Reports, 2017, 7: 8029. DOI:10.1038/s41598-017-08425-9 |
| [14] | Partensky F, Blanchot J, Vaulot D. Differential distribution and ecology of Prochlorococcus and Synechococcus in oceanic waters: a review. Bulletin de l'Institut Océanographique de Monaco, 1999(19): 457-476. |
| [15] | Johnson ZI, Zinser ER, Coe A, McNulty NP, Woodward EMS, Chisholm SW. Niche partitioning among Prochlorococcus ecotypes along ocean-scale environmental gradients. Science, 2006, 311(5768): 1737-1740. DOI:10.1126/science.1118052 |
| [16] | Bouman HA, Ulloa O, Scanlan DJ, Zwirglmaier K, Li WKW, Platt T, Stuart V, Barlow R, Leth O, Clementson L, Lutz V, Fukasawa M, Watanabe S, Sathyendranath S. Oceanographic basis of the global surface distribution of Prochlorococcus ecotypes. Science, 2006, 312(5775): 918-921. DOI:10.1126/science.1122692 |
| [17] | Partensky F, Hess WR, Vaulot D. Prochlorococcus, a marine photosynthetic prokaryote of global significance. Microbiology and Molecular Biology Reviews, 1999, 63(1): 106-127. DOI:10.1128/MMBR.63.1.106-127.1999 |
| [18] | Lange PK, Brewin RJ, Dall'Olmo G, Tarran GA, Sathyendranath S, Zubkov M, Bouman HA. Scratching beneath the surface: a model to predict the vertical distribution of Prochlorococcus using remote sensing. Remote Sensing, 2018, 10(6): 847. DOI:10.3390/rs10060847 |
| [19] | Jiao NZ, Yang YH. Ecological studies on Prochlorococcus in China seas. Chinese Science Bulletin, 2002, 47(15): 1243-1250. DOI:10.1360/02tb9276 |
| [20] | Jiao NZ, Yang YH, Koshikawa H, Harada S, Watanabe M. Microscopic overestimation of heterotrophic bacteria in open waters of China Seas. Journal of Microbiology and Biotechnology, 2001, 11(5): 899-901. |
| [21] | Zhang X, Shi Z, Liu QX, Ye F, Tian L, Huang XP. Spatial and temporal variations of picoplankton in three contrasting periods in the Pearl River Estuary, South China. Continental Shelf Research, 2013, 56: 1-12. DOI:10.1016/j.csr.2013.01.015 |
| [22] | Moore LR, Goericke R, Chisholm SW. Comparative physiology of Synechococcus and Prochlorococcus: influence of light and temperature on growth, pigments, fluorescence and absorptive properties. Marine Ecology Progress Series, 1995, 116(1/3): 259-275. |
| [23] | Kettler GC, Martiny AC, Huang K, Zucker J, Coleman ML, Rodrigue S, Chen F, Lapidus A, Ferriera S, Johnson J, Steglich C, Church GM, Richardson P, Chisholm SW. Patterns and implications of gene gain and loss in the evolution of Prochlorococcus. PLoS Genetics, 2007, 3(12): e231. DOI:10.1371/journal.pgen.0030231 |
| [24] | Voigt K, Sharma CM, Mitschke J, Lambrecht SJ, Vo? B, Hess WR, Steglich C. Comparative transcriptomics of two environmentally relevant cyanobacteria reveals unexpected transcriptome diversity. The ISME Journal, 2014, 8(10): 2056-2068. DOI:10.1038/ismej.2014.57 |
| [25] | Malmstrom RR, Coe A, Kettler GC, Martiny AC, Frias-Lopez J, Zinser ER, Chisholm SW. Temporal dynamics of Prochlorococcus ecotypes in the Atlantic and Pacific oceans. The ISME Journal, 2010, 4(10): 1252-1264. DOI:10.1038/ismej.2010.60 |
| [26] | Coleman ML, Sullivan MB, Martiny AC, Steglich C, Barry K, DeLong EF, Chisholm SW. Genomic islands and the ecology and evolution of Prochlorococcus. Science, 2006, 311(5768): 1768-1770. DOI:10.1126/science.1122050 |
| [27] | Rocap G, Distel DL, Waterbury JB, Chisholm SW. Resolution of Prochlorococcus and Synechococcus ecotypes by using 16S-23S ribosomal DNA internal transcribed spacer sequences. Applied and Environmental Microbiology, 2002, 68(3): 1180-1191. DOI:10.1128/AEM.68.3.1180-1191.2002 |
| [28] | Dufresne A, Garczarek L, Partensky F. Accelerated evolution associated with genome reduction in a free-living prokaryote. Genome Biology, 2005, 6(2): R14. DOI:10.1186/gb-2005-6-2-r14 |
| [29] | Scanlan DJ, Ostrowski M, Mazard S, Dufresne A, Garczarek L, Hess WR, Post AF, Hagemann M, Paulsen I, Partensky F. Ecological genomics of marine picocyanobacteria. Microbiology and Molecular Biology Reviews, 2009, 73(2): 249-299. DOI:10.1128/MMBR.00035-08 |
| [30] | Ting CS, Rocap G, King J, Chisholm SW. Cyanobacterial photosynthesis in the oceans: the origins and significance of divergent light-harvesting strategies. Trends in Microbiology, 2002, 10(3): 134-142. DOI:10.1016/S0966-842X(02)02319-3 |
| [31] | Xu K, Grant-Burt JL, Donaher N, Campbell DA. Connectivity among photosystem Ⅱ centers in phytoplankters: patterns and responses. Biochimica et Biophysica Acta (BBA) - Bioenergetics, 2017, 1858(6): 459-474. DOI:10.1016/j.bbabio.2017.03.003 |
| [32] | Fujinuma D, Akutsu S, Komatsu H, Watanabe T, Miyashita H, Iwamoto K, Shiraiwa Y, Islam MR, Koike H, Kawachi M, Kobayashi M. Detection of divinyl chlorophyll a' and divinyl pheophytin a as minor key components in a marine picoplankton Prochlorococcus sp. RCC315. Photomedicine and Photobiology, 2012, 34: 47-52. |
| [33] | Komatsu H, Wada K, Kanjoh T, Miyashita H, Sato M, Kawachi M, Kobayashi M. Unique chlorophylls in picoplankton Prochlorococcus sp. "Physicochemical properties of divinyl chlorophylls, and the discovery of monovinyl chlorophyll b as well as divinyl chlorophyll b in the species Prochlorococcus NIES-2086".. Photosynthesis Research, 2016, 130(1-3): 445-467. DOI:10.1007/s11120-016-0283-5 |
| [34] | Bibby TS, Mary I, Nield J, Partensky F, Barber J. Low-light-adapted Prochlorococcus species possess specific antennae for each photosystem. Nature, 2003, 424(6952): 1051-1054. DOI:10.1038/nature01933 |
| [35] | Stickforth P, Steiger S, Hess WR, Sandmann G. A novel type of lycopene ε-cyclase in the marine cyanobacterium Prochlorococcus marinus MED4. Archives of Microbiology, 2003, 179(6): 409-415. DOI:10.1007/s00203-003-0545-4 |
| [36] | Erdo?an A, Demirel Z, Ero?lu AE, Dalay MC. Carotenoid profile in Prochlorococcus sp. and enrichment of lutein using different nitrogen sources. Journal of Applied Phycology, 2016, 28(6): 3251-3257. DOI:10.1007/s10811-016-0861-0 |
| [37] | Hamada F, Murakami A, Akimoto S. Comparative analysis of ultrafast excitation energy-transfer pathways in three strains of divinyl chlorophyll a/b-containing cyanobacterium, Prochlorococcus marinus. The Journal of Physical Chemistry B, 2015, 119(51): 15593-15600. DOI:10.1021/acs.jpcb.5b10073 |
| [38] | Takaichi S, Mochimaru M, Uchida H, Murakami A, Hirose E, Maoka T, Tsuchiya T, Mimuro M. Opposite chilarity of α-carotene in unusual cyanobacteria with unique chlorophylls, Acaryochloris and Prochlorococcus. Plant and Cell Physiology, 2012, 53(11): 1881-1888. DOI:10.1093/pcp/pcs126 |
| [39] | Saini RK, Nile SH, Park SW. Carotenoids from fruits and vegetables: chemistry, analysis, occurrence, bioavailability and biological activities. Food Research International, 2015, 76: 735-750. DOI:10.1016/j.foodres.2015.07.047 |
| [40] | Kulk G, van de Poll WH, Visser RJW, Buma AGJ. Distinct differences in photoacclimation potential between prokaryotic and eukaryotic oceanic phytoplankton. Journal of Experimental Marine Biology and Ecology, 2011, 398(1/2): 63-72. |
| [41] | Hamada F, Murakami A, Akimoto S. Adaptation of divinyl chlorophyll a/b-containing cyanobacterium to different light conditions: three strains of Prochlorococcus marinus. The Journal of Physical Chemistry B, 2017, 121(39): 9081-9090. DOI:10.1021/acs.jpcb.7b04835 |
| [42] | Campbell DA, Tyystj?rvi E. Parameterization of photosystem Ⅱ photoinactivation and repair. Biochimica et Biophysica Acta (BBA) - Bioenergetics, 2012, 1817(1): 258-265. DOI:10.1016/j.bbabio.2011.04.010 |
| [43] | Bonisteel EM, Turner BE, Murphy CD, Melanson J, Duff NM, Beardsall BD, Xu K, Campbell DA, Cockshutt AM. Strain specific differences in rates of Photosystem Ⅱ repair in picocyanobacteria correlate to differences in FtsH protein levels and isoform expression patterns. PLoS One, 2018, 13(12): e0209115. DOI:10.1371/journal.pone.0209115 |
| [44] | Guidi L, Tattini M, Landi M. How does chloroplast protect chlorophyll against excessive light//Chlorophyll. Intech Open, 2017: 21. |
| [45] | Depège N, Bellafiore S, Rochaix JD. Role of chloroplast protein kinase Stt7 in LHCⅡ phosphorylation and state transition in Chlamydomonas. Science, 2003, 299(5612): 1572-1575. |
| [46] | Rocap G, Larimer FW, Lamerdin J, Malfatti S, Chain P, Ahlgren NA, Arellano A, Coleman M, Hauser L, Hess WR, Johnson ZI, Land M, Lindell D, Post AF, Regala W, Shah M, Shaw SL, Steglich C, Sullivan MB, Ting CS, Tolonen A, Webb EA, Zinser ER, Chisholm SW. Genome divergence in two Prochlorococcus ecotypes reflects oceanic niche differentiation. Nature, 2003, 424(6952): 1042-1047. DOI:10.1038/nature01947 |
| [47] | Xu K, Lavaud J, Perkins R, Austen E, Bonnanfant M, Campbell DA. Phytoplankton σPSⅡ and excitation dissipation; implications for estimates of primary productivity. Frontiers in Marine Science, 2018, 5: 281. DOI:10.3389/fmars.2018.00281 |
| [48] | Bailey S, Mann NH, Robinson C, Scanlan DJ. The occurrence of rapidly reversible non-photochemical quenching of chlorophyll a fluorescence in cyanobacteria. FEBS Letters, 2005, 579(1): 275-280. DOI:10.1016/j.febslet.2004.11.091 |
| [49] | Ogawa T, Mi HL. Cyanobacterial NADPH dehydrogenase complexes. Photosynthesis Research, 2007, 93(1/3): 69-77. DOI:10.1007/s11120-006-9128-y |
| [50] | Zorz JK, Allanach JR, Murphy CD, Roodvoets MS, Campbell DA, Cockshutt AM. The RUBISCO to photosystem Ⅱ ratio limits the maximum photosynthetic rate in picocyanobacteria. Life, 2015, 5(1): 403-417. DOI:10.3390/life5010403 |
| [51] | Fridman S, Flores-Uribe J, Larom S, Alalouf O, Liran O, Yacoby I, Salama F, Bailleul B, Rappaport F, Ziv T, Sharon I, Cornejo-Castillo FM, Philosof A, Dupont CL, Sánchez P, Acinas SG, Rohwer FL, Lindell D, Béjà O. A myovirus encoding both photosystem Ⅰ and Ⅱ proteins enhances cyclic electron flow in infected Prochlorococcus cells. Nature Microbiology, 2017, 2(10): 1350-1357. DOI:10.1038/s41564-017-0002-9 |
| [52] | MacGregor-Chatwin C, Jackson PJ, Sener M, Chidgey JW, Hitchcock A, Qian P, Mayneord GE, Johnson MP, Luthey-Schulten Z, Dickman MJ, Scanlan DJ, Hunter CN. Membrane organization of photosystem Ⅰ complexes in the most abundant phototroph on Earth. Nature Plants, 2019, 5(8): 879-889. DOI:10.1038/s41477-019-0475-z |
| [53] | Wood WHJ, MacGregor-Chatwin C, Barnett SFH, Mayneord GE, Huang X, Hobbs JK, Hunter CN, Johnson MP. Dynamic thylakoid stacking regulates the balance between linear and cyclic photosynthetic electron transfer. Nature Plants, 2018, 4(2): 116-127. |
| [54] | Dufresne A, Salanoubat M, Partensky F, Artiguenave F, Axmann IM, Barbe V, Duprat S, Galperin MY, Koonin EV, Le Gall F, Makarova KS, Ostrowski M, Oztas S, Robert C, Rogozin IB, Scanlan DJ, de Marsac NT, Weissenbach J, Wincker P, Wolf YI, Hess WR. Genome sequence of the cyanobacterium Prochlorococcus marinus SS120, a nearly minimal oxyphototrophic genome. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(17): 10020-10025. DOI:10.1073/pnas.1733211100 |
| [55] | Swan BK, Tupper B, Sczyrba A, Lauro FM, Martinez-Garcia M, González JM, Luo HW, Wright JJ, Landry ZC, Hanson NW, Thompson BP, Poulton NJ, Schwientek P, Acinas SG, Giovannoni SJ, Moran MA, Hallam SJ, Cavicchioli R, Woyke T, Stepanauskas R. Prevalent genome streamlining and latitudinal divergence of planktonic bacteria in the surface ocean. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(28): 11463-11468. DOI:10.1073/pnas.1304246110 |
| [56] | Steglich C, Futschik ME, Lindell D, Voss B, Chisholm SW, Hess WR. The challenge of regulation in a minimal photoautotroph: non-coding RNAs in Prochlorococcus. PLoS Genetics, 2008, 4(8): e1000173. DOI:10.1371/journal.pgen.1000173 |
| [57] | Waldbauer JR, Rodrigue S, Coleman ML, Chisholm SW. Transcriptome and proteome dynamics of a light-dark synchronized bacterial cell cycle. PLoS One, 2012, 7(8): e43432. DOI:10.1371/journal.pone.0043432 |
| [58] | Stazic D, Lindell D, Steglich C. Antisense RNA protects mRNA from RNase E degradation by RNA-RNA duplex formation during phage infection. Nucleic Acids Research, 2011, 39(11): 4890-4899. DOI:10.1093/nar/gkr037 |
| [59] | Zamore PD, Haley B. Ribo-gnome: the big world of small RNAs. Science, 2005, 309(5740): 1519-1524. DOI:10.1126/science.1111444 |
| [60] | Nitzan M, Rehani R, Margalit H. Integration of bacterial small RNAs in regulatory networks. Annual Review of Biophysics, 2017, 46: 131-148. DOI:10.1146/annurev-biophys-070816-034058 |
| [61] | Smirnov A, F?rstner KU, Holmqvist E, Otto A, Günster R, Becher D, Reinhardt R, Vogel J. Grad-seq guides the discovery of ProQ as a major small RNA-binding protein. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(41): 11591-11596. DOI:10.1073/pnas.1609981113 |
| [62] | Chaulk SG, Smith-Frieday MN, Arthur DC, Culham DE, Edwards RA, Soo P, Frost LS, Keates RAB, Glover JNM, Wood JM. ProQ is an RNA chaperone that controls ProP levels in Escherichia coli. Biochemistry, 2011, 50(15): 3095-3106. DOI:10.1021/bi101683a |
| [63] | Zhang A, Wassarman KM, Ortega J, Steven AC, Storz G. The Sm-like Hfq protein increases OxyS RNA interaction with target mRNAs. Molecular Cell, 2002, 9(1): 11-22. DOI:10.1016/S1097-2765(01)00437-3 |
| [64] | Axmann IM, Kensche P, Vogel J, Kohl S, Herzel H, Hess WR. Identification of cyanobacterial non-coding RNAs by comparative genome analysis. Genome Biology, 2005, 6(9): R73. DOI:10.1186/gb-2005-6-9-r73 |
| [65] | Axmann IM, Holtzendorff J, Vo? B, Kensche P, Hess WR. Two distinct types of 6S RNA in Prochlorococcus. Gene, 2007, 406(1/2): 69-78. |
| [66] | Nakamura T, Naito K, Yokota N, Sugita C, Sugita M. A cyanobacterial non-coding RNA, Yfr1, is required for growth under multiple stress conditions. Plant and Cell Physiology, 2007, 48(9): 1309-1318. DOI:10.1093/pcp/pcm098 |
| [67] | Richter AS, Schleberger C, Backofen R, Steglich C. Seed-based IntaRNA prediction combined with GFP-reporter system identifies mRNA targets of the small RNA Yfr1. Bioinformatics, 2010, 26(1): 1-5. DOI:10.1093/bioinformatics/btp609 |
| [68] | Lambrecht SJ, Kanesaki Y, Fuss J, Huettel B, Reinhardt R, Steglich C. Interplay and targetome of the two conserved cyanobacterial sRNAs Yfr1 and Yfr2 in Prochlorococcus MED4. Scientific Reports, 2019, 9: 14331. DOI:10.1038/s41598-019-49881-9 |
| [69] | Lambrecht SJ, Wahlig JML, Steglich C. The GntR family transcriptional regulator PMM1637 regulates the highly conserved cyanobacterial sRNA Yfr2 in marine picocyanobacteria. DNA Research, 2018, 25(5): 489-497. DOI:10.1093/dnares/dsy019 |
| [70] | Morris JJ, Kirkegaard R, Szul MJ, Johnson ZI, Zinser ER. Facilitation of robust growth of Prochlorococcus colonies and dilute liquid cultures by "helper" heterotrophic bacteria. Applied and Environmental Microbiology, 2008, 74(14): 4530-4534. DOI:10.1128/AEM.02479-07 |
| [71] | Laohavisit A, Anderson A, Bombelli P, Jacobs M, Howe CJ, Davies JM, Smith AG. Enhancing plasma membrane NADPH oxidase activity increases current output by diatoms in biophotovoltaic devices. Algal Research, 2015, 12: 91-98. DOI:10.1016/j.algal.2015.08.009 |
| [72] | Andeer PF, Learman DR, Mcilvin M, Dunn JA, Hansel CM. Extracellular haem peroxidases mediate Mn(Ⅱ) oxidation in a marine Roseobacter bacterium via superoxide production. Environmental Microbiology, 2015, 17(10): 3925-3936. DOI:10.1111/1462-2920.12893 |
| [73] | Imlay JA. The molecular mechanisms and physiological consequences of oxidative stress: lessons from a model bacterium. Nature Reviews Microbiology, 2013, 11(7): 443-454. DOI:10.1038/nrmicro3032 |
| [74] | Kim D, Nakashima T, Matsuyama Y, Niwano Y, Yamaguchi K, Oda T. Presence of the distinct systems responsible for superoxide anion and hydrogen peroxide generation in red tide phytoplankton Chattonella marina and Chattonella ovata. Journal of Plankton Research, 2007, 29(3): 241-247. DOI:10.1093/plankt/fbm011 |
| [75] | Zinser ER. Cross-protection from hydrogen peroxide by helper microbes: the impacts on the cyanobacterium Prochlorococcus and other beneficiaries in marine communities. Environmental Microbiology Reports, 2018, 10(4): 399-411. DOI:10.1111/1758-2229.12625 |
| [76] | Ma LY, Calfee BC, Morris JJ, Johnson ZI, Zinser ER. Degradation of hydrogen peroxide at the ocean's surface: the influence of the microbial community on the realized thermal niche of Prochlorococcus. The ISME Journal, 2018, 12(2): 473-484. DOI:10.1038/ismej.2017.182 |
| [77] | Morris JJ, Lenski RE, Zinser ER. The black queen hypothesis: evolution of dependencies through adaptive gene loss. mBio, 2012, 3(2): e00036-12. DOI:10.1128/mBio.00036-12 |
| [78] | Coleman ML, Chisholm SW. Code and context: Prochlorococcus as a model for cross-scale biology. Trends in Microbiology, 2007, 15(9): 398-407. DOI:10.1016/j.tim.2007.07.001 |
| [79] | Biller SJ, Coe A, Chisholm SW. Torn apart and reunited: impact of a heterotroph on the transcriptome of Prochlorococcus. The ISME Journal, 2016, 10(12): 2831-2843. DOI:10.1038/ismej.2016.82 |
| [80] | Azam F, Malfatti F. Microbial structuring of marine ecosystems. Nature Reviews Microbiology, 2007, 5(10): 782-791. DOI:10.1038/nrmicro1747 |
| [81] | Morris JJ, Johnson ZI, Wilhelm SW, Zinser ER. Diel regulation of hydrogen peroxide defenses by open ocean microbial communities. Journal of Plankton Research, 2016, 38(4): 1103-1114. DOI:10.1093/plankt/fbw016 |
| [82] | Morris JJ, Johnson ZI, Szul MJ, Keller M, Zinser ER. Dependence of the cyanobacterium Prochlorococcus on hydrogen peroxide scavenging microbes for growth at the ocean's surface. PLoS One, 2011, 6(2): e16805. DOI:10.1371/journal.pone.0016805 |
| [83] | Zinser ER, Johnson ZI, Coe A, Karaca E, Veneziano D, Chisholm SW. Influence of light and temperature on Prochlorococcus ecotype distributions in the Atlantic Ocean. Limnology and Oceanography, 2007, 52(5): 2205-2220. DOI:10.4319/lo.2007.52.5.2205 |
| [84] | Sher D, Thompson JW, Kashtan N, Croal L, Chisholm SW. Response of Prochlorococcus ecotypes to co-culture with diverse marine bacteria. The ISME Journal, 2011, 5(7): 1125-1132. DOI:10.1038/ismej.2011.1 |
| [85] | Li B, Sher D, Kelly L, Shi YX, Huang K, Knerr PJ, Joewono I, Rusch D, Chisholm SW, van der Donk WA. Catalytic promiscuity in the biosynthesis of cyclic peptide secondary metabolites in planktonic marine cyanobacteria. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(23): 10430-10435. |
