齐艳利1,2,3, 刘晖1,2,3, 周配1,2,3, 高聪1,2,3, 刘立明1,2,3


1. 江南大学食品科学与技术国家重点实验室, 江苏 无锡 214122;
2. 江南大学教育部工业生物技术重点实验室, 江苏 无锡 214122;
3. 江南大学食品微生物制造工程研究室, 江苏 无锡 214122
收稿日期:2020-07-12;修回日期:2020-10-06;网络出版日期:2020-12-11
基金项目:国家自然科学基金创新研究群体科学基金(32021005);国家自然科学基金(32070124, 22038005);国家轻工技术与工程一流学科自主课题(LITE2018-08)
*通信作者:刘立明, Tel/Fax: +86-510-85197875;E-mail: mingll@jiangnan.edu.cn.
摘要:[目的] 研究中介体亚基Med15B(ORF CAGL0H06215g)介导的脂肪酸代谢影响光滑球拟酵母(Candida glabrata)耐受低pH胁迫的生理机制。[方法] 在菌株Δmed15B中过量表达脂肪酸延伸酶基因elo1(ORF CAGL0L08184g)和Δ9去饱和酶基因ole1(ORF CAGL0I00418g),构建过表达菌株Δmed15B/elo1-ole1,然后与菌株Δmed15B和亲本菌株ATCC55对比分析基因elo1和ole1的表达水平、脂肪酸组分比例、细胞膜完整性和耐受能力在pH 2.0和pH 6.0条件下的差异。[结果] 发现,在pH 2.0条件下过表达菌株Δmed15B/elo1-ole1:(1)基因elo1和ole1表达水平比菌株Δmed15B分别上调了4.1倍和3.3倍,与亲本菌株ATCC55相比分别上调了2.5倍和2.2倍;(2)脂肪酸平均链长延长至17.4,高于菌株Δmed15B的16.8和亲本菌株ATCC55的17.1;不饱和脂肪酸与饱和脂肪酸的比值比菌株Δmed15B和亲本菌株ATCC55分别提高了53.1%和41.5%;(3)碘化丙啶(PI)染色细胞数占总检测细胞数的比例,与菌株Δmed15B和亲本菌株ATCC55比较分别下降了60.8%和37.7%;(4)细胞半抑制pH(胁迫)(IC50)值达到pH 2.3,比菌株Δmed15B的pH 3.7和亲本菌株ATCC55的pH 3.1具有更强的耐受能力。[结论] 在菌株Δmed15B中过表达基因elo1和ole1能通过提高长链脂肪酸和不饱和脂肪酸含量,增强细胞对低pH胁迫的耐受能力。
关键词:光滑球拟酵母Med15B长链脂肪酸不饱和脂肪酸低pH耐受能力
Enhancing low pH tolerance of Candida glabrata Δmed15B by overexpressing genes elo1 and ole1
Yanli Qi1,2,3, Hui Liu1,2,3, Pei Zhou1,2,3, Cong Gao1,2,3, Liming Liu1,2,3


1. State Key Laboratory of Food Science and Technology, Jiangnan University, Wuxi 214122, Jiangsu Province, China;
2. Key Laboratory of Industrial Biotechnology, Ministry of Education, Jiangnan University, Wuxi 214122, Jiangsu Province, China;
3. Laboratory of Food Microbial-Manufacturing Engineering, Jiangnan University, Wuxi 214122, Jiangsu Province, China
Received: 12 July 2020; Revised: 6 October 2020; Published online: 11 December 2020
*Corresponding author: Liming Liu, Tel/Fax: +86-510-85197875; E-mail: mingll@jiangnan.edu.cn.
Foundation item: Supported by the Science Fund for Creative Research Groups of the National Natural Science Foundation of China (32021005), by the National Natural Science Foundation of China (32070124, 22038005) and by the National First-Class Discipline Program of Light Industry Technology and Engineering (LITE2018-08)
Abstract: [Objective] To investigate the effect of mediator subunit Med15B mediated fatty acid metabolism on Candida glabrata low pH tolerance. [Methods] Overexpression of genes elo1 and ole1, encoding fatty acid elongase and Δ9 desaturase, in strain Δmed15B we obtained an overexpressed strain Δmed15B/elo1-ole1. The expression level of genes elo1 and ole1, the membrane fatty acid composition, the membrane integrity and stress tolerance of parent strain ATCC55, strain Δmed15B and overexpression strain Δmed15B/elo1-ole1 were determined and compared at pH 2.0 and pH 6.0. [Results] At pH 2.0, the expression level of genes elo1 and ole1 in overexpression strain Δmed15B/elo1-ole1 was increased by 4.1-fold and 3.3-fold compared to those of strain Δmed15B, and increased by 2.5-fold and 2.2-fold compared to those of parent strain ATCC55, respectively. The average acyl-chain length of overexpression strain Δmed15B/elo1-ole1 reached 17.4 longer than that of the deletion strain (16.8) and parent strain (17.1), and the ratio of unsaturated fatty acid to saturated fatty acid (UFA/SFA ratio) increased by 53.1% and 41.5%, respectively. Furthermore, the percentage of propidium iodide (PI)-stained cell in overexpression strain Δmed15B/elo1-ole1 was decreased by 60.8% and 37.7%, respectively, compared to that of strain Δmed15B and parent strain ATCC55. Similarly, overexpression strain Δmed15B/elo1-ole1 exhibited more tolerance to low pH and the half-maximal inhibition (IC50) values for pH stress was reached pH 2.3, which was pH 3.7 of strain Δmed15B and pH 3.1 of parent strain ATCC55. [Conclusion] These results indicated that overexpression genes elo1 and ole1 in strain Δmed15B can enhance cell tolerance to low pH by increasing the content of long chain and unsaturated fatty acids.
Keywords: Candida glabrataMed15Blong chain fatty acidsunsaturated fatty acidslow pH tolerance
光滑球拟酵母是发酵法生产有机酸的重要工业菌株[1]。在利用其发酵生产有机酸的工业过程中,发酵液pH会随着有机酸的不断积累而逐渐降低,成为影响光滑球拟酵母生产有机酸过程经济效益的关键因素[1]。微生物耐受低pH胁迫的生理机制包括:细胞壁和细胞膜的屏障作用、胞内质子动势、DNA遗传水平损伤修复、信号转导系统级联调控作用和特定转录因子激活或失活[2-4]。其中,细胞膜作为隔开细胞质与外界环境的天然屏障,通过调节自身组分含量和比例,调节细胞膜完整性、流动性和通透性,从而耐受低pH胁迫。
微生物细胞膜脂质主要由磷脂、鞘脂和甾醇组成,在脂质双分子层上有跨膜蛋白、锚定蛋白和外周蛋白,因此细胞膜中脂质和蛋白质的物理特性和生理功能影响细胞膜的完整性、流动性和通透性。Konas等[5]计算模拟发现磷脂头部组成特性不同,对乙酸引起的细胞膜损伤的响应不同。在此基础上,Tan等[3]在大肠菌(Escherichia coli)中上调磷脂酰丝氨酸(PS)合成酶PssA的表达水平,提高磷脂酰乙醇胺(PE)的含量,使突变菌株对辛酸(C8)的耐受能力和含量分别提高了29%和46%。而Yin等[4]在酿酒酵母(Saccharomyces cerevisiae)中模块化过表达胞苷二磷酸-二酰甘油(CDP-DAG)合成酶Cds1和CDP-DAG-丝氨酸O-磷脂酰转移酶Cho1,使突变菌株对NaCl胁迫的IC50值达到1.19 mol/L,显著高于亲本菌株的1.01 mol/L。另一方面,Sherkhanov等[5]在E. coli中通过敲除酰基转移酶aas,提高脂酰链平均链长,部分消除了中链脂肪酸积累对细胞的损害并使产量提高了20%。Bergenholm等[6]在S. cerevisiae通过组合过表达乙酰辅酶A羧化酶Acc1、Elo1和Ole1,显著提高可可脂的产量,比亲本菌株提高了5.8倍。除了改造脂质合成路径,Tan等[7]在E. coli中异源表达来源于Pseudomonas aeruginos的顺反异构酶(Cti),引入反式不饱和脂肪酸合成,增强细胞对毒性化合物(如己酸和丁醇)和多种胁迫(如高温和高渗)的耐受能力。由于脂质合成路径的复杂性,调节单个或几个脂质代谢路径基因改变细胞膜磷脂组分影响菌株胁迫耐受能力的策略具有一定的局限性。为此,需要开发通过改造全局转录因子从而广泛调节脂质代谢路径基因响应环境胁迫的方法[8-9]。
中介体复合物(mediator complex)作为真核细胞中重要的基因表达调节蛋白[10],通过与RNA聚合酶Ⅱ以及转录因子相结合从而调节基因的转录过程,在细胞生长和耐受环境胁迫方面发挥重要作用[11]。采用电镜技术(electron microscopy)对酵母中介体进行3D投射图分析发现中介体复合物可分“头部-中部-尾部”三个不同的结构区域,中部与尾部模块相互作用位于中介体上部,头部模块则与之相分离位于底部[12]。随后,中国科学技术大学蔡刚教授课题组通过晶体衍射与单粒子电镜技术,重新定义了中介体三维结构:头部与中部模块位于中介体上部,而尾部处于中介体底部[13]。酵母中介体复合物的头部主要包括:Med6、Med8、Med11、Med17、Med18、Med20、Med22;中部主要包括:Med1、Med4、Med7、Med9、Med10、Med14、Med19、Med21、Med31;尾部主要包括:Med2、Med3、Med5、Med15、Med16[14]。
中介体头部模块的结构与人的颚部相似,Med17亚基的螺旋区域与Med11、Med22互作,在610-660残基处形成β折叠结构,构成上颚结构的内表面;下颚则是主要由Med18-Med20-Med8亚基复合物形成;Med17与Med18的环状结构域互作形成多重螺旋束“Neck”域,这一区域中接合了5个中介体亚基[15-16]。中介体中部模块是由异二聚体Med4/Med9和Med7/Med21通过亚基末端保守螺旋区形成的四聚体,呈现“钩子”与“旋钮”的形态。Med7/Med21的两侧分别连接Med10与Med31,Med31构成了其中的“旋钮”的部分,Med14的N端连接头部模块的螺旋束区,C端则交联头部模块的“颚部”[17]。然而,至今还未见中介体尾部模块结构的系统解析,只知道Med2、Med3和Med15互作形成三聚体尾部子模块[18]。但中介体与转录因子发生相互作用主要是通过尾部亚基的构象变化来完成的,因此中介体尾部亚基在转录调控过程中发挥着重要作用[19]。研究发现,Med15能直接与转录激活因子(Gcn4)、油酸激活转录因子(Oaf1)、耐药转录因子(Pdr1)等[20-23]发生相互作用,招募中介体结合到基因上游激活区域,激活相应靶基因表达,从而参与碳源利用、胁迫响应和多药耐受[23-24]等细胞活动。在前期对中介体亚基Med2、Med3、Med15与Med16进行敲除或过量表达时,发现,Med3、Med15和Med16参与了酸胁迫[19]。更进一步发现在pH 2.0条件下,Med15B能通过下调脂质代谢相关基因的表达水平,影响细胞膜脂质组分,进而影响光滑球拟酵母对低pH的耐受能力[25]。但上述研究没有深入解析Med15-基因表达-脂质组分变化-胁迫抗性之间的关联机制。为此,本研究以Δmed15B为出发菌株,通过表达脂肪酸延伸酶Elo1和去饱和酶Ole1改变长链脂肪酸和不饱和脂肪酸的含量,解析脂质组分变化对细胞耐受低pH胁迫的影响。
1 材料和方法 1.1 材料
1.1.1 菌株与质粒: 光滑球拟酵母ATCC55由Karl Kuchler教授惠赠[26],中介体亚基Med15B菌株Δmed15B在前期研究中构建并保存[25],重组质粒克隆的大肠杆菌JM109和大肠杆菌-光滑球拟酵母穿梭载体pY26由本实验室保存。
1.1.2 培养基与培养方法: 大肠杆菌的培养条件:采用LB培养基,培养温度为37 ℃,接种液体培养基时转速为200 r/min。在筛选带有质粒的JM109时,添加终浓度为100 μg/mL的氨苄青霉素获得抗生素筛选压力培养基。光滑球拟酵母的培养条件:培养温度为30 ℃,接种液体培养基时转速为200 r/min。在YNB (yeast nitrogen base without amino acids)培养基中培养时,需要添加终浓度为200 μg/mL的必需氨基酸。根据实验需要用盐酸调节pH模拟低pH胁迫条件。
1.1.3 主要试剂和工具酶: 主要工具酶有rTaq、Primstar、限制酶、RNA抽提试剂盒、反转录试剂盒、荧光定量PCR试剂盒等购自宝日医生物技术(北京)有限公司(TaKaRa中国);酵母基因组DNA提取、PCR产物回收纯化、质粒提取等试剂盒购自北京天根生物技术公司;同源重组试剂盒购自南京诺唯赞生物科技有限公司;蛋白胨、酵母粉购自英国Oxoid公司;YNB、氨苄青霉素、碘化丙啶(PI,propidium iodide)购自生工生物工程(上海)股份有限公司;化学试剂为分析纯或色谱纯购自上海国药集团。
1.2 光滑球拟酵母过表达菌株Δmed15B/elo1-ole1的构建 以光滑球拟酵母CBS138基因组序列为模板,设计表 1引物用于基因elo1和ole1过表达菌株的构建及鉴定。引物合成和测序工作由天霖生物科技(无锡)有限公司完成。
表 1. 本研究所用引物 Table 1. Primers used in this study
| Primer names | Primer sequences (5′→3′) |
| Elo1-kz-F | TCTAAGTTTTCTAGAACTAGCGCGGCCGCATGACTACCATTATTCCACTACTAAAG |
| Elo1-kz-R | GGCGAAGAATTGTTAATTAAAGATCTTTATCTCTTTCTTCTTCTATCTTTGGC |
| Ole1-kz-F | CGACGGATTCTAGAACTAGTGGATCCATGTCTAGGGTTGAAACCAGAA |
| Ole1-kz-R | TGACATAACTAATTACATGACTCGAGTCAGTTCATGTGTGCTCTCTC |
| Elo1-yz-F | GATGACCTCCCATTGATATTTAAGT |
| Elo1-yz-R | AATTGGAGCGACCTCATG |
| Ole1-yz-F | TGCAACCTGCCTGGAGTA |
| Ole1-yz-R | GACCTAGACTTCAGGTTGTCT |
| Elo1-dl-F | GTAACGAAGGTGCCTGGAC |
| Elo1-dl-R | TGTGGCAGAGTTGGGAAGTA |
| Ole1-dl-F | GCTTTGTTCGCTACTTTCTATT |
| Ole1-dl-F | GCCATTGAGTTGATACAGAAGG |
| β-Actin-F | ACCGCTGCTCAATCTTCC |
| β-Actin-R | GGTTTGCTCCTTTGGTCTATT |
| F: forward primer; R: reverse primer. | |
表选项
根据酵母基因组DNA提取试剂盒说明书提取光滑球拟酵母ATCC55基因组DNA,并用作模板,使用表 1中引物Elo1-kz-F/R和Ole1-kz-F/R扩增带有同源臂的elo1和ole1基因片段并纯化回收。将酵母表达载体pY26用限制酶Not I和Bgl Ⅱ酶切,利用同源重组试剂盒将elo1基因片段插入质粒pY26,转化到E. coli JM109感受态细胞中,在含氨苄青霉素抗性的平板上筛选阳性转化子,使用引物Elo1-yz-F/R进行菌落PCR和测序验证,将正确的重组质粒命名为pY26-elo1。然后,将质粒pY26-elo1用限制性内切酶BamH I和Xho I酶切,利用同源重组试剂盒将ole1基因片段插入质粒pY26-elo1,转化到E. coli JM109感受态细胞中,在含氨苄青霉素抗性的平板上筛选阳性转化子,使用引物Ole1-yz-F/R进行菌落PCR和测序验证,将正确的重组质粒命名为pY26-elo1-ole1。
将重组质粒pY26-elo1-ole1转化至光滑球拟酵母med15Δ的感受态细胞中,在不含有尿嘧啶的YNB平板上筛选阳性转化子,将阳性转化子接种至新鲜的液体培养基并提取重组质粒。以提取的重组质粒和空白质粒为模板,使用引物Elo1-yz-F/R和Ole1-yz-F/R进行PCR验证,得到正确的过表达菌株Δmed15B/elo1-ole1。
1.3 基因elo1和ole1转录水平分析 总RNA提取:将酵母细胞培养至指数生长期,分别转接至pH 6.0和pH 2.0的新鲜YNB培养基中,培养6 h,然后收集菌体(4 ℃,5000 r/min,5 min)。将收集后的酵母菌体经过液氮快速冷冻后研磨,破除细胞壁,使用RNA提取试剂盒提取样品总RNA,并对RNA的浓度和质量进行检测。
cDNA合成:利用反转录试剂盒Prime Script Ⅱ 1st-strand cDNA synthesis kit对总RNA进行反转录合成cDNA。
qRT-PCR:将合成的cDNA浓度稀释至100 ng/μL,将其用于荧光定量PCR (qRT-PCR)的模板,利用表 1中的引物Elo1-dl-F/R、Ole1-dl-F/R和β-Actin-F/R,使用SYBR Premix ExtaqTM试剂盒进行qRT-PCR定量分析基因elo1和ole1的表达水平。基因act1作为本实验的对照组。
1.4 细胞膜脂肪酸组分分析 酵母细胞收集:酵母细胞的培养和收集如1.3.1所述,将收集后的酵母菌体,真空冷冻干燥备用。
脂肪酸提取液的配置:皂化溶液:称取9 g氢氧化钠溶于30 mL超纯水,再加入30 mL甲醇混匀;甲基化溶液:取65 mL 6.0 mol/L的盐酸,与55 mL甲醇混匀;提取溶液:将50 mL己烷和50 mL甲基三丁基乙醚混匀;碱洗液:将1.01 g氢氧化钠溶于100 mL超纯水。
脂质提取及处理:称取冷冻干燥后的菌体50 mg,置于耐热试管中,加入2 mL皂化液,沸水浴30 min,置于冰上快速冷却。加入4 mL甲基化溶液,混匀,80 ℃水浴10 min,置于冰上快速冷却。加入2.5 mL提取溶液,摇匀振荡10 min,静置至溶液分层。将上层溶液转移至新的试管中,加入3 mL碱洗液和几滴饱和氯化钠溶液,用旋涡振荡器涡旋5 min,静置至溶液分层。吸取上层液体于气相样品管中,进行检测。色谱分析条件参考Yang等[27]并作了一定调整,具体如下:使用气相色谱仪(GC-2010AF,日本岛津公司,日本),配备PEG毛细管填充柱;使用氦气作为载气;将1.0 μL样品注入250 ℃的进样器中,初始柱温为100 ℃保持1 min,随后以4 ℃/min的速率升至250 ℃并保持5 min,检测器温度保持在280 ℃;通过比较脂肪酸甲酯的保留时间与已知标准的保留时间,对脂肪酸甲酯进行鉴定。
1.5 细胞膜完整性分析 流式细胞仪染色法:酵母细胞的培养和收集如1.3.1所述,将待测菌液的OD600稀释至0.5,取500 μL,加入5 μL的核酸染料PI,立即混匀并避光孵育5 min,用PBS缓冲液(pH 7.4)洗涤菌体2次,并用500 μL的PBS缓冲液复悬。将复溶后的菌液用流式细胞仪检测,在低流速下分析样品中的20000个细胞,所得数据用FlowJo软件进行分析。核酸染料PI不能进入具有完整细胞膜的细胞,所以,PI染色细胞数占总检测细胞数比例越高,细胞膜完整性越差。
1.6 细胞半抑制pH (胁迫)分析 酵母培养、转接的方法如1.3.1所述,使用浓盐酸调节培养基pH为1.5–6.0,间隔0.5个pH值。测定不同pH条件下酵母细胞存活率,并对酵母细胞存活率进行非线性拟合,得到细胞存活率的曲线图。定义:细胞半抑制pH (胁迫) (IC50)值为酵母细胞存活率为50%时,培养基的pH值。
2 结果和分析 2.1 脂肪酸延伸酶Elo1和去饱和酶Ole1过表达菌株的构建 首先以亲本菌株ATCC55基因组为模板,分别利用表 1中的扩增引物Elo1-kz-F/R和Ole1-kz-F/R获得带有同源臂的基因elo1和ole1。随后通过一步同源重组试剂盒将这2个基因片段依次连接到用限制性酶线性化的表达载体pY26 (pY26属于高拷贝质粒,携带的启动子TEF和GPD属于强启动子,能够高强度地表达其启动的目标基因[28]),成功构建出过表达质粒pY26-elo1-ole1,其中,基因elo1和ole1分别受启动子TEF和GPD调控。再将重组质粒导入菌株Δmed15B中,在尿嘧啶缺陷的YNB平板上筛选过表达菌株Δmed15B/elo1-ole1。分别以空载质粒和过表达阳性转化子中提取的重组质粒为模板,通过验证引物Elo1-yz-F/R和Ole1-yz-F/R进行的PCR扩增验证,过表达基因elo1和ole1重组正确的条带大小分别为1525 bp (图 1:泳道2)和1880 bp (图 1:泳道4),对照条带大小分别为414 bp (图 1:泳道1)和355 bp (图 1:泳道3),上述PCR检测结果表明,基因elo1和ole1的过表达菌株Δmed15B/elo1-ole1构建成功。
 |
| 图 1 PCR鉴定基因elo1和ole1过表达菌株Δmed15B/elo1-ole1 Figure 1 Identification of genes elo1 and ole1 overexpressed strain Δmed15B/elo1-ole1 by PCR. M: DL 2000 marker; lane 1: control of elo1; lane 2: elo1; lane 3: control of ole1; lane 4: ole1. |
| 图选项 |
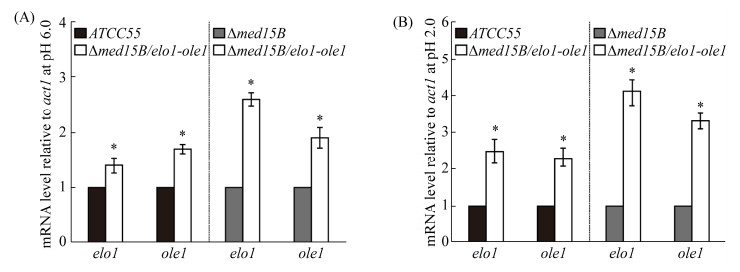
2.2 过表达菌株脂肪酸延伸酶基因elo1和去饱和酶基因ole1表达水平检测 将亲本菌株ATCC55、菌株Δmed15B和过表达菌株Δmed15B/elo1-ole1分别在pH 6.0和pH 2.0的YNB液体培养基中培养6 h。然后利用qRT-PCR检测3株菌基因elo1和ole1的表达水平,结果如图 2所示。在pH 6.0条件下,过表达菌株Δmed15B/elo1-ole1中基因elo1和ole1表达水平分别比亲本菌株ATCC55提高了1.4倍和1.7倍,同时比菌株Δmed15B提高了2.6倍和1.9倍(图 2-A)。另外,随着培养基pH下降,这3株菌中基因elo1和ole1的表达水平都发生显著上调。过表达菌株Δmed15B/elo1-ole1中基因elo1和ole1的表达水平分别比在亲本菌株ATCC55中提高了2.5倍和2.2倍,同时比菌株Δmed15B提高了4.1倍和3.3倍(图 2-B)。上述结果表明在菌株Δmed15B中过表达基因elo1和ole1能弥补Med15B基因缺失所导致的基因表达下调,并进一步提高了基因的表达水平。
 |
| 图 2 不同菌株内基因elo1和ole1的表达水平 Figure 2 The expression level of genes elo1 and ole1 in different strains. Quantitative reverse transcription-PCR (qRT-PCR) tested the mRNA levels of elo1 and ole1 in parent strain ATCC55, strain Δmed15B and overexpression strain Δmed15B/elo1-ole1, calculated relative to the act1 level at pH 6.0 (A) and at pH 2.0 (B). Data represent the means of three biological replicates, and error bars represent the standard deviation. *: P≤0.05. |
| 图选项 |
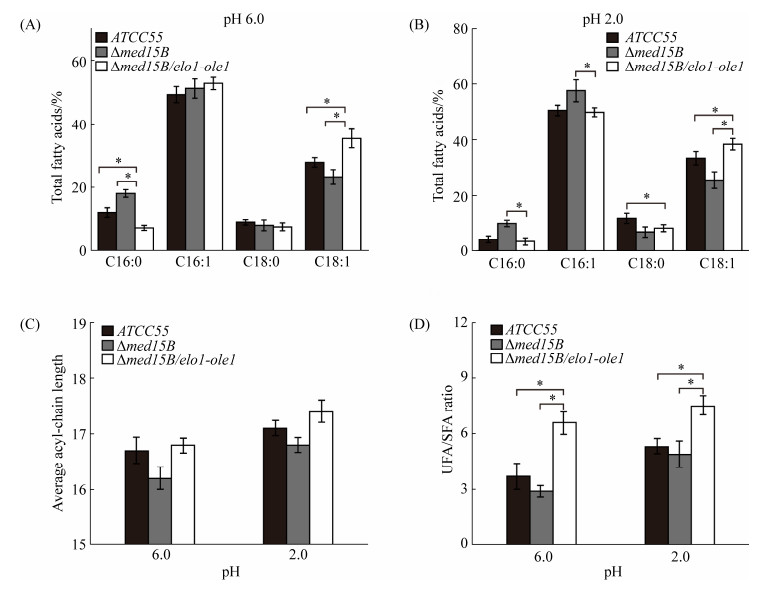
2.3 过表达菌株脂肪酸组分检测 已有研究证明过表达基因elo1和ole1有助于长链脂肪酸和不饱和脂肪酸的合成[6-29],为了检测基因elo1和ole1过表达对细胞膜脂肪酸组分的影响,将亲本菌株ATCC55、菌株Δmed15B和过表达菌株Δmed15B/elo1-ole1分别在pH 6.0和pH 2.0的YNB液体培养基中培养6 h,提取细胞膜脂肪酸进行气质联用检测,结果如图 3所示。在pH 6.0条件下,过表达菌株Δmed15B/elo1-ole1中C16:1和C18:0百分含量与菌株Δmed15B和亲本菌株ATCC55相差不大;C16:0百分含量与菌株Δmed15B和亲本菌株ATCC55对比,分别下降了64.6%和47.1%;而C18:1百分含量则分别提高了51.1%和26.2% (图 3-A)。在pH 2.0条件下,过表达菌株中C18:0百分含量与菌株Δmed15B中相比,没有显著差异;C16:0和C16:1的百分含量与菌株Δmed15B相比,分别下降了66.1%和13.4%;而C18:1百分含量则提高了50.6% (图 3-B)。另外与亲本菌株ATCC55相比,过表达菌株中C16:0和C16:1的百分含量无显著差异,C18:0百分含量下降了29.6%,而C18:1的比例提高16.7% (图 3-B)。进一步对脂肪酸平均链长进行分析,发现过表达菌株Δmed15B/elo1-ole1的脂肪酸平均链长在pH 6.0时为16.9,而在pH 2.0时则增长至17.4;菌株Δmed15B的脂肪酸平均链长在pH 6.0时为16.3,在pH 2.0时增长至16.8;亲本菌株ATCC55脂肪酸平均链长在pH 6.0时为16.8,在pH 2.0时增长至17.1 (图 3-C)。另一方面,在pH 6.0时不饱和脂肪酸(C16:1和C18:1)与饱和脂肪酸(C16:0和C18:0)的比值(UFA/SFA ratio)在过表达菌株Δmed15B/elo1-ole1中为6.6,比菌株Δmed15B的2.9和亲本菌株ATCC55的3.7分别提高了127.6%和78.4%;在pH 2.0条件下,UFA/SFA比值在过表达菌株Δmed15B/elo1-ole1中为7.5,分别比菌株Δmed15B的5.3和亲本菌株ATCC55的4.9提高了53.1%和41.5%。上述结果表明,在菌株Δmed15B中过表达基因elo1和ole1能补偿基因Med15B缺失导致的细胞膜长链脂肪酸和不饱和脂肪酸合成受阻,提高低pH胁迫下细胞膜脂肪酸的平均链长为17.4和UFA/SFA比值为7.5。
 |
| 图 3 基因elo1和ole1过表达影响细胞膜脂肪酸组分 Figure 3 Effect of genes elo1 and ole1 overexpression on the membrane fatty acids. Percentage of fatty acids in different strains at pH 6.0 (A) and pH 2.0 (B). Changes in average acyl-chain length (C) and UFA/SFA ratio (D) at pH 6.0 and pH 2.0. Data represent the means of three biological replicates, and error bars represent the standard deviation. *: P≤0.05. |
| 图选项 |
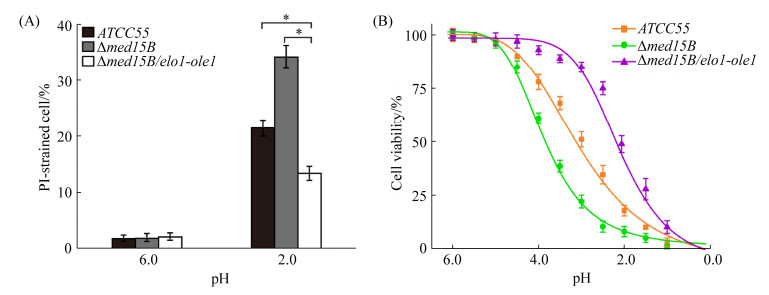
2.4 过表达菌株细胞膜完整性和IC50值检测 为了检测基因elo1和ole1的过表达对细胞膜完整性的影响,将亲本菌株ATCC55、菌株Δmed15B和过表达菌株Δmed15B/elo1-ole1分别在pH 6.0和pH 2.0的YNB液体培养基中培养6 h后,用核酸染料PI对细胞膜破损细胞进行染色,采用流式细胞仪对细胞膜完整性进行定量分析,结果如图 4-A所示。在pH 6.0条件下,PI染色细胞数与总检测细胞数的比值在亲本菌株ATCC55、菌株Δmed15B和过表达菌株Δmed15B/elo1-ole1均低于5%,且3种菌株之间无显著差异。而在pH 2.0条件下,PI染色细胞数与总检测细胞数的比值在3株菌分别升高到21.5%、34.2%、13.4%,过表达菌株Δmed15B/elo1-ole1中PI染色细胞比菌株Δmed15B和亲本菌株ATCC55分别下降了60.8%和37.7%。上述结果表明,在菌株Δmed15B中过表达基因elo1和ole1能有效地弥补Med15B基因缺失导致的细胞膜损伤,增强低pH胁迫下细胞膜完整性。
 |
| 图 4 基因elo1和ole1过表达影响细胞膜完整性和IC50值 Figure 4 Overexpression of genes elo1 and ole1 affects membrane integrity and IC50 value. A: flow cytometry analyses of membrane integrity in different strains at pH 6.0 and pH 2.0, cells were stained with PI; B: analyses of IC50 value in different strains by testing cell viability for each pH value. Data represent the means of three biological replicates, and error bars represent the standard deviation. *: P≤0.05. |
| 图选项 |
进一步通过细胞半抑制pH (胁迫) (IC50)值的计算,检测基因elo1和ole1过表达对细胞低pH胁迫耐受能力的影响,结果如图 4-B所示。亲本菌株ATCC55、菌株Δmed15B和过表达菌株Δmed15B/elo1-ole1对pH胁迫的IC50值分别为pH 3.1、pH 3.7和pH 2.3。根据IC50值的定义,可以发现过表达菌株Δmed15B/elo1-ole1对低pH的耐受能力更强。上述结果表明,过表达基因elo1和ole1能有效地增强低pH条件下细胞膜的完整性,进而改善细胞对低pH的耐受能力。
3 讨论 本研究通过在光滑球拟酵母中介体尾部亚基Med15B缺失菌株(菌株Δmed15B)中过表达脂肪酸延伸酶基因elo1和去饱和酶基因ole1,有效地弥补了低pH条件下Med15B基因缺失引起的基因elo1和ole1表达水平下调[25]。通过进一步对细胞膜脂肪酸组分分析发现,过表达基因elo1和ole1能够提高菌株长链脂肪酸(C18:0和C18:1)和不饱和脂肪酸(C16:1和C18:1)的合成能力[30-31]。在pH 2.0条件下,过表达菌株Δmed15B/elo1-ole1的平均脂肪酸烷基链长达到17.4,UFA/SFA比值达到7.5倍。已有研究发现提高细胞膜长链脂肪酸和不饱和脂肪酸的比例有助于增强胁迫条件下细胞膜的完整性[32-33]。基于过表达菌株中脂肪酸组分的变化,对细胞膜特性进一步分析,发现pH 2.0条件下过表达菌株Δmed15B/elo1-ole1中细胞膜损伤的细胞比例与菌株Δmed15B和亲本菌株ATCC55相比分别下降了60.8%和37.7%。过表达菌株Δmed15B/elo1-ole1的细胞膜完整性增强并使其对pH的IC50值达到pH 2.3,表明其对低pH胁迫耐受能力增强。这些结果与本实验室前期的研究发现相结合,进一步证实了Med15对脂肪酸合成基因的表达和脂肪酸合成起正调控作用,为下一步改造全局转录因子(比如中介体复合物亚基Med3和Med15[8-25]或转录因子Crz1[9])靶向调节代谢路径或信号路径上系列基因的表达,增强菌株的生产能力和耐受能力的策略提供了理论基础和实验依据。
References
| [1] | Liu LM, Xu QL, Li Y, Shi ZP, Zhu Y, Du GC, Chen J. Enhancement of pyruvate production by osmotic-tolerant mutant of Torulopsis glabrata. Biotechnology and Bioengineering, 2007, 97(4): 825-832. DOI:10.1002/bit.21290 |
| [2] | Tan ZG, Khakbaz P, Chen YX, Lombardo J, Yoon JM, Shanks JV, Klauda JB, Jarboe LR. Engineering Escherichia coli membrane phospholipid head distribution improves tolerance and production of biorenewables. Metabolic Engineering, 2017, 44: 1-12. DOI:10.1016/j.ymben.2017.08.006 |
| [3] | Yin NN, Zhu GX, Luo QL, Liu J, Chen XL, Liu LM. Engineering of membrane phospholipid component enhances salt stress tolerance in Saccharomyces cerevisiae. Biotechnology and Bioengineering, 2020, 117(3): 710-720. DOI:10.1002/bit.27244 |
| [4] | Sherkhanov S, Korman TP, Bowie JU. Improving the tolerance of Escherichia coli to medium-chain fatty acid production. Metabolic Engineering, 2014, 25: 1-7. DOI:10.1016/j.ymben.2014.06.003 |
| [5] | Konas RM, Daristotle JL, Harbor NB, Klauda JB. Biophysical changes of lipid membranes in the presence of ethanol at varying concentrations. The Journal of Physical Chemistry B, 2015, 119(41): 13134-13141. DOI:10.1021/acs.jpcb.5b06066 |
| [6] | Bergenholm D, Gossing M, Wei YJ, Siewers V, Nielsen J. Modulation of saturation and chain length of fatty acids in Saccharomyces cerevisiae for production of cocoa butter-like lipids. Biotechnology and Bioengineering, 2018, 115(4): 932-942. DOI:10.1002/bit.26518 |
| [7] | Tan ZG, Yoon JM, Nielsen DR, Shanks JV, Jarboe LR. Membrane engineering via trans unsaturated fatty acids production improves Escherichia coli robustness and production of biorenewables. Metabolic Engineering, 2016, 35: 105-113. DOI:10.1016/j.ymben.2016.02.004 |
| [8] | Lin XB, Qi YL, Yan DN, Liu H, Chen XL, Liu LM. CgMED3 changes membrane sterol composition to help Candida glabrata tolerate low-pH stress. Applied and Environmental Microbiology, 2017, 83(17): e00972-17. |
| [9] | Yan DN, Lin XB, Qi YL, Liu H, Chen XL, Liu LM, Chen J. Crz1p regulates pH homeostasis in Candida glabrata by altering membrane lipid composition. Applied and Environmental Microbiology, 2016, 82(23): 6920-6929. DOI:10.1128/AEM.02186-16 |
| [10] | Poss ZC, Ebmeier CC, Taatjes DJ. The mediator complex and transcription regulation. Critical Reviews in Biochemistry and Molecular Biology, 2013, 48(6): 575-608. DOI:10.3109/10409238.2013.840259 |
| [11] | Allen BL, Taatjes DJ. The mediator complex: a central integrator of transcription. Nature Reviews Molecular Cell Biology, 2015, 16(3): 155-166. DOI:10.1038/nrm3951 |
| [12] | Chadick JZ, Asturias FJ. Structure of eukaryotic mediator complexes. Trends in Biochemical Sciences, 2005, 30(5): 264-271. DOI:10.1016/j.tibs.2005.03.001 |
| [13] | Wang XJ, Sun QQ, Ding ZR, Ji JH, Wang JY, Kong X, Yang JH, Cai G. Redefining the modular organization of the core mediator complex. Cell Research, 2014, 24(7): 796-808. DOI:10.1038/cr.2014.64 |
| [14] | Soutourina J. Transcription regulation by the mediator complex. Nature Reviews Molecular Cell Biology, 2018, 19(4): 262-274. DOI:10.1038/nrm.2017.115 |
| [15] | Cai G, Imasaki T, Yamada K, Cardelli F, Takagi Y, Asturias FJ. Mediator head module structure and functional interactions. Nature Structural & Molecular Biology, 2010, 17(3): 273-279. |
| [16] | Takagi Y, Calero G, Komori H, Brown JA, Ehrensberger AH, Hudmon A, Asturias F, Kornberg RD. Head module control of mediator interactions. Molecular Cell, 2006, 23(3): 355-364. DOI:10.1016/j.molcel.2006.06.007 |
| [17] | Lariviere L, Plaschka C, Seizl M, Petrotchenko EV, Wenzeck L, Borchers CH, Cramer P. Model of the mediator middle module based on protein cross-linking. Nucleic Acids Research, 2013, 41(20): 9266-9273. DOI:10.1093/nar/gkt704 |
| [18] | Ansari SA, Morse RH. Selective role of mediator tail module in the transcription of highly regulated genes in yeast. Transcription, 2012, 3(3): 110-114. DOI:10.4161/trns.19840 |
| [19] | Harper TM, Taatjes DJ. The complex structure and function of mediator. Journal of Biological Chemistry, 2018, 293(36): 13778-13785. DOI:10.1074/jbc.R117.794438 |
| [20] | Ansari SA, Ganapathi M, Benschop JJ, Holstege FCP, Wade JT, Morse RH. Distinct role of mediator tail module in regulation of SAGA-dependent, TATA-containing genes in yeast. The EMBO Journal, 2012, 31(1): 44-57. DOI:10.1038/emboj.2011.362 |
| [21] | Brzovic PS, Heikaus CC, Kisselev L, Vernon R, Herbig E, Pacheco D, Warfield L, Littlefield P, Baker D, Klevit RE, Hahn S. The acidic transcription activator Gcn4 binds the mediator subunit Gal11/Med15 using a simple protein interface forming a fuzzy complex. Molecular Cell, 2011, 44(6): 942-953. DOI:10.1016/j.molcel.2011.11.008 |
| [22] | Thakur JK, Arthanari H, Yang FJ, Chau KH, Wagner G, N??r AM. Mediator subunit Gal11p/MED15 is required for fatty acid-dependent gene activation by yeast transcription factor Oaf1p. Journal of Biological Chemistry, 2009, 284(7): 4422-4428. DOI:10.1074/jbc.M808263200 |
| [23] | Thakur JK, Arthanari H, Yang FJ, Pan SJ, Fan XC, Breger J, Frueh DP, Gulshan K, Li DK, Mylonakis E, Struhl K, Moye-Rowley WS, Cormack BP, Wagner G, N??r AM. A nuclear receptor-like pathway regulating multidrug resistance in fungi. Nature, 2008, 452(7187): 604-609. DOI:10.1038/nature06836 |
| [24] | Kumar V, Waseem M, Dwivedi N, Maji S, Kumar A, Thakur JK. KIX domain of AtMed15a, a mediator subunit of Arabidopsis, is required for its interaction with different proteins. Plant Signaling & Behavior, 2018, 13(2): e1428514. |
| [25] | Qi YL, Liu H, Yu JY, Chen XL, Liu LM. Med15B regulates acid stress response and tolerance in Candida glabrata by altering membrane lipid composition. Applied and Environmental Microbiology, 2017, 83(18): e01128-17. |
| [26] | Roetzer A, Gregori C, Jennings AM, Quintin J, Ferrandon D, Butler G, Kuchler K, Ammerer G, Schuller C. Candida glabrata environmental stress response involves Saccharomyces cerevisiae Msn2/4 orthologous transcription factors. Molecular Microbiology, 2008, 69(3): 603-620. DOI:10.1111/j.1365-2958.2008.06301.x |
| [27] | Yang X, Hang XM, Zhang M, Liu XL, Yang H. Relationship between acid tolerance and cell membrane in Bifidobacterium, revealed by comparative analysis of acid-resistant derivatives and their parental strains grown in medium with and without Tween 80. Applied Microbiology and Biotechnology, 2015, 99(12): 5227-5236. DOI:10.1007/s00253-015-6447-y |
| [28] | Chen XL, Wu J, Song W, Zhang LM, Wang HJ, Liu LM. Fumaric acid production by Torulopsis glabrata: engineering the urea cycle and the purine nucleotide cycle. Biotechnology and Bioengineering, 2015, 112(1): 156-167. DOI:10.1002/bit.25334 |
| [29] | Nasution O, Lee YM, Kim E, Lee Y, Kim W, Choi W. Overexpression of OLE1 enhances stress tolerance and constitutively activates the MAPK HOG pathway in Saccharomyces cerevisiae. Biotechnology and Bioengineering, 2017, 114(3): 620-631. DOI:10.1002/bit.26093 |
| [30] | Fang ZJ, Chen ZX, Wang S, Shi P, Shen YH, Zhang YS, Xiao JH, Huang ZW. Overexpression of OLE1 enhances cytoplasmic membrane stability and confers resistance to cadmium in Saccharomyces cerevisiae. Applied and Environmental Microbiology, 2016, 83(1): e02319-16. |
| [31] | Yu T, Zhou YJJ, Wenning L, Liu QL, Krivoruchko A, Siewers V, Nielsen J, David F. Metabolic engineering of Saccharomyces cerevisiae for production of very long chain fatty acid-derived chemicals. Nature Communications, 2017, 8: 15587. DOI:10.1038/ncomms15587 |
| [32] | Qi YL, Liu H, Chen XL, Liu LM. Engineering microbial membranes to increase stress tolerance of industrial strains. Metabolic Engineering, 2019, 53: 24-34. DOI:10.1016/j.ymben.2018.12.010 |
| [33] | Tan ZG, Black W, Yoon JM, Shanks JV, Jarboe LR. Improving Escherichia coli membrane integrity and fatty acid production by expression tuning of FadL and OmpF. Microbial Cell Factories, 2017, 16(1): 38. DOI:10.1186/s12934-017-0650-8 |
