杨晨, 张为艳, Jung Dawoon, 何山


宁波大学食品与药学学院, 李达三海洋生物医药研究中心, 浙江 宁波 315211
收稿日期:2020-10-30;修回日期:2021-02-02;网络出版日期:2021-02-09
基金项目:国家自然科学基金(41776168)
作者简介:何山, 宁波大学食品与药学学院教授, 研究员, 海洋生物医药浙江省国际科技合作基地主任。主持国家自然科学基金、国家重点研发计划和省市科技项目10余项。发表SCI论文100余篇, 获授权发明专利20余项(转让3项)。"天然药物先导化合物发现及其高效制备方法研究"项目获2019年浙江省自然科学二等奖(第一完成人)。长期从事海洋微生物新资源和生物医药研发。重点关注海洋"未培养"微生物和海洋中光层(深度30-150米)微生物资源, 是我国首位下潜深度达到100米的科学潜水员, 多次带队在南海西沙群岛开展水下科考。近年运用连续流生物反应器、iChip原位培养技术等新型微生物培养技术, 深入挖掘海洋微生物"暗物质", 发现潜在微生物新种300余个.
*通信作者:何山。E-mail: heshan@nbu.edu.cn.
摘要:自然环境中99%微生物在实验室条件下仍是不能被培养的,称之为“未培养”微生物或微生物“暗物质”。对其进行研究不仅有助于认识环境中微生物代谢多样性,丰富生命之树,同时未培养微生物还蕴含着巨大的新基因和新天然产物资源。但传统培养技术的局限性阻碍了“未培养”微生物资源的开发和利用。虽然随着分子生物学技术的发展,可以直接从环境中获得未培养微生物的遗传信息,分析微生物的广泛代谢多样性,但微生物的生理特征和代谢产物等分析仍然需要建立在研究纯菌株的基础上。目前,已经有很多新颖的培养技术被研发,如原位培养技术、共培养技术和连续流生物反应器培养技术等用于挖掘未培养微生物资源。本文主要介绍了连续流生物反应器培养新技术的发展与改进,探讨了“未培养”微生物培养技术及设备的发展方向,以进一步促进“未培养”微生物资源的开发与利用。
关键词:“未培养”微生物培养技术连续流生物反应器微生物资源
Application of continuous flow bioreactor technology in microbial cultivation
Chen Yang, Weiyan Zhang, Dawoon Jung, Shan He


LiDakSum YipYioChin Kenneth Marine Biopharmaceutical Research Center, College of Food and Pharmaceutical Sciences, Ningbo University, Ningbo 315211, Zhejiang Province, China
Received: 30 October 2020; Revised: 2 February 2021; Published online: 9 February 2021
*Corresponding author: Shan He. E-mail: heshan@nbu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (41776168)
Abstract: Ninty nine percentage of microorganisms in the environment remains uncultured, called "uncultured" microorganisms or microbial "dark matter". Unraveling the mysteries of these microbial "dark matter" is especially helpful to understand the diversity of microbes and their metabolic characteristics. DNA genome sequences of microbial "dark matter" could be recovered from the environment samples by population binning of metagenomics and single-cell genomics, independently or combined synergistically. However, microbial functions often cannot be deduced from genetic information alone, and the isolation of uncultured microorganisms remains a powerful approach. Many cultivation-based methods for culturing microorganisms have been developed, such as in situ cultivation, co-culture and continuous flow bioreactor. In this mini-review, we briefly introduce the development of new culture-dependent technology of continuous flow bioreactor and indicate the future research opportunities in this area.
Keywords: "uncultured" microorganismsculture techniquescontinuous flow bioreactormicrobial resources
自然环境中存在的微生物仅有1%甚至 < 1%能在实验室条件下被培养[1]。早在100多年前,科学家在用平板培养环境中微生物时就发现平板上形成的肉眼可见的菌落数和通过显微计数得到的菌落数存在很大差异,在平板上生长的菌落远远少于环境中实际存在的微生物即“平板数异常”现象[2-4]。那些99%在其原生环境中具有代谢活性,但在普通琼脂平板上不能正常增殖、处于休眠状态的微生物[5]被称为“未培养”微生物,该概念最早由Colwell等[6]在1982年提出。
“未培养”微生物蕴含着巨大的未被开发的活性物质资源[7],并且具有新颖的生物和化学特性[8],这些生物资源亟待被挖掘以解决目前天然活性物质短缺等问题[9]。在分子生物学技术的帮助下,“未培养”微生物的功能多样性已经得到了一致的证明;基于环境DNA/RNA分析技术如宏基因组学、单细胞基因组学,也使预测“未培养”微生物的生理生态功能成为可能。虽然这些工具能够证明微生物的代谢多样性,但由于微生物纯培养技术的限制,许多微生物的特性仍然是未知的[10-11]。微生物的功能往往不能单独地从遗传信息中推断出来;而且微生物的生理特征如细胞大小、形态、色素沉着、代谢活性也只能通过活的细胞来进行研究[5, 12],因此,微生物的分离和培养仍然是必不可少的。
“未培养”微生物并不是意味着不能被培养,而是由于对目标微生物生理代谢途径的认识不足和传统的培养技术的局限导致这些“未培养”微生物无法在人工创造的环境里正常生长[13],其局限性主要有以下几方面:(1) 不合适的培养条件如培养基的富营养化、培养时间的不充分[14-15];传统琼脂平板通常利用营养浓度很高的培养基[16],而大多数微生物生长在寡营养的环境中如海洋微生物,营养丰富的培养基反而会抑制它们的生长[17];环境中很多微生物生长缓慢,有些需要培养几个月才能在平板上形成肉眼可见的菌落[18],而短期的培养则无法分离到那些生长速率很低的微生物;(2) 常规的培养方法无法避免生长速率快的菌群对生长速率慢的菌群的抑制作用,并且传统的培养基在灭菌时产生的H2O2对菌落存在抑制作用[19-21];传统培养基的富营养化使生长速率快的菌群在大量生长繁殖时快速消耗培养基中的营养和空间,而生长缓慢的微生物却因养分等因素的限制而无法形成菌落,并且它们在生长过程中产生的过氧化物、超氧化物等有毒代谢产物也会抑制其他微生物的生长;此外,传统的培养基在制备时,琼脂和磷酸盐的混合灭菌同样会产生活性氧,抑制微生物菌落的形成[21];(3) 传统的人工培养基缺乏在自然环境中存在的某些生长因子或信号分子,有些依赖型微生物在缺乏这些生长因子或信号分子的情况下无法进行正常的生长代谢[22]。
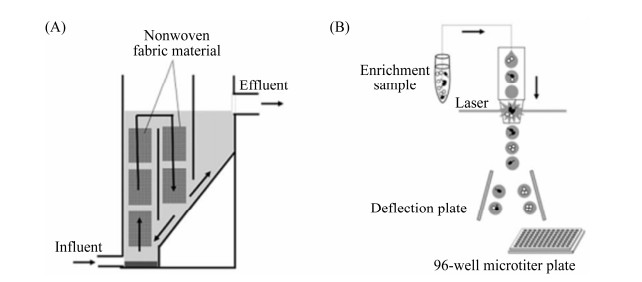
为了挖掘“未培养”微生物潜在的资源,解决传统培养方法存在的问题,很多改进的方法和新技术已被应用于“未培养”微生物的培养。主要有:(1) 改善培养条件如pH、温度、环境气体[22];延长培养时间、使用低营养浓度的培养基[20]和培养基成分的分开灭菌[23];(2) 膜过滤[24]和连续稀释[25-26]降低来自生长速率较快的菌群的不利影响;(3) 培养基中生长因子的添加[27]、依赖型微生物的共培养[28-29]和模拟微生物原生环境的原位培养技术如扩散室、I-chip等[30-32]。虽然通过这些策略已取得一些成果,但仍不足以大幅提高微生物的可培养率。
连续流生物反应器(continuous flow bioreactor)最初被用于处理城市污水。随着不断地改进和发展,几种具有相似基本概念的连续流生物反应器已被成功地应用于具有特定代谢途径的细菌和海绵共生细菌的富集和培养[33-35]。因此,连续流生物反应器是挖掘“未培养”微生物资源的一种强有力的新技术,而且通过连续流生物反应器了解到的培养机制可以为新型培养技术的发展提供新思路。本文综述了目前已经发展的各种连续流生物反应器培养技术,对这些技术进行了评价和技术特点的总结,并为连续流生物反应器技术下一步的发展指明方向。
1 连续流生物反应器的发展与改进 作为微生物培养的新技术,连续流生物反应器最早并不是应用于微生物的培养,而是作为城市污水的后处理技术之一;随着不断改进,已被应用于环境碳氮循环中发挥重要生态作用的细菌菌群如亚硝酸盐氧化细菌、氨氧化细菌和产甲烷微生物群落的富集和培养;最近也有研究者应用该技术培养海绵共生细菌,分离得到的未培养细菌数远远高于传统平板培养。由此可见,连续流生物反应器作为一种新的培养技术具有很好的发展前景。
1.1 连续流生物反应器应用于污水处理
1.1.1 下流式悬挂海绵生物反应器处理污水: 高速无氧反应器普遍应用于处理各种污水,其成本低、所需空间小并且对污水中有机物的去除率高。这些反应器依赖于生物量高浓度的固定,其中,上流式厌氧污泥床(UASB)反应器因其更简便、经济而被认为相对较好,污水经过厌氧处理后残留的有机物会对自然环境中水体造成污染,因此污水中有机物的去除非常重要。
1997年Agrawal等[36]建立了UASB反应器,开发了悬挂海绵反应器作为污水后处理工艺,通过下流式悬挂海绵过滤器(DSH)研究污水中残余有机物的去除和硝化作用并且通过上流淹没式悬挂床生物反应器(USHB)研究无氧条件下污水中的反硝化作用。UASB反应器(47.1 L)由两部分组成:一个高1800 mm,内径150 mm的锥形底圆柱和一个气液固分离器。DHS生物反应器含有3个2 m高的水柱,每个水柱填充120个用尼龙线串成的海绵体(1.5 cm)。USHB反硝化生物反应器的液体体积为770 mL,其中海绵块约占总体积的28% (图 1)。反应器开始运行时,向UASB反应器接种消化过的污水污泥。从UASB反应器流出的污水被泵入DHS生物反应器并依次从3个水柱流过,进入装有海绵块的USHB反应器。实验结果表明,通过悬挂海绵后处理过程,污水中有机物和氮可以被有效去除。但该反应器容量大,且由3个反应器组成,操作繁琐。
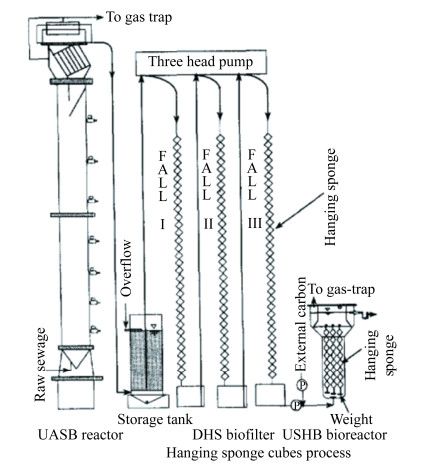 |
| 图 1 下流式悬挂海绵生物反应器结构示意图[36] Figure 1 Schematic diagram of down-flow hanging sponge reactor[36]. |
| 图选项 |
1.1.2 连续流生物反应器用于高温好氧废水处理: 生物废水处理工艺利用混合微生物降解污染物。传统的好氧处理工艺如活性污泥可适当地降解废水中的污染物,但细胞产量高,会产生相当数量的残余生物固体。因此,多种厌氧处理工艺已被开发用于处理含有高浓度有机物的污水。然而,厌氧处理过程以产生低质量的废水而闻名,如果有机酸生产者和产甲烷微生物之间出现不平衡,则可能出现难操作的问题。因此,高温好氧处理工艺应运而生,以处理含有高浓度的可溶性易降解有机物的工业废水[37-38]。2000年,Timothy等[39]将连续流生物反应器应用于工业废水的高温好氧处理,并研究混合的嗜热好氧细菌菌群在反应器中的生长特点、生理特征和群落结构。
连续培养在实验室580 mL的发酵罐、55 ℃环境中进行。发酵罐中接种污泥,将化学需氧量(COD)为3100 mg/L的无菌培养基通过变速控制驱动泵以指定的速率泵入反应器中,连续培养直到生物量浓度达到稳定状态(OD600变化小于5%)。一旦达到稳定状态,反应器再运行3个水力停留时间(HRT)以确保达到了可识别的生理状态。通过对运行了不同水力停留时间的反应器中的化学需氧量和微生物群落的监测发现,随着水力停留时间的增加,反应器中的COD含量显著降低并且微生物优势菌群也发生变化。在有效去除污水中有机物的同时,反应器中高温好氧废水处理的指标如细胞产量、细菌群落转移、细胞反应性和细胞活性等与传统中温处理工艺区别不大。
1.2 连续流生物反应器培养特定微生物群落
1.2.1 厌氧生物滤液反应器富集厌氧氨氧化菌: 厌氧氨氧化(Anammox)工艺被认为是一种经济有效的处理工艺。该工艺可以达到每天8.9 kg N/m3的脱氮率[39],可使反应器的容量比传统系统更小。然而厌氧氨氧化细菌的低生长速率使其不能实用于废水处理。
为了提高厌氧氨氧化菌的生长速率,2006年Isaka团队[33]利用厌氧生物滤液反应器(anaerobic biological filtrated reactor)在短时间内富集了厌氧氨氧化菌。该反应器使用内径为19 mm、高度为177 mm、有效容量为200 mL的圆柱形储罐作为反应池,反应池中装有多孔的聚酯无纺布载体(厚度为0.7 cm)作为厌氧菌的固定床。无纺布在反应池中的填充百分比设置为48%。水力停留时间(HRT)设置为3 h。实验时,将该反应池浸泡于37 ℃水浴中并接种富含厌氧氨氧化菌的种子污泥。从反应池底部连续供应培养基,从顶部排出多余的培养基。通过这种连续培养,反应池中氮转化率逐渐提高,亚硝酸盐含量迅速降低(图 2),表明反应器中的厌氧氨氧化菌随着连续培养的进行而不断增加,成功富集了目标菌群厌氧氨氧化菌,并且能够保持厌氧氨氧化菌的活性。
 |
| 图 2 连续流厌氧生物反应器中流入和流出的氮浓度、氮转化率(A)以及流入和流出的亚硝酸盐浓度(B)随时间的变化[33] Figure 2 Changes of influent and effluent nitrogen concentrations; nitrogen conversion rate (A) and influent and effluent nitrite concentrations (B) during continuous flow cultivation of anammox bacteria against time[33]. |
| 图选项 |
1.2.2 连续流生物反应器培养海洋产甲烷菌: 为了阐明微生物产甲烷作用在海底环境中的重要性,需要对其细胞实体进行鉴定,然而,传统的培养方法难以培养产甲烷微生物。Imachi等在2011年[34]首次报道了下流式悬挂海绵(DHS)反应器用于培养海底沉积物中产甲烷菌。
该下流式悬挂海绵反应器由密闭的聚氯乙烯(PVC)箱(内部容积为10.7 L)和作为微生物细胞的载体材料聚氨酯海绵块构成(3 cm×3 cm×3 cm,孔径0.83 mm)。接种时,于4 ℃下将海绵立方体浸泡在不含任何有机物质的混合沉积物浆液中,然后装入塑料网中以防止海绵压碎(图 3),把包裹好的147个海绵体放进PVC箱里,接种过程中,始终用氮气冲洗浆料和PVC箱。接种后,将PVC盒紧紧盖好,放置于黑暗的10 ℃恒温箱中。将经过氮气净化、pH值为7.5、含有机物质但不含硫酸盐的10 L合成海水通过连接在蠕动泵的自动开/关定时器间歇地从PVC盒顶部供应到其中,并通过分配器分配到海绵载体中,合成海水随后流下,在重力作用下穿过海绵载体,最后被抽出PVC箱。整个反应器在常压下运行。反应器中的水力停留时间为84 h。Imachi等通过DHS反应器作为培养的第一步,在反应器里检测到系统发育多样的微生物,并成功分离到传统方法未分离到的产甲烷菌。用聚氨酯海绵作为微生物的人工载体,是培养海底微生物的理想场所,能够模拟海底沉积物的多孔层(灰层和砂层)。该DHS反应器的显著特点是海绵不会被淹没并自由地放置在大气中[40]。因此,合成海水进入反应器时由于重力作用向下流入海绵,使合成海水不仅流在海绵表面,而且能在海绵内部进行有效交换。这些优势使得DHS生物反应器系统比其他连续流系统产生更大的生物量,促进之后目标微生物的分批培养。最后利用新设计的DHS反应器,成功地从海底沉积物中富集出产甲烷微生物群落并在后续的纯培养中获得了10种厌氧微生物,包括4种产甲烷古菌。但该反应器存在体积庞大,操作较繁琐等问题。
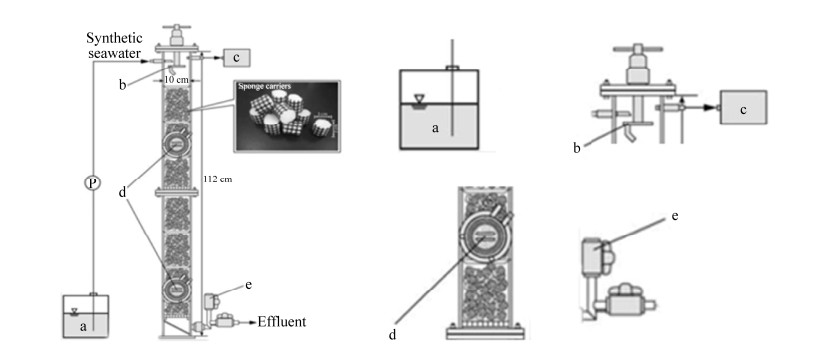 |
| 图 3 产甲烷菌连续流生物反应器结构示意图[34] Figure 3 Schematic diagram of the CF reactor for cultivation of methanogenic community[34]. a: Synthetic seawater tank; b: distributor; c: gas collecting bag; d: sponge carrier sampling port; e: port for the pH/ORP sensor. |
| 图选项 |
1.2.3 连续流生物反应器培养硝化螺旋菌属: 硝化作用是生物地球化学氮循环中的一个重要过程,在污水处理中也得到了广泛的应用。然而,由于硝化螺旋菌的难分离和难培养,人们对其生理特性了解甚少。2014年Fujitani等[41]通过一种连续喂养反应器培养技术选择性地富集硝化螺旋菌(Nitrospira),并通过细胞分选技术将目标菌株与其他菌群分离,最后纯培养得到硝化螺旋菌(图 4)。
 |
| 图 4 硝化螺旋菌属的连续培养(A)和纯化(B)[41] Figure 4 Continuous cultivation (A) and purification (B) for the isolation of Nitrospira[41]. |
| 图选项 |
首先建立一个装有多孔聚酯无纺布材料(厚度为0.7 cm)构成的生物载体的连续进料生物反应器以使细菌保持活性状态,生物反应器的体积为2 L。无机培养基的pH调节为7.8-8.3,水力停留时间(HRT)设为1.6-4.8 h。反应器运行时,连续高流速地将低营养浓度的无机培养基提供到反应器,并且持续给培养物通气以提供过量的氧气。当培养基体积达到反应器最大容量,无活动的细胞,细胞外聚合物质和代谢副产物则被排出反应器。整个实验在23 ℃的环境中进行。定期提高培养基中亚硝酸盐-氮浓度至0.36 mmol/L、1.43 mmol/L和5.71 mmol/L;同时,水力停留时间由4.8 h逐渐缩短至1.6 h。之后定期采用荧光原位杂交技术检测反应池中硝化螺旋菌的比例,发现硝化螺旋菌的比例随着培养时间的增加而增加,甚至高达80%。该反应器能够将与其他硝化细菌共存且生长缓慢的硝化螺旋菌选择性地富集并成功地分离到第一个被分类为硝化螺旋亚菌I的分离物,同时与细胞分选技术相结合提高了目标菌株的分离纯化的效率。因此,该新方法还可用于培养其他具有特定代谢途径和重要生态功能的未培养“k策略者”细菌,具有广泛的应用性。
1.2.4 封闭式下流悬挂海绵反应器培养反硝化厌氧甲烷氧化微生物: 污水的厌氧处理虽然能去除有机物和营养物,但同时也使处理过的废水中含有高浓度的溶解甲烷。因此,经过厌氧前处理的污水需要经过后处理去除甲烷。封闭式下流悬挂海绵反应器处理工艺被认为是污水厌氧后处理中最有前景的工艺之一[42]。反硝化厌氧甲烷氧化微生物能够厌氧氧化甲烷,从而为硝酸盐和亚硝酸盐的反硝化提供电子[43],为污水中甲烷的去除提供了一种新思路[44]。2017年Hatamoto等[45]首次报道了利用封闭式下流悬挂海绵反应器培养反硝化厌氧甲烷氧化(DAMO)微生物来去除污水中氮气和溶解甲烷。
该封闭式下流悬挂海绵反应器包含由15个聚氨酯海绵块(2 cm×2 cm×2 cm)构成的、垂直总长度为50 cm的海绵介质,其总体积为0.12 L,孔隙率为0.985。将它们悬挂在已经接种了污泥的1.25 L玻璃柱中。实验时先将经过氩气和甲烷冲洗过、NaNO2和NaNO3的浓度为0.5 mmol/L的培养基供应到反应器中,之后再将培养基中NaNO2和NaNO3的浓度分别调节到0.7 mmol/L和1.0 mmol/L (图 5)。同时向瓶子和气囊的顶部空间充入甲烷。反应器在30 ℃的恒温箱中运行。运行176 d后,甲烷以2.0 mL/min的流速直接供给反应器。实验结果表明DHS反应器保留了接种污泥中最初的DAMO群落并且将NC10门的DAMO菌在反应器中的丰度提高到50%-70%,其中部分菌株为新种。虽然该反应器富集目标微生物的效果显著,但该反应器运行时间太长,前后长达2年。
 |
| 图 5 DHS反应器操作条件(A, C)和工艺性能(B, D)[45] Figure 5 Operational conditions (A, C) and process performance (B, D) of the DHS reactor[45]. |
| 图选项 |
1.3 连续流生物反应器培养多种微生物
1.3.1 连续流生物反应器培养海绵共生细菌: 连续流生物反应器在富集特定菌群方面具有显著成效。Jung等[46]对反应器简化后首次将其用于培养海绵共生细菌,建立总体积为500 mL、内部装有多孔聚酯非织造布(厚度0.5 cm)[47]的样品室。实验时海绵样品用无菌人工海水冲洗3次,然后用无菌研钵将其研磨成粉并用无菌人工海水制成匀浆,共取200 mL的海绵组织匀浆作为接种剂接种于样品室。盐度3.5%、营养浓度10%的R2A液体培养基由蠕动泵以每天300 mL的容量被供应(水力停留时间为24 h)到样品室中,同时向其中充气以提供氧气。整个反应器于20 ℃环境中运行。实验期间每隔3-7 d测量生物反应器中的总有机碳浓度以监测反应器里的生长条件。同时设置对照组实验(标准直接涂布和原位培养)。实验结果表明,相比于标准直接涂布法1.6%和原位培养技术23%的新种率,其新种率高达45% (图 6)。反应器里的多孔聚酯材料和天然环境的海绵具有相似的结构组织,能为海绵共生细菌提供一个类似于天然海绵的栖息地同时达到将细菌保持在活性状态而不过度生长的目的。虽然反应器中能够培养大量未培养微生物,但附着于多孔聚酯材料上的微生物需要接种到平板上进行进一步的培养和分离,而很多微生物虽然能在反应器中生长,但当它们转移到普通平板上时不一定会形成肉眼可见的菌落。还有一点不足的是需要时常监测保证供应的培养基不能被杂菌污染,否则将影响实验结果。
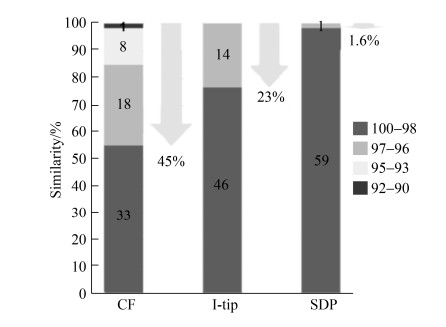 |
| 图 6 不同来源菌株基于16S rRNA基因与GenBank数据库中已知亲缘关系最近的菌株的相似性[46] Figure 6 Similarity based on 16S rRNA gene to the closest known relative of isolates from CF (continuous flow bioreactor), i-tip and SDP (standard direct plating). The numbers in the bar show the number of OTUs at each similar level; the numbers under the arrows show the percentage of new species[46]. |
| 图选项 |
1.3.2 连续流生物反应器在海洋微生物资源挖掘中的应用: 对于培养“未培养”微生物,相较于传统培养方法,连续流生物反应器具有很多优势。反应器不仅能持续为微生物提供新鲜的养料,给微生物提供一个开放的生长环境,还能满足微生物间的相互作用。应用连续流生物反应器培养宁波梅山湾海底沉积物中微生物时,可获得系统发育多样的微生物(图 7),因此连续流作为挖掘海洋微生物资源的一种有效手段,未来还能够更广泛地应用于更多其他的海洋样品。
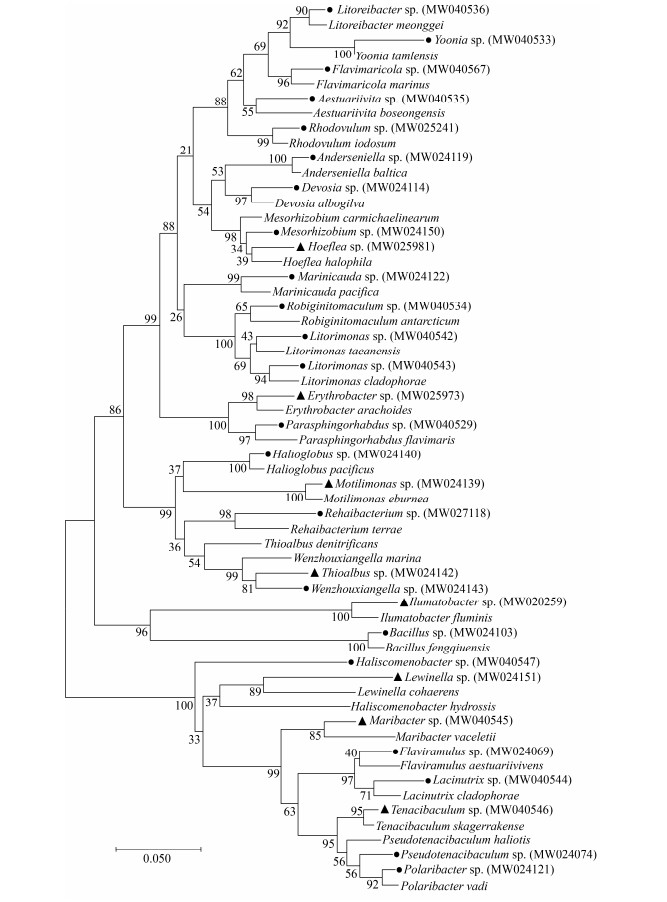 |
| 图 7 连续流生物反应器分离株基于16S rRNA基因序列的系统发育树 Figure 7 Phylogenetic tree based on 16S rRNA gene sequences of novel isolates obtained by CF cultivation. The isolates derived from CF cultivation methods are shown with circle and triangle, others are the closest strains in database of EZBIOCLOUD (https://www.ezbiocloud.net); circle (beach sediment), triangle (sediment with plant) indicate environmental sources of the isolates; the NCBI/GenBank accession numbers for isolates from CF are shown in parentheses; the number at the branch node represents the bootstrap value; the scale represents genetic distance. |
| 图选项 |
2 连续流培养的特点和机制 相比于其他微生物培养技术,连续流生物反应器具有明显优势,可以同时满足目标菌群多种的生长需求。首先生物反应器里放置的多孔纤维材料在培养基中可以提供足够的孔隙空间,为微生物生长提供更大的表面积和更长的细胞停留时间,更有利于微生物在反应器里附着和生长[33];其次,反应器可以对底物浓度和流速实现更灵活的控制。此外,培养基通过蠕动泵在反应器中不断供给而又连续排放,能够在体系里一直保持新鲜,避免微生物在生长过程中产生的有毒代谢产物对体系里其他微生物的抑制[34],最后生物反应器本质上就是多种微生物的共培养,没有破坏微生物之间的联系和种间的信息交流[35],因此相比于传统培养技术能培养更多未培养微生物。
而对于连续流生物反应器富集“未培养”微生物的机制,Jung等[46]对其进行了探究,将通过连续流生物反应器、原位培养和标准直接涂布等不同方法分离得到的细菌进行了生理特征的比较,发现通过连续流培养出的细菌具有以下2个特征:第一,通过连续流培养的细菌生长速度普遍较低;第二,在连续流反应器里生长的菌落的直径远远小于其他方法分离到的菌落(图 8),说明连续流生物反应器创造的生长条件适合那些生长缓慢、对环境较敏感的细菌的生长,而具有这2个特征的菌群往往是受到标准培养和原位培养条件的限制,易被忽略和难分离的未培养菌群。
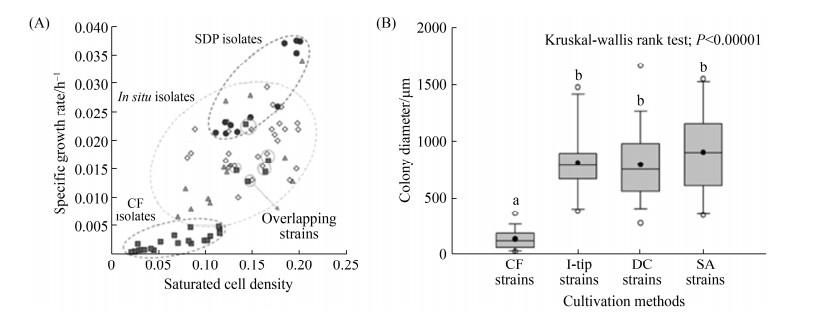 |
| 图 8 不同来源菌株的比生长速率、饱和细胞密度(A)和菌落直径(B)[46] Figure 8 Specific growth rate, saturated cell density (A) and colony diameter (B) of strains from CF (continuous flow bioreactor), in situ cultivation and SDP (standard direct plating)[46]. |
| 图选项 |
3 总结和展望 分子生物学技术特别是基因组学和宏基因组学的发展证明了环境中微生物的多样性,但其中有超过99%的微生物并不能在常规实验条件下得到纯培养,严重阻碍了人们对未培养微生物资源的挖掘和利用,如何革新和开发出更有效的微生物培养技术成为研究者们共同面临的问题。虽然随着微生物纯培养技术的发展,在微生物培养方面也取得了一些实质性的进展,但对于环境中数量庞大、种类繁多的“未培养”微生物群体而言远远不够,在探索微生物多样性及可培养性的过程中仍面临着巨大挑战。虽然连续流生物反应器作为一种新的微生物培养技术,在微生物培养方面已经表现出很多优势,但同时也存在一些不足之处。在未来微生物培养的研究中,连续流培养的研究重点主要在以下几个方面。
3.1 连续流反应器中微生物间相互作用的研究 连续流反应器实际上是样品中所有微生物的共培养,微生物在反应器里可以进行信息的交流。但是我们并不知道具体是哪些菌群之间存在相互作用,如何进行种间信息交流。所以连续流反应器是研究微生物间相互作用强有力的手段。已经有研究者应用microbial co-occurrence networks analysis技术找出特定菌种之间的相互作用[48]。因此未来可以将连续流反应器这种技术结合起来研究特定微生物间相互作用和寻找信息分子,这将会进一步推动微生物的分离培养技术以及连续流培养技术的新发展。
3.2 设计新型的选择培养基 培养基直接影响微生物生长,特异性地添加微生物生长所需的微量元素、生长因子和微生物分泌的信号分子是既简便又有效的方法,但因为缺乏对微生物代谢方式的了解,我们并不知道未培养微生物缺乏什么生长因子。因此未来微生物的研究应侧重于通过新型的培养方法深入地探索微生物可培养的机制然后进行培养基的优化,设计开发出适合不同种微生物的选择培养基,将新型选择培养基结合连续流反应器培养微生物,具有更好的应用前景。
3.3 分子生物学技术与连续流反应器的结合 由于微生物生存环境的复杂性及其群落结构随环境变化而变化的特征,单独依赖某一种或某一类方法研究微生物多样性是远远不够的。因此在连续流生物反应器的基础上,进一步将其与高灵敏的观察检测技术和高效率的细胞分离技术结合起来非常重要。因此,将荧光显微技术、流式细胞技术、可分辨环境样品中细胞生长情况的双链核酸染色等分子生物学技术与纯培养技术结合应用,将会极大提高微生物的可培养性[49-50]。
3.4 连续流反应器中代谢产物的研究 “未培养”微生物的难培养阻碍了我们对“未培养”微生物蕴含的代谢产物的挖掘,而连续流生物反应器可以让一些之前在传统平板上不能生长的微生物得以生长,并且在生长过程中产生各种代谢产物。因此能否利用以及如何利用连续流生物反应器进行微生物代谢的挖掘是一个值得探索的问题,而这也将赋予连续流生物反应器更加深远的意义。
References
| [1] | Ferrari BC, Binnerup SJ, Gillings M. Microcolony cultivation on a soil substrate membrane system selects for previously uncultured soil bacteria. Applied and Environmental Microbiology, 2005, 71(12): 8714-8720. DOI:10.1128/AEM.71.12.8714-8720.2005 |
| [2] | Hugenholtz P, Goebel BM, Pace NR. Impact of culture-independent studies on the emerging phylogenetic view of bacterial diversity. Journal of Bacteriology, 1998, 180(18): 4765-4774. DOI:10.1128/JB.180.18.4765-4774.1998 |
| [3] | Staley JT, Konopka A. Measurement of in situ activities of nonphotosynthetic microorganisms in aquatic and terrestrial habitats. Annual Review of Microbiology, 1985, 39: 321-346. DOI:10.1146/annurev.mi.39.100185.001541 |
| [4] | Amann RI, Ludwig W, Schleifer KH. Phylogenetic identification and in situ detection of individual microbial cells without cultivation. Microbiological Reviews, 1995, 59(1): 143-169. DOI:10.1128/MR.59.1.143-169.1995 |
| [5] | Stewart EJ. Growing unculturable bacteria. Journal of Bacteriology, 2012, 194(16): 4151-4160. DOI:10.1128/JB.00345-12 |
| [6] | Xu HS, Roberts N, Singleton FL, Attwell RW, Grimes DJ, Colwell RR. Survival and viability of nonculturable Escherichia coli and Vibrio cholerae in the estuarine and marine environment. Microbial Ecology, 1982, 8(4): 313-323. DOI:10.1007/BF02010671 |
| [7] | Vartoukian SR, Adamowska A, Lawlor M, Moazzez R, Dewhirst FE, Wade WG. In vitro cultivation of 'unculturable' oral bacteria, facilitated by community culture and media supplementation with siderophores. PLoS One, 2016, 11(1): e0146926. DOI:10.1371/journal.pone.0146926 |
| [8] | Nichols D, Lewis K, Orjala J, Mo S, Ortenberg R, O'Connor P, Zhao C, Vouros P, Kaeberlein T, Epstein SS. Short peptide induces an "uncultivable" microorganism to grow in vitro. Applied and Environmental Microbiology, 2008, 74(15): 4889-4897. DOI:10.1128/AEM.00393-08 |
| [9] | Newman DJ. Predominately uncultured microbes as sources of bioactive agents. Frontiers in Microbiology, 2016, 7: 1832. |
| [10] | van Dorst JM, Hince G, Snape I, Ferrari BC. Novel culturing techniques select for heterotrophs and hydrocarbon degraders in a subantarctic soil. Scientific Reports, 2016, 6: 36724. DOI:10.1038/srep36724 |
| [11] | Gawad C, Koh W, Quake SR. Single-cell genome sequencing: current state of the science. Nature Reviews Genetics, 2016, 17(3): 175-188. DOI:10.1038/nrg.2015.16 |
| [12] | Heath JR, Ribas A, Mischel PS. Single-cell analysis tools for drug discovery and development. Nature Reviews Drug Discovery, 2016, 15(3): 204-216. DOI:10.1038/nrd.2015.16 |
| [13] | Jaisoo K. Review and future development of new culture methods for unculturable soil bacteria. Korean Journal of Microbiology, 2011, 47(3): 179-187. |
| [14] | Chaudhary DK, Khulan A, Kim J. Development of a novel cultivation technique for uncultured soil bacteria. Scientific Reports, 2019, 9(1): 6666. DOI:10.1038/s41598-019-43182-x |
| [15] | Janssen PH, Yates PS, Grinton BE, Taylor PM, Sait M. Improved culturability of soil bacteria and isolation in pure culture of novel members of the divisions Acidobacteria, Actinobacteria, Proteobacteria, and Verrucomicrobia. Applied and Environmental Microbiology, 2002, 68(5): 2391-2396. DOI:10.1128/AEM.68.5.2391-2396.2002 |
| [16] | Davis KER, Sangwan P, Janssen PH. Acidobacteria, Rubrobacteridae and Chloroflexi are abundant among very slow-growing and mini-colony-forming soil bacteria. Environmental Microbiology, 2011, 13(3): 798-805. DOI:10.1111/j.1462-2920.2010.02384.x |
| [17] | Vartoukian SR, Palmer RM, Wade WG. Strategies for culture of 'unculturable' bacteria. FEMS Microbiology Letters, 2010, 309(1): 1-7. |
| [18] | Fan NS, Qi R, Yang M. Current technical progresses in the cultivation for uncultured microorganism. Chinese Journal of Applied and Environmental Biology, 2016, 22(3): 524-530. (in Chinese) 范念斯, 齐嵘, 杨敏. 未培养微生物的培养方法进展. 应用与环境生物学报, 2016, 22(3): 524-530. |
| [19] | Pulschen AA, Bendia AG, Fricker AD, Pellizari VH, Galante D, Rodrigues F. Isolation of uncultured bacteria from Antarctica using long incubation periods and low nutritional media. Frontiers in Microbiology, 2017, 8: 1346. DOI:10.3389/fmicb.2017.01346 |
| [20] | Davis KE, Joseph SJ, Janssen PH. Effects of growth medium, inoculum size, and incubation time on culturability and isolation of soil bacteria. Applied and Environmental Microbiology, 2005, 71(2): 826-834. DOI:10.1128/AEM.71.2.826-834.2005 |
| [21] | Tanaka T, Kawasaki K, Daimon S, Kitagawa W, Yamamoto K, Tamaki H, Tanaka M, Nakatsu CH, Kamagata Y. A hidden pitfall in the preparation of agar media undermines microorganism cultivability. Applied and Environmental Microbiology, 2014, 80(24): 7659-7666. DOI:10.1128/AEM.02741-14 |
| [22] | Kawasaki K, Kamagata Y. Phosphate-catalyzed hydrogen peroxide formation from agar, gellan, and κ-carrageenan and recovery of microbial cultivability via catalase and pyruvate. Applied and Environmental Microbiology, 2017, 83(21): e01366. DOI:10.1128/aem.01366-17 |
| [23] | Ward DM, Weller R, Bateson MM. 16S rRNA sequences reveal numerous uncultured microorganisms in a natural community. Nature, 1990, 345(6270): 63-65. DOI:10.1038/345063a0 |
| [24] | Kato S, Terashima M, Yama A, Sato M, Kitagawa W, Kawasaki K, Kamagata Y. Improved isolation of uncultured anaerobic bacteria using medium prepared with separate sterilization of agar and phosphate. Microbes and Environments, 2020, 35(1). DOI:10.1264/jsme2.me19060 |
| [25] | Imazaki I, Kobori Y. Improving the culturability of freshwater bacteria using FW70, a low-nutrient solid medium amended with sodium pyruvate. Canadian Journal of Microbiology, 2010, 56(4): 333-341. DOI:10.1139/W10-019 |
| [26] | Joseph SJ, Hugenholtz P, Sangwan P, Osborne CA, Janssen PH. Laboratory cultivation of widespread and previously uncultured soil bacteria. Applied and Environmental Microbiology, 2003, 69(12): 7210-7215. DOI:10.1128/AEM.69.12.7210-7215.2003 |
| [27] | Schoenborn L, Yates PS, Grinton BE, Hugenholtz P, Janssen PH. Liquid serial dilution is inferior to solid media for isolation of cultures representative of the Phylum-level diversity of soil bacteria. Applied and Environmental Microbiology, 2004, 70(7): 4363-4366. DOI:10.1128/AEM.70.7.4363-4366.2004 |
| [28] | Bruns A, Cypionka H, Overmann J. Cyclic AMP and acyl homoserine lactones increase the cultivation efficiency of heterotrophic bacteria from the central Baltic sea. Applied and Environmental Microbiology, 2002, 68(8): 3978-3987. DOI:10.1128/AEM.68.8.3978-3987.2002 |
| [29] | Burm?lle M, Johnsen K, Al-Soud WA, Hansen LH, S?rensen SJ. The presence of embedded bacterial pure cultures in agar plates stimulate the culturability of soil bacteria. Journal of Microbiological Methods, 2009, 79(2): 166-173. DOI:10.1016/j.mimet.2009.08.006 |
| [30] | D'Onofrio A, Crawford JM, Stewart EJ, Witt K, Gavrish E, Epstein S, Clardy J, Lewis K. Siderophores from neighboring organisms promote the growth of uncultured bacteria. Chemistry & Biology, 2010, 17(3): 254-264. |
| [31] | Ferrari BC, Winsley T, Gillings M, Binnerup S. Cultivating previously uncultured soil bacteria using a soil substrate membrane system. Nature Protocols, 2008, 3(8): 1261-1269. DOI:10.1038/nprot.2008.102 |
| [32] | Kaeberlein T, Lewis K, Epstein SS. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science, 2002, 296(5570): 1127-1129. DOI:10.1126/science.1070633 |
| [33] | Bollmann A, Lewis K, Epstein SS. Incubation of environmental samples in a diffusion chamber increases the diversity of recovered isolates. Applied and Environmental Microbiology, 2007, 73(20): 6386-6390. DOI:10.1128/AEM.01309-07 |
| [34] | Isaka K, Date Y, Sumino T, Yoshie S, Tsuneda S. Growth characteristic of anaerobic ammonium-oxidizing bacteria in an anaerobic biological filtrated reactor. Applied Microbiology and Biotechnology, 2006, 70(1): 47-52. DOI:10.1007/s00253-005-0046-2 |
| [35] | Imachi H, Aoi K, Tasumi E, Saito Y, Yamanaka Y, Saito Y, Yamaguchi T, Tomaru H, Takeuchi R, Morono Y, Inagaki F, Ken TK. Cultivation of methanogenic community from subseafloor sediments using a continuous-flow bioreactor. The ISME Journal, 2011, 5(12): 1913-1925. DOI:10.1038/ismej.2011.64 |
| [36] | Toit MD, Knopfel S, S k, Ouml; WRONEK FA, Specht I, B, Ouml; CKELER W, Holzapfel WH. A modified continuous flow culture system for studying microbial interactions related to the gastro-intestinal tract: application of the probiotic organism Enterococcus faecium SF68. Bioscience and Microflora, 1998, 17(1): 41-47. DOI:10.12938/bifidus1996.17.41 |
| [37] | Agrawal LK, Harada H, Okui H. Treatment of dilute wastewater in a UASB reactor at a moderate temperature: Performance aspects. Journal of Fermentation and Bioengineering, 1997, 83(2): 179-184. DOI:10.1016/S0922-338X(97)83579-9 |
| [38] | Rozich AF, Colvin RJ. Design and operational considerations for thermophilic aerobic reactors treating high strength wastes and sludges. //Proceedings of the 52nd Industrial Waste Conference May 5-7, CRC Press, 1997: 1-5. |
| [39] | LaPara TM, Konopka A, Nakatsu CH, Alleman JE. Thermophilic aerobic wastewater treatment in continuous-flow bioreactors. Journal of Environmental Engineering, 2000, 126(8): 739-744. DOI:10.1061/(ASCE)0733-9372(2000)126:8(739) |
| [40] | Tandukar M, Uemura S, Ohashi A, Harada H. Combining UASB and the "fourth generation" down-flow hanging sponge reactor for municipal wastewater treatment. Water Science and Technology, 2006, 53(3): 209-218. DOI:10.2166/wst.2006.095 |
| [41] | Uemura S, Harada H. Application of UASB technology for sewage treatment with a novel post-treatment process. Environmental Anaerobic Technology, 2010: 91-112. |
| [42] | Fujitani H, Ushiki N, Tsuneda S, Aoi Y. Isolation of sublineage I?Nitrospira by a novel cultivation strategy. Environmental Microbiology, 2014, 16(10): 3030-3040. DOI:10.1111/1462-2920.12248 |
| [43] | Okubo T, Kubota K, Yamaguchi T, Uemura S, Harada H. Development of a new non-aeration-based sewage treatment technology: Performance evaluation of a full-scale down-flow hanging sponge reactor employing third-generation sponge carriers. Water Research, 2016, 102: 138-146. DOI:10.1016/j.watres.2016.06.035 |
| [44] | Welte CU, Rasigraf O, Vaksmaa A, Versantvoort W, Arshad A, Op den Camp HJM, Jetten MSM, Lüke C, Reimann J. Nitrate- and nitrite-dependent anaerobic oxidation of methane. Environmental Microbiology Reports, 2016, 8(6): 941-955. DOI:10.1111/1758-2229.12487 |
| [45] | Hatamoto M, Sato T, Nemoto S, Yamaguchi T. Cultivation of denitrifying anaerobic methane-oxidizing microorganisms in a continuous-flow sponge bioreactor. Applied Microbiology and Biotechnology, 2017, 101(14): 5881-5888. DOI:10.1007/s00253-017-8315-4 |
| [46] | Dawoon J, Koshi M, Yoichi N, Jeffrey SO, Tomonori K, Akiyoshi O, Akiyoshi O, Yoshiteru A. Cultivation of novel marine sponge-associated bacteria using advanced cultivation techniques: A perspective on possible key mechanisms. Applied and Environmental Microbiology, 2020, 10(3): 23-33. |
| [47] | Fujitani H, Aoi Y, Tsuneda S. Selective enrichment of two different types of Nitrospira-like nitrite-oxidizing bacteria from a wastewater treatment plant. Microbes and Environments, 2013, 28(2): 236-243. DOI:10.1264/jsme2.ME12209 |
| [48] | Xian WD, Salam N, Li MM, Zhou EM, Yin YR, Liu ZT, Ming YZ, Zhang XT, Wu G, Liu L, Xiao M, Jiang HC, Li WJ. Network-directed efficient isolation of previously uncultivated Chloroflexi and related bacteria in hot spring microbial mats. npj Biofilms and Microbiomes, 2020, 6: 20. DOI:10.1038/s41522-020-0131-4 |
| [49] | Zhang XM, Zhang XH. New culture approaches of marine microorganisms. Marine Sciences, 2009, 33(6): 99-104. (in Chinese) 张秀明, 张晓华. 海洋微生物培养新技术的研究进展. 海洋科学, 2009, 33(6): 99-104. |
| [50] | Alain K, Querellou J. Cultivating the uncultured: limits, advances and future challenges. Extremophiles, 2009, 13(4): 583-594. DOI:10.1007/s00792-009-0261-3 |
