梁锦有, 张骏梁

 , 郑晓丽, 陈烁钿, 杨鹏, 廖孟滨, 徐颖
, 郑晓丽, 陈烁钿, 杨鹏, 廖孟滨, 徐颖

深圳大学生命与海洋科学学院, 深圳市海洋生物资源与生态环境重点实验室, 深圳市海洋藻类生物开发与应用工程实验室, 广东 深圳 518060
收稿日期:2020-12-15;修回日期:2021-02-10;网络出版日期:2021-03-09
基金项目:国家重点研发计划(2018YFA0902500);深圳市科技创新委员会项目(KQJSCX20170727101743831)
作者简介:徐颖, 博士, 深圳大学生命与海洋科学学院****。深圳市国家级领军人才; 获2016年度国家自然科学二等奖(第二完成人)。长期从事海洋微生物资源的开发和活性物质的研究, 包括生物膜抑制剂、抗植物致病真菌的绿色农药以及抗炎、抗菌等海洋药物先导物。主持国家863计划子课题、国家自然科学基金等课题。迄今发表SCI论文80余篇, 其中以第一作者或通讯作者发表论文20余篇。部分文章被Nature Middle East、Jacs Spotlights和Faculty 1000专栏报道。以第二发明人拥有美国授权专利2项和中国授权专利2项.
*通信作者:张骏梁。E-mail: junliang_zhang90@hotmail.com;
徐颖。E-mail: boxuying@szu.edu.cn.
摘要:[目的] 研究深圳大鹏半岛海域石磺海牛中可培养的共附生细菌的数量和种类,并对分离获得菌株的代谢产物进行活性筛选。[方法] 通过R2A平板培养、分离纯化和16S rRNA测序,分析鉴定石磺海牛中5个部位可培养细菌;使用分离菌株的菌液及发酵液上清,测定对群体感应信号分子降解的活性和抗生物膜活性。[结果] 从石磺海牛中共分离到215株细菌,归属于87个种,54个属,有16株菌疑似为新菌。分离获得的菌株分布于变形菌门(Proteobacteria,126株),拟杆菌门(Bacteroidetes,44株),厚壁菌门(Firmicutes,34株),放线菌门(Actinobacteria,10株)和浮霉菌门(Planctomycetes,1株)。石磺海牛卵中的细菌数量和种类最为多样。从分离的菌株中筛选出28株菌能够降解群体感应信号分子,8株菌具有抗生物膜活性。[结论] 首次报道了海洋无脊椎动物石磺海牛中具有丰富多样的可培养细菌,包含潜在的新微生物和天然产物资源,为今后研究石磺海牛共附生微生物提供了研究基础和参考。
关键词:石磺海牛共附生细菌多样性活性代谢产物
Isolation and identification of the symbiotic bacteria of Homoiodoris japonica and screen of bioactive metabolite producing bacteria
Liang Jinyou, Zhang Junliang

 , Zheng Xiaoli, Chen Shuotian, Yang Peng, Liao Mengbin, Xu Ying
, Zheng Xiaoli, Chen Shuotian, Yang Peng, Liao Mengbin, Xu Ying

Shenzhen Key Laboratory of Marine Bioresource and Eco-environmental Science, Shenzhen Engineering Laboratory for Marine Algal Biotechnology, College of Life Sciences and Oceanography, Shenzhen University, Shenzhen 518060, Guangdong Province, China
Received: 15 December 2020; Revised: 10 February 2021; Published online: 9 March 2021
*Corresponding author: Junliang Zhang, E-mail: junliang_zhang90@hotmail.com;
Ying Xu, E-mail: boxuying@szu.edu.cn.
Foundation item: Supported by the National Key R & D Program of China (2018YFA0902500) and by the Scientific and Technical Innovation Council of Shenzhen (KQJSCX20170727101743831)
Abstract: [Objective] The present work aims to study the amounts and species of cultivable microbes and bioactive metabolite producing bacteria from a marine invertebrate collected in Shenzhen. [Methods] We dissected the invertebrate into five parts to isolated bacteria by R2A plate and identified them through the 16S rRNA sequence. Then use the culture broth and supernatant to screen the activity of degrading quorum-sensing signal molecules and anti-biofilm activity. [Results] A total of 216 strains of bacteria were isolated from the invertebrate, belonging to 87 species and 54 genera, and 16 strains were suspected to be new bacteria. Distributed in the phylum Proteobacteria (126 strains), Bacteroidetes (44 strains), Firmicutes (34 strains), Actinobacteria (10 strains), and Planctomycetes (1 strain). The number and species of bacteria in the eggs of the invertebrate were the most diverse. Twenty-eight strains were screened to degrade quorum-sensing signal molecules, and eight strains showed anti-biofilm activity. [Conclusion] It is the first report that the marine invertebrate Homoiodoris japonica-associated bacteria contains were abundant and diverse, including potential new species of bacteria and natural product resources, which may provide a research basis for future research on the Homoiodoris japonica-associated microbes.
Keywords: Homoiodoris japonicasymbiotic bacteriadiversitybioactive metabolites
海洋是最为丰富和多样化的生态系统之一,覆盖了地球近70%的面积、囊括95%以上的生物物种[1]。由于海洋环境具有严苛和复杂多变的化学和物理条件,造就了各种各样的生命形式,同时产生了许多化学结构独特、具有潜在生物活性的天然产物[2],成为药物发现和开发的重要来源之一[3]。随着从陆地寻找新的微生物和活性天然产物的难度越来越大,充分挖掘海洋中的生物和化学资源显得日益紧迫[3-4]。
从海洋中发现的新化合物已超过29000个,大部分有着抗肿瘤、抗氧化、抗菌和抗病毒等活性[5],衍生出多个商业化药物和在研的候选分子[6]。这些新的天然产物大部分是从海洋无脊椎动物中获得[7],而海洋无脊椎动物约占海洋现存非微生物物种的50%[8]。但海洋无脊椎动物来源的天然产物产量极低,而无脊椎动物的生物量难以实现可持续和可靠的供应,应用潜力受极大限制[9]。后来发现从海洋动植物中提取的天然产物,与其共附生微生物产生的代谢产物结构相似,人们逐渐认为多数海洋动植物的天然产物是其共附生微生物产生的[5, 10-11]。
海洋共附生微生物是指相对于游离生活在海水等自然环境中的微生物,它们和海洋动植物形成共生、共栖、寄生和附生关系。海洋无脊椎动物中有着丰富多样的共附生微生物[12],例如海绵中的细菌数量可能较海水中的细菌数量高2–4个数量级[13],微生物在珊瑚粘液中的数量可能比在海水中高100–1000倍[14]。海洋无脊椎动物及其共附生微生物形成的共生体在成长、营养互利、抵抗疾病和化学防御等方面起着重要的作用[8, 15-16]。由于共附生微生物及其宿主能够产生结构类似且具有应用潜力的活性天然产物[11],相比直接从宿主获取,利用共附生微生物对活性物质进行深入研究和规模化制备更可行。目前对无脊椎动物共附生微生物的研究主要集中于海绵[17-18]、海参[19]、苔藓虫[20]和珊瑚[21]等,发现了一些新菌新种[22-25],并筛选到具有抗菌、细胞毒、抗病毒、抗炎和抗污损等活性的菌株或天然产物[5, 11]。
群感效应(Quorum Sensing,QS)是广泛存在于细菌之间的一种通过感受细胞密度来调节基因表达的信号交流方式,通过分泌自诱导分子(autoinducer),调节细菌的代谢、毒力因子释放、次级代谢产物合成、生物膜形成等生理变化[26-27]。QS介导的毒力表达、生物膜形成和病原菌的定殖威胁着人类健康、水产养殖和作物栽培[28];细菌形成生物膜后,其对抗生素的敏感性大大降低,难以通过抗菌药物清除,是临床上造成持续感染的重要原因之一[29]。因此,寻找QS抑制剂(小分子化合物或QS淬灭酶)的有望成为干预QS控制细菌毒力或致病性的新手段[30],成为了新的研究热点。已有诸多文献报道了从海洋微生物中发现具有QS抑制活性的小分子或QS淬灭酶[27-28, 31],以及具有抗生物膜的作用的化合物[32-34],可见海洋微生物是挖掘QS抑制剂和抗生物膜活性物质的的宝库。
石磺海牛(Homoiodoris japonica)为腹足纲、裸鳃目的无脊椎动物,分布于我国和日本沿海,生活在潮间带到潮下带浅水区礁石下。目前对石磺海牛的研究较少,对其共附生微生物的分离与研究尚未见报道。本文所用的石磺海牛采集自深圳大鹏自然保护区海域,我们拟对其各部分的共附生细菌进行分离和鉴定,对分离到的菌株进行发酵,筛选能够降解QS信号分子酰基化高丝氨酸内酯(Acyl-homoserine lactone,AHL)及抗生物膜活性的菌株,以期为石磺海牛共附生细菌的多样性研究及活性天然产物的研究提供数据支持。
1 材料和方法 1.1 材料
1.1.1 样品采集: 2017年3月,1份石磺海牛样品采集于广东省深圳市大鹏自然保护区海域(114.61°E,22.55°N),保存于天然海水中,当天送回实验室。
1.1.2 培养基: (1) R2A+海盐培养基(g/L):蛋白胨0.50,Casamino acids 0.50,酵母提取物0.50,葡萄糖0.50,可溶性淀粉0.50,磷酸氢二钾0.30,硫酸镁0.05,丙酮酸钠0.30,琼脂粉15,用一次蒸馏水定容到1 L,pH 7.0±0.2。
(2) marine agar 2216培养基(g/L):蛋白胨5,酵母提取物1,柠檬酸铁0.1000,氯化钠19.4500,氯化镁8.8000,硫酸钠3.2400,氯化钙1.8000,氯化钾0.5500,溴化钠0.1600,溴化钾0.0800,氯化锰0.0340,硼酸0.0220,硅化钠0.0040,氟化钠0.0024,硝酸铵0.0016,磷酸氢二钠0.0080,琼脂粉15,用一次蒸馏水定容到1 L,pH 7.6±0.2。
(3) SGTYP培养基(g/L):可溶性淀粉5,葡萄糖5,胰蛋白胨1,蛋白胨1,酵母提取物1,海盐17,用一次蒸馏水定容到1 L,pH 7.5±0.2。
(4) LB培养基(g/L):胰蛋白胨10,酵母提取物5,氯化钠10,用一次蒸馏水定容到1 L,pH 7.2–7.4。
(5) LB+Glu培养基(g/L):胰蛋白胨10,酵母提取物5,氯化钠10,用一次蒸馏水定容到1 L,pH 7.2–7.4,1.0×105 Pa高压灭菌后加入10 mL过滤除菌的50%葡萄糖溶液,用于抗生物膜活性筛选实验。
1.1.3 活性指示菌株: 紫色杆菌CV026 (Chromobacterium violaceum CV026),耐甲氧西林金黄色葡萄球菌ATCC 43300 (Methicillin- resistant Staphylococcus aureus,MRSA),由本实验室保藏。
1.1.4 主要试剂和仪器: QS信号分子C6-HSL购买自Sigma公司,PIPES缓冲液购买自上海麦克林生化试剂有限公司,2×预混Taq酶购买自宝日医生物技术(北京)有限公司,D2000 plus DNA marker购买自Solarbio公司,引物由生工生物工程(上海)股份有限公司合成。PCR仪、凝胶成像系统、电泳仪购自Bio-Rad公司。
1.2 共附生细菌的分离 在超净工作台中先用无菌海水将1份石磺海牛样品冲洗3次,用无菌手术刀将石磺海牛解剖分成体内液、内脏、嘴、鳃和卵共5个部分,将组织用无菌剪刀剪碎后加入液氮充分研磨破碎。取10 mL无菌海水重悬组织,100×g离心10 min,取上清100×g离心10 min,重复2次,收集上清。上清在5000×g离心15 min,去除上清,加入等体积新鲜的无菌海水重悬沉淀。在5000×g离心15 min,去除上清,沉淀用25%的甘油重悬,分作2份,一份用于微生物分离,另一份置于–80 ℃保存。
取100 μL样品涂布于R2A+海盐固体培养基上,等培养基表面水干燥后用parafilm把平板封住,倒置于25 ℃培养。每隔2天观察1次平板,如有肉眼可见的菌落长出且直径大于1 mm,则挑取菌落划线接种到marine agar 2216平板中,同样用parafilm把平板封住,倒置于25 ℃培养,选择形态不一致的菌落划线传代。传代培养至长出明显单菌落时,再次挑取单菌落划线到新的marine agar 2216培养基平板进行二次和三次纯化,直至平板上的菌落形态一致。分离并纯化好的菌用25%的甘油重悬,编号,保存于–80 ℃。
1.3 细菌的16S rRNA基因序列测定和分析 采用菌落PCR的方法鉴定所分离到的菌株。刮取适当菌体用无菌水重悬至OD595 > 0.2,取100 μL菌液到1.5 mL的EP管,置于沸水中加热5 min后,15000 r/min离心2 min,取2 μL上清作为DNA模板,用通用引物27F:5′-59AGAGTTTG ATCMTGGCTCAG-3′;1492R:5′-TACGGYTAC CTTGTTACGACTT-3′扩增16S rRNA基因[35]。PCR扩增体系:25 μL 2×预混Taq酶;2.5 μL 10 μmol/L 27F引物;2.5 μL 10 μmol/L 1492R引物;2 μL DNA模板;18 μL ddH2O。PCR扩增程序:95 ℃ 5 min;95 ℃ 30 s,56 ℃ 30 s,72 ℃ 2 min,共30个循环;72 ℃ 10 min。
取5 μL扩增产物和1 μL 6×loading buffer混合后上样,经1%琼脂糖凝胶在120 V下电泳15 min后,凝胶在凝胶成像系统中成像,将有条带的阳性扩增产物送到广州艾基生物技术有限公司测序,测序引物为通用引物27F和1492R。将测序峰形良好的结果用SeqMan软件拼接,拼接结果去除前后约40 nt峰形不好的序列,将拼接结果上传到EzBioCloud数据库[36]或NCBI库中比对,记录相似度最高的菌株名称和相似度值,将相似度小于等于97.5%的视为疑似新菌。16S rRNA序列上传至NCBI库中公开。以疑似新菌的16S rRNA序列和其相似度最高的菌株的16S rRNA序列用MEGA 7.0构建进化树,构建算法为Neighbor-Joining,计算模型为Kimura’s two parameter,bootstrap值为1000。
1.4 信号分子AHL降解活性筛选 参考文献[37]使用的方法并稍作修改,信号分子C6-HSL能够使紫色杆菌CV026产生紫色色素,颜色越深则信号分子浓度越高。用marine agar 2216固体平板活化分离的菌株,挑取单菌落接种到SGTYP液体培养基中,28 ℃、200 r/min振荡培养3 d,得菌株发酵液。取0.9 mL菌液加到24孔板,每孔再加入0.1 mL 1 mol/L的PIPES缓冲液(pH 6.8)、1 μL 10 mmol/L的C6-HSL信号分子,阴性对照组为0.9 mL的SGTYP液体培养基。24孔板置于28 ℃、200 r/min振荡培养1 d后,取菌液在15000 r/min下离心2 min,上清待测信号分子的残留量。
将AHL信号分子报告菌株紫色杆菌CV026用LB平板划线活化,28 ℃倒置培养1 d后取单菌落接种到LB液体培养基,28 ℃、200 r/min过夜振荡培养,活化好的菌液用LB液体培养基稀释到OD595=0.1–0.2。取90 μL稀释后的CV026菌液和10 μL保留上清到96孔板,将96孔板置于28 ℃、200 r/min摇床振荡培养12 h。菌液在15000 r/min下离心2 min去除上清,往菌体中加入100 μL DMSO萃取紫色色素后再次在15000 r/min下离心2 min,取上清到酶标板中测OD550,与对照相比吸光值越低,则信号分子被降解程度越大。
1.5 抑制MRSA生物膜形成活性筛选 取1.4菌株发酵液上清,用0.22 μm针式滤器过滤除菌备用。用LB+Glu培养基将过夜摇培的MRSA菌液稀释至菌体浓度约5×105 CFU/mL,24孔板每孔加入0.9 mL菌液,再加入0.1 mL各菌株发酵上清,对照组为SGTYP液体培养基。24孔板置于37 ℃静置培养24 h,小心吸去上清,用无菌PBS小心洗涤2次去除浮游细菌。24孔板底部的生物膜在37 ℃培养箱干燥30 min后,每孔加入1 mL 0.1%的结晶紫室温染色10 min,洗去染液,用1 mL 30%的乙酸溶液溶解染料测OD550。以此吸光值对MRSA形成的生物膜的生物量(biomass)进行定量,评估各菌株代谢产物抑制生物膜形成的活性。
1.6 清除MRSA成熟生物膜活性筛选 用LB+Glu培养基将过夜摇培的MRSA菌液稀释至菌体浓度约5×105 CFU/mL,24孔板每孔加入1 mL菌液于37 ℃静置培养24 h。小心吸去上清,用无菌PBS洗2次去除浮游细菌,每孔分别加入0.9 mL LB+Glu培养基和0.1 mL 1.5小节中各菌株发酵上清,对照组为SGTYP培养基。24孔板继续置于37 ℃静置培养24 h,重复1.5小节中洗涤生物膜、固定、染色和定量的步骤,评估各菌株代谢产物清除成熟生物膜的能力。
2 结果和分析 2.1 石磺海牛共附生细菌的分离、鉴定和多样性分析 采集到的石磺海牛样品如图 1-A所示,其形似石磺,外套膜较宽,覆盖住足盘,背部分布有许多凸起,头部有2个小触须,裸鳃呈羽状5–6叶,三分歧式,鳃腔缘也有小突起围绕,与文献报道的石磺海牛样本基本一致[38]。
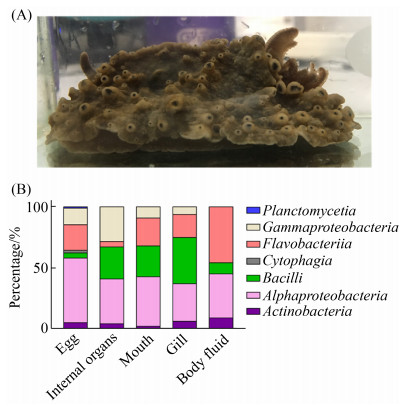 |
| 图 1 石磺海牛各部分可培养的共附生细菌分类统计 Figure 1 The proportion of cultivable bacteria in each part of Homoiodoris japonica. A: The photo of Homoiodoris japonica collected in this research which used to isolate bacteria. B: Classification of bacteria isolated from Homoiodoris japonica at the class level. |
| 图选项 |
从石磺海牛中共分离纯化得到215株细菌,未分离到真菌。通过16S rRNA分子生物学的鉴定,这些菌分别属于87种、54个属、23个科、16个目、7个纲和5个门。这5个门按分离得到的菌株数从多到少依次是变形菌门(Proteobacteria) 126株,拟杆菌门(Bacteroidetes) 44株,厚壁菌门(Firmicutes) 34株,放线菌门(Actinobacteria) 10株和浮霉菌门(Planctomycetes) 1株。
我们将石磺海牛解剖为5个部位分别进行共附生细菌的分离,各部位分离得到的细菌组成见表 1。共附生细菌数量和种类最丰富的部位是卵,共分离得到98株菌、51个种和31个属,涵盖本次分离到的全部7个纲,其中纤维粘网菌纲(Cytophagia)和浮霉菌纲(Planctomycetia)的为卵所特有。共附生细菌丰富程度次之的部位是鳃和内脏,但分离到的细菌数量和种类均不及卵。丰富程度最低的部位是嘴,只分离得到11株,且其中5株的16S rRNA序列比对显示为同一个种。
表 1. 石磺海牛各部位共附生菌组成 Table 1. Classifications of Homoiodoris japonica–associated bacteria in class/genus/species/strain
| Class | Genus/Species/Strains | |||||
| Egg | Internal organs | Gill | Body fluid | Mouth | Total (strains) | |
| Acidimicrobiia | 2/4/5 | 1/1/2 | 1/1/1 | 1/1/1 | 1/1/1 | 10 |
| Alphaproteobacteria | 13/20/51 | 5/7/17 | 10/11/18 | 5/5/5 | 3/3/4 | 95 |
| Bacilli | 1/3/4 | 1/4/12 | 1/3/11 | 1/3/6 | 1/1/1 | 34 |
| Cytophagia | 2/2/2 | – | – | – | – | 2 |
| Flavobacteriia | 8/13/22 | 2/2/2 | 7/8/10 | 3/3/3 | 1/1/5 | 42 |
| Gammaproteobacteria | 4/8/13 | 3/5/13 | 4/4/4 | 1/1/1 | – | 31 |
| Planctomycetia | 1/1/1 | – | – | – | – | 1 |
| Total | 31/51/98 | 12/19/46 | 23/27/44 | 11/13/16 | 6/6/11 | 215 |
| –: no strain isolated. | ||||||
表选项
从纲层面比较各分离部位细菌的组成(图 1-B)。所有分离得到的细菌中,数量最多的是α-变形杆菌纲(Alphaproteobacteria),在各分离部位的菌群组成中均占有很大比例。其次是黄杆菌纲(Flavobacteriia)和芽孢杆菌纲(Bacilli),其中黄杆菌纲在内脏菌群比例中最小,芽孢杆菌纲在卵和嘴的菌群比例中较小。由此看出,石磺海牛各部分的菌群组成有着较大差异。
2.2 共附生细菌中疑似新菌的分析 将各菌株的16S rRNA基因序列在Ezbiocloud数据库搜索比对后,相似度小于等于97.5%的视为疑似新菌。我们发现从石磺海牛中分离得到疑似新菌有16株(表 2),分别归于11个属,占此次分离菌库属总数的20.4%,占菌库株总数的7.4%。其中菌株Z80和相似度最高的模式菌株Olleya algicola 3Alg 18T (KY341922)只有93.38%的相似度,低于不同属的阈值95%,推测Z80为新属新菌。疑似新菌来源最多的部位是嘴和鳃,各分离到6株,卵部位分离到2株,内脏和体内液各分离到1株,疑似新属新菌的Z80分离自鳃。
表 2. 疑似新菌的分离来源及相似度最高的菌株信息 Table 2. The information of the suspected new bacteria and the most similar strain
| Number | Part | Top-Hit strain (Accession) | Similarity/% | Accession No. |
| Z301 | Egg | Primorskyibacter insulae SSK3-2T (KR818861) | 97.10 | MW559462 |
| Z149 | Egg | Alteromonas oceani S35T (MF687202) | 97.50 | MW559441 |
| Z79 | Internal organs | Pseudooceanicola flagellatus DY470T (KF434118) | 97.50 | MW559471 |
| Z80* | Gill | Olleya algicola 3Alg 18T (KY341922) | 93.38 | MW559472 |
| Z114 | Gill | Algibacter aestuarii KYW371T (HQ405792) | 97.00 | MW559436 |
| Z116* | Gill | Phaeobacter porticola P97T (CP016364) | 97.33 | MW559437 |
| Z126* | Gill | Phaeobacter porticola P97T (CP016364) | 97.33 | MW559439 |
| Z223 | Gill | Nitratireductor indicus C115T (KY264918) | 97.30 | MW559454 |
| Z99 | Gill | Pseudooceanicola flagellatus DY470T (KF434118) | 97.50 | MW559476 |
| Z23 | Body fluid | Marinomonas communis LMG 2864T (DQ011528) | 96.60 | MW559455 |
| Z338 | Mouth | Flagellimonas algicola AsT0115T (MK391377) | 96.20 | MW559465 |
| Z359 | Mouth | Flagellimonas algicola AsT0115T (MK391377) | 96.20 | MW559466 |
| Z366 | Mouth | Flagellimonas algicola AsT0115T (MK391377) | 96.20 | MW559467 |
| Z368 | Mouth | Flagellimonas algicola AsT0115T (MK391377) | 96.20 | MW559468 |
| Z378 | Mouth | Flagellimonas algicola AsT0115T (MK391377) | 96.20 | MW559470 |
| Z374 | Mouth | Nitratireductor indicus C115T (KY264918) | 97.30 | MW559469 |
| *: Results of Blast comparison of 16S rRNA in NCBI. | ||||
表选项
对疑似新菌和其最相似模式菌株的16S rRNA序列构建的进化树进行分析显示(图 2),除了Z80外,其他疑似新菌和最相似模式菌株都能很好的归为同一支,因此Z80很可能为一个新属的新菌。整个进化树可以分为3个部分,分别属于Alphaproteobacteria、Gammaproteobacteria和Flavobacteriia三个科,由于前两者均属于变形菌门,而Flavobacteriia属于拟杆菌门,所以前2个科亲缘关系更近,被划分到一个大分支,Flavobacteriia独立一支。
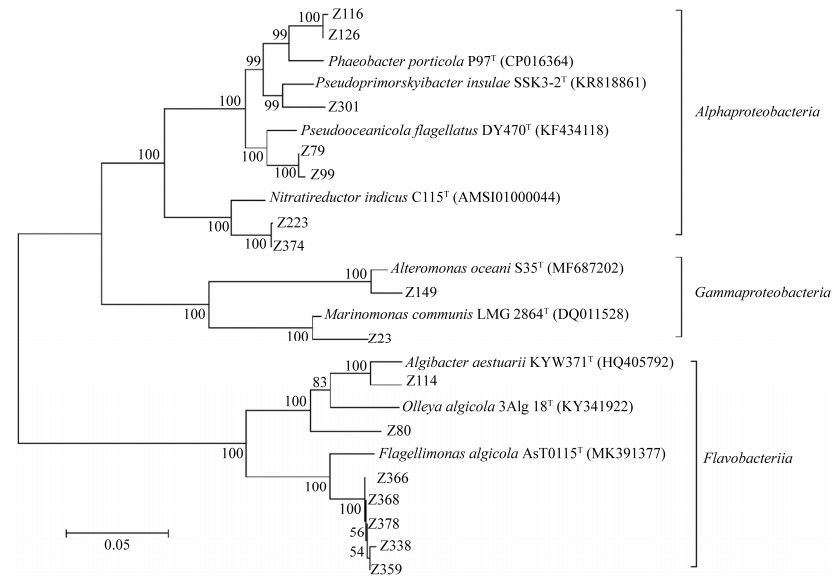 |
| 图 2 疑似新菌和最相似模式菌株16S rRNA序列NJ进化树 Figure 2 Phylogenetic tree based on 16S rRNA gene sequence of the suspected new bacteria and the most similar strain. |
| 图选项 |
2.3 信号分子AHL降解活性筛选 通过筛选得到具有AHL降解活性的菌有28株(表 3),属于20个种,14个属,分别为不动杆菌属(Acinetobacter),交替单胞菌属(Alteromonas),赤杆菌属(Erythrobacter),微杆菌属(Microbacterium),副球菌属(Paracoccus),副红球菌属(Pararhodobacter),假鲁杰氏菌属(Pseudoruegeria),芽孢杆菌属(Bacillus),假海洋菌属(Pseudooceanicola),红球菌属(Rhodococcus),玫瑰单胞菌属(Roseomonas),短状杆菌属(Brachybacterium),Mesoflavibacter和Tenacibaculum(表 3)。其中Mesoflavibacter,Pararhodobacter,Pseudoruegeria,Pseudooceanicola和Brachybacterium五个属目前还未见报道有降解AHL活性的菌株。卵来源的AHL降解活性菌株最丰富,有15株,属于13个种和10个属;其次为鳃部位来源,共计5株、4个种,其中包括疑似新菌Z99。
表 3. 具有AHL降解活性的菌株 Table 3. Strains with AHL degradation activity
| Number | Part | Top-Hit strain | Similarity/% | Accession No. |
| Z205 | Egg | Acinetobacter vivianii NIPH 2168T | 99.85 | MW559450 |
| Z165 | Egg | Alteromonas confluentis DSSK2-12T | 98.50 | MW559444 |
| Z163 | Egg | Bacillus cereus ATCC 14579T | 100.00 | MW559443 |
| Z336 | Egg | Erythrobacter citreus RE35F/1T | 100.00 | MW559464 |
| Z207-2 | Egg | Erythrobacter flavus SW-46T | 99.92 | MW559451 |
| Z238 | Egg | Erythrobacter flavus SW-46T | 99.53 | MW555605 |
| Z185 | Egg | Erythrobacter flavus SW-46T | 99.92 | MW559448 |
| Z166-2 | Egg | Mesoflavibacter sabulilitoris GJMS-9T | 99.62 | MW559445 |
| Z209 | Egg | Microbacterium aquimaris JS54-2T | 100.00 | MW559452 |
| Z151 | Egg | Microbacterium kitamiense Kitami C2T | 98.79 | MW559442 |
| Z203 | Egg | Microbacterium koreense JS53-2T | 99.74 | MW555604 |
| Z322 | Egg | Paracoccus homiensis DD-R11T | 97.93 | MW559463 |
| Z270 | Egg | Pararhodobacter aggregans D1-19T | 100.00 | MW559460 |
| Z210-2 | Egg | Pseudoruegeria aquimaris SW-255T | 98.11 | MW559453 |
| Z178 | Egg | Tenacibaculum litoreum CL-TF13T | 98.04 | MW559447 |
| Z93 | Internal organs | Bacillus anthracis AmesT | 99.85 | MW559474 |
| Z98 | Internal organs | Bacillus anthracis AmesT | 99.85 | MW559475 |
| Z79 | Internal organs | Pseudooceanicola flagellatus DY470T | 97.50 | MW559471 |
| Z221 | Gill | Brachybacterium paraconglomeratum LMG 19861T | 99.45 | MW555606 |
| Z108 | Gill | Mesoflavibacter sabulilitoris GJMS-9T | 99.77 | MW559435 |
| Z122 | Gill | Mesoflavibacter zeaxanthinifaciens DSM 18436T | 100.00 | MW559438 |
| Z85 | Gill | Mesoflavibacter zeaxanthinifaciens DSM 18436T | 99.92 | MW559473 |
| Z99 | Gill | Pseudooceanicola flagellatus DY470T | 97.50 | MW559476 |
| Z20-2 | Body fluid | Bacillus wiedmannii FSL W8-0169T | 100.00 | MW559449 |
| Z230 | Body fluid | Erythrobacter flavus SW-46T | 100.00 | MW559456 |
| Z127 | Body fluid | Rhodococcus qingshengii JCM 15477T | 100.00 | MW559440 |
| Z379 | Mouth | Bacillus wiedmannii FSL W8-0169T | 100.00 | MW555608 |
| Z339 | Mouth | Roseomonas mucosa ATCC BAA-692T | 100.00 | MW555607 |
表选项
2.4 抗MRSA生物膜活性筛选 抗生物膜活性包含了抑制生物膜形成和清除成熟生物膜,通常一些具有抑菌活性的物质也能够抑制生物膜形成,甚至清除成熟的生物膜。我们通过筛选得到8株活性菌株(表 4),抑制生物膜形成活性的菌株有6株,分别为Flavobacterium、Microbacterium、Ruegeria、Tenacibaculum、Bacillus和Marinomonas属的菌;清除MRSA成熟生物膜活性的菌有3株,均来源于Tenacibaculum属。这些活性菌株中,菌株Z171的代谢产物既有抑制生物膜形成的活性,又有清除成熟生物膜的活性;菌株Z259的代谢产物具有较强的抑菌活性,能够抑制MRSA的生长导致无法形成生物膜,因此其抑膜效果是通过抑菌作用实现的。
表 4. 具有抗MRSA生物膜活性的菌株 Table 4. Strains with anti–biofilm activity
| Number | Part | Top–Hit strain | Similarity/% | Accession No. | Inhibition | Eradicate |
| Z259 | Egg | Flavobacterium jejuense EC11T | 99.4 | MW559459 | + | – |
| Z151 | Egg | Microbacterium kitamiense Kitami C2T | 98.8 | MW559442 | + | – |
| Z247 | Egg | Ruegeria scottomollicae LMG 24367T | 99.7 | MW559457 | + | – |
| Z171 | Egg | Tenacibaculum geojense YCS-6T | 99.9 | MW559446 | + | + |
| Z178 | Egg | Tenacibaculum litoreum CL–TF13T | 98.0 | MW559447 | – | + |
| Z25 | Body fluid | Bacillus altitudinis 41KF2bT | 100.0 | MW559458 | + | – |
| Z23 | Body fluid | Marinomonas communis LMG 2864T | 96.6 | MW559455 | + | – |
| Z29 | Body fluid | Tenacibaculum lutimaris TF-26T | 99.9 | MW559461 | – | + |
| +: positive; –: negative. | ||||||
表选项
3 讨论 海洋微生物特别是共附生微生物,是活性天然产物的重要来源之一。研究并获得这些微生物的纯培养,不仅有直接的应用价值,更重要的是能真正去了解这些微生物的生理机能及其生态学意义。截至目前,获得纯培养的微生物仅为冰山一角。基于对16S rRNA的系统进化分析认为,环境中有超过99%的微生物在实验室中“不可培养”[39]。Loceya等利用标度定律(Scaling laws)进行测算认为,地球上有超过1万亿种微生物物种[40]。虽然两种观点均有争议,但都说明绝大多数的微生物属于无法培养或难培养,仍有相当一部分的微生物资源未能挖掘。
由于缺乏对待分离微生物生理代谢的认知,一些传统的培养方法只能保证少数微生物所需的生长条件。一般认为传统的培养方法存在以下问题:培养基富营养化,生长因子缺乏,忽视了生长缓慢的微生物,人为破坏了微生物群落间的关系[41]。提高微生物的可培养性,可以使用稀释后的培养基或寡营养培养基,减少毒性氧物质的危害,维持微生物间的相互作用,添加群体感应信号分子等[42-43]。我们在研究中发现,使用marine agar 2216培养基分离海洋来源微生物时,部分菌落生长速度较快,菌落过大甚至掩盖了其附近生长较慢的菌落,导致许多小的菌落难以分离。而使用寡营养的R2A培养基[44],尽管需要较长培养时间才能观察到菌落形成,但菌落较小,少有菌落覆盖其他菌落的情况。更重要的是,R2A培养基中含有的丙酮酸钠,能缓解毒性氧成分如超氧化物、自由基等对微生物细胞的损伤[45]。许多环境中的微生物处于营养相对贫乏、氧浓度较低、生长代谢缓慢的状态,骤然转移到营养较为丰富的培养基中和空气环境中,可能导致微生物代谢反应不平衡而产生毒性氧物质而导致死亡[46]。因此,R2A培养基能够提高微生物的可培养性,增加分离得到的微生物数量[47]。已有诸多文献报道使用R2A培养基来分离环境中的微生物,获得生物多样性较好的菌株或发现新菌[48-51]。
为了保持共附生微生物的群落状态不变,避免微生物间互利共生关系被打破,从而分离更多可培养细菌。我们将石磺海牛解剖为5个部分,再利用R2A培养基对各部分中可培养细菌进行分离,最终获得215株菌、87个种、7个纲、5个门,其共附生细菌种类丰富,可分离的数量也高于柄海鞘[52]、牡蛎[53]和光裸方格星虫[54]。石磺海牛中种类最丰富的3种菌分别是Bacillus altitudinis (18株)、Sulfitobacter pontiacus (21株)和Pseudovibrio denitrificans (11株),说明这3种菌在石磺海牛中属于优势种。在5个门类中,菌株最多的是变形菌门(126株,占58%以上),而且菌种类最多(50种),说明变形菌门在石磺海牛中占主要优势;浮霉菌门最罕见,仅分离到1株。从石磺海牛各部分共附生微生物的多样性来看,卵来源的细菌数量和多样性最好,其原因还有待深入研究。此外,发现疑似新菌16株,有1株可能为新属新菌;嘴和鳃来源的新菌数量最多,可能是因为石磺海牛进食时接触到的菌比较丰富,所以成为共附生菌也相对丰富。对疑似新菌的鉴定工作已在进行中。
通过初步的活性筛选研究,发现了19株菌能够降解QS信号分子AHL。降解AHL淬灭QS,既能改变竞争菌的生理状态,如运动能力变弱,不能形成生物膜等,降低竞争菌的竞争力,同时还因为宿主没有相对应的信号分子,不会对宿主自身的生理状况产生较大的影响。因此这类具有AHL降解活性的微生物的竞争力可能较强,其生态学意义有待研究。另外还筛选到8株菌具有抗MRSA生物膜活性,目前与这8株菌相似度最高的菌中,暂未见类似活性的报道,值得进一步研究。
综上,本研究利用寡营养和具有缓解毒性氧物质损伤的R2A培养基,对石磺海牛体内液、内脏、嘴、鳃和卵共5个部分的可培养细菌进行了分离、鉴定和活性筛选,首次对石磺海牛共附生细菌的种类和数量进行了研究,并分离到了疑似新菌的菌株,为今后利用石磺海牛共附生微生物及其天然产物资源提供了研究基础和参考。
References
| [1] | Romano S, Jackson SA, Patry S, Dobson ADW. Extending the "one strain many compounds" (OSMAC) principle to marine microorganisms. Marine Drugs, 2018, 16(7): E244. DOI:10.3390/md16070244 |
| [2] | Puglisi MP, Sneed JM, Ritson-Williams R, Young R. Marine chemical ecology in benthic environments. Natural Product Reports, 2019, 36(3): 410-429. DOI:10.1039/C8NP00061A |
| [3] | Molinski TF, Dalisay DS, Lievens SL, Saludes JP. Drug development from marine natural products. Nature Reviews Drug Discovery, 2009, 8(1): 69-85. DOI:10.1038/nrd2487 |
| [4] | Marris E. Marine natural products: drugs from the deep. Nature, 2006, 443(7114): 904-905. DOI:10.1038/443904a |
| [5] | Blunt JW, Copp BR, Keyzers RA, Munro MHG, Prinsep MR. Marine natural products. Natural Product Reports, 2015, 32(2): 116-211. DOI:10.1039/C4NP00144C |
| [6] | Liang X, Luo DM, Luesch H. Advances in exploring the therapeutic potential of marine natural products. Pharmacological Research, 2019, 147: 104373. DOI:10.1016/j.phrs.2019.104373 |
| [7] | Leal MC, Puga J, Ser?dio J, Gomes NCM, Calado R. Trends in the discovery of new marine natural products from invertebrates over the last two decades-where and what are we bioprospecting?. PLoS One, 2012, 7(1): e30580. DOI:10.1371/journal.pone.0030580 |
| [8] | Leal MC, Sheridan C, Osinga R, Dionísio G, Rocha RJM, Silva B, Rosa R, Calado R. Marine microorganism- invertebrate assemblages: perspectives to solve the "supply problem" in the initial steps of drug discovery. Marine Drugs, 2014, 12(7): 3929-3952. DOI:10.3390/md12073929 |
| [9] | Hill RT, Fenical W. Pharmaceuticals from marine natural products: surge or ebb?. Current Opinion in Biotechnology, 2010, 21(6): 777-779. DOI:10.1016/j.copbio.2010.10.007 |
| [10] | Jensen PR, Fenical W. Strategies for the discovery of secondary metabolites from marine bacteria: ecological perspectives. Annual Review of Microbiology, 1994, 48: 559-584. DOI:10.1146/annurev.mi.48.100194.003015 |
| [11] | Piel J. Metabolites from symbiotic bacteria. Natural Product Reports, 2009, 26(3): 338-362. DOI:10.1039/B703499G |
| [12] | Olson JB, Kellogg CA. Microbial ecology of corals, sponges, and algae in mesophotic coral environments. FEMS Microbiology Ecology, 2010, 73(1): 17-30. DOI:10.1111/j.1574-6941.2010.00862.x |
| [13] | Hentschel U, Usher KM, Taylor MW. Marine sponges as microbial fermenters. FEMS Microbiology Ecology, 2006, 55(2): 167-177. DOI:10.1111/j.1574-6941.2005.00046.x |
| [14] | Rosenberg E, Koren O, Reshef L, Efrony R, Zilber-Rosenberg I. The role of microorganisms in coral health, disease and evolution. Nature Reviews Microbiology, 2007, 5(5): 355-362. DOI:10.1038/nrmicro1635 |
| [15] | Lindquist N. Chemical defense of early life stages of benthic marine invertebrates. Journal of Chemical Ecology, 2002, 28(10): 1987-2000. DOI:10.1023/A:1020745810968 |
| [16] | Shnit-Orland M, Kushmaro A. Coral mucus-associated bacteria: a possible first line of defense. FEMS Microbiology Ecology, 2009, 67(3): 371-380. DOI:10.1111/j.1574-6941.2008.00644.x |
| [17] | Knobloch S, Jóhannsson R, Marteinsson V. Co-cultivation of the marine sponge Halichondria panicea and its associated microorganisms. Scientific Reports, 2019, 9: 10403. DOI:10.1038/s41598-019-46904-3 |
| [18] | Knobloch S, Jóhannsson R, Marteinsson V. Bacterial diversity in the marine sponge Halichondria panicea from Icelandic waters and host-specificity of its dominant symbiont "Candidatus Halichondribacter symbioticus". FEMS Microbiology Ecology, 2019, 95(1): fiy220. |
| [19] | Wibowo JT, Kellermann MY, Versluis D, Putra MY, Murniasih T, Mohr KI, Wink J, Engelmann M, Praditya DF, Steinmann E, Schupp PJ. Biotechnological potential of bacteria isolated from the sea cucumber Holothuria leucospilota and Stichopus vastus from Lampung, Indonesia. Marine Drugs, 2019, 17(11): 635. DOI:10.3390/md17110635 |
| [20] | Lopanik NB, Targett NM, Lindquist N. Isolation of two polyketide synthase gene fragments from the uncultured microbial symbiont of the marine bryozoan Bugula neritina. Applied and Environmental Microbiology, 2006, 72(12): 7941-7944. DOI:10.1128/AEM.01277-06 |
| [21] | Sang VT, Dat TTH, Vinh LB, Cuong LCV, Oanh PTT, Ha H, Kim YH, Anh HLT, Yang SY. Coral and coral-associated microorganisms: a prolific source of potential bioactive natural products. Marine Drugs, 2019, 17(8): E468. DOI:10.3390/md17080468 |
| [22] | Yoshizawa S, Tsuruya Y, Fukui YH, Sawabe T, Yokota A, Kogure K, Higgins M, Carson J, Thompson FL. Vibrio jasicida sp. nov., a member of the Harveyi clade, isolated from marine animals (packhorse lobster, abalone and Atlantic salmon).. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(Pt_8): 1864-1870. DOI:10.1099/ijs.0.025916-0 |
| [23] | Romanenko LA, Zhukova NV, Rohde M, Lysenko AM, Mikhailov VV, Stackebrandt E. Glaciecola mesophila sp. nov., a novel marine agar-digesting bacterium. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(3): 647-651. DOI:10.1099/ijs.0.02469-0 |
| [24] | Ivanova EP, Sawabe T, Lysenko AM, Gorshkova NM, Svetashev VI, Nicolau DV, Yumoto N, Taguchi T, Yoshikawa S, Christen R, Mikhailov VV. Pseudoalteromonas ruthenica sp. nov., isolated from marine invertebrates. International Journal of Systematic and Evolutionary Microbiology, 2002, 52(Pt 1): 235-240. |
| [25] | Ivanova EP, Nedashkovskaya OI, Sawabe T, Zhukova NV, Frolova GM, Nicolau DV, Mikhailov VV, Bowman JP. Shewanella affinis sp. nov., isolated from marine invertebrates. International Journal of Systematic and Evolutionary Microbiology, 2004, 54(Pt 4): 1089-1093. |
| [26] | Whiteley M, Diggle SP, Peter Greenberg E. Progress in and promise of bacterial quorum sensing research. Nature, 2017, 551(7680): 313-320. DOI:10.1038/nature24624 |
| [27] | Borges A, Sim?es M. Quorum sensing inhibition by marine bacteria. Marine Drugs, 2019, 17(7): 427. DOI:10.3390/md17070427 |
| [28] | Zhao J, Li XY, Hou XY, Quan CS, Chen M. Widespread existence of quorum sensing inhibitors in marine bacteria: potential drugs to combat pathogens with novel strategies. Marine Drugs, 2019, 17(5): 275. DOI:10.3390/md17050275 |
| [29] | Stewart PS, William Costerton J. Antibiotic resistance of bacteria in biofilms. The Lancet, 2001, 358(9276): 135-138. DOI:10.1016/S0140-6736(01)05321-1 |
| [30] | Defoirdt T. Quorum-sensing systems as targets for antivirulence therapy. Trends in Microbiology, 2018, 26(4): 313-328. DOI:10.1016/j.tim.2017.10.005 |
| [31] | Chen JW, Wang BX, Lu YJ, Guo YQ, Sun JD, Wei B, Zhang HW, Wang H. Quorum sensing inhibitors from marine microorganisms and their synthetic derivatives. Marine Drugs, 2019, 17(2): E80. DOI:10.3390/md17020080 |
| [32] | You JL, Xue XL, Cao LX, Lu X, Wang J, Zhang LX, Zhou SN. Inhibition of Vibrio biofilm formation by a marine actinomycete strain A66. Applied Microbiology and Biotechnology, 2007, 76(5): 1137-1144. DOI:10.1007/s00253-007-1074-x |
| [33] | Bakkiyaraj D, Karutha Pandian ST. In vitro and in vivo antibiofilm activity of a coral associated actinomycete against drug resistant Staphylococcus aureus biofilms. Biofouling, 2010, 26(6): 711-717. DOI:10.1080/08927014.2010.511200 |
| [34] | Khan F, Oloketuyi SF, Kim YM. Diversity of bacteria and bacterial products as antibiofilm and antiquorum sensing drugs against pathogenic bacteria. Current Drug Targets, 2019, 20(11): 1156-1179. DOI:10.2174/1389450120666190423161249 |
| [35] | Hogg JC, Lehane MJ. Identification of bacterial species associated with the sheep scab mite (Psoroptes ovis) by using amplified genes coding for 16S rRNA. Applied and Environmental Microbiology, 1999, 65(9): 4227-4229. DOI:10.1128/AEM.65.9.4227-4229.1999 |
| [36] | Yoon SH, Ha SM, Kwon S, Lim J, Kim Y, Seo H, Chun J. Introducing EzBioCloud: a taxonomically united database of 16S rRNA gene sequences and whole-genome assemblies. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(5): 1613-1617. DOI:10.1099/ijsem.0.001755 |
| [37] | Garge SS, Nerurkar AS. Attenuation of quorum sensing regulated virulence of Pectobacterium carotovorum subsp. carotovorum through an AHL lactonase produced by Lysinibacillus sp. Gs50. PLoS One, 2016, 11(12): e0167344. DOI:10.1371/journal.pone.0167344 |
| [38] | Zhao RY, Cheng JM, Zhao DD. Record on Opisthobranchia from the inter-tidal zone of Dalian. Journal of Northeast Normal University, 1981, 13(1): 43-50. (in Chinese) 赵汝翼, 程济民, 赵大东. 大连潮间带后鳃类软体动物记录. 东北师大学报: 自然科学版, 1981, 13(1): 43-50. |
| [39] | Kaeberlein T, Lewis K, Epstein SS. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science, 2002, 296(5570): 1127-1129. DOI:10.1126/science.1070633 |
| [40] | Locey KJ, Lennon JT. Scaling laws predict global microbial diversity. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(21): 5970-5975. DOI:10.1073/pnas.1521291113 |
| [41] | Zhang ZY, Ding LJ, He S. Research progress on in situ cultivation of uncultured microorganisms. Natural Product Research and Development, 2018, 30(5): 907-913. (in Chinese) 张作艳, 丁立建, 何山. 未培养微生物原位培养技术研究进展. 天然产物研究与开发, 2018, 30(5): 907-913. |
| [42] | Guo B, Wu XL, Qian Y. Approaches for increasing the culturability of microorganisms. Acta Microbiologica Sinica, 2006, 46(3): 508-511. (in Chinese) 郭斌, 吴晓磊, 钱易. 提高微生物可培养性的方法和措施. 微生物学报, 2006, 46(3): 508-511. |
| [43] | Fan NS, Qi R, Yang M. Current technical progresses in the cultivation for uncultured microorganism. Chinese Journal of Applied and Environmental Biology, 2016, 22(3): 524-530. (in Chinese) 范念斯, 齐嵘, 杨敏. 未培养微生物的培养方法进展. 应用与环境生物学报, 2016, 22(3): 524-530. |
| [44] | Reasoner DJ, Geldreich EE. A new medium for the enumeration and subculture of bacteria from potable water. Applied and Environmental Microbiology, 1985, 49(1): 1-7. DOI:10.1128/AEM.49.1.1-7.1985 |
| [45] | Oren A. Pyruvate: a key nutrient in hypersaline environments?. Microorganisms, 2015, 3(3): 407-416. DOI:10.3390/microorganisms3030407 |
| [46] | Bloomfield SF, Stewart GS, Dodd CE, Booth IR, Power EG. The viable but non-culturable phenomenon explained?. Microbiology: Reading, England, 1998, 144(Pt 1): 1-3. |
| [47] | Mizunoe Y, Wai SN, Takade A, Yoshida SI. Restoration of culturability of starvation-stressed and low-temperature- stressed Escherichia coli O157 cells by using H2O2- degrading compounds. Archives of Microbiology, 1999, 172(1): 63-67. DOI:10.1007/s002030050741 |
| [48] | Gao ZH, Ruan SL, Huang YX, Lv YY, Qiu LH. Paraburkholderia phosphatilytica sp. nov., a phosphate-solubilizing bacterium isolated from forest soil. International Journal of Systematic and Evolutionary Microbiology, 2019, 69(1): 196-202. DOI:10.1099/ijsem.0.003129 |
| [49] | Quan XT, Liu QZ, Siddiqi MZ, Im WT. Caballeronia ginsengisoli sp. nov., isolated from ginseng cultivating soil. Archives of Microbiology, 2019, 201(4): 443-449. DOI:10.1007/s00203-018-1577-0 |
| [50] | Sun HJ, Huang HQ, Zhu J, Sun QG, Yu ZH, Bao SX. Isolation and identification of two bacterial strains from marine sponge. Journal of Microbiology, 2010, 30(3): 1-4. (in Chinese) 孙慧洁, 黄惠琴, 朱军, 孙前光, 余中华, 鲍时翔. 2株海绵细菌的分离与鉴定. 微生物学杂志, 2010, 30(3): 1-4. DOI:10.3969/j.issn.1005-7021.2010.03.001 |
| [51] | Lü NN, Shen ZZ, Tao CY, Ou YN, Wang BB, Ruan YZ, Li R, Shen QR. The characteristics of culturable bacterial community in soils and tissue parts of banana. Journal of Nanjing Agricultural University, 2019, 42(6): 1088-1097. (in Chinese) 吕娜娜, 沈宗专, 陶成圆, 欧燕楠, 王蓓蓓, 阮云泽, 李荣, 沈其荣. 蕉园土壤及香蕉植株不同组织可培养细菌的群落特征. 南京农业大学学报, 2019, 42(6): 1088-1097. |
| [52] | Miao TT, Xing X, Du ZJ, Chen GJ. Isolation and phylogenetic analysis of associated bacteria from the styela Clava. Advances in Marine Science, 2012, 30(1): 111-118. (in Chinese) 苗婷婷, 邢翔, 杜宗军, 陈冠军. 柄海鞘共附生细菌的分离培养与系统发育多样性研究. 海洋科学进展, 2012, 30(1): 111-118. DOI:10.3969/j.issn.1671-6647.2012.01.014 |
| [53] | Hou ZM, Chen LC, Wang FW. Diversity of cultivable endophytic microbes isolated from oyster. Food Science and Technology, 2016, 41(7): 20-24. (in Chinese) 侯竹美, 陈廉晨, 王凤舞. 牡蛎共附生可培养微生物多样性的研究. 食品科技, 2016, 41(7): 20-24. |
| [54] | Li XM, Jiang HR, Yin XH, Liu XL, Li SB. Isolation and identification of cultivable microbes to screen bioactive metabolite producing bacteria from Sipunculus nudus in the Beibu Gulf of China. Acta Microbiologica Sinica, 2020, 60(10): 2265-2276. (in Chinese) 李小媚, 江虹锐, 尹秀华, 刘小玲, 李树波. 中国北部湾光裸方格星虫可培养微生物的分离鉴定及其活性代谢物产生菌筛选. 微生物学报, 2020, 60(10): 2265-2276. |
