梁鹏1, 刘博1, 王钰1, 薛莹2, 李备1,2


1. 中国科学院长春光学精密机械与物理研究所, 吉林 长春 130033;
2. 长春长光辰英生物科学仪器有限公司, 吉林 长春 130033
收稿日期:2020-09-16;修回日期:2020-12-03;网络出版日期:2021-02-03
作者简介:李备, 男, 博士, 中国科学院长春光机所研究员, 博士生导师, 生物光子学科带头人。2009年获得英国布里斯托大学博士学位, 曾在卡迪夫大学、牛津大学任研究员, 2017年获得国家高层次人才回国资助, 归国创立长春长光辰英生物科学仪器有限公司, 同时受聘于中国科学院长春光学精密机械与物理研究所应用光学重点实验室。在光学、电子、通信技术方面有着十多年的实践研究经验, 负责项目经费超1500万英镑, 取得了大量世界领先的创新成果, 目前共发表SCI和EI收录的论文30余篇, 专利30余项。带领的长光辰英团队专业方向涵盖光学、机械控制、软件开发、大数据分析、生物等交叉学科领域, 目前致力于开展拉曼单细胞信息技术与单细胞分选技术的研究.
*通信作者:李备。Tel/Fax: +86-431-86708966;E-mail: beili@ciomp.ac.cn.
摘要:自然界相当多的微生物是未培养的,对这些“微生物界暗物质”进行研究,对于研究微生物的进化过程,充分利用微生物资源具有重要意义。对未培养微生物的分离与培养已成为当前国际研究热点,本文对显微操作、荧光活化分选、微流控分选、光镊技术、激光诱导向前转移等可用于微生物分离与培养的技术原理、发展历程进行简单介绍,对比分析了每种分选技术的优点与不足,重点阐述了激光诱导向前转移技术应用于微生物分离的技术优势,并进一步对未来应用于未培养微生物的研究进行了初步展望。
关键词:未培养微生物微生物单细胞分离激光诱导向前转移分离与培养
Application and prospect of single cell sorting technology in isolation and cultivation of microorganisms
Peng Liang1, Bo Liu1, Yu Wang1, Ying Xue2, Bei Li1,2


1. Changchun Institute of Optics, Fine Mechanics and Physics, Chinese Academy of Sciences, Changchun 130033, Jilin Province, China;
2. Hooke Instruments Ltd, Changchun 130033, Jilin Province, China
Received: 16 September 2020; Revised: 3 December 2020; Published online: 3 February 2021
*Corresponding author: Bei Li. Tel/Fax: +86-431-86708966;E-mail: beili@ciomp.ac.cn.
Abstract: Most microorganisms are still unculturable in vitro, therefore studies of the "microbial dark matter" have great significance for investigating the evolution of microorganisms and making the most of microbial resources. Research on the isolation and cultivation of uncultivated microorganisms has become a hot research topic in the world. In this paper, we introduce the principle and development of different single cell sorting techniques, such as micromanipulation, fluorescence activated cell sorting, microfluidic cell sorting, optical tweezer, laser-induced forward transfer, to isolate and culture microorganisms. According to the advantages and disadvantages of each sorting technology, we mainly describe the advantages of laser-induced forward transfer and prospect the application of this technology to the research of uncultivated microorganisms.
Keywords: unculturable microorganismisolation of single microbial celllaser-induced forward transferisolation and cultivation
作为地球上种类最丰富的物种,微生物负责自然界中的化学物质循环,在维持自然界生态平衡中起着至关重要的作用。尽管微生物种类繁多,但能够被培养和分类的微生物只占很小比例,大约为1%[1-3],这极大地阻碍了微生物研究的进展以及人类对微生物资源这个巨大宝库的全面开发和利用。目前普遍认为制约未培养微生物培养的因素主要有:(1)生长速度慢,有些微生物,尤其是生活在营养不充足环境中的微生物自身生长缓慢,难以在培养基上生长为肉眼可见的菌落[4];(2) 培养基组分和培养条件不合适,自然界中大多数微生物生存在寡营养环境中,而实验室培养基的成分通常营养过于丰富[5],而对于那些需要特殊代谢底物才能生长的微生物,我们也知之甚少;(3)微生物间存在相互作用关系,环境中的微生物都是以群体形式生活的,种群间及种群内部的群感效应与实验室的培养环境存在巨大差异[6];(4) 环境中存在“活的非可培养状态” (viable but non-culturable state,VBNC)的微生物,其代谢活性极低,只有在特殊条件下才能将其活化[7]。
针对上述限制未培养微生物培养的因素,科学家们提出了系列方法去研究未培养微生物这一“微生物界暗物质”资源宝库,比如调节培养基法、群体培养法、单细胞分离培养法等。调整培养基法通过调整不同成分和浓度的培养基来探究对未培养微生物的培养条件,其中富集培养法是代表性方法。穆大帅等通过富集培养法成功培养海水沉淀物中的微生物,对培养获得的1251个单克隆进行基因测序,进而对海洋中未培养微生物复苏机制进行研究[8]。群体培养法旨在通过维持微生物群体间的相互作用来实现未培养微生物的培养,例如共培养、原位培养等,Kaeberlein等设计了一种扩散盒来模拟海洋微生物的原位生长环境,该扩散盒核心是一种滤膜,滤膜能够固定住微生物细胞并允许代谢物质、营养物质等小分子通过,将该培养盒置于环境样品的采样点来模拟微生物的原位培养环境,长出菌落后再次分离获得纯培养物[9]。
单细胞分离培养法是指应用专业的单细胞分选设备将单个微生物细胞从群体中分离出来,再对得到的微生物单细胞进行扩大培养。与上述基于“先培养后分离”研究思路的技术不同,单细胞分离培养法提供了一种“先分离后培养”的新策略,理论上可以极大提高分离培养的效率和通量。一方面,在单细胞分离之前,可以先使用荧光标记、拉曼光谱等技术对目标细胞进行识别,更有针对性地将目标细胞从接近原位的环境中分离出来,随后进行扩大培养;另一方面,分离后的微生物单细胞可与共生培养、培养组学等技术结合,实现更高通量的分离培养。更重要的是,对于那些至今无法实现培养的微生物,单细胞分离技术还能提供从基因角度对其进行研究的方法,进而通过代谢等信息促进对未培养微生物培养的研究思路。微生物单细胞测序可以有效地挖掘其种属和功能基因信息,也能够很好地分析不同微生物之间的相互作用关系[6],Swan等通过对海洋样品中的微生物单细胞进行分离测序,获得Deltaproteobacteria cluster SAR324、Gammaproteobacteria clusters ARCTIC96BD-19 和Agg47、Oceanospirillales等菌种的基因信息,并揭示了这些微生物的代谢途径以及其在海洋碳循环中的重要作用[10];Hongoh等利用单细胞分离与基因组技术从白蚁的肠道中发现Candidatus Azobacteroides pseudotrichonymphae等未培养微生物[11]。总之,单细胞分离技术为未培养微生物的研究提供了更多可能性,本文对显微操作、荧光活化分选、微流控分选、光镊技术、激光诱导向前转移等可用于微生物分离与培养的技术进行简要概述,重点阐述激光诱导向前转移技术在微生物研究中的应用进展及创新意义。
1 单细胞分离技术 从复杂样本中分离单个细胞是单细胞研究的普遍需求,对未培养微生物的分离与培养也至关重要,生物成像、生物识别、生物材料等技术的不断进步,促进了一系列单细胞分离技术的产生与发展,这里简要介绍几种常用的单细胞分离技术及其在微生物研究中的应用。
1.1 显微操作技术 1970年起,Johnstone等开始使用微注射器和微毛细管等工具来进行微生物的显微分选[12],但由于显微物镜的放大倍率与工作距离之间的相互制约,几乎不能进行单细胞的分离[13]。随着长工作距离、高放大倍率的显微物镜的出现,显微操作技术有了很大的改进,人们可以通过倒置显微镜对放大1000倍(包括电子放大倍率)的微生物进行观察,结合精准的压力控制装置,可以准确地放置毛细管以实现对微生物细胞的分离[13]。如今也有商业化的显微操作产品,Hohnadel等通过使用商业显微操作仪(Eppendorf Transfer Man 4R),结合倒置显微镜实现了对多种微生物(Escherichia coli、Bacillus cereus、Staphylococcus aureus、Bacteroides vulgatus、Bacillus subtilis、Pseudomonas aeruginosa和Candida albicans)的分离培养,并应用于食品安全领域[14]。Fr?hlich等通过显微操作成功从混合样品中挑取出10个单细胞克隆,培养后经鉴定分别为蜡状芽孢杆菌(Bacillus cereus)、阴沟肠杆菌(Enterobacter cloacae)、大肠埃希菌(Escherichia coli)、金黄色葡萄球菌(Staphylococcus aureus)、硫酸盐还原菌(Desulfovibrio desulfuricans)[13]。Ishoy等通过显微操作对意大利一温泉泥土样品进行单细胞分选,得到金属球菌(Metallosphaem sedula)和硫化叶菌(Sulfolobus solfataricus)[15]。尽管商业化的显微操作分离产品可以通过更换不同孔径的毛细管来实现对不同尺寸微生物的分离,但需要在多次实验中完成。此外显微操作技术只可以分选液体样本,并不适用于固体样本中微生物的分离,且通量较低,难以实现大规模的微生物分离。
1.2 荧光活化细胞分选 荧光活化细胞分选法(fluorescence-activated cell sorting,FACS)是一种发展迅速、广泛使用的细胞分选技术。流式细胞仪(flow cytometer)就是以FACS技术为核心,通过流体控制技术使得细胞悬液成单行流动,检测细胞的荧光信息,并通过压电晶体喷出带有不同电荷的液体来包裹每一个细胞,当包裹着细胞的液体流经电场就会偏转到不同的接收装置中。因其具有测量指标多、检测速度快等优点,被广泛应用于临床医学、细胞学、微生物分离等领域的科学研究中[16]。Zengler等将菌液样本稀释后与融化的琼脂糖混合,形成包裹着微生物单细胞的微滴胶囊,再结合流式细胞仪,将形成的微液滴胶囊分选到含有培养基的96孔板中培养,最终获得纯培养微生物并进行16S rRNA测序,在得到的150株细菌中有71株细菌分类于常见细菌分支中,剩余细菌不存在于环境基因库中,是此前尚未培养的[17]。Scott等通过流式细胞仪实现对厌氧菌(Clostridium thermocellum,Caldicellulosiruptor obsidiansis,Thermotoga maritima,Pyrococcus furiosus)以及生长缓慢微生物的高通量分离培养[18-19]。FACS技术在微生物研究领域发挥了重要作用,但也存在一定局限性。例如,FACS通常要求样品本身具有荧光或对样品进行荧光标记,这需要对目标细胞的表型有预先认知,而未培养微生物通常为完全未知的细胞,难以进行荧光标记;而即便对于那些可以标记荧光的微生物细胞,外源标记也有可能给细胞带来某些影响以至于在实际研究中不能适用。此外,FACS技术需要待分离目标在群落中具有一定浓度,这使其难以对环境中的低丰度微生物进行分离。
1.3 微流控分选 基于微流控芯片的分选研究近些年较为热门。微流控分选(microfluidic isolation)是一种利用微米级的微管道来操控纳升甚至皮升级流体的技术[20],能够精准控制细胞。目前已经有用于单细胞基因组学、转录组学分析的微流控产品[21]。此外很多实验室也开发出多样化微流控芯片设备用于特定的研究,比如中国科学院微生物研究所通过微流控设备与传统培养平皿相结合的方法,实现了微生物细胞的高通量分离与培养,并将其应用于土壤微生物样本,发现了多种能够高效降解多环芳烃的微生物[22]。Hosokawa等通过微流技术在微流控芯片内形成10 pL大小的微液滴,并观察Escherichia coli和Saccharomyces cerevisiae 2种微生物的生长情况,实现了2种微生物的高通量培养[20]。Yin等进一步设计一种Y形微流控芯片,结合拉曼光谱技术对微生物进行识别,通过程序控制芯片通道的压力与拉曼信号同步,将目标微生物输运至指定腔室内[23]。Hmelo等指出微流控芯片在探索微生物微环境中的应用,比如探索不同理化性质下微生物的状态、微生物间的相互作用、微生物抗性、实现单细胞分选以及高通量等相关应用[24]。微流控分选技术的优势在于操作灵活、集成化、速度快等,但由于自然界中的微生物通常形态各异,且含有许多大小不一的微颗粒杂质,在分选时容易发生管道堵塞等问题,限制了此技术在微生物分离培养领域的大规模应用。
1.4 光镊分选 1986年,Ashkin等利用经高数值孔径聚焦的单束激光实现了对电介质微球的三维捕获,标志着光镊技术的诞生[25]。随后科学家对光镊的研究达到空前的重视程度,使其在生物医学、生命科学中得到了广泛应用。与机械镊子相比,光镊是一种以非机械接触的方式来完成对微小物体的夹持、操纵、捕获和固定,近几十年的发展表明,光镊力的大小在0.1皮牛到几百皮牛范围[26],此外通过选用适当波长的激光,可使形成的光镊对物质的热学或化学等效应非常弱,从而几乎不对细胞产生任何损伤。光镊技术所使用的光学力具有靶向性和非侵入性,是能够精确操纵生物样本的基础,从几纳米大小的单分子[27]到几十微米的单个微小器官[28]。光镊技术应用于微生物分选依赖于微流控技术,其优势在于可以结合拉曼光谱等技术,根据微生物的拉曼光谱信息对微生物进行分选。微流控控制微生物样本在芯片中流动,通过光镊捕获之后进行拉曼检测,当检测是目标样品,通过光镊进行分选至收集区通道,而非目标样品则松开后流入废液区(图 1)。Huang等结合光镊和拉曼技术,通过标记所产生的拉曼光谱位移来识别微生物,然后通过光镊移动微生物进行基因测序和微生物培养,在所分选的18个酵母细胞(Saccharomyces cerevisia)和18个荧光菌(Pseudomonas fluorescens)中,分别有11个酵母细胞和7个细胞成功培养[29]。光镊技术在细胞分离中具有低损伤、可视化等优势,但由于设备操作较为复杂,单细胞分离效率较低,在实际应用中受到一定限制。
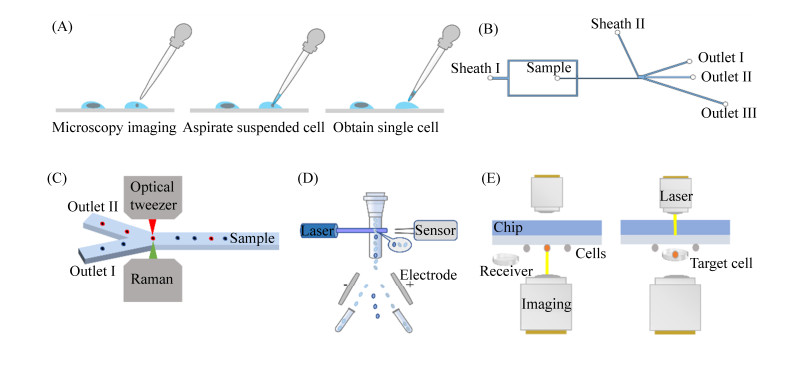 |
| 图 1 几种细胞分选方法 Figure 1 Schematic of several cell isolating methods. A: micromanipulation; B: microfluidic isolating; C: optical tweezers; D: fluorescence-activated cell sorting; E: laser-induced forward transfer. |
| 图选项 |
1.5 基于激光诱导向前转移技术的细胞分选 激光诱导向前转移技术(Laser-Induced Forward Transfer,LIFT)是一种基于光与物质作用的新型可视化微生物细胞分离技术。其通过成像物镜观察锁定目标细胞,通过弹射物镜将激光聚焦到目标细胞进行弹射分选,控制接收装置配合实现目标细胞的接收。与荧光活化细胞分选等分离技术相比,LIFT细胞分选技术由于是镜下分选,有着更高的显微分辨率,可根据目标微生物细胞的形态、尺寸、荧光及拉曼分子指纹等多种特征进行分选,分选后的细胞能够直接用于扩大培养或全基因组扩增及测序等,单细胞获得率高、适用范围广泛。
2 基于激光诱导向前转移技术的单细胞弹射分选与培养方法 2.1 激光诱导向前转移技术原理及发展历程 激光诱导向前转移技术的原理为光与物质的相互作用,当一束光透过透明基底照射到金属或聚合物薄膜上时,部分光会被薄膜吸收转化为内能,薄膜温度升高膨胀变形,甚至液化或者汽化,从而脱离基底产生材料的转移。LIFT技术最早由Bohandy教授在1986年提出,其使用激光照射铜,成功将金属铜转移至硅片基底上[30]。此后,国内外研究者们通过数值分析等仿真手段对不同能量、不同脉宽激光照射不同材料薄膜所产生的热量、温度分布以及材料转移过程机理进行了阐释[31-33],并逐步对LIFT技术进行了优化。LIFT在发明之初,主要被用来转移Cu[33]、AL[34]、Au[35]、Ag以及一些金属化合物[36]等金属材料,应用于微电路的加工制造领域[37-40]。随着LIFT技术的不断改进完善,其可以转移微小物质的特点被逐渐应用到转移一些较脆弱的物质,比如细胞等[41-42]。
2.2 激光诱导向前转移技术在微生物分离培养中的应用 自然界中,比如土壤和海洋沉淀物中存在大量的未培养微生物,许多科学家认为,只要给予微生物合适的矿物质、营养和培养环境,任何微生物都是可培养的,未培养微生物只是人们在实验室中破坏了其原本的生存环境,没有给予其合适培养环境而导致的不可培养。LIFT技术由于其既可以弹射液体,又可以弹射固体的特点,相比于其他微生物分离方法,更能适用于复杂环境中
微生物的分离。此外,由于其采用显微镜下分选策略,可以方便与其他识别技术相结合,比如荧光成像、拉曼光谱等。2013年,Wang等使用LIFT技术精准快速地从复杂的口腔微生物样本中分离出5种细菌;2017年,Huang等结合拉曼光谱与单细胞全基因组测序技术对红海中含有类胡萝卜素的光合细菌,通过拉曼光谱技术识别样本中含有类胡萝卜素的细菌,然后通过LIFT技术将目标细胞弹射并接收,进而进行全基因组测序,来建立单微生物基因型与表型之间的联系,为研究光合菌的类胡萝卜素合成途径提供思路[43],同年还用同样的技术从泰晤士河中分离出抗性微生物并测序分析,建立表型与基因型的对应关系[44];2018年,Jing等从黄海取样,通过LIFT结合拉曼技术成功从中分离出了原位固碳单细胞,并测定其DNA序列以重构其基因组[45];2020年,Yang等通过荧光信息识别铁还原菌,并应用LIFT技术在单细胞水平上将铁还原菌从复杂样品环境中分离,以研究铁还原菌在地球化学循环中的作用[46]。
在应用LIFT技术进行微生物分离培养方面,2002年,Ringeisen等改进LIFT技术,实现了对含有Escherichia coli样本的固态培养基进行弹射, 并且进一步地利用LIFT技术的高精度特点将转移的Escherichia coli排列成想要的图案,同时在对活性进行验证过程中发现LIFT弹射转移过程中并没有对细菌造成明显损伤[47];2014年,Adawiya J. Haider等成功地用LIFT技术实现对液体中Escherichia coli的转移,并对转移接收到的细菌进行培养,验证了LIFT技术可以实现对于液体中微生物的无损弹射转移,此外还验证LIFT转移过程中激光能量、液层厚度、浓度参数对弹射转移细菌的活性影响[48];2015年,为进一步研究未培养微生物的可培养问题,Ringeisen等进一步地直接从土壤中取得环境样本,利用LIFT技术可以转移固体样本的能力将土壤样本进行弹射,并用96孔板接收,结果88个实验组中有45个孔长出微生物群落,且通过计算机控制可以达到每秒弹射20个样本的速度[49];2016年,Taidi等用LIFT技术转移真核生物(Saccharomyces cerevisiae var. bayanus和Chlorella vulgaris)形成一定分布的图案,并通过共聚焦显微镜观察到菌落的生长过程[50]。2019年,Chichkov等也是将LIFT技术应用于土壤微生物的分离[51],同时提出LEMS (laser engineering of microbial system)的概念,并着重对比了LEMS与传统分离提纯相比,在分离速度以及可分离微生物种类上的技术优势[52]。
上述简单介绍了实验室中使用LIFT技术进行微生物分离与培养的有关研究,值得一提的是长春长光辰英生物科学仪器有限公司开发的一款商业化可视化单细胞分选仪,同样是基于LIFT技术弹射分选原理,该仪器主要针对细菌、细胞的可视化无损伤分离与培养,尤其是单细胞层面上的分离与培养。此外,该仪器还可以与荧光显微镜、拉曼光谱仪等耦合,进而深入地研究细菌的代谢信息等,更好地服务于微生物研究,牛津大学团队利用该单细胞分选仪,结合拉曼技术对肠道微生物进行耐药性的有关研究,很好地建立了肠道微生物耐药菌的基因型与表型之间的联系[53]。
2.3 激光诱导向前转移技术用于未培养微生物分离培养有关问题 将LIFT技术用于未培养微生物的分离培养,弹射分选过程是否对微生物活性产生影响、提供合适的培养环境是2个重要因素。欲分析弹射分选过程是否对微生物活性产生影响,整个过程中微生物活性可能受如下几个因素影响:(1) 微生物在弹射分选芯片上的活性;(2) 弹射过程中热、力等因素是否对细胞、细菌有损伤;(3) 接收过程中微生物由高速飞行着陆到接收装置上是否对其有损伤。转移过程中微生物可能受到来自光、热、力等方面的损伤,其中光由于直接作用在金属薄膜上,只有很小部分透过金属薄膜,通过合理控制光的波长与能量,其对细菌活性的影响可以忽略,弹射过程中的热、力则是导致微生物损伤的主要因素,有限远方法计算表明,通过选择合适的中间层材料及厚度,在弹射的瞬间,细菌的温度维持原本温度(室温),同时中间层也能起到缓冲的作用,防止弹射过程中热、力对目标造成损伤。此外,湿度对微生物的活性十分重要,微生物的生长离不开微液体湿润环境,在弹射过程中为微生物样本提供一个微液体,即相对湿润的微环境,以维持微生物基本活性十分重要。在实际使用过程中,我们可以通过添加无生物毒性的一些粘性液体材料处理样本,比如海藻酸钠、甘油等,然后放置在芯片上进行弹射分选。生物学中用于验证细胞、微生物活性的常用方法就是染色法,以酵母细胞为例(图 2),酵母细胞的细胞膜可能是破碎的、渗透的、完好的,对于细胞膜破碎的细胞,我们将无法通过形貌来识别细胞,在染色实验中我们仅考虑后两种情况,对于完好的细胞膜结构,能够阻止染色剂进入细胞膜对细胞核进行染色,因而细胞不会被染色而呈现透明色,对于死亡的或者有损伤的细胞而言,染色剂可以透过细胞膜对细胞核进行染色,因而细胞会呈现深蓝色。为验证微生物在弹射分选过程中是否具有活性,我们可以使用染色剂(亚甲基蓝染色剂)对其进行染色验证,从酵母菌的染色结果可看出通过液体包裹,可以为微生物提供液体环境,维持其生物活性。
 |
| 图 2 不同环境下LIFT分选酵母菌 Figure 2 Isolation of yeast cell by LIFT in different environments. Panels (A-C) show the isolation of yeast cell and dyeing with methylene blue after air drying on a chip. A: before isolating; B: after isolating; C: dyeing result of yeast cell after drying on the chip. Panels (D-F) show the isolation of yeast cell and dyeing with methylene blue in a layer of oil on the chip. D: before isolating; E: after isolating; F: dyeing result of yeast cell in a layer of oil on the chip. |
| 图选项 |
对于弹射分选过程中细胞所受到的损伤,我们难以在微观尺度上直接进行验证,此外,细胞的损伤也分为可修复损伤与不可修复性损伤,为此我们可以通过培养法间接验证微生物的活性在弹射过程中是否受到影响,将培养条件已知的细菌弹射到与之相应的培养基中经过一段时间的培养,可对细菌的活性进行间接验证,对大肠杆菌、枯草杆菌的弹射培养结果(图 3)说明了通过液滴包裹的微生物可以保持活性。
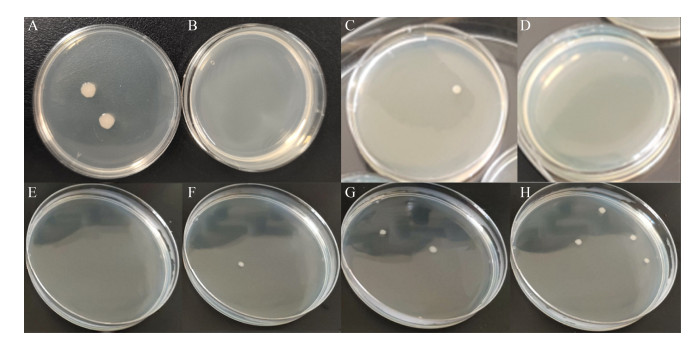 |
| 图 3 不同环境下微生物的LIFT分离培养 Figure 3 Isolation and cultivation of microorganism in different environments by LIFT. Panels (A-B) show the isolation and cultivation of E. coli in a layer of oil, A: colonies grow in experimental group; B: control group. Panels (C-D) show the isolation and cultivation of E. coli in a layer of sodium alginate, C: colony grows in experimental group; D: control group. Panels (E-H) show the isolation and cultivation of Bacillus subtilis in a layer of oil, E: control group; F: isolating 1 single Bacillus subtilis cell and cultivation; G: isolating two single Bacillus subtilis cells and cultivation; H: isolating five single Bacillus subtilis cells and cultivation. |
| 图选项 |
对于未培养微生物而言,适合的培养环境至关重要,LIFT技术的优势在于从复杂环境中分离提取单个微生物细胞,仍不能真正解决未培养微生物的培养难题,但通过LIFT技术将微生物从复杂环境中分离提取,可以结合单细胞全基因组测序等技术,在单细胞层面探索未培养微生物基因信息,推测其代谢通路与培养条件,进而为其他培养方法提供培养思路。
3 总结和展望 未培养微生物作为自然环境中的“暗物质”,对于人类是十分宝贵的自然资源,研究微生物分离培养技术至关重要,而单细胞分选技术为未培养微生物的研究提供了新的思路。目前常见的微生物单细胞分离技术有显微操作、荧光活化分选、微流控分选、光镊分选、基于LIFT的细胞分选等,每种方法各自具有优势及适用范围。显微操作、光镊分选可以大限度地保持细胞的活性,但操作复杂,分离速度慢。荧光活化分选、微流控分选通量高,但微管道对于形态尺寸各异的微生物及杂质颗粒的适应性不足,对于土壤、沉积物等杂质较多的样品中的微生物分离效果不好。而LIFT分选技术由于可实现对液体与固体样品中细胞的转移,能够在更接近原位的状态下完成微生物单细胞分离,且分离不依赖于微管道,可在一次实验中分选不同尺寸、形状的细胞,不受样品中其他颗粒物质的干扰。另外LIFT技术采用镜下可视分选的方式,使其对于低丰度微生物种群的细胞分离也同样适用,极大扩展了其在微生物领域的应用场景。
此外,LIFT细胞分离技术还可以与形态分析、荧光检测、拉曼光谱等细胞识别技术相结合,实现对样品中具有特殊生理功能、代谢活性或特殊形态的微生物单细胞的分离培养,从而更高效地培养出功能性菌株,实现对其功能基因、代谢途径及生态作用的研究,还能加速发酵、制药等工业领域中功能菌的筛选及应用。LIFT细胞分离技术还可以用于微生物互作关系的研究中,即通过在单细胞层面将两个或多个微生物细胞分离至一定环境中进行培养,验证微生物之间是否存在共生、竞争等相互作用,从而进一步探索其调控机制,深入了解微生物生态。未来,LIFT分离技术还可以与培养组学技术相结合,实现未培养微生物的大规模高通量培养研究。
值得注意的是,LIFT技术除了能够在微生物分离培养方面发挥作用之外,还可对分离获得的微生物单细胞进行全基因组扩增,用于单细胞测序研究。这有助于我们从另一个角度去了解那些始终无法培养的微生物种群,通过对其基因信息的分析,推测其代谢通路及培养条件,最终实现未培养微生物的可培养。综上所述,单细胞技术为未培养微生物研究提供了新的策略和可能性,而LIFT细胞分离技术有望在其中发挥重要作用。
References
| [1] | Marcy Y, Ouverney C, Bik EM, L?sekann T, Ivanova N, Martin HG, Szeto E, Platt D, Hugenholtz P, Relman DA, Quake SR. Dissecting biological "dark matter" with single-cell genetic analysis of rare and uncultivated TM7 microbes from the human mouth. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(29): 11889-11894. DOI:10.1073/pnas.0704662104 |
| [2] | Pham VHT, Kim J. Cultivation of unculturable soil bacteria. Trends in Biotechnology, 2012, 30(9): 475-484. DOI:10.1016/j.tibtech.2012.05.007 |
| [3] | Bodor A, Bounedjoum N, Vincze GE, Kis áE, Laczi K, Bende G, Szilágyi á, Kovács T, Perei K, Rákhely G. Challenges of unculturable bacteria: environmental perspectives. Reviews in Environmental Science and Bio/Technology, 2020, 19(1): 1-22. DOI:10.1007/s11157-020-09522-4 |
| [4] | Rappé MS, Connon SA, Vergin KL, Giovannoni SJ. Cultivation of the ubiquitous SAR11 marine bacterioplankton clade. Nature, 2002, 418(6898): 630-633. DOI:10.1038/nature00917 |
| [5] | Simu K, Hagstr?m A. Oligotrophic bacterioplankton with a novel single-cell life strategy. Applied and Environmental Microbiology, 2004, 70(4): 2445-2451. DOI:10.1128/AEM.70.4.2445-2451.2004 |
| [6] | Zhou EM, Li WJ. Uncultivated microorganisms study: methods, opportunities and challenges. Acta Microbiologica Sinica, 2018, 58(4): 706-723. (in Chinese) 周恩民, 李文均. 未培养微生物研究: 方法、机遇与挑战. 微生物学报, 2018, 58(4): 706-723. |
| [7] | Oliver JD. Recent findings on the viable but nonculturable state in pathogenic bacteria. FEMS Microbiology Reviews, 2010, 34(4): 415-425. DOI:10.1111/j.1574-6976.2009.00200.x |
| [8] | Mu DS, Liang QY, Wang XM, Lu DC, Shi MJ, Chen GJ, Du ZJ. Metatranscriptomic and comparative genomic insights into resuscitation mechanisms during enrichment culturing. Microbiome, 2018, 6(1): 230. DOI:10.1186/s40168-018-0613-2 |
| [9] | Kaeberlein T, Lewis K, Epstein SS. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science, 2002, 296(5570): 1127-1129. DOI:10.1126/science.1070633 |
| [10] | Swan BK, Martinez-Garcia M, Preston CM, Sczyrba A, Woyke T, Lamy D, Reinthaler T, Poulton NJ, Masland EDP, Gomez ML, Sieracki ME, DeLong EF, Herndl GJ, Stepanauskas R. Potential for chemolithoautotrophy among ubiquitous bacteria lineages in the dark ocean. Science, 2011, 333(6047): 1296-1300. DOI:10.1126/science.1203690 |
| [11] | Hongoh Y, Sharma VK, Prakash T, Noda S, Toh H, Taylor TD, Kudo T, Sakaki Y, Toyoda A, Hattori M, Ohkuma M. Genome of an endosymbiont coupling N2 fixation to cellulolysis within protist cells in termite gut. Science, 2008, 322(5904): 1108-1109. DOI:10.1126/science.1165578 |
| [12] | Bakoss P. Cloning of leptospires by micromanipulator. Bulletin of the World Health Organization, 1970, 43(4): 599-601. |
| [13] | Fr?hlich J, K?nig H. New techniques for isolation of single prokaryotic cells. FEMS Microbiology Reviews, 2000, 24(5): 567-572. DOI:10.1016/S0168-6445(00)00045-0 |
| [14] | Hohnadel M, Maumy M, Chollet R. Development of a micromanipulation method for single cell isolation of prokaryotes and its application in food safety. PLoS One, 2018, 13(5): e0198208. DOI:10.1371/journal.pone.0198208 |
| [15] | Ish?y T, Kvist T, Westermann P, Ahring BK. An improved method for single cell isolation of prokaryotes from meso-, thermo- and hyperthermophilic environments using micromanipulation. Applied Microbiology and Biotechnology, 2006, 69(5): 510-514. DOI:10.1007/s00253-005-0014-x |
| [16] | Schmid L, Weitz DA, Franke T. Sorting drops and cells with acoustics: acoustic microfluidic fluorescence-activated cell sorter. Lab on a Chip, 2014, 14(19): 3710-3718. DOI:10.1039/C4LC00588K |
| [17] | Zengler K, Walcher M, Clark G, Haller I, Toledo G, Holland T, Mathur EJ, Woodnutt G, Short JM, Keller M. High-throughput cultivation of microorganisms using microcapsules. Methods in Enzymology, 2005, 397: 124-130. |
| [18] | Hamilton-Brehm SD, Vishnivetskaya TA, Allman SL, Mielenz JR, Elkins JG. Anaerobic high-throughput cultivation method for isolation of thermophiles using biomass-derived substrates. Methods in Molecular Biology, 2012, 908: 153-168. |
| [19] | Müller S, Hübschmann T, Kleinsteuber S, Vogt C. High resolution single cell analytics to follow microbial community dynamics in anaerobic ecosystems. Methods, 2012, 57(3): 338-349. DOI:10.1016/j.ymeth.2012.04.001 |
| [20] | Hosokawa M, Hoshino Y, Nishikawa Y, Hirose T, Yoon DH, Mori T, Sekiguchi T, Shoji S, Takeyama H. Droplet-based microfluidics for high-throughput screening of a metagenomic library for isolation of microbial enzymes. Biosensors and Bioelectronics, 2015, 67: 379-385. DOI:10.1016/j.bios.2014.08.059 |
| [21] | Streets AM, Zhang XN, Cao C, Pang YH, Wu XL, Xiong L, Yang L, Fu YS, Zhao L, Tang FC, Huang YY. Microfluidic single-cell whole-transcriptome sequencing. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(19): 7048-7053. DOI:10.1073/pnas.1402030111 |
| [22] | Jiang CY, Dong LB, Zhao JK, Hu XF, Shen CH, Qiao YX, Zhang XY, Wang YP, Ismagilov RF, Liu SJ, Du WB, Parales RE. High-throughput single-cell cultivation on microfluidic streak plates. Applied and Environmental Microbiology, 2016, 82(7): 2210-2218. DOI:10.1128/AEM.03588-15 |
| [23] | McIlvenna D, Huang WE, Davison P, Glidle A, Cooper J, Yin HB. Continuous cell sorting in a flow based on single cell resonance Raman spectra. Lab on a Chip, 2016, 16(8): 1420-1429. DOI:10.1039/C6LC00251J |
| [24] | Wessel AK, Hmelo L, Parsek MR, Whiteley M. Going local: technologies for exploring bacterial microenvironments. Nature Reviews Microbiology, 2013, 11(5): 337-348. DOI:10.1038/nrmicro3010 |
| [25] | Ashkin A, Dziedzic JM, Bjorkholm JE, Chu S. Observation of a single-beam gradient force optical trap for dielectric particles. Optics Letters, 1986, 11(5): 288-290. DOI:10.1364/OL.11.000288 |
| [26] | Neuman KC, Nagy A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods, 2008, 5(6): 491-505. DOI:10.1038/nmeth.1218 |
| [27] | Rai AK, Rai A, Ramaiya AJ, Jha R, Mallik R. Molecular adaptations allow dynein to generate large collective forces inside cells. Cell, 2013, 152(1/2): 172-182. |
| [28] | Favre-Bulle IA, Stilgoe AB, Rubinsztein-Dunlop H, Scott EK. Optical trapping of otoliths drives vestibular behaviours in larval zebrafish. Nature Communications, 2017, 8(1): 630. DOI:10.1038/s41467-017-00713-2 |
| [29] | Huang WE, Ward AD, Whiteley AS. Raman tweezers sorting of single microbial cells. Environmental Microbiology Reports, 2009, 1(1): 44-49. DOI:10.1111/j.1758-2229.2008.00002.x |
| [30] | Bohandy J, Kim BF, Adrian FJ. Metal deposition from a supported metal film using an excimer laser. Journal of Applied Physics, 1986, 60(4): 1538-1539. DOI:10.1063/1.337287 |
| [31] | Hu YX, Cheng H, Xu JX, Yao ZQ. A coupling model to simulate the dynamic process of blister-actuated nanosecond laser-induced forward transfer. Journal of Physics D: Applied Physics, 2017, 50(32): 325305. DOI:10.1088/1361-6463/aa7b08 |
| [32] | Adrian FJ, Bohandy J, Kim BF, Jette AN. A study of the mechanism of metal deposition by the laser-induced forward transfer process. Journal of Vacuum Science & Technology B: Microelectronics and Nanometer Structures, 1987, 5: 1490-1494. |
| [33] | Bohandy J, Kim BF, Adrian FJ, Jette AN. Metal deposition at 532 nm using a laser transfer technique. Journal of Applied Physics, 1988, 63(4): 1158-1162. DOI:10.1063/1.340023 |
| [34] | Schultze V, Wagner M. Laser-induced forward transfer of aluminium. Applied Surface Science, 1991, 52(4): 303-309. DOI:10.1016/0169-4332(91)90072-R |
| [35] | Baseman RJ, Froberg NM, Andreshak JC, Schlesinger Z. Minimum fluence for laser blow-off of thin gold films at 248 and 532 nm. Applied Physics Letters, 1990, 56(15): 1412-1414. DOI:10.1063/1.102484 |
| [36] | Zenou M, Kotler Z. Printing of metallic 3D micro-objects by laser induced forward transfer. Optics Express, 2016, 24(2): 1431-1446. DOI:10.1364/OE.24.001431 |
| [37] | Breckenfeld E, Kim H, Auyeung RCY, Charipar N, Serra P, Piqué A. Laser-induced forward transfer of silver nanopaste for microwave interconnects. Applied Surface Science, 2015, 331: 254-261. DOI:10.1016/j.apsusc.2015.01.079 |
| [38] | Pohl R, Jansink M, R?mer GRBE, in't Veld AJH. Solid-phase laser-induced forward transfer of variable shapes using a liquid-crystal spatial light modulator. Applied Physics A, 2015, 120(2): 427-434. DOI:10.1007/s00339-015-9212-2 |
| [39] | Serra P, Piqué A. Laser-induced forward transfer: fundamentals and applications. Advanced Materials Technologies, 2019, 4(1): 1800099. DOI:10.1002/admt.201800099 |
| [40] | Phipps CR, Zhigilei L, Polynkin P, Gruzdev V, Bohn W, Scharring S, Serra P, Piqué A, Delaporte P, Alloncle AP, Rights M, Kattamis N. Laser-induced forward transfer: fundamentals and applications. Journal of the Optical Society of America B, 2018, 35: 1-33. |
| [41] | Marquez A, Gómez-Fontela M, Lauzurica S, Candorcio-Simón R, Munoz-Martin D, Morales M, Ubago M, Toledo C, Lauzurica P, Molpeceres C. Fluorescence enhanced BA-LIFT for single cell detection and isolation. Biofabrication, 2020, 12(2): 025019. DOI:10.1088/1758-5090/ab6138 |
| [42] | Gruene M, Pflaum M, Hess C, Diamantouros S, Schlie S, Deiwick A, Koch L, Wilhelmi M, Jockenhoevel S, Haverich A, Chichkov B. Laser printing of three-dimensional multicellular arrays for studies of cell - cell and cell - environment interactions. Tissue Engineering Part C: Methods, 2011, 17(10): 973-982. DOI:10.1089/ten.tec.2011.0185 |
| [43] | Song YZ, Kaster AK, Vollmers J, Song YQ, Davison PA, Frentrup M, Preston GM, Thompson IP, Murrell JC, Yin HB, Hunter CN, Huang WE. Single-cell genomics based on Raman sorting reveals novel carotenoid-containing bacteria in the Red Sea. Microbial Biotechnology, 2017, 10(1): 125-137. DOI:10.1111/1751-7915.12420 |
| [44] | Song YZ, Cui L, López JAS, Xu JB, Zhu YG, Thompson IP, Huang WE. Raman-deuterium isotope probing for in-situ identification of antimicrobial resistant bacteria in Thames river. Scientific Reports, 2017, 7(1): 16648. DOI:10.1038/s41598-017-16898-x |
| [45] | Jing XY, Gou H, Gong YH, Su XL, X u, Xu L, Ji YT, Song YZ, Thompson IP, Xu J, Huang WE. Raman-activated cell sorting and metagenomic sequencing revealing carbon-fixing bacteria in the ocean. Environmental Microbiology, 2018, 20(6): 2241-2255. DOI:10.1111/1462-2920.14268 |
| [46] | Gan C, Wu RR, Luo YS, Song JH, Luo DZ, Li B, Yang YG, Xu MY. Visualizing and isolating iron-reducing microorganisms at single cell level. Applied and Environmental Microbiology, 2021, 87: e02192-20. |
| [47] | Ringeisen BR, Chrisey DB, Piqué A, Young HD, Modi R, Bucaro M, Jones-Meehan J, Spargo BJ. Generation of mesoscopic patterns of viable Escherichia coli by ambient laser transfer. Biomaterials, 2002, 23(1): 161-166. DOI:10.1016/S0142-9612(01)00091-6 |
| [48] | Haider AJ, Haider MJ, Majed MD, Mohammed AH, Mansour HL. Effect of laser fluence on a microarray droplets micro-organisms cells by LIFT technique. Energy Procedia, 2017, 119: 256-263. DOI:10.1016/j.egypro.2017.07.078 |
| [49] | Ringeisen BR, Rincon K, Fitzgerald LA, Fulmer PA, Wu PK. Printing soil: a single-step, high-throughput method to isolate micro-organisms and near-neighbour microbial consortia from a complex environmental sample. Methods in Ecology and Evolution, 2015, 6(2): 209-217. DOI:10.1111/2041-210X.12303 |
| [50] | Taidi B, Lebernede G, Koch L, Perre P, Chichkov B. Colony development of laser printed eukaryotic (yeast and microalga) microorganisms in co-culture. International Journal of Bioprinting, 2016, 2(2): 37-43. |
| [51] | Gorlenko MV, Chutko EA, Churbanova ES, Minaev NV, Kachesov KI, Lysak LV, Evlashin SA, Cheptsov VS, Rybaltovskiy AO, Yusupov VI, Zhigarkov VS, Davydova GA, Chichkov BN, Bagratashvili VN. Laser microsampling of soil microbial community. Journal of Biological Engineering, 2018, 12(1): 27. DOI:10.1186/s13036-018-0117-4 |
| [52] | Cheptsov VS, Tsypina SI, Minaev NV, Yusupov VI, Chichkov BN. New microorganism isolation techniques with emphasis on laser printing. International Journal of Bioprinting, 2019, 5(1): 165. |
| [53] | Wang Y, Xu JB, Kong LC, Li B, Li H, Huang WE, Zheng CM. Raman-activated sorting of antibiotic-resistant bacteria in human gut microbiota. Environmental Microbiology, 2020, 22(7): 2613-2624. DOI:10.1111/1462-2920.14962 |
