覃晓云1,2, 李芳3, 王树军3, 刘志媛1,2


1. 海南省热带水生生物技术重点实验室, 海南 海口 570228;
2. 海南大学海洋学院, 海南 海口 570228;
3. 中国热带农业科学院环境与植物保护研究所, 海南 海口 571101
收稿日期:2020-02-24;修回日期:2020-05-18;网络出版日期:2020-05-29
基金项目:雨生红球藻海水驯化研究及陆海结合产业模式试点(ZDYF2019128)
*通信作者:刘志媛, E-mail:liuzhiyuan111@163.com; liuzhiyuan111@hainu.edu.cn.
摘要:[目的] 为建立根癌农杆菌介导的莱茵衣藻快速简便高效的遗传转化体系,本研究以模式生物莱茵衣藻为受体材料,从转化方法和转化子快速鉴定两个方面进行了优化。[方法] 比较了固体培养基共培养转化方法和液体培养基共培养转化方法对根癌农杆菌LBA 4404介导的莱茵衣藻CC425转化效率的影响;研究并比较了(1)首先经过TE裂解再进行PCR(两步法)和(2)不经TE裂解直接进行PCR(一步法)的两种转化子鉴定方法的最佳反应条件和扩增效率。[结果] 农杆菌LBA 4404和莱茵衣藻CC425液体培养基共培养5 d后的转化效果最好,转化率达43.33±1.67个转化子/106个藻细胞。PCR最佳反应条件为:使用高保真DNA聚合酶Taq 1进行扩增;参加PCR反应的细胞密度为5×103-5×106个/mL;TE裂解缓冲液沸水浴20 min(两步直接PCR方法),或者预变性15 min(一步直接PCR方法)。两步法直接PCR的扩增效率优于一步法,但后者反应步骤更简洁。[结论] 本研究建立并优化了农杆菌液体介导莱茵衣藻遗传转化体系,该体系可快速获得遗传转化子,减少转化工作量。
关键词:莱茵衣藻农杆菌介导液体共培养PCR鉴定方法转化子
Optimization of Agrobacterium-mediated transformation of Chlamydomonas reinhardtiii
Xiaoyun Qin1,2, Fang Li3, Shujun Wang3, Zhiyuan Liu1,2


1. Key Laboratory of Tropical Aquatic Biotechnology of Hainan Province, Haikou 570228, Hainan Province, China;
2. Ocean College, Hainan University, Haikou 570228, Hainan Province, China;
3. Institute of Environment and Plant Protection, Chinese Academy of Tropical Agricultural Sciences, Haikou 571101, Hainan Province, China
Received: 24 February 2020; Revised: 18 May 2020; Published online: 29 May 2020
*Corresponding author: Zhiyuan Liu, E-mail:liuzhiyuan111@163.com; liuzhiyuan111@hainu.edu.cn.
Foundation item: Supported by the Study on Seawater Acclimation of Haematococcus pluvialis and Pilot Land-sea Industrial Model (ZDYF2019128)
Abstract: [Objective] To establish a rapid, simple and efficient Agrobacterium-mediated genetic transformation system for Chlamydomonas reinhardtiii, we used the model organism C. reinhardtiii as the receptor material and optimized the Agrobacterium-mediated transformation system of C. reinhardtiii from two aspects: transformation method and transformants identification method. [Methods] We compared the effect of solid co-culture and liquid co-culture on the transformation efficiency of C. reinhardtii CC425 mediated by A. tumefaciens LBA4404. Besides, we analyzed the optimal reaction conditions and amplification efficiency of (1) two-step PCR after TE cleavage, and (2) one-step PCR without TE cleavage. [Results] The highest transformation efficiency was achieved by a 5-day liquid-medium co-culture of Agrobacterium and Chlamydomonas. The transformation rate was 43.33±1.67 transformants/106 algal cells. The optimal reaction conditions were: amplification with high fidelity DNA polymerase Taq 1; the cell density involved in PCR was 5×103–5×106 cells/mL; before amplification, cells were boiled in TE lysis buffer for 20 min (two-step PCR method), or initial denaturation for 15 min (one-step direct PCR method). The amplification efficiency of two-step PCR is better than that of one-step PCR, but the latter is more concise. [Conclusion] Agrobacterium-mediated transformation system of C. reinhardtii was established and optimized, through which rapid genetic transformation can be fulfilled and the workload could be reduced.
Keywords: Chlamydomonas reinhardtiiiAgrobacterium-mediatedliquid co-culturePCR identification methodtransformant
单细胞真核绿藻莱茵衣藻(Chlamydomonas reinhardtii),具有实验操作简单、生长周期短、遗传背景清楚和容易获得各种突变体等优点[1],是众多生物学过程研究的模式物种,已广泛应用于光合作用、遗传与代谢、细胞周期调控、鞭毛功能和组装等生物学过程[2-4]。
莱茵衣藻的遗传转化是获得突变体,进而研究基因功能、遗传代谢等生物学过程的有效手段。目前莱茵衣藻的遗传转化方法主要有玻璃珠法[5]、电击法[6]、基因枪法[7]和农杆菌介导法[8]。
由于农杆菌介导的转化方法操作简单、可转移大片段DNA、插入基因整合拷贝数低、遗传稳定性好、重排少和可优先整合到转录活性区等优点[9],成为当前植物基因工程最常用的方法。然而农杆菌转化效率受颇多因素影响,例如农杆菌株、受体类型、菌与受体共培养的方式、浸染浓度和时间等,因此,转化方法还需根据转化材料特性经过优化,才能提高效率。
快速、简单、高效的转化子PCR鉴定方法是获得高效突变体的关键技术。传统的PCR检测需要细胞培养、收集及基因组DNA提取等步骤。不经过DNA提取和纯化,直接将细胞进行PCR检测的方法可以显著节省时间和成本,提高效率。直接PCR方法已经在细菌和酵母中成熟应用。研究者们在三角褐指藻、亚心型扁藻、球等鞭金藻、盐生杜氏藻、海洋微拟球藻、李氏新月藻中型变种、雨生红球藻、莱茵衣藻、微绿球藻、栅藻和小球藻等单细胞藻类中成功应用直接PCR方法[10-14]。利用玻璃珠研磨[13],或者加入TE、chelex-100、EDTA、Y-PER和2×PCR buffer等裂解液通过加热释放DNA,然后进行PCR扩增鉴定[10-12, 14]。基于现有的方法,针对影响PCR扩增的主要因素,DNA聚合酶、藻细胞浓度和藻细胞破碎时间进行优化,以期提高转化子PCR鉴定方法的效率。
本研究以莱茵衣藻CC425为受体材料,通过比较农杆介导转化的两种转化方法,以及两种不经DNA提取、直接PCR扩增鉴定转化子的方法,优化农杆菌介导转化莱茵衣藻的技术体系,提高了转化效率,为下一步基因功能研究提供技术基础。
1 材料和方法 1.1 材料
1.1.1 藻株、菌株与载体: 莱茵衣藻CC425购自中国科学院水生生物研究所。农杆菌LBA4404由本实验室保存。携带潮霉素抗性基因的植物双元表达载体pCAMBIA 1301由本实验室保存。
1.1.2 培养基: 莱茵衣藻继代培养基:TAP培养基,pH 7.0,固体培养基添加1.5%琼脂粉,高压灭菌后使用。
农杆菌继代培养基:LB培养基,固体培养基添加1.5%琼脂粉,高压灭菌后使用。
诱导培养基:TAP液体培养基高压灭菌后,添加100 μmol/L的乙酰丁香酮(AS,Sigma),pH为7.0。
恢复培养基:TAP培养基高压灭菌后,添加500 μg/mL头孢噻肟钠(Sigma)。
转化子固体筛选培养基:TAP固体培养基高压灭菌后添加15 μg/mL潮霉素(Hyg,Merck)和500 μg/mL头孢噻肟钠。
转化子液体筛选培养基:TAP液体培养基高压灭菌后添加2.5 μg/mL潮霉素和500 μg/mL头孢噻肟钠。
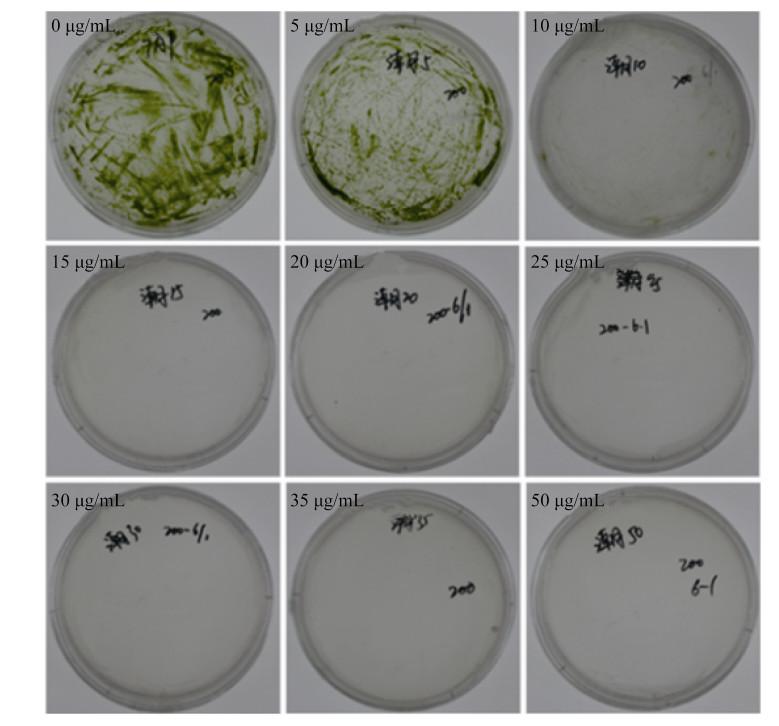
1.2 莱茵衣藻CC425潮霉素抗性浓度筛选 取200 μL指数生长期的莱茵衣藻CC425涂布于分别含有0、5、10、15、20、25、30、35、50 μg/mL Hyg和500 μg/mL头孢噻肟钠的TAP固体培养基上,于25 ℃、55 μmol/(m2·s)、光暗周期为12 h︰12 h (光照︰黑暗)条件下培养。培养5、10、15、30 d后观察藻细胞生长状况。每个处理设置3次重复。
1.3 农杆菌介导转化莱茵衣藻CC425
1.3.1 藻细胞培养: 藻种活化后,接种于TAP液体培养基中,于25 ℃、150 μmol/(m2·s)、光周期为12 h︰12 h (光照︰黑暗)条件下继代培养。
1.3.2 农杆菌侵染用菌液制备: 热击法将表达载体pCAMBIA 1301转入农杆菌LBA 4404菌株中,以潮霉素抗性基因(Hpt)作为目的检测基因,进行PCR鉴定阳性菌株。将含有pCAMBIA 1301载体的农杆菌接种至含有100 μg/mL卡那霉素(Kan,Sigma)的LB液体培养基中,培养至菌液OD600达到0.6左右,5000 r/min离心10 min,收集菌体,重悬到液体诱导培养基中黑暗孵育2 h,准备对莱茵衣藻CC425进行侵染转化。
1.3.3 农杆菌对莱茵衣藻CC425的基因遗传转化体系优化: (1) 固体培养基共培养转化法[8],将200 μL指数生长期(3×106个/mL)莱茵衣藻CC425涂布于固体诱导培养基平板上,3个平行,于25 ℃、55 μmol/(m2·s)、光周期为12 h︰12 h (光照︰黑暗)预培养5 d,再将含载体pCAMBIA 1301的农杆菌LBA 4404悬浮液200 μL均匀涂布浸染,于25 ℃、黑暗共培养2 d。
(2) 液体培养基共培养转化,取10 mL指数生长期(3×106个/mL)的莱茵衣藻CC425和10 mL OD600为0.6左右的农杆菌悬浮液,经4000 r/min离心3 min,去上清,混匀于10 mL液体诱导培养基中,于25 ℃、摇床50 r/min、黑暗共培养3–5 d。
1.3.4 转化子筛选与培养: 将共培养藻细胞,接种到液体的恢复培养基中恢复培养12 h,使农杆菌菌体死亡。然后调节藻细胞浓度为3×106个/mL,取200 μL藻液涂布于固体筛选培养基(含有Hyg和头孢噻肟钠)上培养,直至长出转化子藻落。将转化子接种于液体筛选培养基中活化和培养。培养条件均为55 μmol/(m2·s)、光周期12 h︰12 h (光照︰黑暗)、25 ℃。
1.3.5 转化子PCR鉴定: 收集活化的藻细胞,使用艾德莱基因组DNA提取试剂盒(DN14)提取藻细胞基因组DNA,PCR检测Hpt基因的整合和表达,筛选目的基因插入的转化子藻株。
取1 μL DNA作为模板,加入Taq酶1 U (艾德莱),Hpt基因上下游引物各0.5 μmol/L (表 1),dNTPs 0.2 mmol/L,1×PCR buffer (Mg2+),ddH2O补足至25 μL,用于PCR扩增。PCR扩增程序为:95 ℃ 5 min;95 ℃ 30 s,56 ℃ 30 s,72 ℃ 30 s,35个循环;72 ℃ 8 min。PCR扩增产物用1%的琼脂糖凝胶电泳分析。
表 1. 用于PCR的引物 Table 1. Primers used for PCR
| Primer | Sequence (5′→3′) | Amplification size/bp |
| 18S rRNA | ACCTGGTTGATCCTGCCAGT TCACCTACGGAAACCTTGT | 1770 |
| Hpt | AAAAGTTCGACAGCGTCTC GCGACCTCGTATTGGGAATC | 640 |
表选项
然后利用X-gluc (5-bromo-4-chloro-3-indoyl- b-D-glucuronide,sigma)染色,测定GUS活性,检测PCR筛选的转化藻株Gus基因表达。
1.4 莱茵衣藻CC425转化子PCR检测方法优化
1.4.1 莱茵衣藻CC425 18S rRNA基因快速PCR检测: 以莱茵衣藻18SrRNA基因为目的扩增片段,采取两种方法检测直接PCR方法的扩增效率。
(1) 两步直接PCR法:根据Wan等[11]方法,藻细胞首先在TE中裂解,然后进行PCR反应。取平板生长的藻落或离心收集藻细胞于TE缓冲液中,使藻细胞浓度分别达到每毫升5×103、1×104、5×104、1×105、5×105、1×106、5×106、1×107、5×107、1×108左右,分别进行沸水浴5、10、15、20 min,然后冰浴5 min。取2 μL藻液于PCR管中,加入PCR反应体系至25 μL进行扩增。
(2) 一步直接PCR法:取平板生长的藻落或离心收集藻细胞于PCR管中,使用无酶灭菌水使藻细胞浓度达到每毫升5×103、1×104、5×104、1×105、5×105、1×106、5×106、1×107、5×107、1×108左右,不经过TE裂解和PCR缓冲液裂解细胞步骤,取2 μL藻液于PCR管中,加入PCR反应体系至25 μL,分别进行5、10、15、20 min预变性后,再进行PCR扩增。
PCR反应体系为:Taq酶1 U (Taq 1:艾德莱F8 FastLong Taq;Taq 2:TaKaRa rTaq;Taq 3:艾德莱Taq),18S rRNA上下游引物(表 1)各0.5 μmol/L,dNTPs 0.2 mmol/L,1×PCR buffer (Mg2+),ddH2O补足至25 μL。PCR扩增程序为:95 ℃预变性5–20 min,两步法预变性5 min,95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,35个循环后72 ℃总延伸8 min。PCR扩增产物用1%的琼脂糖凝胶电泳分析。
1.4.2 莱茵衣藻CC425转化子Hpt基因PCR快速检测: 选取1.3.5鉴定的阳性转化子,使用1.4.1优化好的直接PCR方法进行Hpt基因的PCR扩增,以基因组DNA为模板进行Hpt基因的PCR扩增结果作为对照。
1.5 分析方法 采用DPS数据处理系统对数据进行单因素方差分析(One-wayANOVA analysis)。Microsoft Excel 2016进行作图分析。
2 结果和分析 2.1 莱茵衣藻CC425潮霉素抗性浓度筛选 将莱茵衣藻CC425涂布于含不同浓度Hyg和500 μg/mL头孢噻肟钠的TAP固体培养基上。对在不同Hyg浓度的平板上生长10 d的藻落观察发现,在Hyg浓度为5 μg/mL的平板上,藻落可以正常生长,藻细胞密度与对照无显著差异;当Hyg浓度为10 μg/mL时,藻细胞密度显著低于对照;当平板中的Hyg浓度在15 μg/mL以上时,则藻细胞的生长完全受到抑制(图 1)。培养15 d和30 d的结果与10 d的结果一致。因此选择15 μg/mL的Hyg浓度为莱茵衣藻CC425转化子筛选浓度。
 |
| 图 1 莱茵衣藻CC425对潮霉素抗性试验 Figure 1 The resistance of different concentrations of Hyg on C. reinhardtii CC425. |
| 图选项 |
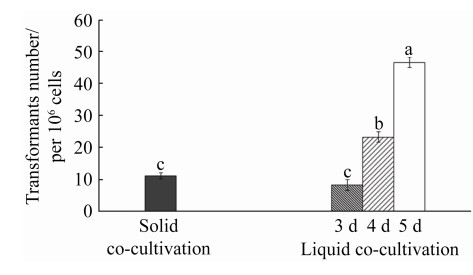
2.2 两种共培养方法对莱茵衣藻CC425转化的影响 分别采用固体培养基共培养转化方法和液体培养基共培养转化方法。前期实验表明,固体共培养的最佳时间为2 d;随着培养时间延长,农杆菌数量增加,会抑制莱茵衣藻CC425生长,影响转化效果。液体共培养则需要3 d才出现转化藻株。因此我们选择固体共培养2 d,液体共培养3、4、5 d,进行转化共培养。将两种方法共培养的藻细胞,恢复培养12 h后分别涂布于固体筛选培养基上弱光培养。
7 d后均得到转化子,采用固体培养基共培养转化方法(图 2)获得11.11±1.67个转化子/106个细胞;采用液体培养基共培养3 d获得的转化子数目(8.33±0.96转化子/106个细胞)和固体共培养获得的无显著性差异(P > 0.05)。而液体共培养4 d (23.33±1.67个转化子/106个细胞)和5 d (46.67±1.67个转化子/106个细胞)获得的转化子数目都显著高于固体共培养获得的转化子数目(P < 0.05),并且液体共培养5 d获得的转化子数目显著高于液体共培养4 d获得的转化子数目(P < 0.05)。
 |
| 图 2 农杆菌不同介导转化方法的效率 Figure 2 Efficiency of different methods of Agrobacterium-mediated transformation of C. reinhardtii CC425. Data are presented as x±s (n=3) and different lower-case letters indicate significant statistical differences in every treatments according to Duncan test (P < 0.05). |
| 图选项 |
对抗性筛选平板上获得的转化子进行Hpt基因的PCR扩增鉴定(图 3),发现固体培养基共培养方法每106细胞可以获得5.00±1.67个阳性转化子,阳性率达44.44%±11.98% (图 4-A–B),固体培养基共培养方法获得的阳性转化子与液体培养基共培养3 d无显著性差异(P > 0.05),但是液体培养基共培养3、4、5 d的阳性率都显著高于固体共培养(P < 0.05)。此外,液体共培养时间不同获得的阳性单克隆不同,共培养5 d (43.33±1.67个/106个细胞)显著性(P < 0.05)高于4 d (21.11±0.96个/106个细胞),并且4 d显著性(P < 0.05)高于3 d (7.22±0.96个/106个细胞),但是不同液体共培养时间的阳性率没有显著性差异(P > 0.05),维持在87.78%–92.85%,阳性率高、稳定且不受共培养时间因素影响。
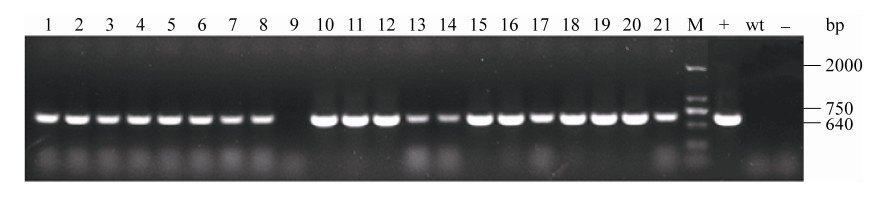 |
| 图 3 莱茵衣藻CC425的Hpt转化子的PCR鉴定 Figure 3 PCR analysis of the transformants contained Hpt in C. reinhardtii CC425. M: marker; lanes 1–21: transformants; +: pCAMBI 1301 plasmid; wt: wild type; –: H2O. |
| 图选项 |
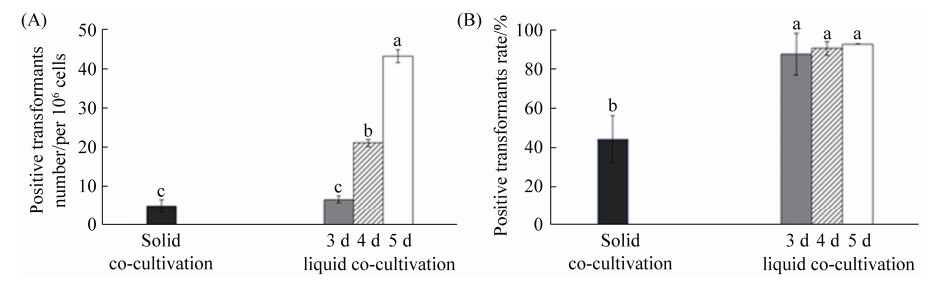 |
| 图 4 莱茵衣藻CC425的Hpt阳性转化子和阳性率 Figure 4 Positive transformants and positive transformants rate of different co-cultivation methods contained Hpt in C. reinhardtii CC425. A: positive transformants of different co-cultivation methods contained Hpt in C. reinhardtii CC425; B: positive transformants rate of different co-cultivation methods contained Hpt in C. reinhardtii CC425. Data are presented as x±s(n=3) and different lower-case letters indicate significant statistical differences in every treatments according to Duncan test (P < 0.05). |
| 图选项 |
为了检测Hpt阳性转化子细胞中Gus基因的表达对PCR鉴定为阳性的转化子进行了Gus表达分析,Gus检测结果显示,藻细胞经X-gluc孵育和乙醇漂白后均呈现出靛蓝色(图 5),表明Gus基因已经整合到莱茵衣藻CC425基因组中。
 |
| 图 5 GUS组织化学染色 Figure 5 The histochemical staining of GUS. |
| 图选项 |
2.3 莱茵衣藻CC425转化子PCR检测方法优化
2.3.1 18S rRNA基因快速PCR检测: 分别采用两步直接PCR和一步直接PCR方法扩增衣藻18S rRNA基因的目的片段,结果表明,DNA聚合酶种类、细胞破碎时间和藻细胞浓度等均影响扩增效率(图 6)。
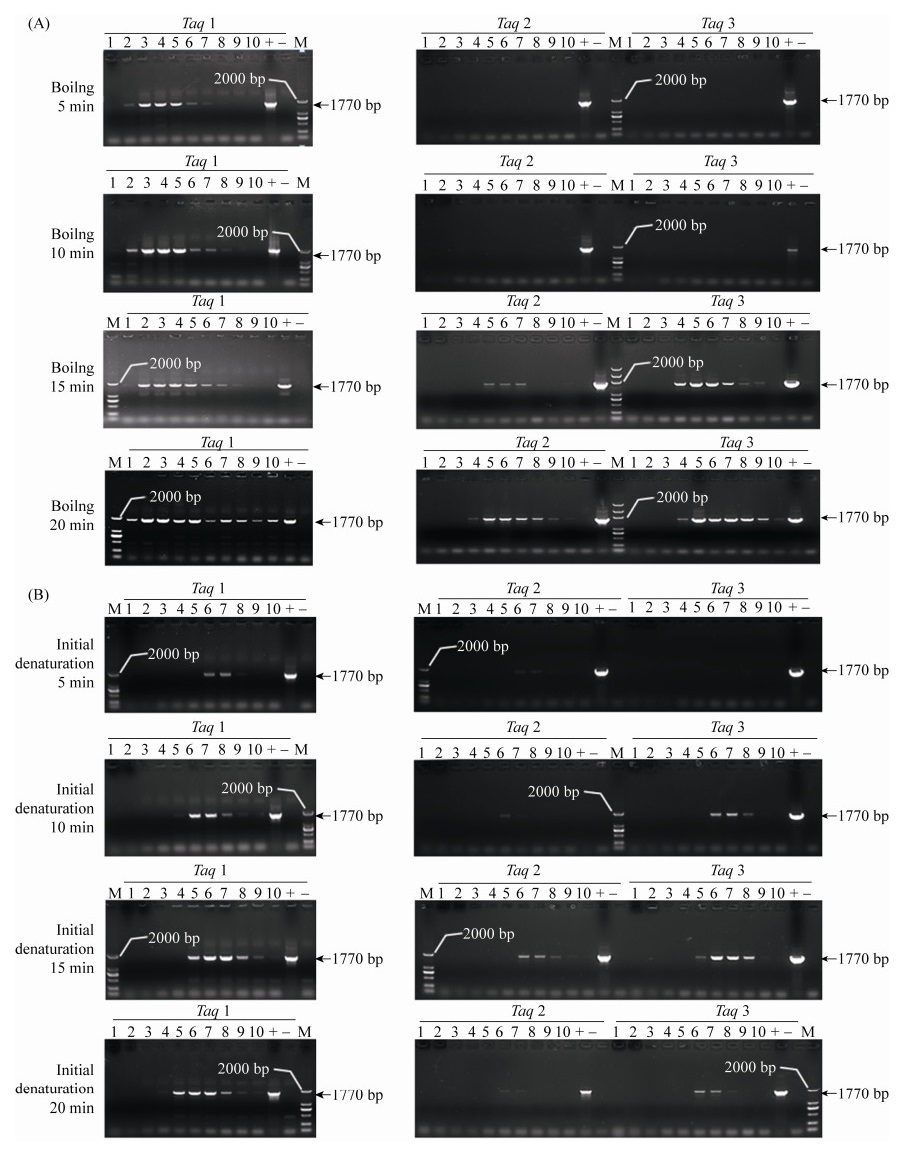 |
| 图 6 两种直接PCR方法的DNA聚合酶种类、细胞浓度和细胞破碎时间的优化 Figure 6 The optimization of DNA polymerase type, cell concentration and cell fragmentation time of two direct PCR methods for C. reinhardtii CC425. A: two-steps PCR method. B: one-step PCR method. Taq 1: high fidelity DNA polymerase (Aidlab); Taq 2 (TaKaRa) and Taq 3 (Aidlab): common DNA polymerase; M: marker; lanes 1–10: the cell concentration of 1×108, 5×107, 1×107, 5×106, 1×106, 5×105, 1×105, 5×104, 1×104, 5×103 cell/mL; +: purified genomic DNA; –: H2O. |
| 图选项 |
先TE裂解再进行PCR扩增的两步PCR法扩增结果表明(图 6-A),TE沸水浴时间为20 min条件下,第一种高保真DNA聚合酶Taq 1的扩增效果最好,所有样品(细胞密度为5×103–1×108个/mL)扩增均获得了清晰条带;而普通DNA聚合酶的有效扩增的细胞密度为5×103–5×106个/mL。
与两步PCR法相比,不经TE裂解的一步PCR法对样品的细胞密度要求较为严格(图 6-B),细胞密度过高不利于扩增。高保真DNA聚合酶的扩增条件为预变性15–20 min,样品细胞密度为5×103–5×106个/mL;其他两种普通DNA聚合酶扩增条件为预变性15 min,样品的最佳细胞密度为5×104–5×105个/mL。
综合上述结果,确定衣藻CC425转化子PCR快速检测的最佳条件为:高保真DNA聚合酶的扩增,细胞密度为5×103–5×106个/mL,TE沸水浴20 min (两步法),或95 ℃预变性15 min (一步法)。
2.3.2 莱茵衣藻CC425转化子Hpt基因的PCR快速检测: 随机选取9个阳性转化子,分别采用两步直接PCR法和一步直接法PCR对Hpt基因进行PCR扩增,两步法采用沸水浴20 min,一步法采用95 ℃预变性15 min,所用细胞密度均为106个/mL,所用聚合酶为Taq 1。以纯化的基因组DNA为模板进行PCR扩增作为参照。结果表明(图 7),3种扩增方法均可获得清晰的Hpt基因目的条带,表明这两种快速的直接PCR方法均可应用于莱茵衣藻CC425转化子的快速检测。
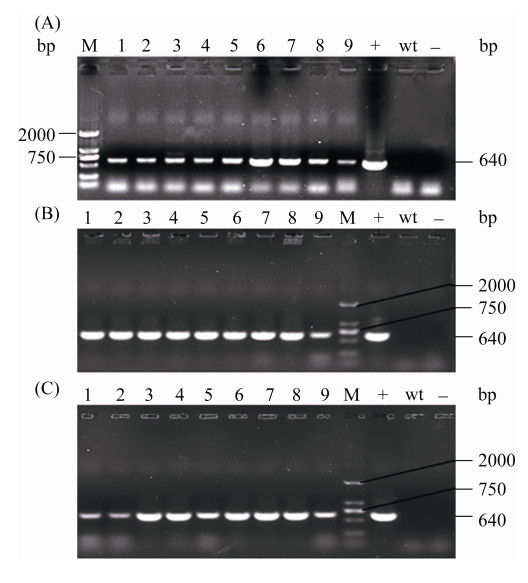 |
| 图 7 不同PCR方法鉴定莱茵衣藻CC425的Hpt转化子 Figure 7 Different PCR methods identified of the transformants contained Hpt in C. reinhardtii CC425. A: purified genomic DNA amplified the Hpt gene; B: two-steps method amplified the Hpt gene; C: one-steps method amplified the Hpt gene. M: marker; lanes 1–9: transformants; +: pCAMBI 1301 plasmid; wt: wild type; –: H2O. |
| 图选项 |
3 讨论 建立高效成熟的转化体系是研究基因功能的重要前提。目前已报道的农杆菌介导转化莱茵衣藻共培养的方法主要采用固体共培养方法[8-9, 15]。Kumar等[8]最先使用农杆菌LBA 4404在固体TAP培养基中转化莱茵衣藻CC-124 (mt–),并在筛选培养基中获得的转化子数目约是玻璃珠法的50倍。而Pratheesh等[9]改进了Kumar的方法,将活化的农杆菌EHA-105在液体TAP培养基中预侵染莱茵衣藻细胞CC-125 (mt+) 30 min,转至固体培养基共培养2 d,转化效率进一步提高。在本实验中,参考Kumar等[8]方法,进行农杆菌LBA 4404介导转化莱茵衣藻CC425藻株,只有6–7个转化子/106个细胞,并且假阳性率比较高。而Mini等[15]参考该方法无法获得耐巴龙霉素的藻落,认为农杆菌菌株、莱茵衣藻藻株和诱导培养基影响转化效率,采用改良的方法,将两株根癌农杆菌C58C1和LBA4404分别与两株莱茵衣藻cw15和CC125在固体AB诱导培养基上共培养2 d,结果LBA4404和C58C1农杆菌菌株都成功转化cw15细胞(分别得到16–31个转化子/108个细胞和25–33个转化子/108个藻细胞),而CC125细胞只有C58C1转化成功(分别得到6–14个转化子/108个细胞),证明使用细胞壁缺陷莱茵衣藻细胞和高毒性农杆菌获得更高的转化效率。为提高转化效率,本研究对其诱导培养的方法进行了改进,采用了液体共培养的方法进行了诱导转化。以期找到一种简便高效的农杆菌介导莱茵衣藻CC425遗传转化体系。
液体共培养法的关键是控制农杆菌的生长和共培养的时间,Ghosh等[16]采用该方法介导香蕉胚性细胞,由于农杆菌污染严重,没能获得转基因植株,而Huang等[17]同样使用液体共培养法介导芭蕉胚性细胞,并稍作改动,在共培养过程中,通过控制摇床转速和更换培养基来控制农杆菌浓度,防止农杆菌过度生长并获得转基因植株。在农杆菌介导的莱茵衣藻转化的报道中,使用了2 d的共培养时间转化藻株[8-9, 15]。然而,在其他植物的结果表明,3–4 d的共培养对植株总体上更有利[18]。长时间共培养的主要缺点是农杆菌过度生长,导致植株坏死和死亡[19],本实验使用固体培养基共培养3 d以上,农杆菌过度生长,完全覆盖在固体抗性平板上,导致莱茵衣藻转化子无法生长而死亡。San等[20]采用2 d液体共培养方法,没有获得转基因植株,这跟本研究的实验结果是一致的,通过延长共培养时间到3 d,可以成功得到转化子,并且共培养5 d的转化率最高,达43.33±1.67个转化子/106个藻细胞。液体共培养的转化率受共培养时间影响,虽然共培养6 d,获得的转化子就比5 d的多(数据未显示),但是抗性培养皿中不能完全抑制农杆菌生长。此外液体共培养的阳性转化率不受共培养时间影响,平均阳性率达87.78%–92.85%。
液体共培养法转化率较高的原因可能是在液体共培养时农杆菌与莱茵衣藻CC425细胞接触更加充分,整个过程中减少莱茵衣藻涂布于固体诱导培养基预培养,农杆菌涂布,藻菌混合细胞洗脱的步骤。相对于固体共培养方法,液体共培养法在操作上更加简便,减少了污染的可能性。农杆菌介导莱茵衣藻转化体系的操作中步骤繁琐,并且农杆菌介导莱茵衣藻转化并没有像在植物转化中减轻转基因重排的表现,所以农杆菌介导的转化在更高或更稳定的表达方面与电击法相比没有表现出明显的优势,但是农杆菌介导转化的单基因位点插入的比例较高,可用于插入突变的实验中[15]。因此优化农杆菌液体共培养介导的转化方法可能对莱茵衣藻的插入突变提供技术基础。
直接PCR方法与传统PCR的比较,传统的PCR检测转化子需要在PCR步骤之前进行多个准备工作,如藻细胞液体培养基中传代培养、细胞收集和裂解、基因组DNA分离和纯化步骤,它是耗时和耗力的步骤,特别是在筛选大量的转基因转化子。直接PCR旨在简化过程,本研究发现,直接PCR方法由3种因素决定,即裂解细胞时间、DNA聚合酶的种类和藻细胞浓度。对于藻细胞裂解,大多数实验方法倾向于采用沸水浴[10-12],本实验的两步PCR结果表明,用该方法进行PCR扩增效果良好。而本研究改良的采用延长预变性时间裂解藻细胞的一步PCR方法,也可以稳定进行扩增。DNA扩增酶影响直接PCR的扩增效率,两种直接PCR结果显示,高保真DNA聚合酶更适用于直接PCR方法体系。藻细胞浓度可能影响直接PCR的效果,两步PCR方法在最佳条件下能稳定扩增5×103–5×108个/mL的藻细胞浓度,而一步PCR方法在最佳条件下能稳定扩增5×103–5×106个/mL的藻细胞浓度。两种直接PCR方法相比较,两步PCR方法的扩增效率更高,更稳定,适用于广范围藻细胞浓度和多次多基因的扩增中。而一步法PCR,不需要从TE裂解液中再次移取藻细胞进行PCR这一步骤,如果进行批量操作,会更省时。
莱茵衣藻的遗传转化和鉴定筛选方法是一个耗时长且工作量大的过程,建立一个快速、简便、高效的转化方法是非常重要的。我们通过莱茵衣藻和农杆菌在液体中完成共培养步骤,减少中间藻预培养、农杆菌涂布和藻菌混合细胞洗脱这些工作量和污染的可能性,并且该方法得到的转化子准确率高,通过延长共培养时间还可以得到更多的转化子。此外通过藻细胞直接PCR的方法减少筛选转化子的工作量,大大缩短筛到转化子的时间。
References
| [1] | Blaby IK, Blaby-Haas CE, Tourasse N, Hom EFY, Lopez D, Aksoy M, Grossman A, Umen J, Dutcher S, Porter M, King S, Witman GB, Stanke M, Harris EH, Goodstein D, Grimwood J, Schmutz J, Vallon O, Merchant SS, Prochnik S. The Chlamydomonas genome project:a decade on. Trends in Plant Science, 2014, 19(10): 672-680. DOI:10.1016/j.tplants.2014.05.008 |
| [2] | Merchant SS, Kropat J, Liu BS, Shaw J, Warakanont J. TAG, You're it! Chlamydomonas as a reference organism for understanding algal triacylglycerol accumulation. Current Opinion in Biotechnology, 2012, 23(3): 352-363. DOI:10.1016/j.copbio.2011.12.001 |
| [3] | Grossman AR, Catalanotti C, Yang WQ, Dubini A, Magneschi L, Subramanian V, Posewitz MC, Seibert M. Multiple facets of anoxic metabolism and hydrogen production in the unicellular green alga Chlamydomonas reinhardtii. New Phytologist, 2011, 190(2): 279-288. DOI:10.1111/j.1469-8137.2010.03534.x |
| [4] | Yang WQ, Catalanotti C, D'Adamo S, Wittkopp TM, Ingram-Smith CJ, Mackinder L, Miller TE, Heuberger AL, Peers G, Smith KS, Jonikas MC, Grossman AR, Posewitz MC. Alternative acetate production pathways in Chlamydomonas reinhardtii during dark anoxia and the dominant role of chloroplasts in fermentative acetate production. The Plant Cell, 2014, 26(11): 4499-4518. DOI:10.1105/tpc.114.129965 |
| [5] | Kindle KL. High-frequency nuclear transformation of Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(3): 1228-1232. DOI:10.1073/pnas.87.3.1228 |
| [6] | Brown LE, Sprecher SL, Keller LR. Introduction of exogenous DNA into Chlamydomonas reinhardtii by electroporation. Molecular and Cellular Biology, 1991, 11(4): 2328-2332. DOI:10.1128/MCB.11.4.2328 |
| [7] | Boynton JE, Gillham NW, Harris EH, Hosler JP, Johnson AM, Jones AR, Randolph-Anderson BL, Robertson D, Klein TM, Shark KB, Sanford JC. Chloroplast transformation in Chlamydomonas with high velocity microprojectiles. Science, 1988, 240(4858): 1534-1538. DOI:10.1126/science.2897716 |
| [8] | Kumar SV, Misquitta RW, Reddy VS, Rao BJ, Rajam MV. Genetic transformation of the green alga-Chlamydomonas reinhardtii by Agrobacterium tumefaciens. Plant Science, 2004, 166(3): 731-738. |
| [9] | Pratheesh PT, Vineetha M, Kurup GM. An efficient protocol for the Agrobacterium-mediated genetic transformation of microalga Chlamydomonas reinhardtii. Molecular Biotechnology, 2014, 56(6): 507-515. DOI:10.1007/s12033-013-9720-2 |
| [10] | Cao MQ, Fu Y, Guo Y, Pan JM. Chlamydomonas (chlorophyceae) colony PCR. Protoplasma, 2009, 235(1/4): 107-110. |
| [11] | Wan MX, Rosenberg JN, Faruq J, Betenbaugh MJ, Xia JL. An improved colony PCR procedure for genetic screening of Chlorella and related microalgae. Biotechnology Letters, 2011, 33(8): 1615-1619. DOI:10.1007/s10529-011-0596-6 |
| [12] | Packeiser H, Lim C, Balagurunathan B, Wu JC, Zhao H. An extremely simple and effective colony PCR procedure for bacteria, yeasts, and microalgae. Applied Biochemistry and Biotechnology, 2013, 169(2): 695-700. DOI:10.1007/s12010-012-0043-8 |
| [13] | Radha S, Fathima AA, Iyappan S, Ramya M. Direct colony PCR for rapid identification of varied microalgae from freshwater environment. Journal of Applied Phycology, 2013, 25(2): 609-613. DOI:10.1007/s10811-012-9895-0 |
| [14] | Liu J, Gerken H, Li YT. Single-tube colony PCR for DNA amplification and transformant screening of oleaginous microalgae. Journal of Applied Phycology, 2014, 26(4): 1719-1726. DOI:10.1007/s10811-013-0220-3 |
| [15] | Mini P, Demurtas OC, Valentini S, Pallara P, Aprea G, Ferrante P, Giuliano G. Agrobacterium-mediated and electroporation-mediated transformation of Chlamydomonas reinhardtii:a comparative study. BMC Biotechnology, 2018, 18: 11. DOI:10.1186/s12896-018-0416-3 |
| [16] | Ghosh A, Ganapathi TR, Nath P, Bapat VA. Establishment of embryogenic cell suspension cultures and Agrobacterium-mediated transformation in an important Cavendish banana cv. Robusta (AAA). Plant Cell, Tissue and Organ Culture (PCTOC), 2009, 97(2): 131-139. DOI:10.1007/s11240-009-9507-0 |
| [17] | Huang X, Huang XL, Xiao W, Zhao JT, Dai XM, Chen YF, Li XJ. Highly efficient Agrobacterium-mediated transformation of embryogenic cell suspensions of Musa acuminata cv. Mas (AA) via a liquid co-cultivation system. Plant Cell Reports, 2007, 26(10): 1755-1762. DOI:10.1007/s00299-007-0376-x |
| [18] | Lopez SJ, Kumar RR, Pius PK, Muraleedharan N. Agrobacterium tumefaciens-mediated genetic transformation in tea (Camellia sinensis[L.] O. Kuntze). Plant Molecular Biology Reporter, 2004, 22(2): 201-202. DOI:10.1007/BF02772730 |
| [19] | Hu Z, Wu YR, Li W, Gao HH. Factors affecting Agrobacterium tumefaciens-mediated genetic transformation of Lycium barbarum L. In Vitro Cellular & Developmental Biology-Plant, 2006, 42(5): 461-466. |
| [20] | Cha TS, Yee W, Aziz A. Assessment of factors affecting Agrobacterium-mediated genetic transformation of the unicellular green alga, Chlorella vulgaris. World Journal of Microbiology and Biotechnology, 2012, 28(4): 1771-1779. DOI:10.1007/s11274-011-0991-0 |
