张琦, 梁雅静, 张宇馨, 陈玲慧, 韩钟娆, 李贝贝, 金一, 何晓青


北京林业大学生物科学与技术学院, 北京 100083
收稿日期:2020-03-15;修回日期:2020-05-12;网络出版日期:2020-07-09
基金项目:国家自然科学基金(31971398,31700633);中国科学院科技服务网络计划(KFJ-STS-ZDTP-036);北京林业大学大学生创新计划(X201910022081)
*通信作者:何晓青. Tel:+86-10-62338830; E-mail:lenahe@bjfu.edu.cn.
摘要:[目的] 为了实现对大肠杆菌靶基因的点突变,本研究将同源重组系统与CRISPR-Cas9技术相结合,探索一种高效、简捷的两步法策略。[方法] 将靶基因的上下游同源臂和标记基因(amp)与pKOV质粒连接,获得pKOV-HR重组质粒。将pKOV-HR转化至大肠杆菌,借助其自身RecA重组系统,介导DNA发生同源重组,获得靶基因敲除菌株。随后,靶基因的点突变采用pSGKP-km和pCasKP-apr双质粒系统。首先,设计与amp结合具有定向引导作用的spacer序列,并利用overlap PCR获得带有靶基因点突变的同源臂,将spacer和同源臂与pSGKP-km质粒连接,获得重组质粒pSGKP-km-spacer-HR;接着,将pSGKP-km-spacer-HR和pCasKP-apr质粒转化至上述敲除菌株;最后,利用质粒表达的Cas9切割蛋白和λ-Red重组蛋白,发生DNA同源重组,获得靶基因点突变菌株。[结果] 利用上述方法,既成功获得了大肠杆菌yjjW敲除菌株D7ΔyjjW::ampR,又实现了yjjW第24位碱基T到C的点突变,获得点突变菌株D7yjjW-24。[结论] 本研究建立了一种高效、简捷的大肠杆菌靶基因点突变方法,amp基因的插入提供了有效的筛选标记,此外该方法建立的重组质粒pSGKP-km-spacer,可应用于同种菌株其他靶基因的点突变,能够缩短后续实验操作,为基因编辑技术的发展提供了科学理论以及实验操作基础。
关键词:CRISPR-Cas9大肠杆菌同源重组点突变
Two-step strategy for point mutation of yjjW in Escherichia coli by gene editing
Qi Zhang, Yajing Liang, Yuxin Zhang, Linghui Chen, Zhongrao Han, Beibei Li, Yi Jin, Xiaoqing He


College of Biological Science and Technology, Beijing Forestry University, Beijing 100083, China
Received: 15 March 2020; Revised: 12 May 2020; Published online: 9 July 2020
*Corresponding author: He Xiaoqing. Tel:+86-10-62338830; E-mail:lenahe@bjfu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31971398, 31700633), by the Science and Technology Service Network Initiative (KFJ-STS-ZDTP-036) and by the Innovation Program for Under-graduate Students in Beijing Forestry University (X201910022081)
Abstract: [Objective] In order to investigate the target genes in Escherichia coli, we used the CRISPR-Cas9 tool combining with homologous recombination to develop a two-step strategy for point mutation. [Methods] First, we assembled the up- and down-stream homologous arms of a gene and the amp gene with the pKOV plasmid to construct the pKOV-HR plasmid. Then the pKOV-HR plasmid was transformed into E. coli and the gene knock-out mutants were obtained by the RecA recombination system of E. coli itself. Subsequently, we applied two plasmids system (pSGKP-km and pCasKP-apr) to handle the point mutations of the target gene. Firstly, we designed a spacer sequence of the amp gene which was able to guide Cas9 proteins. The overlap PCR was used to produce the homologous arm containing point mutations of the target gene. Then the spacer and homologous arm were connected by pSGKP-km plasmid and the plasmid pSGKP-km-spacer-HR was obtained. Next, we transformed this plasmid and pCasKP-apr plasmid into the above-mentioned knock-out mutants. Finally, we got the point mutants by the DNA-cleaving of Cas9 proteins and λ-Red recombinant proteins which were produced by the two plasmids. [Results] By this two-step strategy, we obtained yjjW knockout strains named D7ΔyjjW::ampR, and point mutants named D7yjjW-24 (from T to C the 24th base) successfully. [Conclusion] The study established an efficient method for point mutations of target genes in E. coli. The insertion of amp gene provided an effective screening marker. The recombinant plasmid pSGKP-km-spacer established in this process can be applied to point mutations of other target genes, which will greatly shorten experimental processes. It contributes the scientific theory and practice for the development of gene editing technology.
Keywords: CRISPR-Cas9Escherichia colihomologous recombinationpoint mutation
大肠杆菌(Escherichia coli)作为一种常见的模式菌株,其繁殖迅速,遗传构造研究详尽,已被广泛应用于生物工程领域,用于表达异源蛋白和某些大分子产物等[1],建立和开发基因编辑方法有助于对大肠杆菌的研究和利用[2]。目前对大肠杆菌进行染色体水平上的改造有如下两种方法:一是利用其自身RecA同源重组系统编码的重组蛋白介导DNA发生同源重组;二是通过引入外源重组酶λ-Red或RecET提高同源重组效率;其中上述两种均是通过DNA分子发生双交换实现对靶基因的编辑[3]。这些传统的大肠杆菌基因编辑方法,广泛应用于对靶基因的敲除以及目的基因的插入等,但当对靶基因进行点突变等更加精细的操作时,这些方法存在重组效率低、流程繁琐等缺点,想要大规模应用仍然面临巨大的挑战[4]。
CRISPR (Clustered regularly interspaced short palindromic repeats)技术的出现有效促进了上述问题的解决,能够提高对靶基因编辑的准确性和同源重组效率,逐渐发展成为一种强大的基因编辑工具。CRISPR/Cas系统在微生物中存在广泛,90%的古菌和50%的细菌拥有该系统,作为细菌和古菌抵御外源入侵的一种适应性免疫防御机制。不同微生物自身所携带的CRISPR系统结构有所差异[5–6],根据其前体复合物的类型分为两大类(Class 1和2),其中Class 2结构相对简单只需单个效应蛋白[7–9]。CRISPR-Cas9系统是目前研究最深入也是应用最多的类型(属于Class 2),其结构较为简单,主要由Cas9蛋白、CRISPR RNA (crRNA)、trans-activating (tracrRNA)三部分组成,其中Cas9蛋白是一种核酸内切酶,tracrRNA和crRNA共同结合定向引导Cas9蛋白切割靶基因[10]。随着研究的深入,目前已经把该系统中的crRNA和tracrRNA融合为一条单链引导RNA,即sgRNA[11],该序列的5?端含有20-nt的spacer序列,改变spacer序列,可以使Cas9蛋白识别基因组上不同的PAM序列[12],从而实现对不同靶位点DNA双链的特异性切割。CRISPR-Cas系统中,不同来源的Cas蛋白对应的PAM序列结构也有所不同,其中来源于酿脓链球菌(Streptococcus pyogenes)的spCas9其PAM序列为5?-NGG结构[13]。基因组受损伤的细胞会通过非同源末端连接(nonhomologous end joining,NHEJ)或同源介导的DNA双链修复(homology-directed repair,HDR)对损伤DNA进行修复,借助生物的这种特性,成功将CRISPR-Cas9系统应用于人类疾病治疗等真核生物的基因组编辑中[14–16],相比于真核生物,该系统在原核生物中也展现出强大的编辑功能,包括一些古菌[17]、大肠杆菌(Escherichia coli)[18]、铜绿假单胞菌(Pseudomona aeruginosa)[19]、金黄色葡萄球菌(Staphylococcus aureus)[20]、谷氨酸棒状杆菌(Corynebacterium glutamicum)[21–22]、肺炎克雷伯氏菌(Klebsiella Pneumoniae)[23]等,均成功实现了对靶基因的编辑。
随着CRISPR/Cas系统的出现与应用,使得碱基编辑器开始逐步发展起来,应用于不同领域[24–26],通过对Cas蛋白进行改造,保留与DNA的结合能力,使其只具有切割DNA单链的功能,结合碱基脱氨酶的催化活性,实现核酸链上特定位点碱基的变化[27]。目前应用较多的是DNA碱基编辑器,分为两类,一类是胞嘧啶碱基编辑器(cytosine base editor,CBE),实现C到T的转换[28];另一类是腺嘌呤碱基编辑器(adenine base editor, ABE),实现A到G的转换[29]。虽然碱基编辑器操作简单,但是在原核生物中,碱基编辑器的发展与应用尚不成熟,只在少数细菌中得到应用[19, 30–31],并且多数研究结果显示,碱基编辑器存在较高的脱靶率,编辑窗口有限,仍需进一步的优化。
本研究以细菌互作遗传机制[32–33]得到的显著基因yjjW (T→C)为例,旨在探究一种高效的大肠杆菌靶基因点突变方法。将传统基因编辑方法与CRISPR-Cas9技术相结合,首先,利用同源重组将靶基因替换为amp标记基因;其次,在此基础上设计能够特异性识别amp的spacer序列,利用CRISPR-Cas9系统特异性识别切割amp标记基因,最终获得目的菌株。通过上述两步法,建立一种高效、简捷的靶基因点突变方法。
1 材料和方法 1.1 菌株和质粒 本研究所使用的菌株和质粒详细信息见表 1。
表 1. 本实验所用菌株和质粒 Table 1. Strains and plasmids used in this study
| Strains and plasmids | Characteristics | References |
| Strains | ||
| E. coli D7 (Our laboratory No.) | Original strain | CGMCC (No.1.128) |
| DH5α | For transformation | TransGen Biotech |
| Plasmids | ||
| pKOV | Chloramphenicol resistance (chlR), levansucrase lethal (sacB), temperature- sensitive (rep101) replicon | MiaoLing Bio |
| pMAD | Ampicillin resistance (ampR) | MiaoLing Bio |
| pKOV-HR | pKOV derivative with the repair arms of the yjjW gene | This work |
| pSGKP-km | Kanamycin resistance (kanR), sgRNA expression cassettes, levansucrase lethal (sacB) | Kindly provided by Prof. Quanjiang Ji |
| pCasKP-apr | Apramycin resistance (aprR), expression Cas9 and λ-Red recombination proteins, temperature- sensitive (rep101) replicon | Kindly provided by Prof. Quanjiang Ji |
| pSGKP-km-spacer | pSGKP-km derivative with the spacer of the amp gene | This work |
| pSGKP-km-spacer-HR | pSGKP-km-spacer derivative with the repair arms of the yjjW gene and the mutant yjjW gene | This work |
表选项
1.2 引物 本研究所用的引物见表 2。
表 2. 本实验中使用的引物 Table 2. Primers used in this study
| Primers name | Primers sequence (5′→3′) | Product size/bp |
| yjjW1-U-F | gtacccggggatcgcggccgcCGGTGGGGGAGATCGGTCTGGA | 1053 |
| yjjW1-U-R | aataggggttccgcgCATGCCGGATACGGCCTACGGT | |
| amp-F | gccgtatccggcatgCGCGGAACCCCTATTTGTTTAT | 996 |
| amp-R | cgcgcgtgataagccTTACCAATGCTTAATCAGTGAG | |
| yjjW1-D-F | attaagcattggtaaGGCTTATCACGCGCGGCTGGCG | 952 |
| yjjW1-D-R | cggtcgactctagaggatccGGAAGTGGCGAAGAACATCTGT | |
| Vet1-F | TTCGAAAGCAAATTCGACCCG | 3167 |
| Vet1-R | AACGTGGCGAGAAAGGAAGG | |
| D7-F | GCCGTTTAGTGGCGATGAAG | 3155/3053 |
| D7-R | GGTTTCACCGAACCTGACCT | |
| yjjW2-F | tcctgcagcccgggggatccCGGTGGGGGAGATCGGTCTGGA | 2740 |
| yjjW2-R | gctctagaactagtggatccGGAAGTGGCGAAGAACATCTGT | |
| mut-F | aaatacagcgccggaaataCtaaccggctcaccccg | 1797 |
| mut-R | ggggtgagccggttaGtatttccggcgctgtattt | 978 |
| Vet2-F | ACTTGAAAAAGTGGCACCGAG | 3039 |
| Vet2-R | TCCGGCTCGTATGTTGTGTG | |
| spacer-F | TTGACAGCTAGCTCAGTCCT | 156 |
| spacer-R | GGGCTGCAGGAATTCGATATC | |
| In primers yjjW1-U-F/R, amp-F/R, yjjW1-D-F/R, yjjW2-F/R, the lowercase bases were part of homologous sequences of the connecting fragment, and the uppercase base sequences were sequences complementary to the PCR template. In primers mut-F/R, the uppercase base was a mutant of the yjjW, the lowercase bases were complementary to the PCR template. | ||
表选项
1.3 主要试剂和仪器 氯霉素、氨苄青霉素、卡那霉素、DNA Marker购自北京博迈德技术有限公司;细菌基因组DNA快速提取等试剂盒购自天根生化科技(北京)有限公司;安普霉素、NotⅠ等内切酶购自北京百灵克生物科技有限公司;引物合成和PCR产物测序委托生工生物工程(上海)股份有限公司完成;电转仪、PCR仪,购自Bio-Rad公司。
1.4 大肠杆菌靶基因与标记基因替换实验
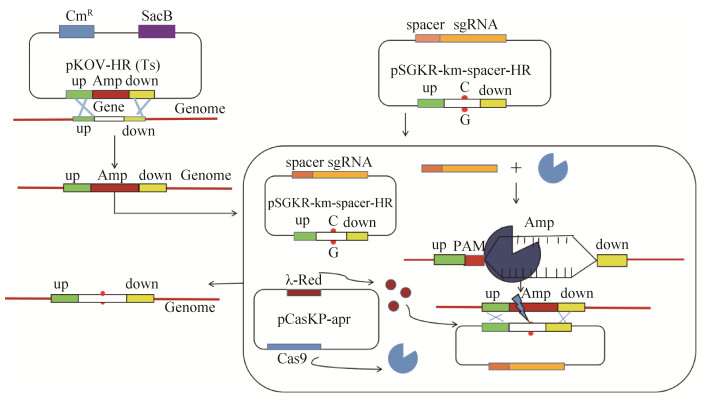
1.4.1 重组质粒的构建: 以敲除E. coli D7 (以下简称D7)基因组的yjjW为例,该部分实验利用的是同源重组系统,采用pKOV质粒进行本阶段实验,该质粒含有蔗糖致死基因(sacB)和温度敏感基因(rep101),最适培养温度为30 ℃,pKOV重组质粒构建方法如下。
首先使用NotⅠ/BamHⅠ对pKOV质粒进行双酶切,获得线性化的质粒片段。然后以D7基因组为模板,分别以yjjW1-U-F/yjjW1-U-R和yjjW1-D-F/yjjW1-D-R为引物扩增得到yjjW的上、下游同源臂,其中上游同源臂的5′端和下游同源臂的3′端通过引物分别引入线性化质粒两端(NotⅠ,BamHⅠ)的一段同源序列,用于与线性化质粒片段的高效连接。以pMAD质粒基因组为模板,使用amp-F/amp-R引物扩增amp片段,amp片段的两端分别含有与上下游同源臂的3′和5′端的同源序列,然后利用重组试剂盒将上下游同源臂、amp片段和线性化质粒片段进行连接,通过热激转化至E. coli DH5α感受态细胞中,30 ℃过夜培养,将长出的单菌落提取质粒基因组,并用Vet1-F/R进行PCR及测序验证,将验证后的重组质粒命名为pKOV-HR。
1.4.2 含标记基因菌株的获取: 将上述构建好的重组质粒pKOV-HR转化至D7感受态细胞中,涂布至含有氨苄青霉素和氯霉素抗性(以下简称双抗)的平板上,30 ℃过夜培养。随机挑取单菌落涂布至双抗平板,42 ℃过夜培养,由于该质粒具有温度敏感基因rep101,在高温培养条件下,不含有同源修复臂的质粒在宿主体内无法正常复制,而含有同源修复臂的重组质粒会与宿主基因组发生同源重组,使得质粒DNA整合至宿主基因组。从42 ℃培养平板上随机挑取单菌落涂布至含有5%蔗糖的氨苄青霉素抗性平板上,30 ℃过夜培养,发生二次同源重组,实现amp标记基因与靶基因的替换,同时pKOV质粒消除。采用96孔板筛选发生重组的菌株,挑取同一单菌落分别接种至氨苄青霉素抗性和氯霉素抗性的LB培养液中,置于30 ℃、150 r/min摇床中过夜培养。筛选在氨苄青霉素抗性培养基中生长但在氯霉素抗性培养基中不生长的菌落,扩大培养,提基因组验证。使用Vet1-F/R进行特异性片段扩增,并且交于生工生物工程(上海)股份有限公司测序,进一步进行结果验证,从而获得靶基因敲除同时带有氨苄青霉素抗性的大肠杆菌D7ΔyjjW::ampR。
1.5 大肠杆菌靶基因点突变实验 本研究的点突变实验,是在上一步实验获得的带有氨苄基因菌株的基础上进行的,采用CRISPR/Cas9双质粒体系实现对靶基因的点突变,其中pSGKP-km带有sgRNA序列以及蔗糖致死基因(sacB),同时作为连接spacer和同源修复臂的载体质粒;pCasKP-apr质粒用于表达Cas9蛋白和λ-Red重组酶,带有温度敏感基因(rep101)。
1.5.1 重组质粒的构建: 以上一步敲除实验获得的大肠杆菌D7ΔyjjW::ampR基因组为模板,在http://www.rgenome.net/设计可以识别amp基因的20-nt的spacer序列(5′-AAAAGGGAATAAGGGCG ACA-3′),在spacer序列的5′端加上与pSGKP-km质粒BsaⅠ酶切位点同源的4个碱基(5′TAGT-),用于spacer与pSGKP-km质粒的连接,然后将spacer序列磷酸化处理,使用T4 DNA连接酶将磷酸化的spacer与线性化的(BsaⅠ) pSGKP-km质粒连接[23],获得pSGKP-km-spacer质粒。采用重叠延伸PCR的方法获得含有点突变(T→C)的yjjW的片段,同时两端带有该基因的上、下游同源修复臂。将同源修复臂与使用BamHⅠ线性化的pSGKP-km-spacer质粒连接,通过热激法转化至大肠杆菌DH5α感受态细胞中,涂布于卡那霉素抗性平板,30 ℃过夜培养获得单菌落,利用Vet2-F/R进行PCR验证以及测序验证,将验证后的重组质粒命名为pSGKP-km-spacer-HR。
1.5.2 点突变菌株的获取: 首先制备已将pCasKP-apr质粒电击转化至D7ΔyjjW::ampR细胞中的大肠杆菌感受态细胞,在制备感受态细胞的过程中添加阿拉伯糖诱导Cas9蛋白和λ-Red重组酶的表达。将pSGKP-km-spacer-HR重组质粒转化至上述感受态细胞中,涂布至添加阿拉伯糖的卡那霉素和安普霉素双抗平板,30 ℃过夜培养。挑取单菌落涂布至含有5%蔗糖的安普霉素抗性平板,30 ℃过夜培养。使用96孔板,挑取同一单菌落分别接种至无抗性、氨苄青霉素抗性和卡那霉素抗性三种LB培养液中,筛选在氨苄青霉素和卡那霉素抗生素培养液中均不生长的菌落,提基因组进行PCR和测序验证,验证结果正确的即为大肠杆菌点突变菌株D7yjjW-24。42 ℃高温过夜培养,消除pCasKP-apr质粒。实验流程如图 1所示。
 |
| 图 1 实验流程图 Figure 1 Flow chart of experiment. |
| 图选项 |
2 结果和分析 2.1 大肠杆菌靶基因与标记基因替换实验
2.1.1 重组质粒的构建: 按方法部分所述引物PCR扩增,获得两端带有同源序列的上、下游同源臂和amp片段(图 2-A、2-B),通过重组克隆试剂盒将这3条片段与线性化的pKOV质粒相连,获得重组质粒pKOV-HR。使用Vet1-F/Vet1-R引物对重组质粒进行PCR扩增,如图 2-C所示,产物(3270 bp)条带在3000 bp左右,随机挑选了3个无非特异性条带且条带较亮的PCR产物送测序,结果显示3片段已成功与质粒相连,重组质粒pKOV-HR构建成功。
 |
| 图 2 pKOV-HR重组质粒构建及验证琼脂糖凝胶电泳图 Figure 2 The construction and validation of pKOV-HR recombinant plasmid. M: marker. A: amplification of upstream and downstream homologous fragments of yjjW gene by PCR; 1–2: downstream homologous arms of yjjW, 3–4: upstream homologous arms of yjjW. B: amplification of ampR fragment by PCR; 1–4: amp gene fragment. C: the PCR validaton of upstream and downstream homologous arms and ampR three fragments connection with linearized pKOV plasmid; 1–5: fusion fragment. |
| 图选项 |
2.1.2 含标记基因菌株的获取: 将pKOV-HR质粒转化至D7感受态细胞中,经30 ℃→42 ℃→ 30 ℃,一系列温度培养之后,重组质粒与宿主基因组发生同源重组,将yjjW替换为ampR基因,随机挑选44个单菌落分别接种至氨苄青霉素和氯霉素抗性培养基中,其中有24个菌落在氨苄青霉素培养基中生长,但在氯霉素培养基中不生长,提取24株菌的基因组使用D7-F/amp-R引物PCR验证,结果显示22株菌成功扩增出目的条带(2255 bp),部分菌株的PCR结果如图 3所示,说明这些菌株已经成功完成了amp标记基因的插入,同时实现了目的基因的敲除,测序结果如图 4所示,表明已成功获得了带有ampR的敲除菌株。
 |
| 图 3 敲除菌株PCR验证 Figure 3 PCR validation of knockout strains. M: marker; 1–3: validation of E. coli D7ΔyjjW::ampR with primers D7-F/amp-R. |
| 图选项 |
 |
| 图 4 敲除菌株测序结果 Figure 4 The sequence validation of knockout strain. |
| 图选项 |
2.2 点突变实验
2.2.1 重组质粒的构建: spacer-F/spacer-R引物的PCR产物及测序结果显示(图 5-A、图 6),spacer已经成功连接在载体质粒上,获得pSGKP-km-spacer质粒,重叠延伸PCR获得带有上、下游同源臂的yjjW点突变片段(图 5-C、5-D),使用重组试剂盒将该片段(图 5-D)与线性化的pSGKP-km-spacer质粒相连,如图 5-B所示,Vet2-F/Vet2-R引物PCR产物的条带在3000 bp左右,说明同源修复臂与载体质粒已经相连,同时测序结果也显示重叠延伸PCR成功实现了yjjW基因的点突变。结果表明已成功获得带有识别amp基因序列的spacer及同源修复臂的重组载体质粒pSGKP-km-spacer-HR。
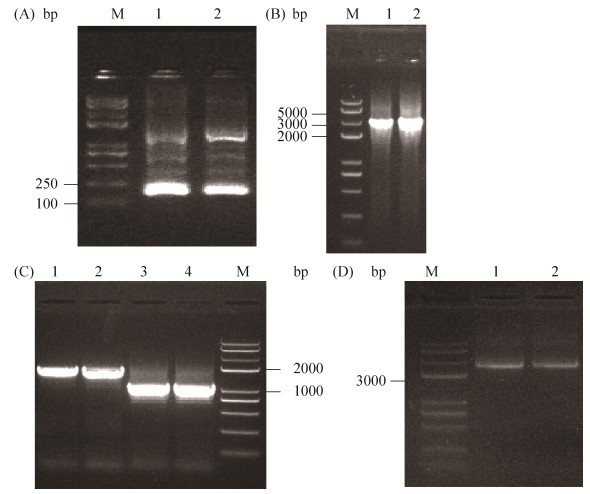 |
| 图 5 重组质粒pSGKP-km-spacer-HR构建及验证琼脂糖凝胶电泳图 Figure 5 The construction and validation of recombinant plasmid pSGKP-km-spacer-HR; M: marker. A: validation of spacer fragment; 1–2: PCR product of pSGKP-km-spacer with primers spacer-F/R; B: validation of homologous repair arm connections with pSGKP-km-spacer; 1–2: PCR product of pSGKP-km-spacer-HR with primers Vet2-F/R PCR; C: overlap PCR of yjjW gene site-directed mutation; 1–2: PCR product with primers yjjW2-F/mut-R PCR; 3–4: PCR product withe primers mut-F/yjjW2-R; D: overlap PCR of yjjW gene site-directed mutation; 1–2: PCR product with primers yjjW2-F/R. |
| 图选项 |
 |
| 图 6 Spacer测序结果 Figure 6 The sequence validation of spacer. |
| 图选项 |
2.2.2 点突变菌株的获取: 将重组质粒pSGKP-km-spacer-HR转化至事先制备好的含有pCasKP-apr质粒的D7ΔyjjW::ampR感受态细胞中,如上述方法操作,最后成功从36个单菌落中筛选到27株菌在相应抗性培养基中不生长,说明pSGKP-km-spacer-HR重组质粒已成功消除,提取27株菌的基因组进行PCR验证,结果显示,这27株菌使用D7-F/amp-R引物均未扩增出任何条带,表明这些菌株均发生了同源重组修复并且已经失去了氨苄青霉素抗性,此外有17株菌使用D7-F/D7-R引物成功扩增出了目的条带(3053 bp),部分菌株PCR结果如图 7所示,从这17株菌中随机挑选4株菌的PCR产物进行测序,部分测序结果如图 8所示,结果显示,这4株菌的yjjW基因均实现了预期的点突变,成功获得了我们所需的突变株,并且成功将pCasKP-apr消除,提取基因组进行PCR测序验证,结果显现该突变株未发生回复突变,表明该实验条件稳定,突变株不易发生回复突变。
 |
| 图 7 点突变菌株验证PCR Figure 7 The validation of point mutant strains. M: marker; 1–3: PCR product of E. coli D7yjjW-24 with primers D7-F/R; 4–6: PCR product of E. coli D7yjjW-24 with primers D7-F/amp-R. |
| 图选项 |
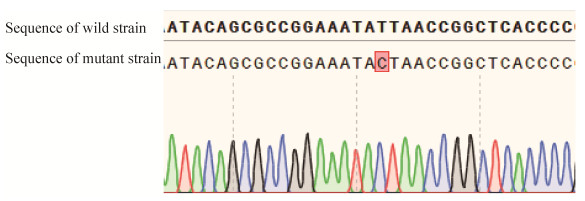 |
| 图 8 yjjW点突变菌株测序结果 Figure 8 The sequence validation of point mutant strain. |
| 图选项 |
3 讨论 CRISPR/Cas9基因编辑技术作为一种新型编辑技术,与传统方法相比具有编辑效率高、操作简便、能完成定点编辑等优点[7]。目前,该方法不断发展与创新,已经成为一种成熟的基因编辑技术,在细菌领域应用广泛,季泉江等运用该方法成功对金黄色葡萄球菌、铜绿假单胞菌等致病菌实现了基因敲除以及基因表达调控,深入了解一些致病菌的耐药性和致病机制[19-20]。CRISPR/Cas9进行基因编辑主要有两种方式,一种是利用质粒载体引入外源CRISPR/Cas9系统;另一种是利用菌体自身所带有的CRISPR/Cas系统进行编辑[34],本研究利用的是第一种方式。
以大肠杆菌yjjW为例,结合CRISPR/Cas9基因编辑技术的优点,探究出一个适用于大肠杆菌靶基因点突变的方法。最初,我们对大肠杆菌靶基因的点突变利用的只是同源重组系统,但结果显示其重组效率低,不能高效筛选到所需菌株。因此,在此基础上结合了CRISPR/Cas9基因编辑技术,利用pSGKP-km/pCasKP-apr双质粒进行靶基因的点突变,结果显示,结合该技术大大提高了获得目的菌株的效率。此外,实验中构建带有可以识别amp spacer序列的pSGKP-km-spacer质粒,还可以快速实现对其他带有amp基因菌株的基因编辑,只需改变同源修复片段,避免了spacer序列的重新设计,同时抗性基因的引入为最后阳性克隆的获取提供了简单的筛选标记,可以快速实现对大肠杆菌其他靶基因的点突变,有效提高了实验效率。
随着研究人员对CRISPR系统的深入研究,发现一些细菌含有Cpf1蛋白,该蛋白与Cas9蛋白相比,具有分子量小,结构简单,能进行多重编辑的优点,并且具有独特的双重切割酶活性,表明该系统未来具有很广的应用前景,未来可能成为与Cas9蛋白功能互补的基因编辑工具[35-37]。虽然CRISPR/Cas系统是目前研究热点之一,但是该系统的研究与应用仍然存在一定的问题,脱靶效应以及PAM序列的识别限制了其广泛应用[38],需要更全面深入的探究其机制才能更好地将该技术应用于各项研究中。
综上所述,我们建立了一种CRISPR/Cas9系统介导的快速、简单和高效的大肠杆菌基因点突变方法,为大肠杆菌基因编辑的研究提供了有效的工具,为本实验室后续验证大肠杆菌互作基因奠定了科学理论和实践基础。同时,该方法的建立,对一些其他生物统计学分析结果的验证提供了参考价值,例如当利用遗传统计模型探究影响微生物某种性状遗传机制时,会获得大量的显著基因(往往是单个碱基的变化),参考该方法的建立,可以快速实现对SNP位点的编辑。
References
| [1] | Zerbini F, Zanella I, Fraccascia D, K?nig E, Irene C, Frattini LF, Tomasi M, Fantappiè L, Ganfini L, Caproni E, Parri M, Grandi A, Grandi G. Large scale validation of an efficient CRISPR/Cas-based multi gene editing protocol in Escherichia coli. Microbial Cell Factories, 2017, 16: 68. DOI:10.1186/s12934-017-0681-1 |
| [2] | Li Q, Wu MX, Guo QH, Shao YR, Yang JJ, Jiang Y, Yang S. Advances in bacterial genome editing techniques. Chinese Bulletin of Life Sciences, 2019, 31(5): 473-492. (in Chinese) 李琦, 武美贤, 郭清华, 邵悠然, 杨俊杰, 蒋宇, 杨晟. 细菌基因组编辑技术进展. 生命科学, 2019, 31(5): 473-492. |
| [3] | Liu J, Liu WF, Tao Y, Ma YH. Novel efficient strategy for continual CRISPR-mediated genome editing in Escherichia coli. Microbiology China, 2018, 45(8): 1621-1631. (in Chinese) 刘姣, 刘伟丰, 陶勇, 马延和. 一种大肠杆菌快速连续CRISPR基因组编辑的新策略. 微生物学通报, 2018, 45(8): 1621-1631. |
| [4] | Bao AK, Bai TH, Zhao TX, Su JH. CRISPR/Cas9:a gene targeting technology and its application in the study of plant genetic function. Acta Prataculturae Sinica, 2017, 26(7): 190-200. (in Chinese) 包爱科, 白天惠, 赵天璇, 苏家豪. CRISPR/Cas9系统:基因组定点编辑技术及其在植物基因功能研究中的应用. 草业学报, 2017, 26(7): 190-200. |
| [5] | Kleinstiver BP, Prew MS, Tsai SQ, Nguyen NT, Topkar VV, Zheng ZL, Joung JK. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology, 2015, 33(12): 1293-1298. DOI:10.1038/nbt.3404 |
| [6] | Qu DF, Shen Y, Zhang CC, Han JZ. Genomics comparison between CRISPR loci and plasmid in Salmonella enteric. Acta Microbiologica Sinica, 2018, 58(2): 209-218. (in Chinese) 曲道峰, 沈杨, 张聪聪, 韩剑众. 沙门氏菌CRISPR位点的结构特征比较. 微生物学报, 2018, 58(2): 209-218. |
| [7] | Hu L, Chen S. Research progress of CRISPR-Cas system in bacteria. Acta Microbiologica Sinica, 2017, 57(11): 1643-1652. (in Chinese) 胡丽, 陈实. 细菌CRISPR-Cas系统的研究进展. 微生物学报, 2017, 57(11): 1643-1652. |
| [8] | Zhu YW, Huang ZW. Recent advances in structural studies of the CRISPR-Cas-mediated genome editing tools. National Science Review, 2019, 6(3): 438-451. DOI:10.1093/nsr/nwy150 |
| [9] | Makarova KS, Wolf YI, Alkhnbashi OS, Costa F, Shah SA, Saunders SJ, Barrangou R, Brouns SJJ, Charpentier E, Haft DH, Horvath P, Moineau S, Mojica FJM, Terns RM, Terns MP, White MF, Yakunin AF, Garrett RA, van der Oost J, Backofen R, Koonin EV. An updated evolutionary classification of CRISPR-Cas systems. Nature Reviews Microbiology, 2015, 13(11): 722-736. DOI:10.1038/nrmicro3569 |
| [10] | 王思涵. S. pyogenes Cas9的表达纯化及其活性抑制剂筛选的研究.哈尔滨工业大学硕士学位论文, 2018. |
| [11] | Qi LS, Larson MH, Gilbert LA, Doudna JA, Weissman JS, Arkin AP, Lim WA. Repurposing CRISPR as an RNA-Guided Platform for Sequence-Specific control of gene expression. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [12] | Guo MH, Ren K, Zhu YW, Tang ZY, Wang YH, Zhang BL, Huang ZW. Structural insights into a high fidelity variant of SpCas9. Cell Research, 2019, 29: 183-192. DOI:10.1038/s41422-018-0131-6 |
| [13] | Cong L, Zhang F. Genome engineering using CRISPR-Cas9 system//Pruett-Miller SM. Chromosomal Mutagenesis. New York: Humana Press, 2015: 197-217. |
| [14] | Shalem O, Sanjana NE, Hartenian E, Shi X, Scott DA, Mikkelsen TS, Heckl D, Ebert BL, Root DE, Doench JG, Zhang F. Genome-Scale CRISPR-Cas9 knockout screening in human cells. Science, 2013, 343(6166): 84-87. |
| [15] | Yan W, Xiang R, Xiang H, Wang XH. Application of CRISPR-Cas9 system in disease treatment. Immunological Journal, 2016, 32(10): 907-911. (in Chinese) 颜雯, 向蓉, 向华, 王晓虎. CRISPR-Cas9技术在疾病治疗中的应用. 免疫学杂志, 2016, 32(10): 907-911. |
| [16] | Zhang S, Wang Z, Lin YP, Rong QQ, Wang LX, Qi XN, Liu H, Wang QH. DNA lesions and repair response caused by genome editing in Saccharomyces cerevisiae. Acta Microbiologica Sinica, 2020. (in Chinese) 张首, 王震, 蔺玉萍, 戎倩倩, 王丽贤, 齐显尼, 刘浩, 王钦宏. 基因组编辑对酿酒酵母DNA的损伤作用及修复响应. 微生物学报, 2020. DOI:10.13343/j.cnki.wsxb.20190431 |
| [17] | Liu T, Wang XJ, Li YJ, Peng N. Systems for the genetic manipulation of Sulfolobus islandicus. Microbiology China, 2019, 46(2): 398-406. (in Chinese) 刘涛, 王晓婕, 李英俊, 彭楠. 冰岛硫化叶菌遗传操作体系的研究进展. 微生物学通报, 2019, 46(2): 398-406. |
| [18] | Zhao DD, Feng X, Zhu XN, Wu T, Zhang XL, Bi CH. CRISPR/Cas9-assisted gRNA-free one-step genome editing with no sequence limitations and improved targeting efficiency. Scientific Reports, 2017, 7: 16624. DOI:10.1038/s41598-017-16998-8 |
| [19] | Chen WZ, Zhang Y, Zhang YF, Pi YS, Gu TN, Song LQ, Wang Y, Ji QJ. CRISPR/Cas9-based Genome editing in Pseudomonas aeruginosa and cytidine deaminase-mediated base editing in Pseudomonas species. iScience, 2018, 6: 222-231. DOI:10.1016/j.isci.2018.07.024 |
| [20] | Chen WZ, Zhang YF, Yeo WS, Bae T, Ji QJ. Rapid and efficient genome editing in Staphylococcus aureus by using an engineered CRISPR/Cas9 system. Journal of the American Chemical Society, 2017, 139(10): 3790-3795. DOI:10.1021/jacs.6b13317 |
| [21] | Zhan ML, Kan BJ, Zhang H, Dong JJ, Xu GC, Han RZ, Ni Y. Comparison of CRISPR-Cpf1 with Cre/loxP for gene knockout in Corynebacterium glutamicum. Microbiology China, 2019, 46(2): 278-291. (in Chinese) 占米林, 阚宝军, 张辉, 董晋军, 许国超, 韩瑞枝, 倪晔. 谷氨酸棒状杆菌CRISPR-Cpf1和Cre/loxP基因敲除技术的比较. 微生物学通报, 2019, 46(2): 278-291. |
| [22] | Peng F, Wang XY, Sun Y, Dong GB, Yang YK, Liu XX, Bai ZH. Efficient gene editing in Corynebacterium glutamicum using the CRISPR/Cas9 system. Microbial Cell Factories, 2017, 16: 201. |
| [23] | Wang Y, Wang SS, Chen WZ, Song LQ, Zhang YF, Shen Z, Yu FY, Li M, Ji QJ. CRISPR-Cas9 and CRISPR-assisted cytidine deaminase enable precise and efficient genome editing in Klebsiella pneumoniae. Applied and Environmental Microbiology, 2018, 84(23): e01834-18. |
| [24] | Hess GT, Tycko J, Yao D, Bassik MC. Methods and applications of CRISPR-Mediated base editing in Eukaryotic genomes. Molecular Cell, 2017, 68(1): 26-43. |
| [25] | Seo H, Kim JS. Towards therapeutic base editing. Nature Medicine, 2018, 24(10): 1493-1495. |
| [26] | Wang Y, Liu Y, Liu J, Guo YM, Fan LW, Ni XM, Zheng XM, Wang M, Zheng P, Sun JB, Ma YH. MACBETH:multiplex automated Corynebacterium glutamicum base editing method. Metabolic Engineering, 2018, 47: 200-210. |
| [27] | Zhao YW, Jiang WH, Deng ZX, Wang ZJ, Lu YH. Development and application of base editors in bacterial genome editing. Microbiology China, 2019, 46(2): 319-331. (in Chinese) 赵亚伟, 姜卫红, 邓子新, 汪志军, 芦银华. 碱基编辑器的开发及其在细菌基因组编辑中的应用. 微生物学通报, 2019, 46(2): 319-331. |
| [28] | Zheng K, Wang Y, Li N, Jiang FF, Wu CX, Liu F, Chen HC, Liu ZF. Highly efficient base editing in bacteria using a Cas9-cytidine deaminase fusion. Communications Biology, 2018, 1: 32. |
| [29] | Gaudelli NM, Komor AC, Rees HA, Packer MS, Badran AH, Bryson DI, Liu DR. Programmable base editing of A?T to G?C in genomic DNA without DNA cleavage. Nature, 2018, 559(7714): E8. |
| [30] | Gu TN, Zhao SQ, Pi YS, Chen WZ, Chen CY, Liu Q, Li M, Han DL, Ji QJ. Highly efficient base editing in Staphylococcus aureus using an engineered CRISPR RNA-guided Cytidine deaminase. Chemical Science, 2018, 9(12): 3248-3253. |
| [31] | Arazoe T, Kondo A, Nishida K. Targeted nucleotide editing technologies for microbial metabolic engineering. Biotechnology Journal, 2018, 13(9): 1700596. |
| [32] | Zhang ZR, Li JT, Liang YJ, Zhang Q, Ji Y, He XQ. Analysis of interaction mode between Escherichia coli and Staphylococcus aureus by systems mapping. Microbiology China, 2019, 46(2): 292-300. (in Chinese) 张佐然, 李金婷, 梁雅静, 张琦, 金一, 何晓青. 利用系统作图(Systems mapping)研究大肠埃希菌和金黄色葡萄球菌的互作遗传机制. 微生物学通报, 2019, 46(2): 292-300. |
| [33] | Chen N, Zhu J, Ye MX, Jin Y, He XQ, Wu RL. Interactions between Escherichia coli and Staphylococcus aureus determined by genome-wide association analysis. Acta Microbiologica Sinica, 2017, 57(4): 526-538. (in Chinese) 陈南, 朱璟, 叶梅霞, 金一, 何晓青, 邬荣领. GWAS研究大肠杆菌和金黄色葡萄球菌种间互作进化机制. 微生物学报, 2017, 57(4): 526-538. |
| [34] | Pyne ME, Bruder MR, Moo-Young M, Chung DA, Chou CP. Harnessing heterologous and endogenous CRISPR-Cas machineries for efficient markerless genome editing in Clostridium. Scientific Reports, 2016, 6: 25666. |
| [35] | 杨建争. Cpf1的小分子抑制剂的筛选与复合物结晶的研究.哈尔滨工业大学硕士学位论文, 2018. |
| [36] | Guo T, An XM. Application and comparison of CRISPR-Cas9 system and CRISPR-Cpf1 system in multigenome editing. Chinese Journal of Cell Biology, 2019, 41(11): 2234-2244. (in Chinese) 郭婷, 安新民. 多重基因组编辑中CRISPR-Cas9系统和CRISPR-Cpf1系统的应用和比较. 中国细胞生物学学报, 2019, 41(11): 2234-2244. |
| [37] | Adiego-Pérez B, Randazzo P, Daran JM, Verwaal R, Roubos JA, Daran-Lapujade P, van der Oost J. Multiplex genome editing of microorganisms using CRISPR-Cas. FEMS Microbiology Letters, 2019, 366(8): fnz086. |
| [38] | Fu JH, Yang FY, Xie HH, Gu F. Application and optimization of CRISPR/Cas system in bacteria. Chinese Journal of Biotechnology, 2019, 35(3): 341-350. (in Chinese) 傅俊豪, 杨发誉, 谢海华, 谷峰. 细菌中CRISPR/Cas系统的应用和优化. 生物工程学报, 2019, 35(3): 341-350. |
