孟凡凡1,2, 胡盎3, 王建军1,2


1. 中国科学院南京地理与湖泊研究所, 湖泊与环境国家重点实验室, 江苏 南京 210008;
2. 中国科学院大学, 北京 100049;
3. 湖南农业大学资源环境学院, 湖南 长沙 410128
收稿日期:2020-05-03;修回日期:2020-06-26;网络出版日期:2020-07-20
基金项目:国家自然科学基金(91851117)
作者简介:王建军, 中国科学院南京地理与湖泊研究所研究员, 博导, 研究方向为水域微生物生态学与全球变化。南京师范大学本科毕业, 中国科学院南京地理与湖泊研究所获硕士和博士学位。中国科学院生态环境研究中心、奥克拉荷马大学博士后、赫尔辛基大学高级研究员。先后赴日本、芬兰、西班牙和美国等地的研究所/高校访学。主持或参与国家自然科学基金、中科院前沿重点项目、国家重点研发、美国国家地理等项目。获得江苏省科学技术奖一等奖、Publon同行评议奖等。中华海外生态****协会理事、中国生态学会水体生态专业委员会委员、中国生态学学会微生物生态专业委员会委员。Functional Ecology等杂志编委, 40多个SCI刊物审稿人。已在Nature Communications、ISME Journal、Microbiome、Global Ecology and Biogeography等刊物发表 60多篇SCI论文.
*通信作者:王建军, E-mail:jjwang@niglas.ac.cn.
摘要:微生物性状是指与其存活、生长和繁殖紧密相关的一系列核心属性,这些属性能够反映微生物对环境变化的响应,进而影响微生物的物种分布格局、群落构建机制以及相应的生态系统功能。越来越多的研究表明,相比于微生物分类学信息,微生物性状可以在种群、群落和生态系统尺度等视角扩展我们对微生物生态过程的理解,并提供生态模式的机理性解释。本文回顾微生物性状研究的发展历程,总结近年来基于微生物性状研究的前沿科学问题,比如微生物性状的分类和测定方法、基于性状的功能多样性定义及应用、性状与物种分布格局和群落构建机制的关系、性状对生物多样性和生态系统功能的影响以及对环境变化的响应等。尽管微生物性状研究已经延伸到生态学领域的各个方面,有力推动着各个前沿科学问题的研究发展,但是仍然面临很多机遇与挑战。因此,本文也从研究方法和研究方向等方面对未来基于微生物性状的研究提出了展望。
关键词:性状功能多样性物种分布群落构建生态系统功能
Microbial traits shed light on species distributions, assembly processes and ecosystem functions
Fanfan Meng1,2, Ang Hu3, Jianjun Wang1,2


1. State Key Laboratory of Lake Science and Environment, Nanjing Institute of Geography and Limnology, Chinese Academy of Sciences, Nanjing 210008, Jiangsu Province, China;
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of Resources and Environment, Hunan Agricultural University, Changsha 410128, Hunan Province, China
Received: 3 May 2020; Revised: 26 June 2020; Published online: 20 July 2020
*Corresponding author: Jianjun Wang, E-mail:jjwang@niglas.ac.cn.
Foundation item: Supported by the National Natural Science Foundation of China (91851117)
Abstract: Microbial traits are core attributes closely related to microbial survival, growth and reproduction. These attributes could reflect the microbial responses to environmental changes, and then affect species distributions, community assembly processes, and relevant ecosystem functions. There is a growing recognition that, compared to microbial taxonomic information, microbial traits could expand our understanding of microbial ecological processes and provide mechanistic explanations of ecological patterns at the scales of population, community and ecosystem. Here, we address important ecological themes based on microbial traits in recent years, including the classification and determination of microbial traits, functional diversity and applications, the relationship between microbial traits and species distribution and community assembly, the effects of microbial traits on biodiversity and ecosystem functions and the response of microbial traits to global change. Although previous studies on microbial traits have been extended to all aspects of ecology and promoted the research and development of various frontier scientific problems, there are still many opportunities and challenges. Thus, we also provide perspectives such as research methods and directions based on microbial traits.
Keywords: microbial traitsfunctional diversityspecies distributionsassembly processesecosystem functions
微生物是陆地及水域等生态系统的重要组成部分,其中水圈(海洋、湖沼、河流、冰川、地下水等)中居住着地球一半以上的微生物,蕴含着地球上最大的微生物组[1-2]。水圈微生物在碳、氮、硫等元素的生物地球化学循环中发挥关键驱动作用[3-4],但是,人们对其在自然环境中的驱动过程和机理仍缺乏足够的认识[1]。这主要是因为微生物之间存在水平基因转移(horizontal gene transfer),不同的微生物可以执行相似的代谢功能,其群落通常表现出较高的“功能冗余”(functional redundancy)[5-6],使得基于分类学(taxonomy)的研究框架不一定反映出微生物物种多样性减少或群落物种组成变化对其介导的生态过程的显著影响或对环境变化的响应[7]。因此,微生物生态学转而关注基于微生物性状(trait)的功能生态学(functional ecology)研究框架[7-8],比如功能生物地理学[9](functional biogeography)等。通过将这类研究框架应用于水圈微生物,可以进一步深入了解水圈微生物的群落形成、环境互作、生态功能以及对环境变化的响应等。
性状或功能性状(functional trait)是植物、动物和微生物等对外界环境长期适应和进化后所呈现出来的可度量的特征,通过影响有机体的生存、生长和繁殖进而影响其对环境的适应性和功能[10]。其中,微生物性状的研究历史悠久,早期微生物学家通过微生物的表型特征鉴别不同的微生物类群。相比于动植物而言[10],微生物性状的研究相对滞后,这主要是因为微生物种类繁多、体积微小且绝大多数不可培养[11],使得其性状通常难以测定。但是,随着新兴的分子生物学技术,如基因组学、转录组学、蛋白质组学和代谢组学等技术的蓬勃发展,有关微生物种群和群落多样性的数据越来越多[12],给微生物性状的定性和定量研究带来了新的机遇,推动着基于性状的微生物生态学的发展[8]。
当前,基于微生物性状的研究涉及多个层次,从个体水平到生态系统水平,并延伸到微生物生态学研究的各个领域。本文主要关注以下这些前沿的科学问题[13-15],包括:(1)微生物性状的分类与测量;(2)微生物性状与系统发育之间的关系;(3)基于微生物性状的功能多样性沿环境梯度的分布格局及变化规律;(4)基于性状研究微生物的物种分布格局和局域、区域以及全球尺度下的群落构建机制;(5)基于性状研究微生物对生物多样性和生态系统功能的影响以及对环境变化的响应等。本文回顾总结近年来微生物性状研究的发展历程、论述当前研究面临的机遇与挑战,并展望未来具有潜力的研究方向,以供国内相关领域的同行参考。
1 微生物性状的分类与测量方法 1.1 微生物性状的分类
1.1.1 基因型性状与表型性状: 微生物性状可以分为基因型性状(genotypic traits)和表型性状(phenotypic traits)。基因型性状编码微生物的功能潜力。在微生物类群中,基因型性状主要表现为基因组中是否存在特定的功能基因或代谢途径[16]。此外,还包括GC含量、基因数量、有效基因组大小以及16S rRNA基因拷贝数等[17-18]。微生物群落被认为是环境过滤后由一系列功能基因编码的功能文库[19]。在微生物群落中,除了宏基因组中功能基因或代谢途径的绝对丰度以外[16],功能基因变异的多样性(如相对丰度或均匀度等)也可以视为基因型性状[20-21]。
表型性状是基因型性状在微生物类群或群落层次的表观特征,与生态系统过程之间存在更直接的联系[16]。在微生物类群中,表型性状主要包括微生物的形态和生理特征(例如细胞大小、形状、运动性、孢子形成、生长速率和化学计量等)、环境偏好(例如需氧量、最佳pH、最佳温度和耐盐度等)、代谢能力(例如纤维素降解、某些酶的产生等)和病毒抗性等。在微生物群落中,表型性状通常对应与生物多样性和生态系统功能相关的群落功能性状,例如底物利用、气体产生、化合物降解和生物过程的时间稳定性等[16]。
1.1.2 浅层性状与深层性状: 由于不同性状的生物化学特性和遗传复杂性不同,其系统发育保守性存在差异,从而可将性状分为浅层性状(shallow traits)和深层性状(deep traits)。其中,深层性状通常涉及多个功能基因的相互作用,在系统发育中高度保守,例如对pH和盐度的环境偏好;而浅层性状通常涉及较少的功能基因,系统发育保守性相对较差,例如对碳和磷等底物的利用[8]。这是因为浅层的简单性状可能被噬菌体、质粒或转座子携带,通过水平基因转移在微生物物种或个体水平上对基因组进行修饰[22]。性状的系统发育保守性差异可以用来预测微生物的群落组成变化;具体而言,对深层性状或浅层性状产生影响的环境变化将分别在广泛或更精细的分类水平上改变微生物的群落组成[8]。
1.1.3 响应性状和效应性状: 在研究微生物群落如何响应环境变化并影响生态系统功能时,关键在于区分群落的响应性状(response trait)和效应性状(effect trait)[23]。其中,响应性状体现了物种对环境变化的响应和承受能力,决定了其面对新环境时的丰度变化,因此响应性状的集合构成了群落组成变化的基础;而效应性状则体现了物种实现和影响生态系统功能的能力,因此效应性状的集合通常是生态系统功能变化的可靠预测因子[24-25]。与初始群落相比,具有响应性状的物种在受到环境变化影响后,其效应性状的变化程度将决定群落对生态系统功能的影响程度[26]。对于响应性状和效应性状的研究选择,取决于研究者对涉及效应性状的特定生态过程的实现还是对涉及响应性状的跨空间、时间和环境梯度的生态过程的稳定性感兴趣[16]。
1.2 微生物性状的测量方法 微生物性状的测量方法可分为直接方法和间接方法。直接方法是指通过直接观察表型来表征微生物性状[27],主要包括研究微生物形态性状(如细胞形状和细胞壁结构)的传统显微镜技术和培养技术[28-29];量化微生物对各种底物或应激源生理反应(如呼吸)的表型阵列技术(phenotypic arrays);通过分析微生物产生的代谢物,进而获取其生理性状的代谢组学技术;通过分析给定微生物样品产生的蛋白质,进而获取微生物群落生理性状的宏蛋白质组学技术等[30]。由于微生物群落层次的表型性状通常是由多种微生物的相互作用引起的,因此相比于微生物类群层次的表型性状往往难以测量和预测[16]。新的技术方法可以根据微生物群落内的活动或生理状态测量群落水平的表型性状。例如,定量化稳定同位素探针技术(quantitative stable isotope probing)可以在群落层次量化利用同位素标记的特定底物的微生物类群的性状[31]。目前,我们正尝试通过同位素标记葡萄糖中的碳原子和水中的氢原子等测定不同微生物类群的碳源同化速率和生长速率,从效应值(effect size)角度建立新的研究技术方法从而定量获取微生物物种对环境变化的响应特征。
间接方法是指通过基因组学、宏基因组学、转录组学和宏转录组学等组学技术进行基因测序和分析来间接量化微生物性状[27]。这些方法依赖于将目标基因序列与基因或蛋白质的功能注释数据库(如京都基因与基因组百科全书KEGG和蛋白质直系同源簇COGs等)进行比较,从而推测基因序列的功能和微生物的功能潜力。微生物群落层次的基因型性状主要通过两类方法进行描述。一类方法是通过群落内微生物的分类组成进行推断,常用的软件包括PICRUST[32]、Tax4Fun[33]和Vikodak[34]等。第二类方法是通过对宏基因组学或宏转录组学的测序结果进行功能基因或代谢途径的功能归类[35-36]。一旦确定了群落的宏基因组含量,还可以估算出一些基因型性状,如GC含量及其变化、有效基因组大小或宏基因组功能含量等[17]。
2 微生物功能多样性及分布模式 功能多样性(functional diversity)是生物多样性的一个组成部分,是联系生物多样性和生态系统功能的关键要素。尽管功能多样性的重要性越来越被认同,但是其定义却多种多样,如“生态系统中生物所形成和执行的功能类型、数量及其分布”[37],“功能多样性是影响生态系统功能和运行的生物多样性的功能组分”[38]等。本研究认为,功能多样性是特定生态系统中物种功能性状的大小、范围及分布,也称功能性状多样性(functional trait diversity),强调群落中物种功能的差异[39]。自1981年首次提出微生物功能多样性指数[40]以来,人们日益认识到,相比于只包括物种数和相对多度的物种多样性,功能多样性可以提供更有力的理论检验[41-42],用于解释和预测若干重要的生态学问题[15]。
2.1 功能多样性指数 功能多样性主要通过基于功能性状计算的功能多样性指数进行描述(表 1)。目前被广泛认可的指数包括功能多样性指数(functional diversity,FD)[39]、功能丰富度(functional richness,FRic)、功能均匀度(functional evenness,FEve)和功能相异度(functional divergence,FDiv)[43]等。此外,功能发散度(functional dispersion,FDis)[44]、功能特征多样性指数(functional attribute diversity,FAD2)[45]、RaoQ指数[46]和CWM指数(community weighted mean)[47]等也被用于描述功能多样性。
表 1. 功能多样性指数及其含义 Table 1. Functional diversity index and its meaning
| Index | Meaning | Reference |
| FD | The total branch length of a functional dendrogram. | [39] |
| FRic | The amount of functional space filled by the community. | [43] |
| FEve | The evenness of abundance distribution in a functional trait space. | [43] |
| FDiv | The divergence in the distribution of abundance in a functional trait space. | [43] |
| FDis | The mean distance in multidimensional trait space of individual species to the centroid of all species. | [44] |
| FAD2 | The sum of the squared Euclidian distances the species are apart. | [45] |
| RaoQ | The quadratic entropy of Rao that incorporates both the relative abundances of species and a measure of the pairwise functional differences between species. | [46] |
| CWM | Community-level weighted means of trait values. | [47] |
表选项
功能多样性指数的一般计算框架如图 1所示:(1)获得物种的功能性状矩阵;(2)基于欧几里得距离(Euclidean distance)或高尔距离(Gower’s distance)等将功能性状矩阵转换为物种对之间的功能距离矩阵;(3)将功能距离矩阵聚类生成树状图或由主成分分析生成多维的功能空间;(4)结合物种丰度或生物量信息和物种在功能空间中的位置,计算群落的功能多样性指数[48]。在计算功能多样性指数时,用来表征功能丰富度(FRic)和功能发散度(FDis、RaoQ)的多样性指数对性状数量变化表现出较高的敏感性,而功能均匀度(FEve)和功能相异度(FDiv)受性状数量变化的影响较小。因此使用功能多样性解释生态现象和生态过程时,不仅需要考虑具体性状值,而且需要考虑性状数量[49]。
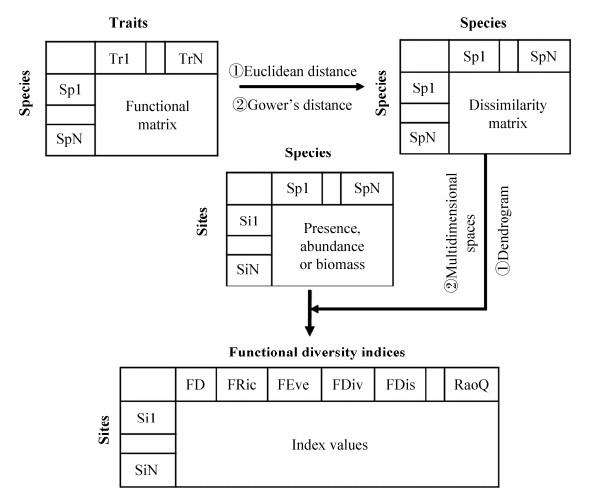 |
| 图 1 基于性状的功能多样性的计算 Figure 1 Calculation of functional diversity based on traits. Sp: Species; Tr: Trait; Si: Site |
| 图选项 |
2.2 功能多样性指数与物种或系统发育多样性指数的关系 生物多样性包括三个维度,即物种多样性、系统发育多样性和功能多样性[16]。传统的物种多样性指数仅包含物种数和相对多度的信息,如物种丰富度、Simpson指数和Shannon-Weiner指数等,其忽视了物种自身的差异在群落构建和生态系统功能维持过程中的作用和贡献的相对大小。系统发育多样性指数是从进化的角度认识群落内物种的多样性格局,通常采用Faith’s PD(群落中物种的系统发育树的所有分支长度之和)[50]作为多样性指数。此外,MPD和MNTD指数以及标准化指数NRI和NTI等也被用来表征系统发育多样性[51-52]。功能多样性指数则将物种的多度和性状分布结合在一起。由于物种在生态过程中的作用和贡献往往与性状密切相关,且功能多样性考虑了物种之间的相互作用以及对环境响应的差异,因此与物种或系统发育多样性相比能够更好地揭示生态过程。此外,由于微生物性状在系统发育上或多或少存在保守性[8],因而PD也可以被用来反映功能多样性[52],并在此基础上衍生出基于功能性状所构建的系统进化树进行计算的功能多样性指数FD[39]。
2.3 功能多样性的分布模式 随着环境或地理梯度的变化,微生物功能多样性通常表现出一定的分布模式。通过认识微生物的多样性分布模式有利于预测环境变化对生态系统的影响[53]。但是,由于研究区域和尺度不同,其分布模式随着环境或地理梯度的变化往往表现出不同的变化趋势。以海拔梯度为例,微生物功能多样性随着海拔升高会表现出上升、下降或单峰等趋势,这和不同地区不同海拔梯度下的地形条件、土壤环境、气候因子以及地上植被状况存在密切关系[54]。借助功能基因芯片(GeoChip)技术,研究发现在草地生态系统中,土壤微生物中与碳循环、氮循环及压力相关功能基因的相对丰度在不同海拔间差异明显,海拔越高,压力相关的功能基因相对丰度越高[55-56];在森林生态系统中,土壤微生物功能基因的海拔分布模式与物种多样性分布不一致,与碳循环和氮循环相关的功能基因在林线交错带具有更高的多样性,且林线两侧土壤微生物的功能基因多样性存在差异[57-56]。相比于土壤微生物,目前针对水体微生物功能多样性的海拔分布模式鲜有报道。近期,我们通过对亚欧山地溪流生物膜中包括细菌、古菌以及真菌等微生物的研究[53],发现其功能基因多样性在气候因子的调节作用下随着海拔升高而单调下降,并且这一分布模式在挪威、西班牙和中国等多个研究区域得到验证;在该研究的基础上,我们进一步发现在未来的气候模式下,亚欧大陆河流水体微生物的功能基因多样性变化将主要发生在中高纬地区。具体而言,与现状相比,这些地区在2060-2080年期间的功能基因多样性将最高增加30%,功能基因的组成变化将高达35%。
3 基于性状的微生物生态学研究 3.1 研究思路 基于性状的微生物生态学研究思路如图 2所示:物种库(species pool)经过环境过滤后形成局域的微生物群落,基于群落中物种的相对丰度和性状信息计算微生物的功能多样性,进而(1)描述物种和群落水平的性状在环境梯度和空间尺度上的分布;(2)解释物种的空间分布格局和群落构建机制;(3)预测微生物性状对环境变化的响应和生态系统功能的影响[9]。
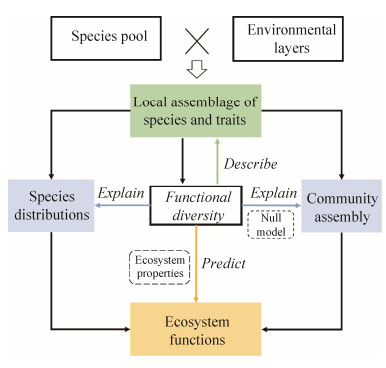 |
| 图 2 基于性状的微生物生态学研究思路 Figure 2 Research ideas of microbial ecology based on traits |
| 图选项 |
3.2 性状与物种分布格局 微生物的表型性状通常难以测量和获取,基因组信息为识别和量化微生物的相关性状提供了便利。由于一些基因型性状在整个生命树中是共有的,因此可以用来理解与生态进化和适应相关的特定策略的权衡[58-59]。具体而言,基因型性状与微生物生活方式[60]、生长速率[61]、细菌表型[62]、细菌生活史策略[63]、海洋细菌的营养策略[64]和土壤细菌的栖息地宽度[65]之间存在关联。
3.2.1 基因组大小: 对于微生物而言,基因组大小是垂直遗传、水平基因转移以及基因复制和丢失的产物[66]。因此,基因组大小至少部分反映了微生物的生活方式和对环境所施加的外部压力的适应性[67]。居住在可变或异质环境中的微生物,如土壤微生物,往往比生活在恒定或稳定环境中的微生物,如生活在宿主体内的寄生虫,拥有更大和更多功能的基因组[68]。此外,较大的基因组更容易受到来自远缘生物水平基因转移的影响,从而形成一些理论假设:基因组大小与环境异质性之间存在相关性,生活在复杂环境中的微生物群落增加了对更大基因组的需求,通过增加对同一环境中相互作用的系统发育距离较远的有机体基因的摄取,进而扩大了自身的基因组[69]。
3.2.2 核糖体基因拷贝数: 核糖体基因拷贝数是与细菌生态策略相关的一系列生活史特征之一[70],它可能在微生物群落构建中发挥重要作用[71]。在资源丰富时,核糖体基因拷贝数与微生物的最大生长速率和响应时间存在联系。在资源匮乏时,拥有多个基因拷贝数意味着竞争成本,这体现了生活史的权衡。此外,核糖体基因拷贝数还可以通过影响细胞化学计量和食物网相互作用而对生态系统功能产生广泛影响[72],因此了解核糖体基因拷贝数的生物地理学对于预测生态系统对环境变化的响应很重要。
3.2.3 转运蛋白: 微生物对盐度或极端pH环境的适应性通常与其细胞表面拥有或表达的转运蛋白的互补有关[73]。在pH梯度下,针对土壤细菌群落功能基因的研究表明,适应较高pH土壤的微生物通常是因为其多种转运蛋白(如ABC转运蛋白)的含量更高,可以直接吸收底物和辅因子[74]。
3.3 性状与群落构建机制 微生物群落是联系微生物生态学与动植物宏观生态学的基础,其结构和构建是微生物生态学领域长期关注的问题[75]。功能性状在群落生态学中正在被越来越多地用于群落形成过程的检验和量化[76]。对细菌群落的研究发现群落的构建是基于功能基因而非物种[75]。在局域群落内,功能性状通过影响物种沿环境梯度的排列、种间竞争以及群落内的资源分配而影响群落的物种共存格局。一方面,具有相似功能性状的物种间具有相似的资源需求,竞争更加激烈,因而在群落中的多度不高;另一方面,具有最适合某类生境功能性状的物种或种群得以在该生境下大量存活繁殖,因而在群落中会具有很高的多度[15]。通过分析功能性状在空间和时间上的分布格局可以验证不同生态和进化驱动因素在时空尺度上对群落构建的重要性[77-78]。进一步研究表明,使用功能性状验证不同的群落构建机制时,应同时将物种的存在与否与多度数据纳入影响因素中[15]。
在有关微生物群落构建机制的研究中,部分研究支持了中性理论[79-80],认为微生物群落结构是由随机过程决定的。当然,也有研究强调生态位和确定性过程的重要性[81]。支持生态位理论的研究,往往集中于验证功能性状在不同群落间存在显著差异以及群落内共存物种间的性状距离比随机模型模拟的性状格局更加发散;支持中性理论的研究,则往往集中于验证群落的实际功能性状格局是否和模拟的由扩散限制引起的空间聚集格局相吻合[15]。本研究组认为,群落构建可能是随机的生态漂变和生态位分化共同作用的过程。当前,更多的研究开始关注整合生态位和中性理论探究随机作用和确定性作用的相对贡献[82-83]。研究人员通过检验由随机过程导致的性状相似性和离散性与实际群落中物种性状分布的差异,即群落构建中的零检验(null model)[84],进而区分生态位理论(确定性过程)和中性理论(随机过程)在驱动微生物群落构建方面的相对重要性。如果由随机过程导致的群落物种性状分布与现实群落中的物种性状分布之间没有明显差异,这表明随机过程是群落构建的决定因素;如果在观察的群落中,性状相似性比随机的预期要高,这表明微生物的性状是由环境选择决定的,即确定性过程是群落构建的决定因素[75, 78]。
3.4 性状与生态系统功能 基于性状方法研究生态系统功能的优势在于特定性状与生态系统功能紧密相关[10]。微生物的大多数代谢性状可以被视为效应性状,因为它们直接影响生态系统中的营养循环过程(例如碳水化合物降解和磷酸盐获取等)和微量气体排放过程(例如甲烷生成和甲烷氧化等)。因此,生态系统中是否存在具有某些特定性状的微生物以及其数量多少,可能影响生态系统的功能和效率[8]。此外,环境因子与性状共同影响着自然条件下生态系统的功能,且性状会受到环境因子的影响,随环境因子的变化而改变[85],因此深入理解其动态变化对精确预测生态系统如何调节和应对环境变化尤为重要。
3.4.1 微生物功能多样性与生态系统功能: 微生物生物多样性和生态系统功能之间的关系(biodiversity and ecosystem functioning,BEF)主要通过测量由标记基因方法衍生出的物种多样性和群落组成的变化进行推断,但是这种推断并不能说明微生物多样性和生态系统功能之间的因果关系[86]。对微生物生物多样性和生态系统功能关系机制的研究需要进一步表征微生物在生态系统中的功能表现和作用[87]。微生物的性状恰恰表征了它们在生态系统中的功能表现,比如对气候条件的适应能力、获取资源的能力以及与群落中其他微生物相互作用的能力等。因此,利用性状的丰度、多样性和分布等信息有助于阐明微生物多样性的潜在机制并且解释和预测生物多样性对生态系统功能的影响[15]。
研究表明,微生物生物多样性在不同生境和空间尺度上与生态系统功能呈正相关关系[88-89]。生物多样性对生态系统功能的积极影响可以归因于两种与生态位相关的机制,即选择效应(selection effects)和互补效应(complementarity effects)[16]。选择效应反映了高竞争性物种对群落整体功能的影响,某些关键性状的存在或者丰富可能会不成比例地影响生态系统功能[90]。互补效应强调具有互补性状的物种的存在,在资源生态位上的差异会导致竞争的减少和群落生态位的增加,使得微生物对限制资源的使用更加有效[86],从而对生态系统功能产生积极影响。此外,当某些物种以有利于其他物种的方式改变环境条件时,还会发生促进效应(facilitation effects)[91],例如根瘤菌与豆科植物的共生,提高了豆科植物的固氮效率。
功能多样性指数可以用来评估生物多样性对生态系统功能的影响。例如,功能相异度(functional divergence)表示群落内性状值的差异性,该指数越高,说明生态位互补程度越强,资源的利用效率提高,生态系统功能增强[37];功能分散度(functional dispersion)被认为是物种相互作用的结果,其量化可以提高对物种相互作用在调节生态系统功能中重要性的认识[9]。由于生态系统功能评估在很大程度上受到功能性状选择和功能多样性指数算法的影响[92],为了更好地获取功能多样性指数,最大的挑战是定义哪些微生物性状对于生态系统功能或特定的生态系统功能而言很重要以及如何测量这些相关性状[86]。此外,人们已经认识到通过更好地描述微生物性状之间的权衡关系有助于定义微生物的生活史策略,同时可以减少需要考虑的性状维度[86]。
总体而言,在基于性状的研究框架下将生态生理研究与当代分子技术相结合可以增强我们将微生物生物多样性与生态系统过程联系起来的能力。需要注意的是,基于性状的研究并非是取代基于分类学的研究,这是因为描述微生物之间性状共存和权衡所需的信息需要建立在分类学的基础上[86]。
3.4.2 微生物性状对环境变化的响应及其对生态系统功能的影响: 由于性状的生物地理格局将生物体的功能和环境联系在一起,理论上可以通过性状的变化预测生物体、群落和生态系统对环境变化的响应[26, 93]。通过关注微生物与环境相互作用的生活史策略,有助于将微生物生态学和生态系统功能联系起来[94]。基于性状的微生物生活史策略可以分为三种:高产Y (high yield),资源获取A (resource acquisition)和胁迫耐受S (stress tolerance)[94]。在资源丰富和环境压力较小的情况下有利于高产策略[95],此时微生物最大限度地提高用于生物合成的资源摄取比例。在资源匮乏的情况下有利于资源获取策略,此时微生物以牺牲产量为代价增加资源获取[94]。当微生物被暴露在极端环境时将具有与胁迫耐受性相关的性状,而以牺牲其他性状为代价。例如,在高酸度或者高盐度的环境中,微生物通过改变细胞膜的结构和组成来维持细胞的完整性和渗透平衡[96]。
基于性状的响应和效应框架(trait-based response-effect framework)可以通过区分群落对环境变化的响应(由响应性状预测)和该变化对生态系统过程的影响(由效应性状预测)来预测环境变化对生态系统功能的影响[26]。研究发现,微生物的响应性状在系统发育上存在保守性[8]。一项研究采用rRNA丰度作为响应性状表征草地土壤微生物对水分突变的短期响应策略[97],发现细菌的响应策略在门水平得到高度保留,且疣微菌和放线菌的响应比变形菌更为迅速。另一项研究采用将微生物类群之间的遗传距离与响应相似性相关联的方式来量化草地叶片凋落微生物对干旱和氮添加的响应策略[25]。结果显示,真菌类群在更精细的分类水平上对干旱和氮添加的响应保守,且对干旱的响应比对氮添加的响应更为保守,这可能因为微生物的抗旱性状(包括特殊的细胞壁结构和渗透调节等)比响应氮添加的性状涉及更多的功能基因[8]。
4 微生物性状研究面临的机遇与挑战 当前,国内外****在微生物性状研究领域已经开展了大量的工作,取得了丰硕的成果。基于性状的研究方法可以在种群、群落和生态系统等不同层次上扩展我们对微生物生态过程的理解,并提供生态模式的机理性解释[76]。当然,这一研究框架在提供机遇的同时仍然面临很多挑战。
4.1 机遇 数据整合是微生物生态学的优势,依赖数据库的开发来存储、维护和共享大量的微生物数据[98-99]。目前,微生物的表型数据和功能基因注释正在被添加到现有的或者新的数据库中,使得性状信息更容易检索和比较[100-103]。蛋白质注释数据库的增长也有助于更精确的功能预测[104]。但是,不同研究之间数据收集和处理方法的差异使得在区域或者全球范围内进行综合分析存在困难[105]。因此,性状数据的进一步整合还需要开发用于收集和处理大量原始数据的标准化方法,从而使不同研究之间的数据收集更加标准化且更具可比性[27]。
4.2 挑战
4.2.1 性状多样性: 由微生物生活方式和代谢方式的多样性导致的性状多样性是性状方法标准化使用面临的挑战。一方面,许多已知的性状可能还具有潜在的适应意义;另一方面,许多具有潜在适应意义的性状可能没有被记录[106]。因此当我们试图对基因序列进行功能注释时,由于部分基因序列相关的蛋白质很少通过实验进行表征,因此它们与任何已经被表征的蛋白质可能都不完全匹配,从而不能通过同源性来预测其功能[107]。此外,性状的多样性导致研究人员在进行性状研究时,通常根据自己的研究兴趣对性状进行选择。
因此,基于性状的微生物生态学研究应试图寻找微生物适应环境变化的一组核心性状,将大量的潜在性状减少为一组数量较少,但始终与微生物生态策略相关的可测量性状,从而使得研究人员集中精力研究,进而解决时间和预算限制等问题[27]。与此同时,考虑到部分功能多样性指数对性状数量变化表现出较高的敏感性[49],因此在适当减少性状的同时,还应考虑性状数量对功能多样性指数的影响。
4.2.2 基因型与表型和环境的匹配: 由于表型性状具有较高的复杂性,通常涉及到基因组多个功能基因的相互作用,因此很难通过是否存在特定的功能基因推测微生物的功能潜力,或者仅通过基因组信息获知其代谢途径[8]。例如,氨单加氧酶基因(amoA)与甲烷单加氧酶基因(pmoA)同源,因此amoA和pmoA基因的共存表明微生物可能进行甲烷氧化、氨氧化或者同时进行这两种生物化学过程[108]。此外,功能性状需要在可培养的微生物中进行验证,但是由于绝大多数微生物的不可培养性,使得基于基因组信息推测的功能性状可能被错误地注释[109]。
在自然环境下可以对植物性状进行原位测量,但是对微生物却难以适用[8]。存放在公共数据库中的微生物基因组信息通常缺少自然条件下对应的表型数据和环境数据[110],从而给这些数据与基因组信息的整合和下游分析带来困难。因此,我们需要进一步整合宏基因组学、实验培养和实地测量技术,将微生物分类、系统发育和功能基因等信息与测定的表型性状和环境偏好联系起来[73]。
5 展望 关于微生物个体的研究表明,微生物的个体性(如细胞间的表型变异性)可能是驱动微生物适应和进化的重要因素[111]。因此,为了进一步了解微生物个体在自然环境中的潜在重要性,我们建议对微生物个体在自然环境中的表型变异进行分析,并研究这种变异的功能潜力以及微生物类群能否从中获益。与此同时,新的微生物生理学研究技术,如蛋白质非标记定量技术(label-free approaches)、稳定同位素探针技术(stable isotope probing)和底物类似物探针技术(substrate analogue probing)等能够无损伤地测量从自然环境中获得的单个微生物细胞的基因表达和代谢活动等信息[112],为微生物个体层次的研究提供了客观条件。
基于性状的研究还应促进群落生态学和生态系统生态学之间的融合[76]。为了更好地搭建性状研究与宏观生态学研究的桥梁,需要从更高层次提出新的概念,如生态系统性状(ecosystem trait),即以单位土地面积为基础,在群落尺度能体现生物对环境适应性和生产力优化的性状,由植物群落性状、动物群落性状和微生物群落性状等组成[113],借此推动传统性状研究与宏观生态学研究的结合。
References
| [1] | Huang L, Feng XL, Du QS, Dong XZ, Liu SJ, Wen MZ, Dai X. Focusing on key scientific issues of microbiome research in hydrosphere:NSFC major research plan for microbes in hydrosphere. Bulletin of Chinese Academy of Sciences, 2017, 32(3): 266-272. (in Chinese) 黄力, 冯雪莲, 杜全生, 东秀珠, 刘双江, 温明章, 戴欣. 水圈微生物重大研究计划:聚焦水圈微生物组研究的核心科学问题. 中国科学院院刊, 2017, 32(3): 266-272. |
| [2] | Liu SJ, Shi WY, Zhao GP. China microbiome initiative:opportunity and challenges. Bulletin of Chinese Academy of Sciences, 2017, 32(3): 241-250. (in Chinese) 刘双江, 施文元, 赵国屏. 中国微生物组计划:机遇与挑战. 中国科学院院刊, 2017, 32(3): 241-250. |
| [3] | Chen SC, Sun GX, Yan Y, Konstantinidis KT, Zhang SY, Deng Y, Li XM, Cui HL, Musat F, Popp D, Rosen BP, Zhu YG. The Great Oxidation Event expanded the genetic repertoire of arsenic metabolism and cycling. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(19): 10414-10421. DOI:10.1073/pnas.2001063117 |
| [4] | Wu QL, Jiang HL. China lake microbiome project. Bulletin of Chinese Academy of Sciences, 2017, 32(3): 273-279. (in Chinese) 吴庆龙, 江和龙. 中国湖泊微生物组研究. 中国科学院院刊, 2017, 32(3): 273-279. |
| [5] | Louca S, Jacques SMS, Pires APF, Leal JS, Srivastava DS, Parfrey LW, Farjalla VF, Doebeli M. High taxonomic variability despite stable functional structure across microbial communities. Nature Ecology & Evolution, 2016, 1(1): 0015. |
| [6] | Louca S, Parfrey LW, Doebeli M. Decoupling function and taxonomy in the global ocean microbiome. Science, 2016, 353(6305): 1272-1277. DOI:10.1126/science.aaf4507 |
| [7] | Green JL, Bohannan BJM, Whitaker RJ. Microbial biogeography:from taxonomy to traits. Science, 2008, 320(5879): 1039-1043. DOI:10.1126/science.1153475 |
| [8] | Martiny JBH, Jones SE, Lennon JT, Martiny AC. Microbiomes in light of traits:a phylogenetic perspective. Science, 2015, 350(6261): aac9323. DOI:10.1126/science.aac9323 |
| [9] | Violle C, Reich PB, Pacala SW, Enquist BJ, Kattge J. The emergence and promise of functional biogeography. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(38): 13690-13696. DOI:10.1073/pnas.1415442111 |
| [10] | Violle C, Navas ML, Vile D, Kazakou E, Fortunel C, Hummel I, Garnier E. Let the concept of trait be functional!. Oikos, 2007, 116(5): 882-892. DOI:10.1111/j.0030-1299.2007.15559.x |
| [11] | Stewart EJ. Growing unculturable bacteria. Journal of Bacteriology, 2012, 194(16): 4151-4160. DOI:10.1128/JB.00345-12 |
| [12] | Bahram M, Hildebrand F, Forslund SK, Anderson JL, Soudzilovskaia NA, Bodegom PM, Bengtsson-Palme J, Anslan S, Coelho LP, Harend H, Huerta-Cepas J, Medema MH, Maltz MR, Mundra S, Olsson PA, Pent M, P?lme S, Sunagawa S, Ryberg M, Tedersoo L, Bork P. Structure and function of the global topsoil microbiome. Nature, 2018, 560(7717): 233-237. DOI:10.1038/s41586-018-0386-6 |
| [13] | Antwis RE, Griffiths SM, Harrison XA, Aranega-Bou P, Arce A, Bettridge AS, Brailsford FL, de Menezes A, Devaynes A, Forbes KM, Fry EL, Goodhead I, Haskell E, Heys C, James C, Johnston SR, Lewis GR, Lewis Z, Macey MC, McCarthy A, McDonald JE, Mejia-Florez NL, O'Brien D, Orland C, Pautasso M, Reid WDK, Robinson HA, Wilson K, Sutherland WJ. Fifty important research questions in microbial ecology. FEMS Microbiology Ecology, 2017, 93(5): fix044. |
| [14] | Sutherland WJ, Freckleton RP, Godfray HCJ, Beissinger SR, Benton T, Cameron DD, Carmel Y, Coomes DA, Coulson T, Emmerson MC, Hails RS, Hays GC, Hodgson DJ, Hutchings MJ, Johnson D, Jones JPG, Keeling MJ, Kokko H, Kunin WE, Lambin X, Lewis OT, Malhi Y, Mieszkowska N, Milner-Gulland EJ, Norris K, Phillimore AB, Purves DW, Reid JM, Reuman DC, Thompson K, Travis JMJ, Turnbull LA, Wardle DA, Wiegand T. Identification of 100 fundamental ecological questions. Journal of Ecology, 2013, 101(1): 58-67. |
| [15] | Liu XJ, Ma KP. Plant functional traits-concepts, applications and future directions. Scientia Sinica Vitae, 2015, 45(4): 325-339. (in Chinese) 刘晓娟, 马克平. 植物功能性状研究进展. 中国科学:生命科学, 2015, 45(4): 325-339. |
| [16] | Escalas A, Hale L, Voordeckers JW, Yang YF, Firestone MK, Alvarez-Cohen L, Zhou JZ. Microbial functional diversity:from concepts to applications. Ecology and Evolution, 2019, 9(20): 12000-12016. DOI:10.1002/ece3.5670 |
| [17] | Barberán A, Fernández-Guerra A, Bohannan BJM, Casamayor EO. Exploration of community traits as ecological markers in microbial metagenomes. Molecular Ecology, 2012, 21(8): 1909-1917. DOI:10.1111/j.1365-294X.2011.05383.x |
| [18] | Fierer N, Barberán A, Laughlin DC. Seeing the forest for the genes:using metagenomics to infer the aggregated traits of microbial communities. Frontiers in Microbiology, 2014, 5: 614. |
| [19] | Boon E, Meehan CJ, Whidden C, Wong DHJ, Langille MGI, Beiko RG. Interactions in the microbiome:communities of organisms and communities of genes. FEMS Microbiology Reviews, 2014, 38(1): 90-118. DOI:10.1111/1574-6976.12035 |
| [20] | Huang QY, Briggs BR, Dong HL, Jiang HC, Wu G, Edwardson C, De Vlaminck I, Quake S. Taxonomic and functional diversity provides insight into microbial pathways and stress responses in the saline Qinghai Lake, China. PLoS One, 2014, 9(11): e111681. DOI:10.1371/journal.pone.0111681 |
| [21] | Powell JR, Welsh A, Hallin S. Microbial functional diversity enhances predictive models linking environmental parameters to ecosystem properties. Ecology, 2015, 96(7): 1985-1993. DOI:10.1890/14-1127.1 |
| [22] | Young JPW. Bacteria are smartphones and mobile genes are apps. Trends in Microbiology, 2016, 24(12): 931-932. DOI:10.1016/j.tim.2016.09.002 |
| [23] | Naeem S, Wright JP. Disentangling biodiversity effects on ecosystem functioning:deriving solutions to a seemingly insurmountable problem. Ecology Letters, 2003, 6(6): 567-579. |
| [24] | Allison SD, Martiny JBH. Resistance, resilience, and redundancy in microbial communities. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(S1): 11512-11519. |
| [25] | Amend AS, Martiny AC, Allison SD, Berlemont R, Goulden ML, Lu Y, Treseder KK, Weihe C, Martiny JBH. Microbial response to simulated global change is phylogenetically conserved and linked with functional potential. The ISME Journal, 2016, 10(1): 109-118. |
| [26] | Suding KN, Lavorel S, Chapin III FS, Cornelissen JHC, Díaz S, Garnier E, Goldberg D, Hooper DU, Jackson ST, Navas ML. Scaling environmental change through the community-level:a trait-based response-and-effect framework for plants. Global Change Biology, 2008, 14(5): 1125-1140. DOI:10.1111/j.1365-2486.2008.01557.x |
| [27] | Lajoie G, Kembel SW. Making the most of trait-based approaches for microbial ecology. Trends in Microbiology, 2019, 27(10): 814-823. DOI:10.1016/j.tim.2019.06.003 |
| [28] | Buchanan RET, Gibbons NE, Bergey. Bergey's manual of determinative bacteriology. Taxon, 1975, 24(2/3): 377-378. DOI:10.2307/1218353 |
| [29] | Nielsen JL, Nielsen PH. Advances in microscopy:microautoradiography of single cells. Methods in Enzymology, 2005, 397: 237-256. DOI:10.1016/S0076-6879(05)97014-6 |
| [30] | Maron PA, Ranjard L, Mougel C, Lemanceau P. Metaproteomics:a new approach for studying functional microbial ecology. Microbial Ecology, 2007, 53(3): 486-493. DOI:10.1007/s00248-006-9196-8 |
| [31] | Hungate BA, Mau RL, Schwartz E, Caporaso JG, Dijkstra P, van Gestel N, Koch BJ, Liu CM, McHugh TA, Marks JC, Morrissey EM, Price LB. Quantitative microbial ecology through stable isotope probing. Applied and Environmental Microbiology, 2015, 81(21): 7570-7581. DOI:10.1128/AEM.02280-15 |
| [32] | Langille MGI, Zaneveld J, Caporaso JG, McDonald D, Knights D, Reyes JA, Clemente JC, Burkepile DE, Thurber RLV, Knight R, Beiko RG, Huttenhower C. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nature Biotechnology, 2013, 31(9): 814-821. DOI:10.1038/nbt.2676 |
| [33] | A?hauer KP, Wemheuer B, Daniel R, Meinicke P. Tax4Fun:predicting functional profiles from metagenomic 16S rRNA data. Bioinformatics, 2015, 31(17): 2882-2884. DOI:10.1093/bioinformatics/btv287 |
| [34] | Nagpal S, Haque MM, Mande SS. Vikodak-a modular framework for inferring functional potential of microbial communities from 16S metagenomic datasets. PLoS One, 2016, 11(2): e0148347. DOI:10.1371/journal.pone.0148347 |
| [35] | Alneberg J, Bjarnason BS, De Bruijn I, Schirmer M, Quick J, Ijaz UZ, Lahti L, Loman NJ, Andersson AF, Quince C. Binning metagenomic contigs by coverage and composition. Nature Methods, 2014, 11(11): 1144-1146. DOI:10.1038/nmeth.3103 |
| [36] | Carvalhais LC, Dennis PG, Tyson GW, Schenk PM. Application of metatranscriptomics to soil environments. Journal of Microbiological Methods, 2012, 91(2): 246-251. DOI:10.1016/j.mimet.2012.08.011 |
| [37] | D??az S, Cabido M. Vive la différence:plant functional diversity matters to ecosystem processes. Trends in Ecology & Evolution, 2001, 16(11): 646-655. |
| [38] | Tilman D. Functional diversity//Levin SA. Encyclopedia of Biodiversity. Elsevier: Academic Press, 2001: 109-120. |
| [39] | Petchey OL, Gaston KJ. Functional Diversity (FD), species richness and community composition. Ecology Letters, 2002, 5(3): 402-411. DOI:10.1046/j.1461-0248.2002.00339.x |
| [40] | Troussellier M, Legendre P. A functional evenness index for microbial ecology. Microbial Ecology, 1981, 7(4): 283-296. DOI:10.1007/BF02341423 |
| [41] | Lamanna C, Blonder B, Violle C, Kraft NJB, Sandel B, ?ímová I, Donoghue II JC, Svenning JC, McGill BJ, Boyle B, Buzzard V, Dolins S, J?rgensen PM, Marcuse-Kubitza A, Morueta-Holme N, Peet RK, Piel WH, Regetz J, Schildhauer M, Spencer N, Thiers B, Wiser SK, Enquist BJ. Functional trait space and the latitudinal diversity gradient. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(38): 13745-13750. DOI:10.1073/pnas.1317722111 |
| [42] | Louca S, Polz MF, Mazel F, Albright MBN, Huber JA, O'Connor MI, Ackermann M, Hahn AS, Srivastava DS, Crowe SA, Doebeli M, Parfrey LW. Function and functional redundancy in microbial systems. Nature Ecology & Evolution, 2018, 2(6): 936-943. |
| [43] | Villéger S, Mason NWH, Mouillot D. New multidimensional functional diversity indices for a multifaceted framework in functional ecology. Ecology, 2008, 89(8): 2290-2301. DOI:10.1890/07-1206.1 |
| [44] | Laliberté E, Legendre P. A distance-based framework for measuring functional diversity from multiple traits. Ecology, 2010, 91(1): 299-305. |
| [45] | Walker B, Kinzig A, Langridge J. Plant attribute diversity, resilience, and ecosystem function:the nature and significance of dominant and minor species. Ecosystems, 1999, 2(2): 95-113. DOI:10.1007/s100219900062 |
| [46] | Botta-Dukát Z. Rao's quadratic entropy as a measure of functional diversity based on multiple traits. Journal of Vegetation Science, 2005, 16(5): 533-540. DOI:10.1111/j.1654-1103.2005.tb02393.x |
| [47] | Lavorel S, Grigulis K, McIntyre S, Williams NSG, Garden D, Dorrough J, Berman S, Quétier F, Thébault A, Bonis A. Assessing functional diversity in the field-methodology matters!. Functional Ecology, 2008, 22(1): 134-147. |
| [48] | Maire E, Grenouillet G, Brosse S, Villéger S. How many dimensions are needed to accurately assess functional diversity? A pragmatic approach for assessing the quality of functional spaces. Global Ecology and Biogeography, 2015, 24(6): 728-740. DOI:10.1111/geb.12299 |
| [49] | Legras G, Loiseau N, Gaertner JC, Poggiale JC, Gaertner-Mazouni N. Assessing functional diversity:the influence of the number of the functional traits. Theoretical Ecology, 2020, 13(1): 117-126. DOI:10.1007/s12080-019-00433-x |
| [50] | Faith DP. Conservation evaluation and phylogenetic diversity. Biological Conservation, 1992, 61(1): 1-10. DOI:10.1016/0006-3207(92)91201-3 |
| [51] | Webb CO. Exploring the phylogenetic structure of ecological communities:an example for rain forest trees. The American Naturalist, 2000, 156(2): 145-155. DOI:10.1086/303378 |
| [52] | Webb CO, Ackerly DD, McPeek MA, Donoghue MJ. Phylogenies and community ecology. Annual Review of Ecology and Systematics, 2002, 33: 475-505. DOI:10.1146/annurev.ecolsys.33.010802.150448 |
| [53] | Picazo F, Vilmi A, Aalto J, Soininen J, Casamayor EO, Liu YQ, Wu QL, Ren LJ, Zhou JZ, Shen J, Wang JJ. Climate mediates continental scale patterns of stream microbial functional diversity. Microbiome, 2020, 8(1): 92. DOI:10.1186/s40168-020-00873-2 |
| [54] | Wang Y, Zong N, He NP, Zhang JJ, Tian J, Li LT. Soil microbial functional diversity patterns and drivers along an elevation gradient on Qinghai-Tibet, China. Acta Ecologica Sinica, 2018, 38(16): 5837-5845. (in Chinese) 王颖, 宗宁, 何念鹏, 张晋京, 田静, 李良涛. 青藏高原高寒草甸不同海拔梯度下土壤微生物群落碳代谢多样性. 生态学报, 2018, 38(16): 5837-5845. |
| [55] | Yang YF, Gao Y, Wang SP, Xu DP, Yu H, Wu LW, Lin QY, Hu YG, Li XZ, He ZL, Deng Y, Zhou JZ. The microbial gene diversity along an elevation gradient of the Tibetan grassland. The ISME Journal, 2014, 8(2): 430-440. DOI:10.1038/ismej.2013.146 |
| [56] | Chu HY, Feng MM, Liu X, Shi Y, Yang T, Gao GF. Soil microbial biogeography:recent advances in China and research frontiers in the world. Acta Pedologica Sinica, 2020, 57(3): 515-529. (in Chinese) 褚海燕, 冯毛毛, 柳旭, 时玉, 杨腾, 高贵锋. 土壤微生物生物地理学:国内进展与国际前沿. 土壤学报, 2020, 57(3): 515-529. |
| [57] | Shen CC, Shi Y, Ni YY, Deng Y, Van Nostrand JD, He ZL, Zhou JZ, Chu HY. Dramatic increases of soil microbial functional gene diversity at the treeline ecotone of Changbai Mountain. Frontiers in Microbiology, 2016, 7: 1184. |
| [58] | Gudelj I, Weitz JS, Ferenci T, Horner-Devine MC, Marx CJ, Meyer JR, Forde SE. An integrative approach to understanding microbial diversity:from intracellular mechanisms to community structure. Ecology Letters, 2010, 13(9): 1073-1084. DOI:10.1111/j.1461-0248.2010.01507.x |
| [59] | Verberk WCEP, van Noordwijk CGE, Hildrew AG. Delivering on a promise:integrating species traits to transform descriptive community ecology into a predictive science. Freshwater Science, 2013, 32(2): 531-547. DOI:10.1899/12-092.1 |
| [60] | Garcia JAL, Bartumeus F, Roche D, Giraldo J, Stanley HE, Casamayor EO. Ecophysiological significance of scale-dependent patterns in prokaryotic genomes unveiled by a combination of statistic and genometric analyses. Genomics, 2008, 91(6): 538-543. DOI:10.1016/j.ygeno.2008.03.001 |
| [61] | Vieira-Silva S, Rocha EPC. The systemic imprint of growth and its uses in ecological (meta)genomics. PLoS Genetics, 2010, 6(1): e1000808. DOI:10.1371/journal.pgen.1000808 |
| [62] | Weimann A, Mooren K, Frank J, Pope PB, Bremges A, McHardy AC. From genomes to phenotypes:traitar, the microbial trait analyzer. mSystems, 2016, 1(6): e00101-16. |
| [63] | Livermore JA, Emrich SJ, Tan J, Jones SE. Freshwater bacterial lifestyles inferred from comparative genomics. Environmental Microbiology, 2014, 16(3): 746-758. DOI:10.1111/1462-2920.12199 |
| [64] | Lauro FM, McDougald D, Thomas T, Williams TJ, Egan S, Rice S, DeMaere MZ, Ting L, Ertan H, Johnson J, Ferriera S, Lapidus A, Anderson I, Kyrpides N, Munk AC, Detter C, Han CS, Brown MV, Robb FT, Kjelleberg S, Cavicchioli R. The genomic basis of trophic strategy in marine bacteria. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(37): 15527-15533. DOI:10.1073/pnas.0903507106 |
| [65] | Barberán A, Ramirez KS, Leff JW, Bradford MA, Wall DH, Fierer N. Why are some microbes more ubiquitous than others? Predicting the habitat breadth of soil bacteria. Ecology Letters, 2014, 17(7): 794-802. DOI:10.1111/ele.12282 |
| [66] | Koonin EV, Wolf YI. Genomics of bacteria and archaea:the emerging dynamic view of the prokaryotic world. Nucleic Acids Research, 2008, 36(21): 6688-6719. DOI:10.1093/nar/gkn668 |
| [67] | Konstantinidis KT, Tiedje JM. Trends between gene content and genome size in prokaryotic species with larger genomes. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(9): 3160-3165. DOI:10.1073/pnas.0308653100 |
| [68] | Guieysse B, Wuertz S. Metabolically versatile large-genome prokaryotes. Current Opinion in Biotechnology, 2012, 23(3): 467-473. DOI:10.1016/j.copbio.2011.12.022 |
| [69] | Cordero OX, Hogeweg P. The impact of long-distance horizontal gene transfer on prokaryotic genome size. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(51): 21748-21753. DOI:10.1073/pnas.0907584106 |
| [70] | Fierer N, Bradford MA, Jackson RB. Toward an ecological classification of soil bacteria. Ecology, 2007, 88(6): 1354-1364. DOI:10.1890/05-1839 |
| [71] | Stevenson BS, Schmidt TM. Life history implications of rRNA gene copy number in Escherichia coli. Applied and Environmental Microbiology, 2004, 70(11): 6670-6677. DOI:10.1128/AEM.70.11.6670-6677.2004 |
| [72] | Weider LJ, Elser JJ, Crease TJ, Mateos M, Cotner JB, Markow TA. The functional significance of ribosomal (r) DNA variation:impacts on the evolutionary ecology of organisms. Annual Review of Ecology, Evolution, and Systematics, 2005, 36: 219-242. DOI:10.1146/annurev.ecolsys.36.102003.152620 |
| [73] | Barberán A, Velazquez HC, Jones S, Fierer N. Hiding in plain sight:mining bacterial species records for phenotypic trait information. mSphere, 2017, 2(4): e00237-17. |
| [74] | Malik AA, Thomson BC, Whiteley AS, Bailey M, Griffiths RI. Bacterial physiological adaptations to contrasting edaphic conditions identified using landscape scale metagenomics. mBio, 2017, 8(4): e00799-17. |
| [75] | Burke C, Steinberg P, Rusch D, Kjelleberg S, Thomas T. Bacterial community assembly based on functional genes rather than species. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(34): 14288-14293. DOI:10.1073/pnas.1101591108 |
| [76] | McGill BJ, Enquist BJ, Weiher E, Westoby M. Rebuilding community ecology from functional traits. Trends in Ecology & Evolution, 2006, 21(4): 178-185. |
| [77] | Severin I, ?stman ?, Lindstr?m ES. Variable effects of dispersal on productivity of bacterial communities due to changes in functional trait composition. PLoS One, 2013, 8(12): e80825. DOI:10.1371/journal.pone.0080825 |
| [78] | Staley C, Gould TJ, Wang P, Phillips J, Cotner JB, Sadowsky MJ. Core functional traits of bacterial communities in the Upper Mississippi River show limited variation in response to land cover. Frontiers in Microbiology, 2014, 5: 414. |
| [79] | Sloan WT, Lunn M, Woodcock S, Head IM, Nee S, Curtis TP. Quantifying the roles of immigration and chance in shaping prokaryote community structure. Environmental Microbiology, 2006, 8(4): 732-740. DOI:10.1111/j.1462-2920.2005.00956.x |
| [80] | Woodcock S, van der Gast CJ, Bell T, Lunn M, Curtis TP, Head IM, Sloan WT. Neutral assembly of bacterial communities. FEMS Microbiology Ecology, 2007, 62(2): 171-180. DOI:10.1111/j.1574-6941.2007.00379.x |
| [81] | Dumbrell AJ, Nelson M, Helgason T, Dytham C, Fitter AH. Relative roles of niche and neutral processes in structuring a soil microbial community. The ISME Journal, 2010, 4(3): 337-345. DOI:10.1038/ismej.2009.122 |
| [82] | Cao P, He JZ. A preliminary theoretical framework of microbial ecology. Acta Ecologica Sinica, 2015, 35(22): 7263-7273. (in Chinese) 曹鹏, 贺纪正. 微生物生态学理论框架. 生态学报, 2015, 35(22): 7263-7273. |
| [83] | He JZ, Ge Y. Recent advances in soil microbial biogeography. Acta Ecologica Sinica, 2008, 28(11): 5571-5582. (in Chinese) 贺纪正, 葛源. 土壤微生物生物地理学研究进展. 生态学报, 2008, 28(11): 5571-5582. |
| [84] | Harvey PH, Colwell RK, Silvertown JW, May RM. Null models in ecology. Annual Review of Ecology and Systematics, 1983, 14: 189-211. DOI:10.1146/annurev.es.14.110183.001201 |
| [85] | Lei LJ, Kong DL, Li XM, Zhou ZX, Li GY. Plant functional traits, functional diversity, and ecosystem functioning:current knowledge and perspectives. Biodiversity Science, 2016, 24(8): 922-931. (in Chinese) 雷羚洁, 孔德良, 李晓明, 周振兴, 李国勇. 植物功能性状、功能多样性与生态系统功能:进展与展望. 生物多样性, 2016, 24(8): 922-931. |
| [86] | Krause S, Le Roux X, Niklaus PA, van Bodegom PM, Lennon JT, Bertilsson S, Grossart HP, Philippot L, Bodelier PLE. Trait-based approaches for understanding microbial biodiversity and ecosystem functioning. Frontiers in Microbiology, 2014, 5: 251. |
| [87] | Wang JT, Egidi E, Li J, Singh BK. Linking microbial diversity with ecosystem functioning through a trait framework. Journal of Biosciences, 2019, 44(5): 109. DOI:10.1007/s12038-019-9928-9 |
| [88] | Delgado-Baquerizo M, Maestre FT, Reich PB, Jeffries TC, Gaitan JJ, Encinar D, Berdugo M, Campbell CD, Singh BK. Microbial diversity drives multifunctionality in terrestrial ecosystems. Nature Communications, 2016, 7(1): 10541. DOI:10.1038/ncomms10541 |
| [89] | Jing X, Sanders NJ, Shi Y, Chu HY, Classen AT, Zhao K, Chen LT, Shi Y, Jiang YX, He JS. The links between ecosystem multifunctionality and above-and belowground biodiversity are mediated by climate. Nature Communications, 2015, 6(1): 8159. DOI:10.1038/ncomms9159 |
| [90] | Hector A, Schmid B, Beierkuhnlein C, Caldeira MC, Diemer M, Dimitrakopoulos PG, Finn JA, Freitas H, Giller PS, Good J, Harris R, H?gberg P, Huss-Dane K, Joshi J, Jumpponen A, K?rner C, Leadley PW, Loreau M, Minns A, Mulder CPH, O'Donovan G, Otway SJ, Pereira JS, Prinz A, Read DJ, Scherer-Lorenzen M, Schulze ED, Siamantziouras ASD, Spehn EM, Terry AC, Troumbis AY, Woodward FI, Yachi S, Lawton JH. Plant diversity and productivity experiments in European grasslands. Science, 1999, 286(5442): 1123-1127. DOI:10.1126/science.286.5442.1123 |
| [91] | Bruno JF, Stachowicz JJ, Bertness MD. Inclusion of facilitation into ecological theory. Trends in Ecology & Evolution, 2003, 18(3): 119-125. |
| [92] | Byrnes JEK, Gamfeldt L, Isbell F, Lefcheck JS, Griffin JN, Hector A, Cardinale BJ, Hooper DU, Dee LE, Emmett Duffy J. Investigating the relationship between biodiversity and ecosystem multifunctionality:challenges and solutions. Methods in Ecology and Evolution, 2014, 5(2): 111-124. DOI:10.1111/2041-210X.12143 |
| [93] | Lavorel S, Garnier E. Predicting changes in community composition and ecosystem functioning from plant traits:revisiting the Holy Grail. Functional Ecology, 2002, 16(5): 545-556. DOI:10.1046/j.1365-2435.2002.00664.x |
| [94] | Malik AA, Martiny JBH, Brodie EL, Martiny AC, Treseder KK, Allison SD. Defining trait-based microbial strategies with consequences for soil carbon cycling under climate change. The ISME Journal, 2019, 14(1): 1-9. |
| [95] | Lipson DA. The complex relationship between microbial growth rate and yield and its implications for ecosystem processes. Frontiers in Microbiology, 2015, 6: 615. |
| [96] | Wood JM. Bacterial responses to osmotic challenges. The Journal of General Physiology, 2015, 145(5): 381-388. DOI:10.1085/jgp.201411296 |
| [97] | Placella SA, Brodie EL, Firestone MK. Rainfall-induced carbon dioxide pulses result from sequential resuscitation of phylogenetically clustered microbial groups. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(27): 10931-10936. DOI:10.1073/pnas.1204306109 |
| [98] | Nilsson RH, Larsson KH, Taylor AFS, Bengtsson-Palme J, Jeppesen TS, Schigel D, Kennedy P, Picard K, Gl?ckner FO, Tedersoo L, Saar I, K?ljalg U, Abarenkov K. The UNITE database for molecular identification of fungi:handling dark taxa and parallel taxonomic classifications. Nucleic Acids Research, 2019, 47(D1): D259-D264. DOI:10.1093/nar/gky1022 |
| [99] | Zhulin IB. Databases for microbiologists. Journal of Bacteriology, 2015, 197(15): 2458-2467. DOI:10.1128/JB.00330-15 |
| [100] | Basenko EY, Pulman JA, Shanmugasundram A, Harb OS, Crouch K, Starns D, Warrenfeltz S, Aurrecoechea C, Stoeckert CJ, Kissinger JC, Roos DS, Hertz-Fowler C. FungiDB:an integrated bioinformatic resource for fungi and oomycetes. Journal of Fungi, 2018, 4(1): 39. |
| [101] | Chen IMA, Markowitz VM, Chu K, Palaniappan K, Szeto E, Pillay M, Ratner A, Huang JH, Andersen E, Huntemann M, Varghese N, Hadjithomas M, Tennessen K, Nielsen T, Ivanova NN, Kyrpides NC. IMG/M:integrated genome and metagenome comparative data analysis system. Nucleic Acids Research, 2017, 45(D1): D507-D516. DOI:10.1093/nar/gkw929 |
| [102] | Nguyen NH, Song ZW, Bates ST, Branco S, Tedersoo L, Menke J, Schilling JS, Kennedy PG. FUNGuild:an open annotation tool for parsing fungal community datasets by ecological guild. Fungal Ecology, 2016, 20: 241-248. DOI:10.1016/j.funeco.2015.06.006 |
| [103] | Reimer LC, Vetcininova A, Carbasse JS, S?hngen C, Gleim D, Ebeling C, Overmann J. BacDive in 2019:bacterial phenotypic data for High-throughput biodiversity analysis. Nucleic Acids Research, 2019, 47(D1): D631-D636. DOI:10.1093/nar/gky879 |
| [104] | Apweiler R, Bairoch A, Wu CH, Barker WC, Boeckmann B, Ferro S, Gasteiger E, Huang HZ, Lopez R, Magrane M, Martin MJ, Natale DA, O'Donovan C, Redaschi N, Yeh LSL. UniProt:the universal protein knowledgebase. Nucleic Acids Research, 2004, 32(S1): D115-D119. |
| [105] | Chu HY, Gao GF, Ma YY, Fan KK, Delgado-Baquerizo M. Soil microbial biogeography in a changing world:recent advances and future perspectives. mSystems, 2020, 5(2): e00803-19. |
| [106] | Sberro H, Fremin BJ, Zlitni S, Edfors F, Greenfield N, Snyder MP, Pavlopoulos GA, Kyrpides NC, Bhatt AS. Large-scale analyses of human microbiomes reveal thousands of small, novel genes. Cell, 2019, 178(5): 1245-1259. DOI:10.1016/j.cell.2019.07.016 |
| [107] | Price MN, Wetmore KM, Waters RJ, Callaghan M, Ray J, Liu HL, Kuehl JV, Melnyk RA, Lamson JS, Suh Y, Carlson HK, Esquivel Z, Sadeeshkumar H, Chakraborty R, Zane GM, Rubin BE, Wall JD, Visel A, Bristow J, Blow MJ, Arkin AP, Deutschbauer AM. Mutant phenotypes for thousands of bacterial genes of unknown function. Nature, 2018, 557(7706): 503-509. DOI:10.1038/s41586-018-0124-0 |
| [108] | Arp DJ, Stein LY. Metabolism of inorganic N compounds by ammonia-oxidizing bacteria. Critical Reviews in Biochemistry and Molecular Biology, 2003, 38(6): 471-495. DOI:10.1080/10409230390267446 |
| [109] | Schnoes AM, Brown SD, Dodevski I, Babbitt PC. Annotation error in public databases:misannotation of molecular function in enzyme superfamilies. PLoS Computational Biology, 2009, 5(12): e1000605. DOI:10.1371/journal.pcbi.1000605 |
| [110] | Konstantinidis KT, Ramette A, Tiedje JM. The bacterial species definition in the genomic era. Philosophical Transactions of the Royal Society B:Biological Sciences, 2006, 361(1475): 1929-1940. DOI:10.1098/rstb.2006.1920 |
| [111] | Ackermann M. Microbial individuality in the natural environment. The ISME Journal, 2013, 7(3): 465-467. DOI:10.1038/ismej.2012.131 |
| [112] | Hatzenpichler R, Krukenberg V, Spietz RL, Jay ZJ. Next-generation physiology approaches to study microbiome function at single cell level. Nature Reviews Microbiology, 2020, 18(4): 241-256. DOI:10.1038/s41579-020-0323-1 |
| [113] | He NP, Liu CC, Piao SL, Sack L, Xu L, Luo YQ, He JS, Han XG, Zhou GS, Zhou XH, Lin Y, Yu Q, Liu SR, Sun W, Niu SL, Li SG, Zhang JH, Yu GR. Ecosystem traits linking functional traits to macroecology. Trends in Ecology & Evolution, 2019, 34(3): 200-210. |
