冷东瑾1,2, 胡晓婧1,2, 李兴1,2, 欧一新1,2, 康前进1,2

 , 白林泉1,2, 邓子新1,2
, 白林泉1,2, 邓子新1,2 1. 上海交通大学生命科学技术学院, 微生物代谢国家重点实验室, 上海 200240;
2. 上海交通大学代谢与发育国际联合合作实验室, 上海 200240
收稿日期:2019-08-06;修回日期:2019-09-17;网络出版日期:2019-11-06
基金项目:国家自然科学基金(31770034,31700027);上海交通大学新进青年教师启动计划(17X100040064)
*通信作者:康前进。Tel:+86-21-34206119;E-mail:qjkang@sjtu.edu.cn.
摘要:[目的] 采用特征次级代谢产物生物合成的保守功能基因探针,定向分离土壤中产生特征次级代谢产物的菌种资源,借助基因转录分析为导向的培养基优化方法,获得目标次生代谢产物。[方法] 首先,根据5种特征次级代谢产物保守的合成功能基因设计简并引物,定向从土壤样品中筛选、分离并纯化菌株。然后,以RT-qPCR为指导开展目标产物的发酵培养基优化;最后,对菌株进行发酵,利用多种色谱技术分离纯化目标天然产物,并结合高分辨质谱与核磁共振等技术对所获得的化合物进行结构鉴定。[结果] 从土壤中筛选得到了一株AHBA合酶基因和环氧化酶基因均为阳性的链霉菌菌株(编号为CQ01819),根据转录分析优化发酵培养基,最终从该菌株分离纯化得到了含有AHBA结构单元的丝裂霉素C、聚醚类抗生素莫能霉素A和缬吲霉素。[结论] 本研究通过菌株的定向分离纯化,筛选得到了产生预期抗生素的浅紫灰链霉菌菌株CQ01819;基于RT-qPCR指导的发酵培养基优化,确定了菌株的发酵条件;获得发酵粗提物后,采用多种色谱整合技术和光谱分析策略,快速分离并鉴定了目标产物。该研究为目标菌种资源的定向筛选、菌株的发酵条件的快速优化和化合物的定向分离提供了较好的参考。
关键词:菌株定向分离链霉菌基因转录分析培养基优选结构鉴定
Targeted exploration of Streptomyces lavendulae subsp. CQ01819 and its secondary metabolites
Dongjin Leng1,2, Xiaojing Hu1,2, Xing Li1,2, Yixin Ou1,2, Qianjin Kang1,2

 , Linquan Bai1,2, Zixin Deng1,2
, Linquan Bai1,2, Zixin Deng1,2 1. State Key Laboratory of Microbial Metabolism, School of Life Sciences and Biotechnology, Shanghai Jiao Tong University, Shanghai 200240, China;
2. Joint International Research Laboratory of Metabolic & Developmental Sciences, Shanghai Jiao Tong University, Shanghai 200240, China
Received: 6 August 2019; Revised: 17 September 2019; Published online: 6 November 2019
*Corresponding author: Qianjin Kang, Tel:+86-21-34206119;E-mail:qjkang@sjtu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31770034, 31700027) and by the Start-up Foundation for New Young Teachers of Shanghai Jiao Tong University (17X100040064)
Abstract: [Objective] To obtain targeted natural products and the bacterial producers based on characteristic biosynthesis genes and transcription level analysis. [Methods] First, we directionally mined the secondary metabolites producers by 5 pair of degenerate primers according to the conserved biosynthesis genes. Then RT-qPCR guided fermentation medium optimization was performed. Finally, the targeted natural products were isolated using different chromatography technologies, and their chemical structures were identified based on High Resolution Mass Spectrometry (HRMS) and Nuclear Magnetic Resonance (NMR). [Results] The AHBA synthase and epoxidase-encoding genes positive Streptomyces lavendulae subsp. CQ01819 was successfully obtained from soil. Targeted compounds purification led to discovering AHBA-derived mitomycin C, polyether antibiotic monensin A and valindolmycin. [Conclusion] Our findings provide a good example for efficiently discovering bacterial resource of expected natural products.
Keywords: directional strains isolationStreptomycesgene transcription analysismedium optimizationstructure elucidation
链霉菌是抗生素的主要产生者,在医药学研究中具有重要的价值[1]。该类微生物产生的次级代谢产物种类丰富且结构多样、复杂,如:Ⅰ型聚酮类化合物中的大环内脂类、多烯类、聚醚类和安莎类等[2];Ⅱ型聚酮类化合物中的蒽环类、角蒽环类、四环素类、xanthone类和金霉酸类等[3];Ⅲ型聚酮类化合物中的查尔酮类等[4];非核糖体肽类化合物中的多肽类和糖肽类[5]等以及核糖体肽类化合物中的lanthipeptide、硫肽类和lasso peptide等[6];此外,还包括了C7N氨基环醇类、聚酮聚肽杂合类和寡糖磷脂类等[7]。这些次级代谢产物中的许多成员均具有显著的抗感染、抗肿瘤、抗氧化和酶抑制剂等活性[8],为人类的健康提供了有效的保障,已成为了当前创新药物开发的重要资源[9]。在目前使用的抗生素中,多烯类抗生素匹马霉素主要用于食品防腐剂和抗真菌药物开发[10-11];安莎类抗生素利福霉素对结核杆菌有抑制作用,在临床上被应用于肺结核的治疗[12];以美登素为小分子开发的抗体偶联药物也已经成功用于HER2阳性乳腺癌晚期患者的治疗[13];以阿卡波糖为代表的氨基环醇类抗生素是治疗Ⅱ型糖尿病的重要药物[14];在聚醚类抗生素中,盐霉素能与Na+、K+金属离子进行螯合,干扰生物膜的正常功能[15-16],特异性杀死上皮肿瘤干细胞[17],莫能菌素有抗鸡球虫病的作用,是兽用饲料的添加剂[18]。
随着病原菌株的不断演化以及临床上抗生素的大量使用,导致了耐药性病原菌的不断出现[19]。寻找新型抗生素越来越迫切,具有严峻的挑战性[20-21]。因此,研究者们通过寻找各种资源、建立多种新方法来开展新型天然产物的发掘。例如,Zipperer等从鼻腔内黏膜中分离出Staphylococcus aureus,该菌可产生一种新型含噻唑烷的环肽类抗生素Lugdunin,能够显著抑制金黄色葡萄球菌和粪肠球菌[22];Brady等将宏基因组Cosmind文库在白色链霉菌J1074中表达,通过表型筛选获得了一个新化合物metatricycloene[23];Wang等利用RecET重组系统介导的DNA片段线性-线性同源重组的方法,将Photorhabdus luminescens染色体中生物合成基因簇实现了克隆,将其在异源宿主中进行了表达,获得了化合物Luminmide A/B[24]。
微生物药物生物合成途径的系统解析为其产生的天然产物新结构创制提供了新的机会[25]。同时,大量微生物次级代谢产物生物合成基因簇的克隆与生物合成途径的解析,验证了各种结构类型化合物的保守功能基因[26],这些基因涵盖了在化合物生物合成中的模块合成基因、组装合成基因、后修饰基因和抗性基因等[27]。正是因为这些保守基因的存在,为定向追踪目标阳性菌株和活性天然产物提供了可能[28]。本课题组前期在微生物天然产物定向发掘方面积累了深厚的研究基础,保存了根据特征化合物生物合成的保守基因设计的多种简并引物,包括负责多烯类聚酮骨架修饰的P450羧化酶(CYP)基因[29]、安莎类3, 5-AHBA合酶基因[30]、C7N氨基环醇环化酶(CYC)基因、聚酮骨架特殊延伸单元的酰基辅酶A还原酶(ADH)基因[31]和聚醚类的环氧化酶(ES)基因等[32]。本研究定向地从重庆市慕江县老瀛山白云观的土壤中进行了放线菌的定向分离,从中筛选出一株AHBA合酶基因和环氧化酶基因均为阳性的浅紫灰链霉菌CQ01819。同时,对该菌株进行了基因转录分析指导的培养基优化,通过大量发酵和化合物的定向分析,获得了丝裂霉素C、莫能菌素A和缬吲霉素三种不同类型的化合物。本文对该菌株的定向分离鉴定、培养基的快速优化以及化合物的定向分离纯化和结构鉴定进行了报道。
1 材料和方法 1.1 材料
1.1.1 样品的采集: 土壤样品于2017年8月16日采集于重庆市慕江县老瀛山白云观(28°59' 5.55'' N,106°44' 20.47'' E,海拔945米),样品采集后24 h内冷藏并空运至实验室,进行分离培养。
1.1.2 主要生化试剂及仪器: 乙酸乙酯、三氯甲烷、甲醇、乙醇和异丙醇购自国药集团化学试剂股份有限公司;乙腈购自安捷伦公司;氘代甲醇购自SIGMA公司;细胞研磨仪Precellys?24购自Bertin Technologies公司;恒温振荡摇床ZQZY- 70BS为上海知楚仪器有限公司生产;电子天平BSA3202S-CW为Sartorius公司生产;旋转蒸发仪Rotavapor R-210购自BUCHI公司;生化培养箱LRH-250A购自韶关市泰宏医疗器械有限公司;高速离心机CR21G Ⅲ为Hitachi公司生产;NanoDrop 2000超微量核酸定量仪购自Thermo Fisher Scientific公司;超净工作台ZHJH-C1115C购自南京远拓科学仪器有限公司;荧光定量PCR仪QuanStudio? 3购自Thermo Fisher Scientific公司;采用Agilent 1260 Infinity高效液相色谱仪进行化合物的分析;化合物的精确分子量在Agilent G6540 Q-TOF上进行测定,核磁数据在德国Bruker公司的Avance Ⅲ 600 MHz核磁共振仪上进行采集。
1.1.3 培养基和试剂: TSBY液体培养基:胰胨豆汤粉30 g,蔗糖103 g,酵母提取物5 g,加蒸馏水至1 L,调节pH值至7.2–7.4,121 ℃灭菌20 min。
YMG培养基:酵母提取物4 g,麦芽提取物10 g,葡萄糖4 g,加蒸馏水至1 L,调节pH值至7.2,115 ℃灭菌20 min。固体培养基添加2.0%琼脂。
NK液体培养基:玉米粉10 g,可溶性淀粉20 g,黄豆饼粉10 g,K2HPO4 0.2 g,NaCl 3 g,NH4Cl3 3 g,加蒸馏水至1 L,调节pH值至7.2,121 ℃灭菌20 min。
M液体培养基:葡萄糖15 g,淀粉5 g,NaCl 5 g,酵母提取物5 g,CaCO3 3 g,加蒸馏水至1 L,调节pH值至7.2,115 ℃灭菌20 min。
J液体培养基:糊精40 g,玉米浆30 g,麦芽提取物10 g,蔗糖10 g,(NH4)2SO4 1g,CaCl2 10 g,加蒸馏水至1 L,调节pH值至7.2,115 ℃灭菌20 min。
H液体培养基:酵母提取物10 g,玉米淀粉5 g,葡萄糖5 g,甘油40 g,K2HPO4 0.5 g,FeSO4·7H2O 0.2 g,加蒸馏水至1 L,调节pH值至7.2,115 ℃灭菌20 min。
17号液体培养基:葡萄糖10 g,黄豆饼粉30 g,玉米浆10 g,甘油5 g,蔗糖10 g,NaCl 5 g,MgCl2 0.2 g,加蒸馏水至1 L,调节pH值至7.2,115 ℃灭菌20 min。
溶菌酶溶液:0.3 mol/L蔗糖,25 mmol/L Tris-Cl (pH值为8.0),25 mmol/L EDTA (pH值为8.0),0.02%溴甲酚绿,RNase (热处理过) 50 μg/mL,加入终浓度为2 mg/mL的溶菌酶溶解。
1.2 菌株分离与培养 将90 mL无菌水倒入250 mL无菌三棱瓶中,在超净工作台中加入10 g土壤样品,在振荡器上充分振荡10 min后静置15 min,收集土壤悬液,然后梯度稀释10–2–10–6倍,各取100 μL稀释液分别均匀涂布到终浓度为0.25 mol/L重铬酸钾的YMG固体培养基上,30 ℃培养7 d,挑取具有不同菌落形态特征的单个菌落,采用平板划线法,重复分离纯化,获得目的菌株。
1.3 放线菌总DNA的少量快速提取方法 将分离纯化后的菌株在YMG固体培养基上30 ℃培养5 d后,刮取少量新鲜菌体于1.5 mL离心管中,加入500 μL 2 mg/mL的溶菌酶,上下颠倒混匀后在37 ℃恒温水浴锅中放置约30 min,再加入50 μL 2%的SDS溶液,缓慢混匀至黏度显著下降。接着加入250 μL苯酚/氯仿,充分混匀后12000 r/min离心5 min,取500 μL上清至新的1.5 mL离心管,加入0.1倍体积的3 mol/L醋酸钠溶液,混合后加入1倍体积的异丙醇,混匀后室温放置5 min,将呈絮状沉淀的DNA挑出,并用70%乙醇洗涤2次后待乙醇完全挥发,加入50 μL无菌水溶解离心管底部的DNA,置于–80 ℃保存备用。
1.4 目的菌株的PCR定向筛选与测序 利用AHBA类、C7N氨基环醇类、多烯类、聚醚类以及含有甲氧基丙二酰-ACP特殊延伸单元的5对简并引物(表 1),采用PCR的方法,对所获得的菌株进行系统筛选。其中,AHBA合酶基因用利福霉素产生菌Amycolatopsis mediterranei S699作为阳性对照,预期PCR产物大小为0.63 kb;CYC基因用井冈霉素产生菌Streptomyces hygroscopicus 5008作为阳性对照,预期PCR产物大小为0.4 kb;CYP基因用两性霉素产生菌Streptomyces nodosus作为阳性对照,预期PCR产物大小为0.35 kb;ES基因用盐霉素产生菌Streptomyces albus XM211作为阳性对照,预期PCR产物大小为0.9 kb;ADH基因用含有安丝菌素产生菌Actinosynnema pretiosum ATCC 31280作为阳性对照,预期PCR产物大小为0.5 kb。
表 1. 用于菌株定向筛选的简并引物及本实验中其他引物 Table 1. Degenerate primers for strain screening and other primers in this work
| The name of conserved genes | Primers | Sequences (5′→3′) | Source |
| 3-amino-5-hydroxybenzoate (AHBA) synthase | AHBA-F AHBA-R | CCSGCCTTCACCTTCATCTCCTC AYCCGGAACATSGCCATGTAGTG | [30] |
| Cyclase (CYC) | CYC-F CYC-R | AAGACMGGBGTSAAC ACSGCCTCGCCGTGCAG | Our lab |
| Cytochrome P450 hydroxylase (CYP) | CYP-F CYP-R | GGATCGGCGACGACCGSVYCG CCGWASAGSAYSCCGTCGTACTT | [29] |
| Epoxidase (ES) | Epo-F Epo-R | GTSACCGTSRTSGAVCGIGA GCTCATSCCRTGSCCGT | [32] |
| Acyl-CoA dehydrogenases (ADH) | ADH-F ADH-R | CAGGGCATGGCCGCSTGGACSGT GCAGGCGCGCAGGATSCCSACRCA | [31] |
| Purpose | |||
| Primers for 16S rRNA PCR analysis | 27 F | AGAGTTTGATCCTGGCTCAG | [33] |
| 1492 R | ACGGCTACCTTGTTACGACT | ||
| Antisense primer for RT-qPCR analysis | AHBA-FF | GGCTCCAACTACCGCATGAA | This work |
| AHBA-RR | ATGGCCATGTAGTGGGAGTG | ||
| Epo-FF | CCCTCGAAGAGGACGTGGT | ||
| Epo-RR hrdB-FF hrdB-RR | ATCCAGCGGCCGCCCTCGAT CATGTCTTCATAAGCCCATG TGCCACAGACTTGAGACTC |
表选项
此步PCR反应采用翊圣公司的2×HieffTM PCR Master Mix(With dye),50 μL反应体系:2×HieffTM PCR Master Mix 25 μL,上、下游引物各2 μL,50% DMSO 5 μL,基因组DNA 1 μL,蒸馏水补齐至50 μL。扩增条件为95 ℃ 5 min;95 ℃ 30 s,65 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 10 min,4 ℃保存。PCR产物经1%琼脂糖凝胶电泳检测后,切胶回收纯化,然后送至派森诺公司进行测序。
1.5 菌株CQ01819菌种鉴定
1.5.1 菌株CQ01819的16S rRNA序列扩增与测序: 挑取少量该菌株新鲜的菌体提取基因组DNA作为PCR反应模板,使用细菌16S rRNA通用引物27 F和1492 R(表 1)进行PCR。此步所用高保真DNA聚合酶为Vazyme公司的Phanta? Max Super-Fidelity DNA Polymerase,配制50 μL PCR反应体系:2 × Phanta? Max Buffer 25 μL,dNTP Mix 2 μL,上、下游引物各2 μL,50% DMSO 5 μL,基因组DNA 1 μL,Phanta? Max Super-Fidelity DNA Polymerase 1 μL,蒸馏水12 μL。扩增条件为95 ℃ 5 min;95 ℃ 30 s,58 ℃ 30 s,72 ℃ 1.5 min,30个循环;72 ℃ 10 min,4 ℃保存。PCR产物预期大小为1.5 kb,经1%琼脂糖凝胶电泳检测后,切胶回收纯化,送至派森诺公司进行测序。
1.5.2 菌株CQ01819系统发育树的构建: 得到测序成功的序列后,手动删除序列头尾质量不佳的部分,使用DNAMAN软件对测序结果进行拼接,利用NCBI网站下载GenBank相似性较高的菌株序列,使用ClustalX进行多序列对比,基因序列之间遗传距离通过MEGA 7.0计算完成,采用Neighbor-Joining (NJ)法[34]构建系统发育进化树,用自展法(Bootstrap)进行检验,自展数据集为1000次。
1.6 细胞破碎法提取菌株CQ01819总RNA 将甘油保存的链霉菌孢子悬液接种至25 mL TSBY液体培养基中,在30 ℃、220 r/min的恒温摇床中培养24 h,制备种子菌液。按1/25的接种量分别将种子菌液接种到5种不同的发酵培养基中,于30 ℃、220 r/min的恒温摇床中培养72 h。取1 mL发酵液,4 ℃离心5 min收集菌体,加入少量直径为1 μm玻璃珠后,取1 mL的Redzol溶液将菌体悬浮,在振荡细胞破碎仪上将其破碎,最后于冰上放置5 min。加入200 μL氯仿混匀,在4 ℃低温下12000 r/min离心15 min。吸取900 μL上清转入新的1.5 mL管中,加入200 μL无水乙醇,上下颠倒混匀后将溶液加入预先准备好的离心柱中,冰上静置3 min,4 ℃、12000 r/min离心1 min,弃掉收集管中的液体。用600 μL缓冲液洗涤2次,弃去收集管中的液体,再离心2 min,最后放在超净工作台中吹干。取50 μL DEPC处理的水进行溶解,再将离心柱12000 r/min离心1 min,重复2次。取2 μL样品经1%琼脂糖凝胶电泳检测RNA完整性,其余样品于–80 ℃冰箱保存备用。
1.7 RT-qPCR导向的培养基优选 将提取的总RNA反转录为cDNA,此步通过Fermentas公司的RevertAidTM H Minus First Strand cDNA Synthesis Kit说明书上的步骤来完成。
接下来用已合成的cDNA进行qPCR,所用试剂盒是Fermentas公司的MaximaTM SYBR Green/ ROX qPCR Master Mix (2×)。反应体系共20 μL,其中包括cDNA 5 μL,上、下游引物各1 μL,2×qPCR Master Mix 10 μL,蒸馏水3 μL。上述体系配好后,在QuanStudio? 3仪器中进行qPCR扩增。运行程序为:50 ℃ 2 min,95 ℃ 10 min;95 ℃ 15 s,60 ℃ 20 s,72 ℃ 20 s,40个循环;4 ℃保存。取3 μL PCR产物经1.5%琼脂糖凝胶电泳检测不同培养基中目的基因的转录情况,进行培养基优选。
1.8 菌株CQ01819的大量发酵 将分离纯化完成的菌株接种于30 mL TSBY液体培养基的250 mL三棱瓶中,在30 ℃、220 r/min的恒温摇床里培养1 d,得到种子液。将种子液按照1/25的比例接种至装有400 mL M液体发酵培养基的2 L三棱瓶中,共发酵8 L,同样条件下发酵4 d。
1.9 化合物的分离纯化 8 L发酵液离心后,将上清菌液用等体积乙酸乙酯萃取3次,萃取液在旋转蒸发仪中进行浓缩,获得发酵液提取物约2 g。
将发酵液提取物经MCI柱分离,用纯水、水-甲醇(7:3)、水-甲醇(1:1)、水-甲醇(3:7)、甲醇流动相顺序洗脱,得到4个组分(P1–P4)。其中P3经硅胶柱层析,用三氯甲烷-甲醇(25:1),三氯甲烷-甲醇(9:1)洗脱,得到2个馏分(L1和L2)。L2经过凝胶Sephadex LH-20柱层析,用甲醇洗脱得化合物1 (4 mg)。P4经正向硅胶柱层析,用三氯甲烷-甲醇(100:3)洗脱,得粗样120 mg。然后,用Agilent 1260 Infinity高效液相色谱仪进行化合物分离条件摸索,流动相为:乙腈-水(1‰的甲酸),流速:0.5 mL/min,均采用全波长扫描。HPLC洗脱条件:0–5 min时15%乙腈洗脱,6–35 min时10%–95%乙腈梯度洗脱,36–45 min时15%乙腈洗脱。最后,经HPLC制备,洗脱得化合物2 (5 mg)和化合物3 (3 mg)。将得到纯化的化合物用Agilent G6540 Q-TOF高分辨质谱仪测定单体化合物的分子量,在Avance Ⅲ 600 MHz核磁共振仪上采集化合物的核磁数据。
2 结果和分析 2.1 放线菌菌株的分离纯化 首先,对不同稀释度的土壤样品进行菌落的计算,确定了2×10–5倍稀释浓度可以长出较均匀分散的菌落。然后,将稀释后的土壤样品均匀涂布在含有0.25 mol/L重铬酸钾的YMG固体培养基上。3 d后菌落开始慢慢长出,培养7 d后,培养基上长出了形态各异的菌落。最后,挑取放线菌单克隆,采用平板划线法对长出的菌落进行重复分离纯化,直至获得纯化培养的菌株。
2.2 放线菌菌株的定向筛选 制备纯化培养菌株的总DNA样品,选取5种不同抗生素生物合成途径中的保守基因探针,分别为:(1)多烯类抗生素中的P450羧化酶(CYP)编码基因,(2)含有AHBA结构单元的抗生素中的AHBA合酶编码基因,(3) C7N氨基环醇类抗生素中的环化酶(CYC)编码基因,(4)含有甲氧基丙二酰-ACP特殊延伸单元的抗生素中的酰基辅酶A还原酶(ADH)编码基因,(5)聚醚类抗生素中环氧化酶(ES)的编码基因,对分离到的菌株进行定向筛选。如图 1所示,编号为CQ01819的菌株在使用AHBA合成酶和聚醚类抗生素的环氧化酶编码基因的引物探针AHBA-F/R和Epo-F/R时,均获得了阳性的PCR结果,条带大小分别为0.63 kb和0.9 kb,其他引物探针的PCR均呈现了阴性结果。将PCR产物进行回收后,进行序列测定。
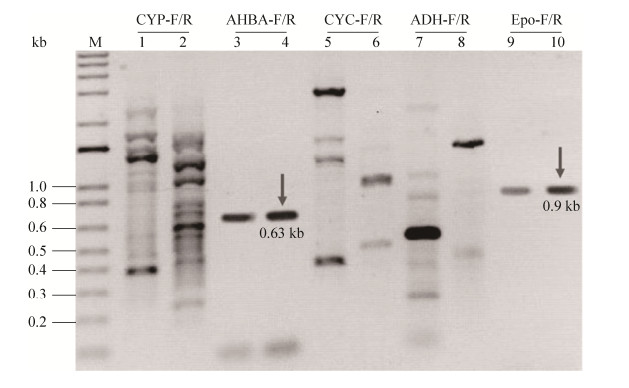 |
| 图 1 菌株CQ01819的PCR定向筛选结果 Figure 1 The result of PCR-directed screening of strain CQ01819. M: Marker; lanes 1 and 2 were amplified with primers CYP-F/R, the templates were gDNA of S. nodosus and CQ01819, the expected PCR products were 0.35 kb; lanes 3 and 4 were amplified with primers AHBA-F/R, the templates were gDNA of A. mediterranei S699 and CQ01819, the expected PCR products were 0.63 kb; lanes 5 and 6 were amplified with primers CYC-F/R, the templates were gDNA of S. hygroscopicus 5008 and CQ01819, and the expected PCR products were 0.4 kb; lanes 7 and 8 were amplified with primers ADH-F/R, the templates were gDNA of A. pretiosum ATCC 31280 and CQ01819, the expected PCR products were 0.5 kb; lanes 9 and 10 were amplified with primer Epo-F/Epo-R, the templates were gDNA of S. albus XM211 and CQ01819, the expected PCR products were 0.9 kb. |
| 图选项 |
因为该部分DNA序列编码了蛋白含有保守结构域的氨基酸残基,所以将测序后得到的DNA序列在NCBI网站进行BLASTX,收集同源性较高的蛋白序列构建系统进化树(图 2和图 3),进一步确定了菌株CQ01819中含有的AHBA合酶CqnH与丝裂霉素生物合成中的AHBA合酶MitA具有较高的同源性,环氧化酶CqnE的序列与莫能菌素生物合成中的环氧化酶具有较高的同源性,表明了该菌株可能具有产生丝裂霉素和莫能菌素的潜力。
 |
| 图 2 AHBA合酶的系统发育树 Figure 2 Phylogenetic tree of AHBA synthases. |
| 图选项 |
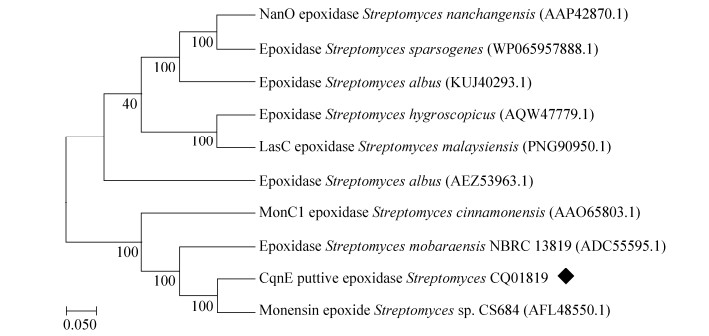 |
| 图 3 环氧化酶的系统发育树 Figure 3 Phylogenetic tree of epoxidases. |
| 图选项 |
2.3 菌株CQ01819的鉴定 以菌株CQ01819基因组DNA为模板,利用细菌16S rRNA通用引物27F/1492R进行PCR扩增,获得了1.5 kb的PCR产物,经胶回收纯化后进行测序。然后,将所获得的基因序列在NCBI上通过BLAST与GenBank数据库的序列进行比对,选取同源性较高的序列,通过MEGA 7.0软件的Neighbor-joining法[34]构建系统发育树,比较结果显示菌株CQ01819与Streptomyces lavendulae 属于同一个分枝(图 4),因此,命名为浅紫灰链霉菌CQ01819 (Streptomyces lavendulae subsp. CQ01819)。
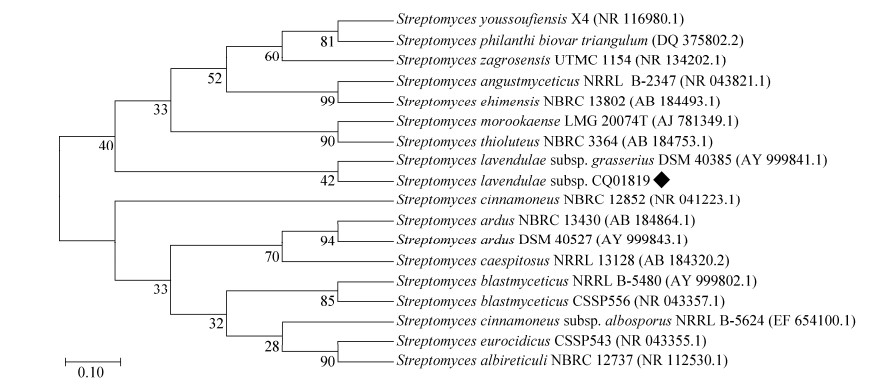 |
| 图 4 浅紫灰链霉菌CQ01819系统发育树 Figure 4 Phylogenetic tree of Streptomyces lavendulae subsp. CQ01819. |
| 图选项 |
2.4 基于RT-qPCR指导的浅紫灰链霉菌CQ01819发酵培养基优选 为了进一步考察浅紫灰链霉菌CQ01819对含有AHBA结构单元与聚醚类化合物的产生能力,选取了5种碳氮源成分差异较大的液体培养基(编号分别为NK、17、H、M和J),利用RT-qPCR指导的检测方法,对该菌株的发酵培养基进行了优选。首先,根据所获得的AHBA合酶编码基因和ES编码基因的序列,设计转录分析的特异性引物。然后,将菌株发酵培养72 h,分别制备发酵样品的cDNA并进行定量。接着,考察了2对引物的特异性,发现溶解曲线峰形较为单一,说明所选用的引物特异性较好(图 5)。最后,以hrdB作为内参分别对这两个目标基因的转录水平进行了PCR分析,结果显示,AHBA合酶基因以及环氧化酶基因在5种不同的液体培养基发酵过程中均有不同程度的表达,其中AHBA合酶基因在17和M两种培养基中的转录水平较高,而环氧化酶基因在NK和M培养基中转录情况较好(图 5)。同时,将获得的PCR产物利用琼脂糖凝胶电泳进行检测,也得到了相同的结果(图 5)。因此,接下来选用M液体培养基作为浅紫灰链霉菌CQ01819大量发酵的培养基。
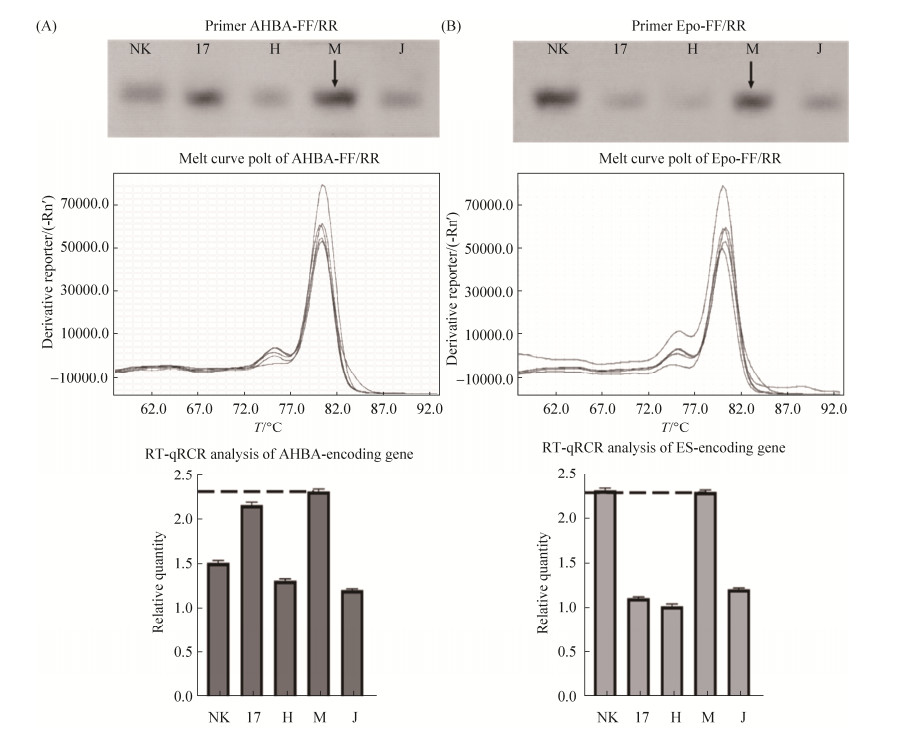 |
| 图 5 AHBA合酶基因(A)和环氧化酶编码基因(B)在不同发酵条件下的转录分析结果 Figure 5 Transcriptional analysis of AHBA synthase (A) and epoxidase (B) encoding gene under different fermentation conditions. |
| 图选项 |
2.5 浅紫灰链霉菌CQ01819的大量发酵与粗提物样品的制备 将保存在–80 ℃冰箱的菌种接种于YMG固体培养基上活化5 d,再接种于含有种子培养基TSBY的250 mL弹簧瓶中,培养24 h后,将种子培养液转接到装有M液体发酵培养基的2000 mL弹簧瓶中,在30 ℃、220 r/min的条件下进行发酵,共计发酵8 L。4 d后,离心收取发酵液,将上清菌液用乙酸乙酯在室温萃取3次,合并萃取液,进行减压浓缩,获得发酵液粗提物约2 g。
接下来将粗提物过滤,经MCI胶柱层析初步分离,再经过正向柱层析、葡聚糖凝胶LH-20色谱柱层析和HPLC对样品进行细致地分离纯化(图 6-A),最终获得化合物1 (4 mg)、化合物2 (5 mg)和化合物3 (3 mg)。
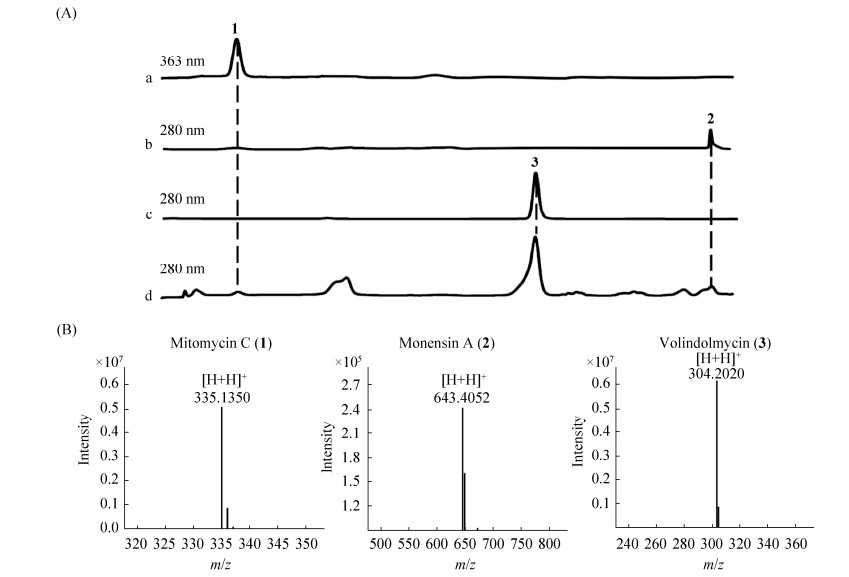 |
| 图 6 发酵总产物及纯化样品的高效液相色谱(A)和纯化样品的高分辨质谱检测结果(B) Figure 6 HPLC analysis of crude extract and purified compounds (1–3) (A) and the results of HR-ESI-MS analysis of compounds (1–3) (B). a: HPLC of purified compound 1; b: HPLC of purified compound 2; c: HPLC of purified compound 3; d: HPLC of total extract. |
| 图选项 |
2.6 化合物结构鉴定 化合物1:深紫色粉末;分子式:C15H18N4O5;HR-ESI-MS m/z: 335.1350 [M+H]+(图 6-B);1H NMR (Methanol-d4,600 MHz)δ:4.67(2H,dd,J=4.4,10.59 Hz,H-13),4.30(1H,t,J=10.9 Hz,H-7),4.16(2H,d,J=13.0 Hz,H-11),3.58(2H,dd,J=4.4,11.2 Hz,H-13),3.48(2H,dd,J=2.0,13.0 Hz,H-11),3.22(3H,s,H-15),2.98(1H,d,J=4.6 Hz,H-9),2.86(1H,dd,J=1.9,4.6 Hz,H-10),1.75(3H,s,H-12);13 C NMR(Methanol-d4,125 MHz)δ:177.0(s,C-1),151.1(s,C-2),107.3(s,C-3),179.0(s,C-4),157.4(s,C-5),110.6(s,C-6),44.4(d,C-7),104.8(s,C-8),37.5(d,C-9),33.5(d,C-10),50.8(t,C-11),8.10(q,C-12),63.1(t,C-13),159.6(s,C-14),50.0(q,C-15)。以上数据与文献一致[35],确认化合物1为丝裂霉素C (图 7)。
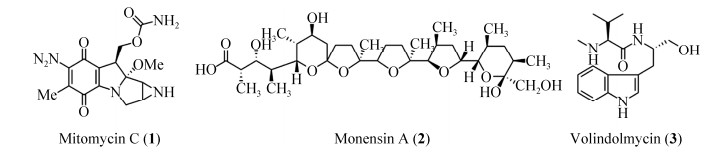 |
| 图 7 化合物1–3的化学结构式 Figure 7 Chemical structures of compounds 1–3. |
| 图选项 |
化合物2: 无色固体;分子式: C34H58O11;HR-ESI-MS m/z: 643.4052 [M+H]+(图 6-B);1H NMR (Methanol-d4,600 MHz)δ: 4.36(2H,br s,H-5),4.16(2H,d,J=10.8 Hz,H-5),4.07(1H,d,J=6.0 Hz,H-17),3.96(1H,d,J=4.2 Hz,H-7),3.85(1H,br s,H-3),3.78(1H,m,H-21),3.73(1H,m,H-13),3.72(1H,m,H-26),2.69(1H,dq,J=13.8 Hz,6.0 Hz,H-2),2.20(1H,m,H-18),2.18(1H,m,H-19),2.02(2H,m,H-15),2.00(1H,m,H-4),1.98(1H,m,H-6),1.96(2H,m,H-11),1.85(2H,m,H-15),1.84(2H,m,H-10),1.68(2H,m,H-14),1.66(2H,m,H-8),1.66(2H,m,H-11),1.59(2H,m,H-23),1.58(2H,m,H-8),1.56(1H,s,H-30),1.55(2H,m,H-14),1.52(1H,s,H-31),1.50(2H,m,H-10),1.33(1H,m,H-19),1.29(2H,m,H-23),1.21(1H,m,H-22),1.21(1H,d,J=6.6 Hz,H-34),1.12(1H,d,J=6.2 Hz,H-32),1.12(1H,d,J=6.2 Hz,H-33),0.98(1H,d,J=6.4 Hz,H-34),0.96(1H,d,J=7.2 Hz,H-29),0.89(1H,d,J=6.0 Hz,H-27),0.88(1H,d,J=6.0 Hz,H-28);13C NMR(Methanol-d4,125 MHz)δ: 174.0(s,C-1),45.6(d,C-2),75.1(d,C-3),42.4(d,C-4),67.0(d,C-5),35.2(d,C-6),70.7(d,C-7),33.7(t,C-8),107.5(s, C-9),38.8(t,C-10),33.8(t,C-11),84.5(s,C-12),82.1(d,C-13),27.0(t,C-14),27.2(t,C-15),84.5(s,C-16),86.9(d,C-17),34.8(d,C-18),33.7(t,C-19),78.2(d,C-20),75.3(d,C-21),32.7(d,C-22),36.7(t,C-23),36.5(d,C-24),97.5(s,C-25),71.5(t,C-26),15.9(q,C-27),16.5(q,C-28),14.5(q,C-29),10.7(q,C-30),27.2(q,C-31),16.5(q,C-32),9.3(q,C-33),8.2(q,C-34)。以上数据与文献一致[36],确定化合物2为莫能菌素A (图 7)。
化合物3:无色固体;分子式:;C17H25N3O2;HR-ESI-MS m/z: 304.2020 [M+H]+(图 6-B);1H NMR(Methanol-d4,600 MHz)δ:7.64(1H,d,J=7.9 Hz,H-5),7.30(1H,d,J=8.1 Hz,H-8),7.09(1H,s,H-10),7.06(1H,m,H-7),7.00(1H,m,H-6),4.33(1H,m,H-1),3.60(2H,dd,J=2.7,5.4 Hz,H-11),3.07(1H,dd,J=14.8,5.8 Hz,H-2),2.90(1H,dd,J=14.6,8.6 Hz,H-2),2.74(1H,d,J=6.3 Hz,H-13),2.02(3H,s,H-17),1.82(1H,dd,J=6.8,13.4 Hz,H-14),0.89(3H,d,J=6.9 Hz,H-16),0.88(3H,d,J=6.9 Hz,H-15);13C NMR (Methanol-d4,125 MHz)δ:53.4(s,C-1),27.9(d,C-2),112.3(d,C-3),129.0(d,C-4),119.5(d,C-5),119.6(d,C-6),122.3(d,C-7),122.1(t,C-8),138.1(s,C-9),124.2(t,C-10),64.7(t,C-11),174.3(s,C-12),71.2(d,C-13),32.3(t,C-14),19.1(t,C-15),19.4(s,C-16),34.5(d,C-17)。以上数据与文献一致[37],确认化合物3为缬吲霉素(图 7)。
3 讨论 放线菌产生了众多具有较强生理活性的天然产物,成为新药发现的宝贵资源,引起了许多研究者的关注[38]。在这些天然产物的生物合成过程中,含有类似或相同结构单元的成员,在其生物合成基因簇中均具有同源性较高的功能蛋白编码基因。因此,围绕具有开发潜力的抗生素对环境样品中的菌株进行定向分离,可快速获得具有产生目标结构化合物的菌株资源。本研究基于实验室前期的积累,利用5种特征化合物的保守基因的简并引物(涵盖了多烯类、安莎类、聚醚类、聚酮特殊延伸单元类和C7N氨基环醇类),采用PCR检测方法,对采自重庆市老瀛山的土壤样品中的微生物进行了定向的分离,快速确定了浅紫灰链霉菌CQ01819中含有AHBA合酶编码基因和醚类抗生素中环氧化酶(ES)编码基因。通过系统进化树分析发现浅紫灰链霉菌CQ01819中的AHBA合酶CqnH聚在了苯AHBA抗生素的分枝中,与丝裂霉素生物合成基因簇中的MitA(AHBA合酶编码基因)的同源性最高。同时,所获得的环氧化酶CqnE也与莫能菌素生物合成基因簇中的环氧化酶同源性最高,这些结果表明该菌株可能具有产生这些化合物的潜力。此外,在对微生物药物的生物合成调控及生物合成途径优化的研究过程中,通过基因转录水平的评估,检测目标基因的表达情况是较为常用的研究策略[39]。为了快速获得AHBA合酶基因和环氧化酶基因表达水平较高的发酵条件,我们以RT-qPCR为指导,用这2个基因的特异性序列,设计转录分析的引物,根据不同的发酵培养基的转录分析结果,迅速优选了浅紫灰链霉菌CQ01819的发酵培养基。接着,对该菌株进行了大量发酵和化合物的定向分离。最后,快速地获得了丝裂霉素C和聚醚类抗生素莫能菌素A,同时获得一个吲哚类化合物缬吲霉素,相关的化学结构均利用NMR进行了结构鉴定。
本研究所获得的3个化合物均具有突出的生物活性,丝裂霉素C含有氮杂环丙烷、氨基甲酸乙酯和苯醌基团,具有显著的抗肿瘤活性。其抗肿瘤的作用机制是丝裂霉素C分子中的氮丙啶与氨甲酰基烷化形成激活态,在DNA小沟中形成链间和链内交联,从而阻碍DNA的复制,抑制癌细胞的分裂,临床上用于治疗包括消化道、肺癌、颈部肿瘤和慢性白血病在内的人体多种恶性肿瘤[40]。莫能菌素具有离子载体功能,能够与一价阳离子Na+、Li+、Ag+、K+等形成络合物,在电中性的情况下将这些阳离子转运穿过细胞膜,起到离子的逆向运输而杀死细胞[41],对一些病原细菌、病毒、真菌和寄生虫等均具有较好的活性,现已经作为治疗鸡球虫病的兽用抗生素使用。缬吲霉素是蛋白质激酶C的抑制剂,是杀鱼菌素的重要生物合成中间体。
综上所述,本研究利用特征化合物的保守基因探针,快速定向地筛选目标菌株是一个发掘菌株资源的有效策略。同时,由基因转录指导的培养基优化是一个迅速确定菌株发酵培养基的方法,可以为定向的化合物分离鉴定提供较好的保障。而且,从浅紫灰链霉菌CQ01819所获得的次级代谢产物结构多样,生物活性较强,具有重要的研究价值。
References
| [1] | Liu R, Deng ZX, Liu TG. Streptomyces species:ideal chassis for natural product discovery and overproduction. Metabolic Engineering, 2018, 50: 74-84. DOI:10.1016/j.ymben.2018.05.015 |
| [2] | Ning XJ, Wang XR, Wu YT, Kang QJ, Bai LQ. Identification and engineering of post-PKS modification bottlenecks for ansamitocin P-3 titer improvement in Actinosynnema pretiosum subsp. pretiosum ATCC 31280. Biotechnology Journal, 2017, 12(11): 1700484. |
| [3] | Risdian C, Mozef T, Wink J. Biosynthesis of polyketides in Streptomyces. Microorganisms, 2019, 7(5): 124. DOI:10.3390/microorganisms7050124 |
| [4] | Lim YP, Go MK, Yew WS. Exploiting the biosynthetic potential of type Ⅲ polyketide synthases. Molecules, 2016, 21(6): E806. DOI:10.3390/molecules21060806 |
| [5] | Süssmuth RD, Mainz A. Nonribosomal peptide synthesis-principles and prospects. Angewandte Chemie International Edition, 2017, 56(14): 3770-3821. DOI:10.1002/anie.201609079 |
| [6] | Arnison PG, Bibb MJ, Bierbaum G, Bowers AA, Bugni TS, Bulaj G, Camarero JA, Campopiano DJ, Challis GL, Clardy J, Cotter PD, Craik DJ, Dawson M, Dittmann E, Donadio S, Dorrestein PC, Entian KD, Fischbach MA, Garavelli JS, G?ransson U, Gruber CW, Haft DH, Hemscheidt TK, Hertweck C, Hill C, Horswill AR, Jaspars M, Kelly WL, Klinman JP, Kuipers OP, Link AJ, Liu W, Marahiel MA, Mitchell DA, Moll GN, Moore BS, Müller R, Nair SK, Nes IF, Norris GE, Olivera BM, Onaka H, Patchett ML, Piel J, Reaney MJT, Rebuffat S, Ross RP, Sahl HG, Schmidt EW, Selsted ME, Severinov K, Shen B, Sivonen K, Smith L, Stein T, Sussmuth RD, Tagg JR, Tang GL, Truman AW, Vederas JC, Walsh CT, Walton JD, Wenzel SC, Willey JM, van der Donk WA. Ribosomally synthesized and post-translationally modified peptide natural products:overview and recommendations for a universal nomenclature. Natural Product Reports, 2013, 30(1): 108-160. |
| [7] | Chen RD, Zhang QB, Tan B, Zheng LJ, Li HX, Zhu YG, Zhang CS. Genome mining and activation of a silent PKS/NRPS gene cluster direct the production of totopotensamides. Organic Letters, 2017, 19(20): 5697-5700. DOI:10.1021/acs.orglett.7b02878 |
| [8] | Nepal KK, Wang GJ. Streptomycetes:surrogate hosts for the genetic manipulation of biosynthetic gene clusters and production of natural products. Biotechnology Advances, 2019, 37(1): 1-20. |
| [9] | Newman DJ, Cragg GM. Natural products as sources of new drugs from 1981 to 2014. Journal of Natural Products, 2016, 79(3): 629-661. |
| [10] | Qi Z, Zhou YC, Kang QJ, Jiang CY, Zheng JT, Bai LQ. Directed accumulation of less toxic pimaricin derivatives by improving the efficiency of a polyketide synthase dehydratase domain. Applied Microbiology and Biotechnology, 2017, 101(6): 2427-2436. DOI:10.1007/s00253-016-8074-7 |
| [11] | Qi Z, Kang QJ, Jiang CY, Han M, Bai LQ. Engineered biosynthesis of pimaricin derivatives with improved antifungal activity and reduced cytotoxicity. Applied Microbiology and Biotechnology, 2015, 99(16): 6745-6752. DOI:10.1007/s00253-015-6635-9 |
| [12] | Kang QJ, Shen YM, Bai LQ. Biosynthesis of 3, 5-AHBA-derived natural products. Natural Product Reports, 2012, 29(2): 243-263. |
| [13] | von Minckwitz G, Huang CS, Mano MS, Loibl S, Mamounas EP, Untch M, Wolmark N, Rastogi P, Schneeweiss A, Redondo A, Fischer HH, Jacot W, Conlin AK, Arce-Salinas C, Wapnir IL, Jackisch C, DiGiovanna MP, Fasching PA, Crown JP, Wülfing P, Shao ZM, Rota Caremoli R, Wu HY, Lam LH, Tesarowski D, Smitt M, Douthwaite H, Singel SM, Geyer Jr CE, KATHERINE Investigators. Trastuzumab emtansine for residual invasive HER2-positive breast cancer. The New England Journal of Medicine, 2019, 380(7): 617-628. DOI:10.1056/NEJMoa1814017 |
| [14] | Xie HX, Zhao QQ, Zhang X, Kang QJ, Bai LQ. Comparative functional genomics of the acarbose producers reveals potential targets for metabolic engineering. Synthetic and Systems Biotechnology, 2019, 4(1): 49-56. DOI:10.1016/j.synbio.2019.01.001 |
| [15] | Jiang CY, Qi Z, Kang QJ, Liu J, Jiang M, Bai LQ. Formation of the ?18, 19 double bond and bis (spiroacetal) in salinomycin is atypically catalyzed by SlnM, a methyltransferase-like enzyme. Angewandte Chemie International Edition, 2015, 54(31): 9097-9100. DOI:10.1002/anie.201503561 |
| [16] | Hama? A, Ca?eque T, Müller S, Mai TT, Hienzsch A, Ginestier C, Charafe-Jauffret E, Codogno P, Mehrpour M, Rodriguez R. An iron hand over cancer stem cells. Autophagy, 2017, 13(8): 1465-1466. DOI:10.1080/15548627.2017.1327104 |
| [17] | Mai TT, Hamai A, Hienzsch A, Ca?eque T, Müller S, Wicinski J, Cabaud O, Leroy C, David A, Acevedo V, Ryo A, Ginestier C, Birnbaum D, Charafe-Jauffret E, Codogno P, Mehrpour M, Rodriguez R. Salinomycin kills cancer stem cells by sequestering iron in lysosomes. Nature Chemistry, 2017, 9(10): 1025-1033. DOI:10.1038/nchem.2778 |
| [18] | Xie XQ, Garg A, Khosla C, Cane DE. Mechanism and stereochemistry of polyketide chain elongation and methyl group epimerization in polyether biosynthesis. Journal of the American Chemical Society, 2017, 139(8): 3283-3292. DOI:10.1021/jacs.7b00278 |
| [19] | Santos-Beneit F, Ordó?ez-Robles M, Martín JF. Glycopeptide resistance:links with inorganic phosphate metabolism and cell envelope stress. Biochemical Pharmacology, 2017, 133: 74-85. DOI:10.1016/j.bcp.2016.11.017 |
| [20] | Baral B, Akhgari A, Mets?-Ketel? M. Activation of microbial secondary metabolic pathways:Avenues and challenges. Synthetic and Systems Biotechnology, 2018, 3(3): 163-178. DOI:10.1016/j.synbio.2018.09.001 |
| [21] | Rossiter SE, Fletcher MH, Wuest WM. Natural products as platforms to overcome antibiotic resistance. Chemical Reviews, 2017, 117(19): 12415-12474. DOI:10.1021/acs.chemrev.7b00283 |
| [22] | Zipperer A, Konnerth MC, Laux C, Berscheid A, Janek D, Weidenmaier C, Burian M, Schilling NA, Slavetinsky C, Marschal M, Willmann M, Kalbacher H, Schittek B, Br?tz-Oesterhelt H, Grond S, Peschel A, Krismer B. Human commensals producing a novel antibiotic impair pathogen colonization. Nature, 2016, 535(7613): 511-516. DOI:10.1038/nature18634 |
| [23] | Iqbal HA, Low-Beinart L, Obiajulu JU, Brady SF. Natural product discovery through improved functional metagenomics in Streptomyces. Journal of the American Chemical Society, 2016, 138(30): 9341-9344. DOI:10.1021/jacs.6b02921 |
| [24] | Wang HL, Li Z, Jia RN, Hou Y, Yin J, Bian XY, Li AY, Müller R, Stewart AF, Fu J, Zhang YM. RecET direct cloning and Redαβ recombineering of biosynthetic gene clusters, large operons or single genes for heterologous expression. Nature Protocols, 2016, 11(7): 1175-1190. DOI:10.1038/nprot.2016.054 |
| [25] | Pérez-Victoria I, Oves-Costales D, Lacret R, Martin J, Sanchez-Hidalgo M, Diaz C, Cautain B, Vicente F, Genilloud O, Reyes F. Structure elucidation and biosynthetic gene cluster analysis of caniferolides A-D, new bioactive 36-membered macrolides from the marine-derived Streptomyces caniferus CA-271066. Organic & Biomolecular Chemistry, 2019, 17(11): 2954-2971. |
| [26] | Blodgett JA, Zhang JK, Yu XM, Metcalf WW. Conserved biosynthetic pathways for phosalacine, bialaphos and newly discovered phosphonic acid natural products. The Journal of Antibiotics (Tokyo), 2016, 69(1): 15-25. |
| [27] | Xu M, Wang YM, Zhao ZL, Gao GX, Huang SX, Kang QJ, He XY, Lin SJ, Pang XH, Deng ZX, Tao MF. Functional genome mining for metabolites encoded by large gene clusters through heterologous expression of a whole-genome bacterial artificial chromosome library in Streptomyces spp. Applied and Environmental Microbiology, 2016, 82(19): 5795-5805. DOI:10.1128/AEM.01383-16 |
| [28] | Shen B. A new golden age of natural products drug discovery. Cell, 2015, 163(6): 1297-1300. DOI:10.1016/j.cell.2015.11.031 |
| [29] | Hwang YB, Lee MY, Park HJ, Han K, Kim ES. Isolation of putative polyene-producing actinomycetes strains via PCR-based genome screening for polyene-specific hydroxylase genes. Process Biochemistry, 2007, 42(1): 102-107. |
| [30] | Zhu N, Zhao PJ, Shen YM. Selective isolation and ansamycin-targeted screenings of commensal actinomycetes from the "maytansinoids-producing" arboreal Trewia nudiflora. Current Microbiology, 2009, 58(1): 87-94. DOI:10.1007/s00284-008-9284-8 |
| [31] | Zhao CH, Ju JH, Christenson SD, Smith WC, Song DF, Zhou XF, Shen B, Deng ZX. Utilization of the methoxymalonyl-acyl carrier protein biosynthesis locus for cloning the oxazolomycin biosynthetic gene cluster from Streptomyces albus JA3453. Journal of Bacteriology, 2006, 188(11): 4142-4147. DOI:10.1128/JB.00173-06 |
| [32] | Jiang CY, Wang HG, Kang QJ, Liu J, Bai LQ. Cloning and characterization of the polyether salinomycin biosynthesis gene cluster of Streptomyces albus XM211. Applied and Environmental Microbiology, 2012, 78(4): 994-1003. |
| [33] | Chen YL, Lee CC, Lin YL, Yin KM, Ho CL, Liu T. Obtaining long 16S rDNA sequences using multiple primers and its application on dioxin-containing samples. BMC Bioinformatics, 2015, 16(S18): S13. |
| [34] | Eickmeyer K, Huggins P, Pachter L, Yoshida R. On the optimality of the neighbor-joining algorithm. Algorithms for Molecular Biology, 2008, 3: 5. DOI:10.1186/1748-7188-3-5 |
| [35] | Sitachitta N, Lopanik NB, Mao YQ, Sherman DH. Analysis of a parallel branch in the mitomycin biosynthetic pathway involving the mitN-encoded aziridine N-methyltransferase. The Journal of Biological Chemistry, 2007, 282(29): 20941-20947. DOI:10.1074/jbc.M702456200 |
| [36] | Pospí?il S, Sedmera P, Vokoun J, Van?k Z, Bud?sínsky M. 3-O-demethylmonensins A and B produced by Streptomyces cinnamonensis. The Journal of Antibiotics, 1987, 40(4): 555-557. |
| [37] | Jin WZ, Cui GL. Valindolmycin, a new indole antibiotic, isolation and structure determination. Chinese Journal of Antibiotics, 1988, 13(6): 397-401. (in Chinese) 金文藻, 崔桂兰. 新抗生素缬吲霉素的分离及结构测定. 中国抗生素杂志, 1988, 13(6): 397-401. |
| [38] | Tan GY, Liu TG. Rational synthetic pathway refactoring of natural products biosynthesis in actinobacteria. Metabolic Engineering, 2017, 39: 228-236. DOI:10.1016/j.ymben.2016.12.006 |
| [39] | Jeong Y, Kim JN, Kim MW, Bucca G, Cho S, Yoon YJ, Kim BG, Roe JH, Kim SC, Smith CP, Cho BK. The dynamic transcriptional and translational landscape of the model antibiotic producer Streptomyces coelicolor A3(2). Nature Communications, 2016, 7: 11605. DOI:10.1038/ncomms11605 |
| [40] | Bass PD, Gubler DA, Judd TC, Williams RM. Mitomycinoid alkaloids:mechanism of action, biosynthesis, total syntheses, and synthetic approaches. Chemical Reviews, 2013, 113(8): 6816-6863. DOI:10.1021/cr3001059 |
| [41] | Huczyński A, Janczak J, ?owicki D, Brzezinski B. Monensin A acid complexes as a model of electrogenic transport of sodium cation. Biochimica et Biophysica Acta (BBA)- Biomembranes, 2012, 1818(9): 2108-2119. DOI:10.1016/j.bbamem.2012.04.017 |
