王宇, 赵洁, 孙志宏, 孙天松, 张和平


内蒙古农业大学乳品生物技术与工程教育部重点实验室, 农业农村部奶制品加工重点实验室, 内蒙古 呼和浩特 010018
收稿日期:2019-08-11;修回日期:2019-11-13;网络出版日期:2019-11-20
基金项目:国家自然科学基金(31430066,31771954)
*通信作者:张和平。Tel:+86-471-4300593;E-mail:hepingdd@vip.sina.com.
摘要:[目的] 嗜热链球菌(Streptococcus thermophilus)是发酵乳制品的基础发酵菌种之一,全基因组水平解析嗜热链球菌的遗传多样性和工业发酵特性对于优良发酵菌株的筛选意义重大。[方法] 本研究通过比较基因组学方法对27株嗜热链球菌的遗传多样性和防御系统进行分析。[结果] 全基因组分析结果显示嗜热链球菌群体内具有较高的遗传多样性;基于核心基因集构建的系统发育树划分为2个分支,其中分支2菌株缺乏完整的组氨酸合成途径,经验证,分支2菌株在缺乏组氨酸的培养基中不能正常生长。通过对嗜热链球菌不同菌株的防御系统进行分析发现,同类型的CRISPR基因座和限制修饰系统在基因组中出现的位置相对固定。CRISPR-Cas系统(P < 0.05,r=0.43)和限制修饰系统(P < 0.01,r=-0.59)的数量与编码转座酶基因的数量均显著相关,表明嗜热链球菌为了阻止外源DNA入侵会进化出多种防御系统来保护自身遗传完整性。此外,分支1菌株的CRISPR-Cas系统数量极显著(P < 0.001)多于分支2,而限制修饰系统无显著差异,表明分支1菌株在噬菌体抗性方面可能更具优势。[结论] 本研究基于核心基因构建的系统发育分析将27株嗜热链球菌分为2个分支,不同分支菌株在组氨酸代谢能力和防御系统方面有一定差异。该研究结果为今后快速筛选优良嗜热链球菌发酵剂提供了新思路。
关键词:嗜热链球菌比较基因组学遗传多样性CRISPR-Cas系统限制修饰系统
Comparative genomics of genetic diversity and defense system in Streptococcus thermophilus
Yu Wang, Jie Zhao, Zhihong Sun, Tiansong Sun, Heping Zhang


Key Laboratory of Dairy Biotechnology and Engineering, Ministry of Education, Key Laboratory of Dairy Products Processing, Ministry of Agriculture, Inner Mongolia Agricultural University, Hohhot 010018, Inner Mongolia Autonomous Region, China
Received: 11 August 2019; Revised: 13 November 2019; Published online: 20 November 2019
*Corresponding author: Heping Zhang, Tel:+86-471-4300593;E-mail:hepingdd@vip.sina.com.
Foundation item: Supported by the National Natural Science Foundation of China (31430066, 31771954)
Abstract: [Objective] Streptococcus thermophilus is one of the most commonly used strains in fermented dairy industry. Therefore, it is important to screen S. thermophilus with good fermentation properties. [Methods] The genetic diversity and defense systems of 27 S. thermophilus genomes were analyzed using comparative genomics. [Results] The genetic diversity of S. thermophilus was high based on whole genome analysis. The phylogenetic tree built based on the core genes was divided into two clades, and the strains in Clade 2 were lack of the complete histidine synthesis pathway, thus could not grow normally in the medium lacking histidine. The analysis of defense systems of S. thermophilus reveals the same type of CRISPR locus and restriction modification system was fixed in the genome relatively. The numbers of CRISPR-Cas (P < 0.05, r=0.43) and restriction modification systems (P < 0.01, r=-0.59) correlated significantly with the number of genes encoding transposases, indicating S. thermophilus has evolved multiple defense systems to protect its genetic integrity by preventing the invasion of exogenous DNA. In addition, the number of CRISPR-Cas system of the Clade 1 strains was significantly (P < 0.001) higher than the Clade 2 strains, whereas there was no significant difference in restriction modification systems. These results suggest that the Clade 1 strains had stronger capacity in resistance to phages. [Conclusion] The phylogenetic analysis based on the core genes was divided into two clades. There were some differences in histidine metabolism and defense system between the different clades, providing a new method for the rapid screening of S. thermophilus starters with excellent fermentation characteristics.
Keywords: Streptococcus thermophiluscomparative genomicsgenetic diversityCRISPR-Cas systemrestriction modification system
嗜热链球菌作为一种重要的工业发酵剂,是链球菌属中唯一公认的安全菌种[1],被广泛应用于各种发酵乳制品中。基因组学研究为解析嗜热链球菌在发酵过程中的许多关键生理功能提供了新的见解,全基因组序列有助于更好地了解嗜热链球菌的生产特性,如胞外多糖合成、代谢途径、产酸能力和防御系统等重要的工业表型性状[1-3]。
噬菌体污染是乳制品发酵过程中最常见的问题,它会导致产酸变慢和发酵失败,并造成巨大的经济损失[4]。因此,菌株抵抗噬菌体污染的能力成为优良商业发酵剂的筛选标准之一[5]。嗜热链球菌为了保护自身免受噬菌体的侵害进化出多种抗噬菌体机制,其中限制修饰系统(restricted modification system,R-M)和CRISPR-Cas系统都是通过专门切割进入宿主细胞的外源DNA来发挥作用[6]。CRISPR位点与噬菌体特异性获得性免疫的关系最早在嗜热链球菌中得到证实[7],其通过获得新的间隔序列以应对噬菌体攻击,具有较强CRISPR介导防御能力的嗜热链球菌菌株在乳制品发酵过程中抵抗噬菌体侵染的能力也较强[8]。此外,CRISPR-Cas系统具有对物种进行基因分型的潜力[9],特别是在优良发酵剂和益生菌的筛选方面[10-11]。与CRISPR-Cas系统不同,R-M系统是细菌生来就有的一种先天性免疫系统。Binetti等[5]对9株商业嗜热链球菌菌株的噬菌体抗性进行研究,发现R-M系统在细胞死亡之前就中断并除去噬菌体感染颗粒,是一种强大且有效的防御机制。此外,R-M系统的多样性对于细菌菌群之间遗传信息的传递非常重要[12]。
近年来,乳酸菌领域内关于遗传多样性和种群结构等微生物基础研究逐渐受到人们的重视。2018年,赵洁[7]对分离自自然发酵乳的185株Streptococcus thermophilus基因组进行多样性分析,结果显示分离自酸牦牛奶和酸牛奶的菌株遗传多样性更高。同年,宋宇琴[13]通过构建200株德氏保加利亚乳杆菌(Lactobacillus delbrueckii subsp. bulgaricus)的泛-核心基因集,发现随着基因组数量的增加,L. delbrueckii subsp. bulgaricus的泛基因集呈现增加的趋势,由此说明该物种具有较高的遗传多样性。基因组学是研究乳酸菌遗传多样性的重要工具,从全基因组水平上了解嗜热链球菌的遗传信息对菌株发酵特性的研究有重要作用。
现代测序技术使得微生物全基因组分析更加便捷。本研究中的菌株S. thermophilus ND07分离自青海地区自然发酵酸牦牛奶样品,本实验室前期的研究揭示S. thermophilus ND07较商业发酵菌株S. thermophilus YC-X11在发酵乳中具有弱后酸化、高黏度和高持水性的特性,具有成为一株优良商业化发酵菌株的潜力。本研究利用比较基因组学手段构建了27株嗜热链球菌的泛-核心基因集,并对其核心、附属和特异基因进行功能注释,为研究嗜热链球菌遗传多样性的研究提供基本思路;同时对嗜热链球菌重要的工业表型性状如氨基酸生物合成、限制性修饰系统以及CRISPR-Cas系统等进行深入分析,为工业生产中快速筛选优良乳品发酵剂提供借鉴。
1 材料和方法 1.1 试验材料 S. thermophilus ND07分离自青海地区自然发酵酸牦牛奶样品, 由内蒙古农业大学乳品生物技术与工程教育部重点实验室提供,其具体的全基因组DNA提取方法和全基因组序列参考钟智等[3]的文章。选择已完成全基因组测序的26株嗜热链球菌进行比较基因组学分析,菌株序列从NCBI数据库(https://www.ncbi.nlm.nih.gov/genome/?term=)下载。26株嗜热链球菌菌株具体信息见表 1。
表 1. 27株完全测序的嗜热链球菌基因组的基本信息表 Table 1. Summary of completely sequenced genomes of S. thermophilus strains
| Strains | Accession | Source | Size/Mb | GC/% | CDSs | rRNAs | tRNAs | ncRNAs | Pseudogenes | CRISPR-Cas system | Restriction- modification (RM) systems | Transposase |
| JM8232 | NC_017581 | Dairy (France) | 1.93 | 38.9 | 2033 | 18 | 67 | 4 | 196 | 2 | 4 | 42 |
| LMG18311 | NC_006448 | Yogurt (UK) | 1.80 | 39.1 | 1925 | 18 | 67 | 4 | 215 | 2 | 4 | 36 |
| CNRZ1066 | NC_006449 | Yogurt (UK) | 1.80 | 39.1 | 1936 | 18 | 67 | 4 | 209 | 1 | 4 | 36 |
| LMD-9 | NC_008532 | Danisco (USA) | 1.86 | 39.1 | 2000 | 18 | 67 | 4 | 230 | 3 | 2 | 52 |
| P1:NC_008500 | ||||||||||||
| P2:NC_008501 | ||||||||||||
| ND03 | NC_017563 | Yak milk (China) | 1.83 | 39.0 | 1968 | 15 | 57 | 4 | 200 | 3 | 2 | 43 |
| MN-ZLW-002 | NC_017927 | Yogurt block (China) | 1.85 | 39.1 | 1982 | 15 | 57 | 4 | 211 | 3 | 3 | 52 |
| ASCC1275 | NZ_CP006819 | ASCRC (Australia) | 1.85 | 39.1 | 1974 | 15 | 55 | 4 | 234 | 4 | 3 | 47 |
| SMQ-301 | NZ_CP011217 | Dairy | 1.86 | 39.1 | 1993 | 18 | 67 | 4 | 220 | 3 | 2 | 52 |
| MN-BM-A02 | NZ_CP010999 | Dairy Fan (China) | 1.85 | 39.0 | 1977 | 15 | 57 | 4 | 224 | 4 | 3 | 50 |
| MN-BM-A01 | NZ_CP012588 | Yogurt block (China) | 1.88 | 39.1 | 2023 | 18 | 67 | 4 | 273 | 3 | 2 | 52 |
| S9 | NZ_CP013939 | Dairy (China) | 1.79 | 39.1 | 1922 | 18 | 67 | 4 | 203 | 2 | 3 | 31 |
| KLDS SM | NZ_CP016026 | Yogurt (China) | 1.86 | 39.1 | 1984 | 18 | 67 | 4 | 224 | 4 | 3 | 49 |
| CS8 | NZ_CP016439 | Rubing (China) | 1.79 | 39.0 | 1924 | 15 | 57 | 4 | 207 | 1 | 3 | 33 |
| KLDS 3.1003 | NZ_CP016877 | Yogurt (China) | 1.90 | 38.9 | 2037 | 18 | 68 | 4 | 271 | 3 | 3 | 39 |
| ND07 | NZ_CP016394 | Yak milk (China) | 1.87 | 39.0 | 1996 | 15 | 57 | 4 | 236 | 4 | 3 | 50 |
| APC151 | NZ_CP019935 | Intestine (Ireland) | 1.84 | 39.1 | 1982 | 18 | 67 | 4 | 206 | 3 | 2 | 45 |
| ST3 | NZ_CP017064 | Commercial dietary | 1.87 | 39.0 | 1982 | 18 | 68 | 4 | 253 | 2 | 3 | 53 |
| supplements (South Korea) | ||||||||||||
| B59671 | NZ_CP022547 | Raw milk (USA) | 1.82 | 39.1 | 1925 | 18 | 67 | 4 | 269 | 2 | 2 | 42 |
| DGCC7710 | NZ_CP025216 | Dairy culture | 1.85 | 39.0 | 1962 | 15 | 56 | 4 | 230 | 4 | 2 | 51 |
| GABA | NZ_CP025399 | Milk (China) | 1.86 | 39.1 | 1952 | 18 | 68 | 4 | 241 | 2 | 3 | 42 |
| EPS | NZ_CP025400 | Milk (China) | 1.81 | 39.0 | 1937 | 18 | 67 | 4 | 240 | 2 | 3 | 32 |
| ST109 | NZ_CP031545 | Raw milk (USA) | 1.79 | 39.2 | 1906 | 18 | 67 | 4 | 246 | 3 | 2 | 37 |
| ST106 | NZ_CP031881 | Raw milk (USA) | 1.86 | 39.3 | 2006 | 18 | 67 | 4 | 324 | 2 | 1 | 68 |
| IDCC2201 | NZ_CP035306 | Cheese (South Korea) | 1.79 | 39.2 | 1916 | 18 | 67 | 4 | 203 | 3 | 3 | 31 |
| ACA-DC 2 | NZ_LT604076 | Yogurt (Greece) | 1.73 | 39.2 | 1847 | 15 | 56 | 4 | 217 | 1 | 4 | 26 |
| NCTC12958 | NZ_LS483339 | UK | 2.10 | 39.0 | 2237 | 15 | 56 | 4 | 308 | 2 | 3 | 51 |
| N4L | NZ_LS974444 | France | 1.83 | 39.1 | 1950 | 18 | 67 | 4 | 247 | 3 | 3 | 40 |
表选项
1.2 组氨酸缺陷试验 将S. thermophilus ND07、MN-BM-A01 (蒙牛乳业(集团)股份有限公司提供,菌粉)和EPS (光明乳业股份有限公司提供)以2%接种量接种于M17液体培养基(Oxoid Ltd.,Basingstoke,United Kingdom)中,42 ℃厌氧培养18 h,扩大培养至三代后将其以2%的接种量接种于添加(含量0.15 g/L)或不添加组氨酸的化学限定培养基(CDM)[14]中,42 ℃厌氧培养,从0 h开始每隔2 h在600 nm处测定OD值。
1.3 基因预测和注释 采用Prokka软件对菌株基因组序列进行基因预测[15],根据预测得到的CDS (编码序列)位置信息提取氨基酸序列,与NCBI非冗余蛋白数据库比对。氨基酸同源性判断阈值:(a) identity:≥60%;(b) E value≤1e-6;(c)比对上的序列长度大于总长度的90%。利用BLASTx对嗜热链球菌核心、附属和特异基因序列进行COG (同源基因簇)功能注释[16],E值截止值为1e-6。随后在KAAS网站[17]通过BBH (bi-directional best hit)方法对27株嗜热链球菌基因组中的蛋白质编码基因进行KEGG注释。
1.4 ANI值和TNI值的计算 平均核苷酸一致性(average nucleotide identity,ANI)不仅可以用来评估物种的遗传多样性程度,还可以用于判断菌株是否为同一个种或亚种[13]。总核苷酸一致性(total nucleotide identity,TNI)是对ANI进行优化之后的一种计算方法,其准确度更高。本研究采用Goris等[18]和Chen等[19]提出的方法来计算嗜热链球菌的ANI值和TNI值。
1.5 比较基因组分析 以S. thermophilus JIM8232为参考菌株,使用Mauve软件[20]对其余嗜热链球菌基因组进行共线性分析。
1.6 Core-Pan基因集及系统发育树的构建 基于前期Prokka软件的基因组预测结果,使用Roary软件(v3.6.1)[21]构建27株嗜热链球菌的泛-核心(Core-Pan)基因集。此外,利用MEGA7软件[22]基于核心基因序列构建邻接树,对嗜热链球菌的系统发育关系进行评估。
1.7 CRISPR-Cas系统和R-M系统分析 使用CRISPR Finder网络在线工具(https://crisprcas.i2bc.paris-saclay.fr/)识别嗜热链球菌基因组中的CRISPR-Cas系统[23];基于REBASE数据库(http://rebase.neb.com/rebase/rebase.html)预测基因组中的R-M系统[24]。并采用Spass软件根据Spearman参数进行相关性分析。
2 结果和分析 2.1 嗜热链球菌基因组的一般特点 27株嗜热链球菌全基因组长度平均为1.85±0.06 Mb,GC含量通常在39%左右。在基因组中共预测到1973±67个CDS,其中(12±1)%个基因突变为假基因。全基因组长度与GC含量显著负相关(P < 0.05,r=-0.44);与CDS数量、假基因数量极显著正相关(P < 0.01,r分别为0.98和0.51)。CDS数量与GC含量显著负相关(P < 0.05,r=-0.40),而与假基因数量极显著正相关(P < 0.01,r=0.52)。此外,除S. thermophilus LMD-9包含2个质粒外,其余嗜热链球菌菌株均不存在质粒。
进一步通过ANI和TNI对嗜热链球菌群体内的序列同源性进行评估。理论上两两序列ANI > 95%、TNI > 70%被视为同一物种。本研究以S. thermophilus JIM8232的全基因组序列为参考,计算了菌株两两间ANI和TNI值,并绘制了热图(图 1)。结果显示,27株嗜热链球菌菌株两两间ANI值均 > 98.27%,TNI值均 > 84.95%。
 |
| 图 1 27株嗜热链球菌的ANI (A)及TNI (B)结果 Figure 1 Heatmap of ANI (A) and TNI (B) based on the sequences of 27 S. thermophilus. |
| 图选项 |
2.2 嗜热链球菌的比较基因组分析 以S. thermophilus JIM8232作为参考序列,通过Mauve软件分析嗜热链球菌基因组之间的保守性和差异性。如图 2所示,嗜热链球菌菌种内遗传稳定,突变重组较少。27株嗜热链球菌菌株中,仅在S. thermophilus MN-BM-A01和MN-ZLW-002中观察到小范围的倒位,而其余菌株仅发生小的插入、缺失或重排等现象,表明这些菌株在进化过程中只发生过小范围的基因重组和转移。通过对发生插入和缺失的区域进行比对,发现这些区域携带编码各种蛋白质的基因,包括假设蛋白质、应激蛋白、噬菌体相关蛋白和生物合成相关蛋白质,这些蛋白质小范围的基因缺失与插入可以使基因组结构多样化,从而有助于基因组获得一些有用的工业表型性状[25-26]。
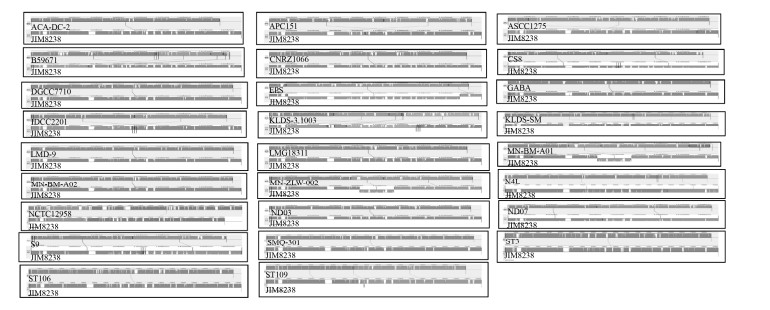 |
| 图 2 嗜热链球菌的共线性分析 Figure 2 Synteny analysis of S. thermophilus using the genome of strain JIM8232 as the reference. |
| 图选项 |
2.3 泛-核心(pan-core)基因集的构建 27株嗜热链球菌的泛基因集包含4139个基因,其中1192个基因为27株菌所共有,构成了嗜热链球菌的核心基因集;其余2947个非核心基因集中,包括1734个附属基因和1213个特异基因。由图 3-A可知,随着基因组数量的增加,核心基因的个数逐渐趋于稳定,而泛基因的个数仍呈现增加的趋势,说明嗜热链球菌基因组为开放式基因组,同时也说明嗜热链球菌具有较高的遗传多样性。
 |
| 图 3 嗜热链球菌遗传多样性分析 Figure 3 Analysis of genetic diversity of S. thermophilus. A: The trend chart of the set of core-pan genes; B: Annotations of core gene, accessory gene and unique gene based on the COG database. COG category: A: RNA processing and modification; J: translation, ribosomal structure and biogenesis; K: transcription; L: replication, recombination and repair; C: energy production and conversion; G: carbohydrate transport and metabolism; E: amino acid transport and metabolism; F: nucleotide transport and metabolism; H: coenzyme transport and metabolism; I: lipid transport and metabolism; P: inorganic ion transport and metabolism; Q: secondary metabolites biosynthesis, transport and catabolism; D: cell cycle control, cell division, chromosome partitioning; M: cell wall/membrane/envelope biogenesis; O: posttranslational modification, protein turnover, chaperones; T: signal transduction mechanisms; U: intracellular trafficking, secretion, and vesicular transport; V: defense mechanisms; R: general function prediction only; S: function unknown; N: cell motility; Z: cytoskeleton. |
| 图选项 |
利用COG数据库对27株嗜热链球菌的核心、附属和特异基因进行功能注释,结果如图 3-B所示。代谢相关基因主要在核心基因中富集,所占比例达到41%,其中E类“氨基酸转运和代谢”的基因数量最多,占核心基因的15.22%。M类“细胞壁/膜/包膜生物合成”和O类“翻译后修饰,蛋白质折叠,伴侣蛋白”与特定环境中的适应性或相互作用有关,其在核心基因中的富集表明这些基因对于嗜热链球菌适应牛奶丰富的营养环境至关重要。附属基因中L类“复制、重组和修复”所占的比例最大,为30.67%;V类“防御机制”主要在附属基因和特异基因中富集,说明某些菌株中可能存在特定的防御机制。
2.4 系统发育分析 为了研究27株嗜热链球菌的群体结构,本研究基于1192个核心基因的核酸序列采用邻接法构建了系统发育树,bootstrap值为1000。如图 4所示,27株嗜热链球菌可划分为2个较为清晰的分支。为进一步了解嗜热链球菌2个分支代谢能力的差异,分别对其核心基因进行KEGG注释(表 2)。通过对2个分支KEGG注释通路的比较发现,分支1和分支2共有22个差异代谢通路,分支1基本涵盖了分支2的所有代谢通路;22个有差异的代谢通路中分支1有67个特异性基因,而分支2仅有6个特异性基因。分支1中的67个特异性基因与次级代谢产物、抗生素和氨基酸的生物合成、碳代谢、半胱氨酸和蛋氨酸代谢、组氨酸代谢以及其他生物合成或代谢途径密切相关,其中参与组氨酸生物合成的基因多达41个。与分支1相比,组成组氨酸操纵子的11个基因hisG、hisZ、hisE、hisI、hisA、hisF、hisH、hisB、hisC、hisK和hisD中仅基因hisK是分支2菌株所共有的,表明分支2菌株可能因缺失编码组氨酸途径多种酶的基因而无法合成组氨酸。因此,选取分支1中的代表菌株S. thermophilus ND07和MN-BM-A01,分支2代表菌株S. thermophilus EPS验证其在组氨酸缺乏情况下的生长情况,结果表明上述3株菌株在含有组氨酸的情况下生长无明显差异(图 5-A);而在组氨酸缺乏的情况下,S. thermophilus EPS表现出明显的生长缺陷(图 5-B),该结果与基因组分析结果一致。此外,在14株嗜热链球菌菌株中检测到胞外蛋白酶prtS基因,并且这14株菌均属于分支1。
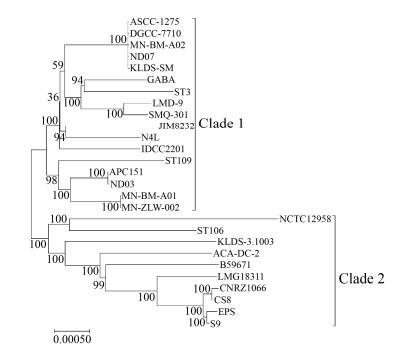 |
| 图 4 27株嗜热链球菌的系统发育关系 Figure 4 Phylogenetic tree constructed based on core genome of 27 S. thermophilus strains. |
| 图选项 |
表 2. 不同分支的嗜热链球菌菌株特异性基因分布 Table 2. Pathway annotation of genes specific to S. thermophilus strains from two clades
| Pathway number | Pathway annotation | No. of unique enzyme in Clade 1 | No. of unique enzyme in Clade 2 |
| ko01100 | Metabolic pathways | 14 | 1 |
| ko01110 | Biosynthesis of secondary metabolites | 12 | 0 |
| ko01130 | Biosynthesis of antibiotics | 2 | 0 |
| ko01230 | Biosynthesis of amino acids | 13 | 0 |
| ko01120 | Microbial metabolism in diverse environments | 1 | 0 |
| ko02010 | ABC transporters | 2 | 2 |
| ko01200 | Carbon metabolism | 1 | 0 |
| ko00270 | Cysteine and methionine metabolism | 2 | 0 |
| ko02020 | Two-component system | 1 | 1 |
| ko00400 | Phenylalanine, tyrosine and tryptophan biosynthesis | 1 | 0 |
| ko00260 | Glycine, serine and threonine metabolism | 1 | 0 |
| ko00300 | Lysine biosynthesis | 1 | 0 |
| ko00030 | Pentose phosphate pathway | 1 | 0 |
| ko03060 | Protein export | 1 | 0 |
| ko03070 | Bacterial secretion system | 1 | 0 |
| ko00450 | Selenocompound metabolism | 1 | 0 |
| ko00350 | Tyrosine metabolism | 1 | 0 |
| ko00401 | Novobiocin biosynthesis | 1 | 0 |
| ko00960 | Tropane, piperidine and pyridine alkaloid biosynthesis | 1 | 0 |
| ko00340 | Histidine metabolism | 9 | 0 |
| ko00730 | Thiamine metabolism | 0 | 1 |
| ko04122 | Sulfur relay system | 0 | 1 |
表选项
 |
| 图 5 嗜热链球菌在不同CDM (添加/不添加组氨酸)的生长曲线 Figure 5 Growth curve of S. thermophilus in different CDM (with/without addition of histidine). A: Strains cultured in complete CDM medium; B: Strains cultured in the CDM medium lacking histidine. |
| 图选项 |
2.5 CRISPR-Cas系统分析 通过CRISPR Finder对27株嗜热链球菌的CRISPR-Cas系统进行分析(图 6-A),发现嗜热链球菌菌株含有四种不同的CRISPR-Cas系统(CRISPR1、CRISPR2、CRISPR3和CRISPR4),其中CRISPR1-Cas和CRISPR3-Cas系统均为Ⅱ-A型,CRISPR2-Cas系统为Ⅲ-A型,CRISPR4-Cas系统为Ⅰ-E型。除S. thermophilus ACA-DC-2仅含有一个Ⅲ-A型CRISPR-Cas系统外,其余嗜热链球菌基因组均含有至少一个Ⅱ-A型CRISPR-Cas系统。
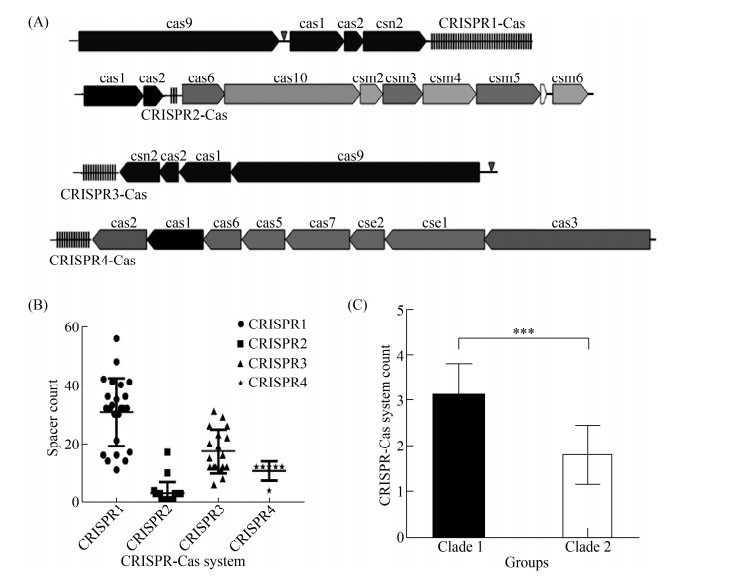 |
| 图 6 嗜热链球菌的CRISPR-Cas系统 Figure 6 CRISPR-Cas system of S. thermophilus. A: Structural diagram of CRISPR-Cas system of S. thermophilus; B: Number of S. thermophilus CRISPR spacers; C: Correlation analysis of two clades of CRISPR-Cas System. ***: P < 0.001. |
| 图选项 |
有趣的是,在含有较少CRISPR-Cas系统的菌株中检测到退化的CRISPR重复序列,S. thermophilus JIM8232中发现退化的CRISPR3重复序列,S. thermophilus B59671中发现退化的CRISPR1重复序列,以及S. thermophilus GABA和ST3中发现退化的CRISPR2重复序列。退化的CRISPR3重复序列位于基因组序列的上游,而重复序列位于基因组的下游,这可能是由于末端重复序列和退化的CRISPR3重复序列之间发生了重组事件,进而造成CRISPR3相关cas基因片段的缺失或插入。相反,退化的CRISPR1和CRISPR2重复序列都位于基因组序列的下游,表明这2个CRISPR系统退化的主要原因可能就是相关cas基因的丢失。
通过比较发现,4个CRISPR基因座在基因组中的位置相对固定。通常,CRISPR1上游的编码基因是假定蛋白,下游是磷酸丝氨酸磷酸酶的编码基因SerB;CRISPR2上游的编码基因是二氢乳清酸脱氢酶B催化亚基,下游的编码基因是乳清酸核苷5′-磷酸脱羧酶;而CRISPR3和CRISPR4上下游的编码基因均为假定蛋白。
S. thermophilus ND07、DGCC-7710、KLDS-SM、MN-BM-A02和ASCC-1275包含全部四种CRISPR-Cas系统,表明它们具有更好的适应性免疫力,在乳品发酵中可以更好的抵御噬菌体侵染。每个CRISPR-Cas系统在不同菌株中都拥有相同的重复序列和cas基因,这表明其抵抗噬菌体的防御机制在同一物种中可能是相似的。然而,CRISPR-Cas系统中的间隔序列具有多态性,间隔序列的数目可以在一定程度上反映该CRISPR基因座的活跃程度。如图 6-B所示,CRISPR1中间隔序列数目的最大值和平均值均为最高,说明嗜热链球菌中CRISPR1最为活跃,CRISPR3次之,CRISPR2的平均值最低,说明嗜热链球菌中CRISPR2活性最低。
此外,在27株嗜热链球菌菌株中注释到大量编码转座酶的基因,这些基因的数量与菌株中CRISPR-Cas系统的数量显著正相关(P < 0.05,r=0.43)。通过对不同分支菌株中所含的CRISPR-Cas系统数量进行比较(图 6-C),发现分支1中菌株的CRISPR-Cas系统数量极显著高于分支2,推测分支1菌株可能具有较强的抵抗噬菌体污染的能力。
2.6 限制修饰系统分析 将27株嗜热链球菌的基因组序列与REBASE数据库进行比较,嗜热链球菌基因组与R-M系统相关的基因数量如表 3所示。S. thermophilus ACA-DC-2、CNRZ1066、JIM8232和LMG 18311包含全部4种R-M系统,却仅包含1-2种CRISPR-Cas系统,由此说明菌株中CRISPR-cas系统的低活性可以通过R-M系统来弥补[27],但是CRISPR-cas系统和R-M系统并没有明显的相关性。
表 3. 嗜热链球菌各个R-M系统的基因数 Table 3. Number of genes in R-M systems found in the S. thermophilus strains
| Strains | Type Ⅰ | Type Ⅱ | Type Ⅲ | Type Ⅳ |
| APC151 | 3 | 6 | 0 | 0 |
| ASCC1275 | 8 | 0 | 1 | 1 |
| DGCC7710 | 9 | 0 | 0 | 1 |
| GABA | 6 | 5 | 2 | 0 |
| IDCC2201 | 3 | 0 | 4 | 1 |
| JIM8232 | 9 | 2 | 2 | 1 |
| KLDS SM | 7 | 0 | 1 | 1 |
| LMD-9 | 5 | 6 | 0 | 0 |
| MN-BM-A01 | 6 | 6 | 0 | 0 |
| MN-BM-A02 | 9 | 0 | 1 | 1 |
| MN-ZLW-002 | 5 | 4 | 1 | 0 |
| N4L | 7 | 4 | 2 | 0 |
| ND03 | 6 | 6 | 0 | 0 |
| ND07 | 8 | 0 | 1 | 1 |
| SMQ-301 | 4 | 4 | 0 | 0 |
| ST109 | 6 | 0 | 0 | 1 |
| ST3 | 3 | 2 | 0 | 1 |
| ST106 | 6 | 0 | 0 | 0 |
| ACA-DC-2 | 3 | 2 | 2 | 1 |
| EPS | 6 | 2 | 2 | 0 |
| KLDS 3.1003 | 6 | 1 | 0 | 1 |
| LMG 18311 | 8 | 4 | 2 | 1 |
| S9 | 3 | 2 | 2 | 0 |
| B59671 | 0 | 3 | 1 | 0 |
| CNRZ1066 | 8 | 5 | 2 | 2 |
| CS8 | 7 | 4 | 2 | 0 |
| NCTC12958 | 6 | 2 | 2 | 0 |
表选项
嗜热链球菌基因组共包含4种R-M系统,除Ⅰ型R-M系统外,其余3种R-M系统在基因组中的位置相对固定。Ⅰ型R-M系统是目前已知的最复杂的一种类型,由DNA甲基转移酶(HsdM)、限制性内切酶(HsdR)和特异性序列绑定识别亚基(HsdS)作为一个蛋白复合体行使限制-修饰功能[28]。本研究除S. thermophilus B59671没有该系统外,其余菌株都包含1-3个完整的Ⅰ型R-M系统,部分嗜热链球菌中有1个R-M系统因其中1个或2个基因发生移码突变而失去功能活性。Ⅱ型R-M系统在细菌中最为普遍,本研究有19株菌包含编码Ⅱ型R-M系统的基因,S. thermophilus JIM8232、KLDS 3.1003和NCTC12958仅存在DNA甲基转移酶,不构成完整的Ⅱ型R-M系统,其余菌株都包含1-3个完整的Ⅱ型R-M系统。Ⅱ型R-M系统的编码基因主要位于假定蛋白、赖氨酸连接酶、UDP-N-乙酰葡糖胺2-差向异构酶、果糖-1, 6-二磷酸醛缩酶和核酸切割酶附近。本研究中共有17株菌有编码Ⅲ型R-M系统的基因,其由mod基因编码的DNA甲基转移酶和由res基因编码的限制性内切酶构成,前者存在于所有菌株中,而后者在6株菌中是缺失的,即这6株菌仅包含一个孤儿甲基转移酶,并没有完整的Ⅲ型R-M系统。此外,S. thermophilus EPS、S9、KLDS SM和ASCC 1275中的DNA甲基转移酶突变为假基因,无实际功能活性。Ⅲ型R-M系统的编码基因通常出现在假定蛋白、谷氨酰胺水解酶和乙醇脱氢酶附近。共有13株菌有IV型R-M系统,其编码基因的上游通常是DNA错配修复蛋白或者DNA错配修复蛋白的水解产物核苷三磷酸,下游通常是转座酶或者假定蛋白,只有S. thermophilus ST3、ST109和IDCC2201这3株菌的下游是赖氨酸连接酶。S. thermophilus ND07发生倒位,其编码基因的上游是转座酶,下游是DNA错配修复蛋白。
此外,与CRISPR-Cas系统不同,菌株中R-M系统的数量与编码转座酶基因的数量极显著负相关(P < 0.01,r=-0.59)。通过对不同分支菌株中所含的R-M系统数量进行比较(图 6-C),发现两个分支的R-M系统数量无显著性差异,说明不同分支嗜热链球菌菌株的先天防御能力大致相同。
3 讨论 全基因组测序有助于基因组组装成完整的基因组序列,从而获得更加准确和深入的基因组注释信息。嗜热链球菌作为自然发酵乳中的关键菌株,其丰富的基因组信息对食品工业中选择合适的嗜热链球菌发酵剂至关重要。
系统发育分析结果将27株嗜热链球菌划分为2个分支,通过对两个分支代谢能力差异的比较发现,分支1和分支2菌株在多数的氨基酸合成方面能力大致相同,但是在组氨酸合成途径以及与组氨酸生物合成相关的基因方面存在较大差异,分支2菌株因缺失编码组氨酸途径多种酶的基因而无法合成组氨酸,该结果与李柏良等[29]分析的结果类似。Fontaine等[30]通过对S. thermophilus LMD-9和LMG18311插入或缺失组氨酸生物合成位点的研究表明,在组氨酸存在时二者的生长情况相似,而在组氨酸不存在时S. thermophilus LMG18311生长明显缓慢。Pastink等[31]对S. thermophilus LMG18311的多种氨基酸缺陷型试验表明,该菌株仅在缺乏组氨酸时不生长,并且基因预测结果表示S. thermophilus LMG18311包含了除组氨酸外所有氨基酸生物合成所需酶的编码基因。本研究在添加或不添加组氨酸的情况下对分支1菌株S. thermophilus ND07、MN-BM-A01和分支2菌株S. thermophilus EPS的生长情况进行测定,发现S. thermophilus EPS在缺乏组氨酸的情况下表现出明显的生长缺陷。S. thermophilus EPS和LMG18311的组氨酸缺陷试验结果都与基因组预测结果一致,由此表明分支2菌株在组氨酸代谢能力方面具有一定的缺陷性。胞外蛋白酶PrtS可以分解乳蛋白产生肽类和氨基酸,使嗜热链球菌能够快速生长[32],但只存在于少数菌株中。本研究27株嗜热链球菌中有14株菌株检测到胞外蛋白酶PrtS的编码基因,而这14株菌均属于分支1,由此可推测分支1菌株在生长性能方面可能更具有优势。
27株嗜热链球菌的泛基因集包含4139个基因,具体包括1192个核心基因、1734个附属基因和1213个特有基因。随着基因组数量的增加,嗜热链球菌的泛基因集呈现上升的趋势,说明嗜热链球菌的遗传物质具有开放性,同时也说明嗜热链球菌具有较高的遗传多样性。此外,嗜热链球菌的开放性基因组可能是由于进化过程中基因的插入或缺失导致的[33]。对核心、附属和特异基因进行COG功能注释,发现L类“复制,重组和修复”主要在附属基因中富集,这可能是由转座酶等移动遗传元件引起的[34]。在27株嗜热链球菌菌株中注释到大量编码转座酶的基因,这些基因的数量与菌株中CRISPR-Cas系统的数量显著正相关(P < 0.05,r=0.43),说明嗜热链球菌进化出多种防御系统是为了在各种移动遗传元件的增殖中存活并维持其遗传完整性[35]。
嗜热链球菌在工业生产中应用时需要面对噬菌体、低酸、高温等各种环境胁迫,其中噬菌体污染是工业发酵中最常见的难题之一。在本研究中,CRISPR1-Cas系统在嗜热链球菌基因组中普遍存在,推测CRISPR1-Cas系统可能在嗜热链球菌中形成一种主要且有效的噬菌体防御机制。此外,CRISPR1-Cas系统中间隔序列数目的最大值和平均值均为最高,其次是CRISPR3-Cas,由此表明新型间隔序列可能在这2个CRISPR-Cas系统中更加频繁地插入,而且当CRISPR1-Cas和CRISPR3-Cas的2个系统共存于同一基因组中时,增强了嗜热链球菌对噬菌体感染的抗性[36]。Hidalgo-Cantabrana等[9]对66株长双歧杆菌的CRISPR-Cas多样性进行分析,发现存在于长双歧杆菌中的CRISPR-Cas系统可作为基因分型的遗传工具,具有CRISPR-Cas系统的菌株是合适的益生菌候选物,这些菌株不仅能增强它们在人体肠道中的存活能力,还能增强对人体肠道有害菌的抗性[37]。
R-M系统存在于超过90%的细菌和古细菌中,是研究最充分的噬菌体防御机制[38],但是嗜热链球菌中R-M系统的研究有限。本研究中最为复杂的Ⅰ型R-M系统在嗜热链球菌中普遍存在,其可能是嗜热链球菌中拮抗噬菌体侵染的重要系统。R-M系统作为一种经典的防御机制,不仅能防止内源DNA降解,还能防止外源DNA如转座子、插入序列、噬菌体和质粒等可移动遗传元件的入侵[39]。与CRISPR-Cas系统不同,菌株中R-M系统的数量与编码转座酶基因的数量极显著负相关(P < 0.01,r=-0.59),说明R-M系统可能通过阻止外源DNA的入侵,而起到保护嗜热链球菌自身遗传物质稳定的作用。
4 结论 本研究从全基因组水平对嗜热链球菌的遗传多样性进行研究,结果显示随着基因组数量的增加,嗜热链球菌的泛基因集仍呈现增加的趋势,说明该物种群体内具有较高的遗传多样性。基于核心基因组序列构建的系统发育树共分为两个分支,不同分支嗜热链球菌菌株的氨基酸合成能力基本相同,仅分支2菌株缺乏多种编码组氨酸生物合成酶的基因,由此表明分支2菌株在组氨酸代谢能力方面具有一定的缺陷性。通过对嗜热链球菌基因组中与环境防御相关的CRISPR-Cas系统和R-M系统进行分析,发现同类型的CRISPR位点和R-M系统在基因组中出现的位置相对固定。此外,分支1菌株的CRISPR-Cas系统数量极显著高于分支2 (P < 0.001),推测分支1菌株可能具有较强的抵抗噬菌体污染的能力;而R-M系统无显著差异,说明不同分支嗜热链球菌菌株的先天防御能力大致相同。
References
| [1] | Goh YJ, Goin C, O'Flaherty S, Altermann E, Hutkins R. Specialized adaptation of a lactic acid bacterium to the milk environment:the comparative genomics of Streptococcus thermophilus LMD-9. Microbial Cell Factories, 2011, 10(1): S22. DOI:10.1186/1475-2859-10-22 |
| [2] | Hols P, Hancy F, Fontaine L, Grossiord B, Prozzi D, Leblond-Bourget N, Decaris B, Bolotin A, Delorme C, Ehrlich SD, Guédon E, Monnet V, Renault P, Kleerebezem M. New insights in the molecular biology and physiology of Streptococcus thermophilus revealed by comparative genomics. FEMS Microbiology Reviews, 2005, 29(3): 435-463. |
| [3] | Zhong Z, Sun TS, Chen YF. Genomic insights into the high exopolysaccharides-producing bacterium Streptococcus thermophilus ND-07. China Dairy Industry, 2018, 46(4): 9-11, 21. (in Chinese) 钟智, 孙天松, 陈永福. 基因组分析揭示Streptococcus thermophilus ND-07富产胞外多糖分子机制. 中国乳品工业, 2018, 46(4): 9-11, 21. |
| [4] | Li W, Wang NN, Zhang DQ, Huo GC. CRISPR detection and protospacer prediction in Streptococcus thermophilus. Modern Food Science and Technology, 2016, 32(10): 252-258. (in Chinese) 李婉, 王娜娜, 张丹青, 霍贵成. 嗜热链球菌CRISPR序列的检测及原间隔序列预测. 现代食品科技, 2016, 32(10): 252-258. |
| [5] | Binetti AG, Suárez VB, Tailliez P, Reinheimer JA. Characterization of spontaneous phage-resistant variants of Streptococcus thermophilus by randomly amplified polymorphic DNA analysis and identification of phage-resistance mechanisms. International Dairy Journal, 2007, 17(9): 1115-1122. DOI:10.1016/j.idairyj.2007.01.007 |
| [6] | Dupuis Mè, Villion M, Magadán AH, Moineau S. CRISPR-Cas and restriction-modification systems are compatible and increase phage resistance. Nature Communications, 2013, 4: 2087. DOI:10.1038/ncomms3087 |
| [7] | 赵洁.自然发酵乳中嗜热链球菌群体遗传学和功能基因组学研究.内蒙古农业大学博士学位论文, 2018. http://cdmd.cnki.com.cn/Article/CDMD-10129-1018881781.htm |
| [8] | Vale PF, Lafforgue G, Gatchitch F, Gardan R, Moineau S, Gandon S. Costs of CRISPR-Cas-mediated resistance in Streptococcus thermophilus. Proceedings of the Royal Society B:Biological Sciences, 2015, 282(1812): 20151270. DOI:10.1098/rspb.2015.1270 |
| [9] | Hidalgo-Cantabrana C, Crawley AB, Sanchez B, Barrangou R. Characterization and exploitation of CRISPR loci in Bifidobacterium longum. Frontiers in Microbiology, 2017, 26(8): 1851. |
| [10] | Briner AE, Barrangou R. Deciphering and shaping bacterial diversity through CRISPR. Current Opinion in Microbiology, 2016, 31: 101-108. DOI:10.1016/j.mib.2016.03.006 |
| [11] | Hidalgo-Cantabrana C, O'Flaherty S, Barrangou R. CRISPR-based engineering of next-generation lactic acid bacteria. Current Opinion in Microbiology, 2017, 37: 79-87. DOI:10.1016/j.mib.2017.05.015 |
| [12] | Humbert O, Dorer MS, Salama NR. Characterization of Helicobacter pylori factors that control transformation frequency and integration length during inter-strain DNA recombination. Molecular Microbiology, 2011, 79(2): 387-401. DOI:10.1111/j.1365-2958.2010.07456.x |
| [13] | 宋宇琴.德氏乳杆菌保加利亚亚种的群体遗传学和功能基因组学研究.内蒙古农业大学博士学位论文, 2018. http://cdmd.cnki.com.cn/Article/CDMD-10129-1018881732.htm |
| [14] | Letort C, Juillard V. Development of a minimal chemically-defined medium for the exponential growth of Streptococcus thermophilus. Journal of Applied Microbiology, 2001, 91(6): 1023-1029. DOI:10.1046/j.1365-2672.2001.01469.x |
| [15] | Seemann T. Prokka:rapid prokaryotic genome annotation. Bioinformatics, 2014, 30(14): 2068-2069. DOI:10.1093/bioinformatics/btu153 |
| [16] | Tatusov RL, Galperin MY, Natale DA, Koonin EV. The COG database:a tool for genome-scale analysis of protein functions and evolution. Nucleic Acids Research, 2000, 28(1): 33-36. |
| [17] | Moriya Y, Itoh M, Okuda S, Yoshizawa AC, Kanehisa M. KAAS:an automatic genome annotation and pathway reconstruction server. Nucleic Acids Research, 2007, 35(S2): W182-W185. |
| [18] | Goris J, Konstantinidis KT, Klappenbach JA, Coenye T, Vandamme P, Tiedje JM. DNA-DNA hybridization values and their relationship to whole-genome sequence similarities. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(1): 81-91. DOI:10.1099/ijs.0.64483-0 |
| [19] | Chen JP, Yang XW, Chen JW, Cen Z, Guo CY, Jin T, Cui YJ. SISP:a fast species identification system for prokaryotes based on total nucleotide identity of whole genome sequences. Infectious Diseases and Translational Medicine, 2015, 1(1): 30-55. |
| [20] | Darling ACE, Mau B, Blattner FR, Perna NT. Mauve:multiple alignment of conserved genomic sequence with rearrangements. Genome Research, 2004, 14(7): 1394-1403. DOI:10.1101/gr.2289704 |
| [21] | Page AJ, Cummins CA, Hunt M, Wong VK, Reuter S, Holden MTG, Fookes M, Falush D, Keane JA, Parkhill J. Roary:rapid large-scale prokaryote pan genome analysis. Bioinformatics, 2015, 31(22): 3691-3693. DOI:10.1093/bioinformatics/btv421 |
| [22] | Kumar S, Stecher G, Tamura K. MEGA7:Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [23] | Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats. BMC Bioinformatics, 2007, 8: 172. DOI:10.1186/1471-2105-8-172 |
| [24] | Roberts RJ, Vincze T, Posfai J, Macelis D. REBASE——a database for DNA restriction and modification:enzymes, genes and genomes. Nucleic Acids Research, 2015, 43(D1): D298-D299. DOI:10.1093/nar/gku1046 |
| [25] | Nishio Y, Nakamura Y, Usuda Y, Sugimoto S, Matsui K, Kawarabayasi Y, Kikuchi H, Gojobori T, Ikeo K. Evolutionary process of amino acid biosynthesis in Corynebacterium at the whole genome level. Molecular Biology and Evolution, 2004, 21(9): 1683-1691. DOI:10.1093/molbev/msh175 |
| [26] | Desiere F, Lucchini S, Brüssow H. Evolution of Streptococcus thermophilus bacteriophage genomes by modular exchanges followed by point mutations and small deletions and insertions. Virology, 1998, 241(2): 345-356. |
| [27] | Alexandraki V, Kazou M, Blom J, Pot B, Tsakalidou E, Papadimitriou K. The complete genome sequence of the yogurt isolate Streptococcus thermophilus ACA-DC 2. Standards in Genomic Sciences, 2017, 12: 18. DOI:10.1186/s40793-017-0227-5 |
| [28] | 薛花.两个新型结核杆菌DNA甲基化酶的鉴定和性质初步分析.中国科学院北京基因组研究所硕士学位论文, 2015. |
| [29] | Li BL, Ding XY, Jin D, Liu F, Meng YY, Li N, Zhao L, Huo GC. Genomic studies of proteolysis system and amino acid biosynthesis pathway in Streptococcus thermophilus KLDS SM. Food Science, 2018, 39(18): 120-126. (in Chinese) 李柏良, 丁秀云, 靳妲, 刘飞, 蒙月月, 李娜, 赵莉, 霍贵成. 基于基因组学分析嗜热链球菌KLDS SM的蛋白质水解系统和氨基酸合成途径. 食品科学, 2018, 39(18): 120-126. |
| [30] | Fontaine L, Dandoy D, Boutry C, Delplace B, De Frahan MH, Fremaux C, Horvath P, Boyaval P, Hols P. Development of a versatile procedure based on natural transformation for marker-free targeted genetic modification in Streptococcus thermophilus. Applied and Environmental Microbiology, 2010, 76(23): 7870-7877. DOI:10.1128/AEM.01671-10 |
| [31] | Pastink MI, Teusink B, Hols P, Visser S, de Vos WM, Hugenholtz J. Genome-scale model of Streptococcus thermophilus LMG18311 for metabolic comparison of lactic acid bacteria. Applied and Environmental Microbiology, 2009, 75(11): 3627-3633. DOI:10.1128/AEM.00138-09 |
| [32] | Tian H, Liang HZ, Huo GC, Etareri ES. Research progress on the property and application of Streptococcus thermophilus. Biotechnology Bulletin, 2015, 31(9): 38-48. (in Chinese) 田辉, 梁宏彰, 霍贵成, Etareri ES. 嗜热链球菌的特性与应用研究进展. 生物技术通报, 2015, 31(9): 38-48. |
| [33] | Sun ZH, Harris HMB, McCann A, Guo CY, Argimón S, Zhang WY, Yang XW, Jeffery IB, Cooney JC, Kagawa TF, Liu WJ, Song YQ, Salvetti E, Wrobel A, Rasinkangas P, Parkhill J, Rea MC, O'Sullivan O, Ritari J, Douillard FP, Ross RP, Yang RF, Briner AE, Felis GE, de Vos WM, Barrangou R, Klaenhammer TR, Caufield PW, Cui YJ, Zhang HP, O'Toole PW. Expanding the biotechnology potential of lactobacilli through comparative genomics of 213 strains and associated genera. Nature Communications, 2015, 6: 8322. DOI:10.1038/ncomms9322 |
| [34] | Schmid M, Muri J, Melidis D, Varadarajan AR, Somerville V, Wicki A, Moser A, Bourqui M, Wenzel C, Eugster-Meier E, Frey JE, Irmler S, Ahrens CH. Comparative genomics of completely sequenced Lactobacillus helveticus genomes provides insights into strain-specific genes and resolves metagenomics data down to the strain level. Frontiers in Microbiology, 2018, 9: 63. DOI:10.3389/fmicb.2018.00063 |
| [35] | Makarova KS, Wolf YI, Koonin EV. Comparative genomics of defense systems in archaea and bacteria. Nucleic Acids Research, 2013, 41(8): 4360-4377. DOI:10.1093/nar/gkt157 |
| [36] | Magadán AH, Dupuis M-è, Villion M, Moineau S. Cleavage of phage DNA by the Streptococcus thermophilus CRISPR3-Cas system. PLoS One, 2012, 7(7): e40913. DOI:10.1371/journal.pone.0040913 |
| [37] | Gogleva AA, Gelfand MS, Artamonova II. Comparative analysis of CRISPR cassettes from the human gut metagenomic contigs. BMC Genomics, 2014, 15: 202. DOI:10.1186/1471-2164-15-202 |
| [38] | Stern A, Sorek R. The phage-host arms race:shaping the evolution of microbes. Bioessays, 2011, 33(1): 43-51. DOI:10.1002/bies.201000071 |
| [39] | Gorrell R, Kwok T. The Helicobacter pylori methylome:roles in gene regulation and virulence. Current Topics in Microbiology and Immunology, 2017, 400: 105-127. |
