张艳军1#, 陈宝婷1#, 张玉梅1, 张逸飞1, 朱宝琳1, 苏盼1, 丁林贤2, 谢乃钧3, 张萍华1


1. 浙江师范大学化学与生命科学学院, 浙江 金华 321004;
2. 浙江师范大学地理与环境科学学院, 浙江 金华 321004;
3. 广西靖西鑫晟茧丝绸科技有限公司, 广西 靖西 533800
收稿日期:2020-01-08;修回日期:2020-03-16;网络出版日期:2020-03-27
基金项目:浙江省自然科学基金(LY19C130002);浙江省肠道微生态重点实验室开放课题(2019KLGM011)
*通信作者:张萍华, Tel:+86-579-82288003;E-mail:zph@zjnu.cn.
#并列第一作者
摘要:[目的] 解析广西蚕沙中细菌群落组成与多样性,为蚕沙中菌种资源发掘和蚕沙的综合利用提供科学依据。[方法] 通过高通量测序技术研究细菌群落组成特征,同时利用常规稀释涂布平板法和基于复苏促进因子(Rpf)的MPN培养系统解析并筛选蚕沙中可培养和活的非可培养(VBNC)状态的优势菌群,并经16S rRNA基因测序对筛选得到的菌株作初步分类鉴定。[结果] 高通量测序表明,广西蚕沙样品中细菌归属于10个门、18个纲、27个目、57个科、96个属,其中4个属的丰度达1%以上,优势菌群为变形菌门(Proteobacteria)肠杆菌属(Enterobacter);通过稀释涂布平板法共获得14个属的33株可培养细菌,其中4个属(Citrobacter、Weissella、Chitinophaga、Pseudoclavibacter)在高通量测序中未被检测到;而在MPN培养系统中,基于复苏促进因子的处理组细菌总数最大检出丰度提高了100倍,并从中共检出21株对Rpf敏感的VBNC菌株,其中6个属(Paenibacillus、Caulobacter、Roseomonas、Pantoea、Erwinia、Acinetobacter)为传统分离法中未能获得的菌属,2个属(Paenibacillus和Caulobacter)在高通量测序中未被检测到。[结论] 蚕沙细菌多样性丰富,不同方法解析蚕沙细菌组成多样性存在一定差异,而且通过添加Rpf首次发现了蚕沙中存在大量过去未被认知的VBNC状态细菌,其中从16S rRNA基因序列同源性推测有3个VBNC菌株为潜在新种。此研究结果为深入挖掘蚕沙中微生物资源提供了新的视角与途径,为蚕沙的综合处理提供了研究基础。
关键词:复苏促进因子活的非可培养状态高通量测序蚕沙细菌多样性
Bacterial diversity and VBNC bacteria in silkworm excrement from Guangxi
Yanjun Zhang1#, Baoting Chen1#, Yumei Zhang1, Yifei Zhang1, Baolin Zhu1, Pan Su1, Linxian Ding2, Naijun Xie3, Pinghua Zhang1


1. College of Chemistry and Life Sciences, Zhejiang Normal University, Jinhua 321004, Zhejiang Province, China;
2. College of Geography and Environmental Sciences, Zhejiang Normal University, Jinhua 321004, Zhejiang Province, China;
3. Xinsheng Silk Technology, Ltd., Jingxi 533800, Guangxi Zhuang Autonomous Region, China
Received: 8 January 2020; Revised: 16 March 2020; Published online: 27 March 2020
*Corresponding author: Pinghua Zhang, Tel:+86-579-82288003;E-mail:zph@zjnu.cn.
Foundation item: Supported by the Zhejiang Province Natural Science Foundation of China (LY19C130002) and by the Open Project of Zhejiang Key Laboratory of Intestinal Microecology (2019KLGM011)
#These authors contributed equally to this work.
Abstract: [Objective] To analyze the composition and diversity of bacterial in silkworm excrement from Guangxi, and to explore microbial resource of silkworm excrement. [Methods] Through high-throughput sequencing technology, we analyzed the composition of bacterial communities in silkworm excrement. Meanwhile, we isolated culturable bacteria and those in viable but non-culturable (VBNC) state in silkworm excrement using spread-plate method and resuscitation promoting factor (Rpf)-based MPN method, and then classified and identified the isolated strains through 16S rRNA gene sequence analysis. [Results] The bacteria detected from silkworm excrement of Guangxi belonged to 10 phyla, 18 classes, 27 orders, 57 families and 96 genera, in which the bacteria abundance of 4 genera was greater than 1% and the dominant microflora was Enterobacter (Proteobacteria). We obtained 33 culturable bacteria from 14 genera through the spread-plate method, in which 4 genera (Citrobacter, Weissella, Chitinophaga, Pseudoclavibacter) were not detected by high-throughput sequencing. The detection abundance of bacteria increased by up to 100 times by Rpf-based MPN method. Also, we found 21 Rpf-sensitive and resuscitation-cultivable VBNC strains from the Rpf-treatment group, in which 6 genera (Paenibacillus, Caulobacter, Roseomonas, Pantoea, Erwinia, Acinetobacter) and 2 genera (Paenibacillus and Caulobacter) were not found through spread-plate method and high-throughput sequencing, respectively. [Conclusion] Our results provide a new perspective and a new approach for exploring and utilizing the microbial resources in silkworm excrement.
Keywords: resuscitation-promoting factorviable but non-culturable (VBNC) statehigh-throughput sequencingsilkworm excrementbacterial diversity
蚕沙是由蚕排出的粪便、幼虫蜕皮物和食剩的残桑等组成的混合物。随着国家“东桑西移”的战略调整,浙江、江苏等传统蚕茧主产区逐步向广西等西部地区转移,蚕桑业已成为西部地区农村脱贫致富的主要产业之一。2018年我国蚕茧产量高达67.9万吨,但每年产生的大约几百万吨的新鲜蚕沙,大多被焚烧或随意弃置,加重环境污染负荷,同时也造成严重的资源浪费。探究蚕沙中菌群组成可为蚕沙的资源化利用及资源微生物开发提供理论基础。目前对于环境样品大多采用传统分离方法分析研究其微生物组成,但是研究报道表明多数自然环境样品中真正能分离到的可培养微生物大约只有0.01%–10%,绝大部分微生物因处于活的但非可培养状态(viable but non- culturable,VBNC)未被有效分离获得并鉴定,从而限制了人们对生态环境中微生物多样性组成的认识与科学应用[1-2]。有研究表明,藤黄微球菌(Micrococcus luteus)在对数生长后期分泌的复苏促进因子(Resuscitation-promoting factor,Rpf)可恢复多种生态环境中处于VBNC状态细胞的复苏生长能力[3-5]。课题组曾利用Rpf从制药废水中成功筛选到1株降解农药毒死蜱效果达70%的荧光假单胞菌(Pseudomonas fluorescens)菌株,可见利用Rpf在各种不同环境中发掘VBNC菌是当今发现新菌种资源与开发环境功能菌的行之有效的途径[6-8]。
迄今为止,对于蚕沙中微生物的研究报道甚少,尤其是蚕沙中是否存在VBNC状态菌的研究尚未见报道。此外,高通量测序因高精度、高通量等特点,正被广泛应用于环境样品中微生物群落多样性的解析[9-11]。因此本研究采用高通量测序技术对蚕沙细菌群落及多样性进行分析,并通过常规稀释涂布平板分离法和基于Rpf的MPN培养系统分离获取蚕沙中可培养细菌和VBNC优势菌群,以期为蚕沙中菌种资源发掘和蚕沙的综合利用提供科学依据。
1 材料和方法 1.1 材料
1.1.1 主要材料: (1) 蚕沙样品:桂蚕二号五龄期新鲜蚕粪,采自广西靖西市武平乡(23°14′ N, 106°22′ E),收集于50 mL无菌离心管内,置冰盒带回实验室,保存至–80 ℃冰箱,备用。(2)菌种:藤黄微球菌(Micrococcus luteus) IAM14879T,由东京大学分子细胞生物学研究所生物资源研究室横田明教授提供。
1.1.2 分离培养基: (1) 弱营养细菌培养基(g/L):蛋白胨5,酵母浸膏0.5,葡萄糖5,NaCl 2.5,琼脂20,pH 7.0–7.2。(2)弱营养MRS培养基(g/L):蛋白胨5,牛肉膏5,酵母提取物2.5,K2HPO4 2,柠檬酸二铵2,乙酸钠5,葡萄糖10,吐温80 1,MgSO4?7H2O 0.5,MnSO4?4H2O 0.25,琼脂20,pH 6.2–6.6。(3)弱营养高氏一号培养基(g/L):可溶性淀粉10,KNO3 1,K2HPO4 0.5,FeSO4 0.01,MgSO4?7H2O 0.5,NaCl 0.5,琼脂20,pH 7.2–7.4。(4)藤黄微球菌复苏培养基、LMM液体培养基参见文献[12],其他培养基参见文献[13]。
1.1.3 主要试剂和仪器: E.Z.N.ATM Mag-Bind Soil DNA Kit试剂盒(Omega,美国);Qubit3.0 DNA检测试剂盒[赛默飞世尔科技(中国)有限公司,美国];Agencourt AMPure XP试剂盒(Beckman coulter,美国);Ezup柱式细菌基因组DNA抽提试剂盒(生工生物工程(上海)股份有限公司,上海)。
PCR仪(BIO-RAD,美国);凝胶成像系统(UVP,美国);Qubit? 3.0荧光计[赛默飞世尔科技(中国)有限公司,美国];酶标仪(Tecan,瑞士)。
1.2 高通量测序法分析蚕沙中细菌群落组成 称取200 mg蚕沙样品于2 mL离心管中,加入1 mL 70%乙醇,混匀后10000 r/min离心3 min,弃上层液体;加入PBS溶液,混匀后10000 r/min离心3 min,弃上层液体后,倒置离心管于吸水纸上1 min,再将样品管放入55 ℃烘箱10 min。预处理后的样品,使用E.Z.N.ATM Mag-Bind Soil DNA Kit试剂盒抽提蚕沙细菌基因组DNA,并用1%琼脂糖凝胶电泳检测DNA完整性。采用引物341F(5′-CCCTACACGACGCTCTTCCGATCTGCCTACGGG NGGCWGCAG-3′)和805R(5′-GACTG GAGTTCCTTGGCACCCGAGAATTCCAGACTACH VGGGTATCTAATCC-3′)扩增16S rRNA基因的V3+V4区,选用Agencourt AMPure XP试剂盒以0.6倍的磁珠对PCR产物进行DNA纯化回收,并利用Qubit3.0 DNA检测试剂盒对回收的DNA精确定量。样品送寄生工生物工程(上海)股份有限公司,在Illumina Miseq PE 300平台进行高通量测序。测序获得的reads经过滤优化处理后,以序列相似度97%为标准,使用Usearch软件根据RDP classifier贝叶斯算法进行OTUs (Operational Taxonomic Units)聚类,对序列进行分类学分析,统计样品的菌群组成。
1.3 利用稀释涂布平板法分离常规可培养细菌 将蚕沙样品进行梯度稀释,选择不同稀释度的稀释液分别涂布至LB培养基、MRS培养基、高氏一号培养基平板上,每个平皿接种100 μL稀释液,每种培养基选择3个不同的稀释梯度,每处理3个重复。将上述培养皿置于30 ℃恒温培养箱中培养,定期观察菌落生长情况。挑取单菌落至相应培养基平板上获得纯培养。纯培养菌液混匀在终浓度10%甘油管中,–80 ℃保藏。
1.4 基于Rpf复苏促进因子的MPN培养系统解析与检出可培养化VBNC状态菌 含Rpf活性蛋白的培养上清液的制备参照本实验室工作流程[1, 12],上清液用0.22 μm滤膜过滤,保存于–20 ℃冰箱备用。采用基于Rpf的MPN培养系统和稀释平板涂布法,分别用弱营养细菌培养基、弱营养MRS培养基和弱营养高氏一号培养基等3种分离培养基,设培养基中含活性Rpf的处理组与灭活Rpf的对照组,利用96孔板为培养容器进行梯度稀释(10–1–10–10),每梯度3个重复,30 ℃恒温培养,用酶标仪定期测定培养液的OD600值,查对MPN表,计算出实验组(含活性Rpf组)与对照组(含灭活Rpf组)的含菌量,以实验组微生物总量/对照组微生物总量的结果(VR)评价Rpf活性丰度,以(处理组细菌总数–对照组细菌总数)/处理组细菌总数×100%计算VBNC菌的复活率。取实验组与对照组中存在浑浊现象的稀释倍数最大的培养液进行稀释涂布培养,定期观察菌落生长情况,挑取单菌落至相应培养基平板上获得纯培养,于–80 ℃冰箱保藏。
1.5 菌株DNA提取与分子生物学鉴定 利用Ezup柱式细菌基因组DNA抽提试剂盒提取菌株DNA,采用通用引物27F (5′-AGAGTTT GATCCTGGCTCAG-3′)和1492R (5′-GGTTACC TTGTTACGACTT-3′)进行16S rRNA基因扩增。扩增产物送至北京擎科新业生物技术有限公司进行测序。将测序结果与Ezbiocloud数据库中已知序列进行BLAST比对,确定其近缘种属关系。
2 结果和分析 2.1 蚕沙细菌群落基本结构组成 样品中共测得有效序列70439条,序列平均长度为429.77 bp,聚类共获得1893个OTUs。测序覆盖率为98%,表明样品中序列被检测出的概率高;Shannon指数稀疏曲线(图 1)趋于平坦,说明测序数据量合理。Alpha多样性指数统计结果显示样品的Chao1指数26963.75,Ace指数75995.25,Shannon指数1.01,Simpson指数0.72,表明菌群丰度高,但多样性不高。
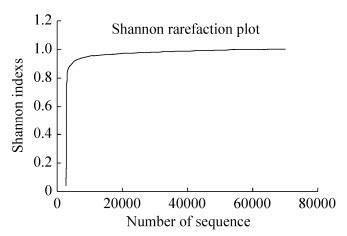 |
| 图 1 Shannon指数稀疏曲线 Figure 1 Shannon rarefaction curve |
| 图选项 |
高通量测序结果表明,从样品中共检出10个门、18个纲、27个目、57个科、96个属。在细菌门水平上,变形菌门(Proteobacteria)的丰度达96.96%,为绝对优势门;其次是厚壁菌门(Firmicutes),丰度为2.86%;其余8个门占比低,还有一些未定门的细菌(图 2)。在属水平上,丰度在1%以上水平的细菌属有肠杆菌属(Enterobacter)、欧文氏菌属(Erwinia)、乳球菌属(Lactococcus)和泛生菌属(Pantoea),分别为84.49%、4.89%、2.10%和1.22%,肠杆菌属占绝对优势,另有4.11%的种属于未分类种群(图 3)。
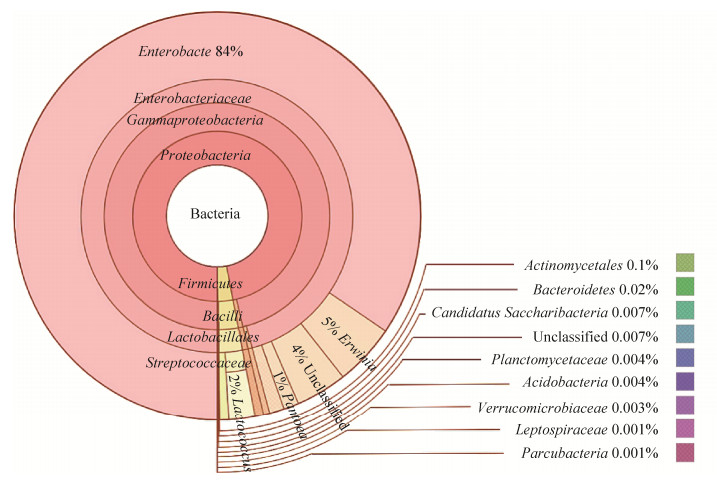 |
| 图 2 多级物种组成图 Figure 2 Multistage species composition |
| 图选项 |
 |
| 图 3 属分类水平菌群分布图 Figure 3 Distribution of microflora at genus classification level |
| 图选项 |
2.2 常规稀释涂布平板法分离与可培养细菌鉴定 3种不同分离培养基分离得到的可培养细菌,经纯化去重复后共获得33个菌株(表 1),经16S rRNA基因测序与比对,菌株归属于4个门、4个纲、8个目、10个科、14个属,其中厚壁菌门为优势门,共15株,占分离菌株的45.45%,其次为变形菌门(8株),放线菌门(9株),拟杆菌门(1株)。在14个属中,芽孢杆菌属(Bacillus)和链霉菌属(Streptomyces)各分离到5株纯培养物,为相对优势菌属。
表 1. 蚕沙可培养细菌分离结果 Table 1. Isolation results of culturable bacteria from silkworm excrement
| Phylum | Class | Order | Family | Genus | Strain |
| Proteobacteria | Gammaproteo- | Enterobacteriales | Enterobacteriaceae | Citrobacter | 2 |
| bacteria | Enterobacter | 3 | |||
| Klebsiella | 1 | ||||
| Xanthomonadales | Xanthomonadaceae | Stenotrophomonas | 1 | ||
| Pseudomonadales | Pseudomonadaceae | Pseudomonas | 1 | ||
| Firmicutes | Bacilli | Bacillales | Staphylococcaceae | Staphylococcus | 4 |
| Bacillaceae | Bacillus | 5 | |||
| Lactobacillales | Enterococcaceae | Enterococcus | 3 | ||
| Lactococcus | 2 | ||||
| Leuconostocaceae | Weissella | 1 | |||
| Actinobacteri | Actinobacteria | Micrococcales | Microbacteriaceae | Microbacterium | 3 |
| Pseudoclavibacter | 1 | ||||
| Streptomycineae | Sterptomycetaceae | Streptomyces | 5 | ||
| Bacteroidete | Sphingobacteria | Sphingobacteriales | Chitinophagaceae | Chitinophaga | 1 |
表选项
2.3 基于Rpf的MPN培养系统分离与可培养化VBNC菌鉴定 基于添加Rpf的MPN培养系统,在3种培养基的培养结果如表 2所示。在培养基中添加Rpf,其活性丰度(VR)大于5,细菌总数检出丰度是相应对照组的14–100倍,VBNC菌的复活率在92.9%以上,说明在蚕沙中存在大量的对Rpf敏感能复苏生长成为可培养化的VBNC状态菌。
表 2. 基于MPN培养系统添加Rpf的3种培养基中VBNC菌复活率比较 Table 2. Comparison of reactivation rate of VBNC bacteria in three media added with Rpf based on the MPN culture system
| Culture medium | Rpf activity abundance (VR) | VBNC bacteria resurrection rate/% |
| Weak nutrient bacteriaculture medium | 1.5×108/7.3×106=20.55 | 95.1 |
| Weak nutrition MRS medium | 2.1×108/1.5×107=14 | 92.9 |
| Weak nutrition Gao’s No.1 medium | 9.3×106/9.3×104=100 | 99.0 |
表选项
取基于添加Rpf的MPN培养系统获得的培养液进行稀释涂布培养,共分离纯化得到29个菌株,分别归属于厚壁菌门和变形菌门,其中加Rpf的实验组获得21株VBNC菌,而加灭活Rpf的对照组获得8株可培养细菌,结果见表 3。这21株VBNC菌归属于3个纲、6个目、7个科和9个属,其中优势菌群为γ-变形菌纲(Gammaproteobacteria)的肠杆菌目(Enterobacteriales)。而对照组的可培养细菌分布在2个纲、3个目、5个科、5个属。16S rRNA基因测序结果表明,处理组与对照组只出现2个相同的属,分别为外杆菌属(Exiguobacterium)和肠球菌属(Enterococcus)。外杆菌属的VBNC菌株有2株,分别是Ex. enclense和Ex. indicum,对照组的可培养细菌有3株,分别是1株Ex. acetylicum和2株Ex. indicum的近缘种;肠球菌属的VBNC菌株有3株,都是En. faecalis的近缘种,而对照组的可培养细菌为2株,分别是En. lactis和En. faecium的近缘种,其他分离到的菌株均为非同属种的异质菌株。
表 3. 基于Rpf在MPN培养系统中检出的VBNC菌和可培养细菌分离结果 Table 3. Isolation results of VBNC and culturable bacteria in Rpf-based MPN culture system
| Category | Class | Order | Family | Genus | Strains |
| +Rpf | Alphaproteobacteria | Caulobacterales | Caulobacteraceae | Caulobacter | 2 |
| Rhodospirillales | Acetobacteraceae | Roseomonas | 1 | ||
| Gammaproteobacteria | Enterobacteriales | Enterobacteriaceae | Pantoea | 5 | |
| Enterobacter | 4 | ||||
| Erwinia | 2 | ||||
| Pseudomonadales | Moraxellaceae | Acinetobacter | 1 | ||
| Bacilli | Bacillales | Exiguobacteriaceae | Exiguobacterium | 2 | |
| Paenibacillaceae | Paenibacillus | 1 | |||
| Lactobacillales | Enterococcaceae | Enterococcus | 3 | ||
| –Rpf | Alphaproteobacteria | Rhizobiales | Methylobacteriaceae | Methylobacterium | 1 |
| Rhizobiaceae | Rhizobium | 1 | |||
| Bacilli | Bacillales | Exiguobacteriaceae | Exiguobacterium | 3 | |
| Bacillaceae | Bacillus | 1 | |||
| Lactobacillales | Enterococcaceae | Enterococcus | 2 |
表选项
在分离获取的29株细菌中,其中有8株VBNC菌与1株可培养菌的16S rRNA基因序列与近缘模式菌株的相似度大于97%、低于99%,如表 4所示。其中CSCXZR6-1与CSCXZR6-4的近缘模式菌株均为Erwinia aphidicola DSM 19347(T),相似度分别为98.96%与98.77%;CSCFZR3-2与CSCRZN7-1的近缘模式菌株都是Enterococcus gallinarum NBRC 100675(T),相似度分别为98.97%与98.90%;CSCFZR4-1、CSCXZR6-2与相应的近缘模式菌株的相似度分别为98.75%、98.80%。而CSCXZR8-1、CSCXZR6-3、CSCXZR7-10与相应的近缘模式菌株的相似度分别为98.61%、98.47%、97.97%,根据16S rRNA基因序列相似度小于98.65%,可以认定为可能的新种或新属的标准[14],这3株VBNC菌株显示为潜在的新物种。
表 4. 分离菌株与近缘种模式菌株的16SrRNA基因序列相似性比较 Table 4. Comparison of 16S rRNA gene sequence similarity between isolated and related species
| Category | Strains | Login number of DDBJ | The closest type strains | Similarity/% |
| VBNC | CSCFZR4-1 | LC484735 | Paenibacillus tylopili MK2(T) | 98.75 |
| VBNC | CSCXZR6-2 | LC484722 | Exiguobacterium enclense NIO-1109(T) | 98.80 |
| VBNC | CSCXZR8-1 | LC500798 | Caulobacter segnis ATCC 21756(T) | 98.61 |
| VBNC | CSCXZR6-3 | LC484723 | Enterobacter huaxiensis 090008(T) | 98.47 |
| VBNC | CSCFZR3-2 | LC484734 | Enterococcus gallinarum NBRC 100675(T) | 98.97 |
| VBNC | CSCXZR6-1 | LC484721 | Erwinia aphidicola DSM 19347(T) | 98.96 |
| VBNC | CSCXZR6-4 | LC484724 | Erwinia aphidicola DSM 19347(T) | 98.77 |
| VBNC | CSCXZR7-10 | LC500466 | Acinetobacter soli CIP 110264(T) | 97.97 |
| Culturable | CSCRZN7-1 | LC484786 | Enterococcus gallinarum NBRC 100675(T) | 98.90 |
表选项
实验中采用了2种分离方法,即常规稀释涂布分离法、基于MPN培养系统中添加失活Rpf对照组分离获得的可培养细菌和添加活性Rpf实验组分离得到的可培养化VBNC细菌,菌株组成在属水平上的分布情况如图 4所示。结果表明所采用的分离方法均能获得肠球菌属(Enterococcus)细菌;常规法与加Rpf实验组均能分离到肠杆菌属(Enterobacter)细菌;常规法与加失活Rpf的对照组均能分离到芽孢杆菌属(Bacillus)细菌;添加活性Rpf和添加失活Rpf的培养基均能获得外杆菌属细菌;而添加活性Rpf的实验组还分离得到了类芽孢杆菌属(Paenibacillus)、柄杆菌属(Caulobacter)、玫瑰单胞菌属(Roseomonas)、泛生菌属(Pantoea)、欧文氏菌属(Erwinia)和不动杆菌属(Acinetobacter)等6个属的菌株,表明在蚕沙中存在较大比例的VBNC菌,同时也证实了这些VBNC菌无法在常规分离培养基和对照组培养基中被检测分离。
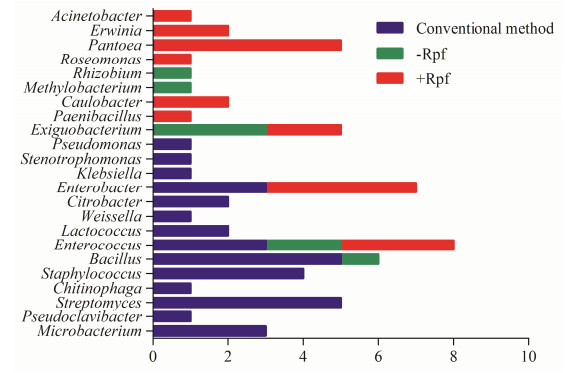 |
| 图 4 常规分离法和Rpf+MPN分离法菌株在属水平上的比较 Figure 4 Comparison of strains isolated by conventional method and Rpf+MPN method at genus level |
| 图选项 |
3 讨论 蚕粪便中含有大量的蚕肠道菌,通过平板稀释培养计数测得蚕沙细菌和放线菌数量达105/g,而真菌数量达104/g[15],已有研究者从蚕沙中分离到能分解纤维素的短小芽孢杆菌(Bacillus pumilus)[16],在蚕沙高温堆肥中分离到对磷有显著溶解、能促进小白菜生长和增产、并对桑椹镰刀菌等真菌也有明显拮抗作用的枯草芽孢杆菌SEM-9菌株[17]。可见,在蚕沙中存在具有潜在应用价值的益生菌。然而要发掘更多更有价值的益生菌并加强蚕沙综合利用的前提就需要全面解析蚕沙中的菌群多样性。本研究利用高通量测序技术对采自广西靖西市武平乡的蚕沙进行细菌菌群多样性分析,结果表明该蚕沙中细菌归属10个门、18个纲、27个目、57个科、96个属,变形菌门为绝对优势门(96.96%),其次是厚壁菌门(2.86%),其余门类都占比很低;在属水平上,肠杆菌属为优势菌属,其次是欧文氏菌属、乳球菌属和泛生菌属。该结果与郑天瑶等[18]研究结果存在一定的差异。Yeruva等[19]对3个品种的桑蚕中肠细菌群落多样性研究发现不同桑蚕品种的细菌多样性和比例不同,由此推测蚕沙中细菌组成可能因家蚕品种差异而存在区别,同时地理位置、气候条件甚至饲料[20]等对蚕沙细菌组成也会造成较大影响。
高通量测序是目前较为准确反映环境样品中微生物组成的有效技术手段,但其缺点是无法获取用于后续研究的菌种资源。本研究通过常规稀释涂布平板分离法共获得33株可培养细菌,其中鉴定得到的柠檬酸杆菌属(Citrobacter)、魏斯氏菌属(Weissella)、噬几丁质菌属(Chitinophaga)与假棍状杆菌属(Pseudoclavibacter)等在高通量测序技术中未检测到,推测与蚕沙样品中这些非优势菌群的丰度较低有关。
虽然稀释平板涂布分离法是一种获取样品中有用微生物、特别是优势菌种的良好方法,但研究报道显示多种环境样品(土壤、污水、海洋底泥等)可培养微生物数量只占其总量的0.01%–10%。样品中大量的微生物因处于VBNC状态而未被认知,极大地限制了对微生物多样性的科学认知。本研究利用复苏促进因子Rpf,首度发现了在蚕沙中存在大量的VBNC菌,可培养化细菌总量是常规分离方法的14–100倍,共得到21株VBNC菌株,其中类芽孢杆菌属、柄杆菌属、玫瑰单胞菌属、泛生菌属、欧文氏菌属和不动杆菌属等6个属细菌均未能在常规分离培养基和对照组培养基上被分离到,同时类芽孢杆菌属和柄杆菌属在高通量测序中也未被检测到,推测这些菌受Rpf的复苏与生长促进作用,恢复了部分处于VBNC状态菌的生长繁殖能力,从而成为可培养化的菌株。此外,本研究共发现3株VBNC菌株的16S rRNA基因序列同源性低于98.65%,而源自VBNC菌的发现是目前获得新种分类群的有效手段[12, 21-22],因此后续拟将这3个菌株作为可培养化VBNC潜在新种菌株,进行多相分类法鉴定和新物种资源开发研究。
综上所述,3种方法均证实蚕沙中存在着丰富的细菌多样性,不同方法解析蚕沙细菌总量丰度比例、种群组成时存在显著差异,而且通过添加Rpf首次在蚕沙中发现了高通量测序以及常规分离方法均未能检测出的处于VBNC状态的菌株,这为更科学地认知蚕沙中的微生物多样性组成、挖掘更多潜在的菌种资源和蚕沙在多领域的综合资源化利用提供更为科学详实的菌种生物多样性信息与理论基础。
References
| [1] | Ding LX. Studies on the isolation of viable but non-culturable bacteria and the phylogenetic analysis of the genus Aquaspirillum. Doctor Dissertation of the University of Tokyo, 2004. |
| [2] | 苏晓梅.多氯联苯污染环境样品中非可培养状态菌的复苏及形成机理研究.浙江大学博士学位论文, 2016. |
| [3] | Colwell RR, Brayton PR, Grimes DJ, Roszak DB, Huq SA, Palmer LM. Viable but non-culturable Vibrio cholerae and related pathogens in the environment:implications for release of genetically engineered microorganisms. Biotechnology, 1985, 3(9): 817-820. |
| [4] | Mukamolova GV, Kaprelyants AS, Young DI, Young M, Kell DB. A bacterial cytokine. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(15): 8916-8921. DOI:10.1073/pnas.95.15.8916 |
| [5] | Mukamolova GV, Turapov OA, Kazarian K, Telkov M, Kaprelyants AS, Kell DB, Young M. The rpf gene of Micrococcus luteus encodes an essential secreted growth factor. Molecular Microbiology, 2002, 46(3): 611-621. DOI:10.1046/j.1365-2958.2002.03183.x |
| [6] | Ding LX, Zhang PH, Hong HC, Lin HJ, Yokota A. Cloning and expression of Micrococcus luteus IAM 14879 Rpf and its role in the recovery of the VBNC state in Rhodococcus sp. DS471. Acta Microbiologica Sinica, 2012, 52(1): 77-82. (in Chinese) 丁林贤, 张萍华, 洪华嫦, 林红军, 横田明. 藤黄微球菌Rpf活性蛋白的制取及其对红球菌VBNC菌体的复苏作用. 微生物学报, 2012, 52(1): 77-82. |
| [7] | Jin Y, Gan GJ, Yu XY, Wu DD, Zhang L, Yang N, Hu JD, Liu ZH, Zhang LX, Hong HC, Yan XQ, Liang Y, Ding LX, Pan YL. Isolation of viable but non-culturable bacteria from printing and dyeing wastewater bioreactor based on resuscitation promoting factor. Current Microbiology, 2017, 74(7): 787-797. DOI:10.1007/s00284-017-1240-z |
| [8] | Wang BB, Wang QL, Liu WX, Liu XY, Hou JY, Teng Y, Luo YM, Christie P. Biosurfactant-producing microorganism Pseudomonas sp. SB assists the phytoremediation of DDT-contaminated soil by two grass species. Chemosphere, 2017, 182: 137-142. DOI:10.1016/j.chemosphere.2017.04.123 |
| [9] | Caporaso JG, Lauber CL, Walters WA, Berg-Lyons D, Lozupone CA, Turnbaugh PJ, Fierer N, Knight R. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(Supplement 1): 4516-4522. |
| [10] | Jia YL, Lin BS, Liao Z, Wang LF, Huang ZX, Song ZZ, Mei L, Lin ZX. Diversity of endophytic nitrogen-fixing bacteria community in the roots of Pennisetum sp. of typical habitats. Acta Microbiologica Sinica, 2020, 60(1): 148-160. (in Chinese) 贾雨雷, 林标声, 廖真, 汪丽芳, 黄在兴, 宋昭昭, 梅兰, 林占熺. 典型生境巨菌草根内生固氮菌的群落多样性. 微生物学报, 2020, 60(1): 148-160. |
| [11] | Mavromatis K, Land ML, Brettin TS, Quest DJ, Copeland A, Clum A, Goodwin L, Woyke T, Lapidus A, Klenk HP, Cottingham RW, Kyrpides NC. The fast changing landscape of sequencing technologies and their impact on microbial genome assemblies and annotation. PLoS One, 2012, 7(12): e48837. DOI:10.1371/journal.pone.0048837 |
| [12] | Ding LX, Yokota A. Curvibacter fontana sp. nov., a microaerobic bacteria isolated from well water. The Journal of General and Applied Microbiology, 2010, 56(3): 267-271. DOI:10.2323/jgam.56.267 |
| [13] | 沈萍, 陈向东. 微生物学实验. 第4版. 北京: 高等教育出版社, 2007. |
| [14] | Kim M, Oh HS, Park SC, Chun J. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(2): 346-351. |
| [15] | Shi CJ, Chen FY, Zhong YS, Wang YY, Li WC, Lin JR. Study on partial physicochemical and biological properties of silkworm paeces of different instar silkworm. Guangdong Agricultural Sciences, 2013, 40(3): 109-111. (in Chinese) 史才娟, 陈芳艳, 钟杨生, 王叶元, 李文楚, 林健荣. 不同龄期家蚕蚕沙部分理化和微生物学性质研究. 广东农业科学, 2013, 40(3): 109-111. DOI:10.3969/j.issn.1004-874X.2013.03.036 |
| [16] | Cen ZL, Hu JM, Wei SY, He TG, Li TT, Zeng Q. Isolation and identification of cellulose-degrading bacterium HB-2 and characteristics of its enzyme activity. Journal of Southern Agriculture, 2016, 47(12): 2065-2071. (in Chinese) 岑贞陆, 胡钧铭, 韦仕岩, 何铁光, 李婷婷, 曾泉. 蚕沙纤维素降解菌HB-2菌株的分离、鉴定及酶活性分析. 南方农业学报, 2016, 47(12): 2065-2071. |
| [17] | Li QR, Liao ST, Zhi HY, Xing DX, Xiao Y, Yang Q. Characterization and sequence analysis of potential biofertilizer and biocontrol agent Bacillus subtilis strain SEM-9 from silkworm excrement. Canadian Journal of Microbiology, 2019, 65(1): 45-58. |
| [18] | Zheng TY, Su SL, Cai HD, Dai XX, Ouyang Z, Duan JA. Silkworm excrement bacterial communities diversity in different instars based on 16S rDNA sequence analysis. China Journal of Chinese Materia Medica, 2017, 42(16): 3098-3105. (in Chinese) 郑天瑶, 宿树兰, 蔡红蝶, 戴新新, 欧阳臻, 段金廒. 基于16S rDNA测序技术分析不同龄期蚕沙中菌群多样性. 中国中药杂志, 2017, 42(16): 3098-3105. |
| [19] | Yeruva T, Vankadara S, Ramasamy S, Lingaiah K. Identification of potential probiotics in the midgut of mulberry silkworm, Bombyx mori through metagenomic approach. Probiotics and Antimicrobial Proteins, 2019, doi: 10.1007/s12602-019-09580-3. |
| [20] | Xiang YQ, Wang XQ, Feng W, Zhou W, Xie HX, Wan YJ. Comparative analysis of the composition of dominant intestinal microflora in silkworm reared with different forages. Acta Ecologica Sinica, 2010, 30(14): 3875-3882. (in Chinese) 向芸庆, 王晓强, 冯伟, 周围, 谢洪霞, 万永继. 不同饲料饲养家蚕其肠道微生态优势菌群类型的组成及差异性. 生态学报, 2010, 30(14): 3875-3882. |
| [21] | Yu XY, Zhang L, Ren B, Yang N, Liu M, Liu XT, Zhang LX, Ding LX. Arthrobacter liuii sp. nov., resuscitated from Xinjiang desert soil. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(3): 896-901. |
| [22] | Yu XY, Zhai JY, Wu C, Zhang CY, Shi JY, Ding LX, Wu M. Pseudomonas pharmafabricae sp. nov., isolated from pharmaceutical wastewater. Current Microbiology, 2018, 75(9): 1119-1125. DOI:10.1007/s00284-018-1495-z |
