甘琪, 姜董豪, 张立奎


扬州大学环境科学与工程学院海洋科学研究所, 扬州江苏 225127
收稿日期:2019-06-19;修回日期:2019-07-11;网络出版日期:2019-09-19
基金项目:扬州大学中青年学术带头人项目
*通信作者:张立奎, Tel:+86-514-89795882;E-mail:lkzhang@yzu.edu.cn.
摘要:高温会加快碱基脱氨基反应形成损伤碱基的速率,进一步对脱氨基的碱基进行复制会导致突变。因此,极端嗜热古菌基因组的稳定性面临着其生存高温环境的挑战。胞嘧啶脱氨基形成尿嘧啶,是常见的脱碱基类型,复制DNA中尿嘧啶会造成GC→AT的突变。尿嘧啶DNA糖苷酶(Uracil DNA glycosylase,UDG)是修复DNA中尿嘧啶的关键酶。基于识别底物的特异性,UDG分为6个家族,广泛分布在细菌、古菌、真核生物以及一些病毒中。基因组序列显示,极端嗜热古菌至少编码一种UDG。目前,对于细菌和真核生物的UDG已进行了大量的研究,但是关于极端嗜热古菌UDG的研究相对较少,尚处于初期阶段。本文综述了极端嗜热古菌UDG的研究进展,并对今后的研究提出了展望。
关键词:极端嗜热古菌尿嘧啶DNA糖苷酶碱基切除修复
Research progress of uracil DNA glycosylase from hyperthermophilic archaea
Qi Gan, Donghao Jiang, Likui Zhang


Marine Science&Technology Institute, Department of Environmental Science and Engineering, Yangzhou University, Yangzhou 225127, Jiangsu Province, China
Received: 19 June 2019; Revised: 11 July 2019; Published online: 19 September 2019
*Corresponding author: Likui Zhang, Tel:+86-514-89795882;E-mail:lkzhang@yzu.edu.cn.
Foundation item: Supported by the Academic Leader of Middle and Young People of Yangzhou University Grant
Abstract: High temperatures accelerate a rate at which deamination of a base forms a damaged base, and further replication of the deaminated base results in a mutation. Therefore, genomic stability of hyperthermophilic archaea is facing a challenge of high temperature environments in which they survive. Deamination of cytosine to form uracil is a common deaminated type, and the replication of uracil in DNA causes a mutation in GC→AT. Uracil DNA glycosylase (UDG) is a key enzyme in the repair of uracil in DNA. Based on their substrate specificity, UDGs are divided into six families and are widely distributed in bacteria, archaea, eukaryotes, and some viruses. Genomic sequences show that hyperthermophilic archaea encode at least one UDG. Currently, UDGs have been extensively studied for bacteria and eukaryotes, but research on the hyperthermophilic archaea UDGs is relatively rare and is still in its infancy. In this paper, research progress of hyperthermophilic archaea UDGs was reviewed, and their future research was proposed.
Keywords: hyperthermophilic archaeauracil DNA glycosylasebase excision repair
DNA中脱氨基作用是造成DNA碱基损伤的重要途径之一,主要包括胞嘧啶脱氨基、腺嘌呤脱氨基和鸟嘌呤脱氨基,分别形成尿嘧啶、次黄嘌呤和黄嘌呤(图 1-A)。胞嘧啶脱氨基形成尿嘧啶,是最为常见的类型。由于尿嘧啶能够与腺嘌呤配对,如果在其被修复之前进行了DNA复制,则G-C配对将突变为A-T配对,从而导致DNA的突变(图 1-B)。为了阻止DNA中尿嘧啶引起的突变,细胞已经进化出相应的碱基切除修复(base excision repair,BER)途径对其进行修复[1]。
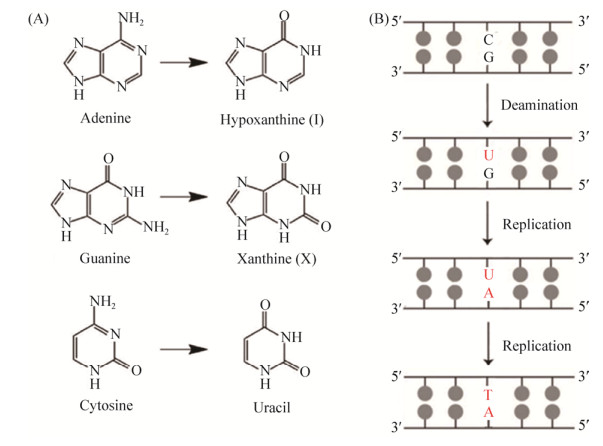 |
| 图 1 常见的DNA脱氨基类型(A)[5]和复制DNA中的尿嘧啶形成GC→AT的突变(B) Figure 1 Common DNA deamination types (A)[5] and GC→AT mutation formed by replication of uracil in DAN (B). |
| 图选项 |
极端嗜热古菌主要生存于深海热液口、火山口、陆地热泉等高温环境,其最适生长温度为80 ℃以上[2]。Sulfolobus acidocaldarius是第一株从黄石公园分离的极端嗜热古菌。目前人们已经发现90多种极端嗜热古菌,大多数属于泉古菌和广古菌[1-2]。极端嗜热古菌中蕴藏着大量未知的生物学过程和功能,研究极端嗜热古菌,不仅有利于理解高温环境下的生命特征及规律,还具有潜在的生物技术开发前景,而且对于分析生命起源和进化也具有重要意义[3]。
由于高温促进了碱基脱氨基的速率[4],因此,生活在高温环境的极端嗜热古菌面临着胞嘧啶脱氨基形成尿嘧啶的严峻挑战。尿嘧啶DNA糖苷酶(Uracil DNA glycosylase,UDG)是启动修复DNA中尿嘧啶的关键酶,广泛分布于细菌,古菌,真核生物和一些病毒中。目前,对于源自细菌和真核生物的UDG进行了大量的研究,但是关于极端嗜热古菌UDG的研究相对较少。本文总结了极端嗜热古菌UDG的研究进展,并对今后的研究进行了一些展望。
1 尿嘧啶DNA糖苷酶的概述 UDG是进化上保守的DNA修复酶,能够去除尿嘧啶[6]进而启动碱基切除修复途径。UDG已经在古菌(例如Sulfolobus solfataricus、Pyrococcus furiosis和Methanococcus jannaschii等)、细菌(例如Escherichia coli、Bacillus subtilis、Neisseria meningitidis、Helicobacter pylori、Mycoplasma pneumonia、Campylobacter jejuni、Deinococcus radiodurans和Mycobacterium tuberculosis等)、真核生物(例如Homo sapiens、Saccharomyces cerivisiae、Caenorhabditis elegans、Drosophila melanogaster和Arabidopsis thaliana等)和大型DNA病毒(例如疱疹病毒和痘病毒等)中得到了研究[7]。
基于其底物特异性,UDG被分为6个家族[8],其系统发育关系如图 2所示。家族Ⅰ UDG,又称为UNG uracil N-Glycosy lase,是UDG超家族中研究最广泛的成员,包括大肠杆菌UDG和人类UDG蛋白。家族Ⅰ UDG的主要生物学功能是去除DNA中的尿嘧啶[9],并能够以相对较低的效率去除5-氟尿嘧啶,但不能切除5-溴或5-碘尿嘧啶[10]。进一步的研究发现,家族Ⅰ UDG切除DNA中尿嘧啶的效率顺序为ssU > U:G > U:A[10]。
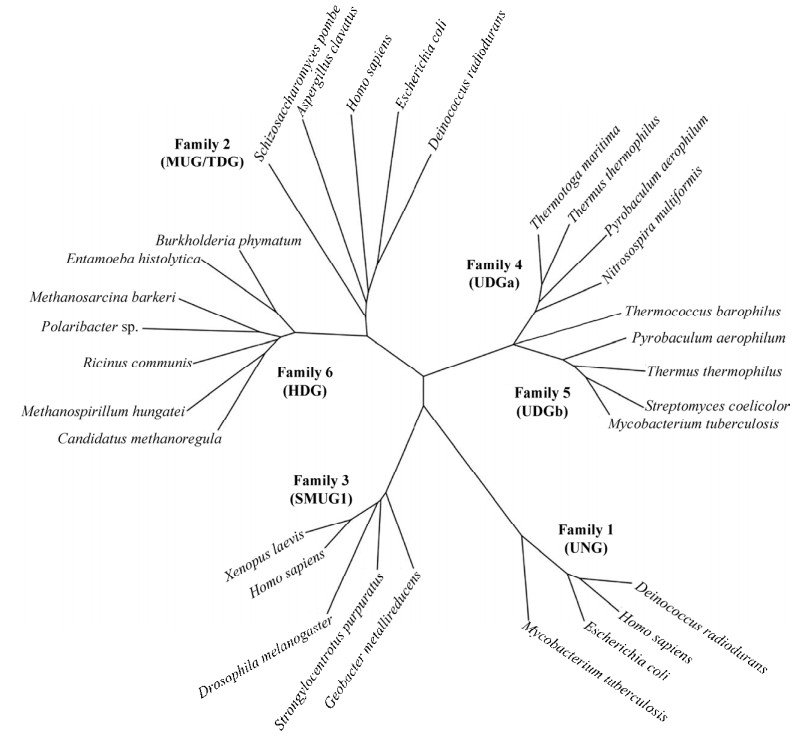 |
| 图 2 UDG家族的系统发育分析[12] Figure 2 Phylogenetic analysis of UDG family[12]. |
| 图选项 |
家族Ⅱ UDG是识别和切割含有尿嘧啶dsDNA中错配的特异性酶,如大肠杆菌错配特异性尿嘧啶糖苷酶(mismatch-specific uracil glycosylase,MUG)和人类胸腺嘧啶DNA糖苷酶(thymine DNA glycosylase,TDG)。E. coli MUG能够有效地切除U:G错配,但对U:A碱基对仅表现出较弱的活性或没有活性[11]。尽管最初认为E. coli MUG能够特异性地从U:G错配中去除U,但后来发现该酶对于携带多种碱基修饰的DNA均具有活性[11]。在高酶浓度下,E. coli MUG类似人类TDG,也能够从T:G错配中切除T[11]。
家族Ⅲ UDG主要存在于高等真核生物中[13-14]。最初发现它对ssDNA具有特异性,又称为sMUG (single-strand specific monofunctional uracil DNA glycosylase)。但后来发现该家族酶不仅能够利用dsDNA作为底物,而且对含尿嘧啶的dsDNA具有更高的亲和力[14]。除了能够从U:G错配和U:A碱基对去除U之外,该家族酶还能够切除5-羟甲基尿嘧啶,但不能切除5-甲基尿嘧啶。
家族Ⅳ和Ⅴ UDG的成员主要源自极端嗜热细菌和古菌,典型的代表为极端嗜热细菌Thermus thermophilus HB8的UDGa和UDGb[15-16],它们具有相似的晶体结构和保守的结构域[17-18]。然而,它们在底物特异性和催化机制方面存在着明显差异。家族Ⅵ的UDG能够去除dsDNA和ssDNA中的尿嘧啶[15],而家族Ⅴ的UDG只能切割dsDNA[16]。此外,家族Ⅴ的UDG不仅能够从G:T错配的DNA中切除T,而且还能够去除DNA中的8oxoG、5-羟甲基尿嘧啶和5-氟尿嘧啶[16]。晶体结构数据表明,家族Ⅵ的UDG与家族Ⅰ的UDG具有相似的结构特征[10]。但是,家族Ⅴ的UDG在活性位点基序上缺少极性的氨基酸残基[18-19]。家族Ⅵ的成员表现出对DNA中次黄嘌呤而非尿嘧啶的糖苷酶活性,从而启动修复DNA中的次黄嘌呤[12]。因此,该家族的成员又被称为HDG (hypoxanthine DNA glycosylase)。
2 极端嗜热古菌尿嘧啶DNA糖苷酶的生化性质 基因组序列显示,极端嗜热古菌至少编码一种UDG,属于家族Ⅵ和Ⅴ(表 1)。目前已经被研究报道的UDG主要是源自极端嗜热广古菌Thermococcus barophilus[20]、P. furiosus[21]、Archaeoglobus fulgidus[22-24]、M. jannaschii[25]和极端嗜热泉古菌Pyrobaculum aerophilum[26]、S. solfataricus[27]、Sulfolobus acidocaldarius[28]和Aeropyrum pernix[29]。另外,S. acidocaldarius和P. aerophilum基因组编码分别属于家族Ⅵ和家族Ⅴ的UDG[28, 30]。表 1比较了极端嗜热古菌UDG最适温度和pH、底物专一性和分类。
表 1. 极端嗜热古菌UDG最适反应温度和pH、底物专一性和分类的比较 Table 1. Comparison of the optimal temperature and pH, substrate specificity and classification of UDGs from hyperthermophilic archaea.
| Organism | Toptimum/℃ | pHoptimum | Substrate specifity | Family | References |
| Euryarchaeota | |||||
| T. barophilus | 55–75 | 5.0–7.0 | U≈U/G > U/T≈U/C > U/A | Unknown | [20] |
| P. furiosus | 70 | 9.0 | U/T≈U/C > U/G≈U/AP≈U/- > U/U≈U/I≈U/A | Ⅳ | [21] |
| A. fulgidus | 80 | 4.8 or 6.2 | U > U/T > U/C=U/G=U/A | Ⅳ | [21-23] |
| M. jannaschii | ND | ND | U/T > U/C > U/G > U/A | Ⅳ | [25] |
| Crenarchaeota | |||||
| P. aerophilum | 70 | ND | G/U > A/U > ssU | Ⅳ | [26] |
| S. solfataricus | 65 | ND | U/G > U > U/A | Ⅳ | [27] |
| A. pernix | 55–65 | 9.0 | U/C=U/G > U/T=U/AP=U/- > U/U=U/I > U/A | Ⅳ | [29] |
| ND: Not determined. | |||||
表选项
生化性质的研究表明,极端嗜热古菌UDG切割尿嘧啶与DNA中脱氧核糖的N-C糖苷键的最适温度均为高温(55–80 ℃)。但是,不同极端嗜热古菌UDG的最适温度不同:A. fulgidus UDG的最适温度为80 ℃,为最高,而A. pernix UDG的最适温度为55–65 ℃,为最低,其余极端嗜热古菌UDG的最适温度居中。因此,极端嗜热古菌UDG能够在高温条件下有效地去除尿嘧啶,从而抵消高温导致尿嘧啶所造成的突变。
极端嗜热古菌UDG切割含有尿嘧啶DNA的效率随pH变化而改变。P. furiosus和A. pernix UDG在pH 9.0表现出最大活性,然而T. barophilus UDG的最适pH为5.0–7.0。但是,重组的A. fulgidus UDG活性的最适pH为4.8,而天然A. fulgidus UDG在pH 6.2时表现出最大活性,这可能与古菌细胞内共价修饰或辅助因子有关,或者其在古菌细胞中表达时具有不同的折叠。
极端嗜热古菌UDG切割尿嘧啶DNA表现出不同的底物专一性。T. barophilus UDG和A. fulgidus UDG切割含有尿嘧啶的ssDNA的效率高于相应的dsDNA,而P. furiosus、P. aerophilum 和A. pernix的UDG则与之相反。需要指出的是,T. barophilus、P. aerophilum、S. solfataricus和A. pernix的UDGs切割含有U/G错配DNA的效率最高。由于C/G配对中胞嘧啶的脱氨作用形成U/G错配,因此它们表现出对U/G错配DNA的最高切割效率,暗示着它们负责去除细胞基因组DNA中由胞嘧啶脱氨基形成的尿嘧啶。最新的研究发现,S. acidocaldarius UDG能够切除RNA中的尿嘧啶[31]。
3 极端嗜热古菌尿嘧啶DNA糖苷酶的结构与功能 极端嗜热古菌S. tokodaii UDG是第一个也是目前唯一一个晶体结构被解析的古菌UDG(图 3-A)。S. tokodaii UDG的晶体结构中存在Fe-S簇,并具有特有的亮氨酸-嵌入环结构[31],这不同于其他家族的UDG。对S. tokodaii UDG亮氨酸-嵌入环的进一步突变分析发现,该酶的氨基酸残基Y170对于底物DNA的识别和催化至关重要[32]。另外,S. tokodaii UDG的晶体结构显示,氨基酸残基E42、N82、H164和F55包裹着尿嘧啶,推测它们可能具有识别尿嘧啶的功能。T. barophilus UDG的E118、N159、H126和Y127分别对应S. tokodaii UDG的E42、N82、H164和F55(图 3-B),暗示着它们具有相似的功能。
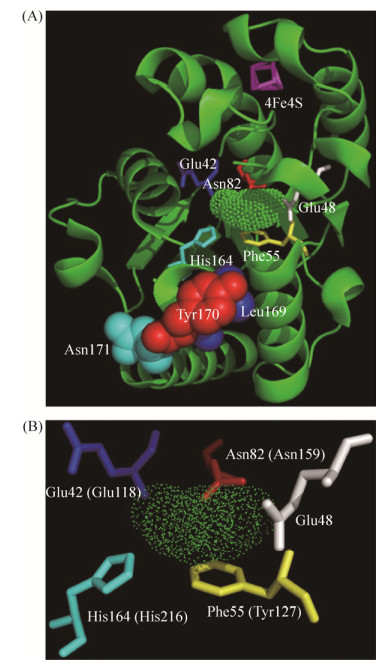 |
| 图 3 S. tokodaii UDG-尿嘧啶复合物的晶体结构(A)及其识别尿嘧啶的氨基酸残基(B)[20] Figure 3 The crystal structure of S. tokodaii UDG-uracil complex (A) and its uracil-recognition residues (B)[20]. The figure was adapted from the S. tokodaii UDG structure (PDB: 4zby) by Pymol[32]. Tba UDG residues are indicated in parentheses. |
| 图选项 |
虽然已对几种极端嗜热古菌UDG进行了突变研究[20, 32-33],但是它们如何识别和切割含尿嘧啶的DNA的机制仍不清楚。对A. fulgidus UDG中Fe-S簇的氨基酸残基突变研究表明,R86A、C85A和C101A突变体仅对双链DNA中的尿嘧啶表现出减弱的活性,而K100A突变体表现出增强的活性[33]。晶体结构显示,家族Ⅵ的UDG,包括极端嗜热细菌Thermus thermophilus[15]和极端嗜热古菌S. tokodaii的UDG[32],类似于家族Ⅰ的UDG[34]。人类UDG是被研究地很清楚的家族Ⅰ UDG的代表。对人类UDG突变研究发现,Q144、N204和H268是切割糖苷键必需的氨基酸残基[34]。人类UDG中的氨基酸残基Q144、D145、N204和H268分别对应极端嗜热古菌T. barophilus UDG中的E118、N159和H216。本实验室研究发现,E118A、N159A和H216A突变体完全丧失了切割活性并保留了较低的结合活性[20],这E118、N159和H216对于尿嘧啶的识别和去除是必需的。
人类UDG的氨基酸残基F158在绝大多数的UDG序列中是完全保守的[34]。该酶的晶体结构显示,F158的苯环与尿嘧啶的嘧啶环堆叠,从而提高了识别尿嘧啶的准确性[34]。然而,F158的功能尚未通过突变分析验证。T. barophilus UDG氨基酸残基Y127位于与人类UDG的F158相同的位置。进一步的研究发现,T. barophilus UDG Y127A突变体表现出与野生型蛋白相似的切割和结合活性[20],暗示着氨基酸残基Y127可能在维持蛋白质构象方面发挥作用。
4 极端嗜热嗜压古菌T. barophilus Ch5尿嘧啶DNA糖苷酶 T. barophilus Ch5分离于大西洋中脊(Logachev油田烟囱,3020 m深)的深海热液区,其最适生长温度为85 ℃,其最适生长压力为40 MPa[35]。T. barophilus Ch5的基因组测序已经完成[36],编码两个UDG(Tba UDG194和Tba UDG247),其对应的基因分别为TbCh5v1_2287和TbCh5v1_0629。
我们实验室最近克隆、表达与纯化了Tba UDG247,并对其生化性质和催化机理进行了研究[19]。研究发现,重组Tba UDG247在65 ℃下仅切割含尿嘧啶的ssDNA和dsDNA,并在70–75 ℃下显示出最佳的切割活性。该酶在4.0至11.0的pH范围内能切割DNA,最佳pH为7.0–9.0。此外,该酶的活性不依赖于二价金属离子; 但是,Zn2+和Cu2+都完全抑制该酶的活性。高NaCl浓度也抑制该酶的活性。另外,该酶去除DNA中的尿嘧啶,具有以下优先顺序:U≈U/G > U/T≈U/C > U/A。动力学结果显示,该酶切割含尿嘧啶的ssDNA和dsDNA的速率相似。
最新的研究发现,Tba UDG247不仅能够切割DNA中尿嘧啶与核糖的N-C糖苷键,从而去除尿嘧啶所产生的AP位点,而且能够切割该AP位点,即具有糖苷酶和AP裂合酶的两种活性。由此可见,该酶不同于已报道的单功能UDG(只具有糖苷酶活性),而类似于双功能的8oxoG DNA糖苷酶(具有糖苷酶活性和AP裂合酶活性)。通过对Tba UDG247与其他极端嗜热古菌和细菌的UDG的氨基酸序列比对,我们发现该酶缺失存在于其他极端嗜热古菌和细菌UDG中一些保守结构域[20]。UDG系统发育树显示,Tba UDG247位于一个独立的分支,介于家族Ⅵ和家族Ⅴ之间(图 2),暗示着该酶为新家族的糖苷酶,这与其表现的不同于其他UDG的生化功能相吻合。
5 总结和展望 极端嗜热古菌生长在80 ℃及以上的高温环境,高温会加快碱基脱氨基反应,形成损伤碱基。胞嘧啶脱氨基生成尿嘧啶,是常见的脱氨基类型。为了修复DNA中的尿嘧啶,极端嗜热古菌细胞至少编码一种UDG,这些UDG与其他参与DNA中尿嘧啶修复的蛋白共同作用,完成了对其的修复,从而使极端嗜热古菌具有与常温微生物相似的突变频率[37]。
一些极端嗜热古菌编码多种UDGs,它们都能够水解尿嘧啶和脱氧核糖之间糖苷键,暗示着它们在细胞中负责切除DNA中的损伤碱基尿嘧啶,并与其他修复蛋白相互作用,协调完成整个尿嘧啶修复的反应。目前,对于极端嗜热古菌中多种UDGs在细胞内的功能、分工方式尚不明确。这些问题的阐明依赖于构建UDGs的基因敲除突变株,测定突变株表型的变化,从而确定它们的生理功能。
极端嗜热古菌编码能够识别DNA中尿嘧啶的DNA聚合酶[38-39],以及负责清除尿嘧啶脱氧核糖核苷三磷酸的水解酶等[40]。通过体外研究这些蛋白修复尿嘧啶损伤碱基的催化反应的分子机制,将有助于揭示极端嗜热古菌如何应对其受到高温引起的脱氨基反应的挑战。
Tba UDG247具有尿嘧啶DNA糖苷酶和AP裂合酶两种活性,不同于已报道的尿嘧啶DNA糖苷酶,可能属于新家族的UDG,其两种活性的作用机制尚不清楚。利用定点突变技术和蛋白质晶体结构解析技术相结合,揭示该酶的作用机制,是值得探讨的课题。
References
| [1] | Wallace SS. Base excision repair:a critical player in many games. DNA Repair, 2014, 19: 14-26. DOI:10.1016/j.dnarep.2014.03.030 |
| [2] | Stetter KO. A brief history of the discovery of hyperthermophilic life. Biochemical Society Transactions, 2013, 41(1): 416-420. DOI:10.1042/BST20120284 |
| [3] | Li YT, Shi HQ, Zhang LK. Research progress of hyperthermophilic archaeal DNA repair endonucleases. Acta Microbiologica Sinica, 2019, 59(10): 1889-1896. (in Chinese) 李玉婷, 史昊强, 张立奎. 极端嗜热古菌DNA修复核酸内切酶的研究进展. 微生物学报, 2019, 59(10): 1889-1896. |
| [4] | Lindahl T, Nyberg B. Heat-induced deamination of cytosine residues in deoxyribonucleic acid. Biochemistry, 1974, 13(16): 3405-3410. DOI:10.1021/bi00713a035 |
| [5] | Chatterjee N, Walker GC. Mechanisms of DNA damage, repair, and mutagenesis. Environmental and Molecular Mutagenesis, 2017, 58(5): 235-263. DOI:10.1002/em.22087 |
| [6] | Zharkov DO, Mechetin GV, Nevinsky GA. Uracil-DNA glycosylase:structural, thermodynamic and kinetic aspects of lesion search and recognition. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 2010, 685(1-2): 11-20. DOI:10.1016/j.mrfmmm.2009.10.017 |
| [7] | Schormann N, Ricciardi R, Chattopadhyay D. Uracil-DNA glycosylases-structural and functional perspectives on an essential family of DNA repair enzymes. Protein Science, 2014, 23(12): 1667-1685. DOI:10.1002/pro.2554 |
| [8] | Slupphaug G, Eftedal I, Kavli B, Bharati S, Helle NM, Haug T, Levine DW, Krokan HE. Properties of a recombinant human uracil-DNA glycosylase from the UNG gene and evidence that UNG encodes the major uracil-DNA glycosylase. Biochemistry, 1995, 34(1): 128-138. |
| [9] | Liu PF, Burdzy A, Sowers LC. Substrate recognition by a family of uracil-DNA Glycosylases:UNG, MUG, and TDG. Chemical Research in Toxicology, 2002, 15(8): 1001-1009. DOI:10.1021/tx020030a |
| [10] | Barrett TE, Savva R, Panayotou G, Barlow T, Brown T, Jiricny J, Pearl LH. Crystal structure of a G:T/U mismatch-specific DNA glycosylase:mismatch recognition by complementary-strand interactions. Cell, 1998, 92(1): 117-129. |
| [11] | O'Neill RJ, Vorob'eva OV, Shahbakhti H, Zmuda E, Bhagwat AS, Baldwin GS. Mismatch uracil glycosylase from Escherichia coli:general mismatch or a specific DNA glycosylase?. The Journal of Biological Chemistry, 2003, 278(23): 20526-20532. DOI:10.1074/jbc.M210860200 |
| [12] | Lee HW, Dominy BN, Cao WG. New family of deamination repair enzymes in uracil-DNA glycosylase superfamily. The Journal of Biological Chemistry, 2011, 286(36): 31282-31287. DOI:10.1074/jbc.M111.249524 |
| [13] | Kavli B, Sundheim O, Akbari M, Otterlei M, Nilsen H, Skorpen F, Aas PA, Hagen L, Krokan HE, Slupphaug G. hUNG2 is the major repair enzyme for removal of uracil from U:A matches, U:G mismatches, and U in single-stranded DNA, with hSMUG1 as a broad specificity backup. The Journal of Biological Chemistry, 2002, 277(42): 29926-29936. |
| [14] | Wibley JEA, Waters TR, Haushalter K, Verdine GL, Pearl LH. Structure and specificity of the vertebrate anti-mutator uracil-DNA glycosylase SMUG1. Molecular Cell, 2003, 11(6): 1647-1659. DOI:10.1016/S1097-2765(03)00235-1 |
| [15] | Starkuviene V, Fritz HJ. A novel type of uracil-DNA glycosylase mediating repair of hydrolytic DNA damage in the extremely thermophilic eubacterium Thermus thermophilus. Nucleic Acids Research, 2002, 30(10): 2097-2102. DOI:10.1093/nar/30.10.2097 |
| [16] | Xia B, Liu YL, Li W, Brice AR, Dominy BN, Cao WG. Specificity and catalytic mechanism in family 5 uracil DNA glycosylase. The Journal of Biological Chemistry, 2014, 289(26): 18413-18426. DOI:10.1074/jbc.M114.567354 |
| [17] | Hoseki J, Okamoto A, Masui R, Shibata T, Inoue Y, Yokoyama S, Kuramitsu S. Crystal structure of a family 4 uracil-DNA glycosylase from Thermus thermophilus HB8. Journal of Molecular Biology, 2003, 333(3): 515-526. DOI:10.1016/j.jmb.2003.08.030 |
| [18] | Kosaka H, Hoseki J, Nakagawa N, Kuramitsu S, Masui R. Crystal structure of family 5 uracil-DNA glycosylase bound to DNA. Journal of Molecular Biology, 2007, 373(4): 839-850. DOI:10.1016/j.jmb.2007.08.022 |
| [19] | Sartori AA, Fitz-Gibbon S, Yang HJ, Miller JH, Jiricny J. A novel uracil-DNA glycosylase with broad substrate specificity and an unusual active site. The EMBO Journal, 2002, 21(12): 3182-3191. DOI:10.1093/emboj/cdf309 |
| [20] | Shi HQ, Gan Q, Jiang DH, Wu YQ, Yin YC, Hou HY, Chen HX, Xu YN, Miao L, Yang ZH, Oger P, Zhang LK. Biochemical characterization and mutational studies of a thermostable uracil DNA glycosylase from the hyperthermophilic euryarchaeon Thermococcus barophilus Ch5. International Journal of Biological Macromolecules, 2019, 134: 846-855. DOI:10.1016/j.ijbiomac.2019.05.073 |
| [21] | Lin LB, Liu YF, Liu XP, Liu JH. Biochemical characterization of uracil-DNA glycosylase from Pyrococcus furiosus. Chemical Research in Chinese Universities, 2012, 28(3): 477-482. |
| [22] | Sandigursky M, Franklin WA. Uracil-DNA glycosylase in the extreme thermophile Archaeoglobus fulgidus. The Journal of Biological Chemistry, 2000, 275(25): 19146-19149. DOI:10.1074/jbc.M001995200 |
| [23] | Knaevelsrud I, Ruoff P, Anensen H, Klungland A, Bjelland S, Birkeland NK. Excision of uracil from DNA by the hyperthermophilic Afung protein is dependent on the opposite base and stimulated by heat-induced transition to a more open structure. Mutation Research/DNA Repair, 2001, 487(3/4): 173-190. |
| [24] | Kn?velsrud I, Kazazic S, Birkeland NK, Bjelland S. The pH optimum of native uracil-DNA glycosylase of Archaeoglobus fulgidus compared to recombinant enzyme indicates adaption to cytosolic pH. Acta Biochimica Polonica, 2014, 61(2): 393-395. |
| [25] | Chung JH, Im EK, Park HY, Kwon JH, Lee S, Oh J, Hwang KC, Lee JH, Jang Y. A novel uracil-DNA glycosylase family related to the helix-hairpin-helix DNA glycosylase superfamily. Nucleic Acids Research, 2003, 31(8): 2045-2055. DOI:10.1093/nar/gkg319 |
| [26] | Sartori AA, Sch?r P, Fitz-Gibbon S, Miller JH, Jiricny J. Biochemical characterization of uracil processing activities in the hyperthermophilic archaeon Pyrobaculum aerophilum. The Journal of Biological Chemistry, 2001, 276(32): 29979-29986. DOI:10.1074/jbc.M102985200 |
| [27] | Dionne I, Bell SD. Characterization of an archaeal family 4 uracil DNA glycosylase and its interaction with PCNA and chromatin proteins. The Biochemical Journal, 2005, 387: 859-863. DOI:10.1042/BJ20041661 |
| [28] | Wang J, Yi GS, Ou J, Liu JH, Liu XP. Cloning, expression, purification and characterization of two uracil-DNA glycosylases from Sulfolobus acidocaldarius. Acta Microbiologica Sinica, 2015, 55(8): 1036-1041. (in Chinese) 王婧, 易刚顺, 欧杰, 刘建华, 刘喜朋. 嗜酸嗜热古菌Sulfolobus acidocaldarius编码尿嘧啶DNA糖苷酶表达, 纯化与酶学特征. 微生物学报, 2015, 55(8): 1036-1041. |
| [29] | Liu XP, Liu JH. Characterization of family Ⅳ UDG from Aeropyrum pernix and its application in hot-start PCR by family B DNA polymerase. PLoS One, 2011, 6(11): e27248. DOI:10.1371/journal.pone.0027248 |
| [30] | Sartori AA, Fitz-Gibbon S, Yang HJ, Miller JH, Jiricny J. A novel uracil-DNA glycosylase with broad substrate specificity and an unusual active site. The EMBO Journal, 2002, 21(12): 3182-3191. DOI:10.1093/emboj/cdf309 |
| [31] | Yi GS, Wang WW, Cao WG, Wang FP, Liu XP. Sulfolobus acidocaldarius UDG can remove dU from the RNA backbone:insight into the specific recognition of uracil linked with deoxyribose. Genes (Basel), 2017, 8(1): 38. DOI:10.3390/genes8010038 |
| [32] | Kawai A, Higuchi S, Tsunoda M, Nakamura KT, Yamagata Y, Miyamoto S. Crystal structure of family 4 uracil-DNA glycosylase from Sulfolobus tokodaii and a function of tyrosine 170 in DNA binding. FEBS Letters, 2015, 589: 2675-2682. DOI:10.1016/j.febslet.2015.08.019 |
| [33] | Engstrom LM, Partington OA, David SS. An iron-sulfur cluster loop motif in the Archaeoglobus fulgidus uracil-DNA glycosylase mediates efficient uracil recognition and removal. Biochemistry, 2012, 51(25): 5187-5197. DOI:10.1021/bi3000462 |
| [34] | Mol CD, Arvai AS, Slupphaug G, Kavli B, Alseth I, Krokan HE, Tainer JA. Crystal structure and mutational analysis of human uracil-DNA glycosylase:structural basis for specificity and catalysis. Cell, 1995, 80(6): 869-878. DOI:10.1016/0092-8674(95)90290-2 |
| [35] | Oger P, Sokolova TG, Kozhevnikova DA, Taranov EA, Vannier P, Lee HS, Kwon KK, Kang SG, Lee JH, Bonch-Osmolovskaya EA, Lebedinsky AV. Complete genome sequence of the hyperthermophilic and piezophilic archaeon Thermococcus barophilus Ch5, capable of growth at the expense of hydrogenogenesis from carbon monoxide and formate. Genome Announcements, 2016, 4(1): e01534-15. |
| [36] | Marteinsson VT, Birrien JL, Reysenbach AL, Vernet M, Marie D, Gambacorta A, Messner P, Sleytr UB, Prieur D. Thermococcus barophilus sp. nov., a new barophilic and hyperthermophilic archaeon isolated under high hydrostatic pressure from a deep-sea hydrothermal vent. International Journal of Systematic Bacteriology, 1999, 49 Pt 2: 351-359. |
| [37] | Grogan DW, Carver GT, Drake JW. Genetic fidelity under harsh conditions:analysis of spontaneous mutation in the thermoacidophilic archaeon Sulfolobus acidocaldarius. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(14): 7928-7933. DOI:10.1073/pnas.141113098 |
| [38] | Abellón-Ruiz J, Ishino S, Ishino Y, Connolly BA. Archaeal DNA polymerase-B as a DNA template guardian:links between polymerases and base/alternative excision repair enzymes in handling the deaminated bases uracil and hypoxanthine. Archaea, 2016, 2016: 1510938. |
| [39] | Abellón-Ruiz J, Waldron KJ, Connolly BA. Archaeoglobus Fulgidus DNA polymerase D:A Zinc-binding protein inhibited by hypoxanthine and uracil. Journal of Molecular Biology, 2016, 428(14): 2805-2813. DOI:10.1016/j.jmb.2016.06.008 |
| [40] | Kerepesi C, Szabó JE, Papp-Kádár V, Dobay O, Szabó D, Grolmusz V, Vértessy BG. Life without dUTPase. Frontiers in Microbiology, 2016, 7: 1768. |
