皮永蕊1

 , 吕永红1, 柳莹1, 唐永政1, 高丽1, 包木太2
, 吕永红1, 柳莹1, 唐永政1, 高丽1, 包木太2 1. 烟台大学海洋学院, 山东 烟台 264005;
2. 中国海洋大学, 海洋化学理论与工程技术教育部重点实验室, 山东 青岛 266100
收稿日期:2018-12-23;修回日期:2019-03-15;网络出版日期:2019-04-03
基金项目:山东省自然科学基金(ZR2018MD018);烟台大学博士科研启动资金(HX2018B32)
作者简介:皮永蕊, 2017年7月,在中国海洋大学获海洋化学工程与技术理学博士学位。2015年9月至2016年8月期间,获国家留学基金委资助,赴加拿大纽芬兰纪念大学学习交流一年。2017年9月进入烟台大学海洋学院工作至今。研究方向为海洋溢油地球生物化学,采用微藻生物学、蛋白质组学、微生物学、基因组学等学科交叉的研究手段,探索菌藻共生体系在溢油生物处理过程中溢油组分的降解规律,微藻生理活性的变化规律,石油烃降解菌群群落结构的演替规律,石油烃化合物生物降解的动力学及其地球化学标志物的变化,碳稳定同位素技术在石油烃化合物微生物降解、迁移过程中的应用。现已在包括Chemical Engineering Journal、Bioresource Technology、Journal of Cleaner Production、Journal of Chemical Technology and Biotechnology、Ecological Engineering在内的学术期刊上发表论文10余篇.
*通信作者:皮永蕊, Tel/Fax:+86-535-2105625, E-mail:pirongrui@163.com.
摘要:在微藻-细菌协同共生的过程中,藻类光合作用释放的氧气被异养微生物利用来矿化水体中的污染物,细菌呼吸为藻类提供二氧化碳作为碳源。近年来,藻类-细菌协同共生体系在污水处理中的应用得到了广泛的研究。本文重点综述了菌藻协同共生体系中微藻与细菌之间的三种相互作用,以及菌藻协同共生体系在废水处理中的应用。菌藻协同共生体系中的微藻与细菌通过营养交换、信号转导及基因转移等相互作用实现共赢。该体系广泛用于处理富营养化、重金属、药物、多环芳烃(polycyclic aromatic hydrocarbons,PAHs)、石油烃化合物等难降解的有机污染的水体。对于氮、磷等营养物质的去除,其主要机理涉及同化作用、厌氧氨氧化作用、硝化与反硝化作用、磷酸化作用等。对重金属、药物、石油烃化合物及其他有机化合物的去除机制主要是生物吸附、生物富集及细胞内外的生物降解。
关键词:微藻-细菌相互作用营养物重金属药物污染物石油烃
Application of microalgae-bacteria symbiosis system in wastewater treatment
Yongrui Pi1

 , Yonghong Lv1, Ying Liu1, Yongzheng Tang1, Li Gao1, Mutai Bao2
, Yonghong Lv1, Ying Liu1, Yongzheng Tang1, Li Gao1, Mutai Bao2 1. School of Ocean, Yantai University, Yantai 264005, Shandong Province, China;
2. Key Laboratory of Marine Chemistry Theory and Technology, Ministry of Education, Ocean University of China, Qingdao 266100, Shandong Province, China
*Corresponding author: Yongrui Pi, Tel/Fax: +86-535-2105625; E-mail:pirongrui@163.com.
Foundation item: Supported by the Natural Science Foundation of Shandong Province (ZR2018MD018), the Doctoral Scientific Research Initial Foundation of Yantai University (HX2018B32)
Abstract: Microalgae play a key role in diverse marine ecosystem, and they also naturally coexist with a large variety of other microorganisms. In this symbiosis, O2 released by algal photosynthesis is utilized by aerobicheterotrophic bacteria to mineralize organic compounds, and bacterial respiration provides CO2 as a carbon source to algae. The algae-bacteria consortia has been extensively studied for applications in wastewater treatment. The algae-bacteria interactions and the application of microalgae-bacteria symbiosis system in wastewater treatment were summarized. According to previous studies, algae-bacteria interactions are categorized into nutrient exchange, signal transduction and gene transfer. The treatment of wastewater, contaminated by nutrients, heavy metals, crude oil, polycyclic aromatic hydrocarbons, and pharmaceutical contaminants through microalgae-bacteria symbiosis system was reviewed. Assimilation, anaerobic ammonia oxidation, nitrification and denitrification, phosphorylation are key mechanisms during inorganic nitrogen and phosphorus removal. The removal mechanisms of heavy metals, crude oil, polycyclic aromatic hydrocarbons, and pharmaceutical contaminants by microalgae-bacteria symbiosis system include bioadsorption, bioaccumulation, and intracellular and extracellular biodegradation.
Keywords: microalgae-bacteriainteractionsnutrientsheavy metalspharmaceutical contaminantspetroleum hydrocarbons
海洋真核生物(微藻类)对地球上的生命有重要的贡献,全球大约50%的碳固定和大气中50%的氧气是由它们贡献的[1]。作为水生生物食物网的基础,微藻在海洋生态系统中扮演着重要的角色。微藻-细菌协同共生系统被广泛用于废水的处理[2],如富营养化的水体[3],重金属污染水体[4],有机化合物[5]甚至是药物[6]。在微藻-细菌协同共生的过程中,藻类光合作用释放的氧气被异养微生物利用来矿化水体中的污染物,细菌呼吸为藻类提供二氧化碳作为碳源。微藻-细菌协同生物系统在废水处理过程中,由于微藻光合作用产氧,对供氧的能量需求较低[7],可以同时去除营养物质和病原体(包括病毒)[8],能够促进后续的下游生物质收获过程,无需使用絮凝剂[9],具有低成本、高效曝气、高效去除污染物、减少温室气体排放等优点[10]。由于这些优势,藻类-细菌群落的协同生物系统被用于处理各种废水。
藻类-细菌联合体在污水处理中的应用得到了广泛的研究[11-12],而最初的研究是在絮凝藻-细菌系统中去除氮[13]。在藻类-细菌群落中,细菌可能利用有机物质来产生氨氮(NH3-N)、磷酸盐和二氧化碳,而藻类可以利用这些物质,以阳光作为能源,通过光合作用合成细胞物质。氧气是光合作用产生的,细菌可以利用光合作用产生的氧气代谢废水中的有机物[14]。藻类和细菌群落中的氧气和二氧化碳循环是共赢的。细菌降解污染物产生的代谢物也能促进藻类的生长。藻类分泌物是细菌的主要碳源(碳水化合物、蛋白质和脂质),此外,微藻的细胞表面可以作为细菌的餐桌栖息地[11]。细菌将有机物分解成矿物质,分泌细胞外代谢物,如生长素和维生素B12,是微藻生长所必需的[15]。因此,利用藻类-细菌联合体修复一些难降解的有机污染物(如原油、抗生素类药物)是非常有利的,能够建立培养微生物之间的协同作用,提高总体吸收效率[16-17]。鉴于传统的工程技术在供氧成本高、自然资源的不完全利用、二次污染物的产生以及某些情况下的技术不实用等缺点,自然系统(藻类-细菌联合体)的自氧化原理可以有效地用于许多污染物的修复。本综述重点介绍了(a)藻类与细菌的相互作用[18],(b)藻类-细菌共生体系在各类污水生物修复中的应用。
1 藻类与细菌的相互作用 许多文献对藻类-细菌的相互作用进行了探究,包括在自然环境或实验室环境。研究揭示了藻类-细菌之间不同类型的相互作用,以形成藻类和细菌之间的特定伙伴关系。根据以往的研究,藻类-细菌相互作用可分为营养交换、信号转导和基因转移[18](图 1)。
 |
| 图 1 藻类-细菌的三种相互作用[18] Figure 1 Three types of algae-bacteria interactions |
| 图选项 |
1.1 营养交换 营养交换是藻类-细菌之间最常见的相互作用。藻类以溶解有机碳(DOC)的形式排泄出部分光合有机物,异养细菌吸收并分解大量藻类DOC。根据与DOC的亲和力和生长动力学,可以选择藻际环境中的DOC分解菌[19]。此外,藻类的尸体也可以作为异养的营养物质。通过研究附着在微藻表面的细菌数量可知,附着在病态微藻上的细菌比健康微藻上的细菌高出两个数量级,达到5×106细菌每克藻鲜重[20]。这一观察结果表明,异养细菌也能同化不受保护的藻类细胞。同样值得注意的是,健康的巨藻能够控制细菌在其表面的定植,而且可能存在抑制细菌过度生长的机制,从而降低了对营养和光照的可及性。此外,一些藻类也会产生颗粒物有机物,例如透明胞外聚合物颗粒(TEPs),研究表明,TEPs决定了异养菌与藻类共生的种类和活性[21]。氮是另一重要元素,固氮蓝细菌(蓝绿藻)是水体生态系统中氮介导相互作用的关键生物。它们中的一些是寄生在宿主真核藻类中的内共生体,它们已经适应了与宿主在基因组水平上的共生互动[22]。在微藻和异养细菌之间也发现了氮介导的相互作用。杜氏藻促生长细菌的筛选表明,一些细菌可能促进微藻氮同化[23]。除了这些营养素之外,还阐明了通过特定维生素的相互作用。例如,真核藻类对只能由原核生物合成的维生素B12具有营养缺陷,研究揭示了维生素B12依赖性藻类和异养细菌之间的相互关系[24-25]。另一方面,一项对藻类相关生物膜的宏基因组研究显示,参与B族维生素生物合成的基因以及用于脂肪酶和酯酶的基因较丰富且具有功能性,表明它们在藻类-细菌联合体中的关键作用[26],这种方法有助于通过这些化合物来发现藻类-细菌之间的相互作用,而用化学方法却难以检测到。
1.2 信号转导 信号转导是藻类-细菌相互作用的另一种形式。在这种情况下,用于它们相互作用的化学物质不是营养物质,而是激活或抑制基因表达和/或生理活动,从而改变它们的行为和生长。藻类和细菌之间的信号转导被分为真核生物和细菌之间的“跨界信号”[27]以及古菌和细菌之间的“跨界信号”[28]。在藻类-细菌的相互作用中,细菌分泌化学信号,诱导藻类的形态发生变化[29]。此外,健康的大型藻类可以抑制其表面多余的生物膜的形成,研究表明藻类能够分泌特定的化学物质来抑制细菌群体感应,这是成熟生物膜形成所必需的机制[30]。众所周知,海藻还能分泌挥发性卤代化合物[31]和脂肪酸[32],其中一些具有抗菌活性。另一方面,据报道,细菌群体效应信号分子(如酰基均丝氨酸内酯)干扰了石莼游动孢子的萌发和生长,而石莼是一种巨藻,会导致人工构建物的生物淤积[33]。此外,已知细菌的不同类群如Shewanella[34]、Streptomyces[35]和Bacillus[36]可以产生杀菌代谢产物。这些研究已经揭示了藻际环境中复杂的信号作用,可以利用生物技术来控制藻类和/或细菌。
1.3 基因转移 基因转移是相邻的微生物之间基因的水平转移,是一个进化过程,例如藻际环境中的藻和细菌。Brembu等[37]在罗布斯塔硅藻叶绿体基因组中发现了水平基因转移的痕迹。Moszczynski等[38]研究表明,甲藻的微管基因组拥有与属于拟杆菌进化枝的噬冷菌(Algoriphagus)和/或噬细胞菌属(Cytophaga)密切相关的基因,表明这些基因发生了转移。一些水平转移的基因赋予藻类关键的功能,使其能更好地在变化的环境中生存,例如,在鸟嘌呤-尿素循环(被认为是从细菌转移的)中编码酶的硅藻基因促进了它们对偶发性氮可用性的代谢反应[39]。此外,真核生物的基因组分析也发现了维生素相关通路的广泛衰减,例如藻类中由藻圈细菌提供的B族维生素的合成[40],这表明藻类和细菌是共同进化的,很可能他们在这种进化过程中建立了特殊的伙伴关系。
2 藻类-细菌互作在污水生物修复中的应用 2.1 去除营养物质 自20世纪90年代以来,人们一直在研究由附着的微藻和细菌组成的联合体从废水中去除营养物,特别是氮、磷等营养物质。Liang等[41]利用小球藻与地衣芽孢杆菌构建了藻类-细菌联合系统,通过6 d的实验结果表明联合系统对NH4+和总磷的去除率达到了78%和92%,远远超过了仅有小球藻的系统(29%和55%)。Su等[10]的研究结果表明,沉淀后的菌藻培养基对废水的化学需氧量、总凯氏氮及总磷的8 d去除率分别为98.2%±1.3%、88.3%±1.6%和64.8±1.0%。Posadas等[42]研究了微藻-细菌生物膜生物反应器对系统中氮、磷的去除,结果显示对氮、磷的去除率达到了70%±8%和85%±9%。Tang等[43]研究了序批式生物膜反应器中菌藻协同系统对生活污水中氮、磷的生物降解,总氮和总磷的去除率达到了65.8%和89.3%,是单纯序批式生物膜反应器去除率的近2倍(38.5%)和3倍(31.9%)。
菌藻协同体系对无机氮的去除机理主要涉及同化作用[3]。氧化态氮的同化如NO3–或NO2–首先还原为NH4+态,然后进一步融入氨基酸,而还原态氮(NH4+)可以被微藻直接同化。具体来说,硝酸盐还原酶利用烟酰胺腺嘌呤二核苷酸(NADH)的还原形式传递两个电子,使硝酸盐转化为亚硝酸盐。然后亚硝酸盐被亚硝酸还原酶转化成铵,使用铁氧还蛋白(Fd)作为电子供体(图 2反应1a和1b)。因此,所有形式的无机氮最终都转化为铵,然后利用谷氨酸和ATP将铵结合到氨基酸中(图 2反应1c)[44-45]。除同化作用外,氨挥发作用、硝化作用和反硝化作用也是无机氮去除过程中的关键机理。在pH > 8的条件下,氨可以氨气的形式从水中挥发(图 2反应2)。硝化作用是将NH4+氧化成NO2–(图 2反应3a)再氧化成NO3–(图 2反应3b),主要是通过氨氧化细菌、氨氧化古菌、亚硝酸盐氧化菌或者是氨氧化细菌完全氧化(图 2反应4)得到的。反硝化与之相反,NO3–还原成NO2–(图 2反应5a)再被还原成N2或者N2O(图 2反应5b),主要是通过反硝化细菌在无氧的水环境中实现的。近几十年来,厌氧氨氧化菌(图 2反应6)和好氧反硝化菌的发现,可以在完全好氧或厌氧条件下直接脱氮。
 |
| 图 2 菌藻协同体系去除N的机理 Figure 2 Nitrogen removal mechanisms by the symbiotic system of microalgae-bacteria |
| 图选项 |
有机氮(如氨基酸和蛋白质)在各种细菌分泌的Gln-合成酶、Glu-2-酮戊二酸氨基转移酶、Glu-脱氢酶等酶的参与下,可以分解成氨(图 2反应8),即所谓的氨化或矿化反应[46]。然后,生成的NH4+可以像上面描述的那样从地表水中除去。
磷在微藻及细菌的代谢过程中起着重要的作用,无机磷(PO43–)可以通过磷酸化作用与有机化合物(如DNA、RNA、脂类)结合[3]。被同化的磷中有很大一部分参与了从ADP产生ATP的过程,同时伴随着一种形式的能量输入(反应式1)[43]。同化的磷除用于微藻和细菌的生长繁殖之外,一些微藻和微生物还能摄取一些磷并将其储存在细胞内。此外,在pH值较高的条件下,磷酸可以与废水中的钙、镁离子形成羟磷灰石,通过与微藻或细菌分泌的胞外聚合物形成氢键而吸附在微藻或细菌的表面。
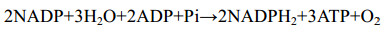 | 公式(1) |
2.3 降解PAHs及原油 一些微藻能够降解原油、PAHs等有机污染物。据报道,海洋硅藻(Skeletonema costatum)和小球藻(Chlorella vulgaris)对多环芳烃及原油的降解有较显著的效果。菌藻共生体系在处理石油烃、PAHs废水方面逐渐得到了关注。Tang等[16]通过4株石油烃降解菌与耐油斜生栅藻(Scenedesmus obliquus GH2)构建了人工微藻-细菌体系,微藻的加入显著提高了石油烃降解菌对原油的降解效果,特别是多环芳烃的降解。Mishamandani等[48]通过海洋硅藻肋骨线藻(Skeletonema costatum)-细菌组合,探究了与海洋硅藻骨架藻共生的石油烃降解菌对原油的反应以及多样性。结果表明,海洋硅藻本身是不能利用碳氢化合物作为自身的碳源和能源的,但是,与硅藻相共生的细菌群落在原油的刺激下,对多环芳烃产生反应并降解,证实了海洋真核浮游植物的生物圈是海洋中尚未开发的生物群落,并发现了能够对石油污染作出反应的新的石油烃降解类群。据报道,天然浮游植物及相关的石油烃降解菌显著提高了原油中毒性更强的PAHs的降解,尤其是分子量较大的二苯并噻吩、4-甲基二苯并噻吩、C28S-三芳香族类固醇[12]。
2.4 降解废水中的药物 世界范围内被药物(PCs)污染的水污染问题日益严重,因其具有较大的生态毒性和健康隐患,已经成为一种新兴的环境问题。微藻生物修复是一种太阳能驱动、生态综合、可持续的资源化利用策略,近年来微藻介导的PCs生物修复受到了科学的关注[6]。微藻去除PCs的机理包括生物吸附、生物富集和细胞内外生物降解。微藻对PCs的吸附包括但不仅限于双氯芬酸、布洛芬、对乙酰氨基酚、美托洛尔、三甲氧苄啶、卡马西平、雌二醇、b-雌二醇、炔雌醇等,吸附量达0%–16.7%。生物积累是由能量驱动的一种吸收底物的代谢过程。藻可以通过生物积累吸收有机污染物和生长养分。生物降解是微藻去除水相有机污染物最有效的途径。微藻通过催化降解复杂的母体化合物形成更简单的分子而达到降解PCs的目的。微藻能够分泌各种胞外聚合物,包括多糖、蛋白质、酶、取代基(多糖-链甲基-乙酰基)和脂质。胞外聚合物可以形成一种水合生物膜基质,通过保持细胞外酶与细胞的紧密结合,使其能够转化为具有溶解性、胶体性或固形性的有机化合物,从而起到体外消化系统的作用。
2.5 降解其他的有机化合物 菌藻共生体系愈来愈广泛地被用于修复各种废水中的有机物,除上述介绍的以外,还包括甲基叔丁基醚[49]、硫氰酸盐[50]、苯并[a]芘[51]、酚类化合物[52]、环烷酸[5]及水杨酸[53]等。通过菌藻共生体系来降解废水中的各种有机污染物,能够使污染物彻底降解,不会产生二次污染,有机物中碳、氮、磷、硫等元素进入相应的地球化学循环,净化环境。与此同时,微藻还可以产生生物质能源,实现双赢。
本课题组利用厌氧折流板反应器(ABR),以Fenton氧化为前处理方法,研究了不同氧环境菌藻共生系统对油田产出水中水解聚丙烯酰胺生物降解的影响,确定了降解水解聚丙烯酰胺的最适条件[54];在厌氧条件下,HPAM在酰胺酶的作用下发生水解,酰胺基团的C-N键断裂,水溶液中的-OH取代-NH2,释放出NH3,产生-COOH、HPAM的酰胺基团作为氮源被微生物利用;HPAM的碳骨架发生断裂,被降解为小分子化合物,作为碳源被微生物利用[55]。
3 结论和展望 菌藻共生体系在大自然中是广泛存在的,本文对菌藻共生体系中微藻与细菌的相互作用以及在废水处理中的应用进行了总结。微藻与细菌通过营养交换、信号转导以及基因转移实现二者的互利共生。菌藻共生体系在废水处理中的应用是非常有价值的,在整个处理过程中不需要额外提供O2便能够实现污染物的安全有效去除、CO2的排放与吸收、营养元素的有效循环及藻类生物量的平衡。
然而,对于产业化的污染水体连续处理,菌藻共生体系中的微藻和细菌需要严格筛选。苯乙烯、酚类化合物等有机污染物浓度高,可抑制微生物降解菌群,PAHs等化合物水溶性低,也会限制生物降解过程。因此,菌藻共生体系处理废水的工业化还有很长的一段路要走,进一步提高该体系的处理效率,或许更能引起企业的关注。
References
| [1] | Field CB, Behrenfeld MJ, Randerson JT, Falkowski P. Primary production of the biosphere:integrating terrestrial and oceanic components. Science, 1998, 281(5374): 237-240. DOI:10.1126/science.281.5374.237 |
| [2] | Quijano G, Arcila JS, Buitrón G. Microalgal-bacterial aggregates:applications and perspectives for wastewater treatment. Biotechnology Advances, 2017, 35(6): 772-781. DOI:10.1016/j.biotechadv.2017.07.003 |
| [3] | Liu JZ, Wu YH, Wu CX, Muylaert K, Vyverman W, Yu HQ, Mu?oz R, Rittmann B. Advanced nutrient removal from surface water by a consortium of attached microalgae and bacteria:a review. Bioresource Technology, 2017, 241: 1127-1137. DOI:10.1016/j.biortech.2017.06.054 |
| [4] | Loutseti S, Danielidis DB, Economou-Amilli A, Katsaros C, Santas R, Santas P. The application of a micro-algal/bacterial biofilter for the detoxification of copper and cadmium metal wastes. Bioresource Technology, 2009, 100(7): 2099-2105. DOI:10.1016/j.biortech.2008.11.019 |
| [5] | Mahdavi H, Prasad V, Liu Y, Ulrich AC. In situ biodegradation of naphthenic acids in oil sands tailings pond water using indigenous algae-bacteria consortium. Bioresource Technology, 2015, 187: 97-105. DOI:10.1016/j.biortech.2015.03.091 |
| [6] | Xiong JQ, Kurade MB, Jeon BH. Can microalgae remove pharmaceutical contaminants from water?. Trends in Biotechnology, 2017, 36(1): 30-44. |
| [7] | Posadas E, Mu?oz R, Guieysse B. Integrating nutrient removal and solid management restricts the feasibility of algal biofuel generation via wastewater treatment. Algal Research, 2017, 22: 39-46. DOI:10.1016/j.algal.2016.11.019 |
| [8] | Mu?oz R, Guieysse B. Algal-bacterial processes for the treatment of hazardous contaminants:a review. Water Research, 2006, 40(15): 2799-2815. DOI:10.1016/j.watres.2006.06.011 |
| [9] | Ryu BG, Kim J, Yoo G, Lim JT, Kim W, Han JI, Yang JW. Microalgae-mediated simultaneous treatment of toxic thiocyanate and production of biodiesel. Bioresource Technology, 2014, 158: 166-173. DOI:10.1016/j.biortech.2014.01.128 |
| [10] | Su YY, Mennerich A, Urban B. Comparison of nutrient removal capacity and biomass settleability of four high-potential microalgal species. Bioresource Technology, 2012, 124: 157-162. DOI:10.1016/j.biortech.2012.08.037 |
| [11] | Ramanan R, Kim BH, Cho DH, Oh HM, Kim HS. Algae-bacteria interactions:evolution, ecology and emerging applications. Biotechnology Advances, 2016, 34(1): 14-29. DOI:10.1016/j.biotechadv.2015.12.003 |
| [12] | Thompson H, Angelova A, Bowler B, Jones M, Gutierrez T. Enhanced crude oil biodegradative potential of natural phytoplankton-associated hydrocarbonoclastic bacteria. Environmental Microbiology, 2017, 19(7): 2843-2861. DOI:10.1111/emi.2017.19.issue-7 |
| [13] | Nambiar KR, Bokil SD. Luxury uptake of nitrogen in flocculating algal-bacterial system. Water Research, 1981, 15(6): 667-669. DOI:10.1016/0043-1354(81)90158-5 |
| [14] | Derry LA, Jacobsen SB. The chemical evolution of Precambrian seawater:evidence from REEs in banded iron formations. Geochimica et Cosmochimica Acta, 1990, 54(11): 2965-2977. DOI:10.1016/0016-7037(90)90114-Z |
| [15] | Salim S, Kosterink NR, Wacka NDT, Vermu? MH, Wijffels RH. Mechanism behind autoflocculation of unicellular green microalgae Ettlia texensis. Journal of Biotechnology, 2014, 174: 34-38. DOI:10.1016/j.jbiotec.2014.01.026 |
| [16] | Tang X, He LY, Tao XQ, Dang Z, Guo CL, Lu GN, Yi XY. Construction of an artificial microalgal-bacterial consortium that efficiently degrades crude oil. Journal of Hazardous Materials, 2010, 181(1/3): 1158-1162. |
| [17] | Wang M, Yang H, Ergas SJ, van der Steen P. A novel shortcut nitrogen removal process using an algal-bacterial consortium in a photo-sequencing batch reactor (PSBR). Water Research, 2015, 87: 38-48. DOI:10.1016/j.watres.2015.09.016 |
| [18] | Kouzuma A, Watanabe K. Exploring the potential of algae/bacteria interactions. Current Opinion in Biotechnology, 2015, 33: 125-129. DOI:10.1016/j.copbio.2015.02.007 |
| [19] | Takemura AF, Chien DM, Polz MF. Associations and dynamics of Vibrionaceae in the environment, from the genus to the population level. Frontiers in Microbiology, 2014, 5: 38. |
| [20] | Weinberger F, Friedlander M, Gunkel W. A bacterial facultative parasite of Gracilaria conferta. Diseases of Aquatic Organisms, 1994, 18: 135-141. DOI:10.3354/dao018135 |
| [21] | Van Oostende N, Moerdijk-Poortvliet TCW, Boschker HTS, Vyverman W, Sabbe K. Release of dissolved carbohydrates by Emiliania huxleyi and formation of transparent exopolymer particles depend on algal life cycle and bacterial activity. Environmental Microbiology, 2013, 15(5): 1514-1531. DOI:10.1111/emi.2013.15.issue-5 |
| [22] | Thompson AW, Foster RA, Krupke A, Carter BJ, Musat N, Vaulot D, Kuypers MM, Zehr JP. Unicellular cyanobacterium symbiotic with a single-celled eukaryotic alga. Science, 2012, 337(6101): 1546-1550. DOI:10.1126/science.1222700 |
| [23] | Le Chevanton M, Garnier M, Bougaran G, Schreiber N, Lukomska E, Bérard JB, Fouilland E, Bernard O, Cadoret JP. Screening and selection of growth-promoting bacteria for Dunaliella cultures. Algal Research, 2013, 2(3): 212-222. DOI:10.1016/j.algal.2013.05.003 |
| [24] | Kazamia E, Czesnick H, van Nguyen TT, Croft MT, Sherwood E, Sasso S, Hodson SJ, Warren MJ, Smith AG. Mutualistic interactions between vitamin B12-dependent algae and heterotrophic bacteria exhibit regulation. Environmental Microbiology, 2012, 14(6): 1466-1476. DOI:10.1111/j.1462-2920.2012.02733.x |
| [25] | Xie B, Bishop S, Stessman D, Wright D, Spalding MH, Halverson LJ. Chlamydomonas reinhardtii thermal tolerance enhancement mediated by a mutualistic interaction with vitamin B12-producing bacteria. The ISME Journal, 2013, 7(8): 1544-1555. DOI:10.1038/ismej.2013.43 |
| [26] | Krohn-Molt I, Wemheuer B, Alawi M, Poehlein A, Güllert S, Schmeisser C, Pommerening-R?ser A, Grundhoff A, Daniel R, Hanelt D, Streit WR. Metagenome survey of a multispecies and algae-associated biofilm reveals key elements of bacterial-algae interactions in photobioreactors. Applied and Environmental Microbiology, 2013, 79(20): 6196-6206. DOI:10.1128/AEM.01641-13 |
| [27] | Hughes DT, Sperandio V. Inter-kingdom signalling:communication between bacteria and their hosts. Nature Reviews Microbiology, 2008, 6(2): 111-120. DOI:10.1038/nrmicro1836 |
| [28] | Shimoyama T, Kato S, Ishii S, Watanabe K. Flagellum mediates symbiosis. Science, 2009, 323(5921): 1574-1574. DOI:10.1126/science.1170086 |
| [29] | Matsuo Y, Imagawa H, Nishizawa M, Shizuri Y. Isolation of an algal morphogenesis inducer from a marine bacterium. Science, 2005, 307(5715): 1598-1598. DOI:10.1126/science.1105486 |
| [30] | Jha B, Kavita K, Westphal J, Hartmann A, Schmitt-Kopplin P. Quorum sensing inhibition by Asparagopsis taxiformis, a marine macro alga:separation of the compound that interrupts bacterial communication. Marine Drugs, 2013, 11(1): 253-265. |
| [31] | Paul C, Pohnert G. Production and role of volatile halogenated compounds from marine algae. Natural Product Reports, 2011, 28(2): 186-195. DOI:10.1039/C0NP00043D |
| [32] | El Shoubaky GA, El Rahman Salem EA. Active ingredients fatty acids as antibacterial agent from the brown algae Padina pavonica and Hormophysa triquetra. Journal of Coastal Life Medicine, 2014, 2(7): 535-542. |
| [33] | Twigg MS, Tait K, Williams P, Atkinson S, Cámara M. Interference with the germination and growth of Ulva zoospores by quorum-sensing molecules from Ulva-associated epiphytic bacteria. Environmental Microbiology, 2014, 16(2): 445-453. DOI:10.1111/1462-2920.12203 |
| [34] | Pokrzywinski KL, Place AR, Warner ME, Coyne KJ. Investigation of the algicidal exudate produced by Shewanella sp. IRI-160 and its effect on dinoflagellates. Harmful Algae, 2012, 19: 23-29. DOI:10.1016/j.hal.2012.05.002 |
| [35] | Luo JF, Wang Y, Tang SS, Liang JW, Lin WT, Luo LX. Isolation and identification of algicidal compound from Streptomyces and algicidal mechanism to Microcystis aeruginosa. PLoS One, 2013, 8(10): e76444. DOI:10.1371/journal.pone.0076444 |
| [36] | Zhao L, Chen L, Yin PH. Algicidal metabolites produced by Bacillus sp. strain B1 against Phaeocystis globosa. Journal of Industrial Microbiology & Biotechnology, 2014, 41(3): 593-599. |
| [37] | Brembu T, Winge P, Tooming-Klunderud A, Nederbragt AJ, Jakobsen KS, Bones AM. The chloroplast genome of the diatom Seminavis robusta:new features introduced through multiple mechanisms of horizontal gene transfer. Marine Genomics, 2014, 16: 17-27. DOI:10.1016/j.margen.2013.12.002 |
| [38] | Moszczyński K, Mackiewicz P, Body? A. Evidence for horizontal gene transfer from bacteroidetes bacteria to dinoflagellate minicircles. Molecular Biology and Evolution, 2012, 29(3): 887-892. DOI:10.1093/molbev/msr276 |
| [39] | Allen AE, Dupont CL, Oborník M, Horák A, Nunes-Nesi A, McCrow JP, Zheng H, Johnson DA, Hu H, Fernie AR, Bowler C. Evolution and metabolic significance of the urea cycle in photosynthetic diatoms. Nature, 2011, 473(7346): 203-207. DOI:10.1038/nature10074 |
| [40] | Helliwell KE, Wheeler GL, Smith AG. Widespread decay of vitamin-related pathways:coincidence or consequence?. Trends in Genetics, 2013, 29(8): 469-478. DOI:10.1016/j.tig.2013.03.003 |
| [41] | Liang ZJ, Liu Y, Ge F, Xu Y, Tao NG, Peng F, Wong M. Efficiency assessment and pH effect in removing nitrogen and phosphorus by algae-bacteria combined system of Chlorella vulgaris and Bacillus licheniformis. Chemosphere, 2013, 92(10): 1383-1389. DOI:10.1016/j.chemosphere.2013.05.014 |
| [42] | Posadas E, García-Encina PA, Soltau A, Domínguez A, Díaz I, Mu?oz R. Carbon and nutrient removal from centrates and domestic wastewater using algal-bacterial biofilm bioreactors. Bioresource Technology, 2013, 139: 50-58. DOI:10.1016/j.biortech.2013.04.008 |
| [43] | Tang CC, Tian Y, Liang H, Zuo W, Wang ZW, Zhang J, He ZW. Enhanced nitrogen and phosphorus removal from domestic wastewater via algae-assisted sequencing batch biofilm reactor. Bioresource Technology, 2018, 250: 185-190. DOI:10.1016/j.biortech.2017.11.028 |
| [44] | Cai T, Park SY, Li YB. Nutrient recovery from wastewater streams by microalgae:status and prospects. Renewable and Sustainable Energy Reviews, 2013, 19: 360-369. DOI:10.1016/j.rser.2012.11.030 |
| [45] | Gon?alves AL, Pires JCM, Sim?es M. A review on the use of microalgal consortia for wastewater treatment. Algal Research, 2017, 24: 403-415. DOI:10.1016/j.algal.2016.11.008 |
| [46] | Simsek H, Kasi M, Ohm JB, Murthy S, Khan E. Impact of solids retention time on dissolved organic nitrogen and its biodegradability in treated wastewater. Water Research, 2016, 92: 44-51. DOI:10.1016/j.watres.2016.01.041 |
| [47] | Kalin M, Wheeler WN, Meinrath G. The removal of uranium from mining waste water using algal/microbial biomass. Journal of Environmental Radioactivity, 2005, 78(2): 151-177. DOI:10.1016/j.jenvrad.2004.05.002 |
| [48] | Mishamandani S, Gutierrez T, Berry D, Aitken MD. Response of the bacterial community associated with a cosmopolitan marine diatom to crude oil shows a preference for the biodegradation of aromatic hydrocarbons. Environmental Microbiology, 2016, 18(6): 1817-1833. DOI:10.1111/1462-2920.12988 |
| [49] | Zhong WH, Li YX, Sun KD, Jin J, Li XZ, Zhang FM, Chen JM. Aerobic degradation of methyl tert-butyl ether in a closed symbiotic system containing a mixed culture of Chlorella ellipsoidea and Methylibium petroleiphilum PM1. Journal of Hazardous Materials, 2011, 185(2/3): 1249-1255. |
| [50] | Ryu BG, Kim J, Farooq W, Han JI, Yang JW, Kim W. Algal-bacterial process for the simultaneous detoxification of thiocyanate-containing wastewater and maximized lipid production under photoautotrophic/photoheterotrophic conditions. Bioresource Technology, 2014, 162: 70-79. DOI:10.1016/j.biortech.2014.03.084 |
| [51] | Warshawsky D, LaDow K, Schneider J. Enhanced degradation of benzo[a]pyrene by Mycobacterium sp. in conjunction with green alga. Chemosphere, 2007, 69(3): 500-506. DOI:10.1016/j.chemosphere.2007.03.031 |
| [52] | Maza-Márquez P, González-Martínez A, Rodelas B, González-López J. Full-scale photobioreactor for biotreatment of olive washing water:structure and diversity of the microalgae-bacteria consortium. Bioresource Technology, 2017, 238: 389-398. DOI:10.1016/j.biortech.2017.04.048 |
| [53] | Mu?oz R, K?llner C, Guieysse B, Mattiasson B. Salicylate biodegradation by various algal-bacterial consortia under photosynthetic oxygenation. Biotechnology Letters, 2003, 25(22): 1905-1911. DOI:10.1023/B:BILE.0000003980.96235.fd |
| [54] | Pi YR, Zheng ZH, Bao MT, Li YM, Zhou YY, Sang GL. Treatment of partially hydrolyzed polyacrylamide wastewater by combined Fenton oxidation and anaerobic biological processes. Chemical Engineering Journal, 2015, 273: 1-6. DOI:10.1016/j.cej.2015.01.034 |
| [55] | Sang GL, Pi YR, Bao MT, Li YM, Lu JR. Biodegradation for hydrolyzed polyacrylamide in the anaerobic baffled reactor combined aeration tank. Ecological Engineering, 2015, 84: 121-127. DOI:10.1016/j.ecoleng.2015.07.028 |
