朱文俊, 毛雪玲, 邱晓挺


宁波大学海洋学院, 应用海洋生物技术教育部重点实验室, 浙江 宁波 315211
收稿日期:2017-12-08;修回日期:2018-01-31;网络出版日期:2018-05-25
基金项目:国家自然科学基金(31400683);宁波大学人才引进启动基金(013-E00843134702,013-E00843144702,013-421504460);宁波大学王宽诚基金;宁波大学添路职业发展基金学生培育项目
*通信作者:邱晓挺, Tel:+86-574-87609572, E-mail:xiaotingqiu@126.com
摘要:RNA聚合酶Ⅱ最大亚基Rpb1的羧基端结构域(carboxyl-terminal repeat domain,CTD)是RNA聚合酶Ⅱ发挥转录延伸功能所必需的,对其执行精确的转录调节功能至关重要。酵母细胞周期蛋白依赖性激酶CTDK-Ⅰ(carboxyl-terminal repeat domain kinase,CTDK-Ⅰ)由CTK1、CTK2和CTK3组成,作用于RNA聚合酶Ⅱ羧基端结构域,动态磷酸化CTD的七肽重复序列(YSPTSPS)来调控转录和翻译。酵母中的特异性蛋白CTK3与特殊的细胞周期蛋白CTK2结合形成异二聚体,再与CTDK-Ⅰ的催化亚基CTK1结合以调节其活性。CTK1作为细胞周期蛋白CDK(cyclin dependent kinase,CDK)的同源蛋白,其结构与功能的研究可拓展人们对CDK蛋白家族的认识;CTK2-CTK3复合物对CTK1调控机制的研究也可为细胞周期蛋白抑制剂的研发提供新的思路。本文简述了酵母CTDK-Ⅰ的功能特点及其亚基的结构与功能以及亚基间的相互作用,并展望了CTDK-Ⅰ复合物的研究前景。
关键词: 细胞周期蛋白依赖性激酶 磷酸化 转录调控 亚基相互作用
Structure and function of yeast RNA polymerase Ⅱ carboxyl-terminal repeat domain kinase CTDK-Ⅰ and its subunits
Wenjun Zhu, Xueling Mao, Xiaoting Qiu


Key Laboratory of Applied Marine Biotechnology of Ministry of Education, School of Marine Sciences, Ningbo University, Ningbo 315211, Zhejiang Province, China
Received 8 December 2017; Revised 31 January 2018; Published online 25 May 2018
*Corresponding author: Qiu Xiaoting, Tel:+86-574-87609572, E-mail:xiaotingqiu@126.com
Supported by the National Natural Science Foundation of China (31400683), by the Talent Plan of Ningbo University (013-E00843134702, 013-E00843144702, 013-421504460), by the K. C. Wong Magna Fund in Ningbo University and by the Student Cultivation Fund of Professional Development in Ningbo University
Abstract: Carboxyl-terminal repeat domain (CTD) of RNA polymerase Ⅱ largest subunit Rpb1 is essential for transcription regulation. Carboxyl-terminal repeat domain kinase (CTDK-Ⅰ) is composed of CTK1, CTK2 and CTK3, acting on RNA polymerase Ⅱ carboxyl-terminal repeat domain and phosphorylating CTD heptapeptide repeat (YSPTSPS) for regulating transcription and translation. The specific protein CTK3 binds to cyclin CTK2 to form a heterodimer, controlling CTK1 activity by binding to CTK1. Structural and functional study of CTK1, a homologous protein of cyclin dependent kinase (CDK), may provide a new idea for the research of CDK family, and analysis of the regulatory mechanism of activation of CTK1 by CTK2-CTK3 complex may offer an innovative method for developing cell cycle protein inhibitors. This article reviews the functional characteristics of CTDK-Ⅰ and the structures and interactions of its subunits and provides a useful guide for the studies of CTDK-Ⅰ complex in the future.
Keywords: carboxy-terminal repeat domain kinase phosphorylation transcription regulation subunit interaction
转录是基因信息表达的初始步骤,其各个阶段均受到精细调控[1]。RNA聚合酶作为转录的执行者,其修饰是转录调控的重要环节,一直是研究焦点[2-3]。RNA聚合酶Ⅱ是RNA聚合酶家族中重要的一员,可以结合在真核生物基因的上游,执行和调控mRNA的转录。其中,RNA聚合酶Ⅱ的最大亚基Rpb1的羧基端结构域是RNA聚合酶Ⅱ发挥转录延伸功能所必需的,对其执行精确的转录调节功能至关重要[4]。CTD中含有一个特殊的重复七肽序列(Tyr1-Ser2-Pro3-Thr4-Ser5-Pro6- Ser7)。在转录过程中,各种转录因子周期性地动态磷酸化七肽序列以调控转录的进行[5]。从目前的研究可知,七肽序列上Ser2和Ser5为主要的磷酸化位点,Tyr1、Thr4和Ser7为次要的磷酸化位点,但是这些磷酸化位点都对转录的调节和细胞正常功能的运行十分重要[6]。
细胞周期性依赖激酶是RNA聚合酶Ⅱ CTD七肽序列众多转录调控因子中的一员。迄今在酵母中已发现了参与CTD动态磷酸化的4种不同的细胞周期蛋白依赖激酶:Kin28、Srb10、Bur1和CTK1[7]。激酶Kin28具有与人源CDK7相似的结构,是起始因子TFIIH的亚基之一,作为mRNA前体的加工因子参与CTD磷酸化过程,还可以招募加帽酶——鸟苷酰基转移酶Ceg1。Ceg1可以与CTD上磷酸化的Ser2或Ser5结合,但只有通过Arg159、Arg185和Lys198残基与磷酸化的Ser5结合的Ceg1才处于能够对mRNA前体进行加工修饰的活性构象[8-10];激酶Srb10能与细胞周期蛋白Srb11结合,形成的Srb10-Srb11复合物与Kin28-Ccl1复合物共同形成转录调节环,在细胞分裂G1期,Kin28-Ccl1与转录因子TFIIH相互作用,促进细胞分裂进入S期,但由于Srb10-Srb11对Kin28-Ccl1的负调节,会导致细胞在G1期发生阻滞现象[11-14]。此外,Srb10-Srb11对于CTD的作用与人源CDK8-cyclinC复合物的功能相似,具有磷酸化CTD七肽序列中的Ser的能力[15-16];激酶Bur1则与CTK1共同发挥作用,Bur1磷酸化Ser2后会刺激CTK1进行随后的磷酸化[10, 17]。酵母的CTK1属于蛋白激酶超家族、Thr/Ser磷酸激酶家族、CDC2/CDKX亚家族,其主要存在于酿酒酵母、接合酵母和念珠地菌等单细胞真核生物中,但在高等多细胞真核生物体内如非洲爪蟾、小麦和人类中也存在底物特异性和受调节方式分化的同源蛋白,这也从侧面说明了CTK1及其同源蛋白在真核生物中的功能多样性和重要性。(图 1-A)。
 |
| 图 1 CTK1的亲缘关系分析与CTK1、CDK9和CDK12激酶结构域的序列比对 Figure 1 Phylogenetic analysis of CTK1 and sequence comparison of kinase domains of CTK1, CDK9 and CDK12. A: phylogenetic analysis of CTK1 from different organisms and other cyclin dependent kinases. Bar, 0.2 protein substitution rate. B: sequence comparison of kinase domain of CTK1 from Saccharomyces cerevisiae and kinase domains of CDK9 and CDK12, both from Homo sapiens. Conserved ATP binding sites and substrate binding sites are labeled by blue and green triangles, respectively. There are so many common sequences among CTK1, CDK9 and CDK12, which suggests that CTK1 is the homologous protein of CDK9 and CDK12. |
| 图选项 |
1 细胞周期蛋白依赖性激酶CTDK-Ⅰ功能的发现 1989年,Lee等[18]发现了具有磷酸化酿酒酵母RNA聚合酶Ⅱ CTD能力的激酶CTDK-Ⅰ,并发现其是由CTK1、CTK2和CTK3三个亚基组成。后来,Sterner等[19]对CDTK-1的3个亚基分别进行敲除或全部敲除使CTDK-Ⅰ部分失效或者全部失效来观察CTDK-Ⅰ对酵母生理活性的影响。结果发现菌株在丧失CTDK-Ⅰ活性条件下出现生长缓慢、形态异常、形成的孢子存在生理缺陷以及不能在低温下生长的现象(冷敏感表型)。磷酸化的RNA聚合酶Ⅱ CTD的特异性抗体实验也揭示了这些突变菌株严重缺少磷酸化形式的CTD[19-20]。Patturajan等[21]进行了突变CTDK-Ⅰ内CTK1亚基的实验,发现CTK1的缺失所导致的CTD在磷酸化过程中七肽重复序列上2号位丝氨酸磷酸化突增的现象在酵母的二次转移时期(酵母生长过程中从厌氧代谢至需氧代谢的过程)消失,这种缺陷与转录时期CTD的不完全磷酸化有关,也从侧面说明了CTDK-Ⅰ活性的缺陷影响了酵母菌对生长所需营养物的利用[22]。上述研究结果表明,CTDK-Ⅰ在酵母细胞中通过动态磷酸化RNA聚合酶Ⅱ的CTD结构域来调节mRNA转录与延伸,CTDK-Ⅰ的突变或者缺失会使酵母细胞在生长过程中出现缺陷。
2 CTDK-Ⅰ亚基的功能 2.1 CTK1亚基的功能 CTK1是CTDK-Ⅰ三个亚基中最重要的亚基,是CTDK-Ⅰ的催化亚基,实际执行磷酸化反应。CTK1主要存在于酵母细胞核内,呈稳态分布[23]。
CTK1 (Saccharomyces cerevisiae)、CDK9 (Homo sapiens)和CDK12 (Homo sapiens)同为细胞周期性依赖激酶,3个蛋白的功能域具有较高的相似性(图 1-B)。3个细胞周期依赖性激酶在调节mRNA转录过程中都可以磷酸化RNA聚合酶Ⅱ的CTD上Ser2位点[24]。不同的是,CDK9磷酸化Ser2主要发生在转录的起始阶段,加强对其他转录延伸因子的招募。除此之外,CDK9还可以磷酸化转录延伸因子Spt5。磷酸化使Spt5的构象发生转变,进而减慢Spt5相对于RNA聚合酶Ⅱ的移动速率,使其在与RNA聚合酶Ⅱ一起移动的过程中与后者脱离,从而影响转录延伸的进行[25-26]。CDK12磷酸化Ser2起调停转录的作用,此外还参与DNA的修复、染色体的组织和细胞周期的调节[27]。其在体外表现出明显的CTD激酶的活性,直接参与磷酸化反应,并且Bartkowiak等[28]的基因敲除实验也表明CDK12的主要功能是磷酸化CTD。相比而言,CTK1可以在酵母细胞内与Spt6 (Spt5同族蛋白)形成前馈回路(feed-forward loop)。当CTD磷酸化准备进行时,Bur1先磷酸化CTD重复序列的Ser2位点,招募并激活具有调节组蛋白甲基化、mRNA加工和调节转录延伸和终止功能的中间蛋白Paf,Paf对Spt6进行招募,然后Spt6与CTD上磷酸化的Ser2相互作用,从而稳定在CTD表面并不断把CTK1招募到RNA聚合酶Ⅱ附近[19],形成CTD结合Spt6与CTK1磷酸化CTD的前馈回路[17]。
在单细胞真核生物酿酒酵母中,CTK1不只是以单体的形式存在于酵母体内来调控RNA聚合酶Ⅱ行使功能,其主要与细胞周期蛋白亚基CTK2 (与人细胞周期蛋白K同源)和一个与已知蛋白没有任何相似性且功能未知的蛋白亚基CTK3 (酵母特异性亚基)共同形成复合物CTDK-Ⅰ,以调节RNA聚合酶Ⅱ末端CTD,因此CTDK-Ⅰ与大多数由催化亚基和调节亚基组成细胞周期蛋白依赖性激酶复合物存在显著差异[29]。
CTK1参与的生理活动还包括mRNA 3′端的形成、细胞核通道的形成、细胞自噬活动以及DNA损伤的修复等[30-31]。van Driessche等[22]发现细胞在葡萄糖浓度较低的条件下生长时,CTK2和CTK3会与Snf1或者Snf1相关的蛋白结合成足以致死的蛋白复合物,但是CTK1的存在会与Snf1形成竞争,优先与CTK2和CTK3结合成复合物CTDK-Ⅰ,保护细胞因为葡萄糖限制而产生致死性的应答活动。乐雁等[32]发现CTK1的缺失会使酵母细胞内的液泡无法与形成的自噬体相融合,而且在酵母CTV选择性自噬(cytosol to vacuole targeting autophagy)过程中,CTK1的缺失会导致反应底物Ape1无法进入到细胞液泡内部,也会使得自噬体标记分子Atg8无法与调节因子Atg3结合,从而影响自噬作用。
在细胞核内,染色质组蛋白的修饰会影响mRNA的转录起始和延伸效率。出现在活性基因转录起始位点附近的组蛋白H3K4位的三甲基化与转录的起始有关,该甲基化由组蛋白甲基转移酶Set1负责调控。在转录起始阶段,Kin28磷酸化CTD七肽序列的Ser5位,各种转录因子和COMPASS (complex of proteins associated with Set1)复合物被招募到编码区域的5′端,Set1局部三甲基化H3K4,促进转录[33-36]。随后在转录起始的结束阶段,CTK1磷酸化CTD的Ser2,COMPASS则与RNA聚合酶Ⅱ脱离[5, 34](图 2)。在CTK1磷酸化CTD调控组蛋白甲基化的过程中,CTD的磷酸化程度是逐步提升的,但是CTD内七肽重复序列磷酸化的顺序并不明确[35],CTK1作为酵母细胞内主要的CTD激酶,其结构和功能的研究对于明确重复序列磷磷化的顺序和理解mRNA的转录起始与延伸的精确分步调控具有重要意义。
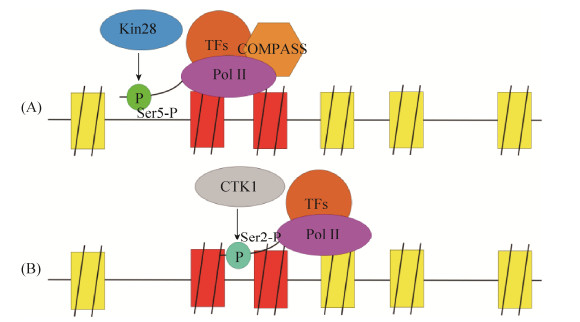 |
| 图 2 组蛋白H3K4转录起始阶段三甲基化模式 Figure 2 Model explaining the pathway of H3K4 trimethylation during transcription initiation. A: the CTD is phosphorylated by Kin28 at serine 5 and various transcription factors and COMPASS are recruited to the 5' portion of coding region. B: the CTD is phosphorylated by CTK1 at serine 2 and COMPASS separate from RNA polymerase Ⅱ. |
| 图选项 |
Rps2是影响mRNA翻译准确性的3个蛋白中的一个,其与Rps5、Rps12共同被定位于密码子-反密码子相互作用区域的对侧,它们可以与16S rRNA的颈环区内高度保守的530个核苷酸片段相互作用以减少翻译误差。这些功能蛋白(Rps2、Rps5和Rps12)的突变可能会引起16S rRNA结构的改变,导致错误编码几率的提高[37-38]。R?ther等[23]证明CTK1的缺失极大影响了翻译的准确性,并结合酿酒酵母80S核糖体的结构说明CTK1在翻译期间通过磷酸化位于核糖体mRNA通道入口位置的Rps2的Ser238残基以改变其构象,来增加翻译的准确性[23, 39]。
2.2 CTK2与CTK3亚基的结构与功能 CTK3是只存在于酵母中的一种与已知蛋白没有任何相似性的特异性蛋白,目前只解析了裂殖酵母CTK3的N端结构域的结构,其完整结构尚未获得。CTK3的N端结构域存在8个α螺旋,包含一个可以与RNA聚合酶Ⅱ CTD结合的类似于CID (CTD-interacting domain)的区域;然而,CTK3 N端结构域的残基Gln21和Lys111面向结合槽的侧链会对CTD结合槽产生封闭作用[7]。并且,CTK3 N端的CID结构域与典型的CID相比较其分子表面的保守性有所不同[7]。典型的CID结构例如Rtt103、Nrd1等,其CTD结合槽具有很强的保守性,但是在CTK3内却只显示了微弱的保守性[6, 40] (图 3)。
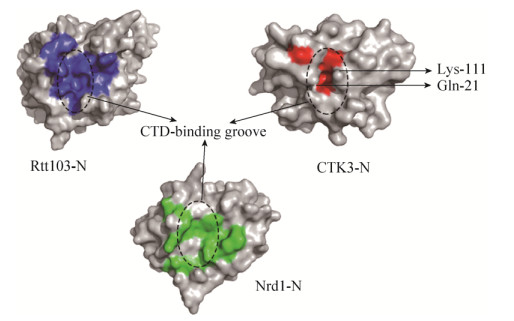 |
| 图 3 CTK3与其同源蛋白的结构比对 Figure 3 Structural comparison of CTK3 and its homologues. Structural comparison of the N-terminal domain of CTK3 from Schizosaccharomyces pombe, Nrd1 and Rtt103, both from Saccharomyces cerevisiae. The structures are shown as surfaces. CTD-binding groove is indicated by a black dashed circle. Residues locating at the surface of Nrd1 and Rtt103 that correspond to CTD interaction are labeled in green and blue, respectively; Residues locating at the surface of CTK3 that correspond to CTD-interacting region are labeled in red. |
| 图选项 |
CTK3 N端结构域还与典型的CID结构域在电荷分布上有所不同。典型的CID结合槽的表面一般呈现正电荷,有助于与带负电荷的CTD相结合,实现磷酸化CTD的目的。但是,CTK3 N端结构域的主要电荷却和CID结合槽不同,呈现为负电荷[7]。因此CTK3 N端结构域与CTD所带电荷相斥,无法相互结合。
CTDK-Ⅰ亚基蛋白CTK3 N端存在一个PEST序列,PEST序列会加强目的蛋白和泛素连接酶的结合,引起CTK3蛋白的泛素化,最后CTK3通过26S蛋白酶体的途径进行降解[41-42],因此根据N端法则,CTK3的半衰期明显要比其他蛋白短。但是,CTK3在酵母细胞中通常会与CTK2相互结合,将其PEST序列包裹进蛋白复合物的内部,从而避免自身被降解。Hautbergue等[29]发现CTK3 C末端大约50个氨基酸的缺失会造成CTK3无法与CTK2相互结合,导致CTK3迅速被泛素-蛋白酶体途径降解。这50个氨基酸不仅用于与CTK2相结合,形成CTK2-CTK3复合物共同调解CTK1的活性,还有助于改变CTK3 N末端部分结构的形态,避免了CTK3和CTK2-CTK3复合物的降解。目前,我们已经成功获得酿酒酵母CTK2-CTK3复合物的晶体并收集了其衍射数据,尝试通过解析复合物的结构来解释CTK3-CTK2复合物的形成保护CTK3不被降解的机制。
CTK2的序列与酵母中的其他细胞周期蛋白依赖性激酶亚基有显著差别,其既不存在引导泛素连接酶的PEST序列也没有支持细胞周期蛋白快速降解的破坏盒结构[29]。奇怪的是CTK2单体在酵母的生理活动中会被快速磷酸化,然后马上被降解,但是其降解方式又不像我们所熟知的G1周期蛋白被泛素-蛋白酶体途径选择性降解,CTK2降解的起始并不需要激酶的识别,也不类似于白细胞的吞噬[29]。这种特殊的形式是否属于一种新的细胞周期蛋白调节机制还有待进一步的研究。
CTK2的降解机制虽然不清楚,但是CTK2可以通过调节CTK3的周转,与CTK3 C末端的50个左右的氨基酸残基相互绑定形成CTK2-CTK3复合物来保证彼此的稳定。Hautbergue等[29]发现只有当CTK2足量表达的时候,CTK3才可能被明显检测到。
因此,CTK2虽然有可能与细胞周期调节有关,但并不直接参与细胞周期相关的生理功能,而是通过调控蛋白质周转活动间接参与到细胞周期活动中。尽管CTK2与CTK3形成与CDK细胞周期蛋白相似的复合体,但是CTK2却不能直接激活细胞周期。与CTK3一样,CTK2和CTK3虽然都可以独自与CTK1激酶相互作用,但并不产生具有活性的复合物。只有CTK2和CTK3与CTK1共同形成的三元复合物CTDK-Ⅰ才具备激酶活性。
综上所述,CTK2和CTK3在CTDK-Ⅰ复合物中参与催化亚基CTK1活性的调控,间接调节转录起始与延伸。此外,CTK2和CTK3不含有核靶向信号,而CTDK-Ⅰ的装配发生在胞质中,CTK1是如何出核参与翻译以及形成的CTDK-Ⅰ复合物又是如何回到细胞核内参与转录的过程还需要进一步研究[23, 29]。
3 展望 CTDK-Ⅰ及其亚基的结构与功能的研究有助于理解酵母细胞周期蛋白依赖性激酶的作用机制以及这类激酶参与并调控酵母的转录活动的方式,这将扩展对真核生物RNA聚合酶Ⅱ调控机制的认识。CTK2和CTK3都是不稳定蛋白,单独存在容易被快速降解且CTK2的降解机制并不清楚。当CTK3与CTK2结合,2个亚基便能保持各自稳定,抗拒降解,但这2个亚基的相互作用界面尚未明确,这需要CTK2-CTK3复合物的结构信息。虽然已知CTK3-CTK2复合物可以激活并调节CTK1的活性,但是CTK3-CTK2对CTK1活性的调节机制——CTK3-CTK2与CTK1的结合引起了后者哪些区域的构象变化尚未知晓,这是否为一种新的细胞周期蛋白调节机制也值得探究。这需要单独的CTK1晶体结构和CTDK-Ⅰ复合物中的CTK1的结构比较并配合相应残基的突变实验来揭示。
随着结晶技术和新的结构解析技术迅速发展,特别是微流体芯片结晶、X射线自由电子激光和冷冻电镜技术的突破性发展,极大地推动了蛋白结构解析技术的进步[43-44]。近年来电子直接探测相机结合冷冻电镜解析高分辨率的蛋白结构的方法使一些难以形成结晶体或者结晶条件难以控制的蛋白的高分辨率结构解析成为可能[45]。
CTDK-Ⅰ作为酿酒酵母体内调控mRNA转录以及延伸的重要酶类,其整体结构和各个亚基结构的研究一直处于停滞不前的阶段,但X-衍射技术的进步、冷冻电镜技术的发展以及结构解析算法的改进为CTDK-Ⅰ及其各亚基结构的解析提供了重要保障[46-47]。CTDK-Ⅰ及其各亚基结构的解析有利于加深人们对RNA聚合酶Ⅱ在转录起始和延伸中受调控机制的认识。CTK3对CTK2的调节机制将随着结构的解析而被揭示,也许这是一种新的细胞周期调节机制。我们将把酿酒酵母CTK2-CTK3复合物与部分CDK家族细胞周期蛋白激酶孵育,研究CTK2-CTK3复合物能否与激活因子竞争性结合CDK以抑制CDK的激酶活性,为细胞周期抑制剂的研究提供新的方法,并可为研究真核生物的细胞周期蛋白依赖性激酶调节亚基的分子进化历程提供思路。
References
| [1] | Maniatis T, Reed R. An extensive network of coupling among gene expression machines. Nature, 2002, 416(6880): 499-506. DOI:10.1038/416499a |
| [2] | Liu PD, Greenleaf AL, Stiller JW. The essential sequence elements required for RNAP Ⅱ carboxyl-terminal domain function in yeast and their evolutionary conservation. Molecular Biology and Evolution, 2008, 25(4): 719-727. DOI:10.1093/molbev/msn017 |
| [3] | Proudfoot NJ, Furger A, Dye JM. Integrating mRNA processing with transcription. Cell, 2002, 108(4): 501-512. DOI:10.1016/S0092-8674(02)00617-7 |
| [4] | Bouchoux C, Hautbergue G, Grenetier S, Carles C, Riva M, Goguel V. CTD kinase I is involved in RNA polymerase I transcription. Nucleic Acids Research, 2004, 32(19): 5851-5860. DOI:10.1093/nar/gkh927 |
| [5] | Porrua O, Libri D. Transcription termination and the control of the transcriptome:why, where and how to stop. Nature Reviews Molecular Cell Biology, 2015, 16(3): 190-202. DOI:10.1038/nrm3943 |
| [6] | Jasnovidova O, Klumpler T, Kubicek K, Kalynych S, Plevka P, Stefl R. Structure and dynamics of the RNAPⅡ CTDsome with Rtt103. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(42): 11133-11138. DOI:10.1073/pnas.1712450114 |
| [7] | Mühlbacher W, Mayer A, Sun M, Remmert M, Cheung ACM, Niesser J, Soeding J, Cramer P. Structure of Ctk3, a subunit of the RNA polymerase Ⅱ CTD kinase complex, reveals a noncanonical CTD-interacting domain fold. Proteins, 2015, 83(10): 1849-1858. DOI:10.1002/prot.24869 |
| [8] | Rodriguez CR, Cho EJ, Keogh MC, Moore CL, Greenleaf AL, Buratowski S. Kin28, the TFⅡH-associated carboxy-terminal domain kinase, facilitates the recruitment of mRNA processing machinery to RNA polymerase Ⅱ. Molecular and Cellular Biology, 2000, 20(1): 104-112. DOI:10.1128/MCB.20.1.104-112.2000 |
| [9] | Bharati AP, Singh N, Kumar V, Kashif M, Singh AK, Singh P, Singh SK, Siddiqi MI, Tripathi T, Akhtar MS. The mRNA capping enzyme of Saccharomyces cerevisiae has dual specificity to interact with CTD of RNA Polymerase Ⅱ. Scientific Reports, 2016, 6: 31294. DOI:10.1038/srep31294 |
| [10] | Qiu HF, Hu CH, Hinnebusch AG. Phosphorylation of the pol Ⅱ CTD by KIN28 enhances BUR1/BUR2 recruitment and ser2 CTD phosphorylation near promoters. Molecular Cell, 2009, 33(6): 752-762. DOI:10.1016/j.molcel.2009.02.018 |
| [11] | Hallberg M, Polozkov GV, Hu GZ, Beve J, Gustafsson CM, Ronne H, Bj?rklund S. Site-specific srb10-dependent phosphorylation of the yeast Mediator subunit Med2 regulates gene expression from the 2-μm plasmid. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(10): 3370-3375. DOI:10.1073/pnas.0400221101 |
| [12] | Nelson C, Goto S, Lund K, Hung W, Sadowski I. Srb10/Cdk8 regulates yeast filamentous growth by phosphorylating the transcription factor Ste12. Nature, 2003, 421(6919): 187-190. DOI:10.1038/nature01243 |
| [13] | Rosonina E, Duncan SM, Manley JL. Sumoylation of transcription factor Gcn4 facilitates its Srb10-mediated clearance from promoters in yeast. Genes & Development, 2012, 26(4): 350-355. |
| [14] | Ohkuni K, Yamashita I. A transcriptional autoregulatory loop for KIN28-CCL1 and SRB10-SRB11, each encoding RNA polymerase Ⅱ CTD kinase-cyclin pair, stimulates the meiotic development of S. cerevisiae. Yeast, 2000, 16(9): 829-846. DOI:10.1002/(ISSN)1097-0061 |
| [15] | Kuchin S, Carlson M. Functional relationships of Srb10-Srb11 kinase, carboxy-terminal domain kinase CTDK-Ⅰ, and transcriptional corepressor Ssn6-Tup1. Molecular and Cellular Biology, 1998, 18(3): 1163-1171. DOI:10.1128/MCB.18.3.1163 |
| [16] | Hengartner CJ, Myer VE, Liao SM, Wilson CJ, Koh SS, Young RA. Temporal regulation of RNA polymerase Ⅱ by Srb10 and Kin28 cyclin-dependent kinases. Molecular Cell, 1998, 2(1): 43-53. DOI:10.1016/S1097-2765(00)80112-4 |
| [17] | Dronamraju R, Strahl BD. A feed forward circuit comprising Spt6, Ctk1 and PAF regulates Pol Ⅱ CTD phosphorylation and transcription elongation. Nucleic Acids Research, 2014, 42(2): 870-881. DOI:10.1093/nar/gkt1003 |
| [18] | Lee JM, Greenleaf AL. A protein kinase that phosphorylates the C-terminal repeat domain of the largest subunit of RNA polymerase Ⅱ. Proceedings of the National Academy of Sciences of the United States of America, 1989, 86(10): 3624-3628. DOI:10.1073/pnas.86.10.3624 |
| [19] | Sterner DE, Lee JM, Hardin SE, Greenleaf AL. The yeast carboxyl-terminal repeat domain kinase CTDK-Ⅰ is a divergent cyclin-cyclin-dependent kinase complex. Molecular and Cellular Biology, 1995, 15(10): 5716-5724. DOI:10.1128/MCB.15.10.5716 |
| [20] | Lee JM, Greenleaf AL. CTD kinase large subunit is encoded by CTK1, a gene required for normal growth of Saccharomyces cerevisiae. Gene Expression, 1991, 1(2): 149-167. |
| [21] | Patturajan M, Conrad NK, Bregman DB, Corden JL. Yeast carboxyl-terminal domain kinase I positively and negatively regulates RNA polymerase Ⅱ carboxyl-terminal domain phosphorylation. Journal of Biological Chemistry, 1999, 274(39): 27823-27828. DOI:10.1074/jbc.274.39.27823 |
| [22] | van Driessche B, Coddens S, van Mullem V, van den Haute J. Glucose deprivation mediates interaction between CTDK-Ⅰ and Snf1 in Saccharomyces cerevisiae. FEBS Letters, 2005, 579(24): 5318-5324. DOI:10.1016/j.febslet.2005.08.057 |
| [23] | R?ther S, Str??er K. The RNA polymerase Ⅱ CTD kinase Ctk1 functions in translation elongation. Genes & Development, 2007, 21(11): 1409-1421. |
| [24] | Bowman EA, Kelly WG. RNA polymerase Ⅱ transcription elongation and Pol Ⅱ CTD Ser2 phosphorylation. Nucleus, 2014, 5(3): 224-236. DOI:10.4161/nucl.29347 |
| [25] | Kim YK, Bourgeois CF, Isel C, Churcher MJ, Karn J. Phosphorylation of the RNA polymerase Ⅱ carboxyl-terminal domain by CDK9 is directly responsible for human immunodeficiency virus type 1 tat-activated transcriptional elongation. Molecular and Cellular Biology, 2002, 22(13): 4622-4637. DOI:10.1128/MCB.22.13.4622-4637.2002 |
| [26] | Zhou MS, Halanski MA, Radonovich MF, Kashanchi F, Peng JM, Price DH, Brady JN. Tat modifies the activity of CDK9 to phosphorylate serine 5 of the RNA polymerase Ⅱ carboxyl-terminal domain during human immunodeficiency virus type 1 transcription. Molecular and Cellular Biology, 2000, 20(14): 5077-5086. DOI:10.1128/MCB.20.14.5077-5086.2000 |
| [27] | Liang KW, Gao X, Gilmore JM, Florens L, Washburn MP, Smith E, Shilatifard A. Characterization of human cyclin-dependent kinase 12(CDK12) and CDK13 complexes in C-terminal domain phosphorylation, gene transcription, and RNA processing. Molecular and Cellular Biology, 2015, 35(6): 928-938. DOI:10.1128/MCB.01426-14 |
| [28] | Bartkowiak B, Liu PD, Phatnani HP, Fuda NJ, Cooper JJ, Price DH, Adelman K, Lis JT, Greenleaf AL. CDK12 is a transcription elongation-associated CTD kinase, the metazoan ortholog of yeast Ctk1. Genes & Development, 2010, 24(20): 2303-2316. |
| [29] | Hautbergue G, Goguel V. Activation of the cyclin-dependent kinase CTDK-Ⅰ requires the heterodimerization of two unstable subunits. Journal of Biological Chemistry, 2001, 276(11): 8005-8013. DOI:10.1074/jbc.M010162200 |
| [30] | Ostapenko D, Solomon MJ. Budding yeast CTDK-Ⅰ is required for DNA damage-induced transcription. Eukaryotic Cell, 2003, 2(2): 274-283. DOI:10.1128/EC.2.2.274-283.2003 |
| [31] | Jona G, Wittschieben B?, Svejstrup JQ, Gileadi O. Involvement of yeast carboxy-terminal domain kinase I (CTDK-Ⅰ) in transcription elongation in vivo. Gene, 2001, 267(1): 31-36. DOI:10.1016/S0378-1119(01)00389-4 |
| [32] | Le Y, Zhou LJ, Yi C. Function of Ctk1 in autophagy process. Chinese Journal of Cell Biology, 2014, 36(7): 877-883. (in Chinese) 乐雁, 周兰姜, 易聪. Ctk1蛋白在自噬过程中的作用. 中国细胞生物学学报, 2014, 36(7): 877-883. |
| [33] | Ng HH, Robert F, Young RA, Struhl K. Targeted recruitment of set1 histone methylase by elongating pol Ⅱ provides a localized mark and memory of recent transcriptional activity. Molecular Cell, 2003, 11(3): 709-719. DOI:10.1016/S1097-2765(03)00092-3 |
| [34] | Wood A, Shukla A, Schneider J, Lee JS, Stanton JD, Dzuiba T, Swanson SK, Florens L, Washburn MP, Wyrick J, Bhaumik SR, Shilatifard A. Ctk complex-mediated regulation of histone methylation by COMPASS. Molecular and Cellular Biology, 2007, 27(2): 709-720. DOI:10.1128/MCB.01627-06 |
| [35] | Piunti A, Shilatifard A. Epigenetic balance of gene expression by Polycomb and COMPASS families. Science, 2016, 352(6290): aad9780. DOI:10.1126/science.aad9780 |
| [36] | Jiang DH, Kong NC, Gu XF, Li ZC, He YH. Arabidopsis COMPASS-like complexes mediate histone H3 lysine-4 trimethylation to control floral transition and plant development. PLoS Genetics, 2011, 7(3): e1001330. DOI:10.1371/journal.pgen.1001330 |
| [37] | Coordes B, Brünger KM, Burger K, Soufi B, Horenk J, Eick D, Olsen JV, Str??er K. Ctk1 function is necessary for full translation initiation activity in Saccharomyces cerevisiae. Eukaryotic Cell, 2015, 14(1): 86-95. DOI:10.1128/EC.00106-14 |
| [38] | Eustice DC, Wakem LP, Wilhelm JM, Sherman F. Altered 40 S ribosomal subunits in omnipotent suppressors of yeast. Journal of Molecular Biology, 1986, 188(2): 207-214. DOI:10.1016/0022-2836(86)90305-0 |
| [39] | Swiercz R, Cheng DH, Kim D, Bedford MT. Ribosomal protein rpS2 is hypomethylated in PRMT3-deficient mice. Journal of Biological Chemistry, 2007, 282(23): 16917-16923. DOI:10.1074/jbc.M609778200 |
| [40] | Morales JC, Richard P, Rommel A, Fattah FJ, Motea EA, Patidar PL, Xiao L, Leskov K, Wu SY, Hittelman WN, Chiang CM, Manley JL, Boothman DA. Kub5-Hera, the human Rtt103 homolog, plays dual functional roles in transcription termination and DNA repair. Nucleic Acids Research, 2014, 42(8): 4996-5006. DOI:10.1093/nar/gku160 |
| [41] | Rogers S, Wells R, Rechsteiner M. Amino acid sequences common to rapidly degraded proteins:the PEST hypothesis. Science, 1986, 234(4774): 364-368. DOI:10.1126/science.2876518 |
| [42] | Rechsteiner M, Rogers SW. PEST sequences and regulation by proteolysis. Trends in Biochemical Sciences, 1996, 21(7): 267-271. DOI:10.1016/S0968-0004(96)10031-1 |
| [43] | Mcpherson A, Cudney B. Optimization of crystallization conditions for biological macromolecules. Acta Crystallographica Section F:Structural Biology Communications, 2014, 70(Pt 11): 1445-1467. |
| [44] | Chayen NE, Saridakis E. Protein crystallization:from purified protein to diffraction-quality crystal. Nature Methods, 2008, 5(2): 147-153. DOI:10.1038/nmeth.f.203 |
| [45] | Bai XC, Fernandez IS, McMullan G, Scheres SHW. Ribosome structures to near-atomic resolution from thirty thousand cryo-EM particles. eLife, 2013, 2: e00461. DOI:10.7554/eLife.00461 |
| [46] | Milazzo AC, Cheng AC, Moeller A, Lyumkis D, Jacovetty E, Polukas J, Ellisman MH, Xuong NH, Carragher B, Potter CS. Initial evaluation of a direct detection device detector for single particle cryo-electron microscopy. Journal of Structural Biology, 2011, 176(3): 404-408. DOI:10.1016/j.jsb.2011.09.002 |
| [47] | Scheres SHW. A bayesian view on Cryo-EM structure determination. Journal of Molecular Biology, 2012, 415(2): 406-418. DOI:10.1016/j.jmb.2011.11.010 |
