刘开辉

 , 张波, 丁小维, 杨学英, 邓百万, 兰阿峰, 彭浩, 牛明月, 米洋
, 张波, 丁小维, 杨学英, 邓百万, 兰阿峰, 彭浩, 牛明月, 米洋 陕西理工大学生物科学与工程学院, 陕西 汉中 723001
收稿日期:2017-11-20;修回日期:2017-12-22;网络出版日期:2018-01-15
基金项目:国家自然科学基金(31100017);陕西省科技厅农业攻关项目(2018NY-156);陕西理工大学科研项目(SLGQD16-06,SLGQD16-07)
*通信作者:刘开辉, Tel/Fax:+86-916-2641661, E-mail:kaihhui168@hotmail.com
摘要:[目的]揭示陕北花马盐湖沉积物原核微生物群落组成,并分析其潜在的耐盐功能基因。[方法]构建盐湖沉积物宏基因组16S rRNA文库和fosmid文库,利用Illumina HiSeq高通量测序及生物信息技术分析细菌古菌群落组成和耐盐菌株(5-5)外源宏基因组的潜在耐盐基因。[结果]获得18978条有效的16S rRNA序列,共5221个OTUs,包括23个门,155个属,其中广古菌门(Euryarchaeota)和变形菌门(Proteobacteria)为优势菌门,盐杆状菌属(Halorhabdus)、盐红菌属(Halorubrum)及假单胞菌属(Pseudomonas)等16个属为优势属,以及嗜盐单胞菌属(Halomonas)、冷弯菌属(Psychroflexus)及不动细菌属(Acinetobacter)等139个属为非优势属。从4126个fosmid文库菌株中筛选出37株耐盐菌株,其中菌株5-5、2E4和2F4对不同浓度的NaCl、CuSO4、ZnSO4及CdSO4具有耐受性,从5-5的外源宏基因组序列中获得61个Unigene,其中12个Unigene的同源基因编码的蛋白质如无机焦磷酸酶、转座酶、亚碲酸钾抗性蛋白及钙调蛋白等广泛参与其他生物的耐盐逆境。[结论]盐湖沉积物中蕴藏着丰富多样的细菌古菌类群以及潜在耐盐功能基因资源。
关键词: 盐湖 宏基因组文库 原核微生物 耐盐基因 高通量测序
Microbial community and salt-tolerant genes of hypersaline sediment from Huama lake revealed by metagenomic sequencing
Kaihui Liu

 , Bo Zhang, Xiaowei Ding, Xueying Yang, Baiwan Deng, Afeng Lan, Hao Peng, Mingyue Niu, Yang Mi
, Bo Zhang, Xiaowei Ding, Xueying Yang, Baiwan Deng, Afeng Lan, Hao Peng, Mingyue Niu, Yang Mi School of Biological Science and Engineering, Shaanxi University of Technology, Hanzhong 723001, Shaanxi Province, China
Received 20 November 2017; Revised 22 December 2017; Published online 15 January 2018
*Corresponding author: Liu Kaihui, Tel/Fax:+86-916-2641661, E-mail:kaihhui168@hotmail.com
Supported by the National Natural Science Foundation of China (31100017), by the Program of Agricultural Scientific and Technological Innovation of Shaanxi Province (2018NY-156) and by the Shaanxi University of Technology Project (SLGQD16-06; SLGQD16-07)
Abstract: [Objective]The aim of this study was to investigate prokaryotic microbial community structures and salt-tolerant genes in hypersaline sediments from Huama salt lake, Northern Shaanxi, China.[Methods]We constructed 16S rRNA library and fosmid library using metagenome of the sediment, and detected prokaryotic microbial distribution and salt-tolerant genes by high-throughput pyrosequencing and bioinformatic analysis.[Results]In total 18978 high-quality reads for 16S rRNA were assigned to 23 phyla, 155 genera and 5221 OTUs. Euryarchaeota and Proteobacteria were the dominant phyla. Sixteen dominant genera such as Halorhabdus, Halorubrum and Pseudomonas and 139 infrequent ones like Halomonas, Psychroflexus and Acinetobacter were explored. A total of 37 salt-tolerant strains were detected from the metagenomic DNA fosmid library containing 4126 strains. The salt-tolerant strains like 5-5, 2E4 and 2F4 exhibited strong tolerance against stresses caused by NaCl, CuSO4, ZnSO4 and CdSO4. High-throughput pyrosequencing of the metagenomic fragment in the strain 5-5 explored 61 unigenes, 23 homogenous genes encoding salt-, or other stress-tolerant proteins in other biological cells, such as inorganic pyrophosphatase, transposase, tellurite resistance protein and calmodulin.[Conclusion]Hypersaline sediments in salt lakes harbor diverse bacterial and arhaeal community and rich salt-tolerant genes.
Keywords: salt lake metagenomic DNA library prokaryotes salt-tolerant genes high-throughput pyrosequencing
盐湖是含盐量较高的内陆咸化湖泊,通常指湖水含盐度ω(NaCl)e > 3.5%的湖泊,包括表面卤水干涸和由含盐沉积与晶间卤水组成的干盐湖[1]。盐湖作为典型的高渗透压环境,不适合大部分生命的生存,而耐/嗜盐极端微生物却在盐湖中广泛分布[2-3]。尽管盐湖中的古菌常以广古菌门(Euryarchaeota)为最优势菌门,细菌则以变形菌门(Proteobacteria)为最丰富类群[2, 4]。然而不同类型、不同盐度的盐湖原核微生物的群落组成不同[5-6]。由于可培养技术在盐湖微生物研究中有很大的局限性,因此利用免培养技术如宏基因组文库的构建及测序分析技术对认识盐湖的微生物群落组成及其耐盐基因资源具有重要作用。
目前发现,嗜盐微生物主要通过两种机制适应高盐环境[7]。一种是通过Na+/H+泵跨膜蛋白将胞内多余的Na+排到胞外,同时利用K+单向转移系统将胞外的K+泵入胞内维持渗透压平衡[8]。这种机制被认为是极端嗜盐古菌适应高盐逆境的一种重要方式,这类功能基因的表达显著受盐诱导[9]。另外,绝大多数耐(嗜)盐菌通过基因组编码的蛋白质在胞内合成耐渗透压化合物如甘油、谷氨酸及脯氨酸等有机物适应盐环境[10-11]。研究人员从一些耐(嗜)盐微生物中检测到不同的耐盐功能基因,这些基因的表达受盐逆境调控[12-14]。近年来,研究人员利用宏基因组技术成功从海洋沉积物及盐湖高盐沉淀样品筛选到一些新的耐盐基因[15-17]。这些工作证实盐湖环境中的极端微生物可能也是挖掘耐盐基因的重要资源库,但大量的耐盐基因仍处于“未知”的状态。
本研究构建陕北花马盐湖沉积物宏基因组16S rRNA文库和fosmid文库,并通过高通量测序技术分析盐湖沉积物原核微生物群落组成及物种多样性,并对其潜在耐盐基因进行分析,该研究为认识盐湖耐/嗜盐细菌适应高盐逆境的功能与机理机制提供依据。
1 材料和方法 1.1 样品采集 盐湖样品HM-10于2011年10月采集于陕北花马盐湖湖底表层高盐沉积物,并置放于50 mL无菌的离心管中。采集的样品迅速放入液氮中冷冻,用干冰盒带回实验室,放置于–80 ℃冰箱中保存,用于宏基因组的分析。
1.2 宏基因组DNA提取 利用Fast DNA? Spin Kit for Soil (MP Biomedicals)土壤DNA提取试剂盒,依说明书抽提并纯化盐湖沉积物(0.5 g样品)微生物基因组总DNA。3次重复抽提的样品DNA合并后并用1%的琼脂糖凝胶电泳检测所提取的基因组。
1.3 16S rRNA基因文库的构建 选用原核微生物16S rRNA基因的通用引物515F/806R扩增16S rRNA基因的V4区[18]。PCR反应条件:94 ℃ 5 min,94 ℃ 1 min,55 ℃ 45 s,72 ℃ 1 min,30个循环,72 ℃ 5 min。PCR反应体系(25 μL):2×PCR buffer 12.5 μL,dNTPs (10 mmol/L) 0.5 μL,基因组DNA 100 ng,515F/806R (50 μmol/L)各1 μL,Taq (5 U/μL) 0.5 μL及8.5 μL ddH2O。同一样品的PCR产物合并后经电泳检测,通过胶回收试剂盒进行纯化,构建16S rRNA基因文库。
1.4 Fosmid文库的构建 宏基因组DNA经1%的低熔点琼脂糖凝胶电泳检测,切取长度为20–40 kb的DNA片段。依照CopyControl? Fosmid Library Production Kit (Epicenter公司)的使用说明对DNA片段进行末端补平和片段的回收纯化,纯化的宏基因组DNA片段同pCC2FOS载体连接,用包装蛋白进行包装,并转导入Escherichia coli EPI300宿主细胞中,涂布于含12.5 mg/L氯霉素的LB平板上。用无菌牙签分别挑取平板上的单菌落至96孔板中,板孔中加入150 μL含12.5 mg/L氯霉素的LB液体培养基,用膜封口于37 ℃温箱中培养过夜备用。随机挑取10个菌株接种于含氯霉素的LB液体培养基中连续培养6 d,利用BAC/PAC大型质粒提取试剂盒(Omega)分别提取1 d和6 d培养物的质粒DNA,通过EcoR Ⅰ和Hind Ⅲ酶切(37 ℃酶切5 h)分析质粒的稳定性。
1.5 耐盐菌株的筛选 在96孔板的每孔中加入200 μL的LB液体培养基(含12.5 mg/L氯霉素和7.5% NaCl),分别转接10 μL活化的单菌株菌悬液,用膜封口于37 ℃温箱中培养48 h,利用酶标仪检测培养物的OD600光密度值,筛选耐盐菌株。取进一步活化的耐盐菌株2E4、2F4及5-5菌悬液(10 μL)分别接种于96孔板上,每孔含200 μL的LB液体培养基(含质粒拷贝诱导剂、12.5 mg/L氯霉素)。上述LB培养基中分别添加不同浓度梯度的NaCl (0–1.3 mol/L)、ZnSO4 (0–1.0 mmol/L)、CuSO4 (0–1.0 mmol/L)和CdSO4 (0–1.0 mmol/L),37 ℃培养48 h,检测培养物的光密度值OD600。对照为含有空载pCC2FOS质粒的E. coli EPI300-C菌株。
1.6 高通量测序及分析 利用Illumina HiSeq2500平台对16S rRNA基因组文库和耐盐菌株5-5的pCC2FOS质粒上插入的宏基因组片段进行高通量测序。在对16S rRNA基因序列分析时,先利用Mothur软件分别去除长度小于200 bp序列、平均质量分值低于25的序列、含有模糊碱基及化学嵌合体,去冗余并挑选unique tag序列[18]。利用分类器RDP classifier对有效序列进行物种注解,选择Confidence Threshold (bootstrapping)参数为0.5进行物种分类。根据97%的序列相似度对有效序列进行同源比对,并对操作分类单元(Operational Taxonomic Units,OTUs)进行群落分析(去除序列为单拷贝的OTU)。在对pCC2FOS质粒插入片段分析过程中,去除污染和低质量序列以及宿主基因组DNA序列,对有效序列进行拼接、组装获得Scaffold,利用Softberry FGENESB软件[19]分析DNA序列上潜在基因的阅读框,将所得基因(Unigene)序列同数据库(Nr,GO和COG)进行比对,并分析其潜在生化功能。
2 结果和分析 2.1 盐湖沉积物原核微生物群落分析 通过Illumina高通量测序从盐湖沉积物中共获得18978条有效的16S rRNA基因序列(共5221个OTUs),提交GenBank获得登录号SRS1404566。这些序列(共23个门)细菌和古菌,其丰度≥1%的优势类群包括广古菌门(Euryarchaeota)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)及放线菌门(Actinobacteria),以广古菌门为最优势菌群(约占OTUs总数的82%),变形菌门次之(占10%),其余优势门的丰度在1%–4%间(图 1-A)。
 |
| 图 1 花马盐湖沉积物原核微生物优势类群的门(A)和属(B)水平分析 Figure 1 Phylum (A)-, genus (B)-level distribution of dominant prokaryotes in hypersaline sediment from Huama salt lake. |
| 图选项 |
在该样品中检测到155个属,16个属为优势属(相对丰度≥1%),如盐杆状菌属(Halorhabdus)、盐红菌属(Halorubrum)、盐杆菌属(Halobacterium)、盐薄片菌属(Halolamina)、Halonotius、假单胞菌属(Pseudomonas)及盐碱单胞菌属(Natronomonas)等。以盐杆状菌为最优势类群(约占OTUs总数的26%),盐红菌属(占13%),盐杆菌属(11%),盐薄片菌属(9%),Halonotius、假单胞菌属(Pseudomonas)及盐碱单胞菌属的丰度在5%–7%之间(图 1-B)。其余139个属为非优势类群如盐单胞菌属(Halomonas)、冷弯菌属(Psychroflexus)及不动细菌属(Acinetobacter)等。
2.2 Fosmid文库的构建及耐盐菌株的筛选 通过构建fosmid文库,共获得4126菌株。从这些菌株中共筛选出37株能在含7.5% NaCl的培养基中生长的耐盐菌株,部分菌株耐受7.5% NaCl的生长状况如图 2所示。培养48 h后,37个耐盐菌株的OD600光密度值为0.21–0.68,表明这些菌株具有耐盐性。其中菌株2E4耐受7.5% NaCl逆境能力较强,2F4则在含盐培养基中生长最缓慢。对照E. coli EPI300-C菌株在含7.5% NaCl培养基中培养,其OD600值无明显变化。上述结果表明,耐盐菌株的pCC2FOS载体上已插入盐湖耐(嗜)盐微生物的耐盐基因片段。
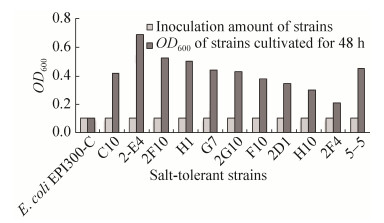 |
| 图 2 Fosmid文库的耐盐菌株筛选 Figure 2 Screenig of salt-tolerant strains of fosmid library. |
| 图选项 |
2.3 耐盐菌株对不同盐逆境的耐受性分析 不同盐胁迫对耐盐菌株生长的影响如图 3所示,随着NaCl浓度增加(0–7.5%),耐盐菌株2E4和2F4的光密度值OD600一直呈现下降趋势,而菌株5-5在培养基盐浓度为1.5 mol/L时,其OD600值达最大(OD600=1.75),之后该值逐步减小(图 3-A)。如图 3-B所示,尽管菌株2F4和5-5对CuSO4有明显的耐受性,但随培养基中CuSO4浓度的增加,它们光密度值逐步减小,然而菌株2E4的光密度值在CuSO4浓度增加到0.2 mmol/L时达到最大值(OD600=1.95),之后随该盐浓度增加而迅速减小。在图 3-C、3-D中,随培养基中ZnSO4和CdSO4浓度增加,菌株2E4、2F4和5-5的光密度值均减小。培养基中CdSO4浓度从0增加到0.2 mmol/L时,2E4和2F4的光密度值锐减超过1倍,而菌株5-5在试验CdSO4浓度下呈现较强的耐受性。尽管菌株2E4,2F4和5-5的光密度值终将随培养基盐离子浓度增加而下降,但同对照比呈现出明显的耐盐性。
 |
| 图 3 部分耐盐菌株对不同盐逆境的耐受分析 Figure 3 Salt tolerance of some salt-tolerant strains against different salt stress. (A), (B), (C) and (D) respectively represents the strains tolerated salt stress caused by addition of different content of NaCl, CuSO4, ZnSO4 and CdSO4. Standard deviation indicated the range of biomass of the strains grown in media supplemented with different concentration of salts. All experiments were performed in triplicate. |
| 图选项 |
2.4 宏基因组测序及Unigene的功能分类 对最耐盐菌株5-5的外源宏基因组片段进行高通量测序,去除无效数据后获得3641189 bp长度的序列,过滤宿主基因序列得到65971 bp的DNA序列,组装获得39个Scaffold,N50为2727 bp和GC含量为44.95%,该宏基因序列已提交GenBank并获得登录号(SRS1411976)。Unigene的GO分类表明(图 4),这些序列属于生物过程(Biological process)、细胞组成(Cellular component)和分子功能(Molecular function),以参与代谢过程(metabolic process)、细胞过程(cellular process)、细胞(cell)、细胞组分(cell part)及结合(binding)的基因较丰富。
 |
| 图 4 基因GO数据库的功能分类 Figure 4 Functional classification of annotated transcripts by GO. |
| 图选项 |
COG功能分析将Unigene分为12类(图 5),其中参与翻译、核糖体结构蛋白基因最丰富(translation, ribosomal structure and biogenesis),其次为复制、重组和修复(replication,recombination and repair)相关基因,细胞运动性(cell motility),胞内分泌和膜泡运输(intracellular trafficking,secretion and vesicular transport)及次生代谢产物的合成、转运和代谢(secondary metabolites synthesis, transform and catabolism)等功能基因。
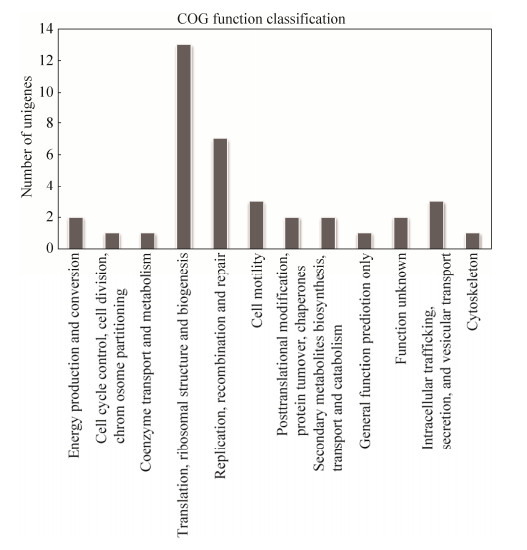 |
| 图 5 基因COG数据库的功能分类 Figure 5 Functional classification of annotated transcripts by COG. |
| 图选项 |
2.5 潜在耐盐基因的筛选及分析 在Nr数据库中对Unigenes进行比对分析,共获得61个功能基因,其中12个基因编码核糖体蛋白(ribosomal protein),5个基因编码转座酶(transposase),4个基因参与DNA复制(DNA replication protein),2个基因属于无机焦磷酸酶(inorganic pyrophosphatase),其他基因编码蛋白如亚碲酸钾抗性蛋白(tellurite resistance protein)、钙调蛋白(calmodulin)、泛素连接酶(ubiquitin-conjugating enzyme)以及脂质转移蛋白(lipid transfer protein)等(表 1)。14个Unigene在数据库中未发现其同源基因,这些可能是未知功能的潜在新基因。23个Unigene同不动细菌属多个物种的功能基因具有高度同源性,暗示该菌株的宏基因组DNA可能来自于不动细菌属细菌,而且该属是该盐湖沉积物的一个非优势微生物类群。经比较分析发现,上述已知功能基因的同源基因编码蛋白质如无机焦磷酸酶、转座酶、亚碲酸钾抗性蛋白、钙调蛋白、参与染色体分离的ATP酶(ATPases involved in chromosome partitioning)、脂质转移蛋白、泛素连接酶E2及植物衰老相关蛋白质(plant senescence-associated protein)等广泛参与其他生物细胞的耐盐逆境反应(表 1)。此外,本研究还发现多个同耐热、耐冷及耐其他逆境相关的功能基因。
表 1. 宏基因组序列的潜在耐盐基因分析 Table 1. Detection of putative salt-tolerant genes of the metagenomic sequence
| Query_ID | Amino acid length | Identity | Function annotation in Nr | Stress tolerance of homologous genes | References |
| PROKKA_00095 | 216 | 0.63 | 60S large subunit ribosomal protein L10 (Piriformospora indica DSM 11827) | + | [20] |
| PROKKA_00104 | 213 | 0.80 | 40S ribosomal protein S5 (Neurospora crassa OR74A) | + | [21] |
| PROKKA_00082 | 122 | 0.77 | 40S ribosomal protein S20 (Mucor circinelloides f. circinelloides 1006PhL) | + | [22] |
| PROKKA_00070 | 191 | 0.67 | Ribosomal protein L6 (Coniophora puteana RWD-64-598 SS2) | + | [23] |
| PROKKA_00054 | 156 | 0.79 | Ribosomal protein S13 (Auricularia delicata TFB-10046 SS5) | + | [24] |
| PROKKA_00006 | 174 | 0.95 | Inorganic pyrophosphatase (Acinetobacter gerneri) | ++ | [25] |
| PROKKA_00007 | 174 | 0.87 | Inorganic pyrophosphatase (Acinetobacter gerneri) | ++ | [25] |
| PROKKA_00024 | 942 | 0.99 | Transposase (Salmonella enterica subsp. enterica serovar Kentucky str. CVM29188) | ++ | [26] |
| PROKKA_00027 | 189 | 0.99 | Transposase (Acinetobacter baumannii) | ++ | [26] |
| PROKKA_00029 | 151 | 0.95 | Transposase (Acinetobacter sp. NIPH 899) | ++ | [26] |
| PROKKA_00123 | 230 | 1.00 | Transposase (Acinetobacter baumannii) | ++ | [26] |
| PROKKA_00038 | 194 | 0.67 | Tellurite resistance protein (Acinetobacter radioresistens) | + | [27] |
| PROKKA_00067 | 196 | 0.94 | Actin-3 (Pyrenophora tritici-repentis Pt-1C-BFP) | ++ | [28] |
| PROKKA_00068 | 207 | 0.54 | Calmodulin (Meyerozyma guilliermondii ATCC 6260) | ++ | [29] |
| PROKKA_00073 | 205 | 0.99 | ATPases involved in chromosome partitioning (Acinetobacter baumannii BJAB0715) | ++ | [30] |
| PROKKA_00094 | 229 | 0.59 | Elongation factor 1-beta (Marssonina brunnea f. sp. ‘multigermtubi’ MB_m1) | + | [31] |
| PROKKA_00083 | 150 | 0.74 | Translation elongation factor EF-1 alpha subunit (Rhodosporidium toruloides NP11) | + | [31] |
| PROKKA_00084 | 702 | 0.58 | Heat shock protein 80 (Piriformospora indica DSM 11827) | + | [32] |
| PROKKA_00093 | 137 | 0.73 | Lipid transfer protein (Uromyces hobsonii) | ++ | [33] |
| PROKKA_00105 | 147 | 0.81 | Ubiquitin-conjugating enzyme E2 (Botryotinia fuckeliana B05.10) | ++ | [34] |
| PROKKA_00111 | 135 | 0.77 | Plant senescence-associated protein (Dacryopinax sp. DJM-731 SS1) | ++ | [35] |
| PROKKA_00125 | 85 | 0.87 | Antitoxin YefM (Wohlfahrtiimonas chitiniclastica) | + | [36] |
| “++” means that homologous genes of unigene exhibited tolerance against salt stress;“+” represents that homologous genes of unigene tolerated other stress. | |||||
表选项
3 讨论 本研究发现,在陕北花马盐湖沉积物中以广古菌门和变形菌门为最优势类群。最近的研究发现,在陕北花马盐湖、苟池盐湖卤水中以及在青海察尔汗盐湖表面沉积物中均以广古菌门和变形菌门菌门为优势菌群[3-4],表明在门分类水平上盐湖水体和表层沉积物优势类群具有相似性,然而在青藏高原茶卡盐湖湖水及表层沉积物中以广古菌门和后壁菌门为优势门[5],以及在青海柴达木盆地达布逊盐湖中以广古菌门和放线菌门最丰富[37]。这可能是由于不同盐湖的地化异质性以及采样的时空差异性导致其微生物群落在不同分类水平上呈现差异的群落格局。
在属水平上,检测到16个优势属和139个非优势属。以盐杆状菌属最丰富(占26%),盐红菌属、盐杆菌属及盐薄片菌属次之。曹露芬等[3]发现,该盐湖卤水中古菌以Halonotius最丰富(41.93%),Halonotius、盐红菌属及盐薄片菌属次之;细菌则以乳球菌属(Lactococcus)、假单胞菌属和Spiribacter为丰富群体。这种优势类群的差异可能由于该盐湖沉淀及其卤水的理化性质不同所致。同一盐湖区的不同盐池中优势微生物丰度有所不同,不同区域盐湖的优势微生物差异更明显[3, 5]。此外,本研究获得多样的非优势属如盐单胞菌属、冷弯菌属及不动细菌属等,这些低丰度微生物的分布具有盐湖的特异性。
从构建的盐湖宏基因组文库中筛选到37个耐盐菌株,对耐盐菌株5-5进行高通量测序及组装。COG功能分析显示,在12个分类中,Unigene被富集到翻译、核糖体结构蛋白、复制、重组和修复以及胞内分泌和膜泡运输的功能类群最多。而Harding等[38]通过对嗜盐菌Halocafeteria seosinensis和非嗜盐菌(Thalassiosira pseudonana和Salpingoeca rosetta)的比较转录组研究发现,在高盐诱导下嗜盐菌H. seosinensis差异表达的基因显著相关于翻译、核糖体结构蛋白基因及胞内分泌和膜泡运输功能基因类群。这说明具有该功能的基因参与生物耐盐。
在5-5菌株宏基因组序列中检测到61个Unigene,12个潜在基因编码的蛋白如焦磷酸酶、转座酶、钙调蛋白、泛素连接酶以及脂质转移蛋白等(表 1)。据文献报道,这些Unigene的同源基因与其他生物耐盐胁迫密切相关,如无机焦磷酸酶参与水稻的耐盐逆境[25]、某些转座酶的表达受盐胁迫或氮素缺乏调控[26]、actin-3蛋白调控拟南芥的钙信号通路响应盐逆境[28]、钙调蛋白参与马铃薯细胞对盐逆境应答[29, 35]、盐湖盐芥(Thellungiella salsuginea)的脂转运蛋白TsnsLTP4的表达受盐胁迫、细胞脱落酸及高低温的显著诱导[33]。最近研究发现,过量表达泛素连接酶UBC1显著增加烟草对隔的抗性,并显著降低该重金属在细胞内的积累[34]。由此可见,这些Unigene参与的途径在增强微生物的耐盐中可能发挥着重要作用。下一步研究将通过转座突变及转录组测序技术分析这些潜在的耐盐基因调控宿主耐盐逆境的功能与机理。
综上,本研究利用Illumina Hiseq 2500高通量测序技术探究了陕北花马盐湖沉积物中原核微生物的群落结构。构建盐湖沉积物宏基因组的fosmid文库,筛选出37株耐盐菌株,并从耐盐菌株5-5的外源宏基因组序列中获得多个潜在耐盐或耐其他逆境的功能基因,该研究将为深入分析外源基因调控宿主耐盐逆境的功能与机理奠定基础。
References
| [1] | Zheng MP. Salt lake resources and eco-environment in China. Acta Geologica Sinica, 2010, 84(11): 1613-1622. (in Chinese) 郑绵平. 中国盐湖资源与生态环境. 地质学报, 2010, 84(11): 1613-1622. |
| [2] | Cao LF, Yin TT, Yuan ZY, Yan YW, Xie FF, Xu XY, Xu FM, Wen HY. Analysis of the microbial community composition of Huama Lake and Gou Lake in Shaanxi Province based on high-throughput sequencing. Letters in Biotechnology, 2016, 27(3): 374-380. (in Chinese) 曹露芬, 殷婷婷, 袁振亚, 严雨薇, 谢凡凡, 许心怡, 徐方敏, 温洪宇. 基于高通量测序的陕西花马池和苟池微生物群落多样性分析. 生物技术通讯, 2016, 27(3): 374-380. DOI:10.3969/j.issn.1009-0002.2016.03.017 |
| [3] | Xie KH, Deng Y, Zhang SC, Zhang WH, Liu JR, Xie YL, Zhang XZ, Huang H. Prokaryotic community distribution along an ecological gradient of salinity in surface and subsurface saline soils. Scientific Reports, 2017, 7(1): 13332. DOI:10.1038/s41598-017-13608-5 |
| [4] | Zhang X, Liu J, Shen GP, Long QF, Han R, Zhu DR. Illumina-based sequencing analysis of microbial community composition in Chaka Salt Lake in Qinghai-Tibet Plateau. Microbiology China, 2017, 44(8): 1834-1846. (in Chinese) 张欣, 刘静, 沈国平, 龙启福, 韩睿, 朱德锐. 基于高通量测序研究青藏高原茶卡盐湖微生物多样性. 微生物学通报, 2017, 44(8): 1834-1846. |
| [5] | Zhong ZP, Liu Y, Miao LL, Wang F, Chu LM, Wang JL, Liu ZP. Prokaryotic community structure driven by salinity and ionic concentrations in plateau lakes of the Tibetan Plateau. Applied and Environmental Microbiology, 2016, 82(6): 1846-1858. DOI:10.1128/AEM.03332-15 |
| [6] | Han R, Zhang X, Liu J, Long QF, Chen LS, Liu DL, Zhu DR. Microbial community structure and diversity within hypersaline Keke Salt Lake environments. Canadian Journal of Microbiology, 2017, 63(11): 895-908. DOI:10.1139/cjm-2016-0773 |
| [7] | Kindzierski V, Raschke S, Knabe N, Siedler F, Scheffer B, Pflüger-Grau K, Pfeiffer F, Oesterhelt D, Marin-Sanguino A, Kunte HJ. Osmoregulation in the halophilic bacterium Halomonas elongata:A case study for integrative systems biology. PLoS One, 2017, 12(1): e0168818. DOI:10.1371/journal.pone.0168818 |
| [8] | Ma YH, Galinski EA, Grant WD, Oren A, Ventosa A. Halophiles 2010:Life in saline environments. Applied and Environmental Microbiology, 2010, 76(21): 6971-6981. DOI:10.1128/AEM.01868-10 |
| [9] | Lin JT, Liang HB, Yan JW, Luo LX. The molecular mechanism and post-transcriptional regulation characteristic of Tetragenococcus halophilus acclimation to osmotic stress revealed by quantitative proteomics. Journal of Proteomics, 2017, 168: 1-14. DOI:10.1016/j.jprot.2017.08.014 |
| [10] | Pocard JA, Smith LT, Smith GM, Le Rudulier D. A prominent role for glucosylglycerol in the adaptation of Pseudomonas mendocina SKB70 to osmotic stress. Journal of Bacteriology, 1994, 176(22): 6877-6884. DOI:10.1128/jb.176.22.6877-6884.1994 |
| [11] | Pollastri S, Savvides A, Pesando M, Lumini E, Volpe MG, Ozudogru EA, Faccio A, de Cunzo F, Michelozzi M, Lambardi M, Fotopoulos V, Loreto F, Centritto M, Balestrini R. Impact of two arbuscular mycorrhizal fungi on Arundo donax L. response to salt stress. Planta, 2017. DOI:10.1007/s00425-017-2808-3 |
| [12] | Yaakop AS, Chan KG, Ee R, Lim YL, Lee SK, Manan FA, Goh KM. Characterization of the mechanism of prolonged adaptation to osmotic stress of Jeotgalibacillus malaysiensis via genome and transcriptome sequencing analyses. Scientific Reports, 2016, 6: 33660. DOI:10.1038/srep33660 |
| [13] | Bidle KA. Differential expression of genes influenced by changing salinity using RNA arbitrarily primed PCR in the archaeal halophile Haloferax volcanii. Extremophiles, 2003, 7(1): 1-7. DOI:10.1007/s00792-002-0289-0 |
| [14] | Liu KH, Ding XW, Narsing Rao MP, Zhang B, Zhang YG, Liu FH, Liu BB, Xiao M, Li WJ. Morphological and transcriptomic analysis reveals the osmoadaptive response of endophytic fungus Aspergillus montevidensis ZYD4 to high salt stress. Frontiers in Microbiology, 2017, 8: 1789. DOI:10.3389/fmicb.2017.01789 |
| [15] | Jeon JH, Lee HS, Kim JT, Kim SJ, Choi SH, Kang SG, Lee JH. Identification of a new subfamily of salt-tolerant esterases from a metagenomic library of tidal flat sediment. Applied Microbiology and Biotechnology, 2012, 93(2): 623-631. DOI:10.1007/s00253-011-3433-x |
| [16] | Sorokin DY, Toshchakov SV, Kolganova TV, Kublanov IV. Halo(natrono)archaea isolated from hypersaline lakes utilize cellulose and chitin as growth substrates. Frontiers in Microbiology, 2015, 6: 942. |
| [17] | Zhang Y, Hao J, Zhang YQ, Chen XL, Xie BB, Shi M, Zhou BC, Zhang YZ, Li PY. Identification and characterization of a novel salt-tolerant esterase from the deep-sea sediment of the South China Sea. Frontiers in Microbiology, 2017, 8: 441. |
| [18] | Caporaso JG, Lauber CL, Walters WA, Berg-Lyons D, Lozupone CA, Turnbaugh PJ, Fierer N, Knight R. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(S1): 4516-4522. |
| [19] | Schloss PD, Westcott SL, Ryabin T, Hall JR, Hartmann M, Hollister EB, Lesniewski RA, Oakley BB, Parks DH, Robinson CJ, Sahl JW, Stres B, Thallinger GG, van Horn DJ, Weber CF. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [20] | Ferreyra MLF, Pezza A, Biarc J, Burlingame AL, Casati P. lant L10 ribosomal proteins have different roles during development and translation under ultraviolet-B stress. Plant Physiology, 2010, 153(4): 1878-1894. DOI:10.1104/pp.110.157057 |
| [21] | Zhang JX, Yuan H, Yang Y, Fish T, Lyi SM, Thannhause TW, Zhang LG, Li L. Plastid ribosomal protein S5 is involved in photosynthesis, plant development, and cold stress tolerance in Arabidopsis. Journal of Experimental Botany, 2016, 67(9): 2731-2744. DOI:10.1093/jxb/erw106 |
| [22] | Pariasca-Tanaka J, Satoh K, Rose T, Mauleon R, Wissuwa M. Stress response versus stress tolerance:A transcriptome analysis of two rice lines contrasting in tolerance to phosphorus deficiency. Rice, 2009, 2(4): 167-185. DOI:10.1007/s12284-009-9032-0 |
| [23] | Bai DM, Zhang JF, Xiao WC, Zheng XF. Regulation of the HDM2-p53 pathway by ribosomal protein L6 in response to ribosomal stress. Nucleic Acids Research, 2014, 42(3): 1799-1811. DOI:10.1093/nar/gkt971 |
| [24] | Liu SL, Adams K. Molecular adaptation and expression evolution following duplication of genes for organellar ribosomal protein S13 in rosids. BMC Evolutionary Biology, 2008, 8: 25. DOI:10.1186/1471-2148-8-25 |
| [25] | Hosseini SA, Gharechahi J, Heidari M, Koobaz P, Abdollahi S, Mirzaei M, Nakhoda B, Salekdeh GH. Comparative proteomic and physiological characterisation of two closely related rice genotypes with contrasting responses to salt stress. Functional Plant Biology, 2015, 42(6): 527-542. DOI:10.1071/FP14274 |
| [26] | Zhao H, Jia FQ, Zhang FC, Wang Y. The transcriptome information analysis of differentially expressed genes of Halostachys caspica under salt stress. Chinese Journal of Bioinformatics, 2014, 12(2): 91-98. (in Chinese) 赵航, 贾富强, 张富春, 王艳. 盐胁迫下盐穗木差异表达基因的转录组信息分析. 生物信息学, 2014, 12(2): 91-98. |
| [27] | Ponnusamy D, Hartson SD, Clinkenbeard KD. Intracellular Yersinia pestis expresses general stress response and tellurite resistance proteins in mouse macrophages. Veterinary Microbiology, 2011, 150(1/2): 146-151. |
| [28] | Zhao Y, Pan Z, Zhang Y, Qu XL, Zhang YG, Yang YQ, Jiang XN, Huang SJ, Yuan M, Schumaker KS, Guo Y. The Actin-related protein2/3 complex regulates mitochondrial-associated calcium signaling during salt stress in Arabidopsis. The Plant Cell, 2013, 25(11): 4544-4559. DOI:10.1105/tpc.113.117887 |
| [29] | Magnan F, Ranty B, Charpenteau M, Sotta B, Galaud JP, Aldon D. Mutations in AtCML9, a calmodulin-like protein from Arabidopsis thaliana, alter plant responses to abiotic stress and abscisic acid. The Plant Journal, 2008, 56(4): 575-589. DOI:10.1111/tpj.2008.56.issue-4 |
| [30] | Guo MX, Wang RC, Wang J, Hua K, Wang YM, Liu XQ, Yao SG. ALT1, a Snf2 family chromatin remodeling ATPase, negatively regulates alkaline tolerance through enhanced defense against oxidative stress in rice. PLoS One, 2014, 9(12): e112515. DOI:10.1371/journal.pone.0112515 |
| [31] | Fu JM, Mom?ilovi? I, Vara Prasad PV. Roles of protein synthesis elongation factor EF-Tu in heat tolerance in plants. Journal of Botany, 2012, 2012: Article ID 835836. |
| [32] | Roychowdhury HS, Wong D, Kapoor M. hsp80 of Neurospora crassa:cDNA cloning, gene mapping, and studies of mRNA accumulation under stress. Biochemistry and Cell Biology, 1992, 70(12): 1356-1367. DOI:10.1139/o92-184 |
| [33] | Sun W, Li Y, Zhao YX, Zhang H. The TsnsLTP4, a nonspecific lipid transfer protein involved in wax deposition and stress tolerance. Plant Molecular Biology Reporter, 2015, 33(4): 962-974. DOI:10.1007/s11105-014-0798-x |
| [34] | Bahmani R, Kim D, Lee BD, Hwang S. Over-expression of tobacco UBC1 encoding a ubiquitin-conjugating enzyme increases cadmium tolerance by activating the 20S/26S proteasome and by decreasing Cd accumulation and oxidative stress in tobacco (Nicotiana tabacum). Plant Molecular Biology, 2017, 94(4/5): 433-451. |
| [35] | Chen HJ, Lin ZW, Huang GJ, Lin YH. Sweet potato calmodulin SPCAM is involved in salt stress-mediated leaf senescence, H2O2 elevation and senescence-associated gene expression. Journal of Plant Physiology, 2012, 169(18): 1892-1902. DOI:10.1016/j.jplph.2012.08.004 |
| [36] | Cherny I, Rockah L, Gazit E. The YoeB toxin is a folded protein that forms a physical complex with the unfolded YefM antitoxin:Implications for a structural-based differential stability of toxin-antitoxin systems. The Journal of Biological Chemistry, 2005, 280(34): 30063-30072. DOI:10.1074/jbc.M506220200 |
| [37] | Zhao WY, Yang J, Dong HL, Wu G, Wang S, Sun YJ, Lai ZP, Jiang HC. Microbial diversity in the hypersaline Dabuxun Lake in Qaidam Basin, China. Earth and Environment, 2013, 41(4): 398-405. (in Chinese) 赵婉雨, 杨渐, 董海良, 吴耿, 王尚, 孙永娟, 赖忠平, 蒋宏忱. 柴达木盆地达布逊盐湖微生物多样性研究. 地球与环境, 2013, 41(4): 398-405. |
| [38] | Harding T, Roger AJ, Simpson AGB. Adaptations to high salt in a halophilic protist:differential expression and gene acquisitions through duplications and gene transfers. Frontiers in Microbiology, 2017, 8: 944. DOI:10.3389/fmicb.2017.00944 |
