何蕴喆1,2, 黄舟1,2, 姜成英1,2,3, 刘双江1,2,3


1.中国科学院微生物研究所, 北京 100101;
2.中国科学院大学, 北京 100049;
3.IMCAS-RCEECAS 环境微生物技术联合实验室, 北京 100101
收稿日期:2017-02-07;修回日期:2017-04-04;网络出版日期:2017-05-16
基金项目:国家自然科学基金(31230003)
作者简介:刘双江,博士,现任中国科学院微生物研究所所长、研究员。现任亚洲研究资源中心网络主席。2000年入选中国科学院“****”。2007年国家****科学基金获得者,目前担任Applied and Environmental Microbiology、Archives of Microbiology编辑,《微生物学报》、《环境科学学报》、《生物工程学报》等杂志编委。在芳烃类化合物的微生物代谢与趋化、微生物分离培养技术及微生物资源收集、极端环境微生物应用等方面,取得了突出成绩,包括:(1) 发现了多个芳烃类化合物转运蛋白和多条代谢途径;(2) 鉴定了催化苯环裂解的2-氨基酚-1, 6-双加氧酶基因,发现了两类新型脱氨酶;(3) 通过改进培养方法和技术,从各种环境中分离获得各种生理类群的微生物菌种,已经鉴定并命名了多个微生物新物种,并建立了2个新属-杨氏菌属(Yangia)和周氏菌属(Zhouia);(4) 揭示了丛毛单胞菌CNB-1对多种芳烃化合物的趋化的新机制
*通信作者:刘双江, Tel:+86-10-64807423, Fax:+86-10-64807421, E-mail:liusj@im.ac.cn
摘要:[目的]MCP2923是睾丸酮丛毛单胞菌(Comamonas testosteroni)CNB-1的一种甲基化趋化受体蛋白,本研究旨在阐明其在CNB-1菌株趋化过程中的作用。[方法]利用游动平板法(swimming plate)检测了CNB-1及其MCP突变菌株CNB-1Δ20、CNB-1ΔMCP2923、CNB-1Δ20/pDSK519-MCP2923、CNB-1Δ20/pBBR1MCS-2-MCP2923和CNB-1Δ20/pBBR1MCS-2-MCP2923ΔLBD对35种芳香族化合物以及9种小分子有机酸的趋化性;进一步利用Agarose-in-plug法表征了MCP2923介导的对芳香族化合物的趋化表型;结合生物信息学分析,对MCP2923配体结合结构域(MCP2923LBD)进行了克隆、表达和纯化,利用等温滴定量热法(ITC)检测了MCP2923LBD与原儿茶酸等11种化合物的相互作用。[结果]CNB-1对原儿茶酸、4-羟基苯甲酸等12种芳香族化合物以及顺乌头酸等9种TCA循环中间代谢产物具有强、中强和弱3个层次的趋化表型;敲除MCP2923基因减弱了菌株对上述趋化诱导物的表型;将MCP2923基因回补到CNB-1Δ20菌株中可回补菌株对15种趋化效应物的表型,敲除MCP2923基因的配体结合区丧失了对15种趋化物的表型回补能力。虽然Agarose-in-plug法检测到了菌株对原儿茶酸和4-羟基苯甲酸的趋化表型,但ITC未能检测到原儿茶酸和4-羟基苯甲酸等11种化合物与MCP2923LBD直接的相互作用。[结论]MCP2923可引发CNB-1菌株对多种芳香族化合物以及小分子有机酸的趋化表型,且MCP2923LBD在这个过程中起关键作用;由于ITC的结果不能证明MCP2923LBD与芳香化合物和小分子有机酸等效应物的直接结合,推测MCP2923引发CNB-1趋化作用的机制不同于已报道的MCP2201和MCP2901的作用方式,其确切机制还有待于进一步深入研究。
关键词: 睾丸酮丛毛单胞菌CNB-1 甲基化受体蛋白(MCP) 细菌趋化性 芳香化合物 三羧酸循环(TCA)
Chemotactic responses towards various organic compounds mediated by MCP2923 in Comamonas testosteroni CNB-1
Yunzhe He1,2, Zhou Huang1,2, Chengying Jiang1,2,3, Shuangjiang Liu1,2,3


1.Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
2.University of Chinese Academy of Sciences, Beijing 100049, China;
3.IMCAS-RCEECAS Joint Laboratory for Environmental Microbial Technology, Beijing 100101, China
Received 7 February 2017; Revised 4 April 2017; Published online 16 May 2017
*Corresponding author: Shuangjiang Liu, Tel:+86-10-64807423, Fax:+86-10-64807421, E-mail:liusj@im.ac.cn
Supported by the National Natural Science Foundation of China (31230003)
Abstract: [Objective]MCP2923 is a putative of Comamonas testosteroni CNB-1. The objective of this study was to experimentally characterize MCP2923 for chemotactic response.[Methods]Using swimming plate assay, we determined chemotaxis towards 35 aromatic compounds and 9 Tricarboxylic Acid Cycle intermediates. Agrose-in-plug was used to screen aromatic chemoattractants that might bind to MCP2923 directly. To study the ligand of MCP2923, Isothermal Titration Calorimetry experiment was done to 11 chemoattractants.[Results]Swimming plate assay showed that CNB-1 responded to 12 aromatic compounds and 9 Tricarboxylic Acid Cycle intermediates that were defined as strong, medium and weak chemoattractants. Knockout MCP2923 gene reduced chemotactic responses to these chemoattractants. Complemented with MCP2923 gene to CNB-1Δ20, chemotaxis toward 15 chemoattractants was restored. Deletion of the ligand binding domain of MCP2923, chemotaxis failed to complement. Isothermal Titration Calorimetry experiment showed no response to the tested 11 compounds, including protocatechuic acid and 4-hydroxybenzoic acid.[Conclusion]MCP2923 mediates chemotaxis towards both aromatic compounds and Tricarboxylic Acid Cycle cycle intermediates by CNB-1. The ligand binding domain of MCP2923 is necessary for triggering chemotaxis toward these chemoattractants.
Key words: Comamonas testosteroni CNB-1 methyl-accepting chemotaxic protein (MCP) bacterial chemotaxis aromatic compounds tricarboxylic acid cycle
细菌的趋化性是细菌趋向对自身有利或者躲避对自身有害的环境因子或者化学物质,从而使细菌处于最适生长环境中的一种行为。因为具有最简单的趋化信号通路,E. coli作为细菌趋化性研究的模式生物[1],其包括5个甲基化受体趋化蛋白(MCP),Tar、Tsr、Tap、Trg和Aer,1个组氨酸激酶CheA,1个响应调控蛋白CheY和1个磷酸脂酶CheZ。细菌的趋化过程可以分为3个部分:(1) 位于细胞膜上的MCP感应趋化效应物,通过自身的构象变化产生信号;(2) 信号通过MCP传递,影响CheA的自磷酸化活性,并进一步影响CheY的磷酸化水平;(3) 磷酸化的CheY与鞭毛蛋白FliM结合,进而影响细菌鞭毛的旋转方向,最终决定了细菌的运动方向。
趋化作用起始于MCP对趋化效应物的感应,MCP可以感应的趋化效应物的种类很多,包括糖类、氨基酸和芳香化合物等营养物质。芳香化合物是自然界中仅次于碳水化合物的第二大碳库[2],可以作为很多微生物生长的碳源和能源,支持细胞生长。目前已经分离鉴定了很多具有芳香化合物降解能力的菌株,例如Pseudomonas putida KT2440[3]、Comamonas testosteroni CNB-1[4]、Sphingobium sp. SYK-6[5]、Cupriavidus necator LMP134等。在许多菌株中,芳香化合物的降解与细菌的趋化性之间存在联系,如Sanmata等人的研究表明Burkholderia sp. SJ98菌株只能对可降解的硝基芳香化合物产生趋化表型[6];另有****证明,芳香化合物本身亦可以激活趋化通路,如Parales等研究表明Pseudomonas putida F1菌株对甲苯等芳香化合物具有明显的趋化表型,当todX等芳香化合物降解基因被敲除后,菌株依然可以对甲苯表现出趋化性[7]。目前对于细菌感应芳香化合物的机制分为代谢依赖型和非代谢依赖型两种,代谢依赖型的趋化包括:(1) 通过Aer类受体感应FAD的氧化还原状态来介导微生物对芳香化合物的趋化性;(2) 通过感应代谢中间产物来介导微生物的趋化性。代谢依赖型趋化过程中MCP不直接与效应物结合,而是与微生物代谢的中间产物相互作用。而非代谢依赖的趋化是指MCP可以直接感应趋化效应物本身,例如,存在于E. coli细胞中的Tar、Tsr、Tap和Trg等MCP蛋白。许多MCP蛋白分子,如McpT[8]、NahY[9]、NbaY和CtpL[10]等分别介导甲苯、萘、2-硝基苯甲酸和对氯苯胺的趋化,但并没有报道与之直接结合的配体,其引发趋化的确切机制还不清楚。
Comamonas testosteroni CNB-1中存在19个MCP,Ni等人构建了敲除全部MCP编码基因的突变菌株CNB-1Δ20、所有MCP的单敲除突变菌株(如:CNB-1ΔMCP2923)和所有MCP的单回补突变菌株(如:CNB-1Δ20/pDSK519-MCP2923),并进一步证明其中的MCP2201和MCP2983可以感应芳香化合物代谢的中间产物从而实现对芳香化合物的趋化[11-12]。在最近的研究中,Huang等人对CNB-1中的MCP2901进行了研究,证明MCP2901可以直接与2, 6-二羟基苯甲酸、2-羟基苯甲酸和柠檬酸结合,这是首次报道MCP与芳香族化合物存在直接相互作用[13]。
本研究对CNB-1菌株中MCP2923所介导的趋化性进行研究。利用游动平板法,首先检测了CNB-1菌株对44种化合物的趋化表型。结果表明,CNB-1菌株对原儿茶酸、4-羟基苯甲酸等12种芳香化合物以及顺乌头酸等9种TCA循环中间代谢产物具有趋化性。对CNB-1ΔMCP2923菌株趋化表型的测定表明,MCP2923基因的缺失使得CNB-1ΔMCP2923的趋化表型相对于原始菌株略有减弱。而进一步将MCP2923基因回补至CNB-1Δ20,结果表明MCP2923基因可以部分回补CNB-1Δ20对效应物的趋化表型。MCP的配体结合结构域(LBD)往往在介导趋化过程中起着关键性的作用,尽管利用ITC技术没有检测到MCP2923LBD与效应物的相互作用,但将MCP2923LBD敲除后,丧失了其对趋化效应物的趋化表型,表明MCP2923LBD在趋化过程中发挥重要作用。
1 材料和方法 1.1 质粒、基因组DNA和相关菌株 本研究使用的菌株和质粒均列于表 1内,CNB-1基因组DNA由本实验室自行提取保存。CNB-1及其突变株的活化或培养使用1.5%琼脂的LB平板或LB液体培养基,需要时加入终浓度为200 μg/mL的卡那霉素,培养温度30℃。E. coli BL21(DE3) 菌株的活化或培养也使用LB培养基,并加入50 μg/mL的卡那霉素。MCP2923利用pDSK519和pBBR1MCS-2进行回补,MCP2923LBD利用pET28a进行表达。
表 1. 本研究使用的菌株和质粒 Table 1. Strains and plasmids used in this study
| Strains/Plasmids | Relevant genotype or description | Source or references |
| Strains | ||
| CNB-1 | [14] | |
| CNB-1Δ20 | All the possible chemoreceptors were disrupted in CNB-1 | [12] |
| CNB-1ΔMCP2923 | MCP2923 was disrupted in CNB-1 | [12] |
| E. coli | ||
| BL21(DE3) | F– ompT hsdSB(rB– mB–) gal dcm (DE3) | Novagen |
| Plasmids | ||
| pDSK519 | Broad-host-range cloning vector; Kmr | [15] |
| pDSK519-MCP2923 | Carries MCP2923 to generate complementation for MCP2923 | |
| pBBR1MCS-2 | Kmr, lacPOZ′broad host vector with R | [16] |
| pBBR1MCS-2-MCP2923 | Carries MCP2923 to generate complementation for MCP2923 | This work |
| pBBR1MCS-2-MCP2923ΔLBD | Ligand binding domain deletion plasmid of MCP2923 | This work |
| pET-28a(+) | Protein expression vector | |
| pET-28a(+)-MCP2923LBD | pET28a derivative for expression of MCP2923LBD (Asn30–Ala188) | This work |
表选项
1.2 主要试剂 PCR反应使用TOYOBO生产的KOD-Plus-Neo高保真酶或康维世纪公司生产的2×Taq MasterMix进行;质粒提取均使用GenStar公司生产的StarPrep Plasmid Miniprep Kit提取试剂盒;PCR产物和酶切产物均使用OMEGA公司生产的E.Z.N.A.TM Gel Extraction Kit或E.Z.N.A.TM Cycle-Pure Kit进行纯化;酶切反应和连接反应所使用的酶均为NEB公司生产,pDSK519和pBBR1MCS-2使用Kpn Ⅰ-HF和Xba Ⅰ进行双酶切,pET28a使用Nde Ⅰ和EcoR Ⅰ-HF进行双酶切,连接反应使用T4 DNA Ligase进行连接。
游动平板法使用的琼脂为BD公司生产的Bacto Agar;Agarose-in-plug法使用Amresco公司生产的低熔点琼脂糖;无机盐培养基(MSB)成分为(g/L):Na2HPO4 1.00、NH4Cl 1.00、KH2PO4 0.50、MgSO4 0.03,pH 8.0;chemotaxis buffer (CB)成分为(mmol/L):NaH2PO4 40.00、EDTA 10.00、甘油0.05%,pH 8.0。
蛋白纯化缓冲液(mmol/L):Na2HPO450、NaCl 300,pH 7.4;透析缓冲液(mmol/L):NaCl 50、MES 5、PIPES 5、咪唑5、Tris 5,pH 8.0。
1.3 趋化性检测 游动平板法:将过夜培养的CNB-1或突变菌株以1%转接于LB培养基,30 ℃过夜培养,室温下离心收集菌体,用CB或MSB培养基洗涤菌体,相同条件下再次离心,弃去上清,重悬后,吸取0.5 μL菌体接种到趋化物浓度为0.15 g/L的MSB半固体平板上(琼脂浓度0.28%),于30 ℃培养箱中培养12–15 h后,观察并记录实验结果。根据趋化圈的直径将菌株的趋化表型定义为强、中强和弱3个层次,强趋化表型:CNB-1菌株的趋化圈直径大于2 cm,以“+++”表示,相应的化合物定义为强趋化效应物;中强趋化表型:CNB-1菌株的趋化圈直径在1–2 cm之间,以“++”表示,相应的化合物定义为中强趋化效应物;弱趋化表型:CNB-1菌株的趋化圈直径小于1 cm,以“+”表示,相应的化合物定义为弱趋化效应物。
Agarose-in-plug法:如上述方法收集细胞,重悬后,接种于溶有0.15 g/L 4-羟基苯甲酸(4HB)的MSB培养基中,培养5 h后,相同条件下收集、洗涤细胞,重悬细胞,调OD600至0.7后进行趋化性检测。
1.4 MCP2923LBD的表达纯化及配体的鉴定 将过夜培养的E. coli BL21-pET28a-MCP2923LBD以1%–2%的接种量转接于1.5 L LB培养基中,培养至OD600为0.6–0.8,0.3 mmol/L IPTG诱导11 h,4离心收集菌体,用蛋白纯化缓冲液洗涤1次,4 ℃再次收菌,并称重,以1:5的质量体积比重悬菌体,超声破碎,4 ℃离心,取上清,过滤后,利用GE公司生产的HiTrap TALON crude进行蛋白纯化,先分别用15 mmol/L和30 mmol/L咪唑进行洗杂,再用150 mmol/L咪唑进行洗脱,纯化样品经SDS-PAGE检测后,利用Millipore生产的Amicon Ultra-15 10K进行超滤浓缩,浓缩后的样品利用GE公司生产的HiLoad 16/60 Superdex 200 pg进一步纯化,再次超滤,浓缩后,过夜透析。
ITC实验利用TA公司生产的Affinity ITC进行。用透析液将上述浓缩样品稀释至100 μmol/L,化合物浓度为5 mmol/L,在25 ℃、75 r/min条件下进行ITC实验。蛋白浓度的测定利用Bio-Rad公司生产的Quick Start Bradford 1× Dye Reagent进行。
2 结果和分析 2.1 CNB-1及其MCP受体突变株对芳香化合物和TCA循环中间代谢产物的趋化表型检测 游动平板法是一种检测细菌趋化性的常用方法,利用该方法对35种芳香族化合物和9种TCA循环中间代谢产物进行了筛选(表 2,图 1),结果表明,CNB-1菌株对12种芳香族化合物和9种TCA循环中间代谢产物具有趋化表型,其中对PCA和4HB具有强趋化表型,PCA和4HB为强趋化效应物,对3HPA、Gen、3HB、Van、Ben、cis-Aco、Mal、Fum和Oxa具有中强趋化表型,这9种化合物为中强趋化效应物,对Vin、Cat、Phe、4FP、GA、α-Ket和iCit具有弱趋化表型,这7种化合物为弱趋化效应物。
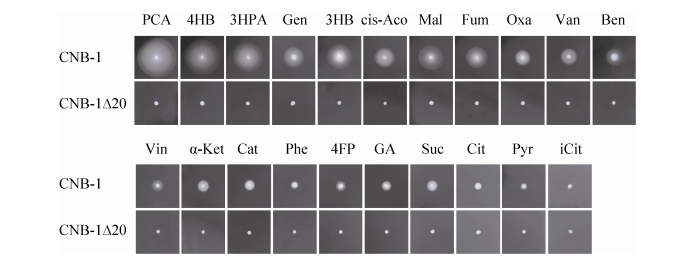 |
| 图 1 游动平板法检测CNB-1对21种化合物的趋化表型 Figure 1 Chemotactic responses to 21 compounds (at final concentrations of 0.15 g/L) by strain CNB-1 and CNB-1Δ20, as determined with swimming plate assays. All tests were performed in triplicate, and one set of bacterial colony photos is displayed. The abbreviations of each compound were listed in Table 2. |
| 图选项 |
表 2. CNB-1对44种化合物的趋化表型检测结果 Table 2. The phenotype of CNB-1 to 44 compounds
| Compounds | abbreviation | phenotype |
| Protocatechuic acid | PCA | +++ |
| 4-Hydroxybenzoic acid | 4HB | +++ |
| 3-Hydroxybenzoic acid | 3HPA | ++ |
| Gentisuric Acid | Gen | ++ |
| 3-Hydroxyphenylacetic acid | 3HB | ++ |
| cis-Aconitic acid | cis-Aco | ++ |
| Malic acid | Mal | ++ |
| Fumaric acid | Fum | ++ |
| Oxaloacetic acid | Oxa | ++ |
| Vanillic acid | Van | ++ |
| Benzoic acid | Ben | ++ |
| Vanillin | Vin | + |
| α-Ketoglutaric acid | α-Ket | + |
| Catechol | Cat | + |
| Phenol | Phe | + |
| p-Hydroxybenzaldehyde | 4FP | + |
| Gallic acid | GA | + |
| Citric acid | Cit | + |
| Pyruvic acid | Pyr | + |
| Succinic acid | Suc | + |
| Isocitric acid | iCit | + |
| 4-Chlorobenzoic acid | 4CB | - |
| Resorcinol | Res | - |
| 4-Hydroxysalicylic acid | 4HS | - |
| 4-Chlorocatechol | 4CP | - |
| Phenylacetic acid | PAC | - |
| m-Toluic acid | 3TC | - |
| 4-Nitrobenzoic acid | 4NB | - |
| 3, 4-Dihychroxyphenylacetic acid | 34DPA | - |
| 4-Methlcatochol | 4MP | - |
| 3-Methlcatochol | 3MP | - |
| p-Dimethylancino benzaldehyde | 4DB | - |
| Biphenyl | Bip | - |
| Diphenylamine | Dip | - |
| m-Phenylenediamine hydrochloride | 3PH | - |
| 5-Sulfosalicylic acid | Kal | - |
| p-Nitroaniline | 4NA | - |
| 4-Hydroxyberzyl alcohol | 4HBA | - |
| m-Nitrochlorobenzerle | 3NCB | - |
| Ethylene-diamine dihydrochloride | EDD | - |
| Metanilic acid | 3ABS | - |
| Chromotropic acid | Chr | - |
| 3-Hydroxybenzyl alcohol | 3HBA | - |
| 2-Hydroxybenzylal alcohol | 2DT | - |
表选项
2.2 突变菌株CNB-1ΔMCP2923的趋化表型减弱 为了验证MCP2923在CNB-1中介导趋化作用,对CNB-1ΔMCP2923菌株的趋化表型进行了检测。结果显示,相对于CNB-1野生株,MCP2923基因的敲除突变株CNB-1ΔMCP2923对20种趋化物的趋化表型减弱,而对异柠檬酸等其他化合物的趋化则没有明显影响(图 2-C)。其中,对Gen、3HB、3HPA和Oxa趋化减弱在25%以上;对4HB和Mal趋化减弱的比例在20%左右;对Ben、PCA、Van、Vin、cis-Aco、Fum和α-Ket趋化减弱的比例在16%左右;对于Cat和Phe趋化减弱的比例分别为26%和39.8%。这表明MCP2923参与了CNB-1对这些化合物的趋化,且多种MCP在这一过程中发挥着作用。
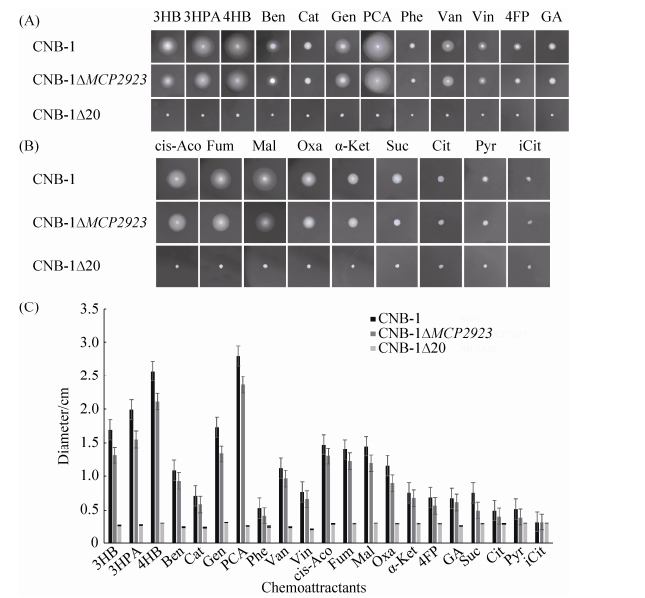 |
| 图 2 游动平板法检测CNB-1Δ2923对21种趋化物的趋化表型 Figure 2 Chemotactic responses to 21 chemoattractants with strain CNB-1 and mutants (CNB-1Δ20 and CNB-1ΔMCP2923). Swiming plate assays were used. All tests were performed in triplicates, and one set of bacterial colony photos is displayed (A) & (B). Averages of colony diameters were plotted, and standard deviations are shown (C). The abbreviations of each compound was listed in Table 2. |
| 图选项 |
2.3 MCP2923基因可以部分回补CNB-1Δ20的趋化表型 在确定敲除MCP2923基因可以减弱趋化表型后,进一步将MCP2923回补到CNB-1Δ20菌株中,以确定MCP2923在CNB-1菌株趋化中的作用(图 3-A,B)。pBBR1MCS-2和pDSK519两种质粒对MCP2923回补结果表明,MCP2923可以回补CNB-1Δ20对上述趋化物中15种的趋化表型,但无法回补CNB-1Δ20对4FP和GA的趋化表型。CNB-1Δ20/pBBR1MCS-2-MCP2923的表型强于CNB-1Δ20/pDSK519-MCP2923,这可能与这2个质粒在宿主菌株的拷贝数差异有关。CNB-1Δ20/ pBBR1MCS-2-MCP2923对3HB、3HPA、4HB和Mal的趋化表型的回补比例分别为23.5%、31.0%、23.4%和27.8% (图 3-C),这与敲除MCP2923基因造成的表型减弱比例大致相同。而对于其他化合物,MCP2923回补产生的表型变化比例均明显大于MCP2923敲除产生的表型变化比例。
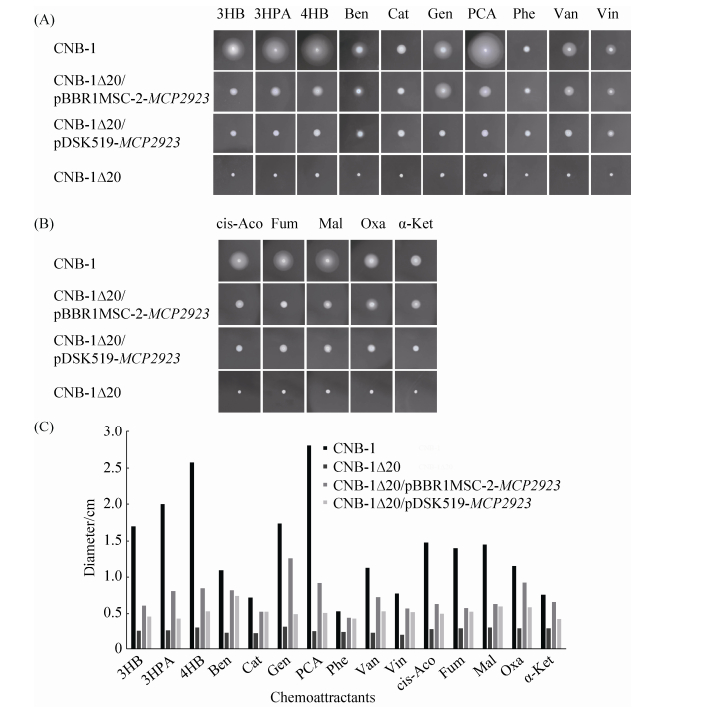 |
| 图 3 游动平板法检测MCP2923基因回补CNB-1Δ20的趋化表型 Figure 3 Chemotactic responses to 15 chemoattractants with strain CNB-1 and mutants (CNB-1Δ20、CNB-1Δ20/pBBR1MCS-2-MCP2923and CNB-1Δ20/pDSK519-MCP2923). Swimming plate assays were used. All tests were performed in triplicate, and one set of bacterial colony photos is displayed (A) & (B). Averages of colony diameters were plotted, and standard deviations are shown (C). The abbreviations of each compound was listed in Table 2. |
| 图选项 |
2.4 利用Agarose-in-plug法检测MCP2923对芳香族化合物的趋化性 游动平板法相对于其他方法具有简单易行的优点,但趋化效应物的浓度梯度形成是依赖代谢的,这意味着如果某一类化合物不能被菌株代谢,则不能使用该方法检测趋化性。对比游动平板法,Agarose-in-plug法依赖趋化物在溶液中扩散形成浓度梯度,随后实验使用plug-in法筛选可能与MCP2923存在直接相互作用的趋化效应物(图 4)。利用Agarose-in-plug法对10种芳香族化合物进行检测,但只检测到了单回补菌株对PCA和4HB的趋化表型,说明MCP2923介导的对芳香化合物的趋化作用,包括了代谢依赖型和非依赖型两种趋化现象。
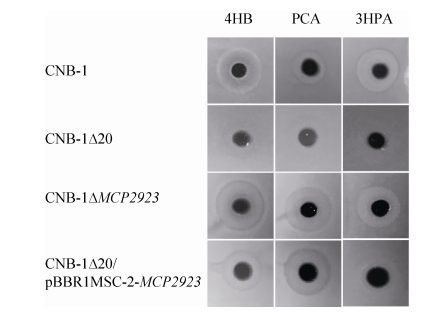 |
| 图 4 Agarose-in-plug法检测MCP2923对芳香族化合物的趋化性 Figure 4 Chemotactic responses of strain CNB-1 and mutants (CNB-1Δ20, CNB-1ΔMCP2923 and CNB-1Δ20/ pBBR1MCS-2-MCP2923) to 4HB, PCA and 3HPA using agarose-in-plug assays. The concentration of each compound used in the assays was 10 mmol/L. The abbreviations of each compound were listed in Table 2. |
| 图选项 |
2.5 MCP2923蛋白分子的跨膜结构和配体结合区的生物信息学分析 根据MCP跨膜区的数目和LBD结构域的位置可以将其分为5种不同的拓扑结构,分别为Ⅰa、Ⅰb、Ⅱ、Ⅲ、Ⅰⅴa和Ⅳb[17]。为了获得MCP2923的拓扑结构,对MCP2923进行了相关分析。DAS是根据疏水性氨基酸序列来预测蛋白跨膜区的生物信息学工具,经DAS预测,MCP2923在第12–29位氨基酸和第192–208位氨基酸具有两段跨膜区(图 5-A)。Pfam数据库和SMART数据库进一步分析也印证了这一预测,结果表明MCP2923具有典型MCP所具有的HAMP结构域和MA结构域(图 5-B)。因此可以认为,MCP2923为2次跨膜,并且配体结合结构域在膜外侧,属于Ⅰa型拓扑结构,LBD结构域的长度为162个氨基酸残基。
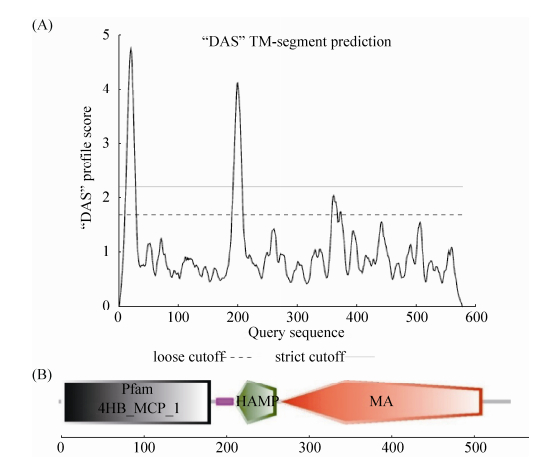 |
| 图 5 MCP2923的结构域和跨膜区分析 Figure 5 The bioinformatic analysis for MCP2923 is performed by DAS server, Pfam database and SMART database. A: the DAS server predict MCP2923 has two transmembrane regions, F12 to L29 and A192 to I208; B: protein domain of MCP2923 is analyzed by Pfam database and SMART database. MCP2923 has HAMP domain and MA domain, representing a typical MCP. |
| 图选项 |
2.6 MCP2923LBD在MCP2923介导的趋化表型中起关键作用 配体结合结构域(ligand binding domain, LBD)是MCP与配体相结合的区域,是趋化信号的起始位置,在细菌的趋化中起着关键作用。为了验证MCP2923LBD对MCP2923功能的必需性,将MCP2923LBD结构域敲除,构建了CNB-1Δ20/ pBBR1MCS-2-MCP2923ΔLBD菌株,利用游动平板法对其趋化性进行了检测(图 6)。结果表明,缺失配体结合结构域的MCP2923无法回补CNB-1Δ20对PCA和Oxa等15种趋化效应物的趋化性。表明MCP2923LBD在MCP2923介导的趋化表型中是必需的。
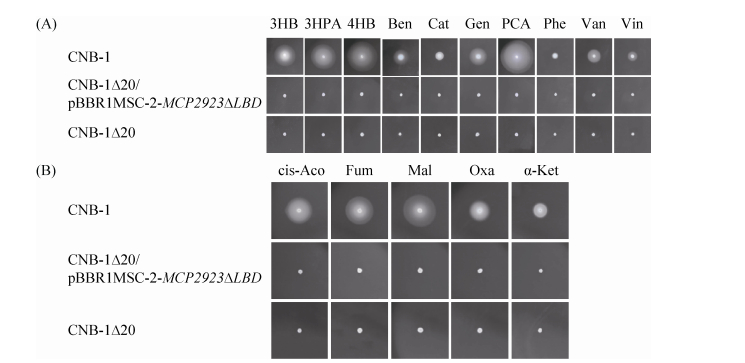 |
| 图 6 敲除MCP2923LBD使MCP2923无法回补CNB-1Δ20趋化表型 Figure 6 Chemotactic responses to 15 chemoattractants with strain CNB-1 and mutants (CNB-1Δ20, CNB-1Δ20/pBBR1MCS-2-MCP2923ΔLBD). Swimming plate assays were used. All tests were performed in triplicate, and one set of bacterial colony photos is displayed (A) & (B). Averages of colony diameters were plotted. The abbreviations of each compound were listed in Table 2. |
| 图选项 |
2.7 MCP2923LBD的纯化及其配体的筛选 为了进一步确定MCP2923是否与上述趋化物存在直接的相互作用,在表型实验证明MCP2923LBD在MCP2923介导的趋化表型中起关键作用后,进一步利用生化实验对上述结论进行了验证。
对6种芳香族化合物(PCA、4HB、3HPA、Gen、Cat和Phe)的滴定结果表明,MCP2923LBD与这6种趋化物均不存在明显的相互作用。虽然在对PCA进行实验时出现了热量变化趋势,但5 mmol/L PCA滴定透析液的对照实验中也出现了热量变化的趋势,因此不能确定PCA与MCP2923LBD具有相互作用。对4种TCA循环中间代谢产物的筛选也没有检测到其与MCP2923LBD之间存在相互作用。
3 讨论 通过游动平板法对44种化合物的筛选,我们发现了CNB-1具有趋化表型的21种趋化效应物,其中包括12种芳香化合物和9种TCA循环中间代谢产物,MCP2923基因可回补CNB-1Δ20对这21种趋化效应物中15种趋化效应物的表型,不能恢复对4FP、GA、Cit、Pyr和Suc等趋化效应物的表型,其中原因尚不清楚。敲除MCP2923基因使突变菌株CNB-1ΔMCP2923相对于野生型菌株表型均略有减弱;缺失LBD的MCP2923不能恢复突变菌株CNB-1Δ20对本实验测定的效应物的趋化性。以上实验结果都表明,MCP2923可以介导CNB-1对多种芳香化合物和小分子有机酸等趋化效应物的趋化表型,并且其LBD结构域在其介导趋化表型的过程中起关键作用。对大多数化合物而言,MCP2923基因敲除造成的趋化圈减弱比例小于MCP2923所回补的趋化圈比例,这在一定程度上说明,CNB-1中MCP的功能存在冗余。有意思的是,虽然从表型实验的结果看,单回补MCP2923无法介导CNB-1对4FP、GA、Cit、Pry和Suc的趋化,但敲除MCP2923却对趋化表型产生了影响,这表明MCP2923对这5种化合物的趋化可能需要多种MCP共同参与。另外,是否利用4HB对菌株进行诱导培养,直接影响着Agarose-in-plug法的实验结果,这一现象与PcaY介导的对芳香族化合物的趋化相似[18]。在最近的研究中,Lopez-Farfan等人利用RT-PCR的方法对P. putida KT2440中的MCP基因的转录水平进行了检测,发现在趋化效应物的存在下,相应MCP基因的转录水平多数上调[19]。结合两种单回补菌株CNB-1Δ20/pDSK519-MCP2923和CNB-1Δ20/ pBBR1MCS-2-MCP2923的表型检测结果以及敲除、回补表型比例存在差异的结果,表明MCP2923的表达水平可能影响菌株的趋化表型。
ITC实验表明MCP2923LBD与所检测的10种化合物均没有相互作用。Iwaki等人实验证明了NbaY可以引发对2-硝基苯甲酸的趋化表型[20],但García等人研究表明,与NbaY具有高相似性的McpP可以结合丙酮酸,但不能与2-硝基苯甲酸相互作用[21]。另外有研究表明,MCP对苯酚的趋化性也不是通过与其LBD结构域结合实现的,而是与MCP跨膜区或HAMP结构域相互作用来介导菌株对苯酚的趋化表型[22]。综上所述,MCP2923可能存在LBD结构域与化合物直接作用以外的其他趋化作用机制,确切的分子机制还有待研究。
微生物的趋化性不仅可以使自身处于有利的环境中,还可以通过自身的趋化运动提高其降解环境中污染物的能力和效率。例如,Paul等人通过模拟环境研究了Ralstonia sp.在土壤中对4-硝基苯酚的降解。结果表明,具有趋化性的SJ98菌株其降解能力强于不具有趋化性的RKJ200菌株[23]。将微生物的趋化性与芳香族化合物的降解能力相结合,可以加强微生物对环境中芳香族化合物的降解能力,治理芳香族化合物造成的环境污染。MCP作为趋化通路中的第一个分子,在微生物的趋化中起着重要的作用。研究MCP不仅有利于我们更好地理解微生物趋化性本身,也有助于构建更加高效的芳香族化合物降解菌株。
References
| [1] | Parkinson JS, Hazelbauer GL, Falke JJ. Signaling and sensory adaptation in Escherichia coli chemoreceptors:2015 update. Trends in Microbiology, 2015, 23(5): 257-266. DOI:10.1016/j.tim.2015.03.003 |
| [2] | Díaz E, Jiménez JI, Nogales J. Aerobic degradation of aromatic compounds. Current Opinion in Biotechnology, 2013, 24(3): 431-442. DOI:10.1016/j.copbio.2012.10.010 |
| [3] | Nelson KE, Weinel C, Paulsen IT, Dodson RJ, Hilbert H, Martins dos Santos VAP, Fouts DE, Gill SR, Pop M, Holmes M, Brinkac L, Beanan M, DeBoy RT, Daugherty S, Kolonay J, Madupu R, Nelson W, White O, Peterson J, Khouri H, Hance I, Chris Lee P, Holtzapple E, Scanlan D, Tran K, Moazzez A, Utterback T, Rizzo M, Lee K, Kosack D, Moestl D, Wedler H, Lauber J, Stjepandic D, Hoheisel J, Straetz M, Heim S, Kiewitz C, Eisen J, Timmis KN, Düsterhoft A, Tümmler B, Fraser CM. Complete genome sequence and comparative analysis of the metabolically versatile Pseudomonas putida KT2440. Environmental Microbiology, 2002, 4(12): 799-808. DOI:10.1046/j.1462-2920.2002.00366.x |
| [4] | Ma YF, Zhang Y, Zhang JY, Chen DW, Zhu YQ, Zheng HJ, Wang SY, Jiang CY, Zhao GP, Liu SJ. The complete genome of Comamonas testosteroni reveals its genetic adaptations to changing environments. Applied and Environmental Microbiology, 2009, 75(21): 6812-6819. DOI:10.1128/AEM.00933-09 |
| [5] | Masai E, Kamimura N, Kasai D, Oguchi A, Ankai A, Fukui S, Takahashi M, Yashiro I, Sasaki H, Harada T, Nakamura S, Katano Y, Narita-Yamada S, Nakazawa H, Hara H, Katayama Y, Fukuda M, Yamazaki S, Fujita N. Complete genome sequence of Sphingobium sp. strain SYK-6, a degrader of lignin-derived biaryls and monoaryls. Journal of Bacteriology, 2012, 194(2): 534-535. DOI:10.1128/JB.06254-11 |
| [6] | Samanta SK, Bhushan B, Chauhan A, Jain RK. Chemotaxis of a Ralstonia sp. SJ98 toward different nitroaromatic compounds and their degradation. Biochemical and Biophysical Research Communications, 2000, 269(1): 117-123. DOI:10.1006/bbrc.2000.2204 |
| [7] | Parales RE, Ditty JL, Harwood CS. Toluene-degrading bacteria are chemotactic towards the environmental pollutants benzene, toluene, and trichloroethylene. Applied and Environmental Microbiology, 2000, 66(9): 4098-4104. DOI:10.1128/AEM.66.9.4098-4104.2000 |
| [8] | Lacal J, Mu?oz-Martínez F, Reyes-Darías JA, Duque E, Matilla M, Segura A, Ortega-Calvo JJ, Jímenez-Sánchez C, Krell T, Ramos JL. Bacterial chemotaxis towards aromatic hydrocarbons in Pseudomonas. Environmental Microbiology, 2011, 13(7): 1733-1744. DOI:10.1111/j.1462-2920.2011.02493.x |
| [9] | Grimm AC, Harwood CS. NahY, A catabolic plasmid-encoded receptor required for chemotaxis of Pseudomonas putida to the aromatic hydrocarbon naphthalene. Journal of Bacteriology, 1999, 181(10): 3310-3316. |
| [10] | Vangnai AS, Takeuchi K, Oku S, Kataoka N, Nitisakulkan T, Tajima T, Kato J. Identification of CtpL as a chromosomally encoded chemoreceptor for 4-chloroaniline and catechol in Pseudomonas aeruginosa PAO1. Applied and Environmental Microbiology, 2013, 79(23): 7241-7248. DOI:10.1128/AEM.02428-13 |
| [11] | Ni B, Huang Z, Wu YF, Fan Z, Jiang CY, Liu SJ. A novel chemoreceptor MCP2983 from Comamonas testosteroni specifically binds to cis-aconitate and triggers chemotaxis towards diverse organic compounds. Applied Microbiology and Biotechnology, 2015, 99(6): 2773-2781. DOI:10.1007/s00253-014-6216-3 |
| [12] | Ni B, Huang Z, Fan Z, Jiang CY, Liu SJ. Comamonas testosteroni uses a chemoreceptor for tricarboxylic acid cycle intermediates to trigger chemotactic responses towards aromatic compounds. Molecular Microbiology, 2013, 90(4): 813-823. DOI:10.1111/mmi.12400 |
| [13] | Huang Z, Ni B, Jiang CY, Wu YF, He YZ, Parales RE, Liu SJ. Direct sensing and signal transduction during bacterial chemotaxis toward aromatic compounds in Comamonas testosteroni. Molecular Microbiology, 2016, 101(2): 224-237. DOI:10.1111/mmi.2016.101.issue-2 |
| [14] | Wu JF, Sun CW, Jiang CY, Liu ZP, Liu SJ. A novel 2-aminophenol 1, 6-dioxygenase involved in the degradation of p-chloronitrobenzene by Comamonas strain CNB-1:purification, properties, genetic cloning and expression in Escherichia coli. Archives of Microbiology, 2005, 183(1): 1-8. DOI:10.1007/s00203-004-0738-5 |
| [15] | Keen NT, Tamaki S, Kobayashi D, Trollinger D. Improved broad-host-range plasmids for DNA cloning in gram-negative bacteria. Gene, 1988, 70(1): 191-197. DOI:10.1016/0378-1119(88)90117-5 |
| [16] | Kovach ME, Elzer PH, Hill DS, Robertson GT, Farris MA, Roop RM 2nd, Peterson KM. Four new derivatives of the broad-host-range cloning vector pBBR1MCS, carrying different antibiotic-resistance cassettes. Gene, 1995, 166(1): 175-176. DOI:10.1016/0378-1119(95)00584-1 |
| [17] | Lacal J, García-Fontana C, Mu?oz-Martínez F, Ramos JL, Krell T. Sensing of environmental signals:classification of chemoreceptors according to the size of their ligand binding regions. Environmental Microbiology, 2010, 12(11): 2873-2884. DOI:10.1111/emi.2010.12.issue-11 |
| [18] | Luu RA, Kootstra JD, Nesteryuk V, Brunton CN, Parales JV, Ditty JL, Parales RE. Integration of chemotaxis, transport and catabolism in Pseudomonas putida and identification of the aromatic acid chemoreceptor PcaY. Molecular Microbiology, 2015, 96(1): 134-147. DOI:10.1111/mmi.2015.96.issue-1 |
| [19] | López-Farfán D, Reyes-Darias JA, Krell T. The expression of many chemoreceptor genes depends on the cognate chemoeffector as well as on the growth medium and phase. Current Genetics. DOI:10.1007/s00294-016-0646-7 |
| [20] | Iwaki H, Muraki T, Ishihara S, Hasegawa Y, Rankin KN, Sulea T, Boyd J, Lau PCK. Characterization of a pseudomonad 2-nitrobenzoate nitroreductase and its catabolic pathway-associated 2-hydroxylaminobenzoate mutase and a chemoreceptor involved in 2-nitrobenzoate chemotaxis. Journal of Bacteriology, 2007, 189(9): 3502-3514. DOI:10.1128/JB.01098-06 |
| [21] | García V, Reyes-Darias JA, Martín-Mora D, Morel B, Matilla MA, Krell T. Identification of a chemoreceptor for C2 and C3 carboxylic acids. Applied and Environmental Microbiology, 2015, 81(16): 5449-5457. DOI:10.1128/AEM.01529-15 |
| [22] | Pham HT, Parkinson JS. Phenol sensing by Escherichia coli chemoreceptors:a nonclassical mechanism. Journal of Bacteriology, 2011, 193(23): 6597-6604. DOI:10.1128/JB.05987-11 |
| [23] | Paul D, Singh R, Jain RK. Chemotaxis of Ralstonia sp. SJ98 towards p-nitrophenol in soil. Environmental Microbiology, 2006, 8(10): 1797-1804. DOI:10.1111/emi.2006.8.issue-10 |
