马紫卉1, 2, 李伟1, 尹文兵1


1. 中国科学院微生物研究所, 真菌学国家重点实验室, 北京 100101;
2. 中国科学院大学, 北京 100449
收稿日期:2015-09-19; 修回日期:2015-12-05;网络出版日期:2015-12-11
基金项目:国家自然科学基金(31470178);中国科学院“****”(Y454011001);北京市自然科学基金(5152018)
通讯作者:尹文兵,E-mail:yinwb@im.ac.cn
摘要: 真菌天然产物是天然药物的重要来源之一,大规模真菌基因组序列测序的完成表明真菌具有产生丰富的次级代谢产物的潜能。然而,许多真菌或生长缓慢,或不适宜在实验室条件下培养,或难以进行遗传操作,或化合物产量极低等,这些因素导致大量有价值的真菌天然产物无法获得。利用异源表达系统对真菌天然产物进行生产是发现新天然产物及解析其生物合成途径的有效手段,并为定向的以合成生物学的手段去合成重要活性分子奠定基础。本文对目前用于真菌天然产物生产的各种异源表达系统进行了综述,并结合最新的DNA组装技术展望了异源表达系统在真菌天然产物研究中的应用价值和前景。
关键词: 天然产物基因簇异源表达合成生物学
Progress in heterologous expression of fungal natural products-A review
Zihui Ma1, 2, Wei Li1, Wenbing Yin1


1. State Key Laboratory of Mycology, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
2. University of Chinese Academy of Sciences, Beijing 100449, China
Abstract:Fungal products are important resources for natural drugs. Availabilities of many fungal genome sequences indicate that fungi have the huge potential to produce secondary metabolites. However, many valuable fungal natural products are hard to obtain under regular lab conditions because fungi are slow to grow, difficult to genetically modify, or have low yield of desired metabolites. Therefore, developing a heterologous expression system has been an efficient approach to discover novel natural product and elucidate the complicated biosynthetic pathways. This review describes several heterologous expression systems that have been successfully used for the expression of fungal biosynthetic gene clusters. Also, we address the application perspective based on the emerging DNA assembly technologies.
Key words: natural productgene clusterheterologous expressionsynthetic biology
活性天然产物是现代新药创制和发现的重要基础,真菌作为活性天然产物的重要来源之一引起了人们极大的关注,主要是因为其丰富的资源、代谢产物结构的多样化及潜在的成药能力,这些代谢产物常常称之为次级代谢产物(secondary metabolite,SM)。对真菌自身而言,这些次级代谢产物有利于真菌在自然环境中竞争营养物质,抵御侵害,实现自我防御,并可作为与环境中生物交流的信号分子[1]。对社会而言,真菌来源的有益天然产物已经在医药行业发挥重大作用,为人类的健康做出重大贡献,如由产黄青霉(Penicillium chrysogenum)产生的抗生素青霉素(penicillin)在二战时期挽救了众多人民的生命,由皱瓣曲霉(Aspergillus rugulosus)产生的棘白菌素B (echinocandin B)作为先导化合物合成的药物阿尼芬净(anidulafungin)和来源于灰黄青霉(P. griseofulvum)的灰黄霉素(griseofulvin)具有抗真菌感染的作用(图 1)。除此之外,还有由包括茄病镰刀菌(Fusarium solani)在内的多种真菌产生的环孢素A (cyclosporin A)可作为免疫抑制剂(图 1),由构巢曲霉(A. nidulans)产生的苔色酸衍生物可用作抗骨质疏松的药物,麦角菌属(Claviceps spp.)真菌产生的生物碱具有抗偏头痛和降血压药物的作用,土曲霉(A. terreus)产生的洛伐他汀(lovastatin)用于降胆固醇药物(图 1)。近些年,也有报道抗癌药物紫杉醇(paclitaxel)(图 1)可由真菌黄灰青霉(P. aurantiogriseum)产生 [1, 2, 3],如果能利用真菌去生产紫杉醇,将会大大节约成本,为社会做出不可估量的贡献。
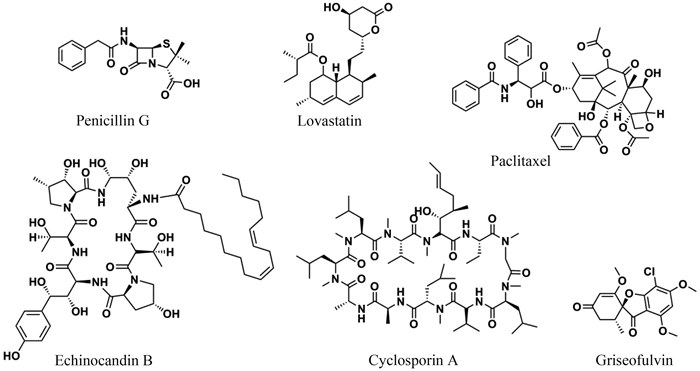 |
| 图 1. 真菌天然产物药物代表[3, 4, 5, 6, 7] Figure 1. Drugs from fungal natural products [3, 4, 5, 6, 7]. |
| 图选项 |
然而,随着社会的发展和技术的日益更新,人们发现新天然产物的脚步却越来越慢,开发成为新药的越来越少,新药的发现面临极大的挑战。归结其原因,不仅仅是资源问题,更主要的是传统的新天然产物的发现方法遇到瓶颈。1953年,Watson和Crick揭示了DNA的双螺旋结构,从而开启了分子生物学时代,天然产物的研究也搭上了分子生物学的快车。在早期研究链霉菌次级代谢产物生物合成的过程中,人们发现一个重要的现象,合成次级代谢产物的基因是成簇存在的,称之为基因簇(gene cluster),这为解析代谢产物生物合成途径奠定了分子基础,该领域的奠基人之一是英国约翰英纳斯中心(John Innes Center)的David Hopwood爵士。随后的研究发现,真菌次级代谢产物的合成基因也遵循此规律,即在染色体上成簇存在,这一发现极大地促进了人们对真菌次级代谢产物生物合成机制的深入研究[2]。这种基因的排列方式可使其高效的完成化学骨架母核合成、底物或化学产物的运输、特定的调节和抑制功能等,典型的基因簇特征可作为从真菌基因组挖掘并寻找有用化合物的重要参考信息[4]。
近年来,基因组测序技术的发展越来越迅速,成本也越来越低,这使得大量真菌基因组测序完成。至2015年9月初,据NCBI数据库公布的数据,已有1146个真菌的基因组测序到contig水平,其中包括606个达到scaffold水平(http://www. ncbi.nlm.nih.gov/genome/)。生物信息学分析发现,真菌基因组中蕴含着丰富的次级代谢产物信息,远远超出我们的预期。以测序完成的几个重要曲霉为例,分析其基因组中合成次级代谢产物的基因簇表明:从烟曲霉(A. fumigatus)中的36个到黑曲霉(A. niger)的79个基因簇不等,然而从基因簇鉴定的比例来看,研究最多的模式真菌构巢曲霉也只有32%被鉴定,还有近70%有待挖掘 (表 1)。这暗示着真菌基因组中蕴含着丰富的天然产物宝藏,将成为发现新天然产物的重要依据,为重新发现新的天然产物打开了一扇大门[8]。尽管如此,人们对真菌的认识却还远远不够,由于大多数真菌的培养困难,其遗传操作体系更是难以建立,这都大大阻碍了人们从真菌中发现新的天然产物的步伐。因此,人们把研究的策略转向遗传背景清晰的异源宿主,利用或开发一个成熟的遗传转化系统、分子操作容易的异源宿主来表达单个生物合成基因或者完整的基因簇。本文将对近年来真菌天然产物在常用的原核和真核异源宿主的表达和生产进展情况进行综述,并结合最新的DNA组装技术展望了异源表达系统在真菌天然产物研究中的应用价值和前景。
表 1. 模式真菌次级代谢产物基因簇的鉴定现状[9]Table 1. Characterization status of secondary metabolite biosynthetic gene cluster from model fungi[9]
| Species | Strains | Size/Mb | Putative SM Core Genes | Identified metabolites | Percent ratio/% |
| Aspergillus nidulans | FGSC A4 | 30.07 | 53 | 17 | 32.1 |
| Aspergillus fumigatus | Af293 | 29.38 | 36 | 11 | 30.6 |
| Aspergillus flavus | NRRL3357 | 36.79 | 62 | 3 | 4.8 |
| Aspergillus niger | CBS513.88 | 37.20 | 79 | 4 | 5.1 |
| Aspergillus terreus | NIH2624 | 29.33 | 65 | 5 | 7.7 |
表选项
1 原核生物表达系统 长期以来,大肠杆菌(Escherichia coli)被用作基因工程和代谢工程的模式菌株,由于其培养条件简单、生长周期短、遗传背景清晰且具备完善的分子生物学操作技术等优势,成为进行异源表达的理想宿主。由于该系统属于原核表达系统,大多原核生物来源的非核糖体多肽类化合物 (Non-Ribosomal Peptides,NRPs)、聚酮类化合物(Polyketides,PKs)和源于聚酮和非核糖体多肽杂合的化合物NRPs/PKs基因簇编码的化合物在此表达系统中可成功获得表达[10]。在真菌天然产物的研究中,大肠杆菌多用于真菌天然产物生物合成途径中关键酶的表达。大肠杆菌作为原核生物表达真菌来源的基因或基因簇存在很大的局限性,大多所表达的单个基因不能产生目标化合物[11],有时获得目标产物还需要对培养条件优化并且添加前体[12]。因此,将大肠杆菌用于体外酶的表达或添加前体的功能验证还是不错的选择。此外,枯草芽孢杆菌(Bacillus subtilis)也作为真菌天然产物的异源宿主,如Schweder等成功表达了来自尖孢镰刀菌(Fusarium oxysporum)的非核糖体环缩肽恩镰孢菌素(enniatin),这是首次将来自真核生物的非核糖体多肽产生基因在枯草芽孢杆菌中表达,而且恩镰孢菌素不在枯草芽孢杆菌体内累积,能够完全排出体外,具有投入到实际发酵生产的优势[13]。
2 酵母表达系统酵母是单细胞真核生物,作为表达系统具有生长快速、培养条件简单和基因操作技术成熟等特点,其分泌途径能够进行正确的蛋白质加工和翻译后修饰,而且没有内毒素。同时,酵母遗传背景清楚,是第一个被测序的真核生物,早在1996年,酿酒酵母(Saccharomyces cerevisiae)的基因组测序工作就已经完成 ,且有多个营养缺陷选择标记的菌株可供使用,因此,被用于真菌中合成次级代谢产物基因或简单基因簇的表达。
近年来,酿酒酵母在真菌天然产物生物合成途径解析和新型天然产物开发等方面均显示了一定的优势。2013年日本Watanabe课题组综述了酿酒酵母作为宿主对真菌天然产物进行成功表达的研究进展,并揭示了酿酒酵母在研究天然产物生物合成酶的催化机制以及挖掘真菌基因组新的次级代谢产物方面发挥的重大作用[14]。该课题组在酿酒酵母中建立起一套异源表达真菌聚酮类化合物与非核糖体多肽的完整体系,利用创建的重叠延伸PCR-酵母同源重组(overlap extension PCR-yeast homologous recombination,ExRec)方法从自然宿主cDNA文库中得到5-12 kb的目的基因,并整合到线性化的载体上。所用酿酒酵母经过改造携带matB基因和npgA基因,matB基因编码的丙二酸单酰辅酶A合成酶(malonyl-CoA synthetase)可作为产物合成的起始或延伸模块,npgA基因编码的PPTase保证载体蛋白的活性。整套系统已经成功将球毛壳菌(Chaetomium globosum)中5个功能未知的基因分别表达,得到6个聚酮类化合物,经过基因敲除实验证明这些基因与cochliodone A的合成相关[14]。从异源表达的基因来源角度看,将不同来源的基因组合表达,是开发新型天然产物的另一条途径。2014年Xu等把来自不同真菌的聚酮类苯二酚内酯(benzenediol lactone,BDL)生物合成途径的聚酮合酶进行组合,依靠酵母异源表达系统,产生出结构多样的聚酮类化合物,这是在天然产物的基础上,经过对生物合成途径的改造得到新的化合物,从而发掘出一个全新的生物活性分子的领域[15]。最近Galanie等设计出一种利用经过改造后能异源表达来自不同物种的多种酶的酵母,以糖为底物在一系列酶的作用下合成吗啡以及其他天然或者半合成的类似物的表达系统,此研究为利用酵母生产天然产物的合成生物学研究提出了一条崭新的思路。在解析真菌天然产物复杂合成途径方面,酵母表达系统也发挥了重要作用,如Saruwatari等利用酵母异源表达体系,首次确定了黄曲霉中生成二聚体化二酮哌嗪生物碱(-)-ditryptophenaline的基因簇及其生物合成途径,并通过体外实验证明DtpCs是一个特殊的细胞色素氧化酶P450,催化闭环反应和碳碳键的形成。2015年Jakubczyk等运用酵母表达体系,确定了日本曲酶(A. japonicus)中easH基因在麦角生物碱中间体裸麦角碱-Ⅰ下游产物cycloclavine生物合成中的作用,结合体外的生化实验,解析部分合成途径,成功鉴定出能催化裸麦角碱-Ⅰ重排形成特殊环丙基的酶,而cycloclavine产量也超过500 mg/L[16]。 此外,酵母也可用于表达植物天然产物,如黄花蒿(Artemisia annua)所产的青蒿素(artemisinin)是治疗疟疾的有效药物,Ro等在酿酒酵母中成功合成重要中间体青蒿酸(artemisinic acid),产量高达 100 mg/L[17]。
尽管酵母在天然产物生物合成以及在合成生物学研究中作为底盘生物发挥着很大作用,但也存在着不足之处,如由于真核基因表达需要去除基因的内含子,而酵母本身缺乏高等的mRNA剪切系统;同时,对于含有3个以上基因的次级代谢产物合成基因簇,其操作难度很大。因此,对于功能更强大的酵母工程菌作为异源宿主的需求迫在眉睫,也需要很长一段路要走。
3 丝状真菌表达系统随着分子技术的发展,丝状真菌被开发作为异源蛋白和小分子化合物的表达宿主[18],与上述提及的大肠杆菌和酵母异源宿主相比,丝状真菌最明显的优势在于能够表达真菌天然产物生物合成的完整基因簇[11],且在克隆基因簇时,不需要去除基因的内含子,丝状真菌宿主大多就能正确地完成来自其它真菌次级代谢产物基因的内含子剪接工作,成功产出目的产物[19, 20]。可见,在多数情况下,具有生产天然产物能力的丝状真菌作为异源宿主来表达真菌天然产物的成功率更高。目前,已有多个真菌成功应用于真菌天然产物的生物合成途径研究和异源表达生产,如构巢曲霉、米曲霉(A. oryzae)和黑曲霉等(表 2)。
表 2. 在异源宿主中成功表达的真菌天然产物Table 2. Fungal natural products successfully produced in heterologous hosts
| Natural products | Donor organism | Heterologous hosts | References |
| 6-MSA | Penicillium patulum | Saccharomyces cerevisiae | [12] |
| Mycophenolic acid | Penicillium brevicompactum | Aspergillus nidulans | [38] |
| Chanoclavine-I | Aspergillus fumigatus | Aspergillus nidulans | [23] |
| Geodin | Aspergillus terreus | Aspergillus nidulans | [24] |
| Neosartoricin B | Trichophyton tonsurans/Arthroderma otae | Aspergillus nidulans | [8] |
| Citrinin | Monascus purpureus | Aspergillus oryzae | [39] |
| Aspyridone | Aspergillus nidulans | Aspergillus oryzae | [30] |
| Terrequinone A | Aspergillus nidulans | Aspergillus oryzae | [31] |
| Monacolin K | Monascus pilosus | Aspergillus oryzae | [31] |
| Penicillin | Penicillium chrysogenum | Neurospora crassa/Aspergillus niger | [33] |
| Enniatin | Fusarium oxysporum | Aspergillus niger | [36] |
表选项
3.1 构巢曲霉表达系统 构巢曲霉是最早完成基因组测序的模式真菌之一,具有丰富的次级代谢产物产生能力,其分子操作容易,遗传转化技术成熟,具有高效的基因打靶体系,是最先进最有效的分子遗传系 统[20]。构巢曲霉还具有有性生殖和无性生殖阶段,在创立多基因突变株时具有强大的优势,已经应用于其它真菌的次级代谢产物生物合成的研究[8]。
为了构巢曲霉更适合作为一个异源表达的宿主,美国堪萨斯大学的Berl Oakley教授、威斯康辛大学麦迪逊分校的Nancy Keller教授和南加州大学的Clay Wang教授3个课题组联合,对构巢曲霉进行改造,建立了一套有效地快速敲除次级代谢产物基因和完整基因簇的方法。为了减少自身的代谢产物背景的影响,构建一个背景简单的菌株,利于异源表达真菌天然产物的检测和分离,他们构建了包括柄曲霉素(sterigmatocystin)和苔色酸在内的许多代谢产物缺失的突变株[19, 21];还在构巢曲霉中建立了一个转化异源次级代谢产物基因簇的有效流程,该流程是通过fusion PCR的原理将片段插入到特定的带有启动子的载体上,常用的启动子有alcA、gpdA等,由于核心基因较大,将其分为2-3个小的DNA片段,各片段相连处设计携带同源序列,以便在酵母中组装成完整的编码序列且成功重组到特定启动子的下游,再在构巢曲霉中进行异源表达。为了转化之后有效筛选转化子,把边侧序列设计成与构巢曲霉孢子颜色变化基因wA的同源序列,这样可将异源基因或基因簇定点整合到wA基因位点上,wA基因被破坏,将导致重组子孢子颜色变为白色而不是绿色,大大提高了筛选效率。利用该宿主,将土曲霉中合成asperfuranone的基因簇成功表达,不仅实现了asperfuranone生物合成途径的完全转移,也通过对该合成途径中的多种基因组合表达进一步揭示出asperfuranone的合成机制[19]。
到目前为止,许多真菌天然产物借助构巢曲霉表达系统成功产出。如2009年Maiya等将来自烟曲霉的烟曲霉毒素基因簇在构巢曲霉中进行超表达,得到了戊烯化二酮哌嗪类tryprostatin B,产量高达250 mg/L[22];2013年,Yin等利用构巢曲霉成功表达了皮肤癣菌的沉默基因簇,产生免疫抑制活性的聚酮类化合物neosartoricins,此研究为不 能操作致病真菌的实验室研究致病菌代谢物生物合成和实验室生长缓慢真菌的代谢产物研究开辟了一条新的道路[8];Ryan等将烟曲霉中的麦角生物碱前体裸麦角碱-Ⅰ(chanoclavine-I)基因簇部分基因在构巢曲霉中异源表达,获得了裸麦角碱-Ⅰ,此研究为理解麦角生物碱生物合成的前期步骤提供了新的方法[23];Nielsen 等将来自于土曲霉的地曲霉素(geodin)完整的基因簇在构巢曲霉中成功表达[24]。2014年,Unkles等利用酵母组装技术克隆了产黄青霉的青霉素基因簇并成功转化到构巢曲霉中,得到青霉素,实现了复杂天然产物合成途径的完全重构,为通过便利的宿主发现新型次级代谢产物提供了新的方法,同时也为人工构建生物合成途径创造了可能。
3.2 米曲霉表达系统 米曲霉既是传统酿造酱油和酒类的菌种,也是非常适用工业生产的安全菌种。米曲霉能将代谢产物大量分泌到培养基,发酵技术成熟,具有放大到实际生产的优势;其基因组背景清晰,具有剪接内含子和进行翻译后修饰等功能[25]。因此,以米曲霉为真菌天然的异源宿主,既能在实验室中发挥优势,也能快速实现工业生产。
米曲霉已经成功应用于真菌天然产物的异源生产。早在1999年,Fujii等将来自黄瓜炭疽病菌(Colletotrichum lagenarium)中与黑色素(melanin)形成相关的PKS1基因在米曲霉中进行了异源表达,在淀粉诱导型启动子的作用下,获得中间产物T4HN[26]。后来陆续有外源基因在米曲霉中进行表达获得目的化合物的报道,如:将来自烟曲霉的alb1 基因在米曲霉中异源表达出化合物naphthopyrone,并同时证明Alb1p聚酮合酶的功能[27];将炭角菌属真菌(Xylaria sp.) pksmt基因在米曲霉中表达,得到了新的吡喃酮化合物[25];将产黄青霉具有抗肿瘤功效的结构最复杂化合物andrastin A基因在米曲霉中成功表达。米曲霉也可有效表达外源的基因簇,如将两个来自简青霉(P. simplicissimum)的 合成吲哚双萜类化合物青霉震颤素(penitrem)所需的基因簇的基因转化至米曲霉中,得到了其中间体和终产物,成功破解了青霉震颤素合成过程中的13步反应[28];来自不同真菌的卵孢白僵菌素(tenellin)、阿菲迪霉素(aphidicolin)、paxilline和aspyridone等化合物在米曲霉中也已经陆续从头合成表达[29, 30],特别是在米曲霉中异源生产的卵孢白僵菌素及其前体的产量是其天然宿主产量的5倍多,展现出该系统的巨大应用潜力。
为了将米曲霉改造为一个通用型的次级代谢产物异源宿主,2012年,Sakai等在全局调控因子laeA前插入一个组成型启动子pgk,得到超表达laeA的米曲霉菌株,利用改造后的菌株分别超表达了源自红曲霉(Monascus purpureus)的聚酮化合物MK和源自构巢曲霉的氨基酸与类异戊二烯的杂合体TQ两个化合物的合成基因[31]。2014年,Oikawa实验室提出了在米曲霉中快速构建真菌代谢产物合成机制的策略,运用此策略成功将来自覃青霉(P. paxilli)和黄曲霉(A. flavus)的化合物aflatrem生物合成所需的7个基因转化到米曲霉中,获得了化合物aflatrem,完成了aflatrem生物全合成;2015年,同样采用此策略将来自费希新萨托菌(Neosartorya fischeri)和米曲霉的沉默的萜烯合酶基因转化到米曲霉中,通过代谢产物分析,确定出二倍半萜合成酶,结合体内体外同位素标记实验成功解析出二倍半萜的生物合成途 径[32]。可见,将生物合成机制在异源宿主米曲霉中重构,不仅可以确定合成过程中每一步酶促反应,还能确定中间体和最终的天然产物,根据获得的一些有效数据能够确定生物合成途径,从而避免选择用繁琐的基因敲除技术来解析整个合成过程,这种简单快速的方法促进了对真菌天然产物的研究,同时也证明了利用米曲霉异源表达体系是研究真菌天然产物及其合成途径的一个有效可靠的方式[28]。
3.3 其它丝状真菌表达系统 黑曲霉是工业上应用较广的另一种重要菌株,和米曲霉均被认证为安全菌株。1990年,首次在丝状真菌中进行异源表达的真菌天然产物青霉素,所选择的异源宿主之一就是黑曲霉[33]。黑曲霉作为异源宿主生产非核糖体多肽具有极大的工业生产优势,2011年,Meyer等在黑曲霉中构建了一个可调节的系统(Tet-on system),这个系统由2个表达模块组成,一个是组成型表达依赖四环素的反式作用因子rtTA2S-M2,另一个模块包括依赖rtTA2S-M2的启动子,控制目的基因的表达,且该系统不受环境中的碳源和氮源的影响,在非诱导环境下不会表达[34]。2012年,Yeh等将构巢曲霉的micA基因转化到黑曲霉中,证明只有一个非核糖体肽合成酶是合成microperfuranone必需 的[35]。2015年,Meyer等将来自尖孢镰刀菌的非核糖体多肽合成酶基因esyn1在Tet-on系统中表达,得到大约5 g/L的环酯肽恩镰孢菌素[36]。以黑曲霉为异源宿主进行真菌天然产物生产方面目前取得了一定进展,如:2014年,Zaehle等将土曲霉酮聚酮合酶基因在黑曲霉中异源表达,得到了苔 色酸、4-hydroxy-6-methylpyranone和 6,7-dihydroxymellein 3种大小和结构均不相同的产 物[37];2015年利用黑曲霉表达了来自土曲霉的土曲霉酮基因,该基因能够合成转录因子TerR,作用于启动子terA,该研究成功阐明了TerR的作用机制。
此外,粗糙脉孢菌(Neurospora crassa)作为异源表达宿主曾成功表达了来自产黄青霉的青霉素基因[33]。迄今为止,越来越多的真菌天然产物可利用多种异源表达系统来进行生产(表 2)。真菌天然产物在模式宿主中进行异源表达生产将是发掘新化合物、不可培养真菌天然产物生产、进行药物前体或药物生产及探究天然产物生物合成途径等研究的有力武器。
4 真菌大片段基因簇克隆技术由于真菌次级代谢产物的相关基因簇的大小长达十几kb至上百kb,核心基因片段较大,用常规的PCR方法和克隆手段难以直接获得,大片段基因克隆技术曾是真菌天然产物异源表达生产的重要限制性因素。为解决这一难题,一些用于克隆真菌大片段基因的技术陆续发展起来,这些技术用于真菌天然产物合成基因簇的克隆和质粒 构建。
2002年,Larionov等提出的基于转化耦联重组(Transformation-associated recombination,TAR)的酵母重组技术,该技术基本原理是将基因簇等分成若干长度为3 kb左右的DNA片段,相连片段间设计重叠区,利用酵母重组高效的DNA同源重组能力和操作的简便性,在酵母中将各片段依次进行同源重组连接,获得预期的DNA长度。但是该方法也有局限,利用此技术难以对长度超过15 kb DNA片段进行操作。本课题组在此基础上对酵母质粒进行改造,目前已经成功获得30 kb的DNA片段(未发表数据)。2007年Nielsen等发明了USER(Uracil-Specific Excision Reagent)克隆技术,利用特异尿嘧啶切除试剂把引物和载体上设计的尿嘧啶碱基切开,结合内切酶导致DNA片段有9个碱基的缺口,使得目标产物和载体上有9个碱基的互补序列,转化大肠杆菌实现DNA的重组。该技术可实现多个片段的组装,大小可达15 kb。如将来自土曲霉的地曲霉素的基因簇仅用两个分别携带12 kb和15 kb目的基因的载体就将地曲霉素合成途径成功在异源宿主构巢曲霉中重构并表达出地曲霉素[24]。2008年,Gibson等利用体外变温两步组装法得到了1/4大小的支原体基因组,后改进为Gibson等温一步法,其原理是5'核酸外切酶切割DNA 使其产生一定长度的3'单链末端,在Phusion 聚合酶与Taq连接酶介导下进行退火,一次实现多个片段的拼接,实现大片段DNA分子的无缝连接,成功组装各种基因、遗传途径和基因组[40]。目前Gibson DNA组装和Golden Gate组装技术已经实现商业化,NEB公司可以买到商用试剂盒。但是,以上技术都是基于PCR扩增,有可能引入碱基突变,导致后续的表达不 成功。
美国洛克菲勒大学的Sean Brady课题组改进了TAR技术,利用PCR free方法,从全基因组中去抓取目标基因簇,已经实现从环境DNA和宏基因组中挖掘出很多新颖化合物,并解析了其生物合成途径[41, 42]。2015年,Keller实验室提出的真菌人工染色体(Fungal artificial chromosomes,FACs)技术,是新发展起来的克隆大片段基因的技术,此技术在细菌人工染色体的基础上引入大肠杆菌复制子和构巢曲霉的自主复制序列构建而成的,因此能够在大肠杆菌和模式真菌构巢曲霉中穿梭复制,并且能够携带的DNA片段高达150 kb。真菌人工染色体能够快捷地从自然宿主基因组DNA上捕获不同大小的基因,通过对转化子末端测序来鉴定是否含有自然宿主的完整基因簇,将携带完整基因簇的真菌人工染色体在构巢曲霉中表达完成天然产物的异源生产。目前已经利用该方法获得了土曲霉的56个基因簇,在异源宿主构巢曲霉中成功表达出土曲霉的astechrome合成途径的中间体terezine D[43]。
当前研究最热门的是CRISPR/Cas9技术,CRISPR-Cas9系统是起源于细菌的古细菌的RNA-介导的实现精准切割定点敲除的技术,该技术几乎能够作用于任何一个基因,已经应用于细菌、细胞、哺乳动物、人等系统,在丝状真菌也已经初步建立[44, 45]。在丝状真菌中,率先成功应用CRISPR/Cas9系统的是里氏木霉(Trichoderma reesei)。2015年,Liu等将带有SV40核定位信号序列的酿脓链球菌(Streptococcus pyogenes) Cas9基因经过密码子优化,通过农杆菌介导的真菌转化,得到表达Cas9的菌株,体外完成转录后的gRNA (guide RNA)盒通过原生质体转化至Cas9表达菌株中,达到定点突变的目的。同时转化携带与靶基因5'和3'侧翼序列同源的供体DNA,通过同源重组,达到基因置换的目的。研究中已经能够同时转化3个供体DNA,虽然同源重组率仅4.2%,但是能够说明CRISPR/Cas9同时定点插入多基因的可行性[46]。Matsu-ura等以粗糙脉孢菌为底盘,利用CRISPR/Cas9系统成功将clr-2的启动子替换为β微管蛋白启动子,也将荧光素酶基因定点整合到csr-1位点[47]。Nodvig等成功将该系统应用于构巢曲霉、黑曲霉、棘孢曲霉(A. aculeatus)、琉球曲霉(A. luchuensis)、巴西曲霉和炭色曲霉(A. carbonarius)等6种丝状真菌。 在此技术开发的基础上,CATCH(Cas9-Assisted Targeting of CHromosome segments)技术是将CRISPR/Cas9系统与Gibson结合,有效组装可达100 kb的细菌基因组片段[48],为克隆大片段DNA序列提供了新的方法。
上述大片段基因克隆技术的建立为真菌天然产物编码基因簇的克隆和真菌天然产物的异源生产提供了重要技术保障,必将大大加快真菌天然产物异源表达生产的研究进程。
5 展 望海量真菌基因组测序数据的出现及其分析显示真菌中沉默的基因簇数远超已经被发现的天然产物,真菌天然产物仍是一个巨大的宝库,亟待人们去发掘。最近提出的Minimum Information about a Biosynthetic Gene cluster (MIBiG) (http://mibig. secondarymetabolites.org/)数据标准能够帮助整理规范检索和添加生物合成的基因簇,这一数据能够让研究者对基因簇的信息获取更加全面快捷。
不断升级优化的大片段构建和转化技术,让真菌异源表达系统更加适用,如利用CRISPR-Cas9系统进行定点敲除,大大推进利用丝状真菌研究新的天然产物的进程[49]。最近张峰课题组从微生物基因组中发现并鉴定了3个新的CRISPR/CAS 亚系统,与已发现的Cas9和Cpf1系统均不同,具有不同的性能,表明CRISPR/Cas系统在基因编辑方面的作用机制是多样的,作为通用型的基因编辑工具还具有巨大的潜力亟待开发[50, 51]。另一方面,构建一个理想的成熟的真菌底盘用于天然产物的异源表达同样重要。首先需要遗传背景清晰的宿主,并对宿主进行改造,敲除部分基因簇,减少非必需基因的干扰,如自身次级代谢产物的合成基因。其次,要结合前沿分子技术,改进异源表达过程中涉及到的组装及敲除等操作方法。另外,应通过灵活运用宿主中的调节元件和转录因子,对外源基因进行调控,使外源基因簇得以表达并且终产物产量提高。
真菌异源表达系统的不断优化和大片段基因克隆技术的发展有利于对新型天然产物的开发和已知天然产物的应用,不仅能够得到中间产物,阐明沉默基因簇的功能,还可以优化其生产条件,提高产量。从研究方向来看,在酵母基因组中加入来自动物、细菌和植物包括罂粟的基因,最终得到吗啡以及其他天然或者半合成的类似物等,这一研究成果进一步预示着利用合成生物学的理念对天然产物在异源宿主表达生产具有重要的指导作用。除了技术的发展,真菌天然产物合成的时空控制的机制研究也是值得期待的一个重要方向,该方向的突破将会使研究者能够从更深层次来改造优化真菌宿主,去实现高等真核生物如植物来源的重要药物在真菌中的工厂化生产。
参考文献
| [1] | Keller NP. Translating biosynthetic gene clusters into fungal armor and weaponry. Nature Chemical Biology, 2015, 11(9): 671-677. |
| [2] | Keller NP, Turner G, Bennett JW. Fungal secondary metabolism-from biochemistry to genomics. Nature Reviews Microbiology, 2005, 3(12): 937-947. |
| [3] | Yang Y, Zhao H, Barrero RA, Zhang B, Sun G, Wilson IW, Xie F, Walker KD, Parks JW, Bruce R, Guo G, Chen L, Zhang Y, Huang X, Tang Q, Liu H, Bellgard MI, Qiu D, Lai J, Hoffman A. Genome sequencing and analysis of the paclitaxel-producing endophytic fungus Penicillium aurantiogriseum NRRL 62431. BMC Genomics, 2014, 15: 69. |
| [4] | Hoffmeister D, Keller NP. Natural products of filamentous fungi: enzymes, genes, and their regulation. Natural Product Reports, 2007, 24(2): 393-416. |
| [5] | Wiemann P, Keller NP. Strategies for mining fungal natural products. Journal of Industrial Microbiology and Biotechnology, 2014, 41(2): 301-313. |
| [6] | Bauer A, Broenstrupt M. Industrial natural product chemistry for drug discovery and development. Natural Product Reports, 2014, 31(1): 35-60. |
| [7] | Banic S, Lunder M. Additive effect of the combination of griseofulvin and ketoconazole against microsporum-canis in vitro. Mycoses, 1989, 32(9): 487-489. |
| [8] | Yin WB, Chooi YH, Smith AR, Cacho RA, Hu Y, White TC, Tang Y. Discovery of cryptic polyketide metabolites from dermatophytes using heterologous expression in Aspergillus nidulans. ACS Synthetic Biology, 2013, 2(11): 629-634. |
| [9] | Guo CJ, Wang CC. Recent advances in genome mining of secondary metabolites in Aspergillus terreus. Frontiers in Microbiology, 2014, 5: 717. |
| [10] | Li J, Neubauer P. Escherichia coli as a cell factory for heterologous production of nonribosomal peptides and polyketides. New Biotechnology, 2014, 31(6): 579-585. |
| [11] | Lazarus CM, Williams K, Bailey AM. Reconstructing fungal natural product biosynthetic pathways. Natural Product Reports, 2014, 31(10): 1339-1347. |
| [12] | Kealey JT, Liu L, Santi DV, Betlach MC, Barr PJ. Production of a polyketide natural product in nonpolyketide-producing prokaryotic and eukaryotic hosts. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(2): 505-509. |
| [13] | Zobel S, Kumpfmuller J, Sussmuth RD, Schweder T. Bacillus subtilis as heterologous host for the secretory production of the non-ribosomal cyclodepsipeptide enniatin. Applied Microbiology and Biotechnology, 2015, 99(2): 681-691. |
| [14] | Tsunematsu Y, Ishiuchi Ki, Hotta K, Watanabe K. Yeast-based genome mining, production and mechanistic studies of the biosynthesis of fungal polyketide and peptide natural products. Natural Product Reports, 2013, 30(8): 1139-1149. |
| [15] | Xu Y, Zhou T, Zhang S, Espinosa-Artiles P, Wang L, Zhang W, Lin M, Gunatilaka AA, Zhan J, Molnar I. Diversity-oriented combinatorial biosynthesis of benzenediol lactone scaffolds by subunit shuffling of fungal polyketide synthases. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(34): 12354-12359. |
| [16] | Jakubczyk D, Caputi L, Hatsch A, Nielsen CA, Diefenbacher M, Klein J, Molt A, Schroder H, Cheng JZ, Naesby M, O'Connor SE. Discovery and reconstitution of the cycloclavine biosynthetic pathway-enzymatic formation of a cyclopropyl group. Angewandte Chemie (International ed. in English), 2015, 54(17): 5117-5121. |
| [17] | Ro DK, Paradise EM, Ouellet M, Fisher KJ, Newman KL, Ndungu JM, Ho KA, Eachus RA, Ham TS, Kirby J, Chang MCY, Withers ST, Shiba Y, Sarpong R, Keasling JD. Production of the antimalarial drug precursor artemisinic Acid in engineered yeast. Nature, 2006, 440(7086): 940-943. |
| [18] | Lubertozzi D, Keasling JD. Developing Aspergillus as a host for heterologous expression. Biotechnology Advances, 2009, 27(1): 53-75. |
| [19] | Chiang YM, Oakley CE, Ahuja M, Entwistle R, Schultz A, Chang SL, Sung CT, Wang CC, Oakley BR. An efficient system for heterologous expression of secondary metabolite genes in Aspergillus nidulans. Journal of the American Chemical Society, 2013, 135(20): 7720-7731. |
| [20] | Yaegashi J, Oakley BR, Wang CC. Recent advances in genome mining of secondary metabolite biosynthetic gene clusters and the development of heterologous expression systems in Aspergillus nidulans. Journal of Industrial Microbiology & Biotechnology, 2014, 41(2): 433-442. |
| [21] | Chiang YM, Ahuja M, Oakley CE, Entwistle R, Asokan A, Zutz C, Wang CC, Oakley BR. Development of genetic dereplication strains in Aspergillus nidulans results in the discovery of aspercryptin. Angewandte Chemie (International ed. in English), 2015, 54: 1-5. DOI: 10.1002/anie.201507097. |
| [22] | Maiya S, Grundmann A, Li S-M, Turner G. Improved tryprostatin B production by heterologous gene expression in Aspergillus nidulans. Fungal Genetics and Biology, 2009, 46(5): 436-440. |
| [23] | Ryan KL, Moore CT, Panaccione DG. Partial reconstruction of the ergot alkaloid pathway by heterologous gene expression in Aspergillus nidulans. Toxins, 2013, 5(2): 445-455. |
| [24] | Nielsen MT, Nielsen JB, Anyaogu DC, Holm DK, Nielsen KF, Larsen TO, Mortensen UH. Heterologous reconstitution of the intact geodin gene cluster in Aspergillus nidulans through a simple and versatile PCR based approach. PLoS One, 2013, 8(8): e72871. |
| [25] | Punya J, Tachaleat A, Wattanachaisaereekul S, Haritakun R, Boonlarppradab C, Cheevadhanarak S. Functional expression of a foreign gene in Aspergillus oryzae producing new pyrone compounds. Fungal Genetics and Biology, 2013, 50: 55-62. |
| [26] | Fujii I, Mori Y, Watanabe A, Kubo Y, Tsuji G, Ebizuka Y. Heterologous expression and product identification of Colletotrichum lagenarium polyketide synthase encoded by the PKS1 gene involved in melanin biosynthesis. Bioscience Biotechnology and Biochemistry, 1999, 63(8): 1445-1452. |
| [27] | Watanabe A, Fujii I, Tsai HF, Chang YC, Kwon-Chung KJ, Ebizuka Y. Aspergillus fumigatus alb1 encodes naphthopyrone synthase when expressed in Aspergillus oryzae. FEMS Microbiology Letters, 2000, 192(1): 39-44. |
| [28] | Liu C, Tagami K, Minami A, Matsumoto T, Frisvad JC, Suzuki H, Ishikawa J, Gomi K, Oikawa H. Reconstitution of biosynthetic machinery for the synthesis of the highly elaborated indole diterpene penitrem. Angewandte Chemie (International ed. in English), 2015, 54(19): 5748-5752. |
| [29] | Tagami K, Liu C, Minami A, Noike M, Isaka T, Fueki S, Shichijo Y, Toshima H, Gomi K, Dairi T, Oikawa H. Reconstitution of biosynthetic machinery for indole-diterpene paxilline in Aspergillus oryzae. Journal of the American Chemical Society, 2013, 135(4): 1260-1263. |
| [30] | Wasil Z, Pahirulzaman KAK, Butts C, Simpson TJ, Lazarus CM, Cox RJ. One pathway, many compounds: heterologous expression of a fungal biosynthetic pathway reveals its intrinsic potential for diversity. Chemical Science, 2013, 4(10): 3845-3856. |
| [31] | Sakai K, Kinoshita H, Nihira T. Heterologous expression system in Aspergillus oryzae for fungal biosynthetic gene clusters of secondary metabolites. Applied Microbiology and Biotechnology, 2012, 93(5): 2011-2022. |
| [32] | Ye Y, Minami A, Mandi A, Liu C, Taniguchi T, Kuzuyama T, Monde K, Gomi K, Oikawa H. Genome mining for sesterterpenes using bifunctional terpene synthases reveals a unified intermediate of di/sesterterpenes. Journal of the American Chemical Society, 2015, (36): 11846-11853. |
| [33] | Smith DJ, Burnham MKR, Edwards J, Earl AJ, Turner G. Cloning and heterologous expression of the penicillin biosynthetic gene-cluster from Penicillium chrysogenum. Nature Biotechnology, 1990, 8(1): 39-41. |
| [34] | Meyer V, Wanka F, van Gent J, Arentshorst M, van den Hondel CAMJJ, Ram AFJ. Fungal gene expression on demand: an inducible, tunable, and metabolism-independent expression system for Aspergillus niger. Applied and Environmental Microbiology, 2011, 77(9): 2975-2983. |
| [35] | Yeh H-H, Chiang Y-M, Entwistle R, Ahuja M, Lee K-H, Bruno KS, Wu T-K, Oakley BR, Wang CCC. Molecular genetic analysis reveals that a nonribosomal peptide synthetase-like (NRPS-like) gene in Aspergillus nidulans is responsible for microperfuranone biosynthesis. Applied Microbiology and Biotechnology, 2012, 96(3): 739-748. |
| [36] | Meyer V, Fiedler M, Nitsche B, King R. The cell factory Aspergillus enters the big data era: opportunities and challenges for optimising product formation. Advances in Biochemical Engineering/Biotechnology, 2015, 149: 91-132. |
| [37] | Zaehle C, Gressler M, Shelest E, Geib E, Hertweck C, Brock M. Terrein biosynthesis in Aspergillus terreus and its impact on phytotoxicity. Chemistry & Biology, 2014, 21(6): 719-731. |
| [38] | Hansen BG, Mnich E, Nielsen KF, Nielsen JB, Nielsen MT, Mortensen UH, Larsen TO, Patil KR. Involvement of a natural fusion of a cytochrome P450 and a hydrolase in mycophenolic acid biosynthesis. Applied and Environmental Microbiology, 2012, 78(14): 4908-4913. |
| [39] | Sakai K, Kinoshita H, Shimizu T, Nihira T. Construction of a citrinin gene cluster expression system in heterologous Aspergillus oryzae. Journal of Bioscience and Bioengineering, 2008, 106(5): 466-472. |
| [40] | Gibson DG, Young L, Chuang R-Y, Venter JC, Hutchison CA, III, Smith HO. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods, 2009, 6(5): 343-345. |
| [41] | Feng Z, Kim JH, Brady SF. Fluostatins produced by the heterologous expression of a TAR reassembled environmental DNA derived type II PKS gene cluster. Journal of the American Chemical Society, 2010, 132(34): 11902-11903. |
| [42] | Chang FY, Ternei MA, Calle PY, Brady SF. Targeted metagenomics: finding rare tryptophan dimer natural products in the environment. Journal of the American Chemical Society, 2015,137(18): 6044-6052. |
| [43] | Bok JW, Ye R, Clevenger KD, Mead D, Wagner M, Krerowicz A, Albright JC, Goering AW, Thomas PM, Kelleher NL, Keller NP, Wu CC. Fungal artificial chromosomes for mining of the fungal secondary metabolome. BMC Genomics, 2015, 16: 343. |
| [44] | Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F. Genome engineering using the CRISPR-Cas9 system. Nature Protocols, 2013, 8(11): 2281-2308. |
| [45] | Nodvig CS, Nielsen JB, Kogle ME, Mortensen UH. A CRISPR-Cas9 system for genetic engineering of filamentous fungi. PLoS One, 2015, 10(7) : e0133085. |
| [46] | Liu R, Chen L, Jiang Y, Zhou Z, Zou G. Efficient genome editing in filamentous fungus Trichoderma reesei using the CRISPR-Cas9 system. Cell Discovery, 2015, 1: 15007. |
| [47] | Matsu-ura T, Baek M, Kwon J, Hong C. Efficient gene editing in Neurospora crassa with CRISPR technology. Fungal Biology and Biotechnology, 2015, 2: 4. |
| [48] | Jiang W, Zhao X, Gabrieli T, Lou C, Ebenstein Y, Zhu TF. Cas9-assisted targeting of chromosome segments CATCH enables one-step targeted cloning of large gene clusters. Nature Communications, 2015, 6: 8101. |
| [49] | Doudna JA, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9. Science (New York, N.Y.), 2014, 346(6213): 1258096. |
| [50] | Shmakov S, Abudayyeh Omar O, Makarova Kira S, Wolf Yuri I, Gootenberg Jonathan S, Semenova E, Minakhin L, Joung J, Konermann S, Severinov K, Zhang F, Koonin Eugene V. Discovery and functional characterization of diverse class 2 CRISPR-Cas systems. Molecular Cell, 2015, 60(3): 385-397. |
| [51] | Zetsche B, Gootenberg JS, Abudayyeh, Slaymaker IM, Makarova KS, Essletzbichler P, Volz SE, Joung J, van der Oost J, Regev A, Koonin EV, Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 2015,63(3): 759-771. |
