刘佳佳,刘钢


中国科学院微生物研究所,真菌学国家重点实验室,北京 100101
收稿日期:2015-11-09;修回日期:2015-12-28;网络出版日期:2015-12-31
基金项目:国家自然科学基金(31470177)
通信作者:Tel: +86-10-64807892; Fax: +86-10-64806017; E-mail: liug@im.ac.cn
摘要: 头孢菌素C由丝状真菌顶头孢霉产生,属于β-内酰胺类抗生素。其经改造后的7-氨基头孢烷酸是头孢类抗生素的重要中间体。头孢类抗生素在国内外抗生素市场中占有巨大的份额,是临床上的主要抗感染药物。随着分子生物学的发展,头孢菌素C的生物合成途径已基本阐明。为提高头孢菌素C的产量和降低生产成本,越来越多的研究者开始关注其较为精细、复杂的调控机制。本文重点对头孢菌素C生物合成及其调控机制的最新进展进行了简述,希望为今后头孢菌素C生产菌株的菌种改造和传统产业的升级换代提供一定的借鉴。
关键词: 顶头孢霉头孢菌素C生物合成调控机制
Advances in the regulation of cephalosporin C biosynthesis - A review
Jiajia Liu,Gang Liu


State Key Laboratory of Mycology, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
Received: 9 November 2015; Revised: 28 December 2015; Published online: 31 December 2015
Supported by the National Natural Science Foundation of China (31470177)
Corresponding author. Tel: +86-10-64807892; Fax: +86-10-64806017; E-mail: liug@im.ac.cn
Abstract:The beta-lactam antibiotic cephalosporin C is produced industrially by Acremonium chrysogenum. Its derivative 7-aminocephalosporanic acid (7-ACA) is the intermediate of most chemical modification cephalosporins that are the most frequently used antibiotics for the therapy of infectious diseases. Due to its importance, the biosynthetic pathway of cephalosporin C has been elucidated in Acremonium chrysogenum. To improve the yield of cephalosporin C and reduce the cost of production, recent studies have been focused on the sophisticated regulation of cephalosporin C biosynthesis. In this review, recent advances in cephalosporin C biosynthesis and regulation are summarized.
Key words: Acremonium chrysogenumcephalosporin Cbiosynthesisregulation
头孢菌素C (简称CPC)是由顶头孢霉(Acremonium chrysogenum)产生的一种β-内酰胺类抗生素[1]。顶头孢霉是上世纪40年代由Brotzu教授在意大利撒丁岛附近海岸分离获得的丝状真菌,并首次描述了顶头孢霉能够产生具有抑菌作用的物质。随后有研究者在其培养液中发现了抗菌物质头孢菌素C (CPC)。1961年CPC结构被Abraham 和Newton解析,并通过X-晶体衍射得到了证实。结构显示头孢菌素C用二氢噻嗪环代替了青霉素中的噻唑环与β-内酰胺环相连,正是由于这种结构上的差异使得头孢菌素C有着更强的稳定性,不易被青霉素酶破坏,能够杀死许多青霉素耐药菌[2]。另外,头孢菌素C的不良反应发生率低,对人、畜较为安全。CPC经化学改造后的7-氨基头孢烷酸(简称7-ACA)是头孢类抗生素,例如头孢噻吩、头孢氨苄等的重要中间体。头孢类抗生素自发现以来一直作为重要的抗感染药物而广泛应用于临床治疗。
来源于CPC的第一代头孢类药物于1964年进入市场,目前已经发展到了第五代。尽管我国也已实现了CPC的产业化,但其发酵水平仍与国外先进水平存在较大差距,而工业生产菌株也均来自比较局限的传统诱变育种[3]。阐明CPC生物合成过程中的分子调控机制,降低生产成本,提高发酵水平已成为本领域国内外关注的热点。本文综述了近年来CPC生物合成及其调控的最新研究进展,旨在从生物学角度为CPC的产业化之路提供新的视角。
1 CPC的生物合成由于CPC在临床上的重要作用,CPC生物合成的研究备受关注。经过几十年的研究,CPC的生物合成途径已基本阐明[4]。同大多数微生物中参与次级代谢产物生物合成的基因相似,参与CPC生物合成的基因也成簇存在于顶头孢霉的染色体上。在顶头孢霉中参与CPC合成的基因分别位于VII号染色体上的“早期”基因簇和位于I号染色体上的“晚期”基因簇。“早期”基因簇中的基因pcbAB和pcbC编码的蛋白(ACV合酶和异青霉素N合酶)分别负责CPC合成的前两步反应:在ACV合酶(ACVS)催化下,L-α-氨基己二酸(L-α-aminoadipic acid)、L-半胱氨酸(L-cysteine)和L-缬氨酸(L-valine)缩合为三肽的ACV,再经异青霉素N合酶(IPN)催化形成异青霉素N(IPN)。cefD1和cefD2位于“早期”基因簇中pcbC的下游,编码青霉素N异构化酶,负责将异青霉素N异构化为青霉素N(PenN)。最后两步反应由“晚期”基因簇中的基因cefEF和cefG编码的扩环酶-羧化酶和DAC乙酰转移酶负责,最终将青霉素N催化为CPC(图 1)。
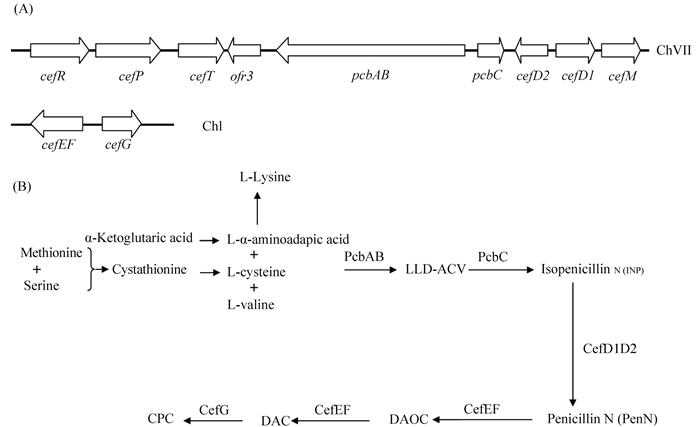 |
| 图 1. 参与头孢菌素C生物合成的基因排布(A)及其生物合成途径(B) Figure 1. The organization of cephalosporin C biosynthetic genes (A) and its biosynthetic pathway (B) in Acremonium chrysogenum. The CPC biosynthetic genes are clustered in two loci of chromosome. The early cluster is located at chromosome VII and the late cluster is located at chromosome I. The coding regions of these genes are indicated by arrows. LLD-ACV, L-α-aminoadipyl-L-cysteinyl-D-valine; DAOC, deacetoxycephalosporin C; DAC, deacetylcephalosporin C; CPC, cephalosporin C. |
| 图选项 |
次级代谢产物的形成会对细胞造成一定的负担,因而产生菌往往会通过一些转运蛋白将次级代谢产物运输出细胞。真菌中,ATP-结合转运蛋白(ATP-binding cassette,ABC)和主要协助转运蛋白超家族(major facilitator superfamily,MFS)在次级代谢产物及其中间代谢产物分泌中起主要运输作用。CPC的产生过程中,也涉及到了一系列的前体物及终产物的转运(图 2)。研究发现,CPC“早期”基因簇中的cefT基因,编码了一种MFS家族膜转运蛋白,增加cefT基因的拷贝数能够显著增加CPC的产量[5]。随后的研究显示,cefT编码一种亲水蛋白,参与了异青霉素N、青霉素N和脱乙酰头孢菌素C的分泌。最近,Teijeira等[6]发现,在CPC“早期”基因簇中cefD1的下游还存在另一个编码MFS家族蛋白的cefM基因,其编码产物含有12个跨膜区,也参与了CPC的产生。在cefM敲除突变株中,CPC产量大幅下降,但是细胞内的青霉素N含量则比野生型菌株高出了7倍。进一步研究发现异青霉素N异构化为青霉素N的过程发生在过氧化物酶体中,cefM参与了青霉素N从过氧化物酶体转运到胞质的过程。有研究显示在CPC“早期”基因簇中,还存在第三个编码膜转运蛋白的基因cefP,其编码产物含有11个跨膜区。cefP敲除突变株中不再有CPC生成,而是积累了大量的异青霉素N,表明cefP参与了异青霉素N异构化为青霉素N的过程[7]。有趣的是,Teijeira等[8]发现,cefP突变株必须由cefP和cefR共同回补才能恢复菌株的野生表型。cefR是位于cefP上游的一个调控基因,在cefR的敲除突变株中,cefEF转录延迟,青霉素N分泌量增加而CPC产量减少,过表达cefR则相反,转录结果表明cefR通过充当基因cefT的抑制子来调控CPC的生物合成。
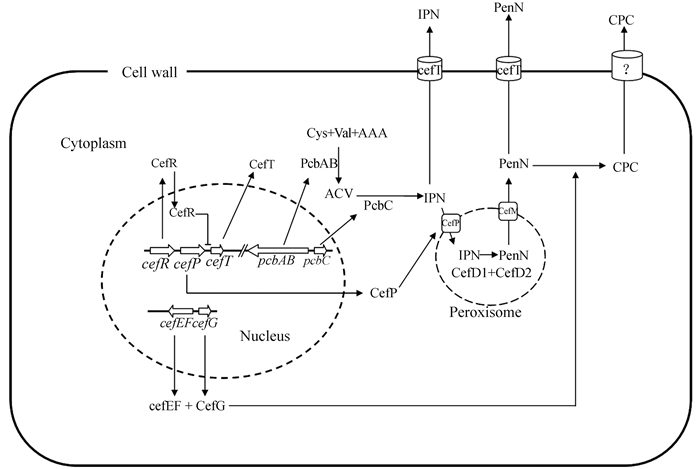 |
| 图 2. 头孢菌素C的生物合成及细胞内运输 Figure 2. Transport of cephalosporin C and its intermediates in A. chrysogenum. The localization of the CefT, CefM, and CefP transporters is indicated. Cys, L-cysteine, Val, L-valine; AAA, L-α-aminoadipic acid; ACV, L-α-aminoadipyl-L-cysteinyl-D-valine; IPN, isopenicillin N; PenN, penicillin N; CPC, cephalosporin C. The transcription of cefT is repressed by CefR. |
| 图选项 |
2 CPC生物合成的调控尽管CPC的生物合成途径已被基本阐明,但是人们对CPC合成过程中复杂的调控机制却知之甚少。原因可能有:(1)和其它丝状真菌相比,顶头孢霉的遗传操作更加困难。顶头孢霉属于半知菌门真菌,生活周期中缺乏有性生殖阶段,因此不能对其进行传统的遗传分析。目前在顶头孢霉中进行的基因功能研究主要依赖于同源重组策略的基因敲除或阻断;(2)顶头孢霉所产的无性孢子较少,因此DNA介导的遗传转化相对困难,并且其发生同源双交换频率很低,进行基因敲除时工作量大;(3)丝状真菌的次级代谢受控于十分复杂的调控网络。
2.1 碳源调控早期用于顶头孢霉CPC产生的发酵培养基中都含有27 g/L的葡萄糖和36 g/L的蔗糖,分别用于保证菌体生长和CPC的合成。在这种培养基中,顶头孢霉对不同碳源的利用有一定的偏好性,蔗糖只是在葡萄糖耗尽之后才开始被利用。同时,发酵过程中CPC只有在顶头孢霉停止生长后才开始产生。此外,工业生产中发现发酵40 h时顶头孢霉菌体停止生长,此后当每隔12 h向培养基中添加1 g/L葡萄糖,或者单独一次性补加20 g/L蔗糖,均可以获得较高产量的CPC。然而,连续不断地将培养基中葡萄糖的浓度补充至8 g/L时就会严重阻碍CPC的生成。在葡萄糖受限的连续培养实验中发现,葡萄糖稀释速率与抗生素产量呈负相关,当葡萄糖稀释速率最低时抗生素产量最高[9]。在发酵培养基中添加不同的碳源对CPC产量的影响也有着显著的差别,提供快速生长的碳源往往会抑制抗生素的生成,且添加的碳源浓度越高,CPC产量越低。速效碳源(如葡萄糖、麦芽糖和果糖等)比迟效碳源(如半乳糖和蔗糖等)更能促进顶头孢霉的生长,但会对抗生素的合成产生强烈抑制。进一步的研究结果表明顶头孢霉在速效碳源下生长对CPC合成产生的负作用主要是因为以下几点:(1) 碳源和氮源代谢产物抑制ACV合酶活性;(2) 速效碳源导致ACV合酶提前降解;(3) 青霉素N转变为头孢菌素之间的酶受到抑制[4]。
在顶头孢霉发酵过程中,抑制扩环酶CefEF和环化酶PcbC的活性会严重影响CPC的产生,速效碳源的添加对环化酶PcbC的影响较大,而对扩环酶CefEF的抑制作用较小[10]。研究还发现在顶头孢霉野生型菌株(ATCC 14553)发酵中添加葡萄糖会导致pcbC 和cefEF的转录量显著降低,同时CPC产量也下降5倍;工业生产菌株(A3/2)在添加速效碳源时抗生素产量也会降低5倍,同时cefEF转录降低,而pcbC转录不变,pcbC和cefEF的翻译以及cefG的转录都不受影响,同时A3/2中pcbC、cefEF和cefG的转录明显高于野生型菌株[11]。有研究显示顶头孢霉含有一种葡萄糖依赖型抑制蛋白CRE1 (Repressor protein CRE1),由基因cre1编码。而在pcbC和cefEF的启动子区含有多个CRE1结合位点;在添加葡萄糖的培养基中,CRE1能够通过直接与启动子区域的结合抑制CPC合成基因pcbC和cefEF的转录,而生产菌株中只有cefEF受到抑制[12]。另外,Shin等[13]发现顶头孢霉发酵中添加甘油可以显著促进CPC的生成,同时pcbC和cefT的转录也随之增加,而且菌丝更倾向于形成节孢子。顶头孢霉发酵后期产生的乙酰水解酶可以降解CPC,降低抗生素产量。乙酰水解酶受葡萄糖、麦芽糖和蔗糖的抑制,而不受甘油和琥珀酸盐的影响,这也是在蔗糖消耗殆尽时需要及时终止发酵的原因。
2.2 氮源调控在顶头孢霉中,高浓度铵盐(NH4+)会强烈的抑制抗生素的产生,因此发酵培养基中更适合以L-天门冬酰胺和L-精氨酸作为氮源。实验发现100 mmol/L以上的铵盐就可以完全阻断CPC的产生。研究表明NH4+可以显著抑制CPC合成中扩环酶的活性,在肉汤培养基发酵过程中,NH4+浓度始终处于较高水平,导致CPC产量较低;在其中加入铵捕获剂磷酸三镁后,扩环酶活性明显增强,CPC产量也随之提高[14]。为阐明铵盐在CPC合成中的调控机制,本课题组在顶头孢霉中克隆到一个氮源调控基因AcareA,AcareA基因缺失导致菌株无法利用包括NO3-、尿酸及多种氨基酸在内的氮源,并在含低浓度NH4+、谷氨酰胺和尿素的培养基上表现出生长缺陷。同时,AcareA突变株中CPC合成相关基因的转录量也明显降低。AcareA编码的蛋白具有典型的Cys2/Cys2锌指结构域,凝胶阻滞实验进一步表明,AcAreA能够直接结合于pcbAB与pcbC之间的双向启动子区[15]。说明一些参与顶头孢霉初级代谢的调控因子对于CPC的生物合成也具有调控作用,这些调控因子很可能在初级代谢和次级代谢的交互调控中发挥关键作用。
2.3 赖氨酸参与的调控CPC主骨架的三肽来源于L-α-氨基己二酸、L-半胱氨酸和L-缬氨酸的缩合。在真菌中,L-α-氨基己二酸(L-α-AAA)是L-赖氨酸形成的前体物。L-赖氨酸由α-酮戊二酸经由L-α-AAA通路形成。在顶头孢霉中高浓度的赖氨酸能够反馈抑制初级代谢和次级代谢产物生物合成共同途径中的高柠檬酸合酶,从而抑制L-α-AAA的形成,并进一步抑制CPC的产生。在产黄青霉(Penicillium chrysogenum)中,L-α-AAA的含量与青霉素 G产量成正比,而L-α-AAA还原酶与青霉素产生有重要联系[16]。Hijarrubia等[17]在顶头孢霉中克隆得到lys2基因,可异源互补产黄青霉中L-α-AAA还原酶基因的缺陷,同时发现该基因编码的L-α-AAA还原酶只在NADPH存在时才有活性,且L-α-AAA还原酶的活性的高低与CPC的产生正好相反。
2.4 甲硫氨酸参与的调控长期以来,甲硫氨酸一直被当作头孢菌素C产生的促进剂用于工业化生产,然而其原因并不清楚[18]。在CPC产生之前,顶头孢霉内源性的甲硫氨酸浓度会瞬时增加2-3倍。尽管甲硫氨酸对于青霉素N和CPC的产生有着显著的促进作用,然而甲硫氨酸并不是顶头孢霉生长所必须的氨基酸,但是却可以充当唯一的氮源或者硫源,其中D-型甲硫氨酸比L-型更能显著促进CPC的产生。细胞间的氨基酸氧化酶类对甲硫氨酸有着一定的降解作用,因此工业生产中添加的甲硫氨酸浓度往往较高,而D-型甲硫氨酸相较于L-型甲硫氨酸则较难被氨基酸氧化酶分解。研究发现在青霉素N形成过程中,同型半胱氨酸和半胱氨酸均不能代替甲硫氨酸。D-型甲硫氨酸并不是β-内酰胺环中D-α-氨基己二酸侧链的前体,而其中的硫却是CPC中硫原子的有效来源。但是,提供硫原子并不是甲硫氨酸在CPC生物合成中的根本作用所在。最早提出甲硫氨酸作为唯一硫源是因为其他含硫复合物替代甲硫氨酸时,使得中间代谢物,例如同型半胱氨酸、胱硫醚和半胱氨酸的活性变低。另外,只有在生长期即刚开始生成CPC时,添加甲硫氨酸才会对CPC合成发挥作用。更有研究显示,在硫源特定的培养基中,顶头孢霉可以利用非硫同源物正亮氨酸代替甲硫氨酸发挥促进CPC合成的效应。和甲硫氨酸一样,正亮氨酸的两种构型均有活性,且D型更强。由于正亮氨酸不含硫,显而易见甲硫氨酸对CPC合成的促进作用不是因为提供了硫原子。研究表明,顶头孢霉发酵过程中添加甲硫氨酸可以刺激ACVS、环化酶和扩环酶的表达。Velasco等发现,甲硫氨酸也可以在转录水平调节CPC的生成,CPC合成相关的基因pcbAB、pcbC和cefEF在甲硫氨酸作用下转录均大幅提高。因此,甲硫氨酸对CPC合成的调控作用主要包括两个方面:(1) 诱导环化酶、扩环酶和ACVS的表达;(2) 为合成CPC所需的半胱氨酸提供硫原子。
此外,顶头孢霉在敲除编码胱硫醚-γ-裂解酶的基因mecB(反转硫途径必须基因)后,在不含甲硫氨酸的培养基中菌体均能正常生长且CPC不再减少,而添加甲硫氨酸后突变株生长受到抑制[19]。Kosalkova等[20]发现在顶头孢霉中高表达mecB后,胱硫醚-γ-裂解酶活性增高2–4倍时,且在含甲硫氨酸的培养基中CPC产量增加了10%-40%,而当该酶增强6倍时,菌体生长非常缓慢且几乎不产抗生素。近年来我们课题组也做了相关研究,发现甲硫氨酸在维持细胞内氧化平衡方面具有重要作用。谷胱甘肽还原酶基因glrA控制了顶头孢霉体内的还原型和氧化型谷胱甘肽的平衡,该基因的缺失增加了顶头孢霉对氧化胁迫的敏感性并使得CPC的合成完全依赖于甲硫氨酸[21]。而另一参与氧化还原的硫氧还蛋白还原酶基因ActrxR1的敲除增强了pcbC、cefEF和cefG的转录水平,使CPC的合成提高近100% [22]。说明不同的氧化还原系统对CPC合成的影响具有不同的机制。然而,目前我们对甲硫氨酸在头孢菌素产生中的促进机制还缺乏深入的了解。
2.5 形态分化与次级代谢的关系丝状真菌的形态分化常与其次级代谢产物的合成具有紧密的联系[23]。顶头孢霉在培养过程中,能产生两种形态的无性孢子—分生孢子和节孢子。分生孢子的形成与其它丝状真菌一致,源于固体平板培养时菌丝分枝末端分化,而节孢子是在液体发酵过程中形成的一种菌丝膨大形态,也称为“酵母”状细胞。发酵过程中营养缺乏或添加甲硫氨酸时,顶头孢霉菌丝形态开始转变,产生节孢子,CPC也开始大量合成,暗示节孢子形成与CPC产生密切相关。Nash等[24]发现当顶头孢霉菌丝分化为节孢子或酵母状细胞时CPC产量最高。另外有研究显示,在含硫酸盐的培养基中顶头孢霉菌丝一般较细,添加半胱氨酸、同型半胱氨酸和胱硫醚时菌丝也呈现细丝状,但是添加甲硫氨酸后,菌丝会膨大形成节孢子,且片段化增加,而这一现象与含甲硫氨酸发酵培养时,开始产生抗生素时菌丝的形态改变一致[25]。工业生产中,也往往利用形成节孢子的现象来判断CPC产量的高低。针对这一现象,目前还没有明确的机制可以解释。我们课题组近期对顶头孢霉分化相关的基因进行了研究。我们利用T-DNA随机插入突变的方法,获得了一系列与形态分化相关的基因。其中,一个和菌丝分隔相关的基因AcsepH的突变导致分生孢子产量下降9倍,而且20%的分生孢子中含有多个细胞核,CPC的产量在突变株中也大幅下降,仅约为野生型的1/10。与其相对应的是pcbC基因转录延迟,cefEF、cefD1和cefD2的转录量明显降低[26]。对另外一个顶头孢霉中与分生孢子产生相关的调控基因AcstuA的研究,发现其可以在转录水平上调控CPC的合成,AcstuA还参与了调控细胞壁的完整性,在AcstuA缺失突变株中,菌丝均呈现不规则膨胀和断裂,而培养基中添加NaCl可以部分修复细胞壁的缺损[27]。对顶头孢霉形态分化与CPC合成之间关系的研究,将加深我们对顶头孢霉中头孢菌素产生的理解。
2.6 分子调控在真菌中参与次级代谢产物生物合成的基因通常成簇存在。真菌次级代谢产物生物合成基因簇往往存在相关的调控基因,这些调控基因编码产物在不同水平上特异地调控簇内相关结构基因、后修饰蛋白编码基因以及转运蛋白编码基因的表达,一些调控基因编码产物也可以作用于簇外基因。在CPC生物合成基因簇中,除最新发现的cefR外,尚未发现其它的调控蛋白编码基因。对CPC生物合成调控的理解主要建立在对一些参与调控生理过程的全局性调控因子的研究上(图 3)。在丝状真菌中,这些全局性调控因子往往指导次级代谢产物的生物合成[28]。在顶头孢霉中,除糖代谢抑制子CRE1外,参与CPC合成调控的还包括PACC、CPCR1等其他调控因子。PH依赖型锌指转录因子PACC参与调控了CPC合成相关基因的转录,PACC与CPC生物合成基因启动子有4个结合位点,2个位于pcbAB-pcbC双向启动子处,另外2个位于cefEF-cefG启动子处[29]。Schmitt和Kück[30]发现顶头孢霉中存在着一种与人类转录调控因子X(RFX)家族同源的蛋白,称之为CPCR1蛋白,与RFX相同,CPCR1的识别位点也是一段不完全的回文序列。随后他们又证明CPCR1至少识别并结合pcbAB和pcbC基因间区的两段序列。利用lacZ作为报告基因,发现当pcbC启动子区的2个CPCR1识别位点都缺失时,报告基因活性大幅下降;另外,在CPCR1敲除株中pcbC转录大幅下降,中间物青霉素N产量也相应减少,但是CPC产量却无变化,CPCR1的高表达和遗传互补也进一步证实了该结果[31],由此可以看出,CPC的生物合成调控是一个较为复杂、精细的过程。此后,研究发现了顶头孢霉中第一个已知靶基因的叉头状转录因子AcFKH1,它具有2个保守区,N-末端叉头相关区(FHA)和C-末端DNA结合区,另外在体外也证实了AcFKH1可以与CPCR1相互作用[31, 32]。Hoff等[33]通过敲除和过表达CPCR1和Acfkh1发现,在顶头孢霉中CPCR1可以控制菌丝片段化以及节孢子的形成,而Acfkh1并不直接参与此过程,在Acfkh1突变株中高表达CPCR1则不能影响节孢子的形成,这说明在顶头孢霉中CPCR1与Acfkh1的相互作用与CPCR1的功能密切相关。Dreyer等[34]在顶头孢霉中发现了构巢曲霉(Aspergillus nidulans)veA同源基因,命名为AcveA。AcveA编码一个全局性调控因子,主要调控CPC生物合成基因的转录水平。在AcveA敲除突变株中cefEF转录大幅下降,CPC产量降低了80%。AcveA过表达实验也证实了这一现象,在AcveA过表达菌株中CPC产量明显上升,CPC合成限速步骤中的早期基因pcbC、晚期基因cefEF以及cefG基因的转录量均大幅提高[35]。
 |
| 图 3. 头孢菌素C生物合成的分子调控 Figure 3. Molecular regulation of cephalosporin C biosynthesis. The positive regulations are shown by arrows, the negative regulations are shown by bars. PACC, pH-dependent regulator; CRE1, glucose repressor; AcAreA, nitrogen regulator; CPCR1, cephalosporin C regulator 1; AcStuA, APSES transcription regulator. |
| 图选项 |
此外,CPC的生物合成同丝状真菌其他次级代谢产物的生物合成一样,还受到温度、pH、光照、活性氧等因素的影响。例如,过量的磷酸盐会减少顶头孢霉CPC生物合成通路中的物质流,从而抑制CPC的产量,同时ACVS、环化酶和扩环酶的活性也受到严重抑制[36]。研究表明,50 mmol/L的磷酸盐对扩环酶抑制率为60%,对ACVS和环化酶抑制率分别为50%和40%。CPC的产生是一个极其耗氧的过程,在这一过程中势必产生大量的活性氧自由基(ROS)。在动植物及酵母菌中ROS会诱导自噬的发生。我们课题组发现,顶头孢霉中存在自噬现象以及和自噬相关的一系列基因。其中,Acatg1基因的缺失在阻断了自噬正常发生的同时,通过稳定PcbC蛋白的持续性表达显著提升了CPC的产量[37]。然而目前人们对丝状真菌自噬的认识还相当有限,对自噬这一真核生物中的主要生理过程在次级代谢产物合成中的调控机制还缺乏理解。
3 展望头孢类抗生素因其较好稳定性和较低毒性,在抗生素药物市场中一直占据着主要的地位。CPC作为头孢类抗生素前体7-ACA的主要来源,其发酵水平直接决定了抗生素药物的价格。CPC的产生是一个极其复杂的过程,涉及到细胞感受外界信号,调控基因的表达,生物合成基因/基因簇的转录激活和翻译表达,一系列酶促反应最终生成终产物并借助于转运蛋白将其分泌到胞外等过程。目前,顶头孢霉中CPC的生物合成途径已基本阐明,但是其生物合成的调控机制还知之甚少。随着基因组测序技术的发展,顶头孢霉基因组测序的完成[38]为分析其调控机制提供了新的视角,比较一些高产菌株的基因组以及转录组[39]等工作必将加深和完善我们对头孢菌素产生的认识。随着我们对顶头孢霉中头孢菌素C产生调控机理认识的不断深入,我们的视野将不再仅仅局限于与头孢菌素C生物合成直接相关的因素以及调控因子上,我们也将更加关注与头孢菌素C合成相关的前体物的形成以及顶头孢霉在产生头孢菌素C过程中的初级代谢和生理变化等各方面,这将有利于提升我们对这一重要的抗生素工业生产菌株的全面了解。此外,顶头孢霉遗传操作系统的逐渐成熟,以及现代生物技术的快速发展也为头孢菌素C生物合成的调控研究提供了便利条件。例如,CRISPRs/CAS9技术的开发以及在丝状真菌的引入[40],使一步敲除顶头孢霉中多个基因成为可能,为从全局水平研究头孢菌素C生物合成的调控提供了条件,为全面改造头孢菌素生产菌株奠定了技术基础。
参考文献
| [1] | Abraham E. Selective reminiscences of β-lactam antibiotics: early research on penicillin and cephalosporins. BioEssays, 1990, 12(12): 601-606. |
| [2] | Martín JF, Ullán RV, García-Estrada C. Regulation and compartmentalization of β-lactam biosynthesis. Microbiology Biotechnology, 2010, 3(3): 285-299. |
| [3] | Hu YJ, Zhu BQ. Research progress on strain improvement of Acremonium chrysogenum by genetic engineering. Hereditas, 2011, 33(10): 1079-1086. (in Chinese)胡又佳, 朱宝泉. 顶头孢霉遗传育种研究进展. 遗传, 2011, 33(10): 1079-1086. |
| [4] | Ozcengiz G, Demain AL. Recent advances in the biosynthesis of penicillins, cephalosporins and clavams and its regulation. Biotechnology Advances, 2013, 31(2): 287-311. |
| [5] | Ullán RV, Liu G, Casqueiro J, Gutiérrez S, Bañuelos O, Martín JF. The cefT gene of Acremonium chrysogenum C10 encodes a putative multidrug efflux pump protein that significantly increases cephalosporin C production. Molecular Genetics and Genomics, 2002, 267(5): 673-683. |
| [6] | Teijeira F, Ullán RV, Guerra SM, García-Estrada C, Vaca I, Martín JF. The transporter CefM involved in translocation of biosynthetic intermediates is essential for cephalosporin production. Biochemical Journal, 2009, 418(1): 113-124. |
| [7] | Ullán RV, Teijeira F, Guerra SM, Vaca I, Martín JF. Characterization of a novel peroxisome membrane protein essential for conversion of isopenicillin N into cephalosporin C. Biochemical Journal, 2010, 432(2): 227-236. |
| [8] | Teijeira F, Ullán RV, Fernández-Aguado M, Martín JF. CefR modulates transporters of beta-lactam intermediates preventing the loss of penicillins to the broth and increases cephalosporin production in Acremonium chrysogenum. Metabolic Engineering, 2011, 13(5): 532-543. |
| [9] | Behmer CJ, Demain AL. Further studies on carbon catabolite regulation of β-lactam antibiotic synthesis in Cephalosporium acremonium. Current Microbiology, 1983, 8(2): 107-114. |
| [10] | Jekosch K, Kück U. Glucose dependent transcriptional expression of the cre1 gene in Acremonium chrysogenum strains showing different levels of cephalosporin C production. Current Genetics, 2000, 37(6): 388-395. |
| [11] | Jekosch K, Kück U. Loss of glucose repression in an Acremonium chrysogenum β-lactam producer strain and its restoration by multiple copies of the cre1 gene. Applied Microbiology and Biotechnology, 2000, 54(4): 556-563. |
| [12] | Jekosch K, Hortschansky P, Kück U. Identification of a minimal cre1 promoter sequence promoting glucose-dependent gene expression in the β-lactam producer Acremonium chrysogenum. Current Genetics, 2008, 53(1): 35-48. |
| [13] | Shin HY, Lee JY, Park C, Kim SW. Utilization of glycerol as cysteine and carbon sources for cephalosporin C production by Acremonium chrysogenum M35 in methionine-unsupplemented culture. Journal of Biotechnology, 2011, 151(4): 363-368. |
| [14] | Shen YQ, Heim J, Solomon NA, Wolfe S, Demain AL. Repression of β-lactam production in Cephalosporium acremonium by nitrogen sources. Journal of Antibiotics, 1984, 37(5): 503-511. |
| [15] | Li JY, Pan YY, Liu G. Disruption of the nitrogen regulatory gene AcareA in Acremonium chrysogenum leads to reduction of cephalosporin production and repression of nitrogen metabolism. Fungal Genetics and Biology, 2013, 61: 69-79. |
| [16] | Esmahan C, Alvarez E, Montenegro E, Martin JF. Catabolism of lysine in Penicillium chrysogenum leads to formation of 2-aminoadipic acid, a precursor of penicillin biosynthesis. Applied and Environmental Microbiology, 1994, 60(6): 1705-1710. |
| [17] | Hijarrubia MJ, Aparicio JF, Casqueiro J, Martín JF. Characterization of the lys2 gene of Acremonium chrysogenum encoding a functional α-aminoadipate activating and reducing enzyme. Molecular and General Genetics, 2001, 264(6): 755-762. |
| [18] | Demain AL, Zhang JY. Cephalosporin C production by Cephalosporium acremonium: the methionine story. Critical Reviews in Biotechnology, 1998, 18(4): 283-294. |
| [19] | Liu G, Casqueiro J, Bañuelos O, Cardoza RE, Gutiérrez S, Martín JF. Targeted inactivation of the mecB gene, encoding cystathionine-γ-lyase, shows that the reverse transsulfuration pathway is required for high-level cephalosporin biosynthesis in Acremonium chrysogenum C10 but not for methionine induction of the cephalosporin genes. Journal of Bacteriology, 2001, 183(5): 1765-1772. |
| [20] | Kosalková K, Marcos AT, Martín JF. A moderate amplification of the mecB gene encoding cystathionine-γ-lyase stimulates cephalosporin biosynthesis in Acremonium chrysogenum. Journal of Industrial Microbiology & Biotechnology, 2001, 27(4): 252-258. |
| [21] | Long LK, Yang J, An Y, Liu G. Disruption of a glutathione reductase encoding gene in Acremonium chrysogenum leads to reduction of its growth, cephalosporin production and antioxidative ability which is recovered by exogenous methionine. Fungal Genetics and Biology, 2012, 49(2): 114-122. |
| [22] | Liu L, Long LK, An Y, Yang J, Xu XX, Hu CH, Liu G. The thioredoxin reductase-encoding gene ActrxR1 is involved in the cephalosporin C production of Acremonium chrysogenum in methionine-supplemented medium. Applied Microbiology and Biotechnology, 2013, 97(6): 2551-2562. |
| [23] | Calvo AM, Wilson RA, Bok JW, Keller NP. Relationship between secondary metabolism and fungal development. Microbiology and Molecular Biology Reviews, 2002, 66(3): 447-459. |
| [24] | Nash CH, Huber FM. Antibiotic synthesis and morphological differentiation of Cephalosporium acremonium. Applied Microbiology, 1971, 22(1): 6-10. |
| [25] | Sándor E, Szentirmai A, Paul GC, Thomas CR, Pócsi I, Karaffa L. Analysis of the relationship between growth, cephalosporin C production, and fragmentation in Acremonium chrysogenum. Canadian Journal of Microbiology, 2001, 47(7): 801-806. |
| [26] | Long LK, Wang YL, Yang J, Xu XX, Liu G. A septation related gene AcsepH in Acremonium chrysogenum is involved in the cellular differentiation and cephalosporin production. Fungal Genetics and Biology, 2013, 50: 11-20. |
| [27] | Hu PJ, Wang Y, Zhou J, Pan YY, Liu G. AcstuA, which encodes an APSES transcription regulator, is involved in conidiation, cephalosporin biosynthesis and cell wall integrity of Acremonium chrysogenum. Fungal Genetics and Biology, 2015, 83: 26-40. |
| [28] | Hoffmeister D, Keller NP. Natural products of filamentous fungi: enzymes, genes and their regulation. Natural Product Reports, 2007, 24(2): 393-416. |
| [29] | Schmitt EK, Kempken R, Kück U. Functional analysis of promoter sequences of cephalosporin C biosynthesis genes from Acremonium chrysogenum: specific DNA-protein interactions and characterization of the transcription factor PACC. Molecular Genetics and Genomics, 2001, 265(3): 508-518. |
| [30] | Schmitt EK, Kück U. The fungal CPCR1 protein, which binds specifically to β-lactam biosynthesis genes, is related to human regulatory factor X transcription factors. The Journal of Biological Chemistry, 2000, 275(13): 9348-9357. |
| [31] | Schmitt EK, Bunse A, Janus D, Hoff B, Friedlin E, Kürnsteiner H, Kück U. Winged helix transcription factor CPCR1 is involved in regulation of β-lactam biosynthesis in the fungus Acremonium chrysogenum. Eukaryotic Cell, 2004, 3(1): 121-134. |
| [32] | Schmitt EK, Hoff B, Kück U. AcFKH1, a novel member of the forkhead family, associates with the RFX transcription factor CPCR1 in the cephalosporin C-producing fungus Acremonium chrysogenum. Gene, 2004, 342(2): 269-281. |
| [33] | Hoff B, Schmitt EK, Kück U. CPCR1, but not its interacting transcription factor AcFKH1, controls fungal arthrospore formation in Acremonium chrysogenum. Molecular Microbiology, 2005, 56(5): 1220-1233. |
| [34] | Dreyer J, Eichhorn H, Friedlin E, Kürnsteiner H, Kück U. A homologue of the Aspergillus velvet gene regulates both cephalosporin C biosynthesis and hyphal fragmentation in Acremonium chrysogenum. Applied and Environmental Microbiology, 2007, 73(10): 3412-3422. |
| [35] | Gong GH, Zhang W, HU YJ. Over-expression of AcveA in Acremonium chrysogenum to improve cephalosporin C production. Chinese Journal of Pharmaceuticals, 2014, 45(8): 719-723. (in Chinese)龚桂花, 张伟, 胡又佳, 朱宝泉. 顶头孢霉中过表达AcveA提高头孢菌素C产量. 中国医药工业杂志, 2014, 45(8): 719-723. |
| [36] | Zhang JY, Wolfe S, Demain AL. Phosphate repressible and inhibitable β-lactam synthetases in Cephalosporium acremonium strain C-10. Applied Microbiology and Biotechnology, 1988, 29(2/3): 242-247. |
| [37] | Wang HT, Pan YY, Hu PJ, Zhu YX, Li JY, Jiang XJ, Liu G. The autophagy-related gene Acatg1 is involved in conidiation and cephalosporin production in Acremonium chrysogenum. Fungal Genetics and Biology, 2014, 69: 65-74. |
| [38] | Terfehr D, Dahlmann TA, Specht T, Zadra I, Kürnsteiner H, Kück U. Genome sequence and annotation of Acremonium chrysogenum, producer of the β-lactam antibiotic cephalosporin C. Genome Announcements, 2014, 2(5): e00948. |
| [39] | Liu Y, Xie LP, Gong GH, Zhang W, Zhu BQ, Hu YJ. De novo comparative transcriptome analysis of Acremonium chrysogenum: high-yield and wild-type strains of cephalosporin C producer. PLoS One, 2014, 9(8): e104542. |
| [40] | Nødvig CS, Nielsen JB, Kogle ME, Mortensen UH. A CRISPR-Cas9 system for genetic engineering of filamentous fungi. PLoS One, 2015, 10(7): e0133085. |
