吴跃梅, 王素英

 , 董世瑞
, 董世瑞天津市食品生物技术重点实验室, 天津商业大学生物技术与食品科学学院, 天津 300134
收稿日期: 2015-05-19; 修回日期: 2015-07-14; 网络出版日期: 2015-09-29
基金项目: 国家自然科学基金(31270050);天津市创新团队项目(TD12-5049)
通信作者: Tel: +86-22-26667562; E-mail: wsying@tjcu.edu.cn
摘要: [目的] 利用16S rRNA和rpoC1基因分子标记研究螺旋藻、节旋藻的系统发育关系,并对其区分能力进行比较。[方法] 以84株螺旋藻、节旋藻为研究对象,对其进行16S rRNA、rpoC1基因序列的扩增、测序及分析,并对构建的系统发育树进行对比。[结果] rpoC1基因序列保守位点所占比例49.7%、平均G+C百分含量47.7%和序列相似度76%-100%明显低于16S rRNA基因序列的79.4%、55.6%和91%- 100%,其变异程度高于16S rRNA基因;基于16S rRNA、rpoC1基因构建的系统发育NJ树拓扑结构基本一致,84株实验藻株分为2个属3个类群,其中仅F-351、F-904-2、F-1070和TJBC14-1藻株为螺旋藻,其余均为节旋藻;虽然2个基因都不能区分形态种和地理种,但rpoC1基因NJ树的置信度(100%)高于16S rRNA基因(99%),属内分群效果也明显优于16S rRNA基因。[结论] 支持了螺旋藻、节旋藻为两个不同属的结论,且在属内分类时rpoC1基因比16S rRNA基因具有更高的区分度。
关键词: 螺旋藻节旋藻16S rRNArpoC1序列分析系统发育
Phylogenetic comparison between Spirulina and Arthrospira based on 16S rRNA and rpoC1 gene
Yuemei Wu, Suying Wang

 , Shirui Dong
, Shirui DongTianjin Key Laboratory of Food and Biotechnology, College of Biotechnology and Food Science, Tianjin University of Commerce, Tianjin 300134, China
Received: 19 May 2015; Revised: 14 July 2015; Published online: 29 September 2015
Supported by the National Natural Science Foundation of China (31270050) and by the Tianjin Innovation Team Programs(TD12-5049)
Corresponding author. Tel: +86-22-26667562; E-mail: wsying@tjcu.edu.cn
Abstract:[Objective] Based on 16S rRNA and rpoC1 gene sequences, the phylogenetic relationship between Spirulina and Arthrospira were studied and compared. [Methods] We amplified, sequenced and analyzed 16S rRNA and rpoC1 of 84 strains. Then the phylogenetic trees were constructed and compared. [Results] The conserved sites percentage, average G+C content and sequence identity of rpoC1 were 49.7%, 47.7%, 76%-100% respectively, significantly lower than 79.4%, 55.6% and 91%-100% of 16S rRNA, and the heterogeneity degree was higher. The trees generated with two different genes showed similar topologies and thus inferred consistent phylogenetic relationships. Eighty-four experimental strains were divided into 3 groups belonging to 2 genera: F-351, F-904-2, F-1070 and TJBC14 were Spirulina and the rest were Arthrospira. Although morphospecies and geographical species could not be distinguished based on 16S rRNA and rpoC1 gene sequences, the bootstrap value of rpoC1 (100%) was higher than that of 16S rRNA (99%). Moreover, clustering effect of rpoC1 for Spirulina and Arthrospirai was better than 16S rRNA. [Conclusion] Spirulina and Arthrospira were different genera, rpoC1 gene has more advantage to distinguish the strains in the same genus than that of 16S rRNA gene.
Key words: SpirulinaArthrospira16S rRNArpoC1sequence analysisphylogeny
螺旋藻(Spirulina)和节旋藻(Arthrospira)均为光能自养型丝状蓝藻。过去以形态学特征如光学显微镜下的细胞长度、宽度、是否有横壁、横壁处是否收缢等为依据的分类学研究,将二者分为了2个属[1, 2]。随后有研究者通过对节旋藻及螺旋藻的形态结构进行对比后发现,无论在光学显微镜还是电子扫描显微镜下二者都有很明显的形态差异[3],但是长期的观察发现藻丝存在多形变异现象[4],给形态为依据的分类带来较多的干扰。后期又出现了生理生化特征如脂肪酸组成、GC含量、对抗生素的敏感性等为依据的分类方法,但这些方法都存在一定的局限性[3, 5, 6, 7]。随着科技的快速发展,进入了以分子生物学为依据的分类时代[8, 9, 10, 11]。目前螺旋藻、节旋藻已完成克隆并用于系统发育分析的基因有:16S rRNA基因、ITS区、藻蓝蛋白操纵子序列、镍铁氢化酶基因、气囊蛋白基因等,这些基因可在一定程度上将两个属区分开,但是对于较低水平的分类鉴定还存在局限。最新的伯杰氏细菌鉴定手册中将蓝细菌分为了5部分,第三部分包括18个属,节旋藻和螺旋藻分别位于第1和第14个属[12]。尽管现在螺旋藻和节旋藻属于独立两个属的观点已被人们接受,但其属名的划分及使用一直存在争议,如同名不同种、同种不同名,这种混乱现象不仅影响了国内外的学术交流,更严重制约了节旋藻产业的发展。因此,迫切需要寻找可有效区分该类群属及其属内种间的分子标记。
RNA聚合酶(RNA polymerase,RNAP)是催化转录合成RNA的重要酶,蓝藻中的RNA聚合酶具有一个其它真细菌所没有的独特核心γ亚单位,由rpoC1基因编码,该基因全长1880 bp,在基因组中以单拷贝形式存在。Palenik运用rpoC1基因对Sargasso海的部分蓝藻分析后发现其结果虽然与16S rRNA基因的类似,但其序列差异更大[13]。Wilson等利用该基因对柱孢藻设计了专一性引物[14]。Fergusson等在卷曲鱼腥藻的研究中也有类似报道[15]。章群等用该基因对微囊藻进行了系统发育分析后发现其序列相似度高达97.6%-100%,不能有效区分地理种和形态种[16]。王捷对念珠藻的研究发现该基因对于不同来源地的藻株有一定区分度,但效果并不理想[17]。虽然运用该基因进行系统发育学研究的蓝藻并不多,GenBank中可参考的序列也较少,对其的分类价值也不一致,但近几年对该基因的研究却越来越多[18, 19, 20]。本研究同时进行了16S rRNA基因及rpoC1基因的克隆,并对其系统发育结果进行对比,探讨了rpoC1基因作为螺旋藻、节旋藻属间及属内分子标记的可操作性。
1 材料和方法 1.1 材料 1.1.1 供试藻株:螺旋藻、节旋藻实验藻株共84 株,主要来源为实验室保藏、分离纯化、惠赠及购买,其地理来源见表 1,其余序列由GenBank下载。
表 1. 实验藻株地理来源 Table 1. The geographic source of the experimental strains
| Strains | Geographic origins | Strains | Geographic origins |
| F-790–F-794 | Jiangxi Province, China | F-971 | Zhejiang Province, China |
| F-351, F-900-1, F-900-2, F-903 | Jiangsu Province, China | TJA, TJB, TJC, THE, TJF, TJG, | |
| F-835, F-1124-1, SDH | Shandong Province, China | TJI, TJJ4, TJSD | Tianjin, China |
| F-902 | Fujian Province, China | HBD | Hebei Province, China |
| F-904-1, F-904-2 | Guangdong Province, China | TJBC15, TJBC17 | Inner Mongolia Autonomous Region, China |
| F-882 | Japan | The others | Unclear |
表选项
1.1.2 主要试剂及仪器:DNA提取及PCR相关试剂均购自北京索莱宝生物科技有限公司;TA克隆试剂及引物由生工生物工程(上海)股份有限公司提供;其他生化试剂均为国产分析纯试剂。iCycle型PCR扩增仪、ChemiDoc XRS型凝胶成像系统,美国伯乐公司;Biowave DNA型蛋白核酸分析仪,英国WPA公司。
1.2 实验方法 1.2.1 藻培养及鲜藻泥的获得:按照参考文献[21]的方法进行藻种培养及藻泥的收集。
1.2.2 DNA提取及扩增:采用CTAB-溶菌酶法进行基因组DNA的提取[21];PCR扩增引物及条件:16S rRNA基因[22]、rpoC1基因[23];扩增反应体系:模板约100 ng,上下游引物(10 µmol/L)各2µL,2×PCR mix 25 µL,ddH2O补足50 µL。对基因组DNA及目的基因PCR扩增产物进行1%琼脂糖凝胶电泳检测。
1.2.3 PCR产物的TA克隆及测序:rpoC1基因完成TA克隆。将PCR扩增产物和菌液送由上海生工生物工程有限公司完成正、反向测序后拼接,同时根据测序峰图,对序列两端可信度较低的序列进行删除。
1.2.4 序列分析、系统进化树构建及对比:84株实验藻株的16S rRNA、rpoC1基因扩增产物经测序、比对截取后获得2个基因选定片段的核苷酸序列。然后从GenBank数据库中下载部分蓝藻的16S rRNA、rpoC1基因序列,用ClustalW2程序进行序列的多重比对及相似度计算,用MEGA 6.05软件排序后人工检查并校正部分排序结果,空位或者缺失位点均当作配对作删除处理,两端切齐后确定有效比对长度。然后进行相关生物信息学分析,并用Kimura-2-parameter模型构建NJ树,构树方法用自展检验估计系统树分支节点的置信度,自展数据集为1000。
2 结果和分析 2.1 基因组DNA提取效果及目的基因PCR扩增检测 部分实验藻株基因组DNA提取及目的基因PCR扩增检测结果见图 1,均得到了单一清晰的条带。基因组DNA分子量约为20 kb,较完整,无降解,OD260/OD280的比值绝大部分在1.7-2.0,仅有个别样品的比值超出此范围,但并不影响后续实验,DNA经适当倍数的稀释后可直接用于PCR扩增。16S rRNA基因及ITS区片段长度约2000 bp,rpoC1基因的片段长度约1000 bp,与文献中报道的其它蓝藻的该基因部分序列长度一致[22, 23],个别藻株扩增条带较弱或无扩增条带时,进行二次扩增直至获得全部实验藻株的PCR扩增产物。
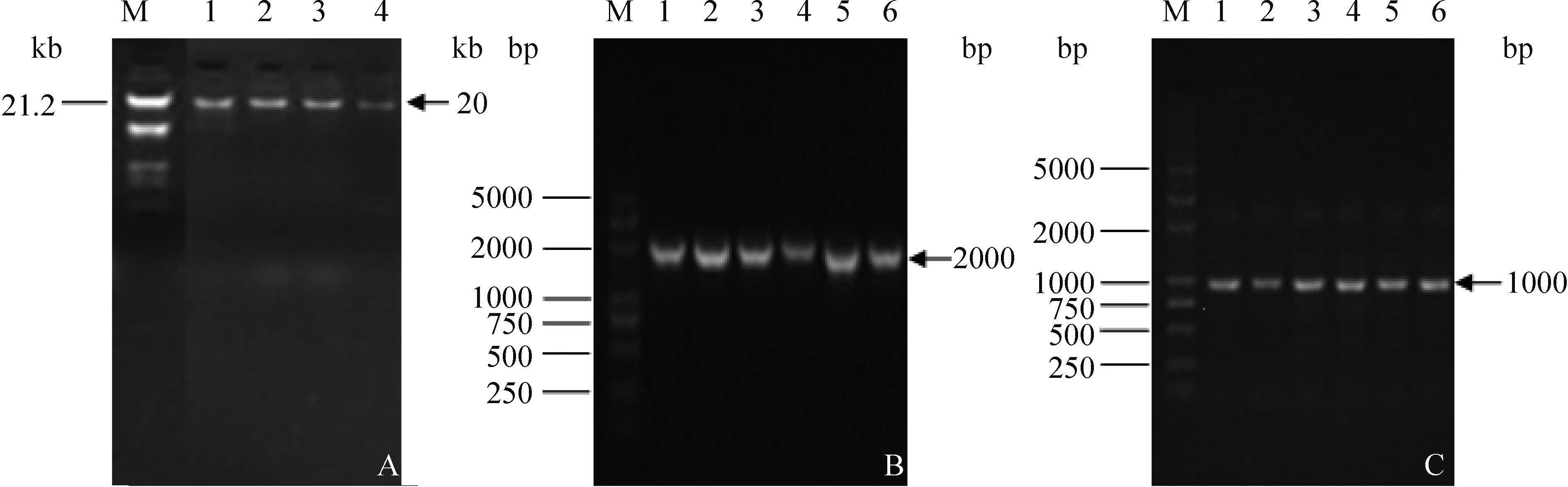 |
| 图 1. 基因组DNA及目的基因PCR扩增琼脂糖凝胶电泳检测图 Figure 1. Agarose gel electrophoresis of genomic DNA and PCR amplification for the target genes. A: Genomic DNA. lane 1-4: TJG1, SDH1, SDH2 and TJI1, respectively; M: Lamda DNA EcoRⅠ/Hind Ⅲ Marker. B: PCR amplification for 16S rRNA gene and ITS region. lane 1-6: TJE1, TJE6, TJE7, TJF1, TJF2 and TJF3, respectively; M: D2000 plus DNA Ladder. C: PCR amplification for rpoC1 gene. lane 1-6: TJB1, TJB2, TJC1, TJC2, HBD1 and HBD2, respectively; M: D2000 plus DNA Ladder. |
| 图选项 |
2.2 基因序列分析 16S rRNA基因及ITS区序列BLAST比对后截取16S rRNA基因序列,以Arthrospira sp. PCC98005为参照,71株实验藻株与它的序列相似性在99%以上,9株实验藻株(HBD4、TJE3、TJE8、TJF4、TJG1、TJJ1、F-791、F-901、F-1124-1)与它的序列相似度在98%-99%,而另外4株F-351、F-904-2、F-1070和TJBC14-1藻株的比对结果较为特殊,与Arthrospira sp. PCC98005的相似度仅为91%左右,说明与之亲缘关系较远。
rpoC1基因序列BLAST比对结果,以Arthrospira sp. PCC98005为参照,80株实验藻株与它的序列相似性在94%-99%,而实验藻株F-351、F-904-2、F-1070和TJBC14-1的比对结果较为特殊,与Arthrospira sp. PCC98005无匹配结果,说明与之亲缘关系较远,而与Cyanothece sp. PCC7424相似度最高,但也仅为80%,与Anabaena cylindrical PCC7122的序列相似度次之,为79%。
经MEGA 6.05软件对序列进行了分析,结果见表 2。
表 2. 生物信息学分析 Table 2. Bioinformatics analysis
| Genes | Effective length/bp | Conservedsites/% | Variablesites/% | Parsim-informationsites/% | Base percentage/% | |||
| T | C | A | G | |||||
| 16S rRNA | 1435 | 79.4 | 20.6 | 15.8 | 19.4 | 23.5 | 24.9 | 32.1 |
| rpoC1 | 948 | 49.7 | 50.3 | 38.7 | 26.0 | 19.3 | 26.3 | 28.4 |
表选项
16S rRNA基因序列的平均GC百分含量明显高于rpoC1基因序列,但其变异位点所占比例却显著低于rpoC1基因,说明rpoC1基因具有更高的变异度。
分析所有序列的碱基百分含量发现:除去3株外类群藻株(Anabaena variabilis ATCC 29413、Synechococcus elongatus PCC 6301和Synechococcus elongatus PCC 7942),99株待分析藻株的16S rRNA基因序列中T、C、A、G四种碱基百分含量分别为19.2%-20.1%,22.4%-22.7%,24.7%- 25.1%,31.8%-32.5%,G+C的百分含量最大56.0%,最小55.6%;4株实验藻株TJBC14-1、F-351、F-1070、F-904-2和3株NCBI数据库中螺旋藻藻株序列的C(23.4%)和A(25.4%-25.7%)百分含量不在上述范围内,其G+C的百分含量最大为55.5%,最小为Spirulina subsalsa CCAP 1475/2的54.3%;各藻株最大G+C百分含量差异为1.7%。除去4株外类群藻株(Anabaena variabilis ATCC 29413、Synechococcus elongatus PCC 6301、Synechococcus elongatus PCC 7942和Cyanothece sp. PCC7424),82株待分析藻株的rpoC1基因序列中T、C、A、G四种碱基百分含量分别为26.1%- 26.8%,18.6%-19.1%,26.1%-26.7%,28.0%-29.0%,而G+C的百分含量最大47.7%,最小46.7%;4株实验藻株TJBC14-1、F-351、F-1070和F-904-2序列的T(22.5%-22.8%)和C(22.8%-22.9%)百分含量不在上述范围内,其G+C的百分含量最大51.3%,最小50.9%;各藻株最大G+C百分含量差异为4.6%。通过分析各藻株之间的最大G+C百分含量差异也可看出rpoC1基因的变异程度要高于16S rRNA基因。
2.3 系统发育分析 基于16S rRNA基因和rpoC1基因构建的螺旋藻、节旋藻系统发育NJ树分别见图 2和图 3,进化分支上只显示支持率大于50%的值。
 |
| 图 2. 基于16S rRNA基因的系统发育NJ树 Figure 2. Phylogenetic NJ tree based on 16S rRNA gene sequence. Bootstrap support values were calculated by 1000 replications. Branches with bootstrap value < 50% are unshown. |
| 图选项 |
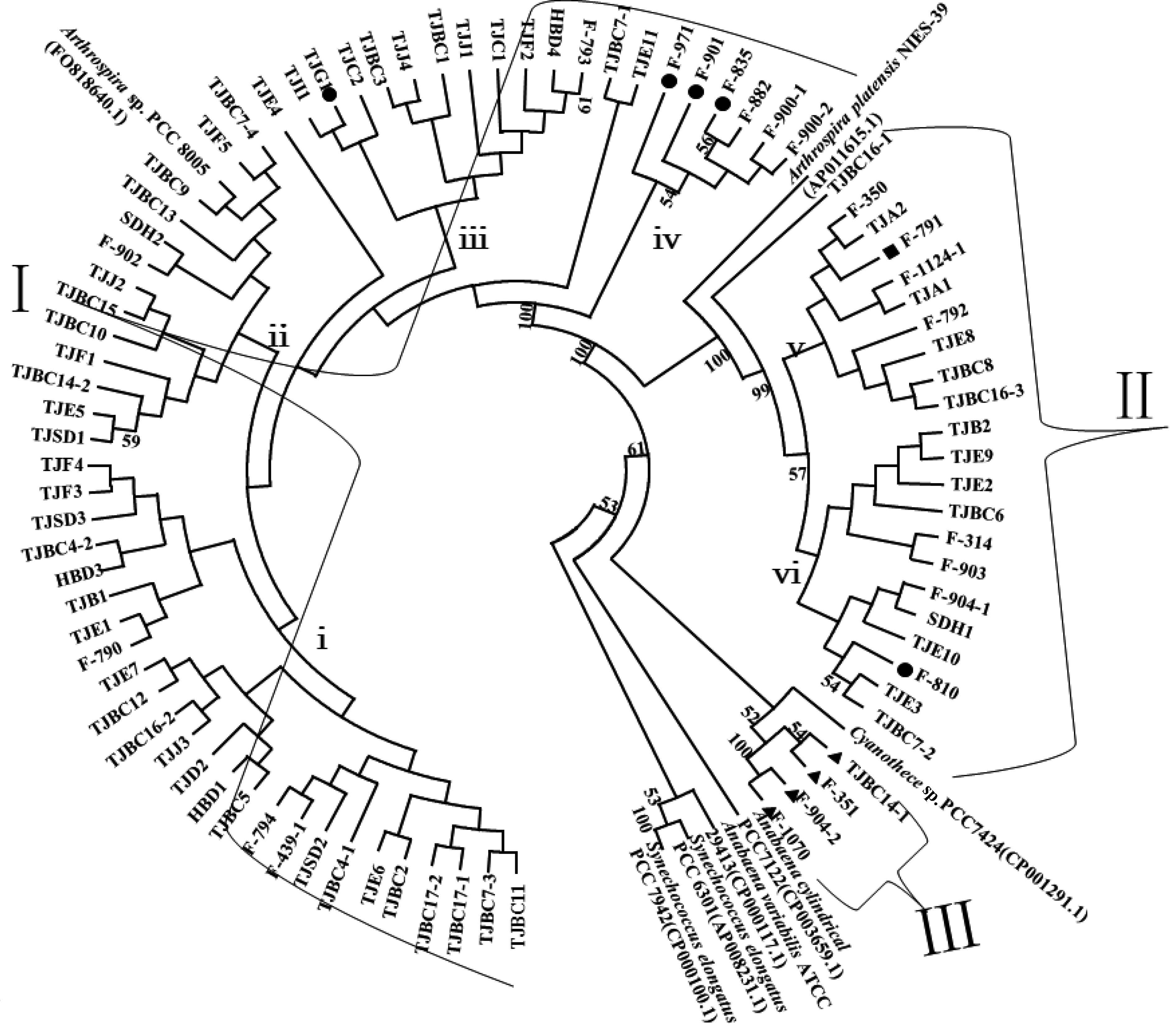 |
| 图 3. 基于rpoC1基因的NJ树 Figure 3. Phylogenetic NJ tree based on rpoC1 gene sequence. Bootstrap support values were calculated by 1000 replications. Branches with bootstrap value < 50% are unshown. |
| 图选项 |
16S rRNA系统发育NJ树将84株实验藻株分为3个类群,且得到了Bootstrap 99%的支持。Ⅰ、Ⅱ类群在Bootstrap 99%的支持率上聚为一个大分支,说明两类群藻株亲缘关系非常近。Ⅰ类群中包括53株实验藻株,GenBank下载序列10条,已经命名的强氏、印度、纺锤和极大节旋藻不同程度混杂在一起,虽然该类群中还有部分藻株分别聚为一个更小的单元(ⅰ、ⅱ、ⅲ),但是其置信度低于50%,认为该聚类结果不可靠;Ⅱ类群中包括27株实验藻株,GenBank下载序列4条,其中3条为钝顶节旋藻、一条为极大节旋藻;Ⅲ类群中所聚类的藻株数较少,仅TJBC14-1、F-351、F-1070和F-904-2为本实验藻株,剩余藻株均为盐泽螺旋藻不同品系。第Ⅲ类群与外类群Anabaena variabilis ATCC29413的遗传距离较近,相似度为89.37%-91.16%。该树的聚类结果与藻株GC含量判断结果一致。
通过序列相似度分析发现:Ⅰ类群、Ⅱ类群群内藻株之间序列相似度在99%-100%,Ⅰ类群、Ⅱ类群之间藻株序列相似度在98%以上,Ⅲ类群群内藻株之间序列相似度在98%以上,Ⅰ类群、Ⅱ类群与Ⅲ类群之间藻株序列相似度在91%-92%。根据茅云翔对节旋藻属的认定:16S rRNA基因序列相似度大于98%[4],判断Ⅰ、Ⅱ类群均为节旋藻品系,Ⅲ类群为螺旋藻品系。Ⅰ类群ⅰ单元序列相似度最高100%,最低99.1%;ⅱ单元最高100%,最低98.5%;ⅲ单元最高99.8%,最低99.1%。在细菌分类领域一般认为,16S rRNA 基因序列同源性大于98.5%,可以认为属于同一种;小于97%,可以认为属于不同的种。但也有人认为细菌如存在种间差异,其16S rRNA 基因序列相似度应低于97%,高于97%时要进一步做DNA-DNA杂交试验,故目前无法对该部分实验藻株做出种的鉴定。但该结果说明实验藻株至少可以分为2个不同的属,支持了其他****关于螺旋藻属、节旋藻属的划分。
基于rpoC1基因构建的系统发育NJ树,其拓扑结构与基于16S rRNA基因构建的基本一致,且得到比16S rRNA基因更高的Bootstrap支持率。通过对各分支中的藻株进行比较后发现:6株实验藻株的聚类位置变化较大,TJG1、F-882、F-835、F-901、F-971由16S rRNA基因NJ树中的Ⅱ类群转移到Ⅰ类群中,同时,F-791则由Ⅰ类群移动到Ⅱ类群中,除此之外其它藻株只是相对位置发生了变化。Ⅰ类群中包括56株实验藻株,Ⅱ类群中包括22株实验藻株,Ⅲ类群藻株未发生变化,但是Ⅲ类群藻株首先与Cyanothece sp. PCC7424聚在一起,而与Anabaena variabilis ATCC29413明显分开,这与BLAST比对结果一致。Ⅰ类群藻株基本被聚为四个更小的分类单元(ⅰ、ⅱ、ⅲ、ⅳ),但只有ⅳ单元的聚类结果得到54%的支持率,其单元内藻株序列相似度为98%以上,结果较为可靠;Ⅱ类群主要被聚为2个更小的单元(ⅴ、ⅵ),得到57%的支持率,但各单元内藻株聚类结果的Bootstrap支持率低于50%,认为聚类结果缺乏一定的可靠性。
通过序列相似度分析发现:Ⅰ、Ⅱ类群群内藻株之间的序列相似度在98%-100%;Ⅰ、Ⅱ类群之间藻株序列相似度在92%-95%;Ⅲ类群群内藻株之间序列相似度在99%以上;Ⅰ类群、Ⅱ类群与Ⅲ类群之间藻株序列相似度在76%左右。根据16S rRNA基因构建的NJ树推断结果:Ⅰ、Ⅱ类群均为节旋藻品系,Ⅲ类群为螺旋藻品系。可以认为92%的rpoC1基因序列相似度为螺旋藻、节旋藻属水平的分类依据,即同一属藻株的rpoC1基因序列相似度应 ≥ 92%。另外基于rpoC1基因序列相似度的第Ⅰ和第Ⅱ类群的群内藻株聚成了置信度较高的更小分类单元,说明rpoC1基因不仅与16S rRNA基因一样可明显将螺旋藻、节旋藻分为两个属,而且比16S rRNA基因的区分度更高。
3 讨论 本研究扩增了84株不同来源螺旋藻、节旋藻的16S rRNA及rpoC1基因的部分序列,并对其序列和系统发育树进行了比较和分析。使用16S rRNA及rpoC1基因都将实验藻株分为了3个类群,2个属,与基于其它基因(ITS区、cpc-F基因、hoxY基因)的系统学分析一样支持了对螺旋藻、节旋藻的属的划分[4, 8, 9, 10]。rpoC1基因对于3个类群划分的置信度均高于16S rRNA基因,可见,在螺旋藻、节旋藻属间或更低水平的分类时,rpoC1基因比16S rRNA基因具有更大的优越性,其原因可能是16S rRNA基因在分子结构上的高度保守性及其在基因组内的多拷贝性,使确定其序列的准确性降低,而同样作为基因水平的分类方法,管家基因rpoC1具有单一拷贝的优越性[13, 17],且rpoC1基因是蛋白质编码基因,它的分子进化主要受到维持氨基酸序列保守性的限制,而由于遗传密码子在第3位核苷酸上所具有的兼并性使得DNA序列可以发生较多替换而不改变其氨基酸序列[24]。因此,在属及其以下分类单元的系统发育分析及分类过程中,编码蛋白质的管家基因常被认为是一种比16S rRNA基因更好的靶标分子。
以分离自天津渤海海滨湿地的实验藻株TJE1-TJE11为例分析形态、地理来源与藻株分类的关系,发现采样时间基本相同、采样地点彼此靠近、形态有差异的11株藻,无论选取哪个基因,这些藻株都不同程度的分散于整个系统发育树中,且聚类结果与形态无关。再如实验藻株TJF1- TJF4虽然都聚在Ⅰ类群中,但彼此形态却有较大差异;分析其它形态种和地理种也是类似结果,说明这2个基因都不能很好地区分地理种和形态种,这一结论与章群等对微囊藻的分析一致[16]。该现象同时说明了螺旋藻、节旋藻形态的多形变异性,即形态不同的藻株或同一形态不同来源的藻株都可能是同一基因型,即以形态学为依据的分类标准存在局限性,不能作为主要分类依据。
目前原核生物的种群确定主要依赖于形态学特征、遗传特征及DNA同源性分析。其中DNA同源性成为细菌新种群确定的必要标准之一,但是依赖于该特征的分析方法存在诸多局限:操作复杂、人力物力消耗大、测定结果误差大、种群地位的确定容易受染色体外DNA的影响等。随着分子技术的不断发展,多基因系统学分析被越来越多地应用于解决系统进化分析,尤其是多序列位点分型(Multilocus Sequence Typing,MLST)技术,该技术通过利用不同位点的保守基因进行比较分析后确定各细菌的分类地位及亲缘关系,为原核生物的相关系统学研究指出了新的方向[25]。研究证明多个基因的联合分析会产生更少的同等简约树,相应分支的Bootstrap支持率也会更高,即使不同分子标记得到的系统发育关系之间有矛盾,通过分析矛盾产生的原因,也可帮助更好的理解系统发育关系。代永东等的研究发现多基因序列联合分析会使得基因树更能准确的推测物种,特别是近缘物种的系统关系[26]。国际细菌分类委员会建议对位于染色体不同位点的持家基因序列(至少5个基因)进行分析,寻找合适的序列相似性水平用于细菌种群的确定,但是该方法还在探索之中。因此,利用基因组中不同位置的多个保守基因来进行螺旋藻、节旋藻的系统发育学研究将会成为今后的研究方向。本研究中2个聚类结果显示大部分藻株为节旋藻,再次说明目前大规模养殖和运用于商业活动中的螺旋藻其分类学地位实际为节旋藻,同时表明16S rRNA基因和rpoC1基因均可用于螺旋藻属、节旋藻属的划分,且在属内藻株的分类中rpoC1基因比16S rRNA基因区分度更高,分类学意义更大,可以作为多基因联合进行系统发育学研究的可选基因之一。
参考文献
| [1] | 胡鸿钧. 螺旋藻生物学及生物技术原理. 上海: 科学出版社, 2003. |
| [2] | Ma ZL, Gao KS. Photoregulation of morphological structure and its physiological relevance in the cyanobacterium Arthrospira (Spirulina) platensis. Planta, 2009, 230(2): 329-337. |
| [3] | Sun P, Shi L, Sun H. Comparison of nutritive value between Spirulina and Arthrospira. Shandong Agricultural Sciences, 2011, (4): 39-42. (in Chinese)孙鹏, 石磊, 孙浩. 螺旋藻与节旋藻营养价值的比较研究. 山东农业科学, 2011, (4): 39-42. |
| [4] | Mao YX, Yang GP, Zhang BH, Zhang XC. Application of the sequences analysis of the 16S rRNA gene and ITS of 16-23S rRNA to the systematic study of the genus Arthrospira and Spirulina. High Technology Letters, 2001, 11(6): 12-18. (in Chinese)茅云翔, 杨官品, 张宝红, 张学成. 16S rRNA 基因与16S-23S rRNA转录单元内间隔区序列分析及其在节旋藻和螺旋藻分类鉴定中的应用. 高技术通讯, 2001, 11(6): 12-18. |
| [5] | Romano I, Bellitti MR, Nicolaus B, Lama L, Mance MC, Pagnotta E, Gambacorta A. Lipid profile: a useful chemotaxonomic marker for classification of a new cyanobacterium in Spirulina genus. Phytochemistry, 2000, 54(3): 289-294. |
| [6] | Wang ZP. Studies on taxonomy and mutants identification by protein SDS-PAGE in Spirulina. Journal of Zhejiang University (Agriculture & Life Sciences), 2000, 26(6): 583-587. (in Chinese)汪志平. 蛋白质SDS-PAGE用于螺旋藻分类及突变体鉴定的研究. 浙江大学学报(农业与生命科学版), 2000, 26(6): 583-587. |
| [7] | Zang XN, Zhang XC, Wang GG, Liu JJ, Zhang XH, Liu B. Comparative study on sensitivities of Arthrospira platensis and Spirulina subsulsa to seven antibiotics. Acta Oceanologica Sinica, 2004, 26(3): 82-92. (in Chinese)臧晓南, 张学成, 王高歌, 刘金姐, 张晓辉, 刘滨. 节旋藻和螺旋藻对7种抗生素敏感性的比较研究. 海洋学报, 2004, 26(3): 82-92. |
| [8] | Zhang XH, Shiraiwa Y, Sui ZH, Zhang XC. Cloning and characterization of hoxY gene from Arthrospira and Spirulina and its application in phylogenetic study. Periodical of Ocean University of China, 2005, 35(6): 1021-1025. (in Chinese)张晓辉, Shiraiwa Y, 隋正红, 张学成. hoxY基因的克隆及其在节旋藻和螺旋藻系统学研究中的应用. 中国海洋大学学报, 2005, 35(6): 1021-1025. |
| [9] | 于海鹏. 螺旋藻藻蓝蛋白裂合酶 cpcE-F基因的克隆、表达及进化分析. 青岛: 青岛科技大学, 2010. |
| [10] | Wang Y, Tai LH, Xie YF, Enhebayaer. Cloning and sequencing of the hoxY genes in three samples of Arthrospira. Journal of Inner Mongolia Medical University, 2014, 36(2): 105-109. (in Chinese)王越, 邰丽华, 谢岳飞, 恩和巴雅尔. 节旋藻(Arthrospira)3个样品镍铁氢化酶hoxY基因的克隆与测序. 内蒙古医科大学学报, 2014, 36(2): 105-109. |
| [11] | 李勇勇. 钝顶节旋藻16S rRNA-ITS分子鉴定及重金属Pb2+对节旋藻影响研究. 曲阜师范大学硕士学位论文, 2013. |
| [12] | Shimamatsu H. Mass production of Spirulina, an edible microalga//Ang Jr PO. Asian Pacific Phycology in the 21st Century: Prospects and Challenges. Netherlands: Kluwer Academic Publishers, 2004: 39-44. |
| [13] | Palenik B. Cyanobacterial community structure as seen from RNA polymerase gene sequence analysis. Applied and Environmental Microbiology, 1994, 60(9): 3212-3219. |
| [14] | Wilson KM, Schembri MA, Baker PD, Saint CP. Molecular characterization of the toxic Cyanobacterium Cylindros permopsis raciborskii and design of a species-specific PCR. Applied and Environmental Microbiology, 2000, 66(1): 332-338. |
| [15] | Fergusson KM, Saint CP. Molecular phylogeny of Anabaena eireinalis and its identifieation in environmental samples by PCR. Applied and Environmental Microbiology, 2000, 66(9): 4145-4148. |
| [16] | Zhang Q, Li ML, Liu HL, Lei LM, Han BP. Phylogenetic relationships of Microcystis based on rpoC1 gene sequences in China. Ecology and Environmental Sciences, 2009, 18(6): 2039-2043. (in Chinese)章群, 李名立, 刘海林, 雷腊梅, 韩博平. 基于rpoC1基因的微囊藻属分子系统学研究. 生态环境学报, 2009, 18(6): 2039-2043. |
| [17] | 王捷. 念珠藻属(蓝藻)的分类及分子系统研究. 山西大学硕士学位论文, 2011. |
| [18] | D'Alelio D, Gandolfi A. Recombination signals in the rpoC1 gene indicate gene-flow between Planktothrix (Cyanoprokaryota) species. Journal of Phycology, 2012, 48(6): 1424-1432. |
| [19] | Churro C, Pereira P, Vasconcelos V, Valério E. Species-specific real-time PCR cell number quantification of the bloom-forming Cyanobacterium Planktothrix agardhii. Archives of Microbiology, 2012, 194(9): 749-757. |
| [20] | Parvathy VA, Swetha VP, Sheeja TE, Leela NK, Chempakam B, Sasikumar B. DNA barcoding to detect chilli adulteration in traded black pepper powder. Food Biotechnology, 2014, 28(1): 25-40. |
| [21] | Wu YM, Wang SY, Dong SR, Wei J, Meng J. Effects of different methods of cell wall-broken on Arthrospira DNA extraction. Research and Exploration in Laboratory, 2015, 34(4): 13-16. (in Chinese)吴跃梅, 王素英, 董世瑞, 魏娟, 孟佳. 不同破壁方法对节旋藻DNA提取效果的影响. 实验室研究与探索, 2015, 34(4): 13-16. |
| [22] | 师德强. 节旋藻新品系TJSD藻种鉴定及其主要营养成分分析. 天津商业大学硕士学位论文, 2009. |
| [23] | Seo PS, Yokota A. The phylogenetic relationships of Cyanobacteria inferred from 16S rRNA, gyrB, rpoC1 and rpoD1 gene sequences. The Journal of General and Applied Microbiology, 2003, 49(3): 191-203. |
| [24] | Mollet C, Drancourt M, Raoult D. rpoB sequence analysis as a novel basis for bacterial identification. Molecular Microbiology, 1997, 26(5): 1005-1011. |
| [25] | Wang FQ, Zhang YF, Liu J, Song AD, Liu QJ, Chen WX. Comparison of phylogeny analysis methods for Rhizobia asolated from Albizia spp., Acacia spp. and Leucaena leucocephala. Acta Microbiologica Sinica, 2008, 48(1): 1-7. (in Chinese)王风芹, 张勇法, 刘杰, 宋安东, 刘全军, 陈文新. 合欢、金合欢和银合欢根瘤菌系统发育研究方法比较. 微生物学报, 2008, 48(1): 1-7. |
| [26] | Dai YD, Yu H, Zeng WB, Yang JY, He L. Multilocus phylogenetic analyses of the genus Isaria (Ascomycota, Cordycipitaceae). Mycosystema, 2015, 34(5): 1-14. (in Chinese)代永东, 虞泓, 曾文波, 杨俊媛, 何璐. 多基因联合分析棒束孢属Isaria系统发育关系(Ascomycota, Cordycipitaceae). 菌物学报, 2015, 34(5): 1-14. |
