闫雷, 徐海


山东大学微生物技术国家重点实验室, 山东 济南 250100
收稿日期: 2015-04-03; 修回日期: 2015-05-15; 优先出版日期: 2015-06-17
基金项目: 国家自然科学基金面上项目(31170112);国家科技重大专项(2013ZX10004217-003);山东大学微生物技术国家重点实验室自主项目
通信作者: 徐海, haixu@sdu.edu.cn
摘要: 喹诺酮类抗菌药物从早期主要用于治疗尿道感染发展到后来治疗肠道感染和呼吸道感染,目前已在临床、畜牧业和水产业中广泛使用,细菌对其耐药性也逐渐呈蔓延趋势,耐药机制日趋复杂。喹诺酮类耐药机制主要分为染色体介导的耐药和质粒介导的耐药,后者对细菌耐药性的广泛传播起着重要作用。1998年首次报道了质粒介导的喹诺酮类耐药机制,即质粒上qnr基因介导的细菌对氟喹诺酮耐药机制,qnr基因可在不同细菌中迅速水平传播,引发的感染不易控制,使得院内感染大范围的流行。此外,qnr基因通常与β-内酰胺类耐药基因相关或存在于复杂整合子中与其它多重耐药基因共同整合,缩小了临床医生治疗相关细菌感染时选药或联合用药的空间,给我们带来了严峻的挑战。本文就qnr基因的发现历史、耐药机理及在国内的流行状况做了详细概述。
关键词: 细菌感染喹诺酮耐药qnr基因质粒介导耐药性的水平转移多重耐药
Classification and prevalence of plasmid-mediated quinolone resistance qnr genes in China-A review
Lei Yan, Hai Xu


The State Key Laboratory of Microbial Techology, Shandong University, Jinan 250100, Shandong Province, China
Received: 3 April 2015; Revised: 15 May 2015; Published online: 17 June 2015
Supported by the National Natural Science Foundation of China (31170112), by the National Science and Technology MajorProject (2013ZX10004217-003), and by the Grant of the State Key Laboratory of Microbial Technology, Shandong University
Corresponding author. Tel: +86-531-88362362; E-mail: haixu@sdu.edu.cn
Abstract:Quinolone antibacterial drugs, developing from the treatment of urinary tract infection in early time and now from the treatment of intestinal infection and respiratory infection, have been widely used in clinical, animal husbandry and aquaculture. Bacteria gradually become resistant to them and resistance mechanism is more and more complicated. Quinolone resistance mechanism is mainly divided into chromosome mediated resistance and plasmid mediated resistance, the latter plays an important role in spreading of antibiotic resistance. In 1998, plasmid mediated quinolone resistance mechanism was reported for the first time, namely the qnr gene mediated fluoroquinolone resistance mechanism. qnr genes can spread rapidly in different bacteria, which causes the infection difficult to control, makes the nosocomial infection popular in a wide range. In addition, qnr genes are usually associated with β-lactamase resistance gene. They exist in complex integron and integrate with the other varieties of resistance genes, which narrows the space of clinical medicine choose or drug combinations use to treat related bacterial infection and brings us a serious challenge. In this review, we provide a detailed overview for the historical discovery, classification, the resistance mechanisms of qnr genes, and the prevalence of those genes in China.
Key words: bacterial infectionsquinolone resistanceqnr genehorizontal transfer of antibiotic resistance by plasmid-mediatedmulti-drug resistance
医药行业不断发展,随之的问题亦接踵而来。抗生素的发现及临床应用曾一度开启了人类抗感染历史的新纪元,而目前又不得不面对耐药性对人类健康的严峻挑战。自从1962年由美国Lesher等开发第一代喹诺酮药物萘啶酸(Nalidixic acid)以来,喹诺酮药物迅速发展且广受欢迎,然而,由于喹诺酮药物的过量使用甚至滥用,造成环境中抗生素的大量残留,使细菌的耐药率不断攀升,喹诺酮耐药问题在人们生活中凸显严重。在国内,喹诺酮类抗菌药物从早期主要用于治疗尿道感染发展到治疗肠道感染和呼吸道感染,如今已在临床、畜牧业和水产业中广泛使用。除染色体基因突变引起的喹诺酮耐药性之外,质粒介导的喹诺酮耐药基因也在人、动物、周围环境[1, 2],肉制品[3],严禁使用抗生素的儿童医院[4],普通医院[5]等地方检测到,其耐药性易于在细菌间相互转移,逐渐呈蔓延趋势,耐药机制日趋复杂。
qnr基因是指能编码一类对萘啶酸或氟喹诺酮药物(Fluoroquinolones)产生抗性蛋白的基因,其编码的蛋白为Qnr,这是一类由串联重复氨基酸构成的蛋白,属于重复五肽家族,一般模式为[Ser、Thr、Ala或Val][Asp或Asn][Leu或Phe][Ser、Thr或Arg][Gly],但并非一成不变。
qnr基因作为质粒介导的喹诺酮耐药性的主要原因,在喹诺酮耐药性的传播中起着重要作用,它能在不同细菌间进行水平传播,若一旦传播到人体病原菌中,可引发不易控制的感染,并大范围流行。qnr基因还能与其他耐药基因共同整合,使得临床治疗用药更加困难。有关喹诺酮抗性基因的研究多与编码β-内酰胺酶的质粒相涉及,比如ESBL基因(SHV、CTX-M或VEB-1类型) 或AmpC基因(FOX-5或DHA-1类型),据报道,1/3-2/3的产超广谱β-内酰胺酶的肠杆菌同时具有氟喹诺酮抗性[6]。
1 qnr基因的发现及分类目前已经命名的qnr基因有qnrA、qnrB、qnrC、qnrD、qnrS、qnrVC六种,而大部分又包含有大量的变体,且其变体的发现速度非常快,共有近100种变体已被发现。
1.1 qnrA真正意义上的质粒介导的喹诺酮耐药基因于1998年首次从一株耐多药的肺炎克雷伯菌(Klebsiella pneumonia)中检测到[7],当时,Martínez-Martínez及同事们在研究一个叫pMG252的质粒,该质粒大小为56 kb,来自于美国Alabama大学病人尿液样本中的一株耐多药肺炎克雷伯菌,接合转移该质粒可使多种不同的革兰氏阴性菌表现出对萘啶酸、环丙沙星及其他喹诺酮药物的耐药性状,将pMG252质粒转移接合到大肠杆菌J53 (E. coli J53)中,萘啶酸的MIC值由4mg/L增加到32 mg/L,对环丙沙星的MIC值由0.008 mg/L增加到0.25 mg/L[7]。由此该基因被命名为qnrA (基因库登录号为AY070235)[8],大小为657bp,编码的蛋白为QnrA,后来由于其他变体的发现,又重新命名为QnrA1。
随后,在中国境内首先发现了QnrA的变体,该变体存在于一株产酸克雷伯菌(Klebsiella oxytoca)中,与QnrA1只有几个氨基酸的不同,命名为QnrA2 (基因库登录号为AY675584)[9]。2005年Laurent Poirel等在海藻希瓦氏菌(Shewanella algae)中又发现3个QnrA的变体,它们与QnrA1相比有2-4个氨基酸的差异,基因中G+C含量与染色体一致为52%[10],这3个QnrA变体被命名为QnrA3 (基因库登录号为DQ058661)、QnrA4 (基因库登录号为DQ058662)、QnrA5 (基因库登录号为DQ058663)。2006年QnrA6在奇异变形杆菌(Proteus mirabilis)和肺炎克雷伯菌中被E. Cambau等发现(基因库登录号分别为DQ151889和DQ435306),与QnrA1相比,奇异变形杆菌中核苷酸有30处不同,导致了5处氨基酸的差异;肺炎克雷伯菌中核苷酸有29处不同,导致了3处氨基酸的差异[11]。后来又在希瓦氏菌属中发现了QnrA7 (基>因库登录号为GQ463707),但其发现过程并未公开。
1.2 qnrS2003年10月初,日本爱知县爆发了一场由食物中弗氏志贺菌(Shigella flexneri 2b)引起的感染疫情,从病人身上采样的8株志贺氏杆菌中,有一株额外地带有氟喹诺酮和头孢菌素II (Cephaloridine)耐药性,通过研究发现,这株菌里有一个携带耐药基因的质粒,能够克隆一种新的耐药基因,该基因能够编码一个218氨基酸的五肽重复蛋白,与QnrA1的氨基酸序列有51%的一致性,后被命名为QnrS (基因库登录号为AB187515),这也是首次报道志贺氏杆菌中的接合质粒[12]。随着后来其他变体的发现,该基因被称为qnrS1。
2006年在美国,Kathryn Gay等在非伤寒沙门氏菌(Non-Typhi Serotypes ofSalmonella enterica)中的质粒中发现了qnrS的变体,编码的氨基酸序列与QnrS1的相似度达到91.3%,后被命名为QnrS2[13](基因库登录号为DQ485530)。而曾经在基因库中保存为qnrS1的在中国广州兽医诊所中发现的qnrS变体,因为与qnrS1有一个密码子差异而被重新命名为qnrS3[14] (基因库登录号为EU077611)。2008年在丹麦国立血清研究所,QnrS4在沙门氏菌(Salmonella enterica)中被发现(基因库登录号为FJ418153),这个新的变体与QnrS1只有1个氨基酸差异(61位V→A)[15],QnrS5与QnrS6于2011年在韩国发现(基因库登录号分别为HQ631377和HQ631376),来源于运动型气单胞菌属(motileAeromonas spp.),与QnrS1相比,QnrS5有6个氨基酸的差异(5位N→R,120位S→T,148位N→H,206位L→Q,207位I→L,216位Y→F),试验中与qnrS2团簇在一起的一个基因,与qnrS2相似度为99%以上,后被命名为qnrS6[16](命名法来源于网站http://www.lahey. org/qnrStudies)。QnrS7与QnrS8虽然已经发现(基因库登录号分别为KF730651和KF730652),其发现历程亦未公开。qnrS9首次报道于2014年,在东南亚采集的一株克雷伯菌中发现[17](基因库登录号为: KF732714)。
1.3 qnrBqnrB是在2004年Jacoby及其同事在研究一株来自印度南部的肺炎克雷伯菌时首次发现的,当时他们发现该基因能够表达出低水平的喹诺酮抗性,但是用PCR的方法不能扩增出qnrA基因,它编码的是一个214或226个氨基酸残基的蛋白质,>随后被命名为qnrB1 (基因库登录号为DQ351241)[18]。
QnrB的变体较多,目前已发现80多种,大部分发现于柠檬酸杆菌(Citrobacter freundii),其基因大小在645 nt左右。与QnrB1相比,大多变异发生在第2(A→T)、20(I→V)、79(S→A或V)、142(I→M)、163(T→S或A)、198(N→S)、212(V→I)位氨基酸上。
1.4 qnrC2009年,在上海华山医院,研究人员发现来自于一个尿路感染病人的一株奇异变形杆菌带有较弱的耐药性,它对大多数抗生素都很敏感,却>对氨苄青霉素(Ampicillin)、新诺明(Sulfamethoxazole)及甲氧苄氨嘧啶(Trimethoprim)有耐药性,通过PCR方法不能扩增出当时已知的qnr基因或者aac(6’)-Ib-cr和qepA基因,而接合转移里面的一个叫pHS10的质粒,受体菌对环丙沙星的药敏值增加了32倍。经过进一步研究发现,该质粒中一个大小为666 bp的基因在起作用,它能编码一种221个氨基酸的蛋白质,该蛋白质与QnrA1、QnrB1、QnrS1和QnrD的序列一致性分别为64%、41%、59%和43%,与QnrVC1和QnrVC2的序列一致性均为67%,后被命名为QnrC[19](基因库登录号为EU917444)。在qnrC基因上游,存在一段新的IS3家族嵌入序列ISPmi1,能编码一种移码突变过的转座酶,使得qnrC基因的转移更简单常见。
目前为止,并未发现qnrC有其他变体。
1.5 qnrD2008年在中国河南地区,研究人员在人身上采集的4株沙门氏菌中发现一个大小约4.3 kb的质粒,接合转移至大肠杆菌内能使其对环丙沙星的最低抑菌浓度提高约32倍,而该质粒并不含有其他喹诺酮耐药基因,它编码一种大小为214个氨基酸的五肽重复蛋白,被命名为[20]qnrD (基因库登录号为FJ228229)。2014年Helga Abgottspon等在肠道沙门氏菌中发现了QnrD2 (基因库登录号为KF055448),与QnrD1有2个氨基酸的差异(Ile189→Thr、Leu202→Phe),但是其耐药水平并无明显差异[21]。
1.6 qnrVCqnrVC是首先在霍乱弧菌(Vibriocholerae)中发现的qnr系列基因,亦能编码一种重复五肽家族的蛋白,如今在别的细菌中也有发现[22]。目前为止,已经发现7种qnrVC的变体,但有一些是以存在细菌染色体中的耐药基因盒(resistance gene cassette)形式被发现的,从质粒中发现的有qnrVC2[23]、qnrVC4[24]、qnrVC5[25]、qnrVC6[26](基因库登录号分别为: AB200915、GQ891757、KC540630、KC202804),其余的qnrVC基因如qnrVC1[23]、qnrVC3[27]、qnrVC7[28]还未有在质粒中发现的相关报道。
2 Qnr蛋白对喹诺酮药物的抗性机制喹诺酮药物的作用过程中,涉及到两个重要的酶,DNA旋转酶(DNA gyrase)和拓扑异构酶Ⅳ(Topoisomerase IV)。它们是细菌正常的生长过程中必不可少的,而喹诺酮类药物正是以这两种酶为结合靶点,起到抑制细菌生长的效果。
DNA旋转酶是由两个A亚基和两个B亚基组合的一种四聚体,这两种亚基分别由gyrA和gyrB基因编码。这种酶能够通过一种涉及ATP的作用,向DNA中引入负超螺旋,来缓解转录和复制过程>中DNA复合体内由于移动引起的拓扑异构压力(图 1-A)。拓扑异构酶Ⅳ属于DNA旋转酶的同系物,也有4个亚基,包括2个C亚基和2个E亚基,分别由parC和parE基因编码(Staphylococcus aureus中为grlA和grlB),拓扑异构酶Ⅳ功能与DNA旋转酶基本相似,但其最主要功能在于解除重复的子染色体之间的连接。
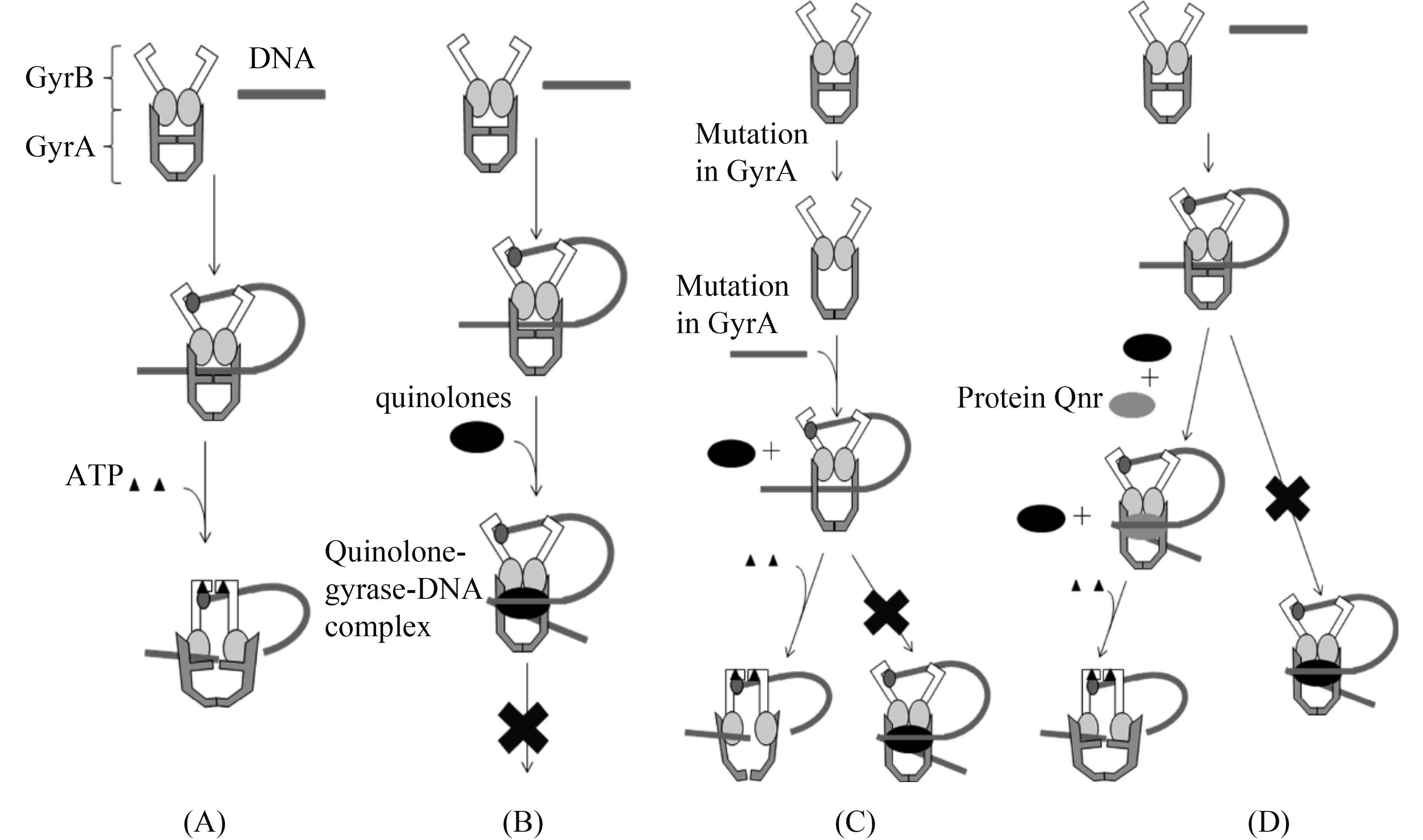 |
| 图 1. 喹诺酮作用机理及耐喹诺酮作用机理示意图 Figure 1. The mechanism of quinolones and quinolone resistance involving DNA gyrase. A: The mechanism of normal DNA gyrase; B: The mechanism of quinolone on DNA gyrase; C: The mechanism of quinolone resistance induced by GyrA mutation; D: The mechanism of quinolone resistance induced by Qnr protection. |
| 图选项 |
>喹诺酮药物能与DNA旋转酶或拓扑异构酶Ⅳ以及细菌DNA互相粘合绑定形成“喹诺酮-酶-DNA”三元复合物(图 1-B),在粘合后,喹诺酮药物会随机诱导酶发生构象的变化,使得DNA受到破坏,同时,喹诺酮药物还阻止了受损DNA的再恢复。在革兰氏阴性和革兰氏阳性菌中,构成三元复合物的酶出现概率并不相同(喹诺酮药物的化学结构对结合靶点的选择性亦有重要影响[29]),对临床菌株的最低抑菌浓度值进行分析以及对DNA旋转酶和拓扑异构酶Ⅳ的活动进行的直接分析表明,在革兰氏阴性菌(如E. coli和Neisseria gonorrhoeae)中,喹诺酮药物的首要结合靶点是DNA旋转酶[30, 31],三元复合物“喹诺酮-DNA旋转酶-DNA”能够快速形成,在复制叉前起作用以抑制DNA复制[32];而在革兰氏阳性菌(S. aureus和S. pneumoniae)中,喹诺酮药物的首要结合靶点是拓扑异构酶Ⅳ[30, 31],三元复合物“喹诺酮-拓扑异构酶Ⅳ-DNA”的形成较之缓慢,位于复制叉之后[32]。
然而,由于抗生素的滥用或者其他作用的影响,导致细菌中基因的突变,产生耐药性并不断遗传。在染色体介导的耐药性机制中,常见的是由于Ⅱ型拓扑异构酶基因的喹诺酮抗性决定区发生突变并不断积累,使得三元复合物不能形成(图 1-C),细菌便表现出对喹诺酮药物的不敏感性,研究表明,大部分突变发生在Ⅱ型拓扑异构酶高度保守的丝氨酸残基上[8]。在质粒介导的喹诺酮耐药机制中,能水平转移的qnr基因为主要原因,它表达产生Qnr蛋白,该蛋白虽然既不能改变细胞内喹诺酮药物的浓度,也不能改变药物的活性,但是它能保护DNA旋转酶或拓扑异构酶Ⅳ不能形成三元复合物(图 1-D),从而免受喹诺酮药物的抑制[8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33]。且Qnr蛋白的浓度能够影响这种保护的程度,相关研究指出,低浓度的Qnr蛋白就能使这种保护即耐药性出现,在通常情况下,DNA旋转酶的抑制需要浓度6 μmol/L (2 μg/mL)的环丙沙星(CIP),但是耐药只需0.5 nmol/L的QnrB1,甚至当>浓度达到5 pmol/L的时候就能出现些许耐药症状[18]。
3 国内对qnr基因流行状况的研究报道自从qnr发现以后,很多科研工作者一直致力于qnr基因的流行调查,以更好地了解其遍及程度来指导医疗卫生等事业的发展。
在国内,很多领域、地区已经检测到qnr基因,包括人畜、水环境、医院、食品,大多研究集中在qnrA、qnrB、qnrS这3种基因上,而含有该基因的菌株大多为肠杆菌属或克雷伯菌属。整合子系统是qnr基因存在的主要方式,qnr基因能随着质粒轻松进入另一宿主,这也是耐药基因能够水平转移的主要原因。虽然质粒介导的喹诺酮耐药性比较微弱,但是通常都能与染色体介导的耐药性互相关联,促进高水平耐药性的选择形成,且能在整合子系统里涉及其他相关耐药基因,提高自身的生存能力。
本实验室曾分离到一株含有复杂Ⅰ型整合子的气单胞菌(Aeromonas puctata 159),对其耐药基因盒阵列结构分析,发现一个新型喹诺酮药物的抗性基因,被命名为qnrVC4,并对此喹诺酮耐药新基因进行了功能验证、定位及转移性研究[24]。同时,我们也对水环境细菌质粒介导的喹诺酮耐药性基因分布和转移性进行初步的调查。从391株不同耐药菌株中筛选了31株具有qnr类基因,其中2株含qnrA基因;12株具有qnrS基因;21株含qnrB基因,5株同时含有qnrB,qnrS基因。在这31株菌中检测到12株菌具有与喹诺酮耐药性相关的aac(6’)-Ib-cr基因。与喹诺酮相关的外排泵基因>qepA也在1株qnrB菌株中和4株qnrS菌株中检测到[34]。
在表 1中,整理搜集了国内近5年来(报道发表于2010年后)对qnr基因的检测报道。
表 1. 近5年来国内对qnr基因的检测报道Table 1. The reports of qnr genes during the past 5 years in China
| References | Type and proportion/isolates | Sampling time and site | Source | Isolated strain |
| UT: Unknow type; UM: Not mentioned. | ||||
| [2] | qnrB:8/78 | 2008.10–11 Hangzhou | Aquatic environments | Citrobacter freundii |
| qnrS1:2/78 | Escherichia coli | |||
| qnrS2:1/78 | Aeromonas punctata | |||
| qnrA1:8/103qnrB:71/103qnrS1:1/103qnrS2:1/103 | 2008.1–12 Beijing,Shanghai,Hangzhou,Wenzhou | Hospital/sputum,urine and bodily secretions | Citrobacter freundii | |
| [24] | DiscoveriedqnrVC4 | 2008 Jinan | Wastewater of hospital | Aeromonas |
| [35] | qnrS:58/223 | 2010.9–2011.10 Beijing | A tertiary-care Hospital | Klebsiella pneumoniae |
| [36] | qnrA1:2/35qnrA2:2/35qnrB2:2/35qnrB6:6/35qnrS1:13/35 | 2009.9–2012.2 Chongqing | Hospital | Enterobactercloacae |
| [37] | qnrS:21/202 | 2012.3–5 Anhui | From chicken cloacae at four different farms | Escherichia coli |
| [34] | qnrA:2/391qnrB:21/391qnrS:12/391 | 2008.3–2009.11 Jinan | River,effluents of Hospital,wastewater treatment plant | Klebsiella spp.,Escherichia coli,Enterobacter spp.,Proteus spp.,Shigella spp. and Citrobacter spp. |
| [38] | qnrD:12/19 | 2010.8–10 Zhejiang | Intensive care units of hospital | Proteus mirabilis |
| [1] | qnrB:1/307qnrS:8/307 | 1993–2010 NM province in China | Humans(feces and urine samples from healthy volunteers or patients) | Escherichia coli |
| qnrB:5/384qnrS:5/384 | From viscera or feces of diseased or healthy chickens | |||
| qnrA:4/198qnrS:17/198 | From viscera or feces of diseased or healthy pigs | |||
| qnrB:2/89qnrS:4/89 | From viscera or feces of diseased or healthy cattle,dogs,ducks,geese | |||
| qnrB:3/44qnrS:9/44 | Environment on different farms,including surface soil,sewage,drinking water and pond water | |||
| [39] | qnrB2:2/146 | 2005–2011 Anhui | Wounds of patient in hospital | Serratia marcescens |
| qnrS2:2/146 | Sputum of patient in hospital | |||
| [3] | qnrA:2/75qnrS:3/75 | 2009.10–2011.5 Henan | Food (meat product) | Enterobacteriaceae |
| [40] | qnrA:12/76qnrB:25/76qnrS:17/76 | 2012.1–9Wenzhou | ICU of hospital | Escherichia coli |
| [41] | qnrA:1/50qnrB: 5/50qnrS:3/50 | 2008–2009Guangzhou | Clinical specimens from hospital | Escherichia coli |
| qnrA:1/30qnrB: 8/30qnrS:10/30 | Klebsiella pneumoniae | |||
| qnrA:13/18qnrB: 5/18qnrS:4/18 | Enterobacter cloacae | |||
| [43] | qnrB:5/245qnrS:1/245 | 2008.12–2011.12Hengshui,Hebei | Sputum of patient in hospital | Escherichia coli |
| [44] | qnrB4:35/176qnrS1:7/176 | 2008.6–2010.12Zhejiang | Hospital | Escherichia coli |
| [45] | qnrB:10/46 | NM Taizhou,Zhejiang | Hospital | Escherichia coli |
| [46] | qnrB:15/143qnrS:27/143 | 2010.8–2011.8Foshan | From viscera or feces of chicken,goose,ducks,pigs | Escherichia coli |
| [47] | UT qnr:9/100 | 2005.1–2008.4 | Clinical patient in hospital | Shigella |
| [48] | qnrA:30/62qnrB:41/62qnrS:7/62 | 2008.1–12Guangzhou | Clinical patient in hospital | Enterobacter cloacae |
| [49] | qnrA:7/226qnrB:17/226qnrS:10/226 | 2008–2011Guangzhou | Hospital/sputum,urine,wound secretion,blood and cervical samples | Enterobacter cloacae |
| [50] | UT qnr:11/29 | NM Haerbing | Sick or dead chicken from chicken farm | Salmonella |
| [51] | qnrA:5/52qnrB2:11/52qnrB4:1/52 | 2004.1–2006.9Anhui | Clinical specimens from hospital | Citrobacter spp. |
| [52] | qnrA:2/62qnrS:5/62 | NMXinxiang | Food (meat,sea food,vegetables and cooked meat) | Salmonella |
| [53] | qnrA:1/37qnrS:4/37 | 2007.1–2008.12Guangzhou | Clinical patient in hospital | Enterobacter cloacae |
表选项
大多数报道中,qnr基因都是与其他耐药基因共同存在,尤其是β-内酰胺酶基因,而由于其他基因的存在会增强细菌对喹诺酮药物的耐药性,这大大提高了临床对耐药患者治疗的难度,这既是医疗卫生科学不得不面对的挑战,更是关系到我们每个人生命健康的重要问题。
4 小结和展望距qnrA1发现短短十几年的时间,qnr基因在全世界范围内被大量报道,尤其是美国、中国等经济发展快速的地方,蔓延速度十分迅速,这必须引起我们足够的重视。
由于废弃物、垃圾等不断地污染我们赖以生存的水环境,有机物堆积形成了细菌等微生物的理想生存地,它们大量繁殖侵略,在环境的自然选择下优胜劣汰,生存能力越来越顽强。而喹诺酮作为全球通用的一种广谱抗生素,在人们平常消炎抗菌中起着重要的作用,其使用量也一直高居不下。抗生素及耐药基因作为环境污染的新成员,摄入人体后有85%以上会以原药或代谢产物的形式经由病人或动物的排泄物排出体外,进入环境,并通过各种方式污染地下水及生态系统。二者结合造成了耐药细菌的广泛蔓延,我们有必要对此感到担心,而不是马马虎虎。
环境的清洁以及抗生素的规范使用,将是我们必须要走的路。《抗菌药物临床应用管理办法》已由卫生部自2012年8月1日起发布施行,但是我们看到其执行效力并不严格,若相关部门能加强监督管理,相信能取得不错的成绩。锻炼身体,增强抵抗力是最有效、最能根除该问题的方法,新一代青少年羸弱的身体素质是抗生素药物广泛使用的一大助长因子。求木之长者,必固其根本;欲流之远者,必浚其泉源,对于我们每个人而言,加强自身身体锻炼,使自己变得更加强壮,将会大大减少病菌在体内生存繁殖的机会,这便是我们个人对解决细菌耐药问题的最根本贡献。
新药物的开发利用亦是有效的解决途径,也是相关科研工作者们努力的方向。首先,从产抗生素的菌源问题着手,海洋植物和无脊椎动物活性成分的研究越来越受到关注,但是作为地球上>最丰富的微生物资源宝库,海洋资源受到的重视还远远不够。海洋环境具有高盐、高压、低温、寡营养等特点,在长期的进化发展过程中,这些特殊的海洋环境因子使海洋微生物获得不同于陆地微生物的代谢途径,从而为人类发现新菌种和新活性化合物提供了广阔的开发空间,它们也将成为新抗生素的潜在菌源。其次,抗生素作用的靶点的改变也是新型药物开发的一大方向,以前由于认识的不足,细菌中已知靶标的数目太少,然而随着基因组测序工程的发展,科学家已经完成了人类、多种致病与非致病微生物的全基因组序列测定工作,因此以靶标筛选抗生素的障碍被逐渐清除,致病微生物中的所有基因都有可能作为潜在的靶标,这为不同靶标抗生素的开发提供了有力的保障。最后,中药的发展使用亦是空间较广的一大方向,中药由于治病效果较慢、口味较苦而被人们越来越忽视,但是,中药有它独到的好处,副作用小,对环境亦无重大危害。如果能在可能的范围内,使用中药来代替不特别需要的西药,从人体自身和环境两方面都会有较好的结果。
本实验室作为微生物技术国家重点实验室的重要组成部分,我们一直致力于环境微生物以及耐药微生物方面的工作,且在质粒介导的喹诺酮耐药基因的相关研究工作中取得良好成绩,其中,qnrVC4的发现以及水环境qnr基因多样性分析便是我们科研团队的优秀代表,接下来,我们准备进行周围、医院水环境中各种喹诺酮耐药基因(包括qnr基因、aac(6′)-Ib-cr基因、外排泵基因qepA)的定量分析,以及它们跟β-内酰胺酶基因之间是否存在定量关系进行探索研究。然而,我们面临的挑战与日俱增,新的问题也将不断出现,一人之力微不足道,大家必须共同面对耐药细菌这一医疗难题,以求取得更好的成绩,为人们的生活提供更进一步的保障。
参考文献
| [1] | Chen X, Zhang W, Pan W, Yin J, Pan Z, Gao S, Jiao X. Prevalence of qnr, aac(6')-Ib-cr, qepA, and oqxAB in Escherichia coli isolates from humans, animals, and the environment. Antimicrobial Agents and Chemotherapy, 2012, 56(6): 3423-3427. |
| [2] | Zhang R, Ichijo T, Huang YL, Cai JC, Zhou HW, Yamaguchi N, Nasu M, Chen GX. High prevalence of qnr and aac(6')-Ib-cr genes in both water-borne environmental bacteria and clinical isolates of Citrobacter freundii in China. Microbes and Environments, 2012, 27(2): 158-163. |
| [3] | Jiang XB, Yu T, Wu N, Meng HC, Shi L. Detection of qnr, aac(6')-Ib-cr and qepA genes in Escherichia coli isolated from cooked meat products in Henan, China. International Journal of Food Microbiology, 2014, 187: 22-25. |
| [4] | Wang AH, Yang YH, Lu Q, Wang Y, Chen Y, Deng L, Ding H, Deng QL, Zhang H, Wang CQ, Liu L, Xu XW, Wang L, Shen XZ. Presence of qnr gene in Escherichia coli and Klebsiella pneumoniae resistant to ciprofloxacin isolated from pediatric patients in China. BMC Infectious Diseases, 2008, 8: 68. |
| [5] | Xiong ZZ, Wang P, Wei YY, Wang H, Cao HX, Huang HS, Li J. Investigation of qnr and aac(6')-Ib-cr in Enterobacter cloacae isolates from Anhui Province, China. Diagnostic Microbiology and Infectious Disease, 2008, 62(4): 457-459. |
| [6] | Dalhoff A. Global fluoroquinolone resistance epidemiology and implictions for clinical use. Interdisciplinary Perspectives on Infectious Diseases, 2012, 2012: 976273. |
| [7] | Martínez-Martínez L, Pascual A, Jacoby GA. Quinolone resistance from a transferable plasmid. Lancet, 1998, 351(9105): 797-799. |
| [8] | Tran JH, Jacoby GA. Mechanism of plasmid-mediated quinolone resistance. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(8): 5638-5642. |
| [9] | Nordmann P, Poirel L. Emergence of plasmid-mediated resistance to quinolones in Enterobacteriaceae. Journal of Antimicrobial Chemotherapy, 2005, 56(3): 463-469. |
| [10] | Poirel L, Rodriguez-Martinez JM, Mammeri H, Liard A, Nordmann P. Origin of plasmid-mediated quinolone resistance determinant QnrA. Antimicrobial Agents and Chemotherapy, 2005, 49(8): 3523-3525. |
| [11] | Cambau E, Lascols C, Sougakoff W, Bébéar C, Bonnet R, Cavallo JD, Gutmann L, Ploy MC, Jarlier V, Soussy CJ, Robert J. Occurrence of qnrA-positive clinical isolates in French teaching hospitals during 2002-2005. Clinical Microbiology and Infection: the Official Publication of the European Society of Clinical Microbiology and Infection Dieseases, 2006, 12(10): 1013-1020. |
| [12] | Hata M, Suzuki M, Matsumoto M, Takahashi M, Sato K, Ibe S, Sakae K. Cloning of a novel gene for quinolone resistance from a transferable plasmid in Shigella flexneri 2b. Antimicrobial Agents and Chemotherapy, 2005, 49(2): 801-803. |
| [13] | Gay K, Robicsek A, Strahilevitz J, Park CH, Jacoby G, Barrett TJ, Medalla F, Chiller TM, Hooper DC. Plasmid-mediated quinolone resistance in non-Typhi serotypes of Salmonella enterica. Clinical Infectious Diseases: an Official Publication of the Infectious Diseases Society of America 2006, 43(3): 297-304. |
| [14] | Strahilevitz J, Jacoby GA, Hooper DC, Robicsek A. Plasmid-mediated quinolone resistance: a multifaceted threat. Clinical Microbiology Reviews, 2009, 22(4): 664-689. |
| [15] | Torpdahl M, Hammerum AM, Zachariasen C, Nielsen EM. Detection of qnr genes in Salmonella isolated from humans in Denmark. The Journal of Antimicrobial Chemotherapy, 2009, 63(2): 406-408. |
| [16] | Han JE, Kim JH, Cheresca CH Jr, Shin SP, Jun JW, Chai JY, Han SY, Park SC. First description of the qnrS-like (qnrS5) gene and analysis of quinolone resistance-determining regions in motile Aeromonas spp. from diseased fish and water. Research in Microbiology, 2012, 163(1): 73-79. |
| [17] | Veldman K, Kant A, Dierikx C, van Essen-Zandbergen A, Wit B, Mevius D. Enterobacteriaceae resistant to third-generation cephalosporins and quinolones in fresh culinary herbs imported from Southeast Asia. International Journal of Food Microbiology,2014, 177: 72-77. |
| [18] | Jacoby GA, Walsh KE, Mills DM, Walker VJ, Oh H, Robicsek A, Hooper DC. qnrB, another plasmid-mediated gene for quinolone resistance. Antimicrobial Agents and Chemotherapy, 2006, 50(4): 1178-1182. |
| [19] | Wang MH, Guo QL, Xu XG, Wang XY, Ye XY, Wu S, Hooper DC, Wang MG. New plasmid-mediated quinolone resistance gene, qnrC, found in a clinical isolate of Proteus mirabilis. Antimicrobial Agents and Chemotherapy, 2009, 53(5): 1892-1897. |
| [20] | Cavaco LM, Hasman H, Xia S, Aarestrup FM. qnrD, a novel gene conferring transferable quinolone resistance in Salmonella enterica serovar Kentucky and Bovismorbificans strains of human origin. Antimicrobial Agents and Chemotherapy, 2009, 53(2): 603-608. |
| [21] | Abgottspon H, Zurfluh K, Nüesch-Inderbinen M, Hächler H, Stephan R. Quinolone resistance mechanisms in Salmonella enterica serovars Hadar, Kentucky, Virchow, Schwarzengrund, and 4, 5, 12: i: -, isolated from humans in Switzerland, and identification of a novel qnrD variant, qnrD2, in S. Hadar. Antimicrobial Agents and Chemotherapy, 2014, 58(6): 3560-3563. |
| [22] | Fonseca EL, Vicente AC. Epidemiology of qnrVC alleles and emergence out of the Vibrionaceae family. Journal of Medical Microbiology, 2013, 62(Pt 10): 1628-1630. |
| [23] | Fonseca éL, Dos Santos Freitas F, Vieira VV, Vicente ACP. New qnr gene cassettes associated with superintegron repeats in Vibrio cholerae O1. Emerging Infectious Diseases, 2008, 14(7): 1129-1131. |
| [24] | Xia RR, Guo XH, Zhang YZ, Xu H. qnrVC-like gene located in a novel complex class 1 integron harboring the ISCR1 element in an Aeromonas punctata strain from an aquatic environment in Shandong Province, China. Antimicrobial Agents and Chemotherapy, 2010, 54(8): 3471-3474. |
| [25] | Liu M, Wong MHY, Chen S. Mechanisms of fluoroquinolone resistance in Vibrio parahaemolyticus. International Journal of Antimicrobial Agents, 2013, 42(2): 187-188. |
| [26] | Liu M, Wong MHY, Chen S. Molecular characterisation of a multidrug resistance conjugative plasmid from Vibrio parahaemolyticus. International Journal of Antimicrobial Agents, 2013, 42(6): 575-579. |
| [27] | Kumar P, Thomas S. Presence of qnrVC3 gene cassette in SXT and class 1 integrons of Vibrio cholerae. International Journal of Antimicrobial Agents, 2011, 37(3): 280-281. |
| [28] | Po KHL, Wong MHY, Chen S. Identification and characterisation of a novel plasmid-mediated quinolone resistance gene, qnrVC7, in Vibrio cholerae of seafood origin. International Journal of Antimicrobial Agents, 2015, 45(6): 667-668. |
| [29] | Pan XS, Fisher LM. Targeting of DNA gyrase in Streptococcus pneumoniae by sparfloxacin: selective targeting of gyrase or topoisomerase IV by quinolones. Antimicrobial Agents and Chemotherapy, 1997, 41(2): 471-474. |
| [30] | Hoshino K, Kitamura A, Morrissey I, Sato K, Kato J, Ikeda H. Comparison of inhibition of Escherichia coli topoisomerase IV by quinolones with DNA gyrase inhibition. Antimicrobial Agents and Chemotherapy, 1994, 38(11): 2623-2627. |
| [31] | Khodursky AB, Zechiedrich EL, Cozzarelli NR. Topoisomerase IV is a target of quinolones in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America, 1995, 92(25): 11801-11805. |
| [32] | Khodursky AB, Cozzarelli NR. The mechanism of inhibition of topoisomerase IV by quinolone antibacterials. The Journal of Biological Chemistry, 1998, 273(42): 27668-27677. |
| [33] | Tran JH, Jacoby GA, Hooper DC. Interaction of the plasmid-encoded quinolone resistance protein QnrA with Escherichia coli topoisomerase IV. Antimicrobial Agents and Chemotherapy, 2005, 49(7): 3050-3052. |
| [34] | Xia RR, Ren Y, Xu H. Identification of plasmid-mediated quinolone resistance qnr genes in multidrug-resistant Gram-negative bacteria from hospital wastewaters and receiving waters in the Jinan area, China. Microbial Drug Resistance, 2013, 19(6): 446-456. |
| [35] | Li B, Yi Y, Wang Q, Woo PCY, Tan L, Jing H, Gao GF, Liu CH. Analysis of drug resistance determinants in Klebsiella pneumoniae isolates from a tertiary-care hospital in Beijing, China. PLoS One, 2012, 7(7): e42280. |
| [36] | Huang SF, Dai W, Sun S, Zhang XJ, Zhang LP. Prevalence of plasmid-mediated quinolone resistance and aminoglycoside resistance determinants among carbapeneme non-susceptible Enterobacter cloacae. PLoS One, 2012, 7(10): e47636. |
| [37] | Li L, Wang BT, Feng S, Li JN, Wu CM, Wang Y, Ruan XC, Zeng MH. Prevalence and characteristics of extended-spectrum β-lactamase and plasmid-mediated fluoroquinolone resistance genes in Escherichia coli isolated from chickens in Anhui province, China. PLoS One, 2014, 9(8): e104356. |
| [38] | Hu YY, Cai JC, Zhang R, Zhou HW, Sun Q, Chen GX. Emergence of Proteus mirabilis harboring blaKPC-2 and qnrD in a Chinese Hospital. Antimicrobial Agents and Chemotherapy, 2012, 56(5): 2278-2282. |
| [39] | Yang HF, Cheng J, Hu LF, Ye Y, Li JB. Plasmid-mediated quinolone resistance in extended-spectrum-β-lactamase- and AmpC β-lactamase-producing Serratia marcescens in China. Antimicrobial Agents and Chemotherapy, 2012, 56(8): 4529-4531. |
| [40] | Cai FF, Xu G, Luo S, Ma YY. The prevalence of qnr gene and drug resistance in Escherichia coli. Chinese Journal of Microecology, 2013, 25(10): 1149-1151, 1167. (in Chinese)蔡芳芳, 许刚, 罗盛, 马泳泳. ICU病区大肠埃希菌中qnr基因的分布与耐药性的研究. 中国微生态学杂志, 2013, 25(10): 1149-1151, 1167. |
| [41] | Zhang WH, Ye HF, Yang YM, Chen HL, Chen YZ, Li HT. Prevalence of resistance gene (qnr) in clinical isolated Enterobacteriaceae and analysis of drug resistance. Chinese Journal of Nosocomiology, 2012, 22(7): 1321-1324. (in Chinese)张伟红, 叶惠芬, 杨银梅, 陈惠玲, 陈英姿, 李焕庭. 肠杆菌科细菌qnr耐药基因的流行现状及耐药分析. 中华医院感染学杂志, 2012, 22(7): 1321-1324. |
| [42] | Zhu HQ, Liao XP, Chen CX, Wang XM, Sun J, Sun Y, Li L, Zhang MJ, Liu YH. Detection of plasmid-mediated quinolone resistance in clinical isolates of Escherichia coli from pet animals. Scientia Agricultura Sinica, 2010, 43(16): 3447-3454. (in Chinese) 朱恒乾, 廖晓萍, 陈朝喜, 王秀梅, 孙坚, 孙迎, 李亮, 张美君, 刘雅红. 宠物源大肠杆菌质粒介导喹诺酮类耐药基因流行性检测. 中国农业科学, 2010, 43(16): 3447-3454. |
| [43] | Hu JY, Du HL, Qiao YM, Guo LF, Yang SJ, Jin YH. Analysis of the antibiotics susceptibility and qnr gene distrabution on Escherichia coli isolated from clinical. Journal of Clinical Pulmonary Medicine, 2013, 18(2): 290-292. (in Chinese)胡景玉, 杜红丽, 乔艳梅, 郭连峰, 杨世金, 金玉怀. 大肠埃希菌药敏分析及qnr基因分布情况研究. 临床肺科杂志, 2013, 18(2): 290-292. |
| [44] | Guo ML, Pan XY, Zeng SF. Detection of plasmid-mediated quinolone resistance determinant qnr in Escherichia coli isolates and its susceptibility. Chinese Journal of Nosocomiology, 2012, 22(9): 1765-1768. (in Chinese) 郭美丽, 潘孝勇, 曾松芳. 大肠埃希菌中质粒介导喹诺酮类耐药基因的检测及其耐药性分析. 中华医院感染学杂志, 2012, 22(9): 1765-1768. |
| [45] | Shu HY, Sun GQ, Fu Y, Wang K, Wang T, Qi YX. Detection of qnrB genes for Escherichia coli and study of resistance mechanisms. Journal of Tropical Medicine, 2014, 14(9): 1131-1133, 1137. (in Chinese) 舒红云, 孙国强, 傅媛, 王凯, 王婷婷, 戚永孝. 大肠埃希氏菌的qnrB基因检测与耐药机制研究. 热带医学杂志, 2014, 14(9): 1131-1133. |
| [46] | Zhuang N, Chen XY, Yue L, Liao XP, Liu YH. Detection of PMQR gene in Escherichia coli isolated from animals. Scientia Agricultura Sinica, 2012, 45(10): 2052-2057. (in Chinese) 庄娜, 陈雪影, 岳磊, 廖晓萍, 刘雅红. 动物源大肠杆菌PMQR基因流行性检测. 中国农业科学, 2012, 45(10): 2052-2057. |
| [47] | Wang YP, Ying CM, Zhang HM, Ye YQ, Yu JP. The determination of plamid-mediated qnr gene in quinolone-resistant Shigella. Laboratory Medicine, 2011, 26(10): 645-647. (in Chinese) 汪雅萍, 应春妹, 张灏旻, 叶杨芹, 于嘉屏. 氟喹诺酮耐药志贺菌中质粒介导qnr基因的检测. 检验医学, 2011, 26(10): 645-647. |
| [48] | Ye HF, Chen HL, Liu P, Chen YY. Survey of prevalence of Enterobacter cloacae qnr gene. China Tropical Medicine, 2011, 11(3): 289-290. (in Chinese)叶惠芬, 陈惠玲, 刘平, 陈玉怡. 广州地区阴沟肠杆菌Qnr基因流行调查. 中国热带医学, 2011, 11(3): 289-290. |
| [49] | Zhou Q, Luo YS, Deng CH, Zhang W, Li MF, Cao KY. Detection and analysis of plamid-mediated qnr gene in Enterobacter cloacae from Guangzhou. Chinese Journal of Clinical Laboratory Science, 2012, 30(9): 704-705. (in Chinese) 周强, 罗娅莎, 邓晨晖, 张文, 黎孟枫, 曹开源. 广州地区阴沟肠杆菌中质粒介导喹诺酮类耐药基因的检测及分析. 临床检验杂志, 2012, 30(9): 704-705. |
| [50] | Liu FP, Zhao YL, Li CW, Liu LX, Li R, Luo PZ, Lu SL, Zhang XY. Drug resistance gene detection and the resistance correlation analysis in Salmonella isolated from chickens. Chinese Journal of Preventive Veterinary Medicine, 2013, 35(8): 627-630. (in Chinese) 刘芳萍, 赵玉林, 李昌文, 刘立新, 李睿, 罗鹏志, 卢斯亮, 张秀英. 鸡源性沙门氏菌耐药基因检测与耐药相关性分析. 中国预防兽医学报, 2013, 35(8): 627-630. |
| [51] | Zhong T, Xu W, Xu YH. Detection of qnr genotypes in clinical isolated citrobacter strains. Acta Universitatis Medicinalis Anhui, 2010, 45(2): 261-265. (in Chinese) 钟涛, 许伟, 徐元宏. 临床分离枸橼酸杆菌qnr基因型检测. 安徽医科大学学报, 2010, 45(2): 261-265. |
| [52] | Liu GS, Yu T. Study on antimicrobial resistance and plasmid-mediated quinolone resistance of foodborne Salmonella isolates. Biotechnology Bulletin, 2014, 46(8): 202-207. (in Chinese) 刘贵深, 于涛. 食源性沙门氏菌耐药性及质粒介导喹诺酮耐药基因检测. 生物技术通报, 2014, 46(8): 202-207. |
| [53] | Huang B, Chen C, Lu HW, Chen LD. Study on drug resistance of Enterobacter cloacae and detection its quinolone-resistant genes. China Tropical Medicine, 2011, 11(7): 785-788. (in Chinese)黄彬, 陈茶, 卢汉威, 陈利达. 阴沟肠杆菌的耐药性及对喹诺酮类耐药基因的检测. 中国热带医学, 2011, 11(7): 785-788. |
