0 引言
【研究意义】陆地棉(Gossypium hirsutum,AADD,2n=4x=52)为异源四倍体棉种,是世界范围内重要的经济作物,约占全球棉花种植总面积的95%[1]。陆地棉由亚洲棉(Gossypium arboreum,AA)和雷蒙德氏棉(Gossypium raimondii,DD)杂交加倍而来[2],在人工驯化选择下,其性状不断按照人类需求的方向进化发展,包括高产、优质、较强耐逆性以及广泛的适应性[3]。SNP(single nucleotide polymorphism)即单核苷酸多态性,是指在基因组水平上由于单个核苷酸变异(转换、颠换、缺失和插入等)引起的DNA序列多态性。SNP在基因组中分布率极高,是很多物种基因组的最常见变异形式[4]。作为最新一代的分子标记,SNP标记由于具有数量多、分布广、遗传的稳定性、易于自动化操作等优势,已成为继SSR标记之后最具潜力的第3代分子标记,近年来已被广泛应用于农作物遗传图谱构建、遗传多样性分析、品种鉴定和分子标记辅助选择育种等[5,6,7],逐渐成为主流的分子标记。【前人研究进展】随着棉花D基因组[2,3,4,5,6,7,8]、A基因组[9]和AD基因组[10,11]测序工作的相继完成以及新一代测序技术、基因芯片技术的迅速发展,加快了分子标记技术的应用,尤其是SNP标记的大规模开发与研究,将对陆地棉复杂性状的基因定位以及优良品种的选育起到巨大的推动作用[12,13]。随着SNP标记技术的逐渐完善,相应的SNP检测技术也不断发展,而SNP芯片作为一种集高通量、微型化和自动化等优点为一体的检测手段,其基本原理是通过将待测样本DNA与固定在载体上的密集的寡核苷酸探针阵列进行等位基因特异性杂交反应,根据释放的荧光信号有无和强弱确定SNP位点。基因组序列中存在4种碱基,SNP可以是二等位多态性,也可以是三或四等位多态性,但实际情况中常以二等位多态性为主,因此便于估计其等位基因频率并进行基因型的自动化分析[14]。目前商业化的SNP芯片主要有两类,分别由美国Affymetrix和Illumina公司开发,其中最具代表性的是Illumina公司开发的Goldengate以及Infinium高通量分析技术。大规模、高通量SNP芯片检测最先在人类群体遗传学研究中得到广泛应用,并在人类关联分析上取得较大进展[15,16]。SNP芯片也被应用于一些家畜全基因组关联分析、QTL定位、候选基因筛选[17,18]。而在棉花中,HULSE-KEMP等[19]基于Infinium技术成功开发出首款包含45 104个陆地棉种内的SNP标记和17 954个陆地棉与其他棉种的种间SNP标记的高密度(63K)芯片,单张芯片可一次性检测24个样品,并用1 156个样本对其进行了验证,分析出38 822个多态性标记。这为棉花中SNP标记的真正大规模检测开辟了先河[20,21]。目前,该芯片已被用于棉花高密度遗传图谱构建和纤维品质、产量、农艺性状的QTL定位[22,23]。另外,高效的数据分析工具对SNP的鉴别是必不可少的。Illumina公司成功开发的GenomeStudio软件,可以分析微阵列和测序产生的数据。对于研究人员来说,GenomeStudio软件能够实现多种应用中的生物变异关联,并将结果以图形显示,这种分型方案适用于所有二倍体物种,在水稻、玉米等植物遗传图谱构建及多样性分析等方面已得到了广泛的应用[24,25]。【本研究切入点】然而,对于异源四倍体的棉花栽培种,亚组间的同源染色体与亚组内的重复序列导致大部分标记具有多拷贝的情况,从而给SNP的准确分型带来了种种困难。通过筛选具有单拷贝特性的SNP位点,可将复杂的多倍体分型转化为二倍体分型,是一种行之有效的手段。【拟解决的关键问题】结合四倍体陆地棉TM-1参考基因组序列信息,筛选一批基因组特异的SNP,推动SNP标记在棉花种质资源鉴定、群体进化分析以及分子标记辅助育种等方面的应用。1 材料与方法
试验于2014年在河北农业大学棉花遗传育种研究室完成。1.1 试验材料
供试材料为遗传背景来源广泛的719份陆地棉种质资源[22],由河北农业大学棉花遗传育种研究室收集保存,其中包括588份来自中国不同省份的陆地棉品种,以及131份来自美国、前苏联等其他国家的陆地棉品种。1.2 DNA提取与质量控制
采用改良CTAB法[26]提取719份材料幼嫩叶片基因组DNA,用0.8%琼脂糖电泳和Nano Drop 2000分光光度计检测所提取DNA的质量,以满足SNP检测的质量要求:即琼脂糖电泳显示DNA条带单一,没有明显弥散;紫外分光光度计检测A260/A280介于1.8—2.0,且DNA浓度>50 ng·μL-1。1.3 SNP检测与数据分析
采用Illumina公司开发的CottonSNP63K芯片,包括63 058个SNP标记。SNP检测参照标准实验流程(基于光纤微珠芯片的Infinium技术)进行,用iScan芯片扫描仪对杂交结果进行扫描,获得原始数据。利用GenomeStudio软件对芯片扫描所获得原始数据进行基因型数据质量控制分析,获得待测四倍体棉花样品SNP位点的基因型。根据中国农业科学院棉花研究所版本Gossypium hirsutum(AD1)genome BGI v1.0与南京农业大学版本G. hirsutum(AD1)genome NBI v1.1两个陆地棉TM-1基因组为参考序列,对CottonSNP63K芯片各SNP位点的侧翼序列分别进行全基因组BLAST比对分析,以筛选基因组特异SNP位点,利用PowerMarker v3.25软件[27]进行遗传分析。2 结果
2.1 四倍体棉花SNP芯片分型
基于CottonSNP63K芯片对供试的719份棉花材料进行基因分型,统计分析结果表明,SNP位点主要表现为以下几种类型:无检出信号的SNP位点(图1)、无多态性的SNP位点(图2)和具有多态性的SNP位点(图3)。而具有多态性的SNP位点分型结果主要表现为以下3种类型:第Ⅰ种是双位点SNP,即在基因组上具有两个拷贝,且这两个拷贝一般成对分布于两条部分同源染色体上,具体可表现为双位点单态(两个拷贝中仅一个拷贝表现出多态)和双位点双态(两个拷贝均表现出多态);其中,双位点单态SNP可Cluster为3种基因型,3种基因型既有可能位于分型图的左半侧(图3-A,从左到右基因型依次为AAAA、AAAB和AABB),也有可能位于分型图的右半侧(图3-B,从左到右基因型依次为AABB、ABBB和BBBB);而双位点双态可Cluster为5种基因型(图3-C,从左到右基因型依次为AAAA、AAAB、AABB、ABBB和BBBB)。第Ⅱ种是多位点SNP,即在基因组上具有两个以上的拷贝,具体表现为多位点多态(多个位点均表现出多态性)和多位点单态(多个位点中仅一个位点表现出多态);图3-D所示为三位点三态的SNP分型图,可Cluster为7种基因型,从左到右基因型依次为AAAAAA、AAAAAB、AAAABB、AAABBB、AABBBB、ABBBBB及BBBBBB。图3-E为多位点单态的SNP分型图,可Cluster为3种基因型,且3种基因型距离很近,难也分辨。第Ⅲ种是单位点SNP(基因组特异SNP),即在基因组上只有一个拷贝,这种基因组特异SNP具有二倍体作物SNP的特性,分型相对简单,可Cluster为3种基因型(图3-F),从左到右基因型依次为AA、AB和BB型,这种基因组特异SNP位点通过软件即可实现自动准确的分型,无需手动对Cluster结果进行优化调整,非常适合于品种指纹图谱的构建。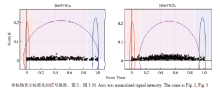 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图 1无检出信号的SNP位点分型图
-->Fig. 1Genotyping patterns of SNP markers without detectable signal
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2无多态性的SNP位点分型图
-->Fig. 2Genotyping patterns of SNP markers without polymorphism
-->
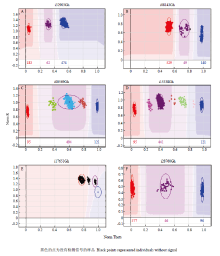 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3多态性SNP位点分型图
-->Fig. 3Genotyping patterns of polymorphic SNP markers
-->
2.2 全基因组特异SNP的筛选
BLAST比对结果表明,中国农业科学院棉花研究所TM-1基因组版本比对获得特异SNP标记为5 474个,占8.6%,其中4 186个SNP标记位于拼接的染色体上,占76.5%,染色体分布情况见表1,At亚组平均每条染色体具有114个基因组特异SNP标记,而Dt亚组平均每条染色体具有208个特异SNP标记,即Dt亚组所含有的特异SNP约为At亚组的两倍。特异SNP标记最多的是Dt_chr9号染色体,多达592个,而对应的部分同源染色体At_chr9在At亚组上也含有最多的单位点SNP标记。特异SNP标记数量最少的是At_chr5,仅有67个。Table 1
表1
表1特异SNP染色体分布情况(BGIv1.0)
Table 1Chromosome distribution of genome-specific SNP markers (BGIv1.0)
| At亚组染色体 Chromosomes of At subgenome | 标记数(个) The number of SNP markers | Dt亚组染色体 Chromosomes of Dt subgenome | 标记数(个) The number of SNP markers | |
|---|---|---|---|---|
| At_chr1 | 124 | Dt_chr1 | 269 | |
| At_chr2 | 104 | Dt_chr2 | 104 | |
| At_chr3 | 71 | Dt_chr3 | 90 | |
| At_chr4 | 187 | Dt_chr4 | 101 | |
| At_chr5 | 67 | Dt_chr5 | 217 | |
| At_chr6 | 79 | Dt_chr6 | 132 | |
| At_chr7 | 76 | Dt_chr7 | 244 | |
| At_chr8 | 85 | Dt_chr8 | 286 | |
| At_chr9 | 237 | Dt_chr9 | 592 | |
| At_chr10 | 77 | Dt_chr10 | 133 | |
| At_chr11 | 124 | Dt_chr11 | 202 | |
| At_chr12 | 103 | Dt_chr12 | 202 | |
| At_chr13 | 145 | Dt_chr13 | 135 | |
| 合计/平均Total/Mean | 1479/114 | 合计/平均 Total/Mean | 2707/208 |
新窗口打开
利用南京农业大学TM-1基因组版本比对获得的基因组特异SNP标记仅为1 850个,占2.9%,其中1 653个SNP标记位于拼接的染色体上,占89.4%,染色体分布情况见表2。At亚组平均每条染色体具有61个SNP标记,Dt亚组平均每条染色体具有66个SNP标记,即At亚组与Dt亚组的特异SNP平均数量是相当的。单位点SNP标记最多的是D05号染色体,为121个,最少的是D11号染色体,仅33个。
Table 2
表2
表2特异SNP染色体分布情况(NBI v1.1)
Table 2Chromosome distribution of genome-specific SNP markers (NBI v1.1)
| At亚组染色体 Chromosomes of At subgenome | 标记数(个) The number of SNP markers | Dt亚组染色体 Chromosomes of Dt subgenome | 标记数(个) The number of SNP markers | |
|---|---|---|---|---|
| A01 | 70 | D01 | 69 | |
| A02 | 46 | D02 | 92 | |
| A03 | 38 | D03 | 39 | |
| A04 | 38 | D04 | 39 | |
| A05 | 69 | D05 | 121 | |
| A06 | 44 | D06 | 76 | |
| A07 | 53 | D07 | 74 | |
| A08 | 98 | D08 | 94 | |
| A09 | 54 | D09 | 41 | |
| A10 | 85 | D10 | 67 | |
| A11 | 65 | D11 | 33 | |
| A12 | 73 | D12 | 46 | |
| A13 | 65 | D13 | 64 | |
| 合计/平均Total/Mean | 798/61 | 合计/平均 Total/Mean | 855/66 |
新窗口打开
以两个陆地棉TM-1基因组版本作为参考序列分别Blast比对获得的特异SNP标记中,共有的特异SNP为1 594个,将这些SNP标记在两个参考基因组上的染色体物理位置信息进行比对,结果表明(表3),13对部分同源染色体中,9对部分同源染色体的对应关系在两个基因组版本中是吻合的,4对部分同源染色体的对应关系在两个版本中不完全吻合,这可能是由于At亚组内的At_chr3与At_chr5,At_chr7与At_chr1,Dt亚组内的Dt_chr5与Dt_chr3,Dt_chr1与Dt_chr7存在较高的亚组内同源性。两个陆地棉TM-1参考基因组比对结果的差异可能是由于基因组序列组装过程中所使用的遗传群体或算法差异等因素所导致。
Table 3
表3
表3两个陆地棉TM-1基因组版本染色体编号对应关系
Table 3Corresponding relationship of chromosomes from two TM-1 genome versions
| 南京农大版本 At亚组 At subgenome of NBI v1.1 | 中棉所版本 At亚组 At subgenome of BGIv1.0 | 南京农大版本 Dt亚组 Dt subgenome of NBI v1.1 | 中棉所版本 Dt亚组 Dt subgenome of BGIv1.0 |
|---|---|---|---|
| A01 | At_chr2 | D01 | Dt_chr2 |
| A02 | At_chr3 | D02 | Dt_chr5 |
| A03 | At_chr5 | D03 | Dt_chr3 |
| A04 | At_chr12 | D04 | Dt_chr12 |
| A05 | At_chr9 | D05 | Dt_chr9 |
| A06 | At_chr10 | D06 | Dt_chr10 |
| A07 | At_chr7 | D07 | Dt_chr1 |
| A08 | At_chr4 | D08 | Dt_chr4 |
| A09 | At_chr6 | D09 | Dt_chr6 |
| A10 | At_chr11 | D10 | Dt_chr11 |
| A11 | At_chr1 | D11 | Dt_chr7 |
| A12 | At_chr8 | D12 | Dt_chr8 |
| A13 | At_chr13 | D13 | Dt_chr13 |
新窗口打开
2.3 核心SNP位点的筛选与指纹图谱构建
以两个陆地棉TM-1基因组版本所共有的1 594个特异SNP作为候选标记,利用719份材料对这些SNP位点进行全面评估与筛选。2.3.1 SNP分型效果评估 GenomeStudio软件获得
的GenTrain score值为0—1.00,该数值反应了3种基因型Cluster分型结果的准确性,score值越大,数据点的准确性与可靠性越高。1 594个特异SNP的GenTrain score值统计结果如下(图 4):score值≤0.60的SNP位点仅有133个,占8.3%;score值在0.6—0.7的SNP位点有92个,占5.8%;score值>0.7的位点有1 369个,占85.9%,表明绝大部分基因组特异SNP分型效果较为理想。
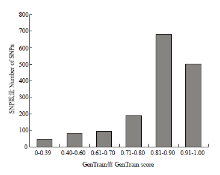 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4基于1 594个特异SNP的GenTrain值分布情况
-->Fig. 4Distribution of 1 594 genome-specific SNPs based on GenTrain score
-->
2.3.2 SNP分型检出率评估 Call frequency值反映受检所有样品在每个SNP位点的检出率,1 594个特异SNP的call frequency值统计结果如图5所示,在719份材料中call frequency值≥0.95共有1 520个,其中完全检出的有1 466个,占92%;call frequency值在0.01—0.94的SNP位点有31个,占1.9%;完全未检出的SNP位点有43个,仅占2.7%,表明绝大部分基因组特异SNP具有极好的稳定性与重复性。
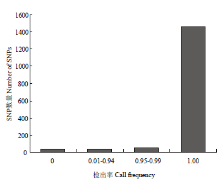 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5基于1 594个特异SNP的检出率分布情况
-->Fig. 5Distribution of 1 594 genome-specific SNPs based on call frequency
-->
2.3.3 SNP多态性评估 最小等位基因频率(minor allele frequency,MAF)可反映各SNP位点在受检样品中的遗传多样性水平,1 594个特异SNP的MAF值<0.2的SNP位点达1 037个,占65.1%;MAF值≥0.2的SNP位点有557个,仅占34.9%(图6),表明陆地棉遗传背景比较狭窄,大部分SNP位点多样性较低。
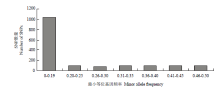 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6基于1 594个特异SNP的最小等位基因频率分布情况
-->Fig. 6Distribution of 1 594 genome-specific SNPs based on minor allele frequency
-->
2.3.4 核心SNP位点筛选 基于以上评估结果,综合考虑分型效果、检出率及多态性3个评价指标,即筛选score值≥0.7,call frequency值≥0.95,且MAF值≥0.2的SNP位点,共获得471个分型效果理想、检出率高且多态性较高的特异SNP位点(定义为核心SNP位点)。在471个SNP位点中,去除41个位于scaffold片段上的位点,其余430个位于染色体上。进一步考虑到标记间的连锁程度,剔除物理位置邻近且遗传多样性与分型结果完全相同的37个连锁标记,最后获得393个核心SNP位点,平均每条染色体为15个,染色体具体分布情况见图7。A亚组中,核心位点最多的是A01,达28个,最少的是A06,仅5个;D亚组中,核心位点最多的是D02,达30个,最少的是D03,仅4个(表4)。核心SNP位点在染色体上的不对称分布可能是由于染色体的大小不同、遗传重组率差异等因素造成的。
Table 4
表4
表4393个核心SNP位点基于陆地棉TM-1基因组(NBI v1.1)的分布情况
Table 4Distribution of 393 core genome-specific SNPs based on TM-1 genome
| At亚组 At subgenome | 核心SNP数量 The number of core SNPs | Dt亚组 Dt subgenome | 核心SNP数量 The number of core SNPs | |
|---|---|---|---|---|
| A01 | 28 | D01 | 18 | |
| A02 | 7 | D02 | 30 | |
| A03 | 11 | D03 | 4 | |
| A04 | 7 | D04 | 13 | |
| A05 | 26 | D05 | 11 | |
| A06 | 5 | D06 | 8 | |
| A07 | 15 | D07 | 22 | |
| A08 | 16 | D08 | 23 | |
| A09 | 25 | D09 | 10 | |
| A10 | 17 | D10 | 23 | |
| A11 | 14 | D11 | 5 | |
| A12 | 16 | D12 | 6 | |
| A13 | 23 | D13 | 10 | |
| 合计/平均Total/Mean | 210/16 | 合计/平均 Total/Mean | 183/14 |
新窗口打开
Table 5
表5
表5核心SNP位点评价情况
Table 5Evaluation of genome-specific core SNP loci
| SNP评价指标 Evaluation index | 最大值 Maximum | 最小值 Minimum | 平均值 Mean |
|---|---|---|---|
| GenTrain 值GenTrain score | 1.00 | 0.70 | 0.86 |
| 检出率Call frequency | 1.00 | 0.97 | 0.99 |
| 最小等位基因频率 Minor allele frequency (MAF) | 0.50 | 0.20 | 0.36 |
| 基因多样性Gene diversity | 0.50 | 0.32 | 0.45 |
| 多态性信息含量 Polymorphism information content (PIC) | 0.37 | 0.27 | 0.35 |
新窗口打开
2.3.5 品种特征指纹图谱构建 利用393个核心SNP组合构建了719份资源材料的特征DNA指纹图谱,每行代表一个品种,每列代表一个SNP标记(图8),结果表明,除个别材料之间遗传背景高度相似、基因型完全一致外,97%以上的材料之间均能实现准确有效的鉴别。基于GenomeStudio软件的基因型数据统计结果显示(表5),平均score值达0.9,call frequency值达1.00,MAF值达0.4;基于PowerMarker统计结果(表5),gene diversity平均值为0.45,PIC平均值为0.35。表明这套核心SNP标记同时具备理想的分型效果、高检出率及高多态性的优点,完全满足棉花品种DNA身份鉴定对高质量标记的要求,可以应用于大量样品的指纹数据库构建及遗传多样性分析。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图7393个核心SNP位点的染色体分布图
-->Fig. 7Chromosome distribution of 393 core SNP loci
-->
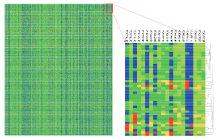 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图8品种特征指纹图谱的总体图与局部放大图
-->Fig. 8Global and local enlarged image of variety DNA fingerprints
-->
3 讨论
SSR标记在品种指纹图谱构建方面一直发挥着重要作用[28,29]。随着基因组学的快速发展,重要农作物的指纹图谱构建开始倾向于应用SNP标记,两种标记技术均适合品种指纹分析,各自具有优缺点,且可优势互补。SSR标记的优势表现为:(1)SSR标记一般不受选择压的影响,为中性变异位点,属于最适合品种鉴定的标记;(2)就单个位点而言,SSR比SNP展示了更高的多态性;(3)SSR技术相对成熟,研究基础较强,易推广应用;(4)单个样品检测成本低。而SSR标记技术也存在两个方面的局限性:(1)不同平台之间数据不能直接比较、整合,需要设立参照样品;(2)检测通量相对较低,且引物位点数量增加时,检测工作量和成本都随之增加。相比之下,SNP标记技术的优势:(1)易实现数据间比较整合,代表基因组中最小的遗传变异单元,并且数据统计相对简单;(2)易实现高通量检测,位点检测通量可达成千上万。当前SNP标记技术局限性主要为仪器成本较高,推广应用较难。因此,应继续发挥SSR标记技术的作用,并积极研发推进SNP检测技术。目前,SNP检测技术已被国际种子检验协会(ISTA)、国际植物新品种保护联盟(UPOV)、国际种子联盟(ISF)等国际组织推荐为品种身份鉴定的辅助方法[30,31]。SNP标记检测手段较多,目前主流的SNP分型技术均是基于高通量的检测平台,主要包括位点高通量的芯片检测平台与样品高通量的检测平台,如本研究所采用的棉花CottonSNP63K芯片是由美国、澳大利亚等国家的研究人员与Illumina公司共同合作开发的全球第一款商业化的棉花SNP芯片产品,一次试验即可实现对棉花全基因组63 058个SNP位点的同时检测,所获得的遗传信息数据量是SSR标记所远不能及的。目前该芯片产品已被应用于棉花全基因组关联分析、高密度遗传连锁图谱的绘制及QTL定位等研究中[22,23]。
SNP标记高通量的检测技术与数据分析的实现主要是基于其二等位变异的遗传特性:利用两种不同的荧光基团分别标记两种不同的等位变异,通过荧光检测系统,即可实现两种纯合与一种杂合基因型的有效鉴别,这种分型方案适用于所有二倍体物种,在水稻、玉米等植物遗传图谱构建及多样性分析等方面已得到了广泛的应用[24,25]。然而,对于多倍体作物,亚组间的同源染色体与亚组内的重复序列导致大部分标记具有多拷贝的特性,多拷贝的特性给SNP的准确分型带来了诸如上述的种种困难,而通过筛选具有单拷贝特性的SNP位点,可将复杂的多倍体分型转化为二倍体分型,是一种有效的手段。本研究结合两个已发表的四倍体陆地棉参考基因组为参考序列,比较两个基因组中共同存在的SNP位点,避免不同基因组组装上差异,使得到的SNP位点更可靠,最终筛选出一批基因组特异的SNP。然而,由于陆地棉A亚组与D亚组具有极高的同源性,导致特异SNP数量不足10%,且符合品种鉴定需求的高质量标记更少。由于目前陆地棉四倍体基因组测序工作才刚完成[10,11],可供筛选的SNP标记数量仍然有限,为满足大规模检测与指纹数据库构建的需要,还需进行大量SNP标记的开发与筛选工作[32],以达到理想的品种鉴定效果。同时,随着棉花功能基因组学和基因工程研究的快速发展,众多的已知功能基因将得到克隆和测序。而SNP标记相比其他分子标记,与功能基因的关联度更高,更容易开发到与性状相关的功能标记,从而将标记和性状联系起来,为SNP标记在棉花品种鉴定中的应用展现了更加广阔的前景。
4 结论
利用CottonSNP63K芯片对719份陆地棉种质资源进行SNP基因分型,筛选出393个基因组特异的SNP,并进一步利用这些核心SNP构建了719份资源材料的特征DNA指纹图谱。The authors have declared that no competing interests exist.
