0 引言
【研究意义】棉花是中国重要的经济作物之一,改善棉花光合作用和延缓棉花衰老是提高棉花产量的有效途径。而叶片的叶绿素含量既能直接影响棉花的光合效率,也是衡量棉花衰老程度的重要指标[1]。有研究表明,植株衰老阶段的叶绿素降解特征,是影响作物高产的重要指标之一[2-3]。因此,对棉花叶片叶绿素含量相关的QTL位点进行基因定位,挖掘其优异等位变异,对发展棉花高产育种具有重要意义。【前人研究进展】目前,连锁分析已经应用于棉花叶绿素含量相关的QTL定位研究。SONG等[4]、SARANGA等[5]分别通过海陆杂交组合BC群体和F2群体各检测到2个和7个叶绿素含量相关的QTL;宋美珍[6]用叶绿素含量等生化指标研究短季棉早熟不早衰的遗传机制,发现早熟不早衰类型的亲本的叶绿素含量高于早衰类型,并检测到4个叶绿素含量相关的QTL;秦鸿德等[7]用四交分离作图群体的F2:3家系对棉花叶绿素含量进行分析,检测到3个与叶绿素含量相关的QTL,认为其遗传效应以加性效应为主;张建等[8]在陆地棉重组近交家系F2:7群体中,检测到4个与叶绿素质量分数相关的QTL;郑巨云等[9]、戎福喜等[10]分别用陆地棉F2:3家系和海陆渗透系构建的群体各检测到1个与叶绿素含量相关的QTL。【本研究切入点】近年来,关联分析(association analysis)的方法被广泛用于植物数量性状QTL研究中,如拟南芥[11]、水稻[12]、玉米[13-14]等。与传统的连锁分析相比,关联分析以自然群体为材料,构建周期短,可以检测到同一座位的多个等位基因,定位精确[15-16]。NIE等[17]、SU等[18-20]通过全基因组关联分析(genome-wide association study,GWAS)的方法对棉花相关农艺性状进行了基因定位,表明在棉花中运用关联分析对数量性状进行基因定位是一种非常有效的方法。但是利用关联分析对棉花叶绿素含量进行基因定位的研究仍鲜见报道。【拟解决的关键问题】以185份陆地棉品种(系)构成的自然群体为材料,选用137对多态性引物,对3年6个环境中的棉花倒4叶叶绿素相对含量进行关联分析,并挖掘与叶片叶绿素含量相关的优异等位变异,以期为棉花分子标记辅助育种提供技术支持。1 材料与方法
1.1 试验材料和田间种植
试验选取185份陆地棉材料组成自然群体[20](表1),于2013年和2014年在中国农业科学院棉花研究所安阳试验站种植,各设置2个播期(春播和夏播),播种日期分别为2013年4月26日、2013年5月23日、2014年5月1日和2014年5月25日;2016年设置安阳和黄冈2个种植点,播种日期分别为2016年4月25日和2016年4月17日。试验采用常规田间管理,完全随机区组试验设计,设置3个重复。Table 1
表1
表1185份陆地棉材料来源
Table 1The origin regions of 185 upland cotton accessions
| 来源地 Origin region | 数量/份 Number | 来源地 Origin region | 数量/份 Number | |
|---|---|---|---|---|
| 中国北京 Beijing, China | 4 | 中国辽宁 Liaoning, China | 14 | |
| 中国河北 Hebei, China | 17 | 中国山东 Shandong, China | 15 | |
| 中国河南 Henan, China | 98 | 中国山西 Shanxi, China | 7 | |
| 中国湖北 Hubei, China | 4 | 中国陕西 Shaanxi, China | 2 | |
| 中国湖南 Hunan, China | 1 | 中国新疆 Xinjiang, China | 10 | |
| 中国江西 Jiangxi, China | 1 | 美国 America | 12 |
新窗口打开
1.2 表型数据调查
从棉花打顶后第一天开始,使用叶绿素计(SPAD-502Plus,Konica Minoita,Japan)测定185份陆地棉材料倒4叶的叶绿素相对含量(SAPD),每个重复材料每10 d测量一次,共测量3次(打顶后0、10和20 d)。具体测量方法为每个材料随机选取5株,每株测量3次,取测量的平均值作为该材料的叶绿素相对含量。1.3 基因组DNA的提取
采用改良的CTAB法[21]提取棉花幼嫩叶片的基因组DNA。从CMD(Cottonmarker database)数据库(http://www.cottonmarker.org)选取137对均匀覆盖陆地棉26条染色体的SSR引物[22]进行群体扩增,所有引物由生工生物工程(上海)股份有限公司合成。PCR反应体系为模板DNA 30 ng、10×PCR buffer 1.0 μL、10 mmol·L-1 dNTP 0.2 μL、10 mmol·L-1 SSR引物 2 μL、5 U·μL-1Taq DNA聚合酶,加ddH2O补足10 μL。Taq聚合酶和dNTP均购自北京全式金生物技术有限公司。PCR反应程序为95℃ 5 min;94℃ 30 s,58℃ 45 s,45 s,29个循环;72℃ 10 min;10℃保存。扩增产物用浓度8%的聚丙烯酰胺凝胶电泳分离,并参考张军等[23]方法进行银染。1.4 数据统计和分析
1.4.1 SSR标记分析 根据SSR扩增产物的电泳结果,用0,1读带法进行带型统计,迁移率相同的带记为1,无带记为0;不同基因型,用小写字母a、b、c等区分。1.4.2 多态性信息含量(polymorphism information content,PIC)分析 根据SSR扩增产物的带型统计结果,采用Power Maker V3.25软件[24]计算各位点的多态性信息含量(PIC),并用Excel表格进行统计分析。
1.4.3 群体结构分析 采用STRUCTURE 2.3.4软件[25]进行群体结构分析,同时计算每个材料的Q值(即第i个材料基因变异源于第K个亚群的概率),以备后续关联分析。参数设置为:K值范围为1—10,重复10次;MCMC(Markov Chain Monte Carlo)的2个参数分别设为50 000次和100 000次。由于STRUCTURE 软件得的原始数据不能很直观地确定K值,因此,需要计算K值的对数变化率ΔK来更为准确地判断K值[26]。通过StructureHarves(http://taylor0.biology.ucla.edu/structureHarvester/)在线工具[27]收集STRUCTURE软件产生的结果,并作ΔK的变化规律图,以此确定合适的群体数目,同时得到该K值下的Q矩阵。
1.4.4 表型数据分析 采用R语言的ANOVA方法对表型数据进行描述性统计分析,运用R语言的‘lme4’包[28]计算SPAD在3个不同时期的遗传力。
1.4.5 关联分析 运用TASSEL 3.0软件[29]提供的一般线性模型(general linear model,GLM)和混合线性模型(mixed liner model,MLM)2种方法分别对3个时期的SAPD均值和SSR标记进行关联分析。GLM分析需要的Q矩阵来自STRUCTURE软件的结果,MLM分析中需要的Kinship矩阵由TASSEL软件计算得到。
1.5 优异等位变异的挖掘
参考BRESEGHELLO等[30]的方法,通过等位位点与无效等位变异(null allele)相比较,判断该等位位点的表型效应。SSR位点等位变异表型效应的计算公式为:ai=∑xij/ni-∑Nk/nk
其中,ai代表第i个等位变异的表型效应值,xij为携带第i个等位变异的第j材料性状表型测定值,ni为具有第i等位变异的材料数目,Nk为携带无效等位变异的第k个材料的表型测定值,nk为具有无效等位变异的材料数目。若ai值为正,则认为该等位变异为增效等位变异,反之为减效等位变异[31]。
2 结果
2.1 表型数据分析
对185份陆地棉材料在3个时期(打顶后0、10和20 d)及各自6个环境中的SPAD进行方差分析(表2)。在3个不同的测定时期,SPAD的平均值分布范围分别为46.50—55.01、46.02—55.80和42.90—52.86,标准差分别为2.45—3.63、2.51—4.94和3.10—6.91,极差分别为13.07—19.50、13.17—21.67和15.37—30.90。在各个测定时期内,6个环境的叶绿素相对含量均呈极显著正相关(P<0.01),说明叶绿素含量在多个环境下重复性较好。虽然3个时期SPAD平均值变化不大,但是标准差和极差都随测着测定时期的推移依次增大,说明越到后期,棉花品种间叶绿素含量的差异越明显。3个时期的偏度和峰度都比较小,符合近正态分布,可以用作后续关联分析。3个测定时期性状的广义遗传力分别为62.76%、57.77%和62.66%,说明叶绿素含量的遗传力稳定且比较高,受环境影响较小。Table 2
表2
表23个时期不同环境下SPAD值统计表
Table 2The statistics of SPAD value in different environments of 3 stages
| 时期 Stage (d) | 环境 Environment | 平均值 Mean | 标准差 SD | 最小值 Min | 最大值 Max | 极差 Range | 偏度 Skewness | 峰度 Kurtosis | 遗传力 H2 (%) |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 2013AYCB | 47.46 | 2.98 | 38.77 | 58.27 | 19.50 | 0.02 | 0.89 | 62.76 |
| 2013AYXB | 48.81 | 2.47 | 42.23 | 55.30 | 13.07 | -0.14 | 0.04 | ||
| 2014AYCB | 55.01 | 2.85 | 44.20 | 63.15 | 18.95 | -0.39 | 1.35 | ||
| 2014AYXB | 52.09 | 2.68 | 45.10 | 59.73 | 14.62 | 0.00 | -0.39 | ||
| 2016AY | 48.23 | 3.63 | 39.56 | 56.26 | 16.70 | -0.26 | -0.57 | ||
| 2016HG | 46.50 | 2.45 | 39.43 | 53.62 | 14.19 | 0.33 | 0.61 | ||
| 10 | 2013AYCB | 49.07 | 4.94 | 36.08 | 57.75 | 21.67 | -0.74 | -0.16 | 57.77 |
| 2013AYXB | 50.21 | 2.99 | 41.00 | 59.30 | 18.30 | -0.25 | 0.23 | ||
| 2014AYCB | 55.80 | 3.35 | 45.85 | 63.95 | 18.10 | -0.28 | -0.28 | ||
| 2014AYXB | 55.22 | 2.81 | 48.58 | 63.50 | 14.92 | 0.28 | -0.15 | ||
| 2016AY | 51.75 | 3.16 | 40.16 | 57.63 | 17.48 | -0.95 | 1.19 | ||
| 2016HG | 46.02 | 2.51 | 40.24 | 53.42 | 13.17 | 0.29 | -0.01 | ||
| 20 | 2013AYCB | 46.78 | 6.91 | 27.97 | 58.87 | 30.90 | -0.56 | -0.30 | 62.66 |
| 2013AYXB | 51.96 | 4.31 | 35.20 | 60.73 | 25.53 | -0.65 | 0.89 | ||
| 2014AYCB | 48.87 | 5.78 | 30.00 | 60.70 | 30.70 | -0.39 | 0.09 | ||
| 2014AYXB | 50.24 | 5.37 | 29.93 | 60.53 | 30.60 | -0.72 | 0.96 | ||
| 2016AY | 52.86 | 3.10 | 42.96 | 58.33 | 15.37 | -0.95 | 0.78 | ||
| 2016HG | 42.94 | 3.56 | 33.87 | 51.18 | 17.31 | -0.08 | -0.16 |
新窗口打开
2.2 遗传多样性和群体结构分析
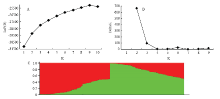
利用筛选出的137对多态性引物进行群体扩增,共检测到355个等位变异,平均每个标记检测到2.59个等位变异。其中HAU2146标记和NAU2083标记检测到的等位变异最多,分别为12个和8个。多态性信息含量(PIC)可以用来衡量基因变异程度的高低,BOTSTEIN等[32]认为,当PIC<0.25时,为低度多态性信息引物,0.25<PIC<0.50时,为中度多态性信息引物,PIC>0.50 时为高度多态性信息引物。对137对引物进行PIC分析,其平均值为0.67,变化范围为0.01—0.95,PIC>0.50的高度多态性引物有115个,占总量的83.94%。其中,PIC最高的标记为HAU2146(0.95)和NAU2083(0.93)。分析结果表明,所选研究群体的遗传多样性较高、SSR标记多态性较好。用STRUCTURE2.3.4软件对185份陆地棉材料进行群体结构分析,并计算相应材料的Q值。由于LnP(D)值随K值的增大而逐渐增大(图1-A),且没有明显的拐点,无法准确判断K的取值,所以参照EVANNO等[26]的方法进行亚群数量的判定。在K=2时,ΔK有最大值(图1-B),所以将材料分为2个亚群(图1-C)。其中一个亚群包含95份材料(占总材料的51%);另一个亚群包含90份材料(占总材料的49%)。获得的Q矩阵将作为关联分析的协变量,以消除群体结构对关联分析结果的影响。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1185份陆地棉材料的群体结构 A:LnP(D)值随K值变化的折线图;B:ΔK值随K值变化的折线图;C:基于SSR标记的185份陆地棉品种群体遗传结构图
-->Fig. 1The population structure of 185 upland cotton accessions A: Chart of LnP (D) changing with K values; B: ΔK value based on structure analysis; C: Population structure of 185 upland cotton accessions based on SSR markers
-->
2.3 SPAD与SSR标记的关联分析
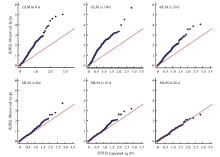
用TASSEL软件的GLM方法和MLM方法,在打顶后0、10和20 d 3个时期,分别对185份材料的SPAD值和355个多态性位点进行关联分析。通过GLM方法(表3、图2)分析关联到22个与SPAD值显著相关的位点(P<0.001),-lg(P)的取值范围为3.04—5.69,表型变异解释率为5.28%—10.85%,平均值为7.24%。贡献率最高的位点是SWU0529a和NAU998c,贡献率分别为10.85%和10.48%。其中变异位点NAU2083e在打顶后10和20 d都被检测到,平均贡献率为7.84%;变异位点NAU998c在0、10和20 d都被检测到,平均贡献率为8.86%。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2GLM和MLM两种模型下的Q-Q plot图
-->Fig. 2Quantile-quantile plots of the GLM and MLM
-->
Table 3
表3
表3GLM和MLM方法关联分析结果
Table 3Association analysis results by the GLM and MLM
| 位点 Locus | GLM | MLM | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 d | 10 d | 20 d | 0 d | 10 d | 20 d | |||||||
| -lg(P) | R2(%) | -lg(P) | R2(%) | -lg(P) | R2(%) | -lg(P) | R2(%) | -lg(P) | R2(%) | -lg(P) | R2(%) | |
| BNL1604d | 3.25 | 6.73 | 2.11 | 4.39 | ||||||||
| BNL3665 | 3.19 | 5.58 | ||||||||||
| BNL226a | 2.22 | 4.59 | ||||||||||
| CER0060a | 2.09 | 4.02 | ||||||||||
| CER0112b | 3.67 | 7.37 | 2.61 | 5.35 | ||||||||
| CGR5258c | 2.05 | 3.76 | 2.28 | 4.16 | ||||||||
| DPL0524d | 3.58 | 6.28 | ||||||||||
| HAU0251 | 3.12 | 5.56 | ||||||||||
| HAU1185b | 3.4 | 6.82 | 2.6 | 5.49 | ||||||||
| HAU1202a | 2.04 | 4.13 | ||||||||||
| HAU3318b | 4.39 | 9.06 | 2.16 | 4.08 | ||||||||
| MGHES-73a | 3.04 | 5.28 | 2.1 | 3.87 | ||||||||
| NAU1042d | 3.35 | 6.09 | ||||||||||
| NAU2083e | 4.52 | 8.54 | 3.56 | 6.42 | 2.06 | 3.88 | 2.62 | 5.22 | 2.09 | 3.84 | ||
| NAU2092e | 3.18 | 5.53 | 2.04 | 3.72 | ||||||||
| NAU2173a | 3.28 | 5.91 | ||||||||||
| NAU2173b | 3.21 | 5.77 | ||||||||||
| NAU2806a | 2.03 | 3.78 | 2.62 | 5.11 | ||||||||
| NAU998c | 4.6 | 9.36 | 5.69 | 10.48 | 3.86 | 6.73 | 2.8 | 5.65 | 2.93 | 5.77 | 2.21 | 4.01 |
| PGML01548c | 4.81 | 9.99 | ||||||||||
| PGML01548b | 3.82 | 7.73 | ||||||||||
| SWU0402b | 3.22 | 6.58 | ||||||||||
| SWU0529a | 5.02 | 10.85 | ||||||||||
| SWU0662d | 3.91 | 7.97 | 3.73 | 8.06 | ||||||||
| SWU0923b | 3.98 | 7.41 | 3.19 | 6.74 | ||||||||
| SWU0987b | 3.04 | 5.41 | 2.1 | 3.92 | ||||||||
| SWU1132a | 4.04 | 7.55 | 2.62 | 5.09 | ||||||||
新窗口打开
通过MLM方法(表3、图2)分析共关联到17个与SPAD值显著相关的位点(P<0.01),-lg(P)的取值范围为2.09—3.73,表型变异解释率为3.72%—8.58%,平均为4.72%。贡献率最高的位点是SWU0923b和SWU0662d,贡献率分别为8.06%和6.74%。其中变异位点NAU2083e和NAU998c可以在0、10和20 d被检测到,平均贡献率分别为4.31%和5.14%;变异位点CGR5258c可以在打顶后10和20 d被检测到,平均贡献率为3.96%;变异位点NAU2806a可以在打顶后10和20 d被检测到,平均贡献率为4.54%。
比较GLM方法和MLM方法的关联结果,GLM方法关联到22个显著性位点,MLM关联到17个显著性位点,两者同时关联到的位点有12个(表4),仅在GLM方法下关联到的位点有10个,仅在MLM方法下关联到的位点有5个。其中变异位点NAU998 c在2种方法3个时期都被检测到。
Table 4
表4
表4GLM方法和MLM方法共同关联到的12个显著性位点在3个时期的表型效应
Table 4Phenotypic effect of the 12 significant loci at 3 stages by the GLM and MLM
| 位点 Locus | 时期 Stage (d) | ||
|---|---|---|---|
| 0 | 10 | 20 | |
| NAU998c | -1.46 | -2.54 | -3.52 |
| NAU2083e | 0.21 | -1.41 | -3.48 |
| MGHES-73a | 0.07 | -1.47 | -2.53 |
| SWU0923b | -0.61 | -1.04 | -1.05 |
| HAU1185b | -1.08 | -0.94 | -0.96 |
| BNL1604d | -1.02 | -0.40 | -0.66 |
| SWU0662d | -0.71 | 0.44 | 0.83 |
| CER0112b | 0.79 | 1.15 | 1.41 |
| NAU2092e | 0.30 | 0.06 | 1.63 |
| SWU1132a | -0.13 | 1.87 | 3.53 |
| SWU0987b | -0.46 | 2.05 | 4.65 |
| HAU3318b | -1.07 | 2.55 | 5.94 |
新窗口打开
2.4 叶绿素含量相关的优异等位变异的发掘
对GLM方法和MLM方法共同关联到的12个显著性等位变异,分别计算其表型效应值,发掘与叶绿素含量相关的优异等位变异(表4)。在打顶后0、10和20 d 3个时期,12个变异位点表型效应的范围不同,分别是-0.71—0.79、-2.54—2.55和-3.52—5.94。在打顶后0 d,有4个位点表现为增效效应,8个位点表现为减效效应,且增效效应和减效效应的效应值绝对值差别不大;但是,在打顶后10 d,增效效应和减效效应的位点各有6个,其中增效效应最大的位点是HAU3318b(2.55)和SWU0987b(2.05),减效效应最大的位点是NAU998c(-2.54)和MGHES-73a(-1.74);在打顶后20 d,增效效应和减效效应的位点也各有6个,其中增效效应最大的位点是HAU3318b(5.94)和 SWU0987b(4.65),减效效应最大的位点是NAU998c(-3.52)和NAU2083e(-3.48)。用2个增效效应最大的位点(HAU3318b和SWU0987b)对材料进行筛选,并比较其在打顶后0、10和20 d 3个时期SPAD值的差异。检测到HAU3318b和SWU0987b 2个增效效应位点的材料(ZX2)有53份,2个增效效应位点都未检测到的材料(ZX0)有46份。ZX2和ZX0在打顶后0 d的SPAD均值差异不大(图3-A),而在打顶后 10和20 d,ZX2的SPAD均值都显著比ZX0的SPAD均值高(图3-B、图3-C);根据材料的表型数据,在ZX2中筛选了一份典型材料——新棉33B,其SPAD值在3个时期分别为52.68(0 d)、55.15(10 d)和55.67(20 d)。说明陆地棉倒4叶在打顶后10 d已经出现衰老表型的差异,此时的SPAD值可以作为棉花品种衰老性状的指标。同时,检测到HAU3318b和SWU0987b是2个提高叶片叶绿素含量的优异等位变异,获得携带2个优异等位变异的载体材料53份,获得携带2个优异等位变异的典型材料一份。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3携带两个增效等位变异的材料在打顶后0、10和20 d 3个时期的SPAD值 ZX2:同时检测到2个增效等位变异的材料;ZX0:2个增效等位变异都未检测到的材料
-->Fig. 3The SPAD of cotton materials carrying two positive alleles in 0, 10 and 20 days after topping ZX2: The carrier materials of two positive alleles; ZX0: The materials that don’t carry two positive alleles
-->
2.5 关联分析结果与前人QTL定位结果的比较
结合前人叶绿素含量相关QTL定位的研究结果[4-10,33],利用e-PCR程序[34]将统计得到的25对SSR标记和本研究得到的10对(2对SSR标记未能成功匹配)显著性标记匹配到参考基因组[35]上(图4)。35对SRR标记被匹配到19条染色体上(A01、A02、A03、A06、A07、A08、A09、A10、A13、D01、D03、D05、D06、D07、D08、D09、D10、D11、D13)。与前人研究结果相比,本研究在5条新的染色体上发现了标记位点,即A01(SSR标记为NAU2083、NAU2092)、A02(SSR标记为NAU3318)、A03(SSR标记为NAU998)、A08(SSR标记为SWU0932)、A09(SSR标记为MGHES-73)。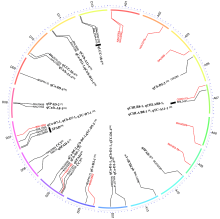 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4综合前人研究与本研究的35对SSR标记的环形物理图谱染色体上的物理距离单位为Mb;红色SSR标记为本研究获得,黑色SSR标记为前人研究获得
-->Fig. 4Circus physical map of 35 SSR markers from this study and previous studies The unit of physical distance is Mb for the chromosomes; the red SSR markers got from this study, and the black SSR markers got from previous studies
-->
3 讨论
叶绿素是植物进行光合作用的主要物质基础,是作物高产的重要指标。目前,关于棉花叶片叶绿素含量的基因定位研究的较少,为了从全基因组水平上定位与叶绿素含量相关的基因,本研究采用关联分析的方法,用覆盖棉花26条染色体的137对多态性SSR引物对棉花叶片叶绿素含量进行了基因定位,并挖掘其优异的等位变异及典型材料。本研究用筛选到的137对多态性引物,187份陆地棉材料中共检测到355个等位变异,根据BOTSTEIN等[32]的分类方法,137对引物中有83.94%的引物为高度多态性引物(PIC>0.50),说明本研究的SSR引物含有丰富的遗传信息并且所选群体的遗传多样性很好,有利于后续的关联分析和发掘优异等位变异。由于环境对数量性状的影响很大,高遗传力有利于关联分析结果的准确性[36]。本研究在3年6个环境下,分3个时期对棉花倒4叶叶绿素相对含量(SPAD)进行了测量,统计结果显示,SPAD的广义遗传力在3个时期约为60%,遗传力稳定并且比较高,这为提高关联分析结果的准确性提供了有利条件。
为降低群体结构和亲属关系造成的假阳性结果[37],本研究计算了Q矩阵和K矩阵,并同时用GLM方法和MLM方法进行关联分析。分析结果显示(表3、图2),MLM方法关联到的位点(17个)比GLM方法关联到的位点(22个)要少,说明GLM方法的假阳性率高于MLM方法。但是,MLM方法和GLM方法共同关联到的位点仅有12个,所以通过2种方法相互比较印证,也可以有效的减少假阳性结果。值得注意的是,不同变异位点在同时期的表型效应有差异,而且有的变异位点在不同时期的表型效应值差异很大,甚至是效果相反。如位点NAU998c在3个时期都表现减效效应,且绝对值不断增大,而位点HAU3318b在打顶后0 d表现为减效效应,在打顶后10和20 d都表现为增效效应,绝对值也不断增大。这种现象可能是由于叶绿素含量相关的基因在棉花叶片不同生育时期是特异性表达造成的,具体原因还需要进一步的科学研究。
本研究首次利用了关联分析的方法对棉花叶绿素含量进行基因定位,将关联分析结果与前人QTL定位结果进行比较发现,D05是标记较为集中的染色体,王鹏等[33]、郑巨云等[9]分别在D05染色体定位到一个叶绿素b含量相关的QTL(qCb-D05-3,附近SSR标记为NAU3252)和一个 SPAD值相关的QTL(qSPAD1,附近SSR标记为NAU1221),本研究在这2个QTL间关联到一个显著性SSR标记NAU1185,而且与qSPAD1(NAU1221)相距仅大约2 Mb,说明这一区段附近很可能存在与叶绿素含量相关的基因位点,可以进行深入的分析验证。
通过计算与SPAD显著关联位点的表型效应,本研究发掘了SPAD相关的优异等位变异HAU3318b和SWU0987b。在棉花育种改良工作中,可以直接选取含有优异等位变异的材料作为亲本,有利于提高选择效率。
4 结论
共获得355个多态性位点,检测到12个与陆地棉叶片叶绿素含量(SPAD)相关的显著性位点。发掘2个可以提高棉花叶片叶绿素含量的优异等位变异HAU3318b和SWU0987b,获得 53份携带2个优异等位变异的析料,获得一份携带2个优异等位变异的典型材料,可用于陆地棉分子标记辅助育种。The authors have declared that no competing interests exist.
