0 引言
【研究意义】土壤盐渍化是全球农业生产面临的一大问题。盐渍化不仅导致农作物产量和品质的显著降低,而且严重限制了农业土地的利用[1]。世界上已有超过2 000万公顷的土地盐渍化[2]。由于气候变化和灌溉不合理,盐渍地面积仍在以每年近百万公顷的速度不断增加[3]。油菜是中国主要经济作物之一,近年来盐渍化严重影响了油菜的生产。油菜耐盐性相关遗传因子的发现及耐盐候选基因的挖掘对指导培育耐盐性油菜品种从而利用大面积的盐渍地具有重要意义。【前人研究进展】盐胁迫是一类典型的非生物胁迫。植物对盐胁迫的耐受反应是个复杂的过程,包括对外界盐信号的感应和传递,特异转录因子的激活和下游控制生理生化应答的效应基因的表达[4]。在盐胁迫条件下,植物多种应答机制参与调节,如渗透调节、信号传导调节、激素调节等[5-7]。SOS(salt overly sensitive)信号转导途径是目前研究最清楚的耐盐机理之一[8]。土壤中Na 盐过多会引起植物体内离子不平衡和离子毒害, SOS途径的重要生理功能即在盐胁迫下进行离子稳态调节和提高植物的耐钠性。SOS途径基因介导信号转导过程中包含6个关键基因(SOS1、SOS2、SOS3、SOS4、SOS5和SOS6)。这6个关键基因通过不同途径调控植物的耐盐性。SOS1是质膜定位的Na+/H+反向运输体,主要在控制细胞质Na+维持低浓度中起主要作用[9-10]。SOS2、SOS3主要通过对SOS1的调控,实现细胞内外K+、Na+的调节,从而增强植物的耐盐性。除此之外,SOS2还可以调控其他运输体的活性, 如调控H+/Ca2+反向运输体、液泡膜H+-ATPase和液泡膜Na+/H+反向运输体来增加植物的耐盐性[11-13]。SOS4不仅可以通过调节SOS1,还可以通过调节其他离子转运蛋白活性来提高植物耐盐性。另外,SOS4还可以通过盐胁迫下促进根毛的发育来提高耐盐性[14]。SOS5主要通过促进细胞壁发育和增强胞间连接而提高植物的耐盐性[15]。SOS6则通过调节细胞渗透胁迫和氧化胁迫来提高植物的耐盐性[16]。植物耐盐性是一个非常复杂的性状。在许多植物中,已发现控制耐盐性的遗传因子。在拟南芥、大麦、水稻、小麦、黑麦草等植物中,目前已通过传统QTL定位、全基因组关联分析或重测序的方法发现了控制耐盐性的遗传因素[17-23]。REN等[19]在拟南芥的萌发过程中,发现了一个耐盐QTL—RAS1,可以通过提高脱落酸敏感性负向调节耐盐性。通过全基因组重测序结合MutMap分析方法,在水稻上也快速定位了耐盐突变体hst1,找到控制水稻耐盐性状的基因[23]。近期研究报道发现,在油菜上,通过全基因组关联分析的方法检测到62个耐盐相关位点并鉴定了一个候选基因BnnaTSN1[1]。除此之外,还有许多通过转基因提高植物耐受性的成功例子:耐受的nax2导入系小麦,在盐渍地上的产量提高了25%[20]。ZHOU等[24]将SeNHX1和甜菜碱合成酶BADH协同转入烟草,发现转基因植物受到盐胁迫的影响比野生型植物小;QIAO等[25]将长穗偃麦草的AeNHX1转化拟南芥并使其在液泡膜表达,发现转基因植物的耐盐性也有所提高;BRINI等[26]将小麦的TNHX1转入拟南芥,转基因株在盐胁迫下长势良好,而野生型的生长受到抑制,叶片大面积枯萎。将油菜耐盐基因BnRCH转入拟南芥,能够提高转基因拟南芥的耐盐性[27]。【本研究切入点】尽管目前国内外关于植物耐盐机制已取得很大进展,但在油菜上,关于耐盐遗传机制和分子机理的研究较少。发芽期是油菜生长发育的起始阶段,也是对盐胁迫最敏感的时期之一[28]。由于受盐胁迫的影响,油菜的发芽率、发芽期的根长、整株鲜重等会降低[29]。【拟解决的关键问题】本研究通过对307份不同遗传背景的甘蓝型油菜种质资源进行发芽期耐盐性鉴定,并在此基础上对油菜耐盐相关性状与SNP标记进行全基因组关联分析,寻找与耐盐性关联的SNP位点并进一步筛选可能与油菜耐盐性有关的候选基因。为揭示甘蓝型油菜耐盐遗传机制提供有价值的信息,为耐盐油菜品种的培育提供遗传理论指导。1 材料与方法
1.1 材料
所用的关联分析群体包含307个具有不同遗传背景的甘蓝型油菜高世代自交系。其中包括51个来自欧洲的冬性甘蓝性油菜品种,以及195个中国的半冬性品种和61个春性品种。所用发芽试验的种子均为2014年自交套袋收获的种子。1.2 试验处理及表型数据考察
每份参试材料取圆润饱满、健康的种子100粒,其中50粒作为试验组,50粒作为对照组,均匀放入培养皿中。培养皿内垫有三层滤纸用以保持水分,盖上皿盖以保持培养皿内的湿度。为确定培养天数及最佳NaCl培养液浓度,选取冬性、半冬性和春性品种各两份进行预试验。处理液浓度分别为0.1%、0.3%、0.6%、1%、1.5%和2%的NaCl溶液,对照液为去离子水。发芽试验在西南大学油菜中心组培室内进行,昼夜温度恒定25℃,光照/黑暗时间为12 h/12 h,光照强度为7 000 lx,相对湿度为60%[30]。通过预试验发现,当NaCl溶液溶度在1%—1.5%时,种子受影响变化程度最大,因此,最终决定以1.2% NaCl溶液作为检测液;从培养第2天开始观察,可发现到第6天,种子基本已发芽完成,故培养观察天数定为7 d。将307份材料试验组以1.2%NaCl溶液25 mL作为培养液,对照组以去离子水25 mL作为培养液。3次重复。每天观察油菜种子的萌发情况,播种后次日起每天统计发芽数并记录。于第7天打开皿盖,每皿随机选取生长一致的10株幼苗测定根长和单株鲜重称重,并做好相关记录。根长用WinRHIZO根系扫描测定系统测定,将完整根系样本放入专用根盘,用镊子调整根的位置避免交叉重叠,用扫描仪对不同品种根系图像进行扫描并保存于电脑中,利用WinRhizo Pro 2004a软件分析图像,参照顾东祥等[31]的方法提取相应的根长度。发芽率= 发芽种子数/参试种子数×100%。将盐胁迫下各性状相对值作为评价耐盐的指标。盐胁迫下各性状的相对值=1.2%NaCl处理测定值/CK测定值× 100% [32-33]。
1.3 SNP基因型
利用Illumina公司油菜60K SNP芯片对307份油菜材料进行基因型分析获取SNP标记,排除最小基因型频率(minor allele frequency,MAF)低于0.05 和SNP得率(call frequency)小于80%的标记,一共有33 186个SNP标记用于随后的关联分析。1.4 全基因组关联分析
利用SPAGeDi v1.4软件对该群体307份甘蓝型油菜进行亲缘关系(relative kinship)分析,并计算亲缘关系值的矩阵(K矩阵)[34]。利用软件STRUCTURE v2.3.4[35],基于贝叶斯数学模型对关联群体进行群体结构分析。假设群体内存在的亚群数目K的范围为1—10,运用该软件对每个K值进行5次模拟运算,将模拟参数迭代(length of burn-in period)和蒙特卡罗迭代(markov chain monte carlo,MCMC)均设置为10 000次循环,并在混合模型下进行运算。最后根据STRUCTURE软件运算得到的后验概率值和2个连续的后验概率值的变化速率(ΔK)来确定群体中存在的亚群数目[36]。为有效排除假关联的影响,在本研究中,将群体结构和材料间的亲缘关系考虑进关联分析中,同时进行了PCA+K模型、Q+K模型以及K模型3种混合线性模型分析和比较,根据所有SNP的-lg(P)观察值和期望值,确定每个性状GWAS分析的最佳模型。采用TASSEL 5.0软件[37],基于最优模型的关联分析结果,利用QQman绘制曼哈顿图。以1/标记数设为显著关联SNP阈值[38],本研究的显著水平值为-lg(P)>4.52。
1.5 候选基因的预测
在关联分析的基础上,利用已公布的甘蓝型油菜基因组测序数据库(http://www.genoscope.cns.fr/blat- server/cgi-bin/colza/webBlat),结合使用BLAST软件[39],在参数E-Value≤1e-10的条件下,对显著性关联的SNP标记与甘蓝型油菜基因组进行比对作图[40],将与耐盐性状显著关联的SNP标记定位到油菜的基因组上,进而在显著性关联的SNP位点侧翼序列200 kb范围内提取出基因。进一步利用目前油菜基因组的注释信息及其与拟南芥基因的共线性关系,根据拟南芥中已经明确功能的耐盐相关基因,筛选出候选区段内与耐盐相关的油菜同源基因。2 结果
2.1 耐盐相关性状的表型变异
通过对307份甘蓝型油菜进行耐盐相关性状分析(图1)。在盐胁迫下,根长、鲜重、发芽率相对值均呈连续性分布。这表明这些耐盐相关性状受多基因控制,属于数量性状遗传。3个性状在盐胁迫下的相对值均表现出广泛的表型变异(表1)。根长相对值变异幅度为0.064—0.994,平均值为0.811,变异系数为16.19%。整株鲜重相对值变异幅度为0.01—0.947,平均值为0.760,变异系数为19.73%。发芽率相对值变异幅度为0—0.997,平均值为0.160,变异系数为129.85%。Table 1
表1
表1耐盐相关性状的表型变异
Table 1Phenotypic variations of salt tolerance related traits
| 性状 Traits | 最小值 Min | 最大值 Max | 平均数±标准差 Mean±SD | 变异系数 CV (%) |
|---|---|---|---|---|
| 根长 Root length | 0.064 | 0.994 | 0.811±0.131 | 16.19 |
| 鲜重 Fresh weight | 0.01 | 0.947 | 0.760±0.150 | 19.73 |
| 发芽率 Germination rate | 0 | 0.997 | 0.160±0.208 | 129.85 |
新窗口打开
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1耐盐相关性状的频率分布图
a:根长;b:鲜重;c:发芽率
-->Fig. 1Phenotype frequency distribution of salt tolerance related traits
a: Root length; b: Fresh weight; c: Germination rate
-->
2.2 亲缘关系分析和群体结构分析
在关联分析前,首先利用高密度覆盖甘蓝型油菜全基因组的5 700个SNP标记,分析了群体材料间亲缘关系和群体结构。结果表明,在307份材料中,约55%在材料之间亲缘关系值等于0(图2),这表明整个群体材料间的亲缘关系较弱,符合进行全基因组关联分析的要求。群体结构分析表明,在K=3处观察到ΔK值最大,所以该自然群体的适宜亚群数确定为3 (图3-a)。基于全基因组33 186个SNP标记的PCA分析结果表明,前两种成分解释的遗传方差分别为16.48%的8.45%,这307份甘蓝型油菜材料被划分为3个亚群,各个亚群分别主要由冬性、春性和半冬性品种构成(图3-b)。因此,将K=3时生成的Q矩阵,用于性状与标记的关联分析。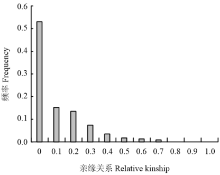 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2关联群体中307份甘蓝型油菜材料的亲缘关系分析
-->Fig. 2Distribution of relative kinship values of 307 B. napus inbred lines in association mapping panel
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3307份甘蓝型油菜的群体结构分析
a:K取不同值时ΔK 的对应分布;b:主成分PC1、PC2的散点分布
-->Fig. 3Analysis of population structure in 307 B. napus
a: Estimated ΔK between successive K from 1 to 9; b: Plots of the first two principal components(PC1 and PC2)
-->
2.3 关联分析模型的选取
采用TASSEL 5.0软件MLM模型程序,对307份甘蓝型油菜进行耐盐性状的全基因组关联分析。为有效排除假关联的影响,将群体结构和材料间的亲缘关系考虑进关联分析中,同时进行了PCA+K模型、Q+K模型以及K模型3种混合线性模型的分析。通过比较3种模型下Q-Q图的分布看出(图4),发芽率和根长这两个性状在PCA+K模型下的-lg(P)值分布最接近预测值,因此,这两性状选择PCA+K模型作为最终分析模型来寻找关联位点。而鲜重在Q+K模型下的-lg(P)值分布最接近预测值,故鲜重性状选择Q+K模型作为最终分析模型来寻找关联位点。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4耐盐相关性状在K模型、Q+K模型及PCA+K模型下的Q-Q散点图
a:根长;b:鲜重;c:发芽率
-->Fig. 4Quantile-quantile plots of model K, model Q+K and model PCA+K for the three salt tolerance related traits
a: Root length; b: Fresh weight; c: Germination rate
-->
2.4 耐盐性状与SNP的全基因组关联分析(GWAS)
对307份材料耐盐各性状的相对值分别与SNP标记进行全基因组关联分析,结果表明,与根长显著关联的SNP标记共检测到164个,除了C04染色体外,其余18条染色体上都检测到了与根长关联的位点。这些位点可解释的表型变异率为10.44%—23.84%(图5-a)。与鲜重显著关联的SNP标记共检测到23个,分别位于A02、A03、A04、A06、A07、A09、C01、C02、C03、C06、C07和C08染色体上。这些位点可解释的表型变异率为8.42%—18.59%(图5-b)。与发芽率显著关联的SNP标记共检测到38个,分别位于A01、A03、A04、A05、A06、C03、C04、C05和C08染色体上。这些位点可解释的表型变异率为17.85%—31.81%(图5-c)。表2包含与根长、鲜重、发芽率最显著关联的SNP位点信息,分别位于染色体A08、A02和A06,贡献率分别为23.84%、18.59%和31.81%。2.5 耐盐候选基因预测
通过对甘蓝型油菜3个耐盐性状的全基因组关联分析,利用已公布的甘蓝型油菜基因组测序结果,将与耐盐性状显著关联的SNP标记定位到油菜的基因组上,进而在显著性关联的SNP位点侧翼序列200 kb范围内提取基因。进一步利用目前油菜基因组的注释信息及其与拟南芥基因的共线性关系,根据拟南芥中已经明确功能的耐盐相关基因,筛选出候选区段内与耐盐相关的油菜同源基因。本研究共筛选出50个与耐盐性相关的候选基因(表3)。在这些候选基因中,发现了大量的转录因子,包括MYB、WRKY、ABI1、bZIP、ERF1、CZF1,以及XERICO。候选基因中还包含了下游受盐胁迫诱导的基因,这些基因涉及不同的功能,主要包括NHX1、PTR3、CAT1、HKT、CAX1、ACER、STH、STO等。其中在根长和发芽率2个不同耐盐性状的GWAS分析结果中均筛选出了位于A03染色体上的基因BnaA03g14410D。另外有两组串联重复基因,分别是位于A03染色体上的BnaA03g18900D和BnaA03g18910D,位于C09染色体上的BnaC09g19080D、BnaC09g19090D和BnaC09g19100D。除了串联重复基因之外,在50个候选基因里还包含了2个距离非常近(中间只间隔2个基因)的重复基因BnaC02g39600D和BnaC02g39630D。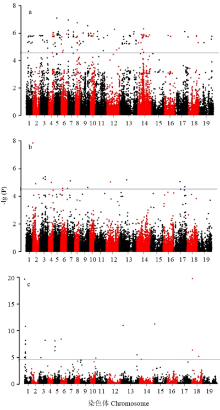 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5耐盐相关性状的全基因组关联分析
a:根长;b:鲜重;c:发芽率。显著性阈值为-lg(1/SNP标记个数),为4.52
-->Fig. 5Genome-wide association study of salt tolerance related traits
a: Root length; b: Fresh weight; c: Germination rate. The significance threshold -lg(P) = 4.52
-->
Table 2
表2
表2关联分析中检测到的最显著SNP位点
Table 2Genome-wide significant association signals detected in root lenth, fresh weight and germination rate, respectively
| 性状 Trait | SNP代号 SNP Code | 染色体 Chr. | SNP 位置 SNP Site | P 值 P value | 显著性 -lg (P) | 贡献率 R2 |
|---|---|---|---|---|---|---|
| 根长 Root lenth | SNP19218 | A08 | 7141913 | 2.05E-06 | 5.6890307 | 0.23842 |
| 鲜重 Fresh weight | SNP4206 | A02 | 552308 | 1.34E-08 | 7.87364127 | 0.18592 |
| 发芽率 Germination rate | SNP14184 | A06 | 2681244 | 4.56E-09 | 8.34146395 | 0.31805 |
新窗口打开
Table 3
表3
表3显著性SNP 位点侧翼序列200 kb内的耐盐候选基因
Table 3Salt tolerance candidate genes in regions of 200 kb flanking sequence from the significant SNP loci
| 性状 Trait | SNP位置 SNP position | 显著性 -lg (P) | 油菜基因 Bna. genes | 拟南芥同源基因 ATH. genes | 距SNP距离 Distance | 基因功能 Gene function | 参考文献 Reference |
|---|---|---|---|---|---|---|---|
| 根长 Root length | A01:7913112 | 4.650 | BnaA01g15250D | At4g26080 | 112.9 | ABI1 | [41] |
| 根长 Root length | A01:7913112 | 4.650 | BnaA01g15290D | At1g64280 | 98.9 | NPR1 | [42] |
| 根长 Root length | A01:7913112 | 4.650 | BnaA01g15750D | At4g26630 | 160.8 | DEK domain protein | [43] |
| 根长 Root length | A01:16459849 | 5.238 | BnaA01g23930D | At3g23250 | 76.2 | MYB15 | [44] |
| 根长 Root length | A01:16459849 | 5.238 | BnaA01g23940D | At3g23240 | 54.9 | ERF1 | [45] |
| 根长 Root length | A01:16459849 | 5.238 | BnaA01g23960D | At3g23230 | 29.2 | Integrase-type DNA-binding superfamily protein | [46] |
| 根长 Root length | A01:21190485 | 5.738 | BnaA01g31420D | At3g10800 | 183.6 | BZIP28 | [47] |
| 根长 Root length | A03:6450763 | 5.777 | BnaA03g13820D | At2g30250 | 117.3 | WRKY25 | [48] |
| 根长 Root length | A03:6450763 | 5.777 | BnaA03g14410D* | At2g31380 | 191.9 | STH | [49] |
| 根长 Root length | A03:8962400 | 5.767 | BnaA03g18900D** | At2g40140 | 37.4 | CZF1 | [50] |
| 根长 Root length | A03:8962400 | 5.767 | BnaA03g18910D** | At2g40140 | 36.3 | CZF1 | [50] |
| 根长 Root length | A03:8962400 | 5.767 | BnaA03g19270D | At2g40880 | 139.2 | CYSA | [51] |
| 根长 Root length | A03:24129001 | 5.299 | BnaA03g46800D | At4g24560 | 129.6 | UBP16 | [52] |
| 根长 Root length | A03:24129001 | 5.299 | BnaA03g47350D | At4g25380 | 121.6 | SAP10 | [53] |
| 根长 Root length | A04:14085477 | 5.733 | BnaA04g17420D | At2g0250 | 78.7 | WRKY25 | [48] |
| 根长 Root length | A04:16618792 | 5.983 | BnaA04g21770D | At2g38010 | 90.7 | Neutral/alkaline non-lysosomal ceramidase | [54] |
| 根长 Root length | A04:16618792 | 5.983 | BnaA04g21850D | At2g38170 | 11.1 | CAX1 | [55] |
| 根长 Root length | A04:16618792 | 5.983 | BnaA04g22040D | At2g38470 | 70.1 | WRKY33 | [56] |
| 根长 Root length | A04:14881039 | 6.009 | BnaA04g18330D | At2g31380 | 190.8 | STH | [49] |
| 根长 Root length | A04:15124491 | 5.738 | BnaA04g19410D | At2g33380 | 178.9 | RD20 | [57] |
| 根长 Root length | A05:9674529 | 5.821 | BnaA05g15160D | At2g04240 | 102.5 | XERICO | [58] |
| 根长 Root length | A06:19876588 | 5.896 | BnaA06g28930D | At5g27150 | 86.1 | NHX1 | [59] |
| 根长 Root length | A07:19491505 | 5.682 | BnaA07g26500D | At1g67580 | 30.2 | Protein kinase | [60] |
| 根长 Root length | A08:7460700 | 5.745 | BnaA08g07400D | At3g17000 | 44.6 | UBC32 | [61] |
| 根长 Root length | A09:8876364 | 5.967 | BnaA09g15290D | At1g56600 | 19.5 | GolS2 | [62] |
| 根长 Root length | A09:8876364 | 5.967 | BnaA09g15400D | At1g56340 | 85.3 | CRT1a | [63] |
| 根长 Root length | A10:1980375 | 5.750 | BnaA10g03880D | At1g05850 | 84.6 | POM1 | [64] |
| 根长 Root length | A10:1980375 | 5.750 | BnaA10g03990D | At1g06040 | 134.9 | STO | [65] |
| 根长 Root length | C01:11162115 | 5.529 | BnaC01g16410D | At4g24560 | 100.8 | UBP16 | [52] |
| 根长 Root length | C02:42327739 | 4.866 | BnaC02g39600D** | At5g27150 | 162.1 | NHX1 | [59] |
| 根长 Root length | C02:42327739 | 4.866 | BnaC02g39630D** | At5g27150 | 180.3 | NHX1 | [59] |
| 续表3 Continued table 3 | |||||||
| 性状 Trait | SNP位置 SNP position | 显著性 -lg (P) | 油菜基因 Bna. genes | 拟南芥同源基因 ATH. genes | 距SNP距离 Distance | 基因功能 Gene function | 参考文献 Reference |
| 根长 Root length | C03:7800453 | 5.157 | BnaC03g15510D | At5g52310 | 40.9 | LTI78/RD29A | [66] |
| 根长 Root length | C03:11587656 | 5.697 | BnaC03g21360D | At2g38470 | 46.1 | WRKY33 | [56] |
| 根长 Root length | C03:12907557 | 5.511 | BnaC03g22970D | At2g40880 | 177.7 | CYSA | [51] |
| 根长 Root length | C03:35335923 | 5.733 | BnaC03g51050D | At5g63310 | 161.8 | NDPK2 | [67] |
| 根长 Root length | C07:16918488 | 4.876 | BnaC07g11080D | At2g26430 | 100.7 | RCY1 | [68] |
| 根长 Root length | C09:42936769 | 5.745 | BnaC09g40820D | At5g17170 | 170.2 | ENH1 | [69] |
| 根长 Root length | C09:16062335 | 5.265 | BnaC09g19080D** | At5g46050 | 100.5 | PTR3 | [70] |
| 根长 Root length | C09:16062335 | 5.265 | BnaC09g19090D** | At5g46050 | 98.2 | PTR3 | [70] |
| 根长 Root length | C09:16062335 | 5.265 | BnaC09g19100D** | At5g46050 | 90.0 | PTR3 | [70] |
| 鲜重 Fresh weight | A02:10251208 | 5.254 | BnaA02g17290D | At1g75540 | 178.0 | STH2 | [71] |
| 鲜重 Fresh weight | A02:20556629 | 4.965 | BnaA02g27740D | At3g25990 | 69.0 | Homeodomain-like protein | [72] |
| 鲜重 Fresh weight | A02:20556629 | 4.965 | BnaA02g28060D | At3g26420 | 179.4 | ATRZ-1A | [73] |
| 鲜重 Fresh weight | A03:11534668 | 5.789 | BnaA03g24340D | At4g10310 | 162.9 | HKT1 | [74] |
| 鲜重 Fresh weight | A06:7733718 | 4.690 | BnaA06g14520D | At1g20630 | 115.4 | CAT1 | [75] |
| 鲜重 Fresh weight | C03:6848993 | 4.623 | BnaC03g14320D | At5g55230 | 36.5 | MAP65-1 | [76] |
| 鲜重 Fresh weight | C07:21411579 | 5.181 | BnaC07g15270D | At1g20630 | 178.3 | CAT1 | [75] |
| 发芽率 Germination rate | A01:2489643 | 5.011 | BnaA01g05110D | At4g31720 | 90.4 | TAFII15 | [77] |
| 发芽率 Germination rate | A01:2783526 | 8.091 | BnaA01g06180D | At4g30960 | 85.9 | SIP3 | [78] |
| 发芽率 Germination rate | A03:6610774 | 4.909 | BnaA03g14410D* | At2g31380 | 31.9 | STH | [79] |
| 发芽率 Germination rate | C05:2486406 | 11.160 | BnaC05g05160D | At1g07380 | 18.9 | AtACER | [54] |
新窗口打开
3 讨论
本研究通过对307个甘蓝型油菜的3个耐盐相关性状的全基因组关联分析,共检测出164个与相对根长显著关联的SNP,23个与相对鲜重显著关联的SNP,38个与相对发芽率显著关联的SNP。根据性状与标记的关联结果,3个耐盐相关性状都检测到较多的位点,这表明油菜的耐盐性为微效多基因控制。YONG等[1]利用一个包含85份油菜材料的自然群体,通过全基因组关联分析的方法检测到了62个耐盐相关位点。将本研究结果中的显著关联的SNP位点与前人研究结果进行比较,发现有10个SNP位点相距很近。这10个SNP位点为A03:23458534、A04:9941507、A05:2490459、A06:1463625、A09:28059070、A10: 11505183、C03:22628438、C03:6032749、C04:2463595和C07:21411579。这些与耐盐性状显著关联的SNP标记,在育种试验中可优先选择贡献率较大的标记,进一步验证后用于油菜耐盐改良。在本研究中,挖掘出可能与甘蓝型油菜耐盐性有关的50个候选基因。在这些候选基因中,发现了大量的转录因子,包括MYB、WRKY、ABI1、bZIP、ERF1、CZF1,以及XERICO。前人研究表明,这些转录因子主要是在盐胁迫的早期感应机制下游发挥重要作用,主要通过进一步调控下游各种基因的表达最终影响植物的耐盐性[79-80]。除了转录因子,候选基因中还包含了下游受盐胁迫诱导的基因,主要包括NHX1、PTR3、CAT1、HKT、CAX1、ACER、STH、STO等,这些基因涉及不同的功能,例如NHX1是液泡膜定位的Na+/H+交换器,主要与离子运输与离子稳态平衡有关[10,81]。HKT1则主要是在根-冠Na+的分配上起重要作用,拟南芥中AtHKT1;1的表达受到基本亮氨酸拉链转录因子bZIP24的负调控[82],对bZIP24的RNA干涉植株则耐盐性提高。除此之外,本研究发现的候选基因中还有除了转录因子之外的其他耐盐相关调节因子,如UBP16。研究表明,UBP16可能是Na+运输过程中的一个重要调控因子,UBP16缺陷的突变体植株在叶片中会积累更高的Na+,表现出盐害超
敏感[52]。在本研究筛选的50个候选基因中,发现有2个基因UBC32与ENH1,这两个基因在前人研究中也预测到与油菜耐盐性相关。这些耐盐候选基因的发现,对耐盐油菜育种具有重要意义。值得一提的是,本研究中根长和发芽率2个不同性状的GWAS结果筛选出了A03染色体上一个共同的耐盐候选基因BnaA03g14410D。该基因在拟南芥中的同源基因为STH(At2g31380),STH是耐盐基因STO的同源基因[83]。STO最早被证实能够补偿盐敏感的酵母钙调磷酸酶缺陷突变体的生长缺陷[84-85]。因此,长期以来人们一直认为STO也可能与植物耐盐性紧密相关[83]。在拟南芥中已报道,过量表达STO的转基因植株根的长度比野生型增加33%—70%,过量表达STO可以增强植株对盐的耐性,STO通过结合一个MYB转录因子基因,赋予拟南芥耐盐性[85]。
另外,在50个候选基因中包含有两组串联重复基因,分别是位于A03染色体上的BnaA03g18900D和BnaA03g18910D,位于C09染色体上的BnaC09g19080D、BnaC09g19090D、BnaC09g19100D。除了串联重复基因之外,在50个候选基因里还包含了2个距离非常近(中间只间隔2个基因)的重复基因BnaC02g39600D和BnaC02g39630D。这三组重复基因功能分别为CZF1、PTR3和NHX1。在高粱基因组测序的研究结果发现,高粱对干旱的耐受性可能与基因和microRNA的重复有关[86]。在本研究中,这些耐盐相关的串联重复基因的发现进一步暗示,在基因突变和自然选择的作用下,重复基因的进化可使生物更好地适应环境变化。
4 结论
通过油菜耐盐性状的全基因组关联分析,获得225个与耐盐性状显著关联的SNP位点,筛选出50个可能与油菜耐盐性有关的候选基因。The authors have declared that no competing interests exist.
