 ,1, 邱艳红
,1, 邱艳红 ,2,3, 张晓飞2, 刘慧2, 杨静静2, 张建2,3, 张海军2,3, 徐秀兰2,3, 温常龙
,2,3, 张晓飞2, 刘慧2, 杨静静2, 张建2,3, 张海军2,3, 徐秀兰2,3, 温常龙 ,2,3
,2,3The Detection of Citrullus lanatus Cryptic Virus Using TaqMan-qPCR Method
ZHAO LiQun ,1, QIU YanHong
,1, QIU YanHong ,2,3, ZHANG XiaoFei2, LIU Hui2, YANG JingJing2, ZHANG Jian2,3, ZHANG HaiJun2,3, XU XiuLan2,3, WEN ChangLong
,2,3, ZHANG XiaoFei2, LIU Hui2, YANG JingJing2, ZHANG Jian2,3, ZHANG HaiJun2,3, XU XiuLan2,3, WEN ChangLong ,2,3
,2,3通讯作者:
责任编辑: 岳梅
收稿日期:2021-03-1接受日期:2021-04-9
| 基金资助: |
Received:2021-03-1Accepted:2021-04-9
作者简介 About authors
赵立群,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (1953KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
赵立群, 邱艳红, 张晓飞, 刘慧, 杨静静, 张建, 张海军, 徐秀兰, 温常龙. TaqMan探针法实时荧光定量PCR检测西瓜潜隐病毒. 中国农业科学, 2021, 54(20): 4337-4347 doi:10.3864/j.issn.0578-1752.2021.20.008
ZHAO LiQun, QIU YanHong, ZHANG XiaoFei, LIU Hui, YANG JingJing, ZHANG Jian, ZHANG HaiJun, XU XiuLan, WEN ChangLong.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】西瓜潜隐病毒(Citrullus lanatus cryptic virus,CiLCV)是近年来发现的一种新型双链RNA病毒,可引起西瓜植株叶片皱缩和马赛克状坏死[1,2]。2013年最早在以色列被发现和报道,2017年在我国河南省首次被检出[1,2]。最近,该病毒陆续在欧洲、亚洲等多个国家被检出,而且呈现检出率高、种子传播等特点[1,3]。目前,已有多项研究报道指出CiLCV常与西瓜花叶病毒(watermelon mosaic virus,WMV)、小西葫芦黄花叶病毒(zucchini yellow mosaic virus,ZYMV)、黄瓜绿斑驳花叶病毒(cucumber green mottle mosaic virus,CGMMV)、甜瓜蚜传黄化病毒(melon aphid-borne yellows virus,MABYV)、甜瓜坏死斑点病毒(melon necrotic spot virus,MNSV)混合发生,极易对西瓜生产造成严重危害[1,2,3];该病毒能否侵染其他葫芦科作物尚不明确,亟需开展系统研究。因此,建立一种准确、灵敏、快速的CiLCV检测技术,监测主要葫芦科作物西瓜、甜瓜、黄瓜、南瓜砧木的种苗携带CiLCV情况,可从源头防止病害的发生和传播,对于病害的科学防控和抗病育种具有重要意义。【前人研究进展】2013年,以色列研究者利用高通量测序技术发现感染MNSV的西瓜植株上存在一种新型双链RNA病毒,即CiLCV,该病毒依赖RNA的RNA聚合酶(RNA dependent RNA polymerase,RdRP)和外壳蛋白(capsid protein,CP)序列分别与萝卜潜隐病毒(Raphanus sativus cryptic virus,RsCV)、辣椒潜隐病毒(pepper cryptic virus,PCV)同源性较高,因此被命名为西瓜潜隐病毒[1]。2017年,XIN等[3]进一步获得了CiLCV的全基因组序列,并建立了CiLCV RT-PCR检测技术,通过对66份西瓜样品进行检测,其中37份样品检出了CiLCV,检出率高达56%,且10个不同品种的西瓜种子均可传播CiLCV。目前,CiLCV的检测方式仅有RT-PCR和高通量测序技术[1,2,3]。尽管RT-PCR技术具有检测便捷、操作简单的优势,但检测灵敏度不强,对于低丰度病毒样品的检测效率不高。高通量测序技术虽然具有检测灵敏度强、检测范围广、效率高的特点,但检测过程耗时费力,成本较高,不适于口岸或田间进行快速检测和鉴定[4]。近年来,实时荧光定量PCR技术因其高灵敏度、高特异性以及可定量分析等优点,成为检测植物病原的重要技术之一[4,5,6,7,8,9,10,11]。王艳娇等[5]研究建立了柑橘脉突病毒(citrus vein enation virus,CVEV)的实时荧光定量PCR检测技术;丁天波等[6]应用实时荧光定量PCR技术建立了番茄褪绿病毒(tomato chlorosis virus,ToCV)鉴定体系;李文学等[8]建立了葡萄霜霉病菌(Plasmopara viticola)的实时荧光定量PCR检测技术;贺振等[11]建立了莲藕中甘薯潜隐病毒(sweet potato latent virus-lotus,SPLV-lotus)实时荧光定量PCR检测技术。另外,基于TaqMan探针法实时荧光定量PCR检测技术(TaqMan-qPCR)具有显著降低荧光背景信号干扰的优势,显著增强了检测病毒的灵敏度、稳定性和准确性[12,13,14,15]。王英超等[14]利用TaqMan-qPCR技术建立的大蒜黑腐病菌(Embellisia allii)检测方法灵敏度可达140 pg。【本研究切入点】近年来,农作物种苗产业发展迅速,特别是蔬菜种苗年交易量约3 000亿株,产值近2 000亿元。种苗带毒现象已经成为影响产业发展的重要问题,但目前有关种苗带毒或带菌的研究鲜有报道。亟需建立CiLCV快速、高效检测方法。【拟解决的关键问题】根据CiLCV保守序列设计特异性扩增引物,构建高灵敏度、稳定性的TaqMan-qPCR检测技术;利用该技术研究我国主要葫芦科作物种苗携带CiLCV情况,为从源头控制该病传播避免其扩散流行提供技术支撑。1 材料与方法
试验于2019年2月至2021年2月在北京市农林科学院蔬菜种质改良北京市重点实验室和农业农村部蔬菜种子质量监督检验测试中心完成。1.1 试验材料
CiLCV为本研究组2019年在北京发现,供试CGMMV、黄瓜花叶病毒(cucumber mosaic virus,CMV)、甜瓜内源病毒(Cucumis melo endornavirus,CMEV)、瓜类褪绿病毒(cucurbit chlorotic yellows virus,CCYV)、南瓜花叶病毒(squash mosaic virus,SqMV)、番茄斑萎病毒(tomato spotted wilt virus,TSWV)、WMV、ZYMV为实验室保存。12份西瓜、甜瓜、黄瓜与南瓜砧木种苗为北京、山东、辽宁3个省(直辖市)葫芦科作物种苗主产区随机选取,作为检测鉴定种苗携带CiLCV的试验材料(表1)。
Table 1
表1
表1主要葫芦科作物待测种苗信息表
Table 1
| 序号 Series | 葫芦科作物种苗 Cucurbits seedling | 来源 Origin | 序号 Series | 葫芦科作物种苗 Cucurbits seedling | 来源 Origin | |
|---|---|---|---|---|---|---|
| 1 | 西瓜种苗Watermelon seedling | 北京Beijing | 7 | 黄瓜种苗Cucumber seedling | 辽宁Liaoning | |
| 2 | 西瓜种苗Watermelon seedling | 北京Beijing | 8 | 甜瓜种苗Melon seedling | 北京Beijing | |
| 3 | 西瓜种苗Watermelon seedling | 北京Beijing | 9 | 甜瓜种苗Melon seedling | 辽宁Liaoning | |
| 4 | 西瓜种苗Watermelon seedling | 山东Shandong | 10 | 南瓜砧木种苗Rootstock pumpkin seedling | 北京Beijing | |
| 5 | 黄瓜种苗Cucumber seedling | 北京Beijing | 11 | 南瓜砧木种苗Rootstock pumpkin seedling | 山东Shandong | |
| 6 | 黄瓜种苗Cucumber seedling | 山东Shandong | 12 | 南瓜砧木种苗Rootstock pumpkin seedling | 辽宁Liaoning |
新窗口打开|下载CSV
1.2 引物设计与合成
根据GenBank公布的CiLCV两条链的基因序列,设计了该病毒RT-PCR检测的特异引物对、基于TaqMan探针法的PCR引物和探针、用于扩增dsRNA1和dsRNA2全长的引物对。引物均由生工生物工程(上海)股份有限公司合成,引物序列见表2。Table 2
表2
表2CiLCV检测引物序列
Table 2
| 引物名称 Primer name | 序列 Sequence (5′-3′) | 目的 Purpose |
|---|---|---|
| CiLCV-R1-F | AGAATTTTCCCCCAGTCAAC | CiLCV-dsRNA1全长克隆 Full length cloning of CiLCV-dsRNA1 |
| CiLCV-R1-R | AGAAGGTAAGGGTTAAATAAC | |
| CiLCV-R2-F | AGAATTTTCCCCCAGTCAAC | CiLCV-dsRNA2全长克隆 Full length cloning of CiLCV-dsRNA2 |
| CiLCV-R2-R | AGAAGGTAAGGGTTAAATAAC | |
| CiCLV-588-F | TCCAGACGTTGGCTACACAC | RT-PCR检测CiLCV CiLCV detection using RT-PCR |
| CiCLV-588-R | ATTGCGAACCTCTCAGGTGG | |
| DCiLCV-F1 | AGAGGCACAGATGATGGATTTGG | TaqMan-qPCR检测CiLCV CiLCV detection using TaqMan-qPCR |
| DCiLCV-P1 | AGCAGACCTTGAATTGATTACGGGTTC | |
| DCiLCV-R1 | CATCCAGAAAGGCACTGCTCAT | |
| DCiLCV-F2 | TGCCATTGAGACTGCTGTTCC | TaqMan-qPCR检测CiLCV CiLCV detection using TaqMan-qPCR |
| DCiLCV-P2 | AGTACGACATGTATGGGGTCGCGC | |
| DCiLCV-R2 | TCTGGGTACACTAACTGTCGGATC |
新窗口打开|下载CSV
1.3 植物总RNA提取
利用TRIZOL法(Invitrogen,美国)提取植物总RNA。具体方法为将叶片加液氮速冻后研磨成粉末状,并加入1 mL的Trizol试剂和200 μL氯仿振荡混匀;离心后取上清,加入等体积的异丙醇,放于-20℃冰箱沉淀;最后,用75%的乙醇洗涤沉淀两次,溶于50 μL DEPC处理的ddH2O中,-20℃保存备用。1.4 高通量测序
小RNA建库试剂盒购自Illumina公司(TruSeq Small RNA Sample Prep Kits,美国)。将提取的植物总RNA 利用Agilent 2100进行质检,合格的样品连接5′和3′接头,并进行反转录和PCR,产物经过电泳和回收后上机测序,测序平台为Illumina Hiseq 2000(Illumina 公司,美国)。测序的数据(raw data)经过过滤杂质等获得clean reads;将clean reads通过软件Velvet进行短序列组装(k-mer值设为17)[16]。组装的重叠群(contig)与NCBI GenBank数据库中的病毒信息进行比对,从而分析出潜在的已知病毒或类病毒,或者发现新的病毒或类病毒;并进一步通过生物学试验进行验证。1.5 RT-PCR
利用HiScript® II One Step RT-PCR Kit(Dye Plus)(诺唯赞,中国)进行RT-PCR反应。50 μL反应体系:2×One Step Mix(Dye Plus)25 μL,上下游引物(10 μmol·L-1)各2 μL,RNA模板1 μL(100 ng·μL-1),One Step Enzyme Mix 2.5 μL,ddH2O补足50 μL。反应条件:50℃预变性30 min;94℃预变性3 min;94℃变性30 s,55℃退火30 s,72℃延伸30 min,30个循环;72℃延伸10 min,4℃ ∞。PCR产物经1.5%琼脂糖凝胶电泳检测后,凝胶成像系统分析结果。1.6 克隆与测序
利用CiLCV全长克隆引物对CiLCV-R1-F/R和CiLCV-R2-F/R(表2)进行RT-PCR扩增,将PCR产物回收并克隆到PCE2TA/Blunt-Zero vector载体上(诺唯赞,中国),获得质粒pCiLCV-R1和pCiLCV-R2。测序后获得该病毒dsRNA1和dsRNA2全长序列信息,并与XIN等[3]报道的CiLCV病毒序列进行同源性比对分析。1.7 TaqMan-qPCR检测
1.7.1 TaqMan-qPCR反应体系和程序 利用实时荧光定量PCR仪CFX96 Touch(伯乐公司,美国)进行CiLCV TaqMan-qPCR检测技术研究。一步法TaqMan-qPCR使用HiScript II One Step qRT-PCR Probe Kit(诺唯赞,中国)。反应体系:2×One Step Q Probe Mix 10 μL;One Step Q Probe Enzyme Mix 1 μL;50×ROX Reference Dye 1 0.4 μL;正向引物(10 μmol·L-1)0.4 μL;反向引物(10 μmol·L-1)0.4 μL;探针(10 μmol·L-1)0.4 μL;ddH2O补足20 μL。反应程序:50℃反转录15 min;95℃变性30 s,95℃变性10 s,60℃退火30 s(收集荧光信号),40个循环。普通TaqMan-qPCR采用2×Real Star Probe Fast Mixture(with ROXII)(ABI公司,美国)。反应体系:2×Real Star Probe Fast Mixture(with ROXII),10 μL;正向引物10 μmol·L-1,0.5 μL;反向引物 10 μmol·L-1,0.5 μL;探针(10 μmol·L-1)0.5 μL;ddH2O补足20 μL。反应程序:95℃变性2 min,95℃变性15 s,60℃退火30 s(收集荧光信号),40个循环。1.7.2 TaqMan-qPCR特异性检测 以感染CiLCV的西瓜叶片总RNA为阳性对照,以感染CGMMV、CMV、CMEV、CCYV、SqMV、TSWV、WMV、ZYMV的植物总RNA为试验材料,进行TaqMan-qPCR检测分析,根据其他病毒是否呈现有效扩增,综合确定TaqMan-qPCR技术的检测特异性。
1.7.3 TaqMan-qPCR灵敏度检测 以CiLCV dsRNA1的克隆载体pCiLCV-R1为模板(浓度约为11.8 ng·μL-1,溶液1),用无菌水以10倍梯度进行稀释,获得溶液2—7,浓度依次约为11.8×10-1—11.8×10-6 ng·μL-1。
以溶液1—7为模板,按照普通TaqMan-qPCR反应体系进行方法的灵敏度检测。以CiLCV克隆载体pCiLCV-R1的拷贝数为横坐标,循环数Cq值为纵坐标,绘制标准曲线。质粒拷贝数=[质粒浓度(ng·μL-1)×质粒体积(μL)×6.02×1023]/[(载体长度bp+片段长度bp)×660 g·mol-1]。同时,利用上述稀释溶液进行PCR检测,比较TaqMan-qPCR检测灵敏度。
2 结果
2.1 西瓜田间症状与病毒鉴定
2019年在北京西瓜田进行病害调查时发现部分西瓜植株表现出叶片卷曲、畸形和变小等症状(图1-A、1-B和1-C)。提取疑似感染病毒病害的西瓜叶片的总RNA,并进行小RNA测序。获得初始reads约为24 545 770条,测序数据过滤后获得22 860 908条,占总测序数据的93.14%。进一步对获得序列的长度和数量进行分析,发现获得的小RNA的长度主要分布在18—30 nt,其中长度为24 nt的小RNA含量约为12 514 525 reads,占总clean reads的54.74%;其次为21 nt的小RNA含量约为3 413 397,占clean reads的14.93%(图1-D),表明测序数据可达到后续分析要求。图1
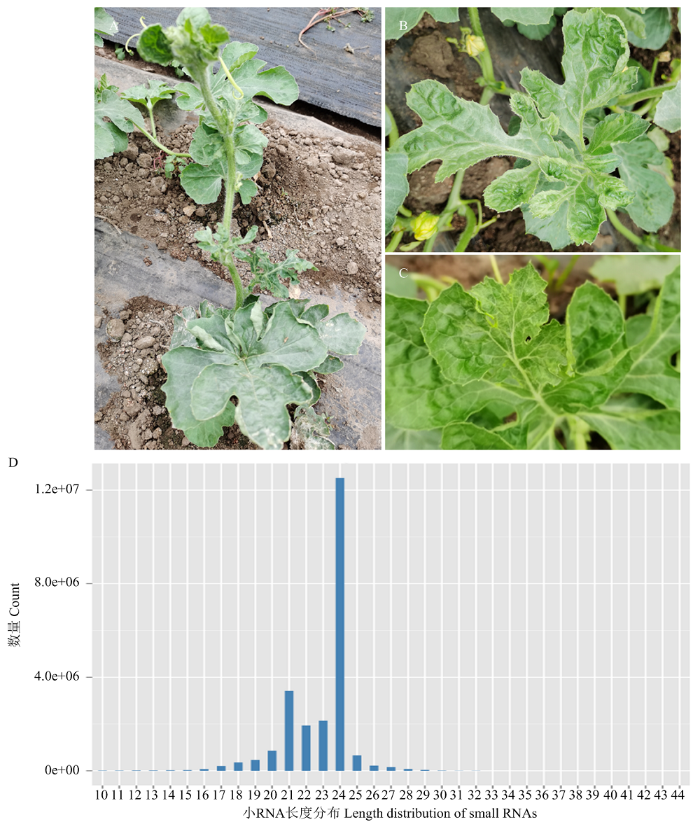 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1西瓜植株感染CiLCV病毒症状及小RNA测序片段长度分布
A、B/C为西瓜植株感染CiLCV症状;D为小RNA测序片段长度分布图
Fig. 1The symptoms of watermelon infected with CiLCV and length distribution of small RNAs sequencing
A、B/C are the symptoms of watermelon infected with CiLCV;D is the length distribution of small RNAs sequencing
使用Velvet 软件(k-mer值设为17)对小RNA进行组装,获得contig与NCBI数据库进行BLASTn和BLASTx比对,发现有17条contig序列与CiLCV基因组具有同源性,未发现其他病毒。根据NCBI上发表的CiLCV序列,设计可特异性扩增RNA1部分序列的引物对CiLCV-588-F/R(表2),利用RT-PCR技术对样品进行扩增验证,获得588 bp的PCR产物(图2),通过测序和BLASTn分析,发现与CiLCV的dsRNA1高度同源(GenBank number KY081285)。
图2
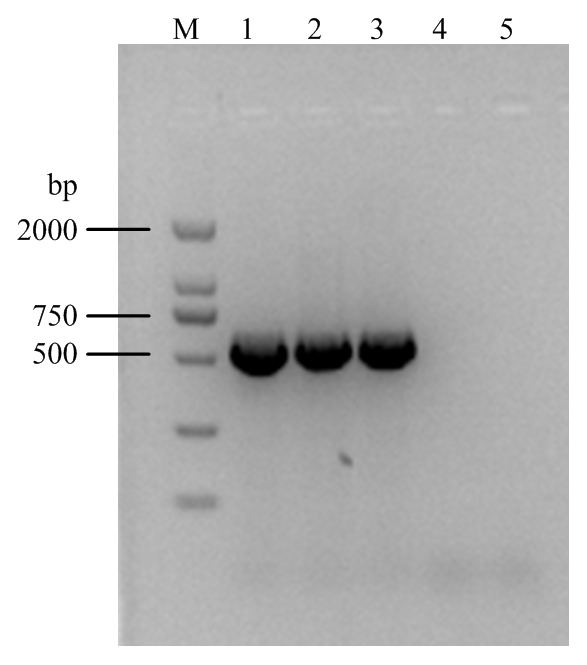 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2CiLCV的RT-PCR检测
M:DL2000 marker;1—3:样品1—3的PCR扩增产物The PCR amplified product of samples 1-3;4、5:阴性对照Negative control group
Fig. 2The detection of CiLCV by RT-PCR
CiLCV属于双分病毒科(Partitiviridae),其基因组由两条dsRNA片段组成。其中dsRNA-1全长1 603 nt,含有一个编码RdRp的ORF;dsRNA-2全长1 466 nt,含有一个编码CP的ORF。根据已发表的CiLCV基因组序列信息,设计引物对CiLCV-R1-F/R和CiLCV-R2-F/R(表2)用于扩增CiLCV的dsRNA1和dsRNA2全长,并克隆到载体上进行测序。分别获得约1 603和1 466 bp的产物,经过比对分析发现与XIN等[3]在河南省鉴定出的CiLCV dsRNA1和dsRNA2序列信息(GenBank number KY081285、KY081284)同源性分别高达99.4%和99.8%。
2.2 CiLCV TaqMan-qPCR检测特异性
设计基于CiLCV-dsRNA1的两对探针引物(序列见表2),建立了CiLCV的TaqMan-qPCR检测技术。利用两对探针引物对CiLCV阳性样品进行TaqMan-qPCR扩增检测,以其他8种常见病毒(CGMMV、CMV、CMEV、CCYV、SqMV、TSWV、WMV、ZYMV)样品作为对照,研究引物的特异性。结果表明两对探针在CiLCV阳性样品中存在有效扩增(蓝色和红色曲线);但探针1特异性在含有其他8种病毒样品中有扩增(绿色曲线),探针2在其他样品无有效扩增(绿色曲线)(图3-A);表明探针2的特异性优于探针1;此外,阳性样品利用探针1的检测Cq值约20.51(蓝色曲线),而探针2的检测Cq值约15.85(红色曲线),说明探针2的灵敏度也高于探针1。因而,将阳性样品的总RNA(浓度约100 ng·μL-1)进行10倍梯度稀释,检测两个探针的灵敏度。结果证明当总RNA稀释到1×10-1 ng·μL-1时,探针1的Cq值为31.34(蓝色),而探针2的Cq值为27.75;而当总RNA稀释到1×10-2 ng·μL-1时,探针2的Cq值接近32,表明探针2的灵敏度优于探针1(图3-B)。图3
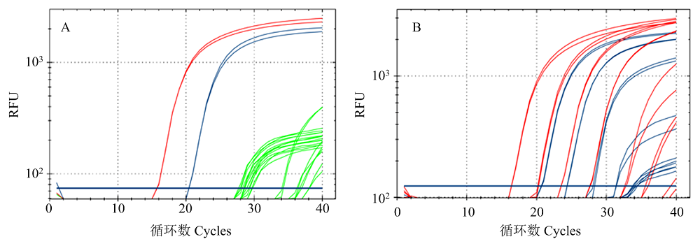 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3两对检测 CiLCV的探针特异性(A)和灵敏度(B)分析
蓝色和红色曲线分别对应CiLCV的探针1和探针2的扩增结果,绿色曲线代表其他8种病毒扩增曲线
Fig. 3Analysis of specificity (A) and sensitivity (B) of detecting CiLCV with two sets of probes
The blue and red curves indicate the amplification results of primer set 1 and primer set 2 for CiLCV, respectively, while the green curves indicate the amplification results of the other eight viruses
2.3 CiLCV TaqMan-qPCR检测灵敏度
由图3可知,探针2在检测感病样品的特异性和灵敏度上显著优于探针1。为进一步研究CiLCV TaqMan-qPCR检测技术的最低检测限度,排除不同感病样品总RNA对检测结果的影响,进一步构建了CiLCV的克隆载体建立探针2的标准曲线。利用RT-PCR技术扩增CiLCV-dsRNA1全长,并克隆到载体上。获得CiLCV的质粒pCiLCV-R1,浓度约为11.8 ng·μL-1,根据质粒拷贝数公式可计算出溶液1中1 μL质粒的拷贝数为2×109。以此为溶液1,进行10倍梯度稀释,依次获得溶液2—7。以溶液1—7为模板进行TaqMan-qPCR。由建立的标准曲线可知(图4-B),随着CiLCV样品浓度降低,Cq值逐渐递增且两者之间呈现良好线性关系,标准曲线斜率为-3.506,决定系数R2=0.996,建立的方程为y=-3.506x+32.608(其中y代表Cq值,x代表拷贝数)。结果表明,TaqMan- qPCR检测技术最低可检出溶液7,即最低可检测出2×103个拷贝的病毒(图4-A)。而RT-PCR引物对质粒pCiLCV-R1进行扩增时,溶液5可见清晰扩增条带,溶液6时仅有微弱且不稳定的扩增条带(图5)。表明TaqMan-qPCR技术检测CiLCV的灵敏度是RT-PCR技术的100倍。图4
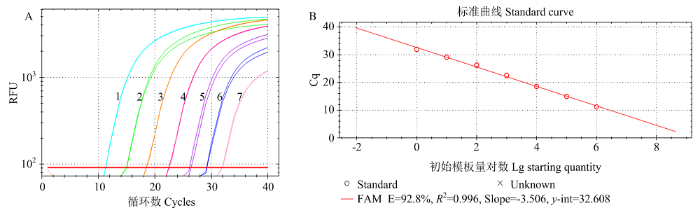 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4CiLCV的TaqMan-qPCR检测灵敏度分析(A)和标准曲线建立(B)
溶液1—7的模板浓度依次为11.8×100—11.8×10-6 ng·μL-1
Fig. 4The analysis of detection sensitivity of TaqMan-qPCR (A) and establishment of standard curve (B) for CiLCV
The template concentration of solution 1-7 is 11.8×100-11.8×10-6 ng·μL-1
图5
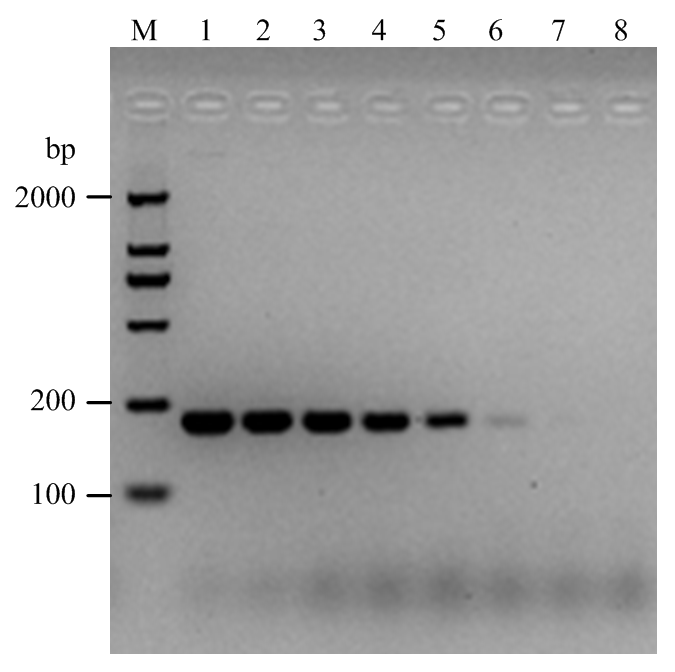 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5CiLCV的RT-PCR灵敏度检测
M:DL2000 marker;1—7:溶液1—7的PCR扩增产物The PCR results for sample 1-7;8:阴性对照Negative control
Fig. 5The detection sensitivity of RT-PCR for CiLCV
2.4 主要葫芦科作物种苗携带CiLCV情况
CiLCV可以通过种子传到种苗上,因而存在CiLCV随着种子、种苗运输在国内扩散的风险。随机抽取了北京、山东、辽宁等我国北方葫芦科作物主产区种苗,利用TaqMan-qPCR检测种苗携带CiLCV情况,重点考察不同地区不同来源的西瓜、黄瓜、甜瓜与南瓜(砧木)是否携带CiLCV。以表1中列出的12份葫芦科作物种苗为样品,每份样品混合提取植物总RNA,应用已建立的CiLCV RT-PCR和TaqMan-qPCR方法检测种苗携带CiLCV情况。结果发现有1份来自北京的西瓜种苗携带CiLCV,而其余11份葫芦科作物种苗同阴性对照(无菌水)均未出现扩增峰型(图6-A),该结果与RT-PCR鉴定结果一致(图6-B),表明我国葫芦科作物主产区种苗携带CiLCV情况总体不高,而且除西瓜外的其他葫芦科作物如甜瓜、黄瓜和南瓜砧木种苗均未检出CiLCV,特别是作为葫芦科作物重要的嫁接砧木南瓜种苗未检出CiLCV。但北京西瓜种苗检出CiLCV的现象值得业界高度警惕。
图6
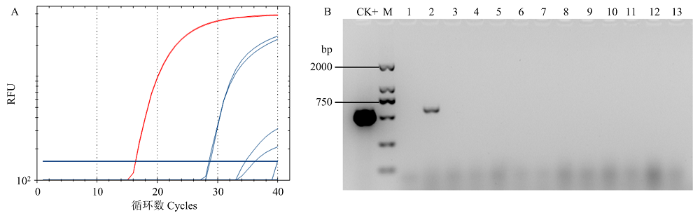 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6主要葫芦科作物种苗携带CiLCV的TaqMan-qPCR 检测(A)和RT-PCR检测(B)
A:红色曲线表示阳性对照样品扩增结果,蓝色曲线表示12份待测样品扩增结果,最左侧蓝色曲线表示感病样品扩增结果The red curves indicate the amplification results of positive control samples, the blue curves indicate the amplification results of the 12 test samples, and the left blue curve presents the positive sample。B:CK+:阳性样品Positive control;M:DL2000 marker;1—12:样品1—12的PCR扩增产物The PCR results for sample 1-12;13:阴性对照Negative control
Fig. 6The detection of CiLCV in main cucurbits crop seedlings with TaqMan-qPCR (A) and RT-PCR (B)
3 讨论
目前,RT-PCR技术、实时荧光定量PCR技术(RT- qPCR)、环介导等温核酸扩增技术(loop-mediated isothermal amplification,LAMP)均为检测植物病毒的有效手段。然而,多项研究证明实时荧光定量PCR技术与RT-PCR技术相比在检测灵敏度方面具有显著优势[4,5,6,7,8,9,10,11]。王艳娇等[5]建立的实时荧光定量PCR能够检出70 copies/μL的CVEV,检测灵敏度是RT-PCR的100倍;丁天波等[6]利用ToCV的外壳蛋白序列建立的实时荧光定量PCR检测技术最低可检出2.7×103 copies/μL,灵敏度是RT-PCR的100倍;贺振等[11]建立的SPLV-lotus实时荧光定量PCR技术能够检测到70 copies/μL的病毒,灵敏度是RT-PCR的100倍;高利利等[17]基于ToCV的HSP(heat shock protein)序列建立的实时荧光定量PCR检测技术扩增效率高达99.8%。另外,尽管LAMP技术具有灵敏度高、特异性好、扩增时间短等优点,应用在柑橘叶斑驳病毒(citrus leaf blotch virus,CLBV)、苹果褪绿叶斑病毒(apple chlorotic leaf spot virus,ACLSV)、甘薯羽状斑驳病毒(sweet potato feathery mottle virus,SPFMV)、植物青枯菌(Ralstonia solanacearum)、草莓轻型黄边病毒(strawberry mild yellow edge virus,SMYEV)、柑橘衰退病毒(citrus tristeza virus,CTV)等检测上[18,19,20,21,22,23,24]。但与实时荧光定量PCR技术相比,LAMP技术存在引物设计较为繁琐,检测成本较高,容易产生假阳性,而且难以进行定量分析的问题[8,25-26]。而且,基于TaqMan探针法的实时荧光定量qPCR技术具有更好的检测灵敏度,王英超等[14]利用TaqMan-qPCR技术建立的大蒜黑腐病菌检测方法灵敏度可达140 pg。本研究建立的TaqMan-qPCR检测技术,对CiLCV具有较高扩增特异性,检测灵敏度可达到2×103 copies,为RT-PCR检测技术的100倍,这一结论与前人研究结果基本一致。综上,实时荧光定量PCR技术是一种操作便捷,特异性强,灵敏度高,并且可以进行定量检测鉴定病害的高效技术。本研究建立的TaqMan-qPCR技术能够快速、精准、高效鉴定CiLCV。近年来,农作物种苗产业发展迅速,种苗传播成为当前病害远距离扩散的重要方式,然而有关种苗带毒或带菌的研究报道较少[27,28,29]。有研究表明葫芦科作物种子可以携带多种病毒,如CGMMV、ZYMV、CMV、SqMV、MNSV以及CiLCV等[1-3,27]。吴会杰等[27]研究发现,葫芦科作物西瓜、甜瓜种子的CGMMV带毒率在93.85%以上,而传毒率则为2.83%以下。尽管有研究表明大多数种传病毒的传毒率通常不高,但只要携带病毒的种子存在就有可能引起作物的重大损失[29,30,31]。DINANT等[31]研究发现,莴苣种子种传率超过0.1%,就足以造成莴苣花叶病毒(lettuce mosaic virus,LMV)的流行。近年来,我国西瓜、甜瓜等葫芦科作物主产区暴发CGMMV,已成为制约西瓜、甜瓜产业发展的重大难题,多个主产区陆续检出CGMMV,变成了疫区,导致这一问题的原因可能是由于种苗带毒和远距离流通引起的。本研究中北京抽取的一份西瓜种苗携带CiLCV,鉴于植物病毒可以通过多种途径进行传播,如农事操作、昆虫媒介等,一旦带毒种苗流入生产环节,很可能造成病毒大规模的扩散和暴发,将对育苗企业或菜农造成潜在重大损失。因此,在育苗早期或育苗企业全面开展基于高效精准检测技术的病毒鉴定和监测预警,是从源头解决种苗带毒和远距离传播的关键。
有研究指出植物感病早期或者处于潜育期的感病叶片往往不表现症状,而且叶片内病毒含量太低,难以采用传统生物学检测方法进行鉴定[8]。李文学等[8]建立的葡萄霜霉病菌实时荧光定量PCR技术,可以检测到病原菌侵入叶片6 h后的低侵染量,为葡萄霜霉病的早期诊断与预测提供了技术支持。本研究构建的CiLCV TaqMan-qPCR检测技术灵敏度高、特异性强,可以应用于检测西瓜和其他葫芦科作物种子、种苗、植株,同时能够满足检测低丰度病毒样品的需求。不同寄主同种病毒的传毒率会不同,种子传毒率还受环境条件、母株感病时间及其交互作用的影响[8,29]。本研究中甜瓜、黄瓜和南瓜砧木种苗均未检出CiLCV,这一结果与SELA等在美国和厄瓜多尔的研究结论一致[1]。仅有来自北京一家育苗企业(序号1)的一份西瓜种苗检出了CiLCV,表明我国葫芦科作物主产区种苗的CiLCV发病率总体不高,该结果与SELA等[1]在美国和XIN等[3]在我国河南的研究结论略有不同,推测原因是CiLCV传播至我国时间不长尚未造成大面积扩散。另外,本研究中CiLCV发病率不高可能与抽取样本数量有限有关。因此,应尽快对我国葫芦科作物主产区开展CiLCV发病情况检测鉴定,尽早切断该病毒的传播扩散,防止其迭代传播造成更强致病性。而本研究建立的CiLCV TaqMan-qPCR检测技术可为将来开展种子、种苗带毒快速精准鉴定提供可靠技术支持。
4 结论
针对我国最近发现的西瓜潜隐病毒(CiLCV)建立了基于TaqMan-qPCR的检测技术,该检测技术特异性强、灵敏度高,能够满足快速、精准、定性、定量鉴定需求,适于口岸和实验室高效鉴定。我国葫芦科作物主产区的西瓜种苗携带CiLCV,尽管与国外相比总体上发病率不高,但仍亟需引起重视,建议尽快开展该病毒大面积检测鉴定,尽早控制其传播途径,避免其扩散流行对产业造成更大损失。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
DOI:10.1007/s11262-013-0937-8URL [本文引用: 9]
DOI:10.1016/j.virusres.2017.02.009URL [本文引用: 4]
DOI:10.1007/s00705-017-3461-7URL [本文引用: 8]
[本文引用: 3]
[本文引用: 3]
[本文引用: 4]
[本文引用: 4]
[本文引用: 4]
[本文引用: 4]
[本文引用: 2]
[本文引用: 2]
[本文引用: 7]
[本文引用: 7]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 4]
[本文引用: 4]
[本文引用: 1]
DOI:10.3748/wjg.v11.i4.508URL [本文引用: 1]
[本文引用: 3]
[本文引用: 3]
DOI:10.1016/S2095-3119(15)61134-0URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1002/rmv.v18:6URL [本文引用: 1]
DOI:10.1006/bbrc.2001.5921URL [本文引用: 1]
[本文引用: 3]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
DOI:10.1016/0966-842X(96)10016-0URL [本文引用: 1]
DOI:10.1111/ppa.1992.41.issue-5URL [本文引用: 2]
