2.
Development and Application of Plasmid Reference Molecule for Genetically Modified Rapeseed Screening
LI Jun1, LI Xia-ying2, WANG Jing-qian2, Zhai Shanshan1, CHEN Zi-yan2, GAO Hong-fei1, LI YunJing1, WU Gang1, ZHANG Xiu-jie2, WU Yu-hua11. 2.
责任编辑: 李莉
收稿日期:2019-08-7接受日期:2019-11-13网络出版日期:2020-04-01
| 基金资助: |
Editor:
Received:2019-08-7Accepted:2019-11-13Online:2020-04-01
作者简介 About authors
李俊,E-mail:lijuner126@126.com。

摘要
关键词:
Abstract
Keywords:
PDF (3168KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
李俊, 李夏莹, 王颢潜, 翟杉杉, 陈子言, 高鸿飞, 李允静, 吴刚, 张秀杰, 武玉花. 转基因油菜筛查阳性质粒分子的研制及应用[J]. 中国农业科学, 2020, 53(7): 1322-1337 doi:10.3864/j.issn.0578-1752.2020.07.003
LI Jun, LI Xia-ying, WANG Jing-qian, Zhai Shanshan, CHEN Zi-yan, GAO Hong-fei, LI YunJing, WU Gang, ZHANG Xiu-jie, WU Yu-hua.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】转基因油菜是四大转基因作物之一,是食用油和饲料饼粕的重要来源,已在加拿大、美国、澳大利亚等国家大规模商业化种植。为了满足食用油和饼粕供应,中国每年需从国外进口油菜籽,2015年进口量超过500万吨,其中90%以上为转基因菜籽。中国也是油菜种植大国,转基因研究活跃,不断有品种进入田间试验阶段。转基因油菜是中国转基因生物安全监管的重要对象。转基因筛查检测是转基因生物安全监管的第一步,以转基因作物中常用的启动子、终止子和目的基因为检测靶标,通过筛查样品中常用基因元件的有无,可初步判定样品中是否含有转基因成分[1]。在转基因筛查时,如果设置的筛查靶标不能充分覆盖已有转基因产品,会导致部分转基因产品的漏检。而且,在进行转基因筛查检测时,常常面临缺乏标准样品的难题。因此,研究转基因油菜的筛查检测策略,并根据筛查靶标研制筛查检测用阳性质粒分子,对中国的转基因油菜安全监管具有重大意义。【前人研究进展】2004年,中国批准进口转基因油菜籽用作加工原料,为了对转基因油菜进行安全监管,一直大力推动转基因油菜检测技术标准体系的建设,现在中国已初步建立起转基因油菜检测技术标准体系,包括国家标准(GB/T)[2,3]、农业农村部公告[4,5,6,7,8]、出入境检验检疫行业标准[9,10,11,12]等。标准方法主要是基于基因组DNA扩增的PCR方法,检测靶标包括花椰菜病毒的35S启动子(P-CaMV35S)、胭脂碱合成酶NOS终止子(T-NOS)等调控元件[4,11],新霉素磷酸转移酶基因(NPTⅡ)、膦丝菌素乙酰转移酶基因(Bar)、膦丝菌素乙酰转移酶基因(PAT)等功能基因[5,6,7,8,9,10,11,12,13],以及批准进口的MS8、RF3、GT73等转化体[11]。虽然针对各个基因元件都发布了标准方法,但在转基因油菜筛查检测时,应该检测哪些靶标,还没有明确的规定。各家转基因检测实验室在检测转基因油菜时,都是自行设置检测靶标,导致实验室间的检测结果缺乏可比性。张丽等[13]曾基于商业化转基因油菜中基因元件使用频率的分析,确定了转基因油菜的筛查检测策略,认为检测P-CaMV35S、玄参花叶病毒35S启动子(P-FMV35S)、Bar、5-烯醇式丙酮酸莽草酸-3-磷酸合酶基因(CP4-EPSPS)、PAT和T-NOS共6个基因元件,可100%检测出批准进口转基因油菜品种。刘冰[14]研究认为检测P-CaMV35S、T-NOS、Bar、PAT、CP4-EPSPS和NPTⅡ 6个靶标元件的组合,理论上可筛查92%的已知商业化转基因油菜转化事件。随着转基因油菜新品种的推出,采用这两个筛查策略均会导致部分转基因油菜品种发生漏检。标准物质或标准样品是进行转基因检测的物质基础,应用标准物质可保证检测结果的准确性、可靠性和可比性。基体标准物质的研制和生产受原材料供应等方面的限制,而质粒标准分子的研制则不依赖转基因原材料,因此,质粒标准分子成为基体标准物质的最佳替代品,适宜在转基因定性检测中用作阳性标准样品[15]。迄今为止,中国仅研制出了一个转基因油菜T45质粒标准物质(GBW(E)100340),该标准物质仅能用于油菜T45转化体的特异识别和定量,不能用于转基因油菜筛查检测。为了满足转基因油菜的检测需求,LI等[16]将RF1、RF2、MS1、MS8、Topas 19/2、Oxy235、RT73和T45共8个转化体的靶标序列融合构建到一个质粒分子上,研制出一个多靶标的油菜质粒分子,但该质粒分子不能用于转基因油菜筛查检测。WU等[17]将P-CaMV35S、P-FMV35S、胭脂碱合成酶NOS启动子(P-NOS)、Bar、NPTII、潮霉素磷酸转移酶基因(HPT)、磷酸甘露糖异构酶基因(Pmi)、T-NOS、花椰菜病毒的35S终止子(T- CaMV35S)、TL-DNA基因7终止子(T-g7)和E9基因3’终止子(T-e9)11个外源基因元件和玉米、油菜、大豆、棉花、水稻和小麦六大作物内标基因的靶标序列,融合构建到一个质粒上,研制出一个通用的筛查质粒分子,但该分子不是针对转基因油菜研制,在应用时会导致个别转基因油菜品种的漏检。徐俊锋等[18]构建了油菜筛查质粒分子pMD-rape,以HMG I/Y作为油菜内标准基因,携带P-CaMV35S、P-FMV35S、花药组织特异基因TA29启动子(PTa29)、P-NOS、T-NOS、T-CaMV35S和T-g7 7个检测靶标序列,覆盖90%的转基因油菜品种,但该质粒分子只适用于通过农业部公告发布的标准,不适用于国家标准(GB/T)和出入境检验检疫行业标准(SN/T)。【本研究切入点】前期研究虽然曾提出转基因油菜的筛查检测策略,但存在检测参数设置不合理导致个别转化体漏检的问题。相应地,构建的转基因油菜筛查质粒分子也只能覆盖大部分转基因油菜品种。因此,在转基因油菜安全监管工作中,现有转基因油菜筛查策略和筛查用标准样品均难以满足现阶段检测需求。【拟解决的关键问题】本研究通过分析商业化转基因油菜和试验阶段转基因油菜的常用基因元件,提出转基因油菜的筛查检测策略,研制转基因油菜筛查检测用质粒标准分子,为转基因油菜的筛查检测提供阳性对照。进一步完善中国的转基因油菜检测、监测技术标准体系,有效发挥标准方法在转基因生物安全监管中的作用,提高中国对非授权转基因油菜产品非法扩散的检测、监测水平,为中国的转基因生物安全监管提供有效的技术支撑。1 材料与方法
1.1 材料
转基因油菜标准物质GT73(0304-B2)、RF1(0711-B2)、MS1(0711-A3)、RF2(0711-C2)、MS8(0306-F6)、RF3(0306-G5)、OXY235、T45(0208-A5)、Topas19 /2(0711-D3)、MON88302(1011-A)购自美国油脂化学家协会(American Oil Chemists’ Society,AOCS);转基因油菜73496(ERM- BF434b)的标准物质购自欧盟的标准物质与测量研究所(institute for reference materials and measurements,IRMM)。用pUC18质粒作为标准质粒分子的载体骨架。PCR引物和探针由上海生物工程技术服务有限公司合成。
1.2 质粒分子构建
确定转基因油菜筛查元件的核苷酸序列后,将各基因元件的核苷酸序列拼接到一起,送到上海生物工程有限公司人工合成核苷酸序列,将合成的核苷酸序列构建到pUC18质粒分子上。然后对质粒分子进行重测序,确定核苷酸序列的准确性。1.3 基因组DNA的提取
用Qiagen的基因组DNA提取试剂盒提取标准样品的基因组DNA。用紫外分光光度计测定所提取基因组DNA的纯度和浓度,要求OD260/OD230>2.0,OD260/OD280为1.8—2.0。1.4 普通PCR检测
在普通PCR仪C1000 TouchTM (Bio-rad,USA)上进行PCR反应,PCR反应体系为25 μL,含20 ng基因组DNA、1×PCR buffer、5 mmol·L-1 MgCl2,200 μmol·L-1 dNTPs、400 nmol·L-1引物、1单位Taq酶(TaKaRa,Shiga,Japan)。PCR反应程序为94℃ 2 min;94℃ 20 s,60℃ 30 s,72℃ 30 s,35个循环;72℃ 2 min。2%琼脂糖凝胶电泳检测PCR产物,EB染色后,在凝胶成像仪中观测结果(Bio-rad,USA)。1.5 实时荧光PCR检测
在实时荧光PCR仪CFX96TM上进行实时荧光PCR反应,PCR反应体系为20 μL,含20 ng基因组DNA、1×TaqMan Universal PCR Master Mix(ABI,USA)、400 nmol·L-1 引物、200 nmol·L-1探针。反应程序为50℃预酶切2 min;95℃ UNG灭活10 min;95℃ 15 s,60℃ 1 min,50个循环。用CFX Manager™软件(Bio-rad,USA)分析数据。
2 结果
2.1 转基因油菜产业化和研发现状分析
查阅国际农业生物技术应用服务组织(The International Service for the Acquisition of Agri-biotech Applications,ISAAA)的“GMO Approved Database”数据库(Table 1
表1
表1ISAAA数据库中登记的转基因油菜品种
Table 1
| 类型 Type | 数目 Number | 名称 Name |
|---|---|---|
| 独立转化体 Event | 26 | 23-18-17、23-198、61061、73496、DHA Canola、GT200、GT73、Topas 19/2(HCN10、HCN92)、T45、MON88302、MPS961、MPS962、MPS963、MPS964、MPS965、MS1、MS11、MS8、OXY235、PHY14、PHY23、PHY35、PHY36、RF1、RF2、RF3 |
| 复合性状品种 Gene-stacked variety | 14 | 73496×RF3、HCN28×MON88302、MON88302×MS8×RF3、MON88302×RF3、MS1×MON88302、MS1×RF1、MS1×RF2、MS1×RF3、MS8×MON88302、MS8×RF3、MS8×RF3×GT73、RF1×MON88302、HCN92×MON88302、RF2×MON88302 |
新窗口打开|下载CSV
中国批准了耐除草剂油菜Ms1×Rf1、Ms1×Rf2、Ms8×Rf3、T45、Topas19/2、Oxy-235、GT73、RF3和MON88302 9个品种进口用作加工原料,涉及10个独立转化体MS1、RF1、RF2、MS8、RF3、T45、OXY235、Topas 19/2、GT73和MON88302。在中国曾做过安全评价,但还未批准进口的有2个品种,分别是MS8×RF3×GT73和73496,涉及1个新转化体73496。
2.2 确定转基因油菜筛查元件
确定转基因油菜筛查的基因元件要考虑2个因素,一个是基因元件的使用频率高;另一个是组合使用不同基因元件可达到最高的品种覆盖率,最好对已知品种覆盖率达到100%。统计分析转基因油菜中使用的基因元件,ISAAA数据库中有26个独立转基因油菜转化体,能查阅到完整遗传转化信息的有19个,包含中国已批准进口的10个转化体(MS1、RF1、RF2、MS8、RF3、T45、OXY235、Topas 19/2、MON88302和GT73)和可能将批准进口的转化体73496。转基因油菜不育系MS11拟取代MS8,用于商业化生产,但没有查阅到其完整的遗传转化信息。ISAAA数据库中提供的信息显示,MS11含有barnase表达框、Bar表达框和barstar表达框。LMO数据库中另外登记的28个转化体中有2个转化体含有PAT,6个转化体含有NPTⅡ,其余转化体未提供明确的外源元件及基因信息。油菜作为四大转基因作物之一,中国转基因研究非常活跃,在1992—2015年间有497篇与转基因油菜相关的论文发表[19]。在基因工程研究中,除了用Bar、NPTⅡ作为选择标记外,还大量用HPT作为选择标记。通过分析查阅到的转基因油菜品系中常用的外源元件和标记基因的使用频率,以及常用遗传转化载体中含有的调控元件和标记基因,确定用9个基因元件进行转基因油菜的筛查检测,分别是(1)P-CaMV 35S、(2)P-FMV 35S、(3)Bar、(4)PAT、(5)修饰的mCP4-EPSPS、(6)NPTⅡ、(7)T-NOS、(8)马铃薯蛋白酶抑制因子Ⅱ终止子(T-PinⅡ)和(9)HPT。9个基因元件在20个已知信息油菜转化体中的分布如表2所示。对这9个基因元件进行检测,可实现20个已知转基因油菜品种、LMO数据库中已知遗传转化信息品种的全覆盖,批准进口品种覆盖2—3次。通过将HPT作为检测参数,可覆盖部分实验室研究阶段的材料。
Table 2
表2
表29个筛查元件在油菜转化体中的分布
Table 2
| 编号 No. | 转化体 Event | P-CaMV35S | P-FMV35S | Bar | mCP4-EPSPS | HPT | NPTⅡ | PAT | T-NOS | T-PinⅡ |
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | T45 | √ | √ | |||||||
| 2 | GT73 / RT73 | √ | √ | |||||||
| 3 | GT200 | √ | √ | |||||||
| 4 | MS8 | √ | √ | |||||||
| 5 | MS1 | √ | √ | √ | ||||||
| 6 | RF1 | √ | √ | √ | ||||||
| 7 | RF2 | √ | √ | √ | ||||||
| 8 | RF3 | √ | √ | |||||||
| 9 | OXY235 | √ | √ | |||||||
| 10 | TOPAS19/2 | √ | √ | √ | ||||||
| 11 | Falcon GS40/ 90pHoe6/A | √ | √ | |||||||
| 12 | Liberator L62 | √ | √ | |||||||
| 13 | 23-18-17, 23-198 | √ | √ | √ | ||||||
| 14 | HCN10 | √ | √ | |||||||
| 15 | PHY14,PHY35 | √ | √ | |||||||
| 16 | PHY36 | √ | √ | |||||||
| 17 | 73496 | √ | ||||||||
| 18 | MON88302 | √ | ||||||||
| 19 | 61061 | √ | √ | |||||||
| 20 | MS11 | √ |
新窗口打开|下载CSV
2.3 确定9个筛查元件的靶标序列
9个筛查元件的完整核苷酸序列信息如表3所示,在转基因检测中,每个筛查元件仅有部分核苷酸序列用作检测靶标,因此,本研究仅将各元件的检测靶标序列构建到质粒分子中。在确定9个筛查元件的靶标序列时,要求拟构建的质粒分子不仅可用作普通PCR检测的阳性对照,还可用作实时荧光PCR检测的阳性对照;不仅适用于农业部发布标准,还适用于GB/T、SN/T和欧盟标准。查阅9个筛查元件的检测标准,包括GB/T检测标准[2,3]、农业农村部发布的检测标准(农业农村部公告)[4,5,6,7,8]、SN/T检测标准[9,10,11,12]和欧盟标准[20,21,22,23,24,25,26,27,28,29,30,31,32,33],收集各标准中针对这9个筛查元件的普通PCR方法和实时荧光PCR方法(表3)。分析各标准中普通PCR引物对和实时荧光PCR引物/探针组合在筛查元件核苷酸序列中的位置,确定拟构建到质粒上的各筛查元件的靶标核苷酸序列。选用HMG I/Y和CruA作为转基因油菜内标基因。各筛查元件的靶标序列长度及油菜内标基因HMG I/Y和CruA的核苷酸序列信息如表3所示。Table 3
表3
表3各筛查元件和油菜内标基因的核苷酸序列、标准方法信息
Table 3
| 元件/基因 Element/Gene | 序列长度 Length (bp) | 登录号 Accession No. | 靶标长度 Target size (bp) | PCR类型 PCR type | 引物/探针 Primer/Probe | 序列 Sequence(5′-3′) | 产物大小 Amplicon size (bp) | 标准 Standards |
|---|---|---|---|---|---|---|---|---|
| P-CaMV35Sq | 835 | AF485783.1 | 250 | CT-PCR | F | GCTCCTACAAATGCCATCATTGC | 195 | 1782-3-2012 [4] SN/T 1197-2016 [11] ISO 21569:2005 [20] |
| R | GATAGTGGGATTGTGCGTCATCCC | |||||||
| 35S-cf3 | CCACGTCTTCAAAGCAAGTGG | 123 | QL-ELE-00-004[21] | |||||
| 35S-cr4 | TCCTCTCCAAATGAAATGAACTTCC | |||||||
| RT-PCR | 35S-QF | CGACAGTGGTCCCAAAGA | 74 | 1782-3-2012[4] SN/T 1201-2014[9] SN/T 2705-2010[10] | ||||
| 35S-QR | AAGACGTGGTTGGAACGTCTTC | |||||||
| 35S-QP | TGGACCCCCACCCACGAGGAGCATC | |||||||
| F | GCCTCTGCCGACAGTGGT | 82 | GB/T 19495.4-2018[2] SN/T 1197-2016[11] SN/T 1204-2016[12] QL-ELE-00-012[22] | |||||
| R | AAGACGTGGTTGGAACGTCTTC | |||||||
| P | CAAAGATGGACCCCCACCCACG | |||||||
| p35s-F | ATTGATGTGATATCTCCACTGACGT | 101 | GB/T 33526-2017[3] | |||||
| p35s-R | CCTCTCCAAATGAAATGAACTTCCT | |||||||
| p35s-P | CCCACTATCCTTCGCAAGACCCTTCCT | |||||||
| NPTⅡ | 795 | AF485783.1 | 669 | CT-PCR | TN5-1 | GGATCTCCTGTCATCT | 173 | QL-ELE-00-003[23] |
| TN5-2 | GATCATCCTGATCGAC | |||||||
| APH2 short | CTCACCTTGCTCCTGCCGAGA | 215 | SN/T 1197-2016[11] | |||||
| APH2 reverse | CGCCTTGAGCCTGGCGAACAG | |||||||
| NPTIIF68 | ACTGGGCACAACAGACAATCG | 289 | 1782-2-2012[7] | |||||
| NPTIIR356 | GCATCAGCCATGATGGATACTTT | |||||||
| RT-PCR | F | AGGATCTCGTCGTGACCCAT | 183 | GB/T 19495.4-2018[2] SN/T 1197-2016[11] SN/T 2705-2010[10] SN/T 1201-2014[9] SN/T 1204-2016[12] | ||||
| R | GCACGAGGAAGCGGTCA | |||||||
| P | CACCCAGCCGGCCACAGTCGAT | |||||||
| qNPTIIF63 | CTATGACTGGGCACAACAGACA | 101 | 1782-2-2012[7] | |||||
| qNPTIIR163 | CGGACAGGTCGGTCTTGACA | |||||||
| qNPTIIFP90 | CTGCTCTGATGCCGCCGTGTTCCG | |||||||
| P-FMV35S | 596 | JN400388.1 | 393 | CT-PCR | FMV-1 | AAGCCTCAACAAGGTCAG | 196 | QL-ELE-00-010[25] |
| FMV-2 | CTGCTCGATGTTGACAAG | |||||||
| FMV35S-F1 | AAGACATCCACCGAAGACTTA | 210 | 1782-3-2012 [4] SN/T 1197-2016 [11] ISO 21569:2005 [20] | |||||
| FMV35S-R1 | AGGACAGCTCTTTTCCACGTT | |||||||
| RT-PCR | pFMV-F | CAAAATAACGTGGAAAAGAGCT | 78 | QL-ELE-00-015[24] SN/T 1197-2016[11] | ||||
| pFMV-R | TCTTTTGTGGTCGTCACTGC | |||||||
| pFMV | CTGACAGCCCACTCACTAATGC | |||||||
| FMV35S-QF | AAGACATCCACCGAAGACTTA | 210 | 1782-3-2012[4] SN/T 1201-2014[9] SN/T 2705-2010[10] | |||||
| FMV35S-QR | AGGACAGCTCTTTTCCACGTT | |||||||
| FMV35S-Qp | TGGTCCCCACAAGCCAGCTGCTCGA | |||||||
| F | CGAAGACTTAAAGTTAGTGGGCATCT | 79 | GB/T 19495.4-2018[2] SN/T 1204-2016[12] | |||||
| R | TTTTGTCTGGTCCCCACAA | |||||||
| P | TGAAAGTAATCTTGTCAACATCGAGCAGCTGG | |||||||
| mCP4-EPSPS | 1368 | JN400388.1 | 793 | CT-PCR | CP4 Synthetic F | GCATGCTTCACGGTGCAA | 108 | QL-ELE-00-019[26] |
| CP4 Synthetic R | TGAAGGACCGGTGGGAGAT | |||||||
| F | GACTTGCGTGTTCGTTCTTC | 204 | SN/T 1197-2016[11] | |||||
| R | AACACCGTTGAGCTTGAGAC | |||||||
| mCP4ESF | ACGGTGAYCGTCTTCCMGTTAC | 333 | 1861-5-2012[5] | |||||
| mCP4ESR | GAACAAGCARGGCMGCAACCA | |||||||
| RT-PCR | F | GCAAATCCTCTGGCCTTTCC | 146 | GB/T 19495.4-2018[2] | ||||
| R | CTTGCCCGTATTGATGACGTC | SN/T 1204-2016[12] | ||||||
| P | TCATGTTCGGCGGTCTCGCG- | SN/T 1197-2016[11] | ||||||
| Bar | 552 | MH973511.1 | 433 | CT-PCR | Pat-Bar Fwd | CGTCAACCACTACATCGAGACAA | 69 | QL-ELE-00-022[27] |
| Pat-Bar Rev | GTCCACTCCTGCGGTTCCT | |||||||
| bar-F | GAAGGCACGCAACGCCTACGA | 262 | 1782-6-2012[6] | |||||
| bar-R | CCAGAAACCCACGTCATGCCA | |||||||
| F | ACCATCGTCAACCACTACATCG | 430 | SN/T 1197-2016[11] | |||||
| R | GCTGCCAGAAACCCACGTCAT | |||||||
| RT-PCR | RapB-F1 | ACAAGCACGGTCAACTTCC | 60 | QL-ELE-00-014 [28] SN/T 1204-2016 [12] | ||||
| RapB-R1 | GAGGTCGTCCGTCCACTC | |||||||
| RapB-S1 | TACCGAGCCGCAGGAACC | |||||||
| F | ACAAGCACGGTCAACTTCC | 175 | GB/T 19495.4-2018[2] SN/T 1197-2016[11] SN/T 2705-2010[10] SN/T 1201-2014[9] | |||||
| R | ACTCGGCCGTCCAGTCGTA | |||||||
| P | CCGAGCCGCAGGAACCGCAGGAG | |||||||
| PAT | 579 | DQ156557.1 | 499 | CT-PCR | Pat-Pat Fwd | CCGCGGTTTGTGATATCGTT | 109 | QL-ELE-00-021[29] |
| Pat-Pat Rev | TCTTGCAACCTCTCTAGATCATCAA | |||||||
| PAT-F | GAAGGCTAGGAACGCTTACGA | 262 | 1782-6-2012[6] | |||||
| PAT-R | CCAAAAACCAACATCATGCCA | |||||||
| F | GTCGACATGTCTCCGGAGAG | 191 | SN/T 1197-2016[11] | |||||
| R | GCAACCAACCAAGGGTATC | |||||||
| RT PCR | Pat-F | CGCGGTTTGTGATATCGTTAAC | 108 | QL-ELE-00-025[30] | ||||
| Pat-R | TCTTGCAACCTCTCTAGATCATCAA | |||||||
| Pat-P | AGGACAGAGCCACAAACACCACAAGAGTG | |||||||
| PAT-KVM-5 | TTGAGGGTGTTGTGGCTGGTA | 68 | QT-ELE-00-002[31] | |||||
| Pat1-p | TGTCCAATCGTAAGCGTTCCT | |||||||
| PAT-KVM-6 | CTTCCAGGGCCCAGCGTAAGCA | |||||||
| F | GTCGACATGTCTCCGGAGAG | 191 | GB/T 19495.4-2018 [2] SN/T 1197-2016 [11] SN/T 2705-2010 [10] SN/T 1201-2014 [9] SN/T 1204-2016 [12] | |||||
| R | GCAACCAACCAAGGGTATC | |||||||
| P | TGGCCGCGGTTTGTGATATCGTTAA | |||||||
| T-NOS | 256 | AF485783.1 | 256 | CT-PCR | tNOS_NN_Fwd | GATTAGAGTCCCGCAATTATACATTTAA | 69 | QL-ELE-00-018[32] |
| tNOS D REV | TTATCCTAGKTTGCGCGCTATATTT | |||||||
| HA-nos118-f | GCATGACGTTATTTATGAGATGGG | 118 | SN/T 1197-2016[11] | |||||
| HA-nos118-r | GACACCGCGCGCGATAATTTATCC | |||||||
| NOS-F1 | GAATCCTGTTGCCGGTCTTG | 180 | SN/T 1197-2016[11] | |||||
| NOS-R1 | TTATCCTAGTTTGCGCGCTA | 1782-3-2012[4] | ||||||
| RT-PCR | 180-F | CATGTAATGCATGACGTTATTTATG | 84 | QL-ELE-00-011[33] SN/T 1204-2016[12] | ||||
| 180-R | TTGTTTTCTATCGCGTATTAAATGT | |||||||
| Tm-180 | ATGGGTTTTTATGATTAGAGTCCCGCAA | |||||||
| NOS-F2 | ATCGTTCAAACATTTGGCA | 165 | 1782-3-2012 [4] GB/T 19495.4-2018 [2] GB/T 33526-2017 [3] SN/T 1197-2016 [11] SN/T 2705-2010 [10] | |||||
| NOS-R2 | ATTGCGGGACTCTAATCATA | |||||||
| NOS-P | CATCGCAAGACCGGCAACAGG | |||||||
| T-PINⅡ | 310 | KP784700.1 | 310 | RT-PCR | F | GACTTGTCCATCTTCTGGATTGG | 105 | 未发表 Unpublished |
| R | CACACAACTTTGATGCCCACAT | |||||||
| P | AGTGATTAGCATGTCACTATGTGTGCATCC | |||||||
| HPT | 1026 | AF234296.1 | 474 | CT-PCR | HptF226 | GAAGTGCTTGACATTGGGGAGT | 472 | 1782-2-2012[7] |
| HptR697 | AGATGTTGGCGACCTCGTATT | |||||||
| RT-PCR | qHptF286 | CAGGGTGTCACGTTGCAAGA | 110 | |||||
| qHptR395 | CCGCTCGTCTGGCTAAGATC | |||||||
| qHptFP308 | TGCCTGAAACCGAACTGCCCGCTG | |||||||
| HMG I/Y | 950 | AF127919 | 206 | RT-PCR | HMG I/Y-F | GGTCGTCCTCCTAAGGCGAAAG | 99 | 2031-9-2013 [8] |
| HMG I/Y-R | CTTCTTCGGCGGTCGTCCAC | |||||||
| HMG I/Y-P | CGGAGCCACTCGGTGCCGCAACTT | |||||||
| CT-PCR | hmg-F | TCCTTCCGTTTCCTCGCC | 206 | |||||
| hmg-R | TTCCACGCCCTCTCCGCT | |||||||
| CruA | 3113 | X14555 | 150 | RT-PCR | QCRUAF | GGCCAGGGCTTCCGTGAT | 101 | |
| QCRUAR | CCGTCGTTGTAGAACCATTGG | |||||||
| QCRUAP | AGTCCTTATGTGCTCCACTTTCTGGTGCA | |||||||
| CT-PCR | CruAF398 | GGCCAGGGCTTCCGTGAT | 150 | |||||
| CruAR547 | CTGGTGGCTGGCTAAATCGA |
新窗口打开|下载CSV
2.4 转基因油菜筛查质粒分子的构建
将9个筛查元件和2个油菜内标基因的靶标序列拼接到一起,获得一条长4 433 bp的融合序列(图1)。将融合序列送到上海生工进行全序列人工合成,将人工合成的序列通过EcoRⅤ酶切位点插入pUC18质粒分子中,构建转基因油菜筛查质粒分子pYCSC-1905(图2)。然后将构建的转基因油菜筛查质粒分子pYCSC-1905进行全分子测序,测得的序列与预期序列完全一致。图1
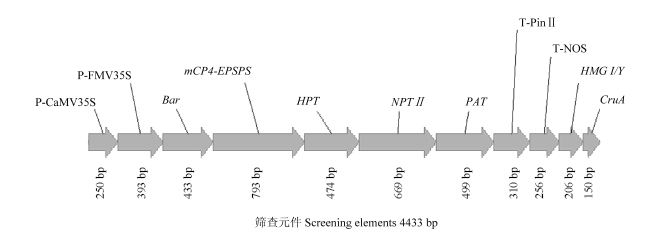 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1筛查元件和油菜内标基因融合序列示意图
Fig. 1Schematic diagram of the fusion sequence of screening elements and the rapeseed reference genes
图2
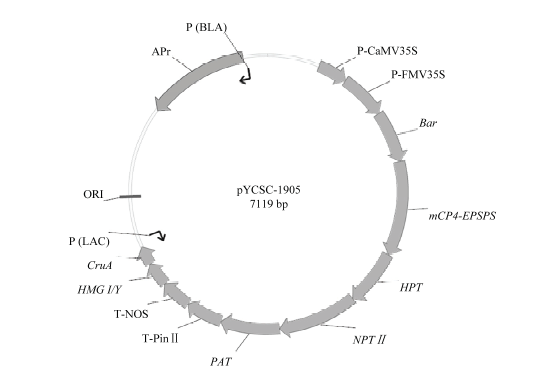 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2转基因油菜筛查质粒分子结构示意图
Fig. 2Schematic diagram of the plasmid molecular structure for transgenic rapeseed screening
合成9个筛查元件和2个油菜内标基因的1个引物/探针组合(电子附表1),进行实时荧光PCR分析。将质粒分子pYCSC-1905梯度稀释,浓度依次为106、105、104、103和102 copies/μL。将梯度稀释的质粒溶液用作标准品,分别进行筛查元件和油菜内标基因的实时荧光PCR扩增。扩增结束后,根据质粒模板拷贝数的对数与Ct值间的对应关系分别绘制各个靶标的标准曲线,计算出标准曲线的斜率,将标准曲线的斜率带入公式E=$10^{\frac{1}{slope}}-1$,计算出以筛查质粒分子作为模板,各个靶标的扩增效率(图3)。9个筛查元件和2个油菜内标基因的扩增效率均在90%—110%,符合标准对实时荧光PCR扩增效率的要求。扩增效率测试结果表明,融合构建到质粒分子pYCSC-1905上的筛查元件和油菜内标基因的核苷酸序列没有干扰引物/探针与模板序列的特异识别,pYCSC-1905适合用作转基因油菜普通PCR和实时荧光PCR筛查检测的质控样品。
图3
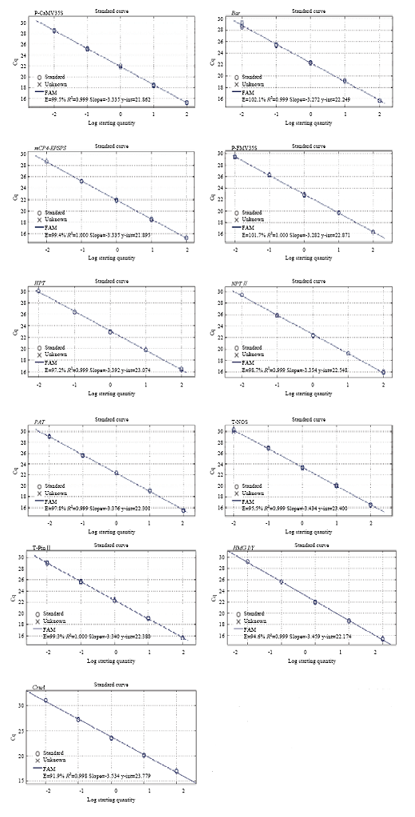 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图39个筛查元件和2个油菜内标基因的标准曲线绘制和扩增效率检测
E表示扩增效率,R2表示标准曲线的决定系数,slope表示标准曲线的斜率,y-int 表示标准曲线的截距
Fig. 3Standard curves and amplification efficiencies of nine screening elements and two rapeseed reference genes
E indicates amplification efficiency, R2 indicates regression coefficient, slope indicates the slope of standard curve, y-int indicates the intercept of standard curve
2.5 转基因油菜筛查质粒分子的应用
将转基因油菜GT73、MS1、RF1、MS1、RF2、MS8、RF3、OXY235、T45、Topas19 /2、MON88302和73496的基因组DNA,作为测试样品,将质粒分子pYCSC-1905设为阳性对照,进行转基因油菜普通PCR和实时荧光PCR筛查检测,引物、探针序列见电子附表1。将普通PCR产物进行凝胶电泳分析,各筛查元件和油菜内标基因在阳性对照和相应的转基因油菜样品中均扩增出预期产物,在不含有相应元件的样品中没有扩增产物(图4)。实时荧光PCR扩增结果表明,各筛查元件和油菜内标基因在阳性对照和相应的转基因油菜样品中均有典型扩增曲线,在不含有相应元件的样品中没有扩增曲线(数据未给出)。普通PCR和实时荧光PCR的筛查检测结果一致,阳性质粒分子pYCSC-1905的每个靶标均获得预期扩增,实际应用结果进一步证实pYCSC-1905适合用作转基因油菜普通PCR和实时荧光PCR筛查检测的质控样品。图4
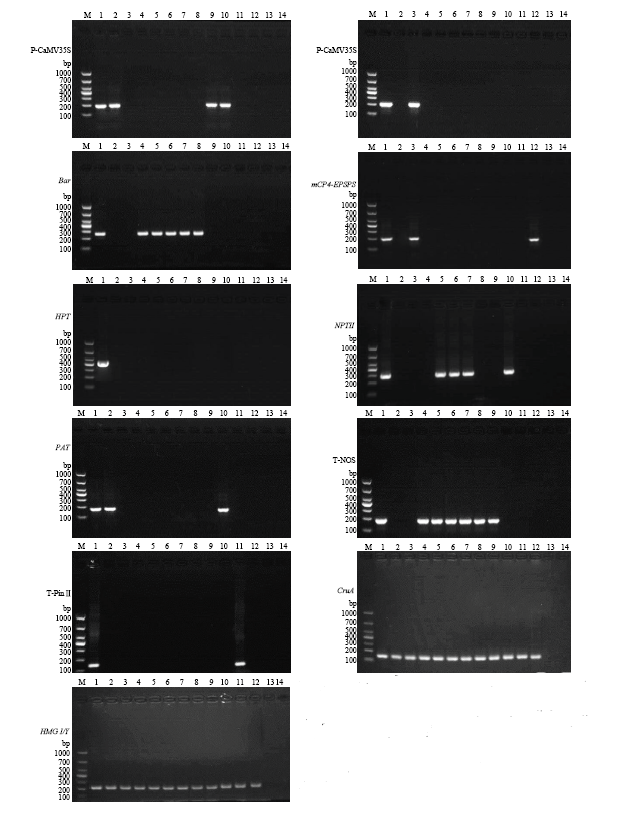 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图412个转基因油菜品种中基因元件的检测
M:Marker;1:p-YCSC-1905;2:T45;3:GT73;4:MS8;5:MS1;6:RF1;7:RF2;8:RF3;9:OXY235;10:Topas 19/2;11:73496;12:MON88302;13:阴性对照中油821;14:空白对照
Fig. 4Screening of gene elements in 12 transgenic rapeseed varieties
M: Marker; 1: p-YCSC-1905; 2: T45; 3: GT73; 4: MS8; 5: MS1; 6: RF1; 7: RF2; 8: RF3; 9: OXY235; 10: Topas 19/2; 11: 73496; 12: MON88302; 13: Negative control ZY821; 14: Blank
3 讨论
转基因生物安全监管一方面要监管批准进口的转基因产品是否按规定流通和使用;另一方面要监管中国未批准进口的转基因产品是否非法进入市场或田间。其中,对非法转基因产品的检测和监测是转基因生物安全监管工作的重中之重。非法(非授权)转基因产品是指未经批准而释放到市场上的转基因产品,包括在别的国家批准而在中国并未获批的转基因材料、田间非法种植的仅批准进口用作加工原料的转基因材料,以及尚处于试验阶段的转基因材料。由于转基因产品存在潜在的环境和食用安全风险,一旦发生非授权转基因产品的非法扩散,将扰乱正常贸易秩序,在消费者中引发恐慌情绪,对国民经济、生活产生一系列的恶劣影响。转基因检测是转基因生物安全监管的技术基础,在转基因检测中尽量降低漏检转基因成分的几率,才能保证转基因生物安全监管的有效性。中国转基因油菜研究活跃,存在很大的非法扩散风险。本研究通过分析转基因油菜中基因元件的使用,制定出了组合检测9个基因元件(P-CaMV 35S、P-FMV 35S、Bar、PAT、CP4-EPSPS、NPTⅡ、HPT、T-NOS、T-PinⅡ)对转基因油菜进行筛查检测的策略,采用本筛查策略可以覆盖已知信息的转基因油菜品种,并且通过筛查HPT,最大程度覆盖了试验阶段的转基因产品。为了给筛查检测提供通用的标准样品,本研究还研制出了转基因油菜筛查质粒分子pYCSC-1905,本质粒分子可为不同的标准方法提供阳性对照,包括农业部公告、GB/T、SN/T和欧盟标准中涉及的普通定性PCR方法和实时荧光PCR方法,具有良好的广适性。组合使用本研究建立的转基因油菜筛查策略和筛查阳性质粒分子pYCSC-1905,不仅可筛查出中国批准进口的转基因油菜,还可筛查出非授权的转基因油菜品种。
4 结论
建立了转基因油菜筛查策略,并研制出与转基因油菜筛查策略配套的阳性质粒分子pYCSC-1905,为转基因油菜筛查和各基因元件标准方法的使用提供了一个通用标准样品。本筛查策略实现了目前已知信息转基因油菜品种的全覆盖,降低了转基因油菜筛查检测中漏检个别品种的几率。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 9]
[本文引用: 9]
[本文引用: 4]
[本文引用: 4]
[本文引用: 9]
[本文引用: 9]
[本文引用: 4]
[本文引用: 4]
[本文引用: 5]
[本文引用: 5]
[本文引用: 6]
[本文引用: 6]
[本文引用: 4]
[本文引用: 4]
[本文引用: 8]
[本文引用: 8]
[本文引用: 9]
[本文引用: 9]
[本文引用: 20]
[本文引用: 20]
[本文引用: 10]
[本文引用: 10]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
URL [本文引用: 2]
URL [本文引用: 2]
URL [本文引用: 2]
URL [本文引用: 2]
URL [本文引用: 2]
URL [本文引用: 2]
URL [本文引用: 2]
URL [本文引用: 1]
URL [本文引用: 2]
URL [本文引用: 2]
URL [本文引用: 2]
URL [本文引用: 2]
URL [本文引用: 2]
