 ,1
,1The QTL Analysis of Single Fruit Weight Associated Traits in Melon Based on CAPS Markers
LIU XiangYu1, ZHANG YuShu1, LIU Liu1, LIU Shi1, GAO Peng1, WANG Di2, WANG XueZheng ,1
,1通讯作者:
收稿日期:2018-10-31接受日期:2019-02-25网络出版日期:2019-05-01
| 基金资助: |
Received:2018-10-31Accepted:2019-02-25Online:2019-05-01
作者简介 About authors
刘相玉,Tel:0451-55191317;E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (2272KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
刘相玉, 张裕舒, 刘柳, 刘识, 高鹏, 王迪, 王学征. 基于CAPS标记的甜瓜单果重相关性状QTL分析[J]. 中国农业科学, 2019, 52(9): 1601-1613 doi:10.3864/j.issn.0578-1752.2019.09.011
LIU XiangYu, ZHANG YuShu, LIU Liu, LIU Shi, GAO Peng, WANG Di, WANG XueZheng.
0 引言
【研究意义】甜瓜(Cucumis melo L.)是葫芦科甜瓜属植物,世界重要的园艺作物之一。主要种植在温带和热带国家,并以亚洲为主要产地。在我国,甜瓜的种植面积和产量均为世界首位,同时我国也是甜瓜的消费大国,因此,对甜瓜展开育种研究具备广大的市场优势与前景。甜瓜的性状中,甜瓜的单果重、果实长度、果实宽度、果实形状及可溶性固形物含量等性状均是影响甜瓜产量及品质的重要因素。本试验在基因组重测序技术广泛应用的背景下,利用二代测序技术构建高覆盖度的CAPS标记,建立高密度遗传图谱,针对甜瓜果实单果重相关的系列性状进行基因定位,为果实单果重相关基因的挖掘奠定基础。【前人研究进展】近些年,国内外****对甜瓜栽培措施、生理生化、遗传规律等方面展开了大量的研究。其中部分结果表明,甜瓜的单果重、果实长度、果实宽度及果肉厚度等性状主要受遗传因素影响[1,2,3,4,5,6]。并且甜瓜果实的相关性状均为数量性状,受多个主效及微效基因控制。遗传图谱是基因定位研究的重要工具,基于不同类型的分子标记,研究者们构建了多张遗传连锁图谱和整合图谱[7,8,9,10],并成功定位到与果实产量、单果重、果实长度、果实宽度及果实形状相关的多个QTL位点。ZALAPA[11]等在探究甜瓜产量相关性状时共检测到包括单株果重、单果平均重在内5个性状的37个QTL位点,试验结果表明甜瓜种质资源中高度分枝类型的基因更具增产潜力。RAMAMURTHY等[12]以蛇形甜瓜品系和厚皮甜瓜品系为亲本,构建了包含200个单株的F2群体,对单果重和果实形态等数量性状进行QTL分析,共检测到31个QTL位点,其中23个位点与前人研究成果一致,新定位到了8个有关单果重、可溶性固形含量等性状的QTL位点,在第8号染色体上检测到2个和单果重有关的位点贡献率分别为20.60%和12.8%,为主效QTL。DIAZ等[13]以栽培甜瓜和野生甜瓜杂交后代为群体,定位到与单果重、果实长度、果实宽度有关的QTL位点各4个,在2号、4号、6号及8号条染色体上位点呈簇出现,且定位区间相同或相近,可能存在一因多效的现象。单果重作为甜瓜产量的重要指标之一,结合分子育种技术探究与单果重有关性状的遗传机理,可以加快育种进程。【本研究切入点】尽管前人已对甜瓜果实性状展开了系统的研究,但一部分性状的研究仍需进一步完善和扩充。在当前对甜瓜单果重QTL分析的报道中,由于传统分子标记的特点,遗传图谱密度较低,并且后代群体性状分离范围较小,因此总体上基因定位精度较低。随着技术的不断更新进步,对甜瓜单果重的遗传机理亟需进一步的研究。【拟解决的关键问题】本研究利用果型与单果重具有显著差异的甜瓜栽培品系M4-130和野生甜瓜品系X207为亲本构建F2:3群体,基于甜瓜全基因组高通量测序数据开发CAPS分子标记构建甜瓜遗传连锁图谱,结合田间表型数据对甜瓜单果重、果实长度、果实宽度、果肉厚度、果形指数及可溶性固形物含量等性状作QTL分析,探究甜瓜单果重相关性状遗传规律,并为定位区间进行初步的功能与代谢通路注释,以期为单果重相关性状的进一步定位和基因克隆提供依据。1 材料与方法
1.1 试验材料
母本材料为M4-130,纯系,果实长椭圆形、果皮厚、果实大(单果重833.0 g)。父本材料为X207,野生品种,纯系,果实圆形、果皮薄、小果型(单果重23.0 g)(图1)。
图1
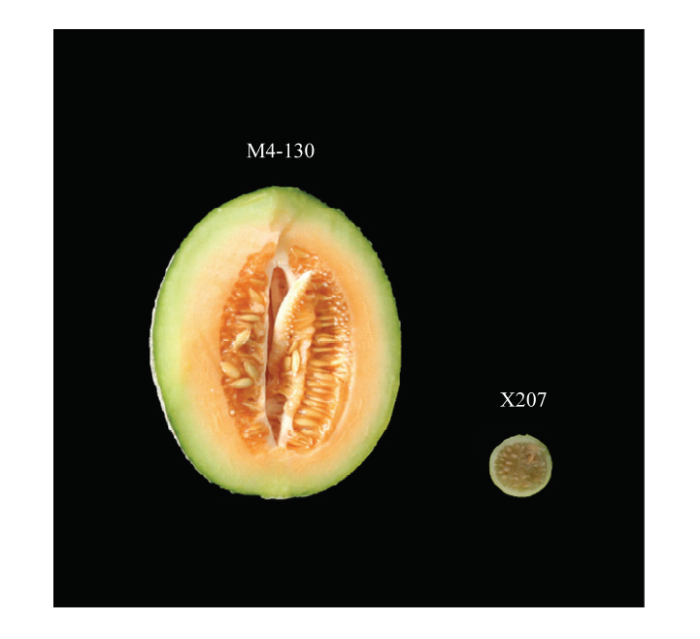 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1M4-130、X207果实纵切图
Fig. 1M4-130 and X207 longitudinal cut of fruit
1.2 田间试验设计
田间试验于2017年3月至2018年9月进行。2017年3—9月,于向阳基地播种P1、P2、F1代各30株,每个小区种10株,3次重复。随机种植F2代群体400株,分株编号自交授粉,获得F3代种子。2018年3—9月播种100个F2:3家系,每个家系种植10株,田间种植株行距35 cm×50 cm,单蔓整枝,第13—15节位留单果,采收果实用以田间表型数据调查及遗传分析。1.3 果实性状的调查方法
根据《甜瓜种质资源描述规范和数据标准》对P1、P2、F1、F2及F2:3代群体果实相关性状进行调查,包括单果重、果实长度、果实宽度、果肉厚度、果型指数、可溶性固形物含量。其中,果实长度、果实宽度、果肉厚度也是决定单果重的重要指标。参照《甜瓜种质资源描述规范和数据标准》[14]对每个果实每个性状测量3次,取平均值作为结果。性状调查方式:单果重:电子天平测量,精确到0.1 g;果实长度:直尺测量,取果实纵径最长处,精确到0.1 cm;果实宽度:直尺测量,取果实横径最长处,精确到0.1 cm;果肉厚度:直尺测量,上、中、下部厚度取平均值,精确到0.1 cm;果型指数:果实长度与果实宽度比值;可溶性固形物含量:手持糖度仪测量。1.4 群体基因组DNA的提取及CAPS分子标记开发
1.4.1 群体基因组DNA的提取 采集P1、P2、F1及F2代生长点附近嫩叶,分别标号,分袋保存于实验室-80℃冰箱,用以基因组DNA的提取。采用改良的CTAB法提取甜瓜基因组DNA[15]。提取后的DNA用1%的琼脂糖凝胶电泳及超微紫外分光光度计SMA3000检验纯度和质量。1.4.2 CAPS分子标记的开发及筛选 以亲本基因组重测序数据为依据,以发布的甜瓜基因组数据为参考,利用东北农业大学西甜瓜实验室自编Perl语言脚本提取两亲本间的SNP位点前后约500 bp的碱基序列,利用SNP2CAPS[16]软件查找双亲存在差异的酶切位点,并在酶切位点上下游100—500 bp设计引物,将SNP位点转化为CAPS标记。
在甜瓜全基因组的12条染色体上均匀选取400个CAPS位点,并用Primer6软件进行引物设计。利用亲本及F1代DNA对已开发的CAPS标记进行PCR扩增和酶切检验。CAPS标记PCR扩增体系:2 μL模板DNA,上、下游引物各1 μL,0.2 μL Taq酶,0.3 μL dNTPs,1 μL Taq Buffer,4.5 μL超纯水。扩增程序采用降落PCR扩增[17]:94℃预变性7 min;94℃变性1 min,60℃退火30 s,72℃延伸1 min,30个循环,每循环降低0.5℃;最后,94℃变性1 min,45℃退火30 s,72℃延伸1 min,72℃终止延伸7 min。
酶切体系为:PCR扩增产物5 μL,0.2 μL限制性内切酶,1 μL限制性内切酶缓冲液,3.8 μL超纯水。酶切反应在水浴锅中进行,具体酶切温度根据不同限制性内切酶最适温度设定。酶切产物用1%琼脂糖凝胶电泳检测。
1.5 遗传连锁图谱的构建及QTL分析
试验以F2代分离群体为遗传连锁图谱的作图群体,以F2:3代性状的家系平均值作为家系性状值进行QTL定位。利用IciMapping V3.3对具有多态性且应用于F2代基因分型的CAPS分子标记构建遗传连锁图谱。用复合区间作图法构建遗传连锁图谱,并对F2:3群体进行QTL分析,以1.0 cM步行速度在全基因组扫描,LOD>2.5为可检测QTL位点存在的阈值。QTL命名方式为性状英文缩写+连锁群+QTL编号。2 结果
2.1 群体性状的表型分析
两亲本在单果重、果实长度、果实宽度、果肉厚度及可溶性固形物含量等性状上都有较大的差异,M4-130在单果重、果实长度、宽度及果肉厚度均大于X207,可溶性固形物小于X207。如表1所示,F1群体单果重、果实长度、宽度、果肉厚度及可溶固形物含量均介于双亲之间,果形指数存在超亲分离现象。F2:3分离群体各家系各性状数值介于双亲之间(图2),各性状数值基本符合正态分布(图3)。相关性分析表明,甜瓜单果重与果实长度、果实宽度、果肉厚度及可溶性固形物均呈显著正相关,与果形指数呈负相关(表2)。Table 1
表1
表1甜瓜单果重性状双亲值及在F2:3群体中的分布
Table 1
| 性状 Trait | 亲本及F1均值 The mean value of and F1 generation | F2:3群体 Distribution in F2:3 population | ||||||
|---|---|---|---|---|---|---|---|---|
| 母本 M4-130 | 父本 X207 | F1 | 均值 Mean | 标准差 SD | 范围 Range | 峰度 SEK | 偏度 SES | |
| 单果重 Fruit weight (g) | 833.0±7.64 | 21.67±2.87 | 226.67±50.58 | 155.92 | 8.76 | 33.00—590.00 | 6.57 | 2.08 |
| 果实长度 Fruit length (cm) | 14.20±0.40 | 3.63±0.23 | 9.83±1.27 | 7.87 | 1.92 | 3.80—13.30 | 0.09 | 0.43 |
| 果实宽度 Fruit width (cm) | 11.40±0.20 | 3.30±0.17 | 6.57±0.31 | 6.01 | 1.42 | 3.00—11.00 | 0.79 | 0.74 |
| 果肉厚度 Fruit thickness (cm) | 3.10±0.10 | 0.33±0.06 | 1.13±0.15 | 1.16 | 0.34 | 0.30—2.30 | 0.58 | 0.41 |
| 果形指数 Fruit shape index (%) | 1.25±0.01 | 1.10±0.12 | 1.49±0.12 | 1.32 | 0.22 | 0.90—2.10 | 0.65 | 0.72 |
| 可溶性固形物含量Soluble solids (%) | 8.17±0.35 | 10.5±0.79 | 10.00±2.20 | 9.46 | 1.81 | 5.90—14.40 | -0.55 | 0.30 |
新窗口打开|下载CSV
图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2亲本、F1及F2:3群体果实纵切图
Fig. 2Parent, F1 and F2:3 group fruit longitudinal cut map
图3
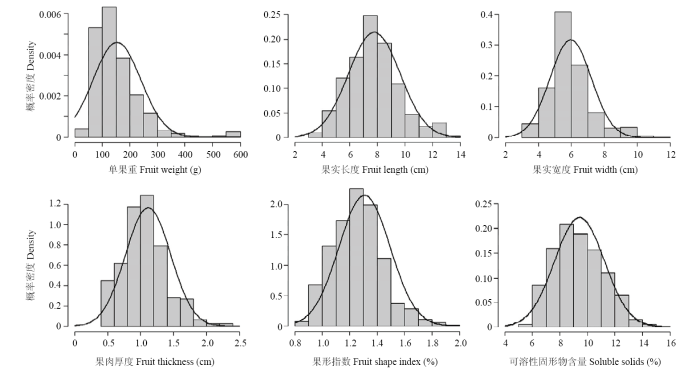 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3F2:3群体相关性状频次直方图
Fig. 3Histogram for the fruit traits of F2:3 population
Table 2
表2
表2甜瓜果实各性状相关性分析
Table 2
| 性状 Trait | 单果重 Fruit weight | 果实长度 Fruit length | 果实宽度 Fruit width | 果肉厚度 Fruit thickness | 果形指数 Fruit shape | 可溶性固形物含量 Soluble solids |
|---|---|---|---|---|---|---|
| 单果重 Fruit weight | 1 | |||||
| 果实长度 Fruit length | 0.873** | 1 | ||||
| 果实宽度 Fruit width | 0.891** | 0.832** | 1 | |||
| 果肉厚度 Fruit thickness | 0.866** | 0.813** | 0.854** | 1 | ||
| 果形指数 Fruit shape | -0.018 | 0.284** | -0.278** | -0.065 | 1 | |
| 可溶性固形物含量 Soluble solids | 0.745** | 0.706** | 0.685** | 0.648** | 0.034 | 1 |
新窗口打开|下载CSV
2.2 遗传连锁图谱的构建
2.2.1 甜瓜基因组DNA提取 利用改良的CTAB法提取甜瓜基因组DNA,1%琼脂糖凝胶电泳检测,图4为电泳检测结果。父母本、F1及F2代分离群体DNA浓度及纯度均可用于分子标记的检测,无蛋白质和RNA杂质,对分子标记检测不存在影响。图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4甜瓜基因组DNA
Fig. 4DNA electrophoregram of muskmelon
2.2.2 CAPS标记的筛选与验证 基于亲本的测序结果,利用Primer6在甜瓜12条染色体上均匀设计400对CAPS引物。利用提取的亲本及F1代DNA,对设计的400对CAPS引物作PCR和酶切反应,验证引物的多态性。图5为部分引物经PCR和酶切反应后产物的电泳条带。验证结果显示,400对CAPS引物中具有多态性的为185对,多态率为46.25%。引物命名为所在染色体号+引物编号。
图5
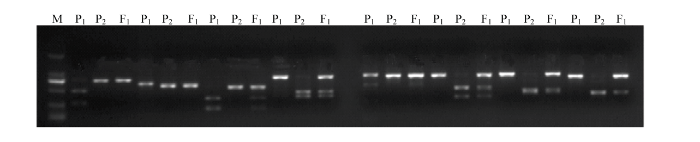 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5CAPS引物在亲本及F1多态性表现
M:标记条带;P1:父本条带;P2:母本条带;F1:F1代条带
Fig. 5Polymorphism of CAPS markers among parental materials and F1 generation
M: The band of marker; P1: Band of female parent; P2: Band of male parent; F1: Band of F1 generation
2.2.3 甜瓜遗传连锁图谱的构建 利用QTL IciMapping V3.3软件对筛选出具有多态性的CAPS标记作遗传图谱,排除条带不清晰及偏分离标记,用于作图的标记共185对,如图5所示,图谱包含12个连锁群,覆盖基因组长度为1 600.45 cM,标记间平均距离为8.65 cM,连锁群长度介于91.53—166.20 cM。该图谱最长连锁群为7号连锁群,长度为166.20 cM,包含15个CAPS标记,最短连锁群为1号连锁群,长度为91.53 cM,包含13个CAPS标记,包含最多标记的连锁群为10号连锁群,共20个标记(表3和图6)。
Table 3
表3
表3甜瓜遗传连锁图谱基本参数
Table 3
| 染色体 Chr. | 标记数目 No. of Marker | 长度 Genetic distance of linkage groups (cM) | 标记间平均距离 Average distance of linkage groups (cM) |
|---|---|---|---|
| 1 | 13 | 91.53 | 7.04 |
| 2 | 13 | 112.55 | 8.66 |
| 3 | 19 | 121.62 | 6.40 |
| 4 | 17 | 140.25 | 8.25 |
| 5 | 15 | 108.46 | 7.23 |
| 6 | 15 | 160.37 | 10.69 |
| 7 | 15 | 166.20 | 11.08 |
| 8 | 15 | 144.30 | 9.62 |
| 9 | 14 | 155.56 | 11.11 |
| 10 | 20 | 107.28 | 5.36 |
| 11 | 14 | 163.11 | 11.65 |
| 12 | 15 | 129.22 | 8.61 |
| 总计Total | 185 | 1600.45 | 8.65 |
新窗口打开|下载CSV
图6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6甜瓜遗传连锁图谱构建与甜瓜单果重相关性状QTL分析
Fig. 6QTL analysis for melon fruit weight traits on genetic linkage map
2.3 单果重相关性状的QTL分析
2.3.1 单果重QTL分析 如表4所示,共检测到7个QTL位点,分别为于FW3.1、FW4.1、FW5.1、FW6.1、FW8.1、FW8.2及FW11.1总贡献率为87.2730%,LOD值介于2.9070—16.8746,在检测到的7个QTL中,有4个QTL的贡献率大于12%。其中FW8.1的贡献率最大,为25.8774%,LOD值为16.8746,为主效QTL,位于标记8-2883和8-5972之间,距标记8-5972遗传距离为3.93 cM,距标记8-2883遗传距离为2.46 cM。FW3.1的贡献率最小,为2.7054%,LOD值为3.0173,定位标记在3-6670和3-0640之间,距标记3-6670遗传距离为5.50 cM,距标记3-0640遗传距离为5.50 cM。Table 4
表4
表4甜瓜果实相关性状QTL分析及其遗传效力
Table 4
| 性状 Trait | 位点 QTL | 相邻标记 Adjacent markers | 图谱位置 Position | 标记间距 Marker range | LOD | 贡献率(%) R2 | 加性效应 Additive | 显性效应 Dominance |
|---|---|---|---|---|---|---|---|---|
| 单果重 Fruit weight | FW3.1 | 3-6670—3-0640 | 66 | 65.23—74.69 | 3.0173 | 2.7054 | 39.2379 | 32.2348 |
| FW4.1 | 4-9382—4-1311 | 31 | 27.25—31.68 | 3.2739 | 12.3435 | 39.6980 | -14.1269 | |
| FW5.1 | 5-9232—5-5773 | 108 | 105.17—108.46 | 2.9070 | 9.3215 | -31.2002 | 23.5592 | |
| FW6.1 | 6-1444—6-6615 | 160 | 106.62—160.37 | 3.9121 | 14.4755 | 40.7147 | -20.9489 | |
| FW8.1 | 8-2883—8-5972 | 101 | 98.60—104.93 | 16.8746 | 25.8774 | -1.2134 | 184.5905 | |
| FW8.2 | 8-0306—8-2169 | 71 | 65.29—72.09 | 3.3297 | 3.6869 | 3.6869 | 9.8677 | |
| FW11.1 | 11-3692—11-1432 | 48 | 41.93—56.06 | 4.6085 | 18.8628 | 48.3023 | -18.9123 | |
| 果实长度 Fruit length | FL2.1 | 2-2347—2-1803 | 104 | 100.56—109.19 | 6.7436 | 17.0180 | -1.3111 | 0.2646 |
| FL3.1 | 3-6670—3-0640 | 66 | 65.23—74.69 | 3.0160 | 15.1015 | 1.1146 | 0.9123 | |
| FL4.1 | 4-9382—4-1311 | 30 | 27.25—31.68 | 4.2371 | 9.9603 | 0.9998 | -0.2666 | |
| FL5.1 | 5-9232—5-5773 | 107 | 105.17—108.46 | 2.6505 | 5.1442 | -0.6258 | 0.5634 | |
| FL6.1 | 6-1444—6-6615 | 148 | 106.62—160.37 | 3.8379 | 14.1998 | 1.1898 | -0.0833 | |
| FL8.1 | 8-8589—8-8413 | 14 | 00.00—14.22 | 2.8085 | 19.9334 | 1.3147 | -0.5710 | |
| FL11.1 | 11-8315—11-3692 | 35 | 30.15—41.93 | 6.1904 | 15.7421 | 1.2632 | -0.0376 | |
| 果实宽度 Fruit width | FWID3.1 | 3-1192—3-0090 | 34 | 18.50—46.50 | 3.0311 | 11.3927 | 0.6226 | 0.0665 |
| FWID4.1 | 4-1311—4-2962 | 34 | 31.68—42.76 | 3.2242 | 10.1598 | 0.7384 | 0.1435 | |
| FWID8.1 | 8-0306—8-2169 | 71 | 65.29—72.09 | 5.3190 | 18.4669 | 0.7200 | -0.3225 | |
| FWID10.1 | 10-3838—10-0417 | 1 | 0.00—9.81 | 4.0028 | 13.2077 | 0.6771 | 0.0365 | |
| FWID11.1 | 11-8315—11-3692 | 38 | 30.15—41.93 | 4.5617 | 15.8568 | 0.9479 | -0.0865 | |
| 果肉厚度 Fruit thickness | FT6.1 | 6-1444—6-6615 | 151 | 106.62—160.37 | 2.8921 | 12.0802 | 0.1414 | -0.2006 |
| FT11.1 | 11-8315—11-3692 | 35 | 30.15—41.93 | 4.3730 | 15.2309 | 0.2292 | -0.0031 | |
| 果型指数 Fruit shape index | FS2.1 | 2-1803—2-2808 | 112 | 109.19—112.55 | 4.6355 | 22.0413 | -0.1131 | 0.0923 |
| 可溶性固形物 Soluble solids | SS6.1 | 6-1444—6-6615 | 146 | 106.62—160.37 | 2.9412 | 16.4299 | 1.2740 | 0.2516 |
| SS12.1 | 12-5356—12-4957 | 9 | 0.00—19.61 | 3.3407 | 10.0947 | 0.1159 | -1.4154 |
新窗口打开|下载CSV
2.3.2 果实长度QTL分析 如表4所示,共检测到与果实长度相关的7个QTL位点,分别为FL2.1、FL3.1、FL4.1、FL5.1、FL6.1、FL8.1及FL11.1总贡献率为97.0993%,LOD值介于2.6505—19.9334之间,其中FL3.1与单果重FW3.1,FL4.1与单果重FW4.1,FL5.1与单果重FW5.1,FL6.1与单果重FW6.1定位位置一致,由表2可知,果实长度与单果重极显著相关,可能与基因多因一效或一因多效遗传有关[18]。检测到7个QTL贡献率大于5%,其中FL8.1贡献率最高,为19.9334%,定位标记在8-8589和8-8413之间,位点偏向标记8-8413,遗传距离为0.22 cM。FL5.1贡献率最小,为5.1442%,位于标记5-9232和5-5773之间,标记间距离为3.29 cM。
2.3.3 果实宽度QTL分析 检测到与果实宽度相关的QTL位点共5个(表4),分别为FWID3.1、FWID4.1、FWID8.1、FWID10.1及FWID11.1,贡献率均超过10%,其中FWID8.1贡献率最大,为18.4669%,定位在标记8-0306和8-2169之间,距两侧标记遗传距离分别为5.71 cM和1.09 cM,此定位位置与FW8.2位置一致。
2.3.4 果肉厚度QTL分析 检测到与果肉厚度相关QTL位点2个(表4),分别为FT6.1、FT11.1,两位点贡献率均大于10%。FT6.1的贡献率为12.0802%,位于6-1444和6-6615标记之间,位置偏向6-6615侧。FT11.1贡献率为15.2309%,位于11-8315和11-3692标记之间。
2.3.5 果形指数QTL分析 关于果型指数,只检测到1个QTL,在2-1803和2-2808之间,与两侧标记间距分别为2.18 cM和0.55 cM(表4)。
2.3.6 可溶性性固形物含量QTL分析 如表4所示,检测到与可溶性固形物含量相关的到2个QTL位点,分别为SS6.1和SS12.1。SS6.1贡献率为16.4299%,位于标记6-1444和6-6615之间,SS12.1贡献率为10.0947%,位于标记12-5356和12-4957之间。
2.4 基因功能注释及通路注释
在本试验中,性状定位的重叠区间共有5个,由于果实长度、果实宽度、果肉厚度均是衡量单果重的重要指标,性状间具有极显著的相关性,因此,定位区间的重叠与果实性状间极高的相关性呈现了本试验数据的双向验证。其中,有3个定位区间物理距离较小,因此选取4号染色体单果重FW2.1和果实长度FL2.1定位区间,物理距离为298 kb,5号染色体FW5.1和FL5.1定位区间,物理距离为169 kb,8号染色体FW8.2和FWID8.1定位区间1.3 Mb,对区间内的223个基因功能注释及代谢通路注释。表5为区段内223个基因的GO(Gene Ontology)二级功能注释,在GO的3大类别中,代谢过程(Metabolic process)、细胞(Cell)及细胞构成(Cell part)、联结(Binding)与催化活性(Catalytic activity)基因数量占比较高。Table 5
表5
表5223个基因功能注释
Table 5
| GO一级类别 GO Level I Category | GO数据库二级分类编号 GO Level II Category | GO分类注释 Annotation of II level GO term | 基因数量 Gene number |
|---|---|---|---|
| 生物学过程 Biological process | GO: 0008152 | 代谢过程 Metabolic process | 91 |
| GO: 0009987 | 细胞过程 Cellular process | 83 | |
| GO: 0044699 | 单体进程 Single-organism process | 65 | |
| GO: 0065007 | 生物调控 Biological regulation | 31 | |
| GO: 0050896 | 应激反应 Response to stimulus | 20 | |
| GO: 0071840 | 细胞成分组织与合成 Cellular component organization or biogenesis | 19 | |
| GO: 0051179 | 定位 Localization | 16 | |
| GO: 0032502 | 发展过程 Developmental process | 11 | |
| GO: 0032501 | 多细胞生物过程 Multicellular organismal process | 9 | |
| GO: 0000003 | 生殖过程 Reproduction | 6 | |
| GO: 0022414 | 再生过程 Reproductive process | 6 | |
| GO: 0023052 | 信号 Signaling | 4 | |
| GO: 0040007 | 发育过程 Growth | 3 | |
| GO: 0051704 | 多生物过程 Multi-organism process | 3 | |
| GO: 0022610 | 生物粘附 Biological adhesion | 1 | |
| GO: 0098754 | 去毒过程 Detoxification | 1 | |
| 分子功能 Molecular function | GO: 0005488 | 联结 Binding | 95 |
| GO: 0003824 | 催化活性 Catalytic activity | 81 | |
| GO: 0005215 | 转运活性 Transporter activity | 8 | |
| GO: 0001071 | 核酸结合转录因子活性 Nucleic acid binding transcription factor activity | 4 | |
| GO: 0098772 | 分子功能调控 Molecular function regulator | 4 | |
| GO: 0005198 | 结构分子活性 Structural molecule activity | 3 | |
| GO: 0016209 | 抗氧化活性 Antioxidant activity | 3 | |
| GO: 0000988 | 蛋白质结合转录活性 Transcription factor activity, protein binding | 1 | |
| GO: 0004871 | 信号传感器活性 Signal transducer activity | 1 | |
| GO: 0060089 | 分子转导活性 Molecular transducer activity | 1 | |
| 细胞组分 (Cellular Component) | GO: 0005623 | 细胞 Cell | 73 |
| GO: 0044464 | 细胞组分 Cell part | 73 | |
| GO: 0043226 | 细胞器 Organelle | 49 | |
| GO: 0016020 | 细胞膜 Membrane | 30 | |
| GO: 0032991 | 大分子复合物 Macromolecular complex | 26 | |
| GO: 0044425 | 细胞膜组分 Membrane part | 20 | |
| GO: 0044422 | 细胞器组分 Organelle part | 15 | |
| GO: 0005576 | 细胞外区 Extracellular region | 4 | |
| GO: 0030054 | 细胞连接 Cell junction | 4 | |
| GO: 0055044 | 共质体 Symplast | 4 | |
| GO: 0031974 | 膜封闭的管腔 Membrane-enclosed lumen | 3 | |
| GO: 0019012 | 病毒 Virion | 2 | |
| GO: 0044423 | 病毒组分 Virion part | 2 |
新窗口打开|下载CSV
对223个基因进行代谢通路注释并进行富集分析,将显著性0.05水平的富集通路整理成表。表6所示,几丁质降解通路II(chitin degradation II)显著性最高,且基因数量最多。其次还有胆碱生物合成(Choline biosynthesis III)等。几丁质酶是几丁质降解的主要成分酶,几丁质酶可以促进植物的营养生长,同时参与植物生长发育的调控,在细胞分裂、分化及在植物生长发育的信号分子中产生作用。
Table 6
表6
表6显著富集的代谢通路
Table 6
| 通路名称 Pathway | 通路注释 Description of pathway | 基因数目 Gene number | P值 P-value | 基因 Gene |
|---|---|---|---|---|
| PWY-6902 | 几丁质降解II Chitin degradation II | 6 | 3.70E-08 | MELO3C007961, MELO3C007962, MELO3C007963, MELO3C007964, MELO3C007965, MELO3C007966 |
| PWY-3561 | 胆碱生物合成III Choline biosynthesis III | 2 | 0.0112 | MELO3C007944, MELO3C007945 |
| ASPARAGINE-DEG1-PWY | L-天冬胺酰降解I L-asparagine degradation I | 1 | 0.0502 | MELO3C008013 |
| GLYSYN-ALA-PWY | 甘氨酸生物合成III Glycine biosynthesis III | 1 | 0.0403 | MELO3C014568 |
| PWY-6013 | 克螨酯生物合成Crepenynate biosynthesis | 1 | 0.0204 | MELO3C009630 |
| PWY-6515 | 根皮苷生物合成 Phloridzin biosynthesis | 1 | 0.0502 | MELO3C007921 |
| PWY-7039 | 磷脂代谢 Phosphatidate metabolism | 2 | 0.0466 | MELO3C007944, MELO3C007945 |
新窗口打开|下载CSV
3 讨论
3.1 试验材料选择
在试验材料选择方面,本试验利用野生种质资源作为亲本之一。由于野生种质资源具有丰富的多样性,其性状与栽培品种差异大,因此可开发性状数量多且后代群体性状分布范围广。本试验选取野生甜瓜品系X207作为亲本之一,其具有与SEBASTIAN[19]和MICHEL[20]的研究中野生品种记录相一致的特征,主要性状表现为小叶片和小花,茎蔓细小、高度分枝,生长势强,花为雌雄同株,果实卵圆形或圆形,果实小(20.0—50.0 g),种子小,果肉薄且无法食用等。野生品种X207具备的性状特征与栽培品种之间具有极大的遗传背景差异,在单果重等数量性状基因定位方面具有较大的优势。3.2 SNP标记挖掘及转化CAPS标记
随着不断研究,通过综合SNP标记和CAPS标记的优点,形成了一种以SNP位点变异为基础的CAPS标记,利用生物信息学手段从植物基因组重测序数据中分析出大量的SNP位点、酶切位点及相应的限制性内切酶,再利用CAPS手段识别DNA序列的多态性。本研究是基于全基因组重测序数据开发的CAPS标记,多态率为46.25%,因此更加具有针对性。基于双亲重测序数据所开发的CAPS标记更加准确、有效,在各作物间的多态性为39.50%—56.83%[21]。束永俊等[17]基于大豆基因组重测序数据共设计CAPS标记139个,其中79个CAPS标记具有多态性,多态率为56.83%。周慧文等[22]基于西瓜重测序数据及已公布的西瓜全基因组数据,共设计了420个CAPS标记,其中247个CAPS具有多态性,多态率为58.80%。由于CAPS标记可以通过简单的PCR、酶切和琼脂糖凝胶电泳完成,所以将SNP标记转化为CAPS标记,能够提升SNP标记在甜瓜分子育种中的应用进程。明确SNP位点在甜瓜基因组上的分布情况可以更加高效精确的进行甜瓜遗传图谱的构建及基因精细定位。3.3 分子标记开发及图谱构建
本研究利用基于亲本材料全基因组重测序数据开发了185个CAPS标记,构建了一张遗传距离全长为1 600.45 cM,标记间平均距离为8.65 cM的高密度遗传图谱。6号染色体上6-1444和6-6615两标记之间物理距离较远,原因可能是标记空缺区段或测序结果染色体部分缺失,尚需进一步增加筛选密度,通过新标记或其他标记及方法完善构建图谱。本研究中开发的CAPS标记基因组座位已知,可以明确各标记间的物理距离,便于日后遗传图谱的加密及基因精细定位等研究工作。与其他标记相比,CAPS标记具有以下优势:(1)CAPS标记来自亲本间广泛分布的SNP位点,具有分布广、分型准、多态率高等优势。本试验中CAPS引物多态性达46.25%;(2)用1%琼脂糖凝胶电泳对CAPS标记进行检测,操作方便且易于观察;(3)在测序的基因组中有庞大的单碱基突变位点为构建高饱和遗传连锁图谱提供更大可能性;(4)所构建遗传连锁图谱与实际染色体相对应,基因定位更有意义。
3.4 单果重相关性状相关性分析及QTL定位
本试验组合栽培品种与野生品种作为亲本材料,单果重、果实长度、果实宽度及果肉厚度等性状均差异显著。通过F2:3群体相关性分析结果显示,单果重与果实长度、果实宽度、果肉厚度及可溶性固形物含量均呈显著相关,说明单果重受果实长度、果实宽度、果肉厚度及可溶性固形物含量影响较大,甜瓜单果重随着果实长度、宽度及果肉厚度的增加而增加,在定位分析中,这些性状大多数在相同的连锁群上,定位区间相同或相近,这种现象不仅存在于甜瓜中,也存在于其他植物中[23,24,25,26,27,28,29,30]。栾非时等[31]利用美国厚皮甜瓜品系和中国薄皮甜瓜品系构建F2群体,在8号染色体上定位到与1个与单果重有关的QTL位点,贡献率为78.24%。RAMAMURTHY等[12]利用蛇瓜和厚皮甜瓜构建的F2群体进行QTL定位,发现在8号染色体存在2个与单果重相关QTL位点,贡献率分别为20.60%和12.80%,与本试验在8号染色体上检测到的2个QTL位点一致,其中FW8.1的贡献率为25.88%,表明控制甜瓜单果重的主效基因位于8号染色体上。WANG等[32]用不同基因型亲本构建两个F2群体遗传连锁图谱,检测到4个与果实长度有关位点,分别位于2、7、7、8号染色体,贡献率均低于15%,属于微效QTL位点,检测到2个与果实宽度有关的QTL位点,定位到第5、11号染色体上,贡献率均小于15%,属于微效QTL位点,果实长度与本试验2、8染色体结果一致,果实宽度与本试验定位的11号染色体位置相同。栾非时[33]等利用厚皮甜瓜和薄皮甜瓜构建的F2群体进行QTL分析,定位到3个与单果重有关的微效位点,与本试验中5、6、11号染色体定位结果一致。定位到5个与果实长度有关的QTL位点,其中3、5、6、11号染色体的定位结果与本试验结果一致,甜瓜的单果重、果实长度、果实宽度等性状都为数量性状受多基因调控,且受群体类型和环境影响较大。DIAZ[13]等以栽培甜瓜和野生甜瓜杂交后代为群体,单果重、果实长度、果实宽度均定位在2号、4号、6号及8号染色体上,果实厚度相关QTL位点在4号和8号染色体上,果形指数在2号、4号及6号染色体上,在这四条染色体上位点呈簇出现,且定位区间在相同或相邻,而本试验中在3号、4号、6号、8号及11号染色体上均有位点成簇出现,可能存在一因多效的现象。****DIAZ试验材料与本试验所用材料类型相似,均使用野生品种作为亲本之一,其4、6、8号染色体上定位结果与本试验定位结果一致,少数染色体定位结果不一致,可能由于受到栽培环境的影响。
3.5 区间内基因注释
由于单果重性状是多基因控制的数量性状,其差异受到多个基因的直接或间接影响,影响因素十分复杂。并且在当前的基因注释数据库(GO、KEGG)中,区间内基因的注释也较有限,因此难以确定某个基因是否与性状相关,当前只能初步得到区间内基因的功能与所参与通路的注释概况。基因的挖掘仍需要进一步的探索。4 结论
本研究构建了包含185个CAPS标记的遗传图谱,共检测到24个QTL位点,明确了其在连锁群上分布情况及位点遗传效应。检测到与单果重相关位点7个,其中FW8.1的贡献率高达25.88%,LOD值为16.88,为主效QTL;与果实长度相关位点7个,总贡献率为97.10%;与果实宽度相关位点5个,与果肉厚度相关位点2个,与果形指数相关位点1个,与可溶性固形物含量相关位点1个。通过遗传分析和相关性研究,发现单果重与果实长度、果实宽度及果肉厚度极显著相关,定位位点集中在3、4、5、6、8、11号染色体上,且定位区间相同或相近。研究结果为进一步探究甜瓜单果重的遗传机理和主效基因的精细定位及克隆奠定了理论基础。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
DOI:10.1590/S1806-66902011000300011URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1093/nar/gnh006URL [本文引用: 1]
DOI:10.3724/SP.J.1006.2010.00574Magsci [本文引用: 2]

<p><span >采用生物信息学方法将大豆</span><span lang="EN-US" >EST</span><span >序列联配到大豆基因组序列上,挖掘到大豆</span><span lang="EN-US" >EST-SNP</span><span >位点</span><span lang="EN-US" >537</span><span >个。对其靶向基因进行功能注释分析,发现它们主要参与亚细胞定位、蛋白质结合与催化以及代谢等与大豆重要农艺性状形成相关的生物过程。同时开发了简便易行的</span><span lang="EN-US" >SNP</span><span >检测方法,利用</span><span lang="EN-US" >EMBOSS</span><span >软件筛选导致酶切位点改变的</span><span lang="EN-US" >EST-SNP</span><span >,分别以大豆绥农</span><span lang="EN-US" >14</span><span >、合丰</span><span lang="EN-US" >25</span><span >、</span><span lang="EN-US" >Acher</span><span >、</span><span lang="EN-US" >Evans</span><span >、</span><span lang="EN-US" >Peking</span><span >、</span><span lang="EN-US" >PI209332</span><span >、固新野生大豆、科丰</span><span lang="EN-US" >1</span><span >号、南农</span><span lang="EN-US" >1138-2</span><span >的</span><span lang="EN-US" >DNA</span><span >及其混合的</span><span lang="EN-US" >DNA</span><span >为模板,设计引物进行</span><span lang="EN-US" >PCR</span><span >扩增,发现</span><span lang="EN-US" >44</span><span >个</span><span lang="EN-US" >PCR</span><span >产物中有</span><span lang="EN-US" >36</span><span >个测序峰图在预期的</span><span lang="EN-US" >EST-SNP</span><span >位点表现出多态性。酶切分析发现</span><span lang="EN-US" >26</span><span >个</span><span lang="EN-US" >PCR</span><span >产物具有酶切多态性,可以作为</span><span lang="EN-US" >CAPS</span><span >标记。结果表明该</span><span lang="EN-US" >EST-SNP</span><span >挖掘体系及其</span><span lang="EN-US" >CAPS</span><span >标记转化系统具有高效率、低成本等优点,有利于促进大豆的遗传育种研究。</span></p>
DOI:10.3724/SP.J.1006.2010.00574Magsci [本文引用: 2]

<p><span >采用生物信息学方法将大豆</span><span lang="EN-US" >EST</span><span >序列联配到大豆基因组序列上,挖掘到大豆</span><span lang="EN-US" >EST-SNP</span><span >位点</span><span lang="EN-US" >537</span><span >个。对其靶向基因进行功能注释分析,发现它们主要参与亚细胞定位、蛋白质结合与催化以及代谢等与大豆重要农艺性状形成相关的生物过程。同时开发了简便易行的</span><span lang="EN-US" >SNP</span><span >检测方法,利用</span><span lang="EN-US" >EMBOSS</span><span >软件筛选导致酶切位点改变的</span><span lang="EN-US" >EST-SNP</span><span >,分别以大豆绥农</span><span lang="EN-US" >14</span><span >、合丰</span><span lang="EN-US" >25</span><span >、</span><span lang="EN-US" >Acher</span><span >、</span><span lang="EN-US" >Evans</span><span >、</span><span lang="EN-US" >Peking</span><span >、</span><span lang="EN-US" >PI209332</span><span >、固新野生大豆、科丰</span><span lang="EN-US" >1</span><span >号、南农</span><span lang="EN-US" >1138-2</span><span >的</span><span lang="EN-US" >DNA</span><span >及其混合的</span><span lang="EN-US" >DNA</span><span >为模板,设计引物进行</span><span lang="EN-US" >PCR</span><span >扩增,发现</span><span lang="EN-US" >44</span><span >个</span><span lang="EN-US" >PCR</span><span >产物中有</span><span lang="EN-US" >36</span><span >个测序峰图在预期的</span><span lang="EN-US" >EST-SNP</span><span >位点表现出多态性。酶切分析发现</span><span lang="EN-US" >26</span><span >个</span><span lang="EN-US" >PCR</span><span >产物具有酶切多态性,可以作为</span><span lang="EN-US" >CAPS</span><span >标记。结果表明该</span><span lang="EN-US" >EST-SNP</span><span >挖掘体系及其</span><span lang="EN-US" >CAPS</span><span >标记转化系统具有高效率、低成本等优点,有利于促进大豆的遗传育种研究。</span></p>
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1016/j.pbi.2008.12.009URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
.
[本文引用: 1]
Magsci [本文引用: 1]

以圆茄高代自交系106和长茄高代自交系113为亲本,利用其F<sub>2</sub>群体构建了一张包括23个SSR标记和85个AFLP标记,共15个连锁群的复合遗传图谱。该图谱覆盖基因组长度1 007.9 cM,平均图距9.3 cM,长度和密度符合定位标准。采用MapQTL4.0软件并结合MQM作图法对果形QTL进行定位分析,定位到与果形指数相关的两个QTL,位于第1和第12连锁群上,表型贡献率分别为20.8%和41.5%;与果长相关的5个QTL,位于第1、8、11、12、14连锁群上,表型贡献率分别为16.5%、36.8%、9.8%、45.0%和41.9%;与果径相关的两个QTL,位于第1和第5连锁群上,表型贡献率分别为16.2%和15.8%。果形指数、果长和果径QTL同时定位在第1连锁群上,且与AFLP标记M23E21B距离3.5 cM,表明果形性状与该标记紧密连锁。
Magsci [本文引用: 1]

以圆茄高代自交系106和长茄高代自交系113为亲本,利用其F<sub>2</sub>群体构建了一张包括23个SSR标记和85个AFLP标记,共15个连锁群的复合遗传图谱。该图谱覆盖基因组长度1 007.9 cM,平均图距9.3 cM,长度和密度符合定位标准。采用MapQTL4.0软件并结合MQM作图法对果形QTL进行定位分析,定位到与果形指数相关的两个QTL,位于第1和第12连锁群上,表型贡献率分别为20.8%和41.5%;与果长相关的5个QTL,位于第1、8、11、12、14连锁群上,表型贡献率分别为16.5%、36.8%、9.8%、45.0%和41.9%;与果径相关的两个QTL,位于第1和第5连锁群上,表型贡献率分别为16.2%和15.8%。果形指数、果长和果径QTL同时定位在第1连锁群上,且与AFLP标记M23E21B距离3.5 cM,表明果形性状与该标记紧密连锁。
[本文引用: 1]
[本文引用: 1]
DOI:10.3864/j.issn.0578-1752.2011.17.003Magsci [本文引用: 1]

目的】鉴定玉米产量相关性状基因位点及包含有利等位基因的导入系,为了解产量性状形成的遗传基础及针对玉米自交系产量性状的分子设计提供参考和依据。【方法】以QB80和Qi319为供体亲本,掖478为轮回亲本,采用回交结合定向选择,分别构建含有61和72个家系的基础导入系群体。通过2年4点田间试验,利用完备复合区间作图进行产量及其相关性状的QTL(quantitative trait locus,QTL)分析。【结果】4个环境下,在QB80为供体的导入系群体中,共检测到9个性状的49个QTL;在Qi319为供体的导入系群体中,检测到9个性状的42个QTL。在2个及以上环境中均检测到的QTL有16个。同一性状在不同环境下所检测的QTL定位在相同的染色体区域,不同性状的QTL也定位在相同或临近的染色体区域,形成多个QTL富集区。2个群体所检测的QTL位点具有较少的一致性,说明2个供体材料中含有不同的有利基因位点。同时,导入片段中含有利基因的导入系,其相关性状明显得以改良,这些导入系可用于QTL聚合以改良掖478的产量相关性状。【结论】QB80较Qi319与掖478间的遗传差异更大,能检测更多的产量性状QTL;2个导入系群体中含有优良等位基因的导入系可用于QTL聚合改良掖478;QTL富集区是为产量性状基因的克隆提供可供参考的重要染色体区域。
DOI:10.3864/j.issn.0578-1752.2011.17.003Magsci [本文引用: 1]

目的】鉴定玉米产量相关性状基因位点及包含有利等位基因的导入系,为了解产量性状形成的遗传基础及针对玉米自交系产量性状的分子设计提供参考和依据。【方法】以QB80和Qi319为供体亲本,掖478为轮回亲本,采用回交结合定向选择,分别构建含有61和72个家系的基础导入系群体。通过2年4点田间试验,利用完备复合区间作图进行产量及其相关性状的QTL(quantitative trait locus,QTL)分析。【结果】4个环境下,在QB80为供体的导入系群体中,共检测到9个性状的49个QTL;在Qi319为供体的导入系群体中,检测到9个性状的42个QTL。在2个及以上环境中均检测到的QTL有16个。同一性状在不同环境下所检测的QTL定位在相同的染色体区域,不同性状的QTL也定位在相同或临近的染色体区域,形成多个QTL富集区。2个群体所检测的QTL位点具有较少的一致性,说明2个供体材料中含有不同的有利基因位点。同时,导入片段中含有利基因的导入系,其相关性状明显得以改良,这些导入系可用于QTL聚合以改良掖478的产量相关性状。【结论】QB80较Qi319与掖478间的遗传差异更大,能检测更多的产量性状QTL;2个导入系群体中含有优良等位基因的导入系可用于QTL聚合改良掖478;QTL富集区是为产量性状基因的克隆提供可供参考的重要染色体区域。
[本文引用: 1]
[本文引用: 1]
Magsci [本文引用: 1]

<P><FONT face=Verdana>【目的】研究红麻重要产量性状的QTL定位,促进红麻分子辅助育种基础科学研究。【方法】以埃及的阿联红麻与福建农林大学育成的高产抗病优质新品种福红992作为亲本进行杂交,自交衍生的162个F2﹕3家系为材料,通过一年两点的随机区组田间试验,测定了红麻株高、茎粗、节数、单株鲜皮重、单株干皮重和种子千粒重6个重要产量性状的数据,采用混合线性模型的复合区间作图法进行了QTL定位及其遗传互作效应分析。【结果】相关分析结果表明,除种子千粒重外,其它性状之间均存在明显的正相关性。在两地共定位了2个株高QTL、2个茎粗QTL、2个节数QTL、1个单株鲜皮重QTL、2个单株干皮重和2个种子千粒重QTL。【结论】在两地检测到11个QTL,主要集中分布在第6、11、14、9、13、17和4连锁群上,这些QTL在连锁群上分布不均匀,具有集中分布的特点。<BR></FONT></P>
Magsci [本文引用: 1]

<P><FONT face=Verdana>【目的】研究红麻重要产量性状的QTL定位,促进红麻分子辅助育种基础科学研究。【方法】以埃及的阿联红麻与福建农林大学育成的高产抗病优质新品种福红992作为亲本进行杂交,自交衍生的162个F2﹕3家系为材料,通过一年两点的随机区组田间试验,测定了红麻株高、茎粗、节数、单株鲜皮重、单株干皮重和种子千粒重6个重要产量性状的数据,采用混合线性模型的复合区间作图法进行了QTL定位及其遗传互作效应分析。【结果】相关分析结果表明,除种子千粒重外,其它性状之间均存在明显的正相关性。在两地共定位了2个株高QTL、2个茎粗QTL、2个节数QTL、1个单株鲜皮重QTL、2个单株干皮重和2个种子千粒重QTL。【结论】在两地检测到11个QTL,主要集中分布在第6、11、14、9、13、17和4连锁群上,这些QTL在连锁群上分布不均匀,具有集中分布的特点。<BR></FONT></P>
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
