 ,1,*
,1,*Cloning and analysis of BoCDPK14 in self-incompatibility Brasscia olerace
BAI Xiao-Jing1,**, LIAN Xiao-Ping2,**, WANG Yu-Kui1, ZHANG He-Cui1, LIU Qian-Ying1, ZUO Tong- Hong1, ZHANG Yi-Zhong1, XIE Qin-Qin1, HU Deng-Ke1, REN Xue-Song2, ZENG Jing3, LUO Shao-Lan1, PU Min1, ZHU Li-Quan ,1,*
,1,*通讯作者:
第一联系人:
收稿日期:2019-03-15接受日期:2019-06-12网络出版日期:2019-07-09
| 基金资助: |
Received:2019-03-15Accepted:2019-06-12Online:2019-07-09
| Fund supported: |
作者简介 About authors
白晓璟,E-mail:xiamoyanyu33@163.com。

摘要
关键词:
Abstract
Keywords:
PDF (2173KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
白晓璟, 廉小平, 王玉奎, 张贺翠, 刘倩莹, 左同鸿, 张以忠, 谢琴琴, 胡燈科, 任雪松, 曾静, 罗绍兰, 蒲敏, 朱利泉. 甘蓝SI相关基因BoCDPK14的克隆与分析[J]. 作物学报, 2019, 45(12): 1773-1783. doi:10.3724/SP.J.1006.2019.94040
BAI Xiao-Jing, LIAN Xiao-Ping, WANG Yu-Kui, ZHANG He-Cui, LIU Qian-Ying, ZUO Tong- Hong, ZHANG Yi-Zhong, XIE Qin-Qin, HU Deng-Ke, REN Xue-Song, ZENG Jing, LUO Shao-Lan, PU Min, ZHU Li-Quan.
自交不亲和性(self-incompatibility, SI)是植物限制自交衰退、促进杂交优势, 在长期进化过程中形成的一种复杂而完善的重要遗传机制。甘蓝属于典型的孢子体自交不亲和植物, 其分子机制主要集中在SI信号传导元件协同作用抑制自花花粉萌发或花粉管生长。目前研究最多的是由SRK-ARC1- Exo70A1等介导的蛋白质泛素化降解途径。Gu等[1]以SRK胞内激酶域为诱饵, 通过酵母双杂交技术从甘蓝型油菜柱头cDNA文库中鉴定出与SRK结合的靶蛋白——ARC1, 其在自交不亲和信号传递过程中起正调控作用。Vanoosthuyse等[2]和Stone等[3]利用RNAi和反义抑制技术分别抑制琴叶拟南芥(Arabidopsis lyrata)和甘蓝型油菜(Brassica napus) ARC1的表达, 以及在甘蓝型油菜中过表达Exo70A1均只能部分打破自交不亲和性。在拟南芥中, ARC1基因以假性遗传因子存在, 向自交亲和拟南芥中仅仅转入琴叶拟南芥的SCR和SRK也能表现出强自交不亲和性[4,5,6]。由此表明ARC1可能并非SRK唯一下游信号元件。
Elleman等[7]证明油菜柱头乳突细胞质内的钙峰与后续花粉粒的水合相对应, Iwano等[8]在亲和授粉后观察到拟南芥乳突细胞胞质钙浓度变化, 在花粉水化后、花粉萌发前和花粉管穿过柱头细胞壁这3个时段Ca2+浓度显著上升, 胞质Ca2+的急剧增加, 是由相同单倍型的SCR与SRK相互作用造成的, 表明Ca2+在调控柱头应答的信号传导中起重要作用。Dearnaley等[9]测定了自交亲和与自交不亲和花粉授粉后在柱头突触细胞的细胞质和细胞壁中的钙离子流, 发现这些钙离子流与接触花粉粒细胞区域相关, 只在特定的一部分区域形成。因此Ca2+可能介导授粉和SI信号转导过程。
钙依赖蛋白激酶(calcium-dependent protein kinase, CDPK)已被广泛证明参与植物的多种抗逆反应[10,11,12]。目前报道, 植物CDPKs在与Ca2+结合后被激活, 从而磷酸化下游底物, 底物通过与CDPK的互作将Ca2+信号级联放大并向下游传导, 进而调控信号转导途径下游基因的表达, 调控植物逆境胁迫应答等生物学过程。由于授粉过程中的自花花粉刺激柱头产生SI信号传导的过程, 类似于逆境刺激植物细胞产生Ca2+依赖性CDPKs的过程, 那么CDPKs是否有可能通过某种分子过程与自交不亲和相联系呢?本研究通过转录组测序分析甘蓝自花和异花授粉后基因表达情况, 首次成功地筛选到1个受自花授粉诱导上调表达的基因, 它与拟南芥中参与植物逆境信号传导的钙依赖蛋白激酶基因高度同源, 命名为BoCDPK14。然后经克隆、序列分析、生物信息学、特异性表达和原核表达、亚细胞定位、启动子活性分析以及通过构建酵母双杂交cDNA文库, 筛选与其互作的蛋白等深入探索, 证明该基因参与了柱头响应花粉刺激的分子过程, 初步提供一个联系自交不亲和性和植物抗逆性的“桥梁”蛋白质, 为甘蓝SI的深入研究提供了新内容。
1 材料与方法
1.1 材料
甘蓝高代自交不亲和系‘A4’和‘F1’由西南大学十字花科蔬菜研究所提供, 于2017年3月底至4月初, 选取长势相同的‘A4’花蕾, 在开花当天人工去雄, 用成熟的‘A4’和‘F1’花粉对‘A4’植株进行自花和异花授粉。然后进行2种方式取材, 一是分别选取自花、异花授粉0、15、30、60 min的柱头, 立即取下置液氮中保存; 另一处理是同时取花蕾、花瓣、叶片、萼片、花药和柱头置液氮中保存备用。另外选取甘蓝‘A4’花蕾, 使用CloneMiner II cDNA文库构建试剂盒(Invitrogen公司, 美国)构建cDNA文库, 酵母质粒小提试剂盒、各种引物合成及测序等均由上海生工生物工程有限公司完成。1.2 SI甘蓝自花、异花授粉后转录组测序及自花授粉相关特异序列的筛选
收集未授粉柱头, 自花授粉15、30、60 min以及异花授粉15、30、60 min的柱头, 并提取其总RNA, 送北京百迈克公司用Illumina HiSeq高通量测序平台测序分析, 测序类型为PE150, 构建转录组文库进行建库测序, 获得转录组数据。采用FPKM (Fragments Per Kilobase of transcript per Million fragments mapped)作为衡量转录本或基因表达水平的指标。在差异表达基因检测过程中, 将差异倍数Fold Change≥2且错误发现率(False Discovery Rate) FDR<0.01作为筛选标准。根据基因在不同授粉处理后的相对表达量, 筛选在自花授粉后表达差异明显、异花授粉后变化趋势不明显的基因。1.3 生物信息学分析
利用MEGA7软件和NCBI网站(1.4 BoCDPK14基因全长及激酶域的克隆
根据目的基因编码区3°端和5°端的保守序列, 以甘蓝A4柱头cDNA和gDNA为模板, 利用Kodaq 2X PCR MasterMix(abm), 以F: 5°-ATGGGGAATTG CTGTGGAAC-3°和R: 5°-CTCTGCATCGCGATTAT TAGAA-3°为引物扩增BoCDPK14基因全长。同时用NCBI网站和SMARTTable 1
表1
表1基因克隆及定量PCR中的引物
Table 1
| 引物名称 Primer | 引物序列 Primer sequence (5°-3°) | 用途 Use |
|---|---|---|
| GST | F: GATCTGGTTCCGCGTGGATCCATGGGGAATTGCTGTGGAAC R: GATGCGGCCGCTCGAGTCGACCTCTGCATCGCGATTATTAGAA | 基因的原核表达 Prokaryotic expression |
| QRP | F: TCAAGAAAAGAGCACTCAGGG R: GATGTGTACAGATATGGCTACGAAC | 荧光定量PCR引物 qRT-PCR |
| DActin | F: GGCTGATGGTGAAGATATTCA R: CAAGCACAATACCAGTAGTAC | 扩增内参基因 For the internal control |
| CDPK14-GFP | F: AAGTCCGGAGCTAGCTCTAGAATGGGGAATTGCTGTGGAAC R: GCCCTTGCTCACCATGGATCCCTCTGCATCGCGATTATTAGAA | 基因亚细胞定位 Subcellular location |
| CDPK14Δ-BD | F: AGGACCTGCATATGGCCATGGATGGGGAATTGCTGTGGAAC R: CCGCTGCAGGTCGACGGATCCTAACCATGGATGTTCAAGCACTT | 酵母表达 Yeast expression |
| GLR2.8d-AD | F: GTACCAGATTACGCTCATATGAGCCCGACAAGTGAAATTAAAGTAG R: ATGCCCACCCGGGTGGAATTCTTTAAGGAACACCCATGTGTTCTTG | 酵母表达 Yeast expression |
新窗口打开|下载CSV
1.5 原核表达
根据目的基因编码区3°端和5°端的保守序列以及原核表达载体pGEX-4T-1的序列, 用Primer Premier 6.0软件设计引物GST-F/GST-R (表1), 以甘蓝A4柱头cDNA为模板扩增BoCDPK14全长编码区序列, PCR体系含8.5 μL ddH2O、12.5 μL Kodaq 2X PCR MasterMix缓冲液、上下游引物各1 μL和模板2 μL; 反应程序为94℃ 3 min; 94℃ 30 s, 60℃ 30 s, 72℃ 96 s, 40个循环; 72℃延伸5 min, 回收目的片段, 运用快速克隆技术将目的片段连接到pGEX- 4T-1载体上后转化大肠杆菌感受态细胞DH5α, 经菌液PCR筛选阳性克隆后送生工生物工程(上海)有限公司测序。将测序正确的原核表达菌株扩大培养后抽提重组质粒转化表达菌株E. coli Transetta (DE3), 用引物GST-F/GST-R进行PCR检测, 将阳性转化子单克隆按1∶100扩大培养, 取500 μL菌液接种于50 mL含100 μg mL-1氨苄抗性LB培养基中, 摇床37℃, 250 r min-1振荡培养至OD600达到0.6~0.8, 加入IPTG至终浓度为1 mmol L-1, 16℃过夜诱导, 诱导结束后, 收集诱导的菌体, 将所有的样品保存于-80℃, 用于表达产物SDS-PAGE电泳检测。1.6 亚细胞定位表达分析
根据目的基因全长编码区序列和Pro Ligation-Free Cloning Kit (abm)快速克隆技术原理以及GFP融合表达载体PAN580序列设计引物CDPK14- GFP-F和CDPK14-GFP-R (表1), 利用PCR技术扩增得到目的基因的片段。将扩增产物与PAN580载体连接, 构建GFP融合蛋白的瞬时表达载体后转化大肠杆菌感受态细胞DH5α, 筛选阳性克隆后送测序, 测序正确后, 提取质粒, 将重组质粒以及空载质粒用PEG法转入拟南芥原生质体, 培养箱暗培养16 h后, 在激光共聚焦显微镜下观察GFP绿色荧光以研究目的基因的亚细胞定位情况。1.7 组织特异性表达分析
参照RNA提取试剂盒RNAprep Pure Plant Kit说明书提取甘蓝柱头、叶片、花药等组织的RNA, 参照TransScript First-Strand cDNA Synthesis SuperMix说明书反转录合成cDNA, 于-20oC保存备用。以dActin作为内参(表1), 根据获得的BoCDPK14基因全长序列设计荧光定量PCR特异性引物: qRP-F、qRP-R (表1), 以叶片、花蕾、萼片、花瓣、花粉和柱头组织的cDNA为模板, 参照SYBR Premix Ex Taq说明书, 用7500型实时荧光定量PCR仪扩增。同时以未授粉柱头, 自花授粉15、30和60 min和异花授粉15、30和60 min的柱头cDNA为模板, dActin为内参, 在荧光定量PCR仪上对目的基因进行荧光定量PCR。反应体系为20 μL, 含2×SYBR Premix Ex Taq 10 μL、引物各1 μL、cDNA模板1 μL、超纯水至7 μL。扩增程序为95oC预变性1 min; 95oC 15 s, 58oC 15 s, 72oC 30 s共40个循环。每个试验设置3次生物学重复和3次技术重复, 采用2-ΔΔCt法计算目的基因的相对表达水平。1.8 甘蓝酵母双杂交筛选
1.8.1 诱饵蛋白毒性及自激活活性检测 将BoCDPK14基因激酶结构域克隆到pGBDT7载体上作为诱饵, 所用引物为CDPK14Δ-BD-F/CDPK14Δ- BD-R (表1), 将诱饵载体重组质粒pGBDT7- CDPK14Δ和pGBDT7空质粒分别转化酵母Y2HGold感受态细胞, 涂布于SD-Trp平板上, 30℃倒置培养3~5 d, 以转化pGBDT7空质粒的平板作为对照, 观察菌落生长状况。若转化重组质粒的菌落明显比空质粒小, 说明重组质粒有毒性。将重组质粒pGBDT7-CDPK14Δ与pGADT7空载共转化酵母Y2HGold感受态细胞, 转化后的菌液涂布在SD/-Leu/-Trp/-His平板上, 30℃倒置培养3~5 d, 以共转阴性对照pGBDT7-Lam×pGADT7-T和阳性对照pGBDT7-p53×pGADT7-T平板作为对照, 观察菌落显色情况。
1.8.2 文库阳性克隆的初步鉴定、测序和生物信息学分析 根据制造商酵母方案手册(Invitrogen), 使用Clontech双杂交系统进行酵母双杂交筛选, 将共转化后的酵母感受态细胞涂布于TDO (SD/-Leu/ -Trp/-His)固体培养基上, 30℃倒置培养3~5 d, 观察菌落在TDO平板的生长情况, 参考酵母质粒提取试剂盒说明书提取质粒pGADT7载体, 然后以提取的酵母质粒为模板, 以T7 5°-TAATACGACTCACTA TAGGG-3°和3°AD 5°-AGATGGTGCACGATGCACA G-3°为引物进行RCR扩增, 将胶回收产物送上海生工生物工程有限公司测序。将测得的序列在NCBI数据库进行BLAST比对, 找到同源序列, 统计序列长度、基因登录号、基因注释等信息。
1.8.3 点对点回转阳性验证 从阳性克隆中选择BoGLR2.8基因作为候选猎物, 将去除信号肽(1~31 aa)和跨膜结构域(580~960 aa)的基因序列BoGLR2.8d连接到pGADT7载体上, 所用引物为: GLR2.8d-AD- F/GLR2.8d-AD-R (表1), 将其与pGBDT7-CDPK14Δ质粒共转化Y2HGold感受态细胞, 设立阴性对照(pGBDT7-Lam×pGADT7-T)和阳性对照(pGBDT7- p53×pGADT7-T), 将共转化后的酵母Y2HGold细胞涂布于DDO (SD/-Leu/-Trp)和TDO(SD/-Leu/- Trp/-His)平板上, 30℃倒置培养3~5 d, 挑取DDO平板上的单菌落进行PCR检测后, 将DDO平板上的阳性克隆菌斑接种至四缺培养基QDO (SD/-Leu/- Trp/-His/-Ade/X/A)上, 30℃倒置培养3~5 d, 观察其生长情况。
2 结果与分析
2.1 BoCDPK14转录水平分析
根据甘蓝自花和异花授粉0~60 min的柱头转录组数据分析, 成功地筛选到1个受自花授粉诱导上调表达的基因, 它与拟南芥中参与植物逆境信号传导的钙依赖蛋白激酶基因高度同源[13], 命名为BoCDPK14。BoCDPK14基因在未授粉时相对表达量为0.03, 自花授粉60 min后达到最高, 为3.82, 显著上调表达; 而在异花授粉15、30、60 min后其表达量分别约为0.62、0.76、0.72, 其表达量趋于稳定(图1)。上述结果表明参与植物逆境反应的CDPK也可能参与了SI反应。图1
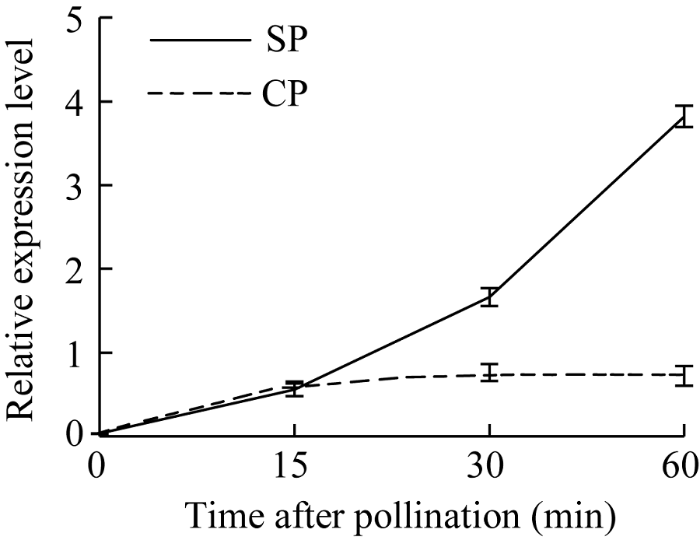 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1甘蓝柱头内响应自花和异花授粉后BoCDPK14表达模式
SP: 自花授粉; CP: 异花授粉。
Fig. 1Expression pattern of BoCDPK14 in stigmas in response to self-pollination and cross-pollination
SP: self-pollination; CP: cross-pollination.
2.2 BoCDPK14的克隆
BoCDPK14 gDNA PCR产物大小约2200 bp, 柱头cDNA PCR产物约1600 bp (图2)。PCR扩增所获得的BoCDPK14 gDNA序列长度为2214 bp, cDNA 序列长度为1599 bp, 包含了BoCDPK14完整的编码框。图2
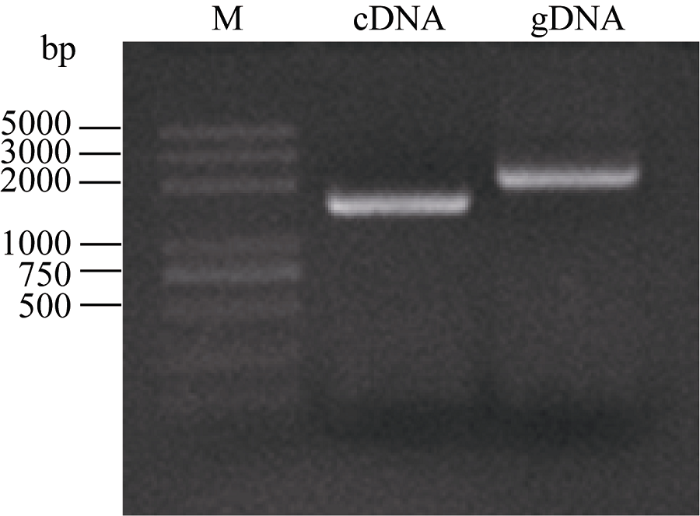 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2甘蓝BoCDPK14基因cDNA和gDNA序列扩增
Fig. 2Amplification of BoCDPK14 gene from cDNA and gDNA of the stigma of Brassica oleracea
由于使用同一对引物同源克隆得到cDNA和gDNA序列相差615 bp, 所以本研究进一步对两者进行了Clustal软件比对分析, 发现该基因由8个外显子(分别为504、124、144、153、116、168、231和159 bp)和7个内含子(分别为91、68、97、89、86、97和87 bp)组成。BoCDPK14基因的开放阅读框(ORF)全长为1599 bp, 编码532个氨基酸残基,相对分子质量为60.4 kD, 理论等电点(pI)为6.73, 不稳定系数为34.02, 属于稳定蛋白。平均亲水性系数为-0.396, 推测为亲水性蛋白。由图3可以看出, 在氨基酸组成中, 亮氨酸、赖氨酸和谷氨酸出现频率最高, 分别占氨基酸总数的8.8%、8.6%和8.6%。带负电荷的氨基酸残基(Asp+Glu)总数为83个, 带正电荷的氨基酸残基(Arg+Lys)为81个。这种偏酸的亲水蛋白, 正是SI反应和抗逆反应所需要的。
图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3甘蓝BoCDPK14 CDNA结构图及其推导的氨基酸序列
下划直线为酪蛋白激酶II磷酸化位点, 虚线框为蛋白激酶C磷酸化位点, 椭圆为N-糖基化位点, 实线框为cAMP或cGMP依赖性蛋白激酶磷酸化位点, 双线框表示N-肉豆蔻酰化位点, 圆点为EF-hand钙结合结构域, 双线表示蛋白激酶ATP结合区, 星星表示丝氨酸/苏氨酸蛋白激酶活性位点。
Fig. 3Gene structure of Brassica oleracea BoCDPK14 and its deduced amino acid sequence
The underline show protein kinase II phosphorylation site; the dashed box show protein kinase C phosphorylation site; the oval show N-glycosylation site; the box show cAMP or cGMP-dependent protein kinase phosphorylation site; the double box show N-myristoylation site; the dot show the EF-hand calcium binding domain; double line show protein kinase ATP binding domain; stars show serine/threonine protein kinase active site.
2.3 BoCDPK14的结构分析
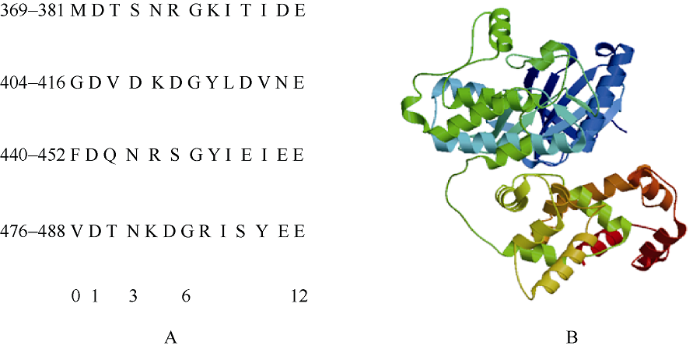
通过Predict protein等软件对CDS的氨基酸编码序列进行在线预测分析, 该蛋白含有4个N-糖基化位点、3个cAMP或cGMP依赖性蛋白激酶磷酸化位点、8个蛋白激酶C磷酸化位点、15个酪蛋白激酶II磷酸化位点、4个N-肉豆蔻酰化位点、1个蛋白激酶ATP结合区、1个丝氨酸/苏氨酸蛋白激酶活性位点、4个EF-hand钙结合结构域(图3), 而已报道的自交不亲和信号传导元件中, SRK属于蛋白激酶类, 其C端具有丝氨酸/苏氨酸蛋白激酶活性位点, 是研究SI信号传导的关键位点。Ca2+结合的核心区域分别位于第369~381、第404~416、第440~452、第476~488位氨基酸残基, 其特点是由12个氨基酸残基组成的α-螺旋环结构, 其中的第1、第3、第6和第12位氨基酸较为保守(图4-A)。在这12个残基的前一位氨基酸(第0位氨基酸)为疏水性氨基酸残基, 由第1、第3、第5、第7、第9和第12位氨基酸残基分别按照X、Y、Z、-Y、-X和-Z空间排列形成五边形双锥结构, 而第12位保守的Asp或Glu的2个氧原子与Ca2+以双齿配体的方式结合, 使EF-hand结构域构象改变, 最终导致靶蛋白的激活或者失活[14,15,16], 说明BoCDPK14可能参与Ca2+信号调控。
图4
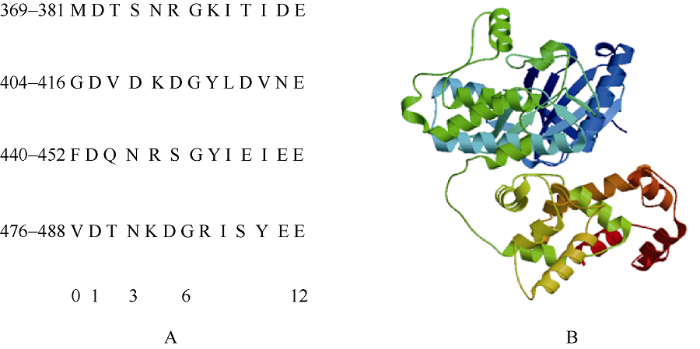 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4BoCDPK14 蛋白Ca2+结合核心区域(A)和BoCDPK14蛋白三维结构预测(B)
A图下排数字表示对这12个氨基酸残基的编号。
Fig. 4Core area of calcium ion binding (A) and predicted three-dimensional structure for BoCDPK14 protein (B)
The figures at lower part in Fig. A are the serial number of the 12 amino acid residues of the core area of calcium ion binding.
利用Swiss-modle在线软件对BoCDPK14蛋白的三级结构进行预测, 得到该蛋白的三维结构(图4-B)。利用MEGA7软件构建BoCDPK14遗传进化树发现BoCDPK14与BrCDPK14亲缘关系最近(图5)。
图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5BoCDPK14与其他物种CDPK14氨基酸序列的系统进化树
Fig. 5Phylogenetic tree of BoCDPK14 and other species CDPK14 amino acid sequence
2.4 BoCDPK14基因的响应元件
利用 BRAD数据库及PlantCARE启动子分析软件在位于BoCDPK14基因起始密码子上游2000 bp的核苷酸序列中预测到胁迫反应、激素反应、代谢调节等多种顺式作用元件(表2), 表明BoCDPK14基因除了响应授粉刺激, 还可能响应包括逆境在内的其他多种信号。Table 2
表2
表2BoCDPK14基因上游调控区顺式作用元件
Table 2
| 相关功能预测 Associated putative function | 启动子顺式作用元件 cis-elements in the promoter region |
|---|---|
| Abscisic acid responsiveness | ABRE |
| Anaerobic induction | ARE |
| DNA binding protein (ATBP-1) | AT-rich element |
| Meristem expression | CAT-box, CCGTCC-box |
| Ethylene-responsive element | ERE |
| Gibberellin-responsive element | GARE-motif |
| Endosperm expression | GCN4_motif, Skn-1_motif |
| MYB binding site | MRE |
| Zein metabolism regulation | O2-site |
| Stress responsiveness | TC-rich repeats, HSE |
| Light responsive | ACE, Box 4, Box I, G-Box, GA-motif, GATA-motif, GTGGC-motif, I-box, 3-AF1 binding site |
新窗口打开|下载CSV
2.5 甘蓝BoCDPK14的原核表达和亚细胞定位
将GST-BoCDPK14重组菌株扩大培养, 并在16℃过夜诱导表达, 诱导结果电泳检测如图6。与未诱导的GST-BoCDPK14 (泳道1)相比, 在80 kD左右, 诱导表达后的泳道2多出1条带, 其蛋白质相对分子质量与GST-BoCDPK14估计值相符。其中pGEX-4T-1空载体表达约26 kD的蛋白, BoCDPK14预测分子大小约为60.4 kD, 二者融合约为86 kD的蛋白, 表明GST-BoCDPK14融合蛋白在原核细胞中诱导表达成功。通过构建GFP-BoCDPK14融合表达载体, 并转化拟南芥原生质体细胞。发现GFP蛋白分布在细胞质和细胞核中, 而GFP-BoCDPK14融合蛋白绿色荧光明显定位在细胞质(图7), 表明该亲水性蛋白定位于细胞质。以上研究结果表明BoCDPK14可能是细胞质内的活性蛋白, 而该蛋白质的胞质亲水特性正是参与SI和抗逆反应所需要的。图6
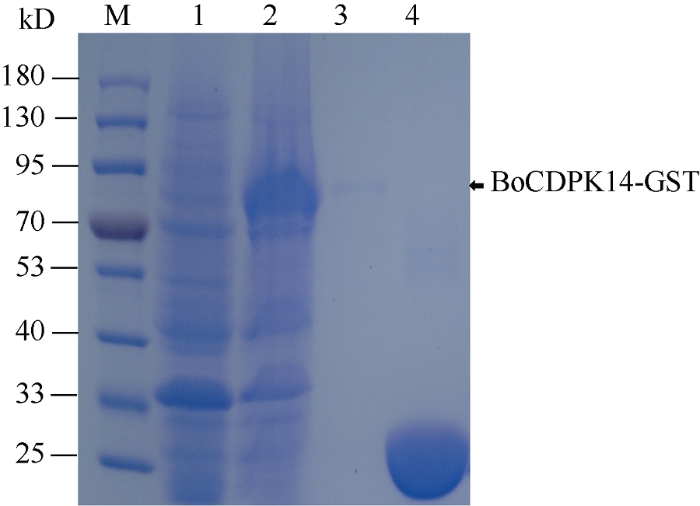 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6BoCDPK14蛋白的原核表达
M: 蛋白相对分子质量标准; 1: BoCDPK14-GST未诱导蛋白; 2: BoCDPK14-GST诱导蛋白; 3: BoCDPK14-GST纯化蛋白; 4: GST蛋白。
Fig. 6Prokaryotic expression protein analysis of BoCDPK14 protein
M: marker; 1: uninduced control; 2: BoCDPK14-GST fusion protein; 3: BoCDPK14-GST purified protein; 4: GST protein.
图7
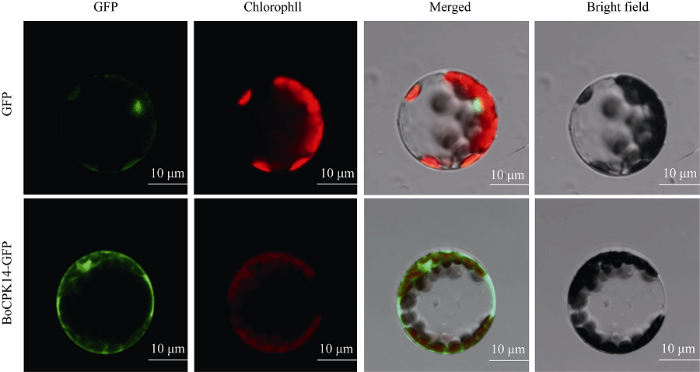 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7BoCDPK14-GFP 融合蛋白的亚细胞定位
绿色表示GFP 蛋白在激光共聚焦显微镜下所发的绿色荧光。
Fig. 7Subcellular localization of BoCDPK14-GFP protein in protoplast cell
GFP protein showed the green fluorescence under confocal laser scanning microscope.
2.6 甘蓝BoCDPK14基因的表达分析
BoCDPK14在甘蓝柱头、花粉、花蕾、花瓣和叶片中表达, 且柱头中的表达量低于花粉(图8-B)。对不同授粉处理后柱头中BoCDPK14的qRT-PCR分析(图8-A)表明, BoCDPK14在自花授粉0~60 min持续上调表达, 在异花授粉过程中变化不大。自花授粉60 min后的相对表达量约为16.09, 约为授粉0 min的13.5倍, 而异花授粉60 min后的相对表达量约为5.36, 前者明显高于后者且相差3倍, 说明自花授粉能显著诱导该基因的表达。该结果与转录组分析结果一致, 说明这个参与植物逆境信号传导的CDPK14基因也可能参与甘蓝SI反应过程。图8
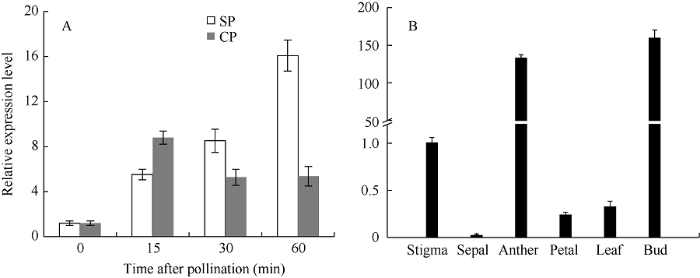 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图8甘蓝柱头内BoCDPK14响应自花和异花授粉后表达模式(A)和BoCDPK14基因在甘蓝不同组织中的表达分析(B)
SP: 自花授粉; CP: 异花授粉。
Fig. 8Expression pattern of BoCDPK14 in stigmas in response to self-pollination and cross-pollination of Brassica oleracea (A) and expression analysis of BoCDPK14 in different organs of Brassica oleracea (B)
SP: self-pollination; CP: cross-pollination.
2.7 甘蓝BoCDPK14功能域分析与截短体构建
NCBI保守域预测表明, BoCDPK14分为4个典型的结构域, 从N端到C端依次为N-末端可变域、催化域(激酶域)、连接域(自抑制域)和C-末端类钙调结构域(图9)。可变域氨基酸长度一般在20~200个不等, 氨基酸水平同源性不高, 保守性差, 多数CDPK的N端含有豆蔻酰化位点, 可被豆蔻酰化或棕榈酰化。催化域具有典型的Ser/Thr蛋白激酶保守序列, 同源性较高, 该区域突变会丧失催化活性。CDPK连接域由20~30个氨基酸残基组成, 当无Ca2+或Ca2+低于某一浓度时, 该区域结合催化区的激酶结构域从而抑制其活性。Ca2+结合区即类钙调结构域, 包含4个可与Ca2+结合的EF手型结构, 能够与Ca2+结合, 引起蛋白结构变化, 解除连接域对激酶区的抑制作用从而激活CDPK的活性[17,18]。为了确定与CDPK14激酶域相互作用的蛋白, 扩增CDPK14截短体的编码序列CDPK14Δ, 利用分子克隆技术将CDPK14Δ序列克隆到pGBDT7载体中。图9
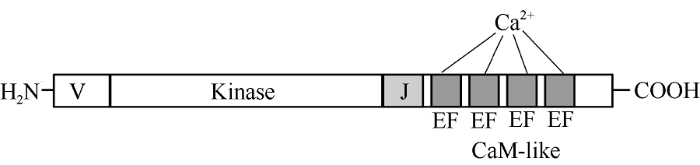 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图9典型钙依赖蛋白激酶(CDPK)结构
V: 可变域; J: 连接域; EF: EF-hand 结构域。
Fig. 9Typical calcium-dependent protein kinase (CDPK) structure
V: variable domain; J: junction domain; EF: EF-hand domain.
2.8 重组诱饵载体毒性及自激活检测
将pGBDT7-CDPK14Δ和pGBDT7空质粒分别转化Y2Hgold酵母感受态细胞, 涂布于SD-Trp平板上, 30℃倒置培养3~5 d。结果发现重组质粒在平板上均可以长出菌落, 且大小和转化pGBDT7空质粒的酵母菌落大小一致, 表明重组质粒没有毒性。将pGBDT7-CDPK14Δ与pGADT7空载共转化酵母Y2HGold感受态细胞, 结果发现只有阳性对照转化的菌落在SD/-Leu/-Trp/-His平板上生长, 其余无斑生长, 说明表达的重组蛋白没有自激活能力。2.9 甘蓝CDPK14激酶结构域与GLR2.8d蛋白相互作用
将酵母文库筛选出的BoGLR2.8d基因连接到pGADT7载体上, 与CDPK14Δ-BD重组质粒共转化酵母Y2Hgold感受态细胞, 分别涂在DDO和TDO固体培养基上, 30℃倒置培养3~5 d, 固体平板有菌落生成(图10)。通过PCR检测, 在同一个单克隆中可以同时扩增出2个目的片段, 说明重组质粒成功转入酵母细胞。为进一步检测蛋白质之间的相互作用, 将SD/-Leu/-Trp平板上的单菌落转移至SD/-Leu/-Trp/-His/-Ade/X-α-gal/AbA平板上, 30℃倒置培养3~5 d, 发现pGBDT7-CDPK14Δ× pGADT7-GLR2.8d和阳性对照能生长且呈蓝色, 而阴性对照菌落不能生长。研究已发现GLR参与了自交不亲和反应[19], 而BoCDPK14激酶结构域在酵母中与BoGLR2.8d (XP_013635321.1)之间存在相互作用(图10), 进一步表明CDPK14也参与了甘蓝SI反应。图10
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图10酵母双杂交验证CDPK14Δ和GLR2.8d之间的相互作用
第1排表示pGADT7-BoGLR2.8d×pGBDT7-BoCDPK14Δ; 第2排表示阳性对照(pGADT7-T×pGBDT7-p53); 第3排表示阴性对照(pGADT7-T×pGBDT7-Lam)。
Fig. 10Interaction of CDPK14Δ and GLR2.8d in yeast two-hybrid assay
The first row indicates pGADT7-BoGLR2.8d×pGBDT7-BoCDPK14Δ; the second row indicates positive control (pGADT7-T×pGBDT7-p53); the third row indicates negative control (pGADT7-T×pGBDT7-Lam). DDO: SD/-Leu/-Trp; TDO: SD/-Leu/-Trp/-His; QDO/X/A: SD/-Leu/-Trp/-His/-Ade/X-α-gal/AbA.
3 讨论
本研究基于以自交不亲和甘蓝经不同授粉处理后的转录组数据, 成功地筛选到1个受自花授粉诱导上调表达的基因, 它与拟南芥中参与植物逆境信号传导的钙依赖蛋白激酶基因高度同源, 命名为BoCDPK14。BoCDPK14-GFP融合蛋白绿色荧光主要出现在细胞质区域。BoCDPK14基因的启动子区域含有胁迫反应、激素反应、代谢调节等多种顺式作用元件, 表明BoCDPK14基因可能参与调控复杂的生物反应过程。Vanoosthuyse等[2]发现钙调素CaM1和CaM2与iSRK (SRK胞内激酶域)能发生相互作用。许俊强等[20]研究发现甘蓝花粉管钙调蛋白CaM12与iSRK相互作用, 而缺失EF-hand模体的钙调蛋白均不能与iSRK相互作用。本文通过蛋白结构分析发现, BoCDPK14含有4个能与Ca2+结合的EF-hand模体,推测BoCDPK14可能通过调控柱头中Ca2+浓度参与SI过程。Sheen等[21]研究发现CDPK可激活胁迫诱导的启动子, 以促进逆境蛋白的表达, 说明CDPKs可能在逆境胁迫信号中起正调节因子的作用, 以调控植物中胁迫信号的转导。Botella等[22]发现在机械刺激等环境胁迫的信号转导过程中, CDPKs发挥重要的调节作用。同时钙依赖蛋白激酶属于蛋白激酶类, 已报道的自交不亲和信号传导元件中SRK属于蛋白激酶类, SRK在自交不亲和反应过程中作为雌性决定因子, 在花粉与柱头的反应中具有重要作用[23]。自交不亲和反应的本质是花粉与柱头识别及信号传导的过程, 而Ca2+作为第二信使, 在植物细胞信号转导过程中发挥重要作用[24]。已有研究表明, 植物体内钙信号的传递主要是通过钙依赖蛋白激酶(CDPK)来完成[25]。Kunz等[26]发现来自花粉管中的钙依赖蛋白激酶可激活花柱传导组织中的S-核糖核酸酶, 该酶通过催化RNA的降解来抑制花粉萌发。Iwano等[19]通过观察自交不亲和拟南芥发现在用相同单倍型的SCR/SP11花粉授粉后, 柱头乳突细胞中Ca2+浓度显著上升, 花粉不能正常萌发生长, 表现自交不亲和性, 用不同单倍型的SCR/SP11花粉授粉后, 柱头乳突细胞中Ca2+浓度上升不明显。当用谷氨酸通道抑制剂AP-5处理柱头表面时, 胞内Ca2+浓度无明显上升, 说明谷氨酸通道调控的钙离子流是自交不亲和反应所必需的。以上结果表明可能存在Ca2+信号途径参与调控自交不亲和性。
有研究表明, 拟南芥AtGLRs具有介导Ca2+跨膜转运和调节胞内Ca2+动态平衡, 调控花粉管的生长, 以及感应和传递外源与内源信号的多重功能, 在烟草和拟南芥中发现GLRs利用D-丝氨酸(D-serine)作为配体介导Ca2+梯度, 最终影响花粉管生长及其形态建成, 在敲除丝氨酸消旋酶基因的拟南芥突变体sr1雌蕊中野生型植物花粉管伸长生长发生缺陷并伴以GLR1.2和GLR3.7的Ca2+通道活性降低[27,28,29,30,31,32,33,34]。说明高等植物GLR家族成员需要特定的配体物质以开启其Ca2+通道功能。本研究通过酵母双杂交试验证明BoCDPK14激酶结构域能与BoGLR2.8d蛋白相互作用。BoCDPK14蛋白可能通过Ca2+结合区结合Ca2+, 激活CDPK激酶活性, 使其与谷氨酸受体通道蛋白BoGLR2.8d相结合, 促使柱头内Ca2+浓度经谷氨酸受体通道(GLR)内流而显著上升, 使花粉不能正常萌发生长, 最终导致自交不亲和反应的发生。
BoCDPK14基因在自花授粉过程中0~60 min持续上调表达, 在异花授粉过程中变化不大。而甘蓝自交不亲和反应主要在开花后30~60 min之内完成[35], 进一步表明该基因可能参与自花授粉后花粉与柱头相互作用的复杂反应过程, 可能跟自花授粉后相同单倍型的SRK与SCR/SP11特异识别后的Ca2+浓度的显著上升有关, 推测该基因可能作为Ca2+信号元件参与甘蓝自花授粉后的反应过程。由于该基因是已知的参与抗逆反应的基因, 所以本文初步提供一个联系自交不亲和性和植物抗逆性的“桥梁”蛋白质, 为SI的深入研究提供了新内容。
4 结论
从甘蓝转录组文库筛选并克隆1个受自花授粉诱导显著上调表达的基因BoCDPK14, 其开放阅读框为1599 bp, 编码533个氨基酸, 编码蛋白大小约为60.4 kD。BoCDPK14蛋白分布于细胞质, 在柱头中的表达量低于花粉中。该基因在自花授粉0~60 min持续上调表达, 在异花授粉过程中表达量变化并不明显。BoCDPK14蛋白可能通过结合Ca2+激活CDPK激酶, 使其与谷氨酸受体通道蛋白BoGLR2.8d结合, 促使柱头内Ca2+浓度显著上升, 花粉不能正常萌发生长, 推测该蛋白是参与SI相关过程的新蛋白。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
