 , 牛陆, 张伟, 杨静, 杜茜, 邢国杰, 郭东全, 李启云, 董英山
, 牛陆, 张伟, 杨静, 杜茜, 邢国杰, 郭东全, 李启云, 董英山
* 通讯作者(Corresponding author): 董英山, E-mail: ysdong@cjaas.com, Tel: +86-431-87063008 第一作者联系方式: E-mail: xdyang020918@126.com, Tel: +86-431-87063044
收稿日期:2016-02-16 接受日期:2016-06-20网络出版日期:2016-07-04基金:
摘要
关键词:大豆; 大豆花叶病毒病; SMV-P3; RNAi介导基因沉默
RNAi-mediated SMV-P3 Silencing Increases Soybean Resistance to Soybean Mosaic Virus
YANG Xiang-Dong
 , NIU Lu, ZHANG Wei, YANG Jing, DU Qian, XING Guo-Jie, GUO Dong-Quan, LI Qi-Yun, DONG Ying-Shan
, NIU Lu, ZHANG Wei, YANG Jing, DU Qian, XING Guo-Jie, GUO Dong-Quan, LI Qi-Yun, DONG Ying-Shan
Fund:
Abstract
Keyword:Soybean; Soybean mosaic virus; SMV-P3; RNAi-mediated gene silencing
Show Figures
Show Figures












大豆花叶病毒(soybean mosaic virus, SMV)病是由大豆花叶病毒引起的一类世界性大豆病害, 也是我国各大豆主产区最重要的病害之一。SMV一般可造成大豆减产10%~30%, 严重年份和地区甚至造成大面积绝产[1, 2, 3]。SMV还常常造成籽粒斑驳, 严重影响大豆籽粒的外观品质和商品价值[4]。在分类学上, SMV属马铃薯Y病毒科(Potyviridae)马铃薯Y病毒属(Potyvirus), 主要通过受侵染的种子和蚜虫传播。随着全球范围内大豆种质资源交流, 新型病毒株系的出现和致病力强弱的变化, 我国大豆抗SMV育种工作面临的形势也更为严重。Yang等[5]对我国境内采集的206个病毒株和589个SMV样本分析表明, 新型重组花叶病毒(recombinant Soybean mosaic virus, SMV-R)出现频率达到16.7%~60.0%, 且主要集中在我国中部和南部大豆种植区。由于化学防治困难且易造成环境安全等问题, 生产上对大豆花叶病毒的防治主要依赖于抗SMV大豆品种的培育。由于SMV存在复杂的株系分化, 大豆对SMV的抗性又具有株系专化性[6], 因此, 寄主的抗性易随着病毒株系的变异而丧失[7, 8, 9], 培育广谱、抗性持久的抗病品种也成为防治大豆花叶病毒病的根本途径。
RNA干扰(RNA interference, RNAi)是植物抵抗外来核酸(病毒、转座子等)入侵以保持自身基因组完整性的一种防御机制[10, 11]。病毒入侵寄主细胞后, 其双链RNA (double-strand RNA, dsRNA)被植物DCL (Dicer-like)核酸酶加工为vsiRNA (virus short interfering RNA), 随后vsiRNA与AGO蛋白结合, 降解与vsiRNAs 互补配对的长链病毒RNA, 从而阻止病毒的进一步侵染[12]。许多研究表明, 在植物中表达部分病毒基因组序列片段, 产生的dsRNA可以有效抑制病毒的侵染[13, 14, 15]。在大豆抗SMV研究方面, Wang等[16]将携带SMV 3° -UTR的外壳蛋白基因CP导入大豆, 其中2个转基因株系对SMV抗性水平较受体品种显著提高。Furutani等[17]将SMV-CP基因导入大豆, 接种鉴定结果表明, 转基因大豆植株对SMV侵染具有较高的抗性。Zhang等[18]和Kim等[19]分别将SMV-NIB和SMV-CP基因序列反向重复片段(inverted repeat, IR)导入大豆, 接种鉴定结果表明, 转基因大豆能够有效抵御SMV的侵染, 其SMV抗性显著高于受体品种。最近, Gao等[3]将转录后基因沉默(post-transcriptional gene silencing, PTGS)的负调控因子SMV HC-Pro基因IR片段导入大豆, 接种鉴定表明, 转基因大豆对SMV抗性显著提高。可见, 利用宿主介导SMV编码基因RNAi沉默是提高大豆抗SMV的有效手段。更为重要的是, 利用RNAi干扰技术为同时抗多种病毒及病毒不同生理小种提供了一条更为有效的技术途径[13, 18, 20]。但目前已有的研究主要是通过RNAi介导SMV-CP或SMV- HC-Pro基因沉默提高大豆抗SMV水平, 而对其他参与SMV侵染、复制、运动等过程关键基因RNAi沉默介导的SMV抗性研究则尚未见到相关报道。
与Potyvirus属其他病毒成员类似, SMV基因组为单链正义RNA[21], 其基因组共编码11个不同功能的成熟蛋白[22]。P3蛋白是由SMV基因组编码的一个关键蛋白。研究表明, P3蛋白不仅在病毒复制和运动过程中发挥着重要的作用, 同时与感染寄主症状表型密切相关, 并在一定程度上决定了病毒侵染的寄主范围[23, 24]。最近研究表明, P3蛋白是携带Rsv1抗性位点大豆基因型的无毒决定因子[25, 26]。携带Rsv4抗性位点大豆基因型的无毒决定因子也位于P3蛋白序列内部, 并在SMV致病过程中发挥着重要的作用[27, 28, 29]。另外, 位于P3蛋白基因顺反子内部编码75个氨基酸的PIPO蛋白对病毒粒子在寄主体内的运动也发挥着关键的作用[30]。本文利用RNAi介导SMV-P3基因沉默研究对大豆抗SMV的影响, 以期为进一步拓宽大豆抗SMV遗传资源基础, 培育广谱、高抗SMV转基因大豆新品种奠定基础。
1 材料与方法1.1 试验材料大豆花叶病毒SC-3株系和受体品种Williams 82分别由吉林省农业科学院植物保护研究所和大豆研究所提供; 受体品种SN9由沈阳农业大学谢甫绨教授惠赠。大肠杆菌DH5α 购自北京TIANGEN公司; 根癌农杆菌(Agrobacterium tumefaciens)菌株EHA101由本实验室保存; 载体质粒pTF101-RBSC由东北师范大学刘宝教授惠赠。TRIzol试剂和Prime-Scrip反转录试剂盒分别购自美国Invitrogen和日本TaKaRa公司; KOD FX高保真酶购自日本TOYOBO公司; ExTaq MasterMix聚合酶购自北京康为试剂公司; 限制性内切酶Hind III、Sac I等购自美国Thermo公司; 除草剂Basta购自北京鼎国生物公司; 草铵膦(Glufosinate-ammonium, 货号45520- 100MG)购自Sigma公司; QuickStix PAT/bar 转基因检测试纸购自美国EnviroLogix公司; 遗传转化实验中所用的培养基基础成分、植物激素、抗生素等购自美国Phyto Tech 和Sigma公司; 测序与引物合成委托北京华大基因公司完成。
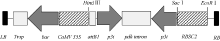
1.2 SMV-P3反向重复片段设计及RNAi表达载体构建利用TRIzol试剂从感染SMV SC-3 病毒的大豆叶片(本实验室保存)中提取总RNA, 并用Prime- Scrip 反转录试剂盒合成cDNA第1链, 保存于-20℃。根据GenBank中已登录SMV株系P3基因保守序列(2529~2822 nt)设计引物, 正向引物为5° -GGTTGTTGGATGCTTTTCTTTC-3° , 反向引物为5° -TCTCTTGATGGGCTTGGTTTCACC-3° 。以合成cDNA第一链为模板, 利用KOD FX 高保真酶扩增目的片段。以pTF101-RBSC质粒为载体骨架, 利用常规分子生物学操作技术将目的片段分别以正向和反向的方式构建到载体中, 构建植物RNAi表达载体pTF101-RBSC-P3, 并送北京华大公司测序验证。构建的重组质粒pTF101-RBSC-P3中, SMV-P3 RNAi片段启动子为菜豆叶片特异启动子RBSC2 (GenBank登录号为AF028707.1), 内含子序列来源于黄菊花属Flaveria trinervia丙酮酸磷酸双激酶pdk基因, 植物筛选标记为草胺膦抗性基因bar (图1)。采用冻融法将载体质粒导入农杆菌菌株EHA101以备转化。
图1
Fig. 1
| Figure OptionViewDownloadNew Window | |
 | 图1 RNAi表达载体pTF101-RBSC-P3 T-DNA区示意图Fig. 1 Schematic representation of the T-DNA region of the recombinant plasmid pTF101-RBSC-P3 |
1.3 农杆菌介导大豆遗传转化采用农杆菌介导法进行大豆遗传转化[31], 转化受体品种为Williams 82和SN9。将萌发培养基GM (B5盐、B5维生素、30 g L-1蔗糖、3.9 g L-1 MES、5 g L-1琼脂粉, pH 5.8)上培养24 h后的大豆种子沿种脐部位剖开, 并在子叶节位置轻微划伤, 然后于农杆菌液中侵染30 min。将侵染后的外植体转移至共培养基CCM (B5盐、B5维生素、30 g L-1蔗糖、3.9 g L-1 MES、1.67 mg L-1 BAP、0.25 mg L-1 GA3、400 mg L-1半胱氨酸、154.2 mg L-1 DTT、200 μ mol L-1 AS、5 g L-1琼脂粉, pH 5.4)上, 于23℃条件下暗培养4 d。将外植体近轴面朝上转移至诱导培养基SIM (B5盐、B5维生素、30 g L-1蔗糖、0.59 g L-1 MES、1.67 mg L-1 BAP、250 mg L-1头孢霉素、100 mg L-1 Timentin、6 mg L-1草丁膦、8 g L-1琼脂粉, pH 5.7)上, 25℃, 16 h/8 h光暗条件下培养2周左右。然后将外植体取出, 切除多余的下胚轴部分, 并转移至诱导培养基SIM上继续培养2~4周。将外植体上产生的丛生芽转移至芽伸长培养基SEM (MS盐、MS维生素、30 g L-1蔗糖、0.59 g L-1 MES、50 mg L-1天门冬氨酸、50 mg L-1 L-谷氨酸、0.1 mg L-1 IAA、0.5 mg L-1 GA3、1.0 mg L-1玉米素核苷、250 mg L-1头孢霉素、100 mg L-1 Timentin、6 mg L-1草丁膦、8 g L-1琼脂粉, pH 5.7)上培养, 培养条件为25℃, 16 h/8 h光暗周期。待抗性芽长至3~5 cm时, 将其切下, 并用1 mg L-1 IBA浸泡30 s, 然后转至生根培养基RM (MS盐、20 g L-1蔗糖、0.59 g L-1 MES、50 mg L-1天门冬氨酸、50 mg L-1 L-谷氨酸、1.0 mg L-1 IBA、3 g L-1植物凝胶, pH 5.6)上继续培养。待抗性芽长出健壮的根时, 移栽至温室中生长结实。
1.4 PCR检测和除草剂喷施鉴定对移栽至温室中的转化再生植株进行PAT/bar试纸检测, 具体检测方法参照产品说明书。采用DNA快速提取法提取转化再生植株叶片DNA[32], 根据植物RNAi表达载体pTF101-RBSC-P3序列设计2对特异性检测引物(P3-F1/P3-R1和Bar-F1/ Bar-R1)。引物P3-F1 (5° -ACCTCAACTCCACCAGC ATC-3° )位于SMV-P3 RNAi片段上游启动子RBSC2, 引物P3-R1 (5° -TACTCTCAACTTTTATCTTCTTCG TC-3° )位于内含子pdk序列。引物对Bar-F1/Bar-R1 (F: 5° -GCACCATCGTCAACCACTACATCGAG-3° , R: 5° - TGAAGTCCAGCTGCCAGAAACCCAC-3° )位于bar基因读码框。预期PCR扩增片段分别为727 bp和441 bp。内标基因Gmactin检测引物为Act-F1/Act-R1 (F: 5° -TTGACTGAGCGTGGTTATTCC-3° , R: 5° -GAT CTTCATGCTGCTGGGTG-3° ), 扩增片段为403 bp。3对引物PCR扩增条件均为95℃ 5 min; 94℃ 30 s, 58℃ 30 s, 72℃ 1 min, 共35个循环; 72℃延伸5 min。扩增产物经1.0%琼脂糖凝胶电泳检测。
对T1~T3代转基因植株后代采用PCR检测和田间喷施除草剂Basta (主要成分为草铵膦)的方法鉴定。PCR检测引物及检测方法同上。待大豆幼苗长出第1组三出复叶时, 喷施500 mg L-1 Basta。7~14 d后调查除草剂抗性情况, 并拔除对除草剂Basta敏感的植株。本研究中获得的PCR阳性和耐除草剂大豆用于后续试验。
1.5 转基因大豆Southern杂交检测选取T1代PCR阳性大豆植株进行Southern杂交检测。采用高盐CTAB法提取转基因大豆和非转化植株叶片总DNA[33]。利用Hind III酶切总DNA (30~50 μ g), 0.8%琼脂糖凝胶电泳分离酶切片段。采用高盐转移法, 将酶切片段转移至带正电荷的Hybond-N+尼龙膜(Amersham, USA)上。根据RNAi载体pTF101- RBSC-P3中的bar基因序列设计引物(引物序列同上), 并扩增探针模板。利用DIG随机引物标记试剂盒标记探针。根据Roche公司的DIG High Prime DNA Labeling and Detection Starter Kit II试剂盒说明书进行探针制备及杂交。杂交温度为42℃, 以2× SSC (含0.1% SDS), 37℃条件下洗膜2次, 每次5 min; 0.5× SSC (含0.1% SDS), 66℃条件下洗膜2次, 每次15 min。然后利用BCIP/NBT在膜上化学显色。
1.6 SMV田间抗性鉴定采用汁液摩擦接种法, 对T2和T3代转基因大豆进行抗SMV鉴定。每个株系种植3垄, 每垄30株, 随机区组设计。待大豆植株第1片三出复叶出现时, 参照Gao等[3]的方法接种SMV SC-3病毒。参照Zhi等[34]提出的SMV病情分级标准, 计算病情指数。高抗(IM), 无可见系统症状, 病情指数为0; 抗病(R), 病情指数在1~20之间; 中抗(MR), 病情指数在21~35之间; 中感(MS), 病情指数在36~50之间; 感病(S), 病情指数在51~70之间; 高感(HS), 病情指数大于70。对受体大豆和转基因大豆的株系生育期(growing period duration, GPD)、叶形(leaf shape, LS)、花色(flower color, FC)、株高(plant height, PlH)、种皮色(seed coat color, SCC)、分枝数(branching number, BN)、节数(node number, NN)、结荚高度(podding height, PoH)、百粒重(100-seed weight, 100SW)、种脐色(hilum color, HC)等表型性状进行调查。
1.7 统计分析利用Microsoft Excel软件, 采用t-检验分析转基因大豆株系与对照受体品种对SMV抗性水平的差异显著性。
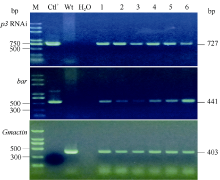
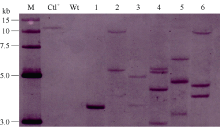
2 结果与分析2.1 转SMV-P3 RNAi大豆植株的获得及分子鉴定 根据GenBank中已登录大豆花叶病毒株系P3基因的保守序列, 选取302 bp片段(2529~2822 nt)作为靶序列, 构建携带该靶序列反向重复片段的RNAi表达载体(图1)。采用农杆菌介导法将SMV-P3RNAi片段导入大豆栽培品种Williams 82和SN9。共转化外植体2876个, 经过分化、筛选和再生, 获得耐6 mg L-1草丁膦再生植株146株。PAT/bar试纸和PCR检测结果表明, 其中83株能扩增出目的条带, 扩增条带分别为727 bp和441 bp, 与预期大小一致, 而对照非转基因植株则没有相应的条带(图2和图3), PCR阳性转化率为2.89%。转基因植株移栽至温室后能够正常开花结实, 且在表型上与对照非转基因植株无明显差异。选取6个PCR阳性转基因大豆植株进行Southern杂交, 杂交探针为bar基因序列。根据载体pTF101-RBSC-P3 T-DNA中Hind III酶切位点的位置, 预期杂交片段最小应为1.5 kb。检测结果表明, 6个转基因大豆植株均可以检测到杂交信号, 而对照非转基因大豆则没有杂交条带出现(图4), 表明外源片段已整合到大豆基因组中。在检测的转基因植株中, 外源T-DNA插入拷贝数为1~4个。
图2
Fig. 2
| Figure OptionViewDownloadNew Window | |
 | 图2 PAT/bar试纸检测Fig. 2 Liberty link strips test |
图3
Fig. 3
| Figure OptionViewDownloadNew Window | |
 | 图3 T0代转基因植株PCR检测 M: DNA标准分子量; Ctl+: 阳性载体质粒pTF101-RBSC-P3; Wt: 未转化植株; 1~6: PAT/bar试纸检测阳性植株B013、B115、B127、B128、B163和B166。Fig. 3 PCR analysis of the T0 transgenic plants M: DNA marker; Ctl+: positive control pTF101-RBSC-P3; Wt: non-transgenic plants; 1-6: transgenic plants B013, B115, B127, B128, B163, and B166 tested by LibertyLink strips. |
图4
Fig. 4
| Figure OptionViewDownloadNew Window | |
 | 图4 T1代转基因大豆Southern杂交检测 M: DNA标准分子量; Ctl+: 阳性载体质粒pTF101-RBSC-P3; Wt: 非转化植株; 1~6: T1代转基因大豆B013、B115、B127、B128、B163和B166。Fig. 4 Southern blot analysis of the T1 transgenic plants M: DNA marker; Ctl+: positive control pTF101-RBSC-P3; Wt: non-transgenic plants; 1-6: T1transgenic plants B013, B115, B127, B128, B163, and B166. |
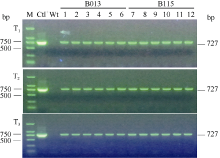
2.2 转基因大豆外源片段遗传稳定性分析除草剂喷施结果表明, 在500 mg L-1 Basta处理条件下, 非转基因大豆很快(7 d左右)出现叶片黄化或枯死现象, 而转基因植株则表现正常(图5)。3个转基因大豆株系B013、B115和B127中, 除草剂抗性植株和敏感植株比例约为3:1, 另外3个转基因大豆株系B128、B163和B166分离比例约为15:1。连续3代PCR检测结果表明, 外源SMV-P3RNAi片段在转基因大豆不同代际间能够稳定遗传(图6)。
图5
Fig. 5
| Figure OptionViewDownloadNew Window | |
 | 图5 T3代转基因大豆耐除草剂鉴定(500 mg L-1Basta)Fig. 5 Herbicide tolerance with 500 mg L-1 of Basta in T3 transgenic lines |
图6
Fig. 6
| Figure OptionViewDownloadNew Window | |
 | 图6 T1~T3代转基因大豆PCR检测 M: DNA标准分子量; Ctl+: 阳性载体质粒pTF101-RBSC-P3; Wt: 非转化植株; 1~6: 转基因大豆株系B013的不同单株; 7~12: 转基因大豆株系B115的不同单株。Fig. 6 PCR of the T1-T3 transgenic lines M: DNA marker; Ctl+: positive control pTF101-RBSC-P3; Wt: non-transgenic plants; 1-6: randomly selected plants of the transgenic lines B013; 7-12: randomly selected plants of the transgenic lines B115. |
2.3 转基因大豆抗SMV鉴定接种SMV SC-3后, 对照非转基因大豆出现叶片严重皱缩、花叶和植株矮化等SMV感染典型症状。转基因大豆植株则表现轻微或是无明显症状(图7和图8), 表明RNAi介导SMV-P3基因沉默显著降低了SMV的侵染及症状发展。也说明SMV-P3基因与SMV感染寄主症状表型密切相关[23, 24]。连续2代抗性鉴定结果表明, 在鉴定的6个株系中, B127和B163对SMV抗性最强, 病情指数降低至4.37~11.11, 另外4个株系对SMV也表现出较高的抗性水平(表1)。6个株系的病情指数均显著低于对照非转基因大豆Williams 82 (病情指数36.81~45.24)和SN9 (病情指数39.80~46.97), 说明转基因大豆显著提高了SMV抗性水平, 且抗性性状能够稳定遗传(表1)。另外, 对转基因大豆田间表型性状调查结果表明, 6个转基因大豆株系在叶形(圆形)、花色(白色)、种皮色(黄色)、种脐色(黑色)、株高、节数、结荚高度、生育期等方面与受体大豆品种没有明显差异(表2)。初步说明, 外源片段的插入并没有对转基因大豆的表型性状造成影响。
图7
Fig. 7
| Figure OptionViewDownloadNew Window | |
 | 图7 摩擦接种SMV SC-3后转基因植株叶片症状表现Fig. 7 Leaf symptom of the transgenic plants inoculated with SMV SC-3 by rub infection |
图8
Fig. 8
| Figure OptionViewDownloadNew Window | |
 | 图8 接种SMV后整株症状表现Fig. 8 Plants of the transgenic lines after inoculation with SMV SC-3 |
表1
Table 1
表1(Table 1)
| 表1 T2和T3代转基因大豆接种SMV SC-3抗性鉴定结果 Table 1 SMV resistance evaluation of the T2 and T3 transgenic lines | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
表2
Table 2
表2(Table 2)
| 表2 转基因大豆株系农艺性状的大田表现 Table 2 Agronomic performance of the transgenic lines in field condition |
3 讨论已有的研究表明, 通过RNAi干扰抑制或阻断SMV关键基因的表达可以显著增强大豆抗SMV能力[3, 18, 19, 35]。在SMV基因组编码的11个不同功能的成熟蛋白中, P3蛋白参与SMV复制和病毒粒子在宿主体内的运动, 并与感染寄主症状表型密切相关[23, 24]。最近的研究结果表明, P3蛋白可能是携带Rsv1和Rsv4抗性位点大豆基因型的无毒决定因子[25, 26, 27, 28, 29]。进一步研究表明, Rsv1编码蛋白可以识别P3蛋白, 并诱发大豆产生Rsv1基因介导的过敏反应(hypersensitive response, HR)[26]。尽管目前尚没有直接证据表明P3蛋白与植物非宿主抗性机制有关, 但也提示P3蛋白在决定大豆宿主范围中发挥着重要的作用, 并与SMV致病性有关[29]。因此, 抑制SMV-P3基因的转录和翻译可以干扰或影响SMV的致病性, 并在一定程度上影响SMV的宿主范围。基于这一思路, 本研究利用宿主RNAi介导SMV-P3基因沉默研究对大豆抗SMV的影响。研究结果表明, 转SMV-P3 RNAi片段大豆在接种SMV SC-3株系后, 其症状表现轻微或无明显症状, 病情指数比受体对照品种显著降低, 表明SMV-P3基因沉默显著降低了SMV侵染或症状发展, 提高了转基因大豆抗SMV的能力。本研究首次报道了RNAi介导SM-P3基因沉默可以显著提高大豆抗SMV水平, 为培育广谱、高抗SMV转基因大豆新品种提供了一条新的路径。
值得注意的是, 尽管抑制SMV-P3基因的表达显著了提高大豆抗SMV能力, 但就本实验的研究结果来看, 转基因大豆对SMV抗性并没有达到免疫水平。这一点与Zhang等[18]和Kim等[19]等研究结果不同。其原因可能与RNAi干扰本身抑制效率有关, 即SMV-P3RNAi并没有100%抑制或降解P3基因的表达; 也可能是抑制P3基因的表达仅对SMV在寄主体内的运动造成干扰, 限制了SMV症状的发展, 但在高水平接种量条件下并没有完全抑制SMV的侵染。不过, 从育种和生产实际角度讲, 低于免疫水平的抗性对于延缓SMV病毒生理小种进化速度, 提高大豆品种持久抗性水平又具有积极的意义。已有的研究表明, 由于位置效应、T-DNA插入拷贝数等因素的影响, 外源基因或片段在转基因植株中易丢失或沉默, 从而导致转基因后代遗传不稳定[36]。本研究结果表明, 转SMV-P3RNAi大豆抗性性状在不同代际间能够稳定遗传。这可能是转基因大豆中外源T-DNA插入拷贝数较少(1~3个)的缘故, 也可能与本研究选用的叶片特异性启动子RBSC2有关。
研究表明, SMV-P3基因在不同SMV株系中存在着较高水平的变异[23, 37]。杨清华[37]对不同来源SMV生理小种P3基因序列同源性和系统发育树分析表明, 来源于中国和韩国的SMV株系P3基因序列同源性相对较高, 核苷酸序列同源性为92.4%~ 98.9%, 氨基酸序列同源性为96%~100%。与来源于美国的SMV株系P3基因序列同源性相对较低, 核苷酸序列同源性为88.50%~99.79%, 氨基酸序列同源性为91.40%~99.86% [37]。本研究根据GenBank中已登录SMV株系P3基因保守序列(2529~2822 nt)设计反向重复片段, 并研究了转SMV-P3RNAi大豆对SMV SC-3株系抗性的影响。由于P3蛋白在决定SMV宿主范围中发挥着重要的作用[23, 24], 转基因大豆对其他SMV株系的抗性是否也能够显著提高, 需要在后续研究中进一步鉴定评价。
4 结论将编码参与SMV运动和影响宿主域范围的P3蛋白基因RNAi片段导入大豆基因组中, 转基因大豆对SMV SC-3抗性水平较受体品种Williams 82和SN9显著提高, 病情指数降低至4.37~18.51, 且抗性能够稳定遗传, 表明RNAi介导SM-P3基因沉默能够显著提高转基因大豆抗SMV水平。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
参考文献View Option
原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | |
| [3] | |
| [4] | |
| [5] | |
| [6] | |
| [7] | |
| [8] | |
| [9] | |
| [10] | |
| [11] | |
| [12] | |
| [13] | |
| [14] | |
| [15] | |
| [16] | |
| [17] | |
| [18] | |
| [19] | |
| [20] | |
| [21] | |
| [22] | |
| [23] | |
| [24] | |
| [25] | |
| [26] | |
| [27] | |
| [28] | |
| [29] | |
| [30] | |
| [31] | |
| [32] | |
| [33] | |
| [34] | |
| [35] | |
| [36] | |
| [37] |
