 , 刘欣, 杜丽璞, 叶兴国, 张增艳
, 刘欣, 杜丽璞, 叶兴国, 张增艳
* 通讯作者(Corresponding author): 张增艳, E-mail:zhangzengyan@caas.cn, Tel: 010-82108781
第一作者联系方式: E-mail:ygkn@163.com
收稿日期:2013-09-16 基金:本研究由国家转基因生物新品种培育科技重大专项(2011ZX08002-001, 2013ZX08002001-004)资助。
摘要抗菌肽是一类具有广谱抗菌性的小分子多肽, 在植物防卫反应中起着重要作用。本研究人工合成了洋葱抗菌肽基因
关键词:洋葱抗菌肽; 转基因小麦; 小麦全蚀病; 抗性
Development and Characterization of
YANG Kun
 , LIU Xin, DU Li-Pu, YE Xing-Guo, ZHANG Zeng-Yan
, LIU Xin, DU Li-Pu, YE Xing-Guo, ZHANG Zeng-Yan
AbstractAntimicrobial peptides, small molecular weight proteins with broad spectrum antimicrobial activity, play important roles in plant defense responses. The open-reading-frame sequence of
Keyword:Antimicrobial peptide from onion; Transgenic wheat; Wheat take-all; Resistance
Show Figures
Show Figures









小麦全蚀病(take-all)又称“黑脚病”和“立枯病”, 是由真菌禾顶囊壳小麦变种( Gaeumannomyces graminisvar. tritici, Ggt)引起的一种世界性小麦土传病害[ 1]。 Ggt以菌丝体形式侵染小麦根部和茎基部, 致使病株的大部分根部和茎基部变黑, 传导组织被堵塞, 分蘖数大大减少, 成穗率降低, 千粒重明显下降、植株枯死, 抽穗后田间出现早枯白穗, 导致减产达40%~60%[ 1, 2], 给小麦生产造成极大威胁。选育和推广抗病小麦品种是控制全蚀病最经济、有效和安全的措施。目前, 生产上大面积推广品种、高代品系对全蚀病的抗性普遍较差, 全蚀病的常规抗病育种进展缓慢。因此, 发掘、利用重要的抗病相关基因, 开展抗全蚀病的小麦基因工程育种非常重要。
抗菌肽(antimicrobial peptides, AMPs)是一类带有正电荷、具广谱抗菌性的小分子多肽的总称, 广泛存在于细菌、病毒、植物及动物体内[ 3]。目前, 大部分植物抗菌肽是从植物种子中分离的, 可以保护植物组织和种子不受病原菌的侵害[ 4], 如硫堇蛋白、植物防卫素、非特异性脂质转运蛋白等能够在体外有效地抑制细菌和真菌病原物的生长[ 5]。将抗菌肽基因导入农作物, 提高农作物的抗病能力[ 4, 6, 7, 8, 9, 10], 是培育作物抗病新品种的策略之一。洋葱抗菌肽Ace-AMP1是从洋葱( Allium cepa L.)种子中分离的一种富含半胱氨酸的非特异性脂质转运蛋白, 能够显著抑制灰霉菌( Botrytis cinerea)、假单胞菌( Pseudomonas syringae)、链格孢菌( Alternaria brassicola)等12种植物病原真菌的生长[ 11]。Bi等[ 9]研究发现, 将 Ace-AMP1基因导入天竺葵, 可提高转基因植株对灰霉菌的抗性。Li等[ 10]将 Ace-AMP1基因导入月季, 增强了转基因月季植株对 Sphaerotheca pannosa菌的抗性。Patkar等[ 4]获得了转 Ace-AMP1基因水稻, 并对4个转基因株系的T1和T2代植株进行水稻稻瘟病、纹枯病及白叶枯病的抗性鉴定, 表明转 Ace-AMP1基因植株能显著减轻这3种水稻病菌的侵染。Roy-Barman等[ 7]将 Ace-AMP1基因通过基因枪法导入春小麦Bobwhite的幼胚中, 鉴定出3个转基因小麦株系, 对T2代小麦的离体叶段接种白粉病菌( Blumeria graminis f. sp. tritici)的结果表明, 转 Ace-AMP1基因小麦的白粉菌感染率为45%~75%, 而未转基因的Bobwhite的白粉菌感染率为100%, 说明 Ace-AMP1表达可增强转基因小麦对白粉病防御能力。该洋葱抗菌肽是否可以提高转基因小麦对全蚀病的防御功能, 尚未见研究报道。
本课题组为揭示洋葱抗菌肽在抗全蚀病小麦基因工程育种上的应用潜力, 按照小麦偏爱密码子人工合成洋葱抗菌肽基因 AcAMP-sn, 其核苷酸序列与洋葱抗菌肽Ace-AMP1编码序列的一致性为88.2%, 氨基酸序列一致性为100%。将人工合成的洋葱抗菌肽基因 AcAMP-sn构建到单子叶植物高效表达载体上, 通过基因枪介导法转化到小麦品种扬麦18中, 并对转基因小麦T0至T4代植株进行分子检测、表达分析及全蚀病抗性鉴定, 以明确转 AcAMP-sn基因小麦在抗全蚀病小麦基因工程育种上的应用潜力。
1 材料与方法1.1 材料以小麦品种扬麦18作为转基因受体材料, 由江苏里下河农业科学研究所程顺和课题组提供。小麦全蚀病病原菌 Ggt引自西北农林科技大学。单子叶高效表达载体质粒pAHC25由本实验室保存。
1.2 AcAMP-sn基因转化载体的构建 根据1995年Cammue等[ 11]发表的序列和小麦偏爱的密码子, 由北京奥科生物技术公司合成基因 AcAMP-sn, 并在该基因的5′端和3′端分别添加 SmaI和 SacI限制内切酶的酶切位点, 连接到pMD18-T vector (TaKaRa)上构建中间载体pMD18-AcAMP-sn, 并克隆到大肠杆菌细胞中。
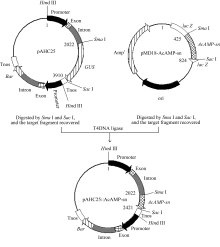
用限制内切酶 Sma I和 SacI (TaKaRa)酶切中间载体质粒pMD18-AcAMP-sn, 回收399 bp目的片段, 同时用 Sma I和 Sac I双酶切单子叶高效组成型表达载体pAHC25, 回收切除 GUS基因的pAHC25载体骨架, 并利用T4连接酶将回收的 AcAMP-sn基因片段与pAHC25载体片段以3∶1摩尔比连接, 构成表达载体pAHC25::AcAMP-sn (图1), 转化到 E. coli菌株Top10感受态细胞中, 通过菌落PCR筛选阳性克隆并测序, 以确定成功构建的转基因表达载体pAHC25::AcAMP-sn。该载体中目标基因 AcAMP-sn的表达受玉米 Ubiqutin启动子控制, Bar基因的表达由另一个 Ubiqutin启动子控制, 可为后续利用Bialaphos筛选转化再生植株提供抗性筛选标记。
图1
Fig. 1
| Figure OptionViewDownloadNew Window | |
 | 图1 pAHC25::AcAMP-sn表达载体的构建Fig. 1 Structure of plant expression vector pAHC25::AcAMP-sn |
1.3 转基因小麦的创制利用徐惠君等[ 12]报道的基因枪介导法, 以人工合成的 AcAMP-sn基因转化扬麦18幼胚愈伤组织1600块, 通过筛选、分化、再生、移栽成苗等步骤, 获得转pAHC25::AcAMP-sn基因小麦扬麦18的T0代植株。从外源 AcAMP-sn基因PCR呈阳性的转基因小麦T0代植株上收获T1代种子, 单株播种T1代转基因植株, 并进行转基因的分子检测和抗病性鉴定; 依此类推。
1.4 小麦DNA提取和外源 AcAMP-sn基因的PCR检测 采用改良酚-氯仿抽提法[ 13], 从转基因小麦及未转基因扬麦18植株0.1 g幼苗叶片中提取基因组DNA, 作为PCR检测的模板。根据转基因载体pAHC25::AcAMP-sn的序列, 设计转基因检测的特异引物UBI-1910U (5′-GCTCTGCCTTCATACGCTA-3′, 位于 Ubiquitin启动子序列上)和AcAMP-sn-2348L (5′-CAGAATGGACGAACAAAGG-3′, 位于 AcAMP-sn基因读码框上), 以转 AcAMP-sn基因小麦DNA为模板, 对T0~T4代转基因植株进行外源目标基因的PCR检测和遗传分析。扩增条件为94℃预变性5 min; 94℃ 30 s, 56℃ 30 s, 72℃ 30 s, 35个循环; 72℃延伸10 min。扩增产物(438 bp)经含2.0% EB的琼脂糖凝胶电泳分析。
1.5 AcAMP-sn基因的转录水平分析 用RNAiso Plus试剂盒(TaKaRa)提取转 AcAMP- sn基因小麦T4代植株根部总RNA, 按RNA PCR Kit (AMV) Ver.3.0 (TaKaRa)说明书合成第一链cDNA。利用ABI PRISMR 7300实时荧光定量RT-PCR仪(ABI, 美国), 以第一链cDNA为模板, 小麦 18S rRNA基因特异引物(F: 5′-GTGACGGGTGACGGA GAATT-3′; R: 5′-GACACTAATGCGCCCGGTAT-3′)为内参照, 调整试材cDNA, 使其浓度一致, 然后用 AcAMP-sn基因的特异引物(F: 5′-CTACGGACTCGG AAGGGC-3′; R: 5′-CCACCAGAATGGACGAACA A-3′)进行半定量RT-PCR分析和实时荧光定量RT-PCR (Q-RT-PCR)。扩增条件为94℃预变性5 min; 94℃ 30 s, 58℃ 30 s, 72℃ 30 s, 共32个循环; 最后72℃延伸5 min, 3次独立重复。Q-RT-PCR分析按照SYBR Premix Ex Taq反应系统(TaKaRa), 反应体系为25 μL, 反应条件为94℃预变性2 min; 然后94℃变性15 s, 60℃退火31 s, 41个循环。对每个反应进行3次独立重复实验。用2-ΔΔCT方法[ 14]计算转基因小麦根部 AcAMP-sn基因的相对表达量(以受体扬麦18为对照)。
1.6 小麦全蚀病抗性鉴定参照菌饼法对转基因株系及受体接种小麦全蚀病菌[ 15], 将接种全蚀病菌的小麦幼苗植于河沙与营养土质量比为1∶2的基质中, 于接种后21 d调查单株的总根长和黑(病)根长。根据Bithell等[ 16]报道的方法, 将全蚀病严重度划分为0~4级, 其黑根面积占总根面积的百分率分别为0、0~10%、10%~30%、30%~60%和60%~100%, 全蚀病病情指数TAI (Take-all index) = (10 × N1 + 30 × N2 + 60 × N3 + 100 × N4) / (N1 + N2 + N3 + N4)。式中, N1~N4为各病级的病株数。采用SPSS19软件进行方差分析。
以小麦 18S rRNA作为内标基因, 利用 Ggt 18S rRNA基因(GenBank登录号为FJ771002)特异性引物(F: 5′-CGAACTCGGTCGTTTAGAGG-3′; R: 5′-GGT ATGTTCACAGGGGTTGG-3′)[ 17], 对转基因植株与受体扬18根中 Ggt转录情况(全蚀病菌的相对含量)进行半定量RT-PCR及Q-RT-PCR分析。半定量RT-PCR扩增条件为94℃预变性5 min; 94℃ 30 s, 58℃ 30 s, 72℃ 30 s, 共32个循环; 最后72℃延伸5 min。Q- RT-PCR分析按照SYBR Premix Ex Taq反应系统(TaKaRa), 反应体系为25 μL, 反应条件为94℃预变性2 min; 然后94℃变性15 s, 60℃退火31 s, 41个循环。对每个反应进行3次独立重复实验。以受体扬麦18为对照, 用2-ΔΔCT方法[ 14]计算转基因小麦根中 Ggt的相对含量。
2 结果与分析2.1 AcAMP-sn基因植物表达载体的构建及转基因小麦的获得 用限制内切酶 SmaI和 SacI分别酶切载体pAHC25质粒DNA和中间载体pMD18-AcAMP-sn, 并回收目的片段, 用T4 DNA连接酶连接回收的载体片段(约7818 bp)和 AcAMP-sn基因片段(399 bp),构建单子叶表达载体pAHC25::AcAMP-sn (8217 bp), 其中 AcAMP-sn基因表达受玉米 Ubiquitin启动子控制。由图1和测序分析结果可知, 构建的重组表达载体pAHC25::AcAMP-sn是正确的, 可用于创制转基因小麦新材料。
通过基因枪将pAHC25::AcAMP-sn轰击小麦推广品种扬麦18的1600个幼胚愈伤组织, 经筛选、分化, 获得89株再生植株, 利用转基因特异性引物进行PCR分析, 检测出阳性植株10株, 转化率为0.62%。
2.2 转 AcAMP-sn基因小麦的PCR检测 利用特异性引物对转 AcAMP-sn基因小麦T0~T4代植株的PCR检测表明, 该引物可以从转 AcAMP-sn基因载体质粒和转 AcAMP-sn基因小麦阳性植株中扩增出438 bp的目的片段。在5个株系(A3、A4、A8、A9和A13)的每代材料中均可以检测到转入的 AcAMP-sn基因(图2-a和2-b), 说明转入的 AcAMP-sn基因在这些转基因小麦株系中能稳定遗传; 在测试的5个抗全蚀病的T4代转基因株系117个植株中均能检测到外源 AcAMP-sn基因, 表明在这5个转基因小麦株系中转入的 AcAMP-sn基因已趋于纯合。
图2
Fig. 2
| Figure OptionViewDownloadNew Window | |
 | 图2 转 AcAMP-sn基因小麦T3(a)及T4(b)代植株的PCR检测结果M: 100 bp DNA ladder; P: 转基因载体质粒pAHC25::AcAMP-sn; CK: H2O; WT: 未转基因扬麦18; 1~15: 转 AcAMP-sn基因T3代植株; A3、A4、A8、A9、A13: 转 AcAMP-sn基因T4代植株。Fig. 2 PCR analyses on T3 (a) and T4 (b) plants of AcAMP-sn transgenic wheatM: 100 bp DNA ladder; P: AcAMP-sn transformed vector plasmid pAHC25::AcAMP-sn as positive control; CK: H2O; WT: untransformed Yangmai 18; 1-15: AcAMP-sn transgenic T3 plants; A3, A4, A8, A9, and A13: AcAMP-sn transgenic T4 plants. |
2.3 转基因植株中 AcAMP-sn基因的转录水平 半定量RT-PCR分析抗病的转基因小麦与未转基因小麦扬麦18根中 AcAMP-sn基因的转录水平表明, 这5个稳定遗传的转基因抗病株系(A3、A4、A8、A9和A13)根中 AcAMP-sn基因的转录水平明显高于受体扬麦18 (图3-a)。进一步利用Q-RT-PCR分析上述5个转基因抗病株系中 AcAMP-sn基因的相对表达量表明, 转基因植株根中 AcAMP-sn基因的相对表达量明显高于未转基因小麦, 但各转基因株系间的表达量有所差异, AcAMP-sn基因在转基因株系A4中的表达量最高、在A9中的表达量最低, 与半定量RT-PCR分析结果基本一致。
图3
Fig. 3
| Figure OptionViewDownloadNew Window | |
 | 图3 转 AcAMP-sn基因小麦中根部 AcAMP-sn基因表达的半定量RT-PCR (a)和Q-RT-PCR (b)分析WT: 未转基因的扬麦18; A3~A13: 转 AcAMP-sn基因株系。 ** 转基因株系与WT有极显著差异( P<0.01)。Fig. 3 Semi-RT-PCR (a) and Q-RT-PCR (b) analysis on AcAMP-sn transcripts in transgenic and wild-type wheat plantsWT: wild-type Yangmai 18; A3-A13: AcAMP-sntransgenic lines. ** Significantly different between AcAMP-sntransgenic line and WT at P<0.01. |
2.4 转基因小麦的全蚀病鉴定对转 AcAMP-sn基因T4代植株的全蚀病抗性鉴定表明, 接种小麦全蚀病菌21 d后未转基因扬麦18出现典型的全蚀病病症(图4), 病根百分比为59.63%, 全蚀病病情指数为72.73; 而5个转 AcAMP-sn基因小麦株系A3、A4、A9、A13和A8的病根百分比为10.46%~19.61%, 全蚀病病情指数分别为18.75、14.29、27.78、28.00和28.75, 均极显著( P<0.01)低于受体扬麦18 (表1)。进一步利用半定量RT-PCR与Q-RT-PCR分析接种 Ggt 21 d的上述转 AcAMP-sn基因小麦根中全蚀病菌的相对含量, 结果表明这些抗病植株中全蚀菌的生物量显著低于未转基因受体, 且 AcAMP-sn基因表达最强的株系(A4)根中全蚀病菌含量最低(图5-a和5-b)。以上结果说明 AcAMP-sn过表达显著增强了转基因小麦对全蚀病的抗性。
图4
Fig. 4
| Figure OptionViewDownloadNew Window | |
 | 图4 接种小麦全蚀病菌21 d后转 AcAMP-sn基因小麦T4代植株与受体扬麦18植株的全蚀病表型WT: 未转基因的扬麦18; A3、A4、A13: 转 AcAMP-sn基因株系。Fig. 4 Phenotypes of take-all in AcAMP-sn transgenic wheat lines and untransformed Yangmai 18 at 21 days after Ggt inoculationWT: untransformed Yangmai 18; A3, A4, and A13: AcAMP-sntransgenic lines. |
表1
Table 1
表1(Table 1)
| 表1 转 AcAMP-sn基因小麦及受体扬麦18的全蚀病鉴定结果 Table 1 Take-all resistance in AcAMP-sn transgenic and control wheat lines |
图5
Fig. 5
| Figure OptionViewDownloadNew Window | |
 | 图5 接种全蚀病菌21 d后转 AcAMP-sn基因与受体小麦扬麦18中该病菌相对丰度的半定量RT-PCR (a)和Q-RT-PCR (b)分析WT为未转基因的扬麦18; 其他为转 AcAMP-sn基因株系。 ** 转基因株系与WT有极显著差异( P<0.01)。Fig. 5 Relative abundance of Ggt in AcAMP-sn transgenic wheat lines relative to that in untransformed Yangmai 18 via semi RT-PCR (a) and Q-RT-PCR (b) at 21 d after inoculationWT is the untransformed Yangmai 18, and other lines are AcAMP-sntransgenic lines. ** Significantly different between AcAMP-sntransgenic line and WT at P<0.01. |
3 讨论Ace-AMP1是从洋葱种子中分离的抗菌肽, 其成熟蛋白与植物非特异性脂质转移蛋白(nsLTP)同源, 76%的氨基酸残基在植物已知的nsLTP中保守[ 11]。研究表明, 植物nsLTP参与植物对细菌、真菌及病毒的防御反应[ 22]。在拟南芥中, nsLTP参与系统获得抗性相关的长距离信号传导过程[ 23]。 Ace-AMP1基因可能参与系统获得抗性(system acquired resistance, SAR), Ace-AMP1过表达使转基因植株体内的SA积累量增加、SAR相关基因上调表达[ 4, 7, 9, 10]。与从萝卜和玉米种子中分离的nsLTP不同, Ace-AMP1无法将脂质体中的磷脂分子转移到线粒体中, 其抗菌机制尚无定论[ 11]。Roy-Barman等[ 7]通过基因枪法将 Ace-AMP1基因导入春小麦Bobwhite中, 通过对T1和T2代转基因小麦的分子鉴定, 获得3个转基因小麦株系, 虽然转入的 Ace-AMP1基因在一定程度上可以提高转基因小麦对白粉病菌的抗性, 但小麦品种Bobwhite的农艺性状较差, 且 Ace-AMP1赋予的白粉病抗性程度和广谱性均未达到直接利用的程度。该抗性鉴定结果与我们转 Ace-AMP1扬麦16对白粉病抗性分析结果(未发表)一致。
小麦全蚀病是小麦重要病害。目前, 可供直接利用的抗性小麦种质匮乏[ 18, 19], 增加了抗病小麦常规育种的难度。利用基因工程的方法, 将抗病基因导入小麦, 为小麦全蚀病抗源的创制和抗性品系(种)的培育提供了新的解决途径。为了解Ace-AMP1在抗全蚀病小麦基因工程育种上的应用潜力, 本研究按照小麦偏爱密码子人工合成洋葱抗菌肽基因 AcAMP-sn, 其核苷酸序列与洋葱抗菌肽编码序列 Ace-AMP1的一致性为88.2%, 编码氨基酸序列一致性为100%; 并利用转基因的方法将抗菌肽基因 AcAMP-sn导入农艺性状优良的小麦推广品种扬麦18中, 通过对T0~T4代转基因小麦逐代、逐株中外源转基因进行PCR\RT-PCR检测, 选育出 AcAMP-sn基因稳定遗传、表达的转 AcAMP-sn基因小麦株系5个; 接种全蚀病菌对这5个株系进行的苗期全蚀病抗性鉴定表明, 与未转基因扬麦18相比, 这5个转 AcAMP-sn基因小麦株系(新种质)的全蚀病抗性显著提高, 且 AcAMP-sn基因表达能够抑制病原菌 Ggt生长, 证明Ace-AMP1在抗全蚀病小麦基因工程育种上具有一定的应用潜力。然而, 由于实验条件所限, 尚未对这5个转 AcAMP-sn基因小麦株系在全蚀病圃进行成熟抗性鉴定, 今后有条件可以进一步开展成熟抗性鉴定。
4 结论通过人工合成基因、基因枪介导法转化小麦扬麦18、对转基因小麦逐代逐株分子检测以及抗病性鉴定, 创制、选育出 AcAMP-sn基因能够稳定遗传、表达、全蚀病抗性提高的转基因小麦新种质5份。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
参考文献View Option
原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | |
| [3] | |
| [4] | |
| [5] | |
| [6] | |
| [7] | |
| [8] | |
| [9] | |
| [10] | |
| [11] | |
| [12] | |
| [13] | |
| [14] | |
| [15] | |
| [16] | |
| [17] | |
| [18] | |
| [19] | |
| [20] | |
| [21] | |
| [22] | |
| [23] |
