 , 陈明
, 陈明 , 徐兆师
, 徐兆师 , 马有志
, 马有志* 通讯作者(Corresponding authors): 马庆, E-mail:qingma66@163.com; 陈明, E-mail:chenming02@caas.cn
第一作者联系方式: E-mail:suruibo19@163.com, Tel: 010-82108750
收稿日期:2013-09-16 基金:本研究由国家高技术研究发展计划(863计划)项目(2012AA10A309)和国家转基因生物新品种培育科技重大专项(2013ZX08002-002)资助。
摘要采用最小表达框技术转化植物可以规避由骨架序列引起的安全风险。核基质结合区序列SAR (scaffold attachment region)可作为边界元件与核基质结合阻挡转基因片段邻近染色质区的作用与影响, 提高外源基因稳定性。本研究在最小表达框序列两端添加SAR序列, 提高小麦最小表达框转基因表达的稳定性, 提高转化基因的表达效率。首先, 以
关键词:SAR序列; 最小表达框转化法; DREB类基因; 基因枪转化法
Improvement of Minimal Gene Cassette Expression Stability by Scaffold Attachment Region (SAR) Sequence in Wheat Transformation
SU Rui-Bo
 , CHEN Ming
, CHEN Ming , XU Zhao-Shi
, XU Zhao-Shi , MA You-Zhi
, MA You-ZhiAbstractThe minimal expression cassette only containing the promoter, coding sequence and terminator sequence is transformed into plants genome, which will reduce the security risks that may be caused by the vector skeleton sequence. Scaffold attachment region (SAR) combining with the nuclear matrix to separate transformed DNA fragment with adjacent genome sequence, which block the influence of the neighboring chromatins and improve the stability of exogenous gene. In this study, a new vector was constructed with the minimal expression cassette flanked with SAR sequences. This vector was used in wheat transformation by micro-particle bombardment aiming at improving stability of exogenous gene expression.
Keyword:SAR sequence; Transformation with minimal expression cassette; DREB-like gene; Gene gun-mediated transformation
Show Figures
Show Figures











基因枪转化法具有转化效率高、无宿主限制、简单易操作等优点, 在小麦遗传转化中得到广泛应用。传统的小麦基因枪转化法多以环形质粒DNA转化为主, 这种方法可能会导致载体骨架序列伴随目的基因同时整合进植物基因组。转化中所使用的环形质粒载体通常含有一些选择标记基因, 如抗生素抗性基因。这些包含抗生素抗性基因的载体骨架序列的整合, 给转基因植物带来了极大的安全隐患。Barro等[ 1]和Cannell等[ 2]分别对基因枪转化获得的6株转基因阳性植株进行Southern-blot分析, 发现这些阳性植株的子二代中各有3株带有氨苄抗性基因 bla。此外, 载体骨架序列中会存在一些潜在的重组热点序列, 引起外源基因不同程度的重排、断裂, 导致遗传不稳定[ 2]; 质粒的复制原点会促进复制介导的基因重排[ 3]; 载体骨架序列中富含A-T的序列会导致DNA片段的多聚化, 从而引起外源基因的多拷贝插入[ 4]。
鉴于环状质粒的载体骨架序列在遗传转化中存在的问题, Fu等[ 5]用包含启动子、编码框和终止子的DNA最小表达框轰击水稻愈伤组织并获得阳性植株, Southern-blot结果显示转基因植株含1~2个外源基因拷贝, 转基因引起的重排反应频率较低。姚琴等[ 6]利用基因枪法进行小麦最小表达框转化, 虽获得了阳性植株, 但没有进一步验证目标基因表达的稳定性。目前用最小表达框转化法获得转基因小麦的成功报道还很少, 我们也发现最小表达框转化法转化小麦时存在转化基因表达稳定性差的问题。为了推进该转化技术的应用, 提高转化基因的表达效率及稳定性是关键。
核基质结合区SAR (scaffold attachment region)可作为DNA的边界元件与核基质结合, 使两个SAR之间的基因片段被界定成一个独立的染色质环状结构, 从而阻挡邻近染色质区的作用与影响, 提高外源基因的稳定性[ 7, 8]。现已从真核蓝藻、酵母、动物、植物及人类中分离出多种SAR序列, 并对其功能进行了验证。采用来自真核蓝藻SAR序列构建的载体, 能够提高转基因的表达水平, 增强外源基因表达的稳定性[ 8]。从酵母中扩增到SAR序列, 并在最小表达框一端或两端加上SAR序列, 采用基因枪法转化烟草悬浮细胞, 结果加SAR序列能提高目的基因的表达, 而且两端加SAR序列比对照(无SAR序列的最小表达框)目的基因的表达量高20倍以上[ 9]。利用来自烟草中的SAR序列, 同样可以增强目的基因的表达[ 10]。
本研究通过在最小表达框两端加SAR序列, 改进了小麦最小表达框转化技术。通过转化 GUS报告基因比较了不加SAR序列的载体与加SAR载体的转化效率及 GUS基因的表达稳定性。并且采用加SAR序列的载体转化抗逆相关基因 GmDREB3, 通过PCR的方法鉴定转基因小麦中插入片段的完整性, 利用real-time PCR方法估算转化基因的拷贝数。结果证实我们改进的最小表达框转化技术可以提高小麦基因枪转化效率, 提高目的基因在转基因小麦中的表达效率及遗传稳定性, 且片段完整性良好。改进的最小表达框转化技术为获得安全转基因小麦新品种、新种质提供了新思路。
1 材料与方法1.1 植物材料烟草( Nicotiana tobacumcv. W38)种植于中国农业科学院作物科学研究所人工气候温室。以科农199为受体的转 GUS基因小麦和以济麦22为受体的转 GmDREB3基因小麦种植于中国农业科学院作物科学研究所可控温室。
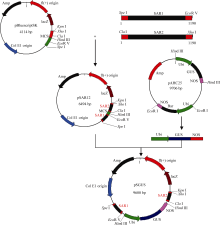
1.2 最小表达框表达载体的构建以烟草W38基因组DNA为模板, 设计引物(表1)扩增SAR序列, 反应条件为94℃ 5 min; 94℃ 30 s, 57℃ 30 s, 72℃ 2 min, 循环35次; 72℃ 10 min。分别将2个SAR序列插入pBluescriptSK载体多克隆位点的5′和3′端, 构建重组载体pSAR12。用 Hind III单酶切pAHC25-GUS回收 GUS基因全长片段, 插入pSAR12构建表达载体pSGUS (图1)。为了转化抗逆相关基因, 设计引物(表1)扩增大豆转录因子基因 GmDREB3, 反应条件为94℃ 5 min; 94℃ 30 s, 58℃ 30 s, 72℃ 50 s, 循环35次; 72℃ 10 min。用 GmDREB3替换pSGUS中的 GUS基因, 构建表达载体pSHGmDREB3。对构建完成的载体均进行酶切验证和测序验证。
表1
Table 1
表1(Table 1)
| 表1 目的基因片段及其对应扩增引物序列 Table 1 Target gene fragment and its corresponding amplification primers |
图1
Fig. 1
| Figure OptionViewDownloadNew Window | |
 | 图1 pSGUS表达载体构建Fig. 1 pSGUS expression vector construction |
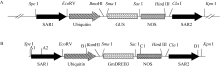
1.3 基因枪法转化小麦用 SpeI和 KpnI双酶切pSGUS质粒和pSHGmDREB3质粒, 分别回收6.6 kb包含 SAR序列、启动子、 GUS报告基因、终止子和 SAR序列的线性片段和5.8 kb包含 SAR序列、启动子、 GmDREB3基因、终止子和 SAR序列的线性片段(图2); 用 EcoR I单酶切pAHC25-Bar质粒, 回收2.89 kb包含启动子、 Bar筛选基因和终止子的线性片段; 用 Hind III单酶切pAHC25-GUS, 回收4.3 kb包含启动子、 GUS基因、终止子的线性片段。将上述胶回收的线性片段产物浓缩为1 μg μL-1, -80℃储存备用。
图2
Fig. 2
| Figure OptionViewDownloadNew Window | |
 | 图2 pSGUS (A)和pSHGmDREB3 (B)表达载体的结构Fig. 2 Structure of pSGUS (A) and pSHGmDREB3 (B) vectors |
将制备好的含 SAR序列和不含 SAR序列的 GUS线性片段和 GmDREB3线性片段, 分别与不含 SAR序列的 Bar基因线性片段按1∶1 ( V/ V)混合制弹, 进行基因枪共转化。其中 GUS基因转化的受体为科农199, GmDREB3基因转化的受体为济麦22, 金粉用量为每枪45 μg, DNA用量为每枪1 μg。轰击后将愈伤组织在原培养基上培养16~18 h, 转移到SD培养基(MS培养基的无机盐成分中添加VB1 1 mg L-1、天门冬酰胺150 mg L-1和2,4-D 2.0 mg L-1)上恢复培养2周。将恢复培养的愈伤组织再转移到筛选分化培养基上培养至愈伤组织分化出幼苗, 将苗转移到生根培养基上继续筛选3次。壮苗后, 待植株生长到15 cm左右时移栽到花盆中, 在可控温室中培养生长, 生长光照设置长日照为 16 h光照/8 h黑暗, 短日照为 10 h光照/14 h黑暗, 室温度设置为18℃。
1.4 转 GUS基因小麦的验证 取转 GUS基因的小麦单株叶片约0.1 g, 采用植物基因组DNA提取试剂盒(TIANGEN, 北京)提取基因组DNA, 并进行 GUS基因的PCR检测。所用检测引物为t-GUS-F/R (表1), 反应条件为94℃ 5 min; 94℃ 30 s, 57℃ 30 s, 72℃ 30 s, 循环35次; 72℃ 10 min。取PCR检测为阳性的转基因植株的根、胚乳、小花置于GUS染色液, 在37℃恒温箱中静置8 h后用75%酒精漂洗5次, 用VHX-600 (Japan)和Nikon8700 (Japan)显微镜观察并照相。
1.5 转 GmDREB3基因小麦的验证及插入片段拷贝数分析 取T0代转 GmDREB3基因的小麦单株叶片(约0.1 g)的基因组DNA进行 GmDREB3基因PCR检测, 检测引物为GmDREB3-F2/R2 (表1), PCR反应条件为94℃ 5 min; 94℃ 30 s, 56℃ 30 s, 72℃ 25 s, 循环35次; 72℃ 10 min。从PCR检测为阳性的植株中随机选取6株, 按照植物总RNA提取试剂盒(TIANGEN, 北京)的操作说明, 提取转基因阳性小麦单株的总RNA, 取5 μg总RNA反转录cDNA第一链。利用RT-PCR的方法检测目的基因 GmDREB3能否正常转录, 引物为GmDREB3-F1/R1 (表1), 反应条件为94℃ 4 min; 94℃ 30 s, 54℃ 30 s, 72℃ 50 s, 循环35次; 72℃ 10 min。为确认含SAR序列的 GmDREB3基因最小表达框在小麦基因组中的插入完整性, 将全长片段(5.3 kb)分为两段, 即5′端- SAR1+启动子(ubi)+GmDREB3和3′端-GmDREB3基因+NOS+SAR2, 对这两段分别进行扩增分析。用A1/C1或A2/C1引物(表1和图2-B)进行最小表达框5′端完整性检测, 用B1/D1引物(表1和图2-B)进行最小表达框3′端完整性检测。PCR反应条件为94℃ 8 min; 94℃ 45 s, 55℃ 30 s, 72℃ 3 min, 循环35次; 72℃ 10 min。
以小麦 puroindoline-b基因为内源参照基因, 利用real-time PCR方法[ 11]确定转基因阳性植株中外源基因 GmDREB3的拷贝数。目的基因检测用引物为t3-GmDREB3 (表1), 内标基因检测用引物为puroin- doline-b (表1)。反应体系包括正反向引物各0.5 μL、10 μL 2×SuperReal PreMix Plus、1 μL 50×Rox Reference Dye (TIANGEN, 北京)、2 μL cDNA模板和6 μL无RNase ddH2O。将配置好的PCR体系置ABI 7500荧光定量PCR仪上, PCR反应程序为95℃ 15 min, 95℃ 15 s, 60℃ 20 s, 72℃ 32 s, 共40个循环。
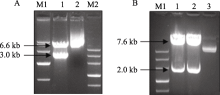
2 结果与分析2.1 pSGUS和pSHGmDREB3载体构建用 KpnI和 SpeI双酶切pSGUS表达载体, 可得到3.0 kb载体骨架片段和6.6 kb目的片段(图3-A), 结果表明pSGUS表达载体构建正确。用 SacI和 BamH I双酶切pSGUS表达载体, 可得到2.0 kb的 GUS基因片段和7.6 kb载体骨架片段(图3-B), 表明pSGUS表达载体结构正确, 且可以通过 SacI和 BamH I双酶切位点将 GUS基因替换为 GmDREB3, 构建pSHGmDREB3表达载体。对pSGUS和pSHGmDREB3载体进行测序(结果未显示)验证, 比对分析表明序列完全正确。
图3
Fig. 3
| Figure OptionViewDownloadNew Window | |
 | 图3 pSGUS重组质粒的酶切A: KpnI和 SpeI双酶切pSGUS表达载体。M1: marker DL10000; 1: pSGUS质粒经 KpnI和 SpeI酶切; 2: pSGUS质粒; M2: marker III。B: SacI和 BamH I双酶切pSGUS表达载体。M: marker III; 1~2: pSGUS质粒经 SacI和 BamH I酶切; 3: pSGUS质粒。Fig. 3 Digested vector of recombinant plasmid pSGUSA: Vector pSGUS digested by KpnI and SpeI. M1: marker DL10000; 1: pSGUS digested by KpnI and SpeI; 2: pSGUS; M2: marker III. B: Vector pSGUS digested by SacI and BamH I. M: marker III; 1-2: pSGUS digested by SacI and BamH I; 3: pSGUS. |
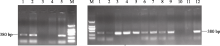
2.2 转 GUS基因小麦PCR检测 将含SAR序列的 GUS基因线性片段(pSGUS片段)与不含SAR序列的 GUS基因线性片段分别与 Bar基因线性片段共转化受体小麦科农199, 经过筛选后获得再生植株(图4-A), 再经壮苗培养(图4-B)和移栽(图3-C), 最终分别获得40株含SAR和31株不含SAR的再生植株。对T0的转基因植株进行PCR检测, 结果31株不含SAR的转 GUS再生植株中, 5株为阳性, 进一步对来自这5个PCR阳性株系的10个T1代单株进行PCR检测, 未获得阳性植株; 而含SAR的40株T0代再生植株中, PCR检测有16株阳性植株, 从来自其中4个随机植株的18个T1代单株中检测出15株PCR阳性植株(图5和表2)。
图4
Fig. 4
| Figure OptionViewDownloadNew Window | |
 | 图4 GUS和pSGUS线性片段小麦转化过程A: 培养; B: 壮苗; C: 移栽。A: regeneration seedlings; B: enhancing seedlings; C: transplanted seedlings.Fig. 4 The transformation process of pSGUS and GUS linear fragments |
图5
Fig. 5
| Figure OptionViewDownloadNew Window | |
 | 图5 T0代转 GUS和pSGUS线性片段小麦PCR检测结果A: 转 GUS小麦PCR检测结果。1~2: 阳性植株; 3: 阴性对照; 4: 受体; 5: 质粒对照; M: DL2000。B: 转pSGUS小麦PCR检测结果; M: DL2000; 1~9阳性植株; 10: 阴性对照; 11: 受体; 12: 质粒对照。Fig. 5 Results of PCR detection of transgenic wheat plants transformed with GUS and pSGUS fragmentsA: PCR detection of GUS transgenic plants. 1-2: positive plants; 3: negative plant; 4: wild type; 5: plasmid control; M: DL2000. B: PCR detection of pSGUS transgenic plants. M: DL2000; 1-9: positive plants; 10: negative plant; 11: wild type; 12: plasmid control. |
表2
Table 2
表2(Table 2)
| 表2 T0代和T1代转 GUS片段和pSGUS片段小麦植株PCR检测结果 Table 2 Testing results of T0and T1generation transgenic wheat plants transformed with GUS and pSGUS fragments | ||||||||||||||||||||||||||||||||||||||
2.3 GUS基因表达稳定性 对T0代5株PCR检测为阳性的 GUS基因(不含SAR序列)植株进行GUS染色, 2株呈阳性; 对来自T0代5个PCR阳性株系的10个T1代单株进行GUS染色, 未获得阳性植株。对含SAR序列的T0代16株阳性植株进行GUS染色, 获得15株阳性植株, 根部染色结果表明染色部位主要集中在根的幼嫩组织(图6-A)。随机选取来自T0代4个PCR阳性株系的18个T1代单株进行GUS染色, 其中15株为阳性植株。小麦胚乳和小花的染色结果表明胚乳全部染成蓝色(图6-B), 小花和颖壳基部均被染成蓝色(图6-C)。结果表明含SAR序列的 GUS片段比不含SAR序列的 GUS片段转化效率高, 而且含SAR序列的 GUS转基因小麦的遗传更加稳定。
受体为科农199。The recipient is Kenong 199.
2.4 转 GmDREB3基因小麦PCR检测及插入片段完整性检测 含SAR序列的 GmDREB3基因线性片段共轰击6045个幼胚(济麦22), 获得130株再生植株, PCR检测获得30株阳性植株(图7-A), 转化率0.50%。随机选取6株阳性植株进行RT-PCR分析, 其中5株为RT-PCR阳性(图7-B)。进一步分析RT-PCR呈阳性的5个株系插入片段的完整性, 引物A1/C1未检测到5′端全长序列, 引物A2/C1 (A2引物距SAR序列起始位点200 bp)检测出4株中扩增出3.6 kb的线性片段(图7-C)。用引物B1/D1对这4株扩增插入片段的3′端, 均检测到2.1 kb的线性片段(图7-D)。对5′端和3′端扩增出的线性片段测序验证, 结果经比对分析后均正确, 表明插入小麦基因组中的最小表达框的线性片段完整性良好。插入片段完整性检测结果(表3)显示, 5′末端SAR序列外侧有200 bp的缺失, 但不影响目的基因的稳定表达。
图6
Fig. 6
| Figure OptionViewDownloadNew Window | |
 | 图6 转pSGUS片段小麦不同部位不同时期 GUS报告基因表达检测结果A: T0代转基因小麦根部染色; B: T1代转基因小麦胚乳染色; C: T1代转基因小麦小花染色。Fig. 6 Staining results of GUS reporter gene in different parts or at different stages of pSGUS transgenic wheat plantsA: root staining of T0 plants; B: endosperm staining of T1 plants; C: flowers staining of T1 plants. |
图7
Fig. 7
| Figure OptionViewDownloadNew Window | |
 | 图7 转 GmDREB3基因小麦分子检测结果A: PCR检测。M: DL2000; 1~6: 阳性株系; 7: 水对照; 8: 受体对照; 9: 质粒对照。B: RT-PCR检测。M: DL2000; 1~5: 阳性株系; 6: 水对照; 7: 受体对照; 8: 质粒对照。C: 插入片段5′端完整性检测。M: marker III; 1~4: 阳性株系; 5: 水对照; 6: 受体对照; 7: 质粒对照。D: 插入片段3′端完整性检测。M: marker III; 1~4: 阳性株系; 5: 水对照; 6: 受体对照; 7: 质粒对照。Fig. 7 Molecular detection results of GmDREB3 transgenic wheatA: PCR result. M: DL2000; 1-6: positive lines; 7: water control; 8: Jimai 22; 9: plasmid control. B: RT-PCR result. M: DL2000; 1-5: positive lines; 6: water control; 7: Jimai 22; 8: plasmid control. C: PCR result of 5′ terminal 5′ insertion fragment. M: Marker III; 1-4: positive lines; 5: water control; 6: Jimai 22; 7: plasmid control. D: PCR result of 3′ terminal of insertion fragment. M: marker III; 1-4: positive lines; 5: water control; 6: Jimai 22; 7: plasmid control. |
表3
Table 3
表3(Table 3)
| 表3 转 GmDREB3小麦插入片段完整性检测结果 Table 3 Test of inserting fragments in GmDREB3 transgenic plants |
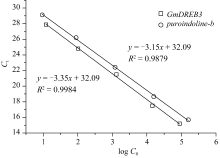
2.5 Real-time PCR确定转 GmDREB3基因小麦的拷贝数 利用定量PCR的方法确定转 GmDREB3基因小麦的拷贝数, 目的基因 GmDREB3的标准曲线相关性较高(图8), 起始模板数的对数值(lg C0,GmDREB3)与 Ct值之间的相关方程为 Ct = -3.35 × lg C0,GmDREB3 31.60。内标基因 puroindoline-b的标准曲线(图8) R2高达0.9879, 起始模板数的对数值(lg C0,pur)与 Ct值之间的相关方程为 Ct = -3.15 × log C0,pur + 32.09。选取6株阳性植株提取基因组DNA, 进行定量PCR分析。将获得的 GmDREB3和 puroindoline-b的 Ct值(表5)及各自标准曲线的斜率( S)和截距( I)代入方程
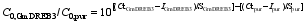
计算出样品起始模板数的比值[ 11]。由于小麦中 puroindoline-b基因为2拷贝, 用 GmDREB3起始模板数与 puroindoline-b起始模板数的比值乘以2, 得到转基因植株中目的基因 GmDREB3的拷贝数。结果显示, 转 GmDREB3基因的6个株系中, 目的基因的拷贝数为1~3 (表4)。
图8
Fig. 8
| Figure OptionViewDownloadNew Window | |
 | 图8 GmDREB3基因和 puroindoline-b基因的标准曲线 Ct: 阈值循环数; lg C0: 起始模板浓度对数。Fig. 8 Standard curves of GmDREB3 and puroindoline-bgenes Ct: Threshold value of cycle number; lg C0: Logarithm value of initial template concentration. |
表4
Table 4
表4(Table 4)
| 表4 GmDREB3和 puroindoline-b基因的 Ct值及 GmDREB3 基因拷贝数估算 Table 4 Ct values of GmDREB3 and puroindoline-b genes and estimation of copy number of GmDREB3gene in transgenic lines | |||||||||||||||||||||||||||||||||||||
3 讨论随着转基因植物产业化的发展, 人们对转基因作物的安全性更加关注, Fu等[ 5]成功地用含目的基因的线性表达框转化水稻, 获得了不含载体骨架序列的转基因植株。Nguyen等[ 12]共转化 CRY1AC基因、 GNA基因和标记基因 hpt的线性表达框, 发现与转化环状质粒对照的转基因植株相比, 转化线性表达框的转基因植株可以积累更多的目标蛋白, 但二者在效率上没有显著差异。Fu等[ 5]和Nguyen等[ 12]都发现, 转化线性表达框的转基因植株都含有1~2个目的基因拷贝, 基因重排现象发生较少。Wang等[ 8]利用电击法将含SAR序列的 CAT基因环形质粒转化到藻类细胞中, 阳性克隆中CAT的表达水平比不含SAR序列的克隆提高4.5倍, 且CAT表达水平的提高并不依赖于 CAT基因拷贝数的升高。Vain等[ 13]分析转GUS质粒T0和T1代阳性植株中GUS的表达活性, 发现含SAR序列的转基因水稻植株中, GUS的表达更稳定, 这也说明SAR序列可以提高环形质粒转基因植株中目的基因的表达稳定性。本研究发现, 含SAR序列的 GUS基因线性表达框在T0代的转化率为1.86%, 高于0.49%的不含SAR序列的T0代转化率(表2)。跟踪检测T1代GUS的表达活性, 发现不含SAR序列的阳性植株均检测不到 GUS基因的表达活性, 含SAR序列阳性植株在T1代仍有83.33%植株显示为GUS阳性(表2和图6)。这表明SAR序列不仅可以提高基因枪法的转化率, 也可以提高最小表达框转基因阳性植株中目的基因的稳定表达。前人研究认为2个SAR之间可以形成位置效应, 提高目的基因表达的稳定性[ 7]。针对这个特性在最小表达框两端分别加上SAR序列, 可以阻止邻近染色质区的顺式调控元件对环内基因影响, 而提高目的基因的稳定。这种结构可以克服植物基因组对外来基因的影响, 因为SAR使外源基因形成独立的转录单位, 使其不受周围基因的影响, 避免位置效应引起的基因失活[ 14, 15]。van der Geest等[ 16]发现, SAR能提高转基因烟草植株中目的基因的总表达水平, 同时可以降低转基因系间的差异, 而基因枪转化时, 转基因在受体中常以多拷贝形式存在, 整合模式较为复杂, 更易导致转基因同源依赖性沉默, SAR有可能通过减弱基因沉默所造成的负影响来提高转基因的表达效率, 促进目的基因的稳定遗传和稳定表达。鉴于SAR序列在植物遗传转化中的优点, 本研究将含SAR序列的 GmDREB3基因转化到济麦22中, 分析T1代阳性植株中目的基因 GmDREB3的转录水平和其线性表达框的完整性, 发现阳性植株中 GmDREB3均可以正常转录, 且除线性表达框5′末端的200 bp丢失外, 其余序列均完整存在。分析转 GmDREB3基因阳性植株的拷贝数, 发现阳性植株均为1~3个拷贝(表4)。此结果证明如果采用不带有SAR序列的线性表达框进行基因枪转化, 线性表达框两端的序列可能会在转化过程中受到损伤, 这种损伤可能会影响转化片段的基因表达及遗传稳定性, 这可能就是我们转化不带有SAR序列的GUS表达框比转化带有SAR序列的表达框的遗传稳定性差的原因, 同时, 带有SAR序列的GUS最小表达框转化效率高于不带有SAR序列的表达框转化效率可能也与线性表达框的缺失相关, 具体的原因需要进一步鉴定。Allen等[ 17]发现, 烟草的SAR序列与酵母的SAR序列相比, 具有更强的结合核基质的亲和力。本研究中选取的是烟草的SAR序列, 是否存在更好的SAR序列资源, 需进一步研究。
4 结论以科农199为受体, 与不带有SAR序列的最小表达框比较, 加 SAR序列的最小表达框pSGUS转化效率明显高, 并且所转化线性片段中 GUS基因可以稳定遗传和表达。以大豆抗旱基因 GmDREB3为目的基因进行转化, 分子检测结果表明目的基因可以稳定表达, 而且插入片段基本完整, 转 GmDREB3基因株系含有较低拷贝数(1~3个)。因而认为改进的最小表达框转化技术与原技术相比可以提高小麦基因枪法转化效率, 减少目的基因的拷贝数, 提高目的基因表达和遗传的稳定性, 增加插入片段的完整性。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
参考文献View Option
原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | |
| [3] | |
| [4] | |
| [5] | |
| [6] | |
| [7] | |
| [8] | |
| [9] | |
| [10] | |
| [11] | |
| [12] | |
| [13] | |
| [14] | |
| [15] | |
| [16] | |
| [17] |
