
 , 熊海容3, 胡晓敏3
, 熊海容3, 胡晓敏3

1. 中国科学院武汉病毒研究所, 湖北 武汉 430071;
2. 中国科学院大学, 北京 100039;
3. 中南民族大学 生命科学学院, 湖北 武汉 430074
收稿日期:2020-09-08;接收日期:2020-12-21
基金项目:湖北省重点专项(No. 2018ABA093),中南民族大学中央专项(No. CZZ20001),中南民族大学人才引进项目(No. YZZ19016) 资助
摘要:噬菌体通过受体结合蛋白(Receptor binding protein,RBP) 结合到细菌表面,其过程需要复杂的原子结构的参与和构象改变。针对噬菌体侵染,细菌发展了多种抗性机制,同时,噬菌体也进化出多种逃逸宿主抗性的机制。对噬菌体与细菌间“吸附-抗吸附-逃逸过程”的探索有助于我们理解噬菌体与细菌共进化的过程,对科学发展噬菌体治疗技术以及噬菌体的生物应用技术具有重要意义。本文概述了噬菌体吸附相关蛋白及吸附发生过程、基于RBP改变的噬菌体逃逸机制和RBP相关的生物技术研究进展。
关键词:噬菌体吸附受体结合蛋白噬菌体逃逸机制噬菌体-宿主互作
Advances of phage receptor binding proteins
Jiahui Sun1,2, Peiling Geng3, Xiaofu Wan1,2, Zhiming Yuan1

 , Hairong Xiong3, Xiaomin Hu3
, Hairong Xiong3, Xiaomin Hu3

1. Wuhan Institute of Virology, Chinese Academy of Sciences, Wuhan 430071, Hubei, China;
2. University of Chinese Academy of Sciences, Beijing 100039, China;
3. College of Life Science, South-Central University for Nationalities, Wuhan 430074, Hubei, China
Received: September 8, 2020; Accepted: December 21, 2020
Supported by: Hubei Province Innovation Major Program, China (No. 2018ABA093), "Fundamental Research Funds for the Central Universities" (No. CZZ20001), "Talent Introduction Project" of South-Central University for Nationalities, China (No. YZZ19016)
Corresponding author: Zhiming Yuan. Tel: +86-27-87197242; E-mail: yzm@wh.iov.cn;
Xiaomin Hu. Tel: +86-27-67842689; E-mail: xiaomin.hu@mail.scuec.edu.cn.
Abstract: Bacteriophages bind to the bacteria receptor through the receptor binding proteins (RBPs), a process that requires the involvement of complex atomic structures and conformational changes. In response to bacteriophage infection, bacteria have developed a variety of resistance mechanisms, while bacteriophages have also evolved multiple antagonistic mechanisms to escape host resistance. The exploration of the "adsorption-anti adsorption-escape process" between bacteriophages and bacteria helps us better understand the co-evolution process of bacteriophages and bacteria, which is important for the development of phage therapeutic technologies and phage-based biotechnologies. This review summarizes the bacteriophage adsorption related proteins, how bacteriophages escape host resistance based on the RBP alternations, and the recent progress of RBP-related biotechnologies.
Keywords: phageadsorptionreceptor binding proteins (RBPs)phage escape mechanismphage-host interaction
噬菌体是专性感染细菌、放线菌、螺旋体和支原体等微生物的病毒,因其具有繁殖快、特异性高、易改造等特性,不仅可用于生物防控,还可以用于基因编辑、表面展示等技术。近年来,新型抗生素制剂研发缓慢、细菌耐药性问题增加,特别是超强耐药菌的出现,使噬菌体获得了越来越多的关注[1]。明确噬菌体与宿主菌的相互作用位点,对于有效开发和应用噬菌体生物制剂以及发展噬菌体相关技术具有重要意义。
吸附(Adsorption) 是噬菌体侵染的第一步,该过程依赖于噬菌体表面的受体结合蛋白以及细菌细胞表面受体,吸附位点的特异性以及吸附能力影响噬菌体的宿主范围及侵染能力。本文对噬菌体吸附及吸附相关蛋白的研究进展和应用进行了简述。
1 噬菌体吸附1.1 噬菌体吸附结构噬菌体结合到细菌表面所需的结构统称为受体结合蛋白。同一噬菌体可以具有多个RBP,其主要功能是识别细菌特异性受体并与之结合。具有水解功能的RBP能水解细菌表面结构,协助核酸注入宿主菌。根据对噬菌体群体RBP的统计,RBP多为尾部结构中的尾刺、尾丝蛋白及基板;少数不具有尾部结构的噬菌体,衣壳蛋白可作为吸附结构[2]。RBP的结构十分模板化,为保守的N端结构域(Amino-terminal)、中间灵活的铰链区(Linker)、末端可吸附和水解细菌受体的C端结构域(Carboxy-terminal)。图 1展示了代表性的4种噬菌体的RBPs结构和识别位点:1)短尾科噬菌体(Podoviridae) P22通过尾刺蛋白gp9的C端识别细菌外膜脂多糖(Lipopolysaccharide,LPS)。2) 肌尾科噬菌体(Myoviridae) T4通过尾丝,包括6根长尾丝与6根短尾丝,共同介导噬菌体吸附:长尾丝的gp37为受体结合域,可以与细菌LPS或外膜蛋白OmpC通过电荷间作用力可逆结合;短尾丝由gp12编码,不参与噬菌体的宿主特异性,与LPS不可逆结合[3]。3) P2为具有基板吸附模式的长尾科噬菌体(Siphoviridae),其不可逆吸附过程吸附依赖于基板[3]。当尾管蛋白的粘附域将噬菌体粘附于细菌表面后,基板RBP会识别细胞壁(Cell wall,CW) 特定糖基,在Ca2+协同作用下,基板蛋白头部结构由C端向上转为向下,暴露更多RBP位点,同时基板RBP将以尾部蛋白为轴心进行200°的螺旋相变,打开基底底部核酸注入通道[4]。4) 微小噬菌体科(Microviridae)的大肠杆菌噬菌体ΦX174没有尾部结构,其衣壳为对称的二十面体,顶点有12个刺突蛋白(Protein G),刺突在识别细菌LPS后从衣壳解离,暴露衣壳F蛋白(Protein F),F蛋白锚定于细胞壁上并协同引导蛋白(Protein H) 发生构象变化,帮助噬菌体完成DNA注入[5]。
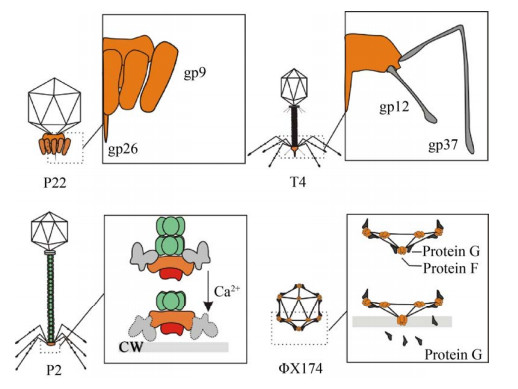 |
| 图 1 四种代表性噬菌体受体结合蛋白的结构(短尾噬菌体科P22;肌尾噬菌体科T4;长尾噬菌体科P2;微小噬菌体科ΦX174) Fig. 1 Structures of RBPs of four bacteriophage represented by Podophage P22, Myophage T4, Siphophage P2, and Microphage ΦX174. |
| 图选项 |
1.2 噬菌体吸附发生过程近年来,通过X射线晶体学以及电镜手段对噬菌体吸附过程的研究表明噬菌体吸附具有复杂的原子水平的细节,且不同类型噬菌体的吸附过程存在差异。T噬菌体是目前吸附过程研究比较深入的噬菌体,被视为最经典的噬菌体吸附模型[6]。T4噬菌体具有可伸缩的尾鞘结构,侵染未发生时长尾丝纤维(Long tail fiber,LTF) 处于回缩状态;当第一个LTF识别到宿主菌LPS或外膜蛋白(OmpC或OmpA) 后,LTF面向细胞表面伸展,并与受体可逆结合,将噬菌体暂时锚定于宿主菌上;在其解离前,第二个LTF将进行伸展并与受体可逆结合;随后噬菌体将重复LTF的“伸展-结合-回缩”过程;当至少2个或3个长尾丝蛋白与细菌可逆结合时,基板将被初步激活并产生构象变化暴露单个短尾丝(Short tail fiber,STF);当STF的gp12与LPS不可逆结合后,基板被进一步激活暴露更多STF并打开核酸释放通道,STFs可将基板拉近细胞膜(Cytoplasmic membrane,CM),噬菌体尾鞘收缩,带动尾管刺入细胞膜(图 2A)[7]。
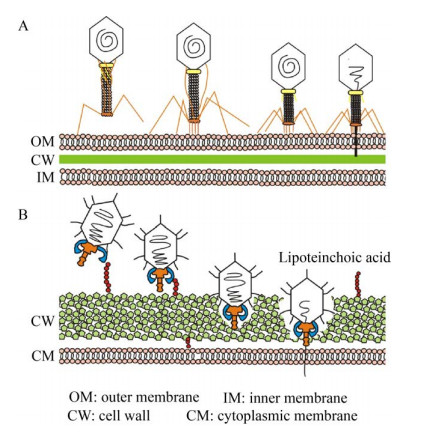 |
| 图 2 T4噬菌体吸附大肠杆菌(A) 及Φ29噬菌体吸附枯草芽孢杆菌(B) 过程 Fig. 2 Schematic models of T4 adsorption to Escherichia coli (A) and Φ29 adsorption to Bacillus subtilis (B). |
| 图选项 |
尽管一些革兰氏阳性菌的RBPs基因组及结构已被解析,但对完整的吸附过程的了解甚少[8]。2017年,Farley等用冷冻电子断层成像术(Cryoelectronic tomography,cryo-ET) 展示了短尾科噬菌体Φ29通过尾部蛋白对壁磷壁酸(Wall teichoic acid,WTA) 和肽聚糖的水解和消化作用克服宿主菌枯草芽孢杆菌肽聚糖层的过程(图 2B)。噬菌体Φ29与宿主菌接触时,尾刺蛋白gp12通过其C端水解酶结构域水解WTA,将噬菌体拉近细菌,并提供能量使病毒方向垂直于细菌表面;衣壳蛋白gp8与头部纤维蛋白gp8.5帮助噬菌体与细菌表面肽聚糖粘附;尾纽(Tail knob) gp13 C端的溶菌酶样结构域可以识别、消化肽聚糖;当噬菌体在肽聚糖层运动时,尾刺蛋白gp12呈现向下的构象变化,促进噬菌体横穿细胞壁;当噬菌体尾部完全嵌入细胞壁,尾部尖端与细胞质膜融合,噬菌体将DNA注入宿主菌[9]。
2 基于RBP改变的噬菌体逃逸机制面对噬菌体的侵染,细菌进化出多种抵抗机制[10],其中大部分抗侵染发生位点是噬菌体吸附阻断[11]。而在自然环境中,噬菌体数量高于细菌数倍,这说明噬菌体也进化出多种逃避细菌抗性的逃逸机制。目前发现的噬菌体逃逸机制主要分为3个方面:识别和适应新的细菌吸附受体、降解受体封阻结构、编码多种RBPs (图 3)。
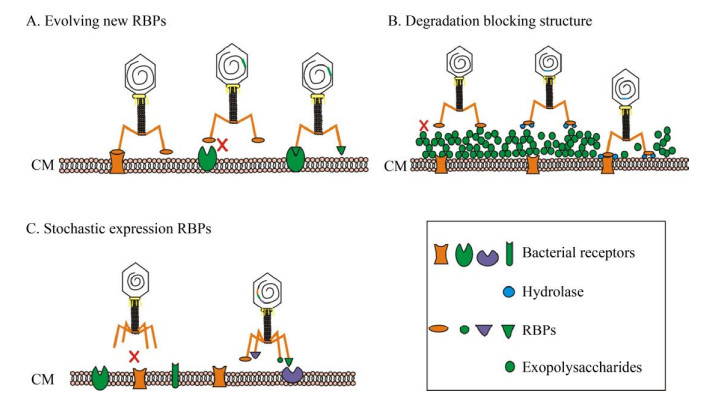 |
| 图 3 基于受体结合蛋白改变的噬菌体的逃逸机制[12] Fig. 3 Escape mechanism of phage based on the modification of RBPs[12]. |
| 图选项 |
2.1 识别和适应新的吸附受体噬菌体可以通过突变RBP编码基因来适应不同受体突变或寻找新的吸附受体(图 3A)。T7和ΦX174的RBPs能根据宿主菌LPS的状态发生适应性突变[13];λ噬菌体的宿主菌吸附受体LamB蛋白表达下调时,噬菌体的gene J在5个位点自发突变,表达新的RBP来识别宿主的表面蛋白OmpF[14]。
2.2 降解受体封阻结构细菌表面受体被胞外多糖(Exopolysaccharides,EPS) 掩盖时,噬菌体通过在RBP末端编码可降解荚膜等结构的水解酶,使细菌受体暴露(图 3B)。大肠杆菌噬菌体K1H、K1G、K1E与K1-5,化脓性链球菌噬菌体H4489A的尾部(尾丝、尾刺等)携带内切酶结构域,可降解透明质酸或糖苷键结构,帮助噬菌体降解荚膜。恶臭假单胞菌噬菌体AF尾刺蛋白(Gp19) C端具有降解EPS的结构域。黏液型铜绿假单胞菌胞外包裹大量具有保护功能的O-乙酰化藻酸,其噬菌体PT-6编码的裂解酶可以水解这些多糖,能有效降低细胞外藻酸盐的黏度和分子量[15]。
具有内切酶活性的RBP的C端结构域编码基因高度保守。例如,AF与T7样噬菌体Φ15尾刺蛋白都具有降解EPS的能力,虽然噬菌体的相似性低,但编码水解活性的区域基因相似度极高;K1E、K1-5与噬菌体63D的RBP末端编码有内切酶,其氨基酸序列相似性高达96%[16]。这暗示噬菌体可能通过水平基因转移获得这种内切酶的特性,这也可以为宿主-噬菌体群体互作和病原菌防控提供一定的线索。
2.3 编码不同受体特异性的RBPs噬菌体可以通过突变或者编码多种不同受体特异性的RBPs来增加侵染几率、扩大宿主谱(图 3C)[17]。肌尾噬菌体Φ92和PVP-SE1至少携带5种不同类型的RBPs,其宿主菌除了大肠杆菌外还有多种沙门氏菌[18]。从Φ92和PVP-SE1这类噬菌体同源蛋白以及相似矩阵上可知,该类噬菌体(rv5样噬菌体) 具有种间和种内基因重组以改变宿主谱的能力。博德特氏菌噬菌体BPP能感染Bvg+相博尔特菌,并能通过自发突变产生变体BMP识别Bvg–相博尔特菌。该过程是由BPP基因组中的差异性反转录因子(Diversity- generating retroelements,DGRs) 与mtd基因簇共同介导的,该系统帮助噬菌体通过“个体微进化”识别宿主相变过程中不同的表面受体;进化的核心是逆转录酶介导的mtd中两个重复序列基因的突变,DGRs通过“转录-逆转录-整合”的方式将供体模板区(Template region,TR) 的序列引入到mtd 3′末端的可变区(Variable region,VR) 中,引起RBP蛋白末端的变异,识别Bvg–相宿主菌[19]。DGRs基因簇在噬菌体及细菌中均被发现,可能与噬菌体的环境适应以及宿主谱的扩大相关。
3 噬菌体RBP与噬菌体生物技术发展特异性高是噬菌体作为生物制剂的优点(仅作用靶标,具有安全性) 也是缺点(宿主谱窄),RBP是研究噬菌体生物技术和优化噬菌体生物制剂的关键:利用RBP的宿主特异性可以进行细菌鉴定及血清型诊断;通过RBP的编辑和驯化,可以对噬菌体宿主谱进行人为选择;将RBP作为抗生素或噬菌体尾类细菌素(Phage tail-like bacteriocins,PTLBs)的载体可以开发综合防治病原菌的生物制剂(图 4,表 1)。
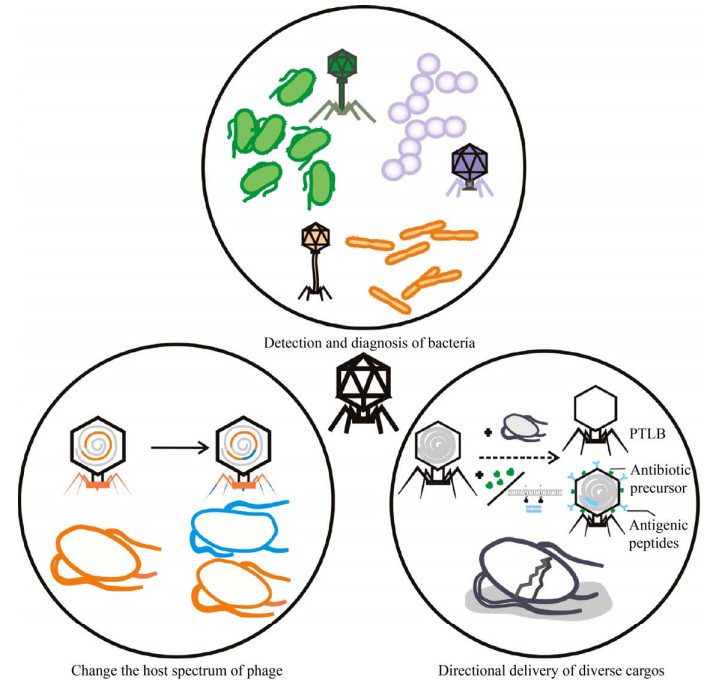 |
| 图 4 噬菌体受体结合蛋白的应用 Fig. 4 Applications of phage RBPs. |
| 图选项 |
表 1 噬菌体吸附蛋白相关生物技术Table 1 Biotechnologies related with phage RBPs
| Categories | Objectives | Main methods | References |
| Detection and diagnosis of bacteria | ① Diagnosis of pathogenic bacteria ② Drug sensitivity detection ③ Distinguish bacterial type | ① Construct luciferase-reporter phage ② Cluster bacteria into different phage type with different lytic pattern | [20-26, 28-29] |
| Change the host spectrum of phage | ① Expand host spectrum ② Narrow host spectrum | ① RBP gene fragments homologous recombination ② Phage display technology | [13, 31-33] |
| Directional delivery of diverse cargos | ① Cell-specific delivery of medicine by VLP ② Cell-specific delivery of antimicrobial peptides and protein toxins ③ Directional delivery of siRNA ④ Defend against strains by PTLB and expand antibacterial spectra of PTLB | ① Coupling of phage hydrophilic aminoglycoside linker and antibiotic precursor using EDC reaction ② Construct chimeric VLP ③ Modify phage with peptide or compound ④ Phage display technology ⑤ RBP gene fragments homologous recombination | [34-37, 39-40] |
表选项
3.1 利用噬菌体RBP进行细菌鉴定与诊断利用噬菌体特异性识别宿主菌的特性可以发展噬菌体诊断试剂,对病原体进行精准高效的鉴定或耐药性检测。例如NCTC12673可以特异性吸附空肠弯曲菌与大肠杆菌,利用该噬菌体RBP的吸附结构域CC-Gp047短肽进行细菌凝集试验,或用EGFP_CC-Gp047复合物对细菌样本进行荧光检测,可在几分钟内完成大肠杆菌与空肠弯曲菌的鉴定。目前,针对结核分枝杆菌[20-21]、鼠疫耶尔森菌[22-23]、大肠杆菌[24]、炭疽杆菌[25]、副溶血性弧菌[26]等多种病原菌都开发了成熟的特异性荧光噬菌体对其进行诊断与鉴定,这种鉴定手段用时短、敏感性高,检测可准确到单个菌落[27]。
不同噬菌体对同一宿主菌的吸附位点不同,产生的裂解表型也有差异,利用噬菌体体系能对细菌进行分型检测[28-29]。O139群霍乱弧菌为强致病性血清型,Chakrabarti等分离到5种O139噬菌体(MAD-5、VE-2、CAL-3、S-2和MS-3),依据它们不同的侵染表型,将O139群霍乱弧菌分成不同的噬菌体型[30]。
3.2 利用噬菌体RBP编辑改变噬菌体宿主谱对噬菌体RBP或其他结构蛋白(衣壳蛋白等)进行编辑可以使噬菌体获得识别吸附其他细菌靶点的能力,扩大噬菌体的宿主范围[13-31]。Cao等从幽门螺旋杆菌免疫过的小鼠淋巴细胞内获得了特异性结合幽门螺旋杆菌的抗原结合片段,并利用噬菌体展示技术,将该片段展示在M13表面,使M13获得了识别和吸附幽门螺旋杆菌的特性[32]。噬菌体WG01与QL01具有不同的宿主范围,其尾丝蛋白的相似性只有59%[31],Chen等以它们的宿主菌DE017为介导,构建了包含QL01不同gp37基因片段的WG01子代嵌合噬菌体,部分子代嵌合噬菌体获得了两种亲代噬菌体的宿主谱,有些则获得了新宿主谱[33]。
在某些情况下,开发高特异性噬菌体制剂对杀灭特定病原、保障生物安全十分重要,RBP可协助噬菌体识别非生产性细菌,利用该特性,可以人工驯化缩小噬菌体宿主谱。根据Holtzman等的研究,T7可以通过尾丝蛋白gp17的自发突变丧失对有完整吸附受体(LPS) 的K-12的有效吸附。T7在两种宿主菌K-12ΔtrxA (噬菌体可吸附,但无法进行生物合成) 与K-12ΔwaaC (LPS部分缺失突变株) 中多次传代驯化后,对K-12ΔwaaC的吸附率增高而对K-12ΔtrxA的吸附率逐渐降低。尽管K-12ΔtrxA表面保留完整的LPS,T7仍丧失了识别宿主菌的能力,这种以非增殖性宿主菌为介导的连续进化系统可以有效缩小噬菌体RBP的受体识别范围,帮助构建高宿主特异性的噬菌体生物制剂[13]。
3.3 RBP参与抗生素或细菌毒素运载利用噬菌体结构蛋白、病毒样颗粒(Virus-like particle,VLP) 作为药物载体,将药物靶向运输到病灶部位,可减少抗生素的细胞毒性,同时可以降低噬菌体生物活剂的生态危害[34-35]。丝状噬菌体(M13、MS2) 是噬菌体载体研究的热点。Yacoby等将氯霉素前体修饰键与M13衣壳蛋白(g8p) 的亲水性氨基糖苷接头通过1-(3-二甲氨基丙基)-3-乙基碳二亚胺盐酸盐(EDC) 偶联,使M13获得了装载氯霉素前体的能力,靶向运载药物至病原菌,并富集于靶细菌周围,血清中酯酶催化M13-氯霉素连接键断裂,抗生素发挥药效[36]。Vaks等利用噬菌体展示技术使M13获得对不同病原菌的吸附能力,并对氯霉素进行了优化,引入新霉素-氯霉素复合物(Neo-CAM),使M13获得了更高的抗生素载量[37]。M13噬菌体有望被改造成标准化、高载量的纳米级抗生素载体。
R型PTLBs与肌尾噬菌体的吸附机制类似,其裂菌谱同样由RBP决定,可通过基因编辑对其进行改造[38]。例如,将大肠杆菌P2噬菌体的尾纤维RBP结构域嵌合到铜绿假单胞菌的R2细菌素中,可以获得对大肠杆菌具有杀菌效果的R2嵌合体;将噬菌体ΦV10尾突C端识别O157抗原的区域嵌合到R2的尾丝N端,R2获得了识别O157的能力[39]。相较于改造噬菌体RBP,对细菌素进行基因工程的操作对象是溶源菌,技术上更成熟简便,而且PTLBs没有头部结构,可以杜绝噬菌体基因组溶源转导造成的细菌毒力因子与耐药性传播的危害。
4 总结与展望噬菌体自发现之初便被作为具有潜力的细菌天然抑制剂被广泛研究开发,不仅用于抗菌抑菌、病原检测,也被用作药物运输、基因编辑的工具。噬菌体RBP是开发噬菌体生物制剂及开展噬菌体细菌互作研究的工作基础。噬菌体生物制剂正逐步向高效化、经济化、生物安全化靠拢,以RBP载体为代表的噬菌体制剂被研发乃至走向临床与应用,有效克服了噬菌体难以医用的限制。随着现代生物学技术手段的发展,各种RBP的类型以及吸附模型逐渐明确,但仍然存在一些经验无法解释的问题。例如,铜绿假单胞菌噬菌体Ab09可以低效侵染吸附受体缺失突变株[41];Chen等在构建广谱嵌合噬菌体时发现,某些RBP的C端结构域相同的嵌合噬菌体具有不同的宿主谱[33]。这说明“RBP-细菌受体”鉴定工作以及吸附模型的建立均存在不足,也许RBP的N端结构域或者尾丝蛋白的三级结构也参与了噬菌体的宿主特异性。因此,我们还需要更深入挖掘噬菌体RBP,解析其亚结构和细菌受体的互作、明确吸附模型,深入探讨噬菌体与细菌的博弈与进化。
参考文献
| [1] | Lin DM, Koskella B, Lin HC. Phage therapy: an alternative to antibiotics in the age of multi-drug resistance. World J Gastrointest Pharmacol Ther, 2017, 8(3): 162-173. DOI:10.4292/wjgpt.v8.i3.162 |
| [2] | Dowah ASA, Clokie MRJ. Review of the nature, diversity and structure of bacteriophage receptor binding proteins that target Gram-positive bacteria. Biophys Rev, 2018, 10(2): 535-542. DOI:10.1007/s12551-017-0382-3 |
| [3] | Nobrega FL, Vlot M, De Jonge A, et al. Targeting mechanisms of tailed bacteriophages. Nat Rev Microbiol, 2018, 16(12): 760-773. DOI:10.1038/s41579-018-0070-8 |
| [4] | Bebeacua C, Tremblay D, Farenc C, et al. Structure, adsorption to host, and infection mechanism of virulent lactococcal phage p2. J Virol, 2013, 87(22): 12302-12312. DOI:10.1128/JVI.02033-13 |
| [5] | Sun YY, Roznowski AP, Tokuda JM, et al. Structural changes of tailless bacteriophage ΦX174 during penetration of bacterial cell walls. Proc Natl Acad Sci USA, 2017, 114(52): 13708-13713. DOI:10.1073/pnas.1716614114 |
| [6] | Silva JB, Storms Z, Sauvageau D. Host receptors for bacteriophage adsorption. FEMS Microbiol Lett, 2016, 363(4): fnw002. DOI:10.1093/femsle/fnw002 |
| [7] | Hu B, Margolin W, Molineux IJ, et al. Structural remodeling of bacteriophage T4 and host membranes during infection initiation. Proc Natl Acad Sci USA, 2015, 112(35): E4919-E4928. DOI:10.1073/pnas.1501064112 |
| [8] | Dunne M, Hupfeld M, Klumpp J, et al. Molecular basis of bacterial host interactions by gram-positive targeting bacteriophages. Viruses, 2018, 10(8): 397. DOI:10.3390/v10080397 |
| [9] | Farley MM, Tu JG, Kearns DB, et al. Ultrastructural analysis of bacteriophage Φ29 during infection of Bacillus subtilis. J Struct Biol, 2017, 197(2): 163-171. DOI:10.1016/j.jsb.2016.07.019 |
| [10] | Azam AH, Tanji Y. Bacteriophage-host arm race: an update on the mechanism of phage resistance in bacteria and revenge of the phage with the perspective for phage therapy. Appl Microbiol Biotechnol, 2019, 103(5): 2121-2131. DOI:10.1007/s00253-019-09629-x |
| [11] | Rost?l JT, Marraffini L. (Ph)ighting phages: how bacteria resist their parasites. Cell Host Microbe, 2019, 25(2): 184-194. DOI:10.1016/j.chom.2019.01.009 |
| [12] | Samson JE, Magadán AH, Sabri M, et al. Revenge of the phages: defeating bacterial defences. Nat Rev Microbiol, 2013, 11(10): 675-687. DOI:10.1038/nrmicro3096 |
| [13] | Holtzman T, Globus R, Molshanski-Mor S, et al. A continuous evolution system for contracting the host range of bacteriophage T7. Sci Rep, 2020, 10: 307. DOI:10.1038/s41598-019-57221-0 |
| [14] | Meyer JR, Dobias DT, Weitz JS, et al. Repeatability and contingency in the evolution of a key innovation in phage lambda. Science, 2012, 335(6067): 428-432. DOI:10.1126/science.1214449 |
| [15] | Glonti T, Chanishvili N, Taylor PW. Bacteriophage-derived enzyme that depolymerizes the alginic acid capsule associated with cystic fibrosis isolates of Pseudomonas aeruginosa. J Appl Microbiol, 2010, 108(2): 695-702. DOI:10.1111/j.1365-2672.2009.04469.x |
| [16] | Cornelissen A, Ceyssens J, Krylov VN, et al. Identification of EPS-degrading activity within the tail spikes of the novel Pseudomonas putida phage AF. Virology, 2012, 434(2): 251-256. DOI:10.1016/j.virol.2012.09.030 |
| [17] | Santos SB, Kropinski AM, Ceyssens J, et al. Genomic and proteomic characterization of the broad-host-range Salmonella phage PVP-SE1: creation of a new phage genus. J Virol, 2011, 85(21): 11265-11273. DOI:10.1128/JVI.01769-10 |
| [18] | Schwarzer D, Buettner FFR, Browning C, et al. A multivalent adsorption apparatus explains the broad host range of phage phi92: a comprehensive genomic and structural analysis. J Virol, 2012, 86(19): 10384-10398. DOI:10.1128/JVI.00801-12 |
| [19] | Morozova V, Fofanov M, Tikunova N, et al. First crAss-like phage genome encoding the diversity-generating retroelement (DGR). Viruses, 2020, 12(5): 573. DOI:10.3390/v12050573 |
| [20] | Mayer O, Jain P, Weisbrod TR, et al. Fluorescent reporter DS6A mycobacteriophages reveal unique variations in infectibility and phage production in mycobacteria. J Bacteriol, 2016, 198(23): 3220-3232. DOI:10.1128/JB.00592-16 |
| [21] | Jain P, Hartman TE, Eisenberg N, et al. φ2GFP10, a high-intensity fluorophage, enables detection and rapid drug susceptibility testing of Mycobacterium tuberculosis directly from sputum samples. J Clin Microbiol, 2012, 50(4): 1362-1369. DOI:10.1128/JCM.06192-11 |
| [22] | Kim S, Kim M, Ryu S. Development of an engineered bioluminescent reporter phage for the sensitive detection of viable Salmonella typhimurium. Anal Chem, 2014, 86(12): 5858-5864. DOI:10.1021/ac500645c |
| [23] | Vandamm JP, Rajanna C, Sharp NJ, et al. Rapid detection and simultaneous antibiotic susceptibility analysis of Yersinia pestis directly from clinical specimens by use of reporter phage. J Clin Microbiol, 2014, 52(8): 2998-3003. DOI:10.1128/JCM.00316-14 |
| [24] | Kim J, Kim M, Kim S, et al. Sensitive detection of viable Escherichia coli O157:H7 from foods using a luciferase-reporter phage phiV10lux. Int J Food Microbiol, 2017, 254: 11-17. DOI:10.1016/j.ijfoodmicro.2017.05.002 |
| [25] | Sharp NJ, Vandamm JP, Molineux IJ, et al. Rapid detection of Bacillus anthracis in complex food matrices using phage-mediated bioluminescence. J Food Prot, 2015, 78(5): 963-968. DOI:10.4315/0362-028X.JFP-14-534 |
| [26] | Peng Y, Jin YQ, Lin H, et al. Application of the VPp1 bacteriophage combined with a coupled enzyme system in the rapid detection of Vibrio parahaemolyticus. J Microbiol Methods, 2014, 98: 99-104. DOI:10.1016/j.mimet.2014.01.005 |
| [27] | Lemire S, Yehl KM, Lu TK. Phage-based applications in synthetic biology. Annu Rev Virol, 2018, 5(1): 453-476. DOI:10.1146/annurev-virology-092917-043544 |
| [28] | Sarkar BL, Bhowmick TS, Das M, et al. Phage types of vibrio cholerae O1 and O139 in the past decade in India. Jpn J Infect Dis, 2011, 64(4): 312-315. |
| [29] | Hamdi S, Rousseau GM, Labrie SJ, et al. Characterization of two polyvalent phages infecting Enterobacteriaceae. Sci Rep, 2017, 7: 40349. DOI:10.1038/srep40349 |
| [30] | Chakrabarti AK, Ghosh AN, Nair GB, et al. Development and evaluation of a phage typing scheme for Vibrio cholerae O139. J Clin Microbiol, 2000, 38(1): 44-49. DOI:10.1128/JCM.38.1.44-49.2000 |
| [31] | Ando H, Lemire S, Pires D, et al. Engineering modular viral scaffolds for targeted bacterial population editing. Cell Syst, 2015, 1(3): 187-196. DOI:10.1016/j.cels.2015.08.013 |
| [32] | Cao J, Sun YQ, Berglindh T, et al. Helicobacter pylori-antigen-binding fragments expressed on the filamentous M13 phage prevent bacterial growth. Biochim Biophys Acta, 2000, 1474(1): 107-113. DOI:10.1016/S0304-4165(00)00005-2 |
| [33] | Chen MM, Zhang L, Abdelgader SA, et al. Alterations in gp37 expand the host range of a T4-like phage. Appl Environ Microbiol, 2017, 83(23): e01576-17. |
| [34] | Ashley CE, Carnes EC, Phillips GK, et al. Cell-specific delivery of diverse cargos by bacteriophage MS2 virus-like particles. ACS Nano, 2011, 5(7): 5729-5745. DOI:10.1021/nn201397z |
| [35] | Lino CA, Caldeira JC, Peabody DS. Display of single-chain variable fragments on bacteriophage MS2 virus-like particles. J Nanobiotechnol, 2017, 15: 13. DOI:10.1186/s12951-016-0240-7 |
| [36] | Yacoby I, Bar H, Benhar I. Targeted drug-carrying bacteriophages as antibacterial nanomedicines. Antimicrob Agents Chemother, 2007, 51(6): 2156-2163. DOI:10.1128/AAC.00163-07 |
| [37] | Vaks L, Benhar I. Antibacterial application of engineered bacteriophage nanomedicines: antibody-targeted, chloramphenicol prodrug loaded bacteriophages for inhibiting the growth of Staphylococcus aureus bacteria//Hurst S. Biomedical Nanotechnology. New York: Humana Press, 2011, 726: 187-206. |
| [38] | Williams SR, Gebhart D, Martin DW, et al. Retargeting R-type pyocins to generate novel bactericidal protein complexes. Appl Environ Microbiol, 2008, 74(12): 3868-3876. DOI:10.1128/AEM.00141-08 |
| [39] | Ritchie JM, Greenwich JL, Davis BM, et al. An Escherichia coli O157-specific engineered pyocin prevents and ameliorates infection by E. coli O157: H7 in an animal model of diarrheal disease. Antimicrob Agents Chemother, 2011, 55(12): 5469-5474. DOI:10.1128/AAC.05031-11 |
| [40] | Sánchez-Sánchez L, Tapia-Moreno A, Juarez-Moreno K, et al. Design of a VLP-nanovehicle for CYP450 enzymatic activity delivery. J Nanobiotechnol, 2015, 13: 66. DOI:10.1186/s12951-015-0127-z |
| [41] | Latino L, Midoux C, Hauck Y, et al. Pseudolysogeny and sequential mutations build multiresistance to virulent bacteriophages in Pseudomonas aeruginosa. Microbiology, 2016, 162(5): 748-763. DOI:10.1099/mic.0.000263 |
