
 , 董庆利1
, 董庆利1 1. 上海理工大学 医疗器械与食品学院,上海 200093;
2. 浙江省农业科学院农产品质量安全与营养研究所 农业农村部农产品质量安全风险评估实验室 (杭州) 农产品质量安全危害因子与风险防控国家重点实验室,浙江 杭州 310021
收稿日期:2020-08-25;接收日期:2020-11-13;网络出版时间:2020-11-26
基金项目:国家重点研发计划(No. 2019YFE0103800),浙江省农业科学院省部共建农产品质量安全国家重点实验室开放基金项目(No. 2010DS700124-KF2001) 资助
摘要:规律成簇间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR) 与其相关蛋白基因系统可通过限制基因的水平转移而有效防御噬菌体等外源基因元件的入侵,不同细菌之间的CRISPR结构有所差异。基于CRISPR系统的差异性,文中对近几年CRISPR在食源性致病菌进化分析、检测与分型、毒力与耐药中的应用研究进行概述,并对基于CRISPR序列特点开发的细菌检测分型方法以及CRISPR与食源性致病菌的毒力、耐药性之间的相关性进行重点总结分析。此外,文中探讨了CRISPR在进化分析、检测与分型、毒力与耐药应用中的不足,提出将CRISPR分型方法标准化、完善与扩充致病菌CRISPR数据库、进一步探究噬菌体与细菌之间的共进化关系等建议,为进一步探索CRISPR功能提供参考。
关键词:CRISPR食源性致病菌进化分析检测与分型毒力与耐药
Application of CRISPR in evolution analysis, detecting and typing, virulence and antibiotic resistance regulation in food-borne pathogens
Zhiye Bai1, Wen Wang2, Xiaofeng Ji2, Yingping Xiao2, Shiqin Zhang1, Zichen Wang1, Hongmei Li1

 , Qingli Dong1
, Qingli Dong1 1. School of Medical Instrument and Food Engineering, University of Shanghai for Science and Technology, Shanghai 200093, China;
2. State Key Laboratory for Managing Biotic and Chemical Threats to the Quality and Safety of Agro-products, MOA Laboratory of Quality & Safety Risk Assessment for Agro-products (Hangzhou), Institute of Quality and Standard of Agricultural Products, Zhejiang Academy of Agricultural Sciences, Hangzhou 310021, Zhejiang, China
Received: August 25, 2020; Accepted: November 13, 2020; Published: November 26, 2020
Supported by: National Key Research and Development Program of China (No. 2019YFE0103800), The Opening Fund of State Key Laboratory of Agricultural Product Quality and Safety, Zhejiang Academy of Agricultural Sciences, China (No. 2010DS700124-KF2001)
Corresponding author: Hongmei Li. E-mail: sunnysand@126.com.
Abstract: Clustered regularly interspaced short palindromic repeats (CRISPR) and its associated protein gene system can limit the horizontal gene transfer, thereby effectively preventing the invasion of foreign gene elements such as bacteriophages. CRISPR arrays of different bacteria are diverse. Based on the differences in the CRISPR system, this review summarizes the application of CRISPR in food-borne pathogen evolution analysis, detection and typing, virulence and antibiotic resistance in recent years. We also address bacterial detection typing method developed based on the characteristics of CRISPR arrays and the association of CRISPR with virulence and drug resistance of food-borne pathogens. The shortcomings of CRISPR in evolution, detection and typing, virulence and resistance applications are analyzed. In addition, we suggest standardizing CRISPR typing methods, improving and expanding the CRISPR database of pathogenic bacteria, and further exploring the co-evolution relationship between phages and bacteria, to provide references for further exploration of CRISPR functions.
Keywords: CRISPRfood-borne pathogensevolutionary analysisdetection and typingvirulence and antibiotic resistance
食源性致病菌是引起食源性疾病的重要因素,它们通过将代谢产物与毒素分泌到食品、水源、土壤等媒介中,造成食品污染,从而导致食物中毒,严重时可致人死亡,是导致食品安全问题的重要原因[1]。常见食源性致病菌如沙门氏菌、金黄色葡萄球菌、副溶血性弧菌、单核细胞增生李斯特氏菌(单增李斯特菌)、铜绿假单胞菌、大肠杆菌O157:H7和志贺氏菌等引起的一系列食源性疾病,威胁着全球的公共卫生和食品安全,因此对食源性致病菌的检测、分型、溯源等进行研究具有重要意义。
研究表明,规律成簇间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR) 是基因组中进化最快的一种元件,分布在约40%的细菌与90%的古细菌中[2-3],其结构的多样性可为细菌的鉴定、分型、进化分析等工作提供可靠依据。CRISPR主要由重复序列(Repeat) 和间隔序列(Spacer) 构成,重复序列几乎一致,体现出细菌进化的保守性,而间隔序列具有高度特异性,其在CRISPR序列中的插入和缺失体现出细菌进化的多态性[4-6]。CRISPR与其相关蛋白基因(CRISPR associated,Cas) 组成CRISPR-Cas系统,该系统可通过噬菌体、质粒等外源基因为原核生物获得免疫能力,从而抵抗噬菌体感染,限制基因的水平转移(Horizontal gene transfer,HGT)[7]。CRISPR免疫的基本能力是在前端序列的近端以极化方式从外源DNA中获得一种新的间隔物,因此,CRISPR阵列中间隔区的位置信息反映了暴露于侵袭性DNA的时间顺序[8-9]。不同菌株之间的CRISPR序列有所差异,利用间隔序列的高度多态性,可作为基因分型的高分辨率生物标记物,以实现对菌株的分型。此外,研究发现CRISPR-Cas系统尤其是CRISPR间隔区的数量与细菌的毒力、耐药基因之间存在一定的关联[10]。
目前,CRISPR作为真核生物强有力的基因编辑工具[11],取得了许多有价值的研究成果,但其在食源性致病菌中进化分析、检测分型、细菌毒力与耐药的调控等方面的应用研究尚处于初步阶段。本文对近年来CRISPR在食源性致病菌中的应用研究内容进行回顾,总结出根据CRISPR序列特点开发的细菌检测分型方法具有高灵敏度、高鉴别力等优势,阐述了CRISPR与食源性致病菌的毒力、耐药性之间的相关性,介绍了CRISPR系统因外源质粒或噬菌体的进化而改变,从而记录细菌的进化以反映菌株之间的进化关系。通过分析不同研究结果总结CRISPR在各方面应用中的优势与不足,以期为进一步探索CRISPR功能与挖掘其应用潜力提供参考。
1 进化分析CRISPR对细菌的基因组进化产生影响,菌株之间的亲缘关系越近则它们的CRISPR间隔序列越相似,反之,菌株之间的亲缘关系越远则CRISPR间隔序列差异越大[12]。有研究发现除间隔序列外,重复序列也与细菌的进化有关,如大肠杆菌的重复序列能引起含有rpoS基因的染色体区域发生复制[13],使大肠杆菌的耐热性提高。CRISPR系统可以赋予细菌适应性免疫功能从而躲避噬菌体的侵袭,这一功能同时促进噬菌体的进化,使噬菌体进化出多种针对CRISPR的逃逸机制[14],表明CRISPR系统在细菌与噬菌体的共进化过程中也发挥了重要作用。
沙门氏菌通过获得与外源质粒和噬菌体等相同的基因序列间隔区来抵御水平基因转移,CRISPR位点所插入的新间隔序列因外源噬菌体和质粒的进化而改变[15],因此CRISPR记录着沙门氏菌的进化,同时也囊括菌株的地理学信息,可根据CRISPR序列的差异判断菌株间的进化关系。除此之外,沙门氏菌的CRISPR系统还能介导其自身短期表型的变化与长期的亚类进化[16]。大肠杆菌的CRISPR系统在不同的系统发育组中有所差异。Touchon等[12]通过分析CRISPR间隔序列组成分布与多位点序列分型进化树之间的关系发现,CRISPR相同的间隔序列几乎存在于亲缘关系近的菌株,而不具有亲缘关系的菌株之间没有相同的间隔序列。Fu等[17]对80株鸡源大肠杆菌的CRISPR1和CRISPR2位点进行研究,分析了所测CRISPR重复序列与间隔序列的数量,并将不同来源的分离株进行对比,结果发现CRISPR1的多态性低于CRISPR2。有研究结果表明产志贺毒素大肠杆菌(Shiga toxin-producing Escherichia coli,STEC) O157:H7血清型菌株由O55:H7血清型菌株进化而来[18],可见CRISPR可记录菌株之间的进化关系,揭示不同来源的菌株之间的相似性与差异性。通过研究克罗诺杆菌的CRISPR-Cas系统发现,其祖先菌株可能同时具有Ⅰ-E型与Ⅰ-F型CRISPR-Cas系统,在漫长的进化过程中,Ⅰ-E亚型得以保留在菌株中,而Ⅰ-F亚型选择性退化[19]。此外,Sun等[20]探究了CRISPR在致病性副溶血性弧菌毒力与进化中的作用,并对208株菌进行了CRISPR分型,结果显示副溶血性弧菌中的间隔序列数量较少,表明副溶血性弧菌处于进化的初期,且CST5型是进化最快的群体。由此可见,CRISPR间隔序列的数量可在一定程度上反映物种的进化程度。
基于上述,食源性致病菌CRISPR的生物信息学分析对于评估食源性致病菌的潜在进化、预测食源性致病菌的暴发具有重要意义。此外,噬菌体可能成为控制食源性致病菌的有效工具[21],且与宿主细菌之间存在相互进化关系,而CRISPR在其中扮演重要角色,但由于基因组的数据库完善程度不够,噬菌体基因组变异迅速,故CRISPR与噬菌体及细菌共进化之间的关系有待继续研究。
2 检测与分型食源性致病菌存在于多种食品中,并在食品生产系统中持续存在,故需要鉴别力高的检测方法对病原菌进行识别。目前对于食源性致病菌的检测多依赖于聚合酶链式反应技术,然而这种方法不能区分是否存在携带所有遗传标记的单一菌株和携带其中一个标记的多个非致病菌株,因此需对病原体的检测技术进行改进。CRISPR测序作为一种定量系统发育工具,可应用于食源性致病菌的检测鉴定。菌株分型为分离株的流行病学调查提供数据信息,用于流行病学监测或菌株进化研究。使用这些数据可以相应地指示暴发的可能来源与感染媒介,并识别病原体的自然宿主[22]。分子分型的要求主要有两点:一是分型结果应适合在实验室之间进行交换与对比;二是分型结果应易于上传至数据库[22]。由于CRISPR间隔序列的多态性与特异性,其间隔序列在不同食源性致病菌种、相同菌种的不同菌株或不同血清型之间差异较大[23],因此CRISPR不仅可以精确地反映菌株或血清型之间的关系,而且可以反映菌株分离宿主环境和地理位置的差异[24],基于此,CRISPR可为开发菌株分型方法提供位点。
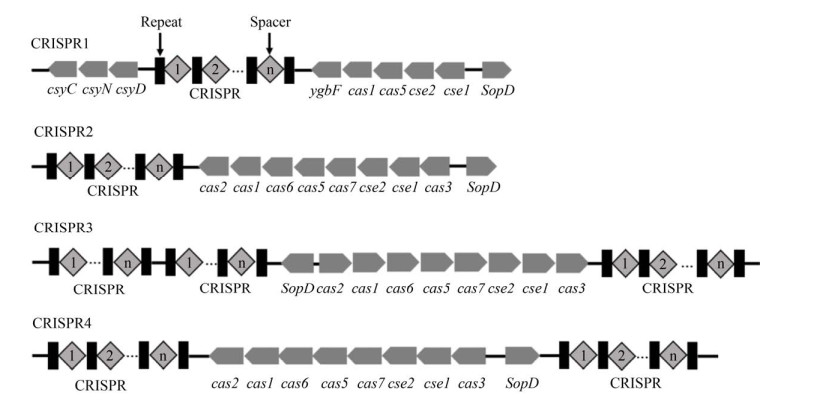
在大肠杆菌CRISPR-Cas系统中,目前已鉴定出4个基因座,分别为CRISPR1、CRISPR2、CRISPR3和CRISPR4[18, 25-27]。Delannoy等[28]以大肠杆菌CRISPR位点作为靶点,利用CRISPR序列的特点设计出一种鉴定菌株的实时聚合酶链式反应(Real-Time PCR) 方法,并通过试验验证了该方法的高灵敏度和高特异性,为CRISPR Real-Time PCR技术在纯细菌和食物及粪便标本上鉴定的应用提供了坚实的基础。从食品等复杂样品中检测STEC的方法目前主要是基于对主要毒力基因stx1、stx2、eae和O-血清群特异基因的PCR检测,然而这种方法缺乏分辨率[22],而CRISPR独特的序列特征可用于包括STEC在内的几种食源性致病菌的菌株检测。Noll等[29]进行了与Delannoy等[28]相似的研究,即建立了一种以CRISPR序列为靶点的实时定量聚合酶链式反应(qPCR) 法检测和定量牛粪便中肠出血性大肠杆菌(Enterohemorrhagic Escherichia coli,EHEC) O157:H7的方法,并证明以CRISPR O157:H7为靶点的检测方法是一种灵敏、高通量的检测牛粪便中大肠杆菌O157:H7血清型的方法。大肠杆菌的CRISPR-Cas系统目前被分为两个亚型,即Ⅰ-E型和Ⅰ-F型[25],Ⅰ-E型系统位于iap和cysH之间以及ygcF和ygcE之间,Ⅰ-F型系统位于clpA和infA之间[22],如图 1所示。Ⅰ-F型系统只在系统发育组B2中发现,并且是组成性表达的[12, 30]。Long等[9]分析了Ⅰ-F型CRISPR-Cas系统在B2群大肠杆菌中的存在情况与多样性,根据CRISPR3和CRISPR4间隔位点的特点将413株具有Ⅰ-F型系统的菌株分为23个基因型,从而证实了CRISPR在基因型分型中的潜力。沙门氏菌的CRISPR-Cas系统同样可分为Ⅰ-E和Ⅰ-F型,有CRISPR1、CRISPR2、CRISPR3和CRISPR4位点[31],如图 2所示。对于沙门氏菌的监测多依赖于用少数菌落的特征代表整体样本,因此对于大量的血清型样品检测难度低,而对于少数血清型样品的检测难度较高。Thompson等[32]利用其间隔序列的血清特异性开发了一种高通量的检测方法,该方法能够在单个样本中识别多个沙门氏菌血清型,且可以检测到混合菌种样品中区分不同的血清型的沙门氏菌菌株。该检测方法具有成本低与敏感性高的优点,可快速检出样品中低丰度的肠炎沙门氏菌,有类似研究也证实了这一点[33-34]。Li等[35]应用CRISPR分型法对分离自不同地区、不同宿主以及不同年限的329株肠炎沙门氏菌进行鉴定分型,将菌株分为18种CRISPR型,并将菌株CRISPR分型与全基因组序列分型相结合,对菌株CRISPR分型结果的可靠性进行了评估,表明CRISPR分型方法具有精确性高与鉴别力强等优势,同时CRISPR也是揭示菌株间系统发育关系的有效工具。有类似研究证实CRISPR分型相较于脉冲场凝胶电泳(Pulsed field gel electrophoresis,PFGE)、多位点序列分型(Multilocus sequence typing,MLST)、多位点可变数目串联重复序列分型(Multiple locus variable-number tandem repeat analysis,MLVA) 等分型方法,样本通量高且操作更为简单,更适合于沙门氏菌的暴发调查[36]。在其他食源性致病菌中也有类似研究,Zeng等[37]对588株克罗诺杆菌分别进行了MLST与CRISPR分析,结果表明CRISPR分型方法是克罗诺杆菌分子流行病学调查中更有效的确定亚型间分型的方法,并且与细菌表型有很好的对应关系。Yeh等[38]在78株空肠弯曲菌中共鉴定出55个不同的CRISPR间隔序列,并对菌株进行CRISPR分型与flaA核苷酸分型,结果表明,CRISPR基因分型的多样性高于flaA核苷酸分型。以上研究均试图将CRISPR作为食源性致病菌分型手段并与其他分型方法对比,研究结果不同程度地证明了基于CRISPR检测分型技术具有高灵敏、易操作等优势,可以作为食源性致病菌分型方法的补充。
 |
| 图 1 大肠杆菌CRISPR-Cas系统结构示意图(A、B为Ⅰ-E亚型,C为Ⅰ-F亚型) Fig. 1 Diagram of Escherichia coli CRISPR-Cas system. A and B are Ⅰ-E subtype, C is Ⅰ-F subtype. |
| 图选项 |
 |
| 图 2 沙门氏菌CRISPR-Cas系统结构示意图(CRISPR 1为Ⅰ-E亚型,CRISPR 2、CRISPR 3、CRISPR 4为Ⅰ-F亚型) Fig. 2 Diagram of Salmonella CRISPR-Cas system. CRISPR 1 is Ⅰ-E subtype, CRISPR 2, CRISPR 3 and CRISPR 4 are Ⅰ-F subtype. |
| 图选项 |
综上所述,基于CRISPR的检测与分型方法在食源性致病菌中有良好的应用前景,但目前数据库不够完善、方法不够成熟,尚未形成标准化,今后的研究可基于全基因组测序结挖掘更多细菌的CRISPR信息,建立食源性致病菌的CRISPR数据库,以充分发挥CRISPR的分型优势。
3 毒力与耐药通常来讲,细菌的毒力因子与其毒力相关。如金黄色葡萄球菌的splA、splB、splE毒力基因可编码丝氨酸蛋白酶导致表皮屏障的损害,seg、sei、sem、sen、seu毒力基因可编码肠毒素导致食物中毒[39];再如eae和stx2的存在是STEC菌株引起出血性结肠炎(Hemorrhagic colitis,HC) 或溶血性尿毒症综合征(Hemolytic uremic syndrome,HUS) 等严重疾病的毒力因子[40]。除毒力基因外,细菌可通过基因水平的突变、整合和转座等多个机制产生耐药性,耐药基因的水平转移作为细菌获得性耐药的一个重要方式已引起了广泛关注。CRISPR系统可通过有效抵抗噬菌体感染及质粒接合转移而限制外源性遗传物质的水平转移,其可能与细菌耐药基因的获取有一定关系。
副溶血性弧菌作为一种革兰氏阴性菌,栖息于海洋、河口和沿海环境,是急性肠胃炎的常见病因[41-43]。目前仅在副溶血性弧菌中发现一个结构为Ⅰ-F型的CRISPR位点,cas基因分布在该位点旁侧,毒力因子tdh与该菌存在的CRISPR-Cas之间存在显著的相关性[26]。有研究表明,CRISPR的缺失使噬菌体通过基因的水平转移以促进毒力基因的插入并增强副溶血性弧菌菌株的毒力[44],另一项研究表明CRISPR与副溶血性弧菌中的噬菌体数量呈负相关[45]。在单增李斯特菌中具有Ⅰ型、Ⅱ型以及RliB CRISPR系统[46-48],其中Ⅱ型CRISPR系统仅在革兰氏阳性菌中存在,而RliB CRISPR系统仅在单增李斯特菌中存在。狄慧玲等[49]的研究表明单增李斯特菌的CRISPR RliB、CRISPR Ⅰ-B和CRISPR Ⅱ-A系统除可抵御噬菌体侵袭外,还可调节细菌毒力、生物膜形成等生理活动,且促使单增李斯特菌朝着高毒力的方向进化。单增李斯特菌CRISPR Ⅱ-A型系统的间隔序列具有极高的多态性[47]。Sampson等[50]研究表明,Ⅱ型CRISPR系统可通过躲避宿主的免疫系统而提升细菌的侵染毒力,少数单增李斯特菌因存在Ⅱ型CRISPR系统而对宿主的危害更大。RliB CRISPR系统可形成茎环结构而调节单增李斯特菌的毒力,但其与菌株毒力的关系还应进一步研究[48]。志贺菌的毒力与CRISPR位点的存在具有相关性,即CRISPR1存在时细菌毒力弱,反之则强[51],但也有研究表明CRISPR系统与志贺菌的毒力基因分布无关[52]。有研究显示[9]细菌中Ⅰ-F型系统的缺失或存在可能影响毒性或抗生素抗性基因的分布,具体关系应增加菌株样本量进行探究。Toro等[26]研究了194株43个血清型的STEC菌株的CRISPR序列,探讨了CRISPR数量与毒力基因之间的潜在关系,结果表明,相较于具有较低致病潜力的菌株,具有较高致病潜力的菌株的CRISPR间隔区数量更少,eae阳性菌株的间隔区数量明显少于eae阴性菌株,Ⅰ-E Cas亚型系统的存在与毒力基因之间没有关联,但间隔区总数与潜在致病性呈负相关。除stx与eae基因之外,有研究也对STEC其他毒力基因或特征基因进行了分析,以确定其与CRISPR序列之间的关系。表 1将菌株来源分为食品、临床、环境3类,在大多数关于STEC毒力基因的文献中,分离菌株的来源以牛肉及牛粪便居多,且不同研究中分离株的毒力基因或特征基因有所差异,除O157血清群外,其他血清群的STEC也可引起食源性疾病。由于我国STEC菌株筛选数量相较于国外较少,因此对STEC菌株毒力基因与CRISPR序列相关性之间的研究在国内依然缺乏。
表 1 STEC菌株毒力基因/特征基因与CRISPR位点情况Table 1 STEC strain virulence gene/characteristic gene and CRISPR loci
| Strain source | Strain numbers | Serogroup/serotype | Virulence gene/characteristic gene | CRISPR locus | References |
| Food, environment, clinical | 194 | O26, O103, O111, O45, O121, O145, O157 | stx1, stx2, eae, hlyA, pagC, sen, nleB, efa-1, efa-2, terC, ureC, iha, aidA-I, nle2-3, nleG6-2, nleG5-2, irp2, fyuA, etc. | CRISPR1/2/3/4 | [26] |
| Food, environment, clinical | 65 | O113:H21 | stx1, stx2, eae, wzx, fliCH21, bfpA, ehxA, katP, espP, etpD, toxB, saa, subA, nleA, astA, irp2, lpfAO113, lpfAO26, iha, terE, ureD, Z2098, Z2099, Z2121, pagC, ent, nleB, nleE, efa1, efa2, etc. | CRISPR1/2a | [53] |
| Food | 115 | O26, O45, O103, O111, O113, O121, O145, O157 | stx1, stx2, eae, ECs848, ECs1322, ECs1323, ECs1326, ECs1561, ECs1568, ECs2226, ECs3857, ECs3858, ECs4552, ECs4553, ECs4557, etc. | CRISPR1/2a | [25] |
| Environment | 178 | O26:H11 | stx1, stx2, eae, espV, iha, terE, lpfA, ehxA, etc. | CRISPR1/2a | [54] |
| Food, clinical | 49 | O91:H21, O91:H10, O91:H14, O91:H9 | stx, fliCH4, fliCH10, fliCH21, etc. | CRISPR1/2a | [55] |
表选项
Baliga等[56]对46株副溶血性弧菌环境分离株的耐药性与CRISPR存在的相关性进行分析,发现除庆大霉素耐药性与CRISPR存在呈显著负相关外,抗生素耐药性与CRISPR与否无显著相关性。Aydin等[57]研究了82株抗生素敏感株和96株多重耐药(Multiple drug resistance,MDR) 大肠杆菌菌株的Ⅰ-E型和Ⅰ-F型CRISPR系统,发现MDR菌株Ⅰ-F型系统较少,因此可推测Ⅰ-F型CRISPR系统与大肠杆菌抗生素敏感性有关,即CRISPR Ⅰ-F型呈阳性时,大肠杆菌的耐药性较低,反之耐药性高。此外,菌株CRISPR间隔区数量与抗生素耐药性之间的关系因抗生素类型而异,如尿致病性大肠杆菌CRISPR间隔区数量与甲氧苄啶/磺胺甲恶唑耐药性呈正相关,与头孢唑林耐药性呈负相关,与氨苄西林耐药性无明显关系[58],这种关系的差异可能是由于每种抗生素的抗性机制不同。毛婷婷[39]对金黄色葡萄球菌26种耐药基因进行分析,发现大部分CRISPR阳性菌株的耐药性高于CRISPR阴性菌株。李琳等[59]发现沙门氏菌的CRISPR序列处在不同抗生素处理的环境中可能会发生变异,故推测CRISPR重复序列的变异和间隔序列的多样性与菌株耐药性有关。Palmer等[60]研究发现,粪肠球菌CRISPR的分布与菌株耐药性存在负相关关系。此外,金黄色葡萄球菌的CRISPR系统能够抑制外源基因的水平转移,并且能够阻止耐药基因在葡萄球菌之间的传播[7]。基于上述研究,CRISPR系统与细菌毒力及耐药具有相关性。CRISPR系统的退化有利于细菌基因组的重组,从而增强其毒力[52],因此分析细菌的CRISPR序列有利于细菌毒力变化机制的研究,因菌株数量与分离地区、时间等的限制,CRISPR系统与细菌毒力相关性应作进一步研究证明。在抗生素选择压力下,CRISPR系统可能因阻碍耐药基因的获取而丢失。但是目前CRISPR系统与细菌耐药关系的研究不多,需大量实验证实其关系,以助于耐药机制的深入探索。到目前为止,关于细菌CRISPR与菌株抗生素耐药性之间的关系未见统一说法,这可能与细菌种类、抗生素种类、宿主、菌株分离时间、地理位置等信息有关,因此对于细菌CRISPR与耐药性之间的关系还应深入研究。此外,利用CRISPR重复序列与间隔序列特点开发抗菌剂具有广阔的应用前景。
4 总结与展望目前,CRISPR在食源性致病菌中的应用主要体现在鉴定检测、分子分型、对细菌毒力的调节以及其与菌株耐药性的关系探究等方面,不同种类的细菌其CRISPR研究的深度也有所不同。CRISPR序列由于其间隔序列的多态性与特异性使其在细菌检测与分型方面显示出分辨率高、灵敏度强等优势,但目前该分型方法未标准化与规范化。CRISPR间隔序列与细菌毒力基因以及耐药基因的关系在不同研究中结论也有所差异,其原因除细菌种类不同之外,可能还有菌株分离地理位置和时间的差异,因此还应继续深入研究。越来越多食源性致病菌的CRISPR序列被揭示,因此菌株之间的进化关系也随之展现,CRISPR通过捕获外源噬菌体或质粒DNA而插入间隔区,亲缘关系越近的菌株CRISPR间隔序列也越相似,但目前对于CRISPR间隔区的缺失原因尚不清楚,需进一步探究。综上所述,CRISPR在食源性致病菌中的应用研究依然有很多待完备之处,可继续挖掘CRISPR强大的功能与应用潜力。
针对CRISPR在食源性致病菌中的应用现状,展望如下:首先,应完善与扩充各种致病菌的CRISPR数据库,充分挖掘其中信息,探究CRISPR与噬菌体及细菌之间的共进化关系;其次,基于目前有不少研究者以CRISPR位点为靶标设计检测致病菌的方法,进一步研究应着眼于形成成套标准化的CRISPR鉴定分型方法,也可将CRISPR与其他分型方法相结合以扩大CRISPR分型的适用范围;最后,利用CRISPR序列特点可开发抗菌剂,以提高杀菌速度与效率。
参考文献
| [1] | 肖芳斌, 刘瑞, 占忠旭, 等. 生物传感器在食源性致病菌检测中应用的研究进展. 生物工程学报, 2019, 35(9): 1581-1589. Xiao FB, Liu R, Zhan ZX, et al. Research progress of biosensors in the detection of foodborne pathogens. Chin J Biotech, 2019, 35(9): 1581-1589 (in Chinese). |
| [2] | Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea. Science, 2010, 327(5962): 167-170. DOI:10.1126/science.1179555 |
| [3] | Sorek R, Kunin V, Hugenholtz P. CRISPR—a widespread system that provides acquired resistance against phages in bacteria and archaea. Nat Rev Microbiol, 2008, 6(3): 181-186. DOI:10.1038/nrmicro1793 |
| [4] | Makarova KS, Haft DH, Barrangou R, et al. Evolution and classification of the CRISPR-Cas systems. Nat Rev Microbiol, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577 |
| [5] | Guzina J, Rodi? A, Blagojevi? B, et al. Modeling and bioinformatics of bacterial immune systems: understanding regulation of CRISPR/Cas and restriction-modification systems. Biol Serb, 2017, 39(1): 112-122. |
| [6] | Rossi CC, Souza-Silva T, Araújo-Alves AV, et al. CRISPR-Cas systems features and the gene-reservoir role of coagulase-negative Staphylococci. Front Microbiol, 2017, 8: 1545. DOI:10.3389/fmicb.2017.01545 |
| [7] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in Staphylococci by targeting DNA. Science, 2008, 322(5909): 1843-1845. DOI:10.1126/science.1165771 |
| [8] | Alkhnbashi OS, Shah SA, Garrett RA, et al. Characterizing leader sequences of CRISPR loci. Bioinformatics, 2016, 32(17): i576-i585. DOI:10.1093/bioinformatics/btw454 |
| [9] | Long JZ, Xu YK, Ou LY, et al. Polymorphism of type Ⅰ-F CRISPR/Cas system in Escherichia coli of phylogenetic group B2 and its application in genotyping. Infect Genet Evol, 2019, 74: 103916. DOI:10.1016/j.meegid.2019.103916 |
| [10] | 梁文娟. 基于CRISPRs的大肠埃希菌分型方法及其与耐药和毒力关系[D]. 郑州: 郑州大学, 2017. Liang WJ. The genotyping method based on CRISPRs and the relationship between the CRISPR/Cas and the virulence and resistant in Escherichia coli[D]. Zhengzhou: Zhengzhou University, 2017 (in Chinese). |
| [11] | 傅俊豪, 杨发誉, 谢海华, 等. 细菌中CRISPR/Cas系统的应用和优化. 生物工程学报, 2019, 35(3): 341-350. Fu JH, Yang FY, Xie HH, et al. Application and optimization of CRISPR/Cas system in bacteria. Chin J Biotech, 2019, 35(3): 341-350 (in Chinese). |
| [12] | Touchon M, Charpentier S, Clermont O, et al. CRISPR distribution within the Escherichia coli species is not suggestive of immunity-associated diversifying selection. J Bacteriol, 2011, 193(10): 2460-2467. DOI:10.1128/JB.01307-10 |
| [13] | Riehle MM, Bennett AF, Long AD. Genetic architecture of thermal adaptation in Escherichia coli. Proc Natl Acad Sci USA, 2001, 98(2): 525-530. DOI:10.1073/pnas.98.2.525 |
| [14] | 傅强, 孙建和, 严亚贤. 细菌CRISPR-Cas系统功能及其与噬菌体相互作用. 微生物学报, 2015, 55(3): 251-257. Fu Q, Sun JH, Yan YX. The functional aspects of bacterial CRISPR-cas systems and interactions between phages and its bacterial hosts—a review. Acta Microbiol Sin, 2015, 55(3): 251-257 (in Chinese). |
| [15] | 沈学怀, 张丹俊, 潘孝成, 等. 沙门氏菌CRISPR序列的结构功能与应用研究进展. 现代农业科技, 2017(20): 236-237, 245. Shen XH, Zhang DJ, Pan XC, et al. Research advance on structure, function and application of CRISPR in Salmonella. Mod Agric Technol, 2017(20): 236-237, 245 (in Chinese). DOI:10.3969/j.issn.1007-5739.2017.20.138 |
| [16] | Fricke WF, Mammel MK, McDermott PF, et al. Comparative genomics of 28 Salmonella enterica isolates: evidence for CRISPR-mediated adaptive sublineage evolution. J Bacteriol, 2011, 193(14): 3556-3568. DOI:10.1128/JB.00297-11 |
| [17] | Fu Q, Su ZX, Cheng YQ, et al. Clustered, regularly interspaced short palindromic repeat (CRISPR) diversity and virulence factor distribution in avian Escherichia coli. Res Microbiol, 2017, 168(2): 147-156. DOI:10.1016/j.resmic.2016.10.002 |
| [18] | Yin S, Jensen MA, Bai JW, et al. The evolutionary divergence of Shiga toxin-producing Escherichia coli is reflected in clustered regularly interspaced short palindromic repeat (CRISPR) spacer composition. Appl Environ Microbiol, 2013, 79(18): 5710-5720. DOI:10.1128/AEM.00950-13 |
| [19] | Zeng HY, Zhang JM, Wu QP, et al. Reconstituting the history of Cronobacter evolution driven by differentiated CRISPR activity. Appl Environ Microbiol, 2018, 84(10): e00267-18. |
| [20] | Sun HH, Li YH, Shi XL, et al. Association of CRISPR/Cas evolution with Vibrio parahaemolyticus virulence factors and genotypes. Foodborne Pathog Dis, 2015, 12(1): 68-73. DOI:10.1089/fpd.2014.1792 |
| [21] | De Melo AG, Levesque S, Moineau S. Phages as friends and enemies in food processing. Curr Opin Biotechnol, 2017, 49: 185-190. |
| [22] | Delannoy S, Beutin L, Fach P. Improved traceability of Shiga-toxin-producing Escherichia coli using CRISPRs for detection and typing. Environ Sci Pollut Res, 2016, 23(9): 8163-8174. DOI:10.1007/s11356-015-5446-y |
| [23] | Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems. Nat Rev Microbiol, 2015, 13(11): 722-736. DOI:10.1038/nrmicro3569 |
| [24] | 申进玲, 吴瑜凡, 崔思宇, 等. 成簇的规律间隔短回文重复序列在致病菌中分型研究及应用. 生物技术, 2017, 27(2): 186-191. Shen JL, Wu YF, Cui SY, et al. Study of clustered regularly interspaced short palindromic repeat sequence subtyping and its application in pathogens. Biotechnology, 2017, 27(2): 186-191 (in Chinese). |
| [25] | Díez-Villase?or C, Almendros C, García-Martínez J, et al. Diversity of CRISPR loci in Escherichia coli. Microbiology, 2010, 156(5): 1351-1361. DOI:10.1099/mic.0.036046-0 |
| [26] | Toro M, Cao GJ, Ju WT, et al. Association of clustered regularly interspaced short palindromic repeat (CRISPR) elements with specific serotypes and virulence potential of Shiga toxin-producing Escherichia coli. Appl Environ Microbiol, 2014, 80(4): 1411-1420. DOI:10.1128/AEM.03018-13 |
| [27] | Touchon M, Rocha EPC. The small, slow and specialized CRISPR and anti-CRISPR of Escherichia and Salmonella. PLoS ONE, 2010, 5(6): e11126. DOI:10.1371/journal.pone.0011126 |
| [28] | Delannoy S, Beutin L, Fach P. Use of clustered regularly interspaced short palindromic repeat sequence polymorphisms for specific detection of Enterohemorrhagic Escherichia coli strains of serotypes O26:H11, O45:H2, O103:H2, O111:H8, O121:H19, O145:H28, and O157:H7 by real-time PCR. J Clin Microbiol, 2012, 50(12): 4035-4040. DOI:10.1128/JCM.02097-12 |
| [29] | Noll LW, Chall R, Shridhar PB, et al. Validation and application of a real-time PCR assay based on the CRISPR array for serotype-specific detection and quantification of enterohemorrhagic Escherichia coli O157:H7 in cattle feces. J Food Prot, 2018, 81(7): 1157-1164. DOI:10.4315/0362-028X.JFP-18-049 |
| [30] | Almendros C, Guzmán NM, Díez-Villase?or C, et al. Target motifs affecting natural immunity by a constitutive CRISPR-Cas system in Escherichia coli. PLoS ONE, 2012, 7(11): e50797. DOI:10.1371/journal.pone.0050797 |
| [31] | 曲道峰, 沈杨, 张聪聪, 等. 沙门氏菌CRISPR位点的结构特征比较. 微生物学报, 2018, 58(2): 209-218. Qu DF, Shen Y, Zhang CC, et al. Genomics comparison between CRISPR loci and plasmid in Salmonella. Acta Microbiol Sin, 2018, 58(2): 209-218 (in Chinese). |
| [32] | Thompson CP, Doak AN, Amirani N, et al. High-resolution identification of multiple Salmonella serovars in a single sample by using CRISPR-SeroSeq. Appl Environ Microbiol, 2018, 84(21): e01859-18. |
| [33] | Fabre L, Hello SL, Roux C, et al. CRISPR is an optimal target for the design of specific PCR assays for Salmonella enterica serotypes Typhi and Paratyphi A. PLoS Negl Trop Dis, 2014, 8(1): e2671. DOI:10.1371/journal.pntd.0002671 |
| [34] | Richards AK, Hopkins BA, Shariat NW. Conserved CRISPR arrays in Salmonella enterica serovar Infantis can serve as qPCR targets to detect Infantis in mixed serovar populations. Lett Appl Microbiol, 2020, 71(2): 138-145. DOI:10.1111/lam.13296 |
| [35] | Li QC, Wang X, Yin KQ, et al. Genetic analysis and CRISPR typing of Salmonella enterica serovar enteritidis from different sources revealed potential transmission from poultry and pig to human. Int J Food Microbiol, 2018, 266: 119-125. DOI:10.1016/j.ijfoodmicro.2017.11.025 |
| [36] | Fabre L, Zhang J, Guigon G, et al. CRISPR typing and subtyping for improved laboratory surveillance of Salmonella infections. PLoS ONE, 2012, 7(5): e36995. DOI:10.1371/journal.pone.0036995 |
| [37] | Zeng HY, Li CS, Ling N, et al. Prevalence, genetic analysis and CRISPR typing of Cronobacter spp. isolated from meat and meat products in China. Int J Food Microbiol, 2020, 321: 108549. DOI:10.1016/j.ijfoodmicro.2020.108549 |
| [38] | Yeh HY, Awad A. Genotyping of Campylobacter jejuni isolates from poultry by clustered regularly interspaced short palindromic repeats (CRISPR). Curr Microbiol, 2020, 77(8): 1647-1652. DOI:10.1007/s00284-020-01965-w |
| [39] | 毛婷婷. 金黄色葡萄球菌CRISPR结构及耐药毒力分子特征[D]. 郑州: 郑州大学, 2019. Mao TT. CRISPR structure and molecular characteristics of resistance genes and virulence genes in Staphylococcus aureus[D]. Zhengzhou: Zhengzhou University, 2019 (in Chinese). |
| [40] | Ethelberg S, Olsen KEP, Scheutz F, et al. Virulence factors for hemolytic uremic syndrome, Denmark. Emerging Infect Dis, 2004, 10(5): 842-847. DOI:10.3201/eid1005.030576 |
| [41] | Li H, Tang R, Lou Y, et al. A comprehensive epidemiological research for clinical Vibrio parahaemolyticus in Shanghai. Front Microbiol, 2017, 8: 1043. DOI:10.3389/fmicb.2017.01043 |
| [42] | Ghenem L, Elhadi N, Alzahrani F, et al. Vibrio Parahaemolyticus: a review on distribution, pathogenesis, virulence determinants and epidemiology. Saudi J Med Med Sci, 2017, 5(2): 93-103. |
| [43] | Paranjpye R, Hamel OS, Stojanovski A, et al. Genetic diversity of clinical and environmental Vibrio parahaemolyticus strains from the Pacific Northwest. Appl Environ Microbiol, 2012, 78(24): 8631-8638. DOI:10.1128/AEM.01531-12 |
| [44] | Yu LH, Teh CSJ, Yap KP, et al. Comparative genomic provides insight into the virulence and genetic diversity of Vibrio parahaemolyticus associated with shrimp acute hepatopancreatic necrosis disease. Infect Genet Evol, 2020, 83: 104347. DOI:10.1016/j.meegid.2020.104347 |
| [45] | Li L, Wong HC, Nong WY, et al. Comparative genomic analysis of clinical and environmental strains provides insight into the pathogenicity and evolution of Vibrio parahaemolyticus. BMC Genomics, 2014, 15: 1135. DOI:10.1186/1471-2164-15-1135 |
| [46] | 狄慧玲, 闫鹤, 石磊. 食源性单核细胞增生李斯特菌CRISPR结构的研究. 现代食品科技, 2014, 30(8): 64-69, 237. Di HL, Yan H, Shi L. Analysis of CRISPR regions in food-borne Listeria monocytogenes. Mod Food Sci Technol, 2014, 30(8): 64-69, 237 (in Chinese). |
| [47] | Di HL, Ye L, Yan H, et al. Comparative analysis of CRISPR loci in different Listeria monocytogenes lineages. Biochem Biophys Res Commun, 2014, 454(3): 399-403. DOI:10.1016/j.bbrc.2014.10.018 |
| [48] | Sesto N, Touchon M, Andrade JM, et al. A PNPase dependent CRISPR system in Listeria. PLoS Genet, 2014, 10(1): e1004065. DOI:10.1371/journal.pgen.1004065 |
| [49] | 狄慧玲. 单核细胞增生李斯特菌分子分型研究及CRISPR/Cas系统解析[D]. 漯河: 华南理工大学, 2014. Di HL. Molecular subtyping and CRISPR/Cas system analysis of Listeria monocytogenes[D]. Luohe: South China University of Technology, 2014 (in Chinese). |
| [50] | Sampson TR, Saroj SD, Llewellyn AC, et al. A CRISPR/Cas system mediates bacterial innate immune evasion and virulence. Nature, 2013, 497(7448): 254-257. DOI:10.1038/nature12048 |
| [51] | 洪丽娟, 张冰, 段广才, 等. CRISPR/Cas系统与志贺菌毒力和耐药的关系及插入序列IS600对cse2表达水平的影响. 微生物学报, 2016, 56(12): 1912-1923. Hong LJ, Zhang B, Duan GC, et al. Role of CRISPR/Cas systems in drug-resistance and virulence and the effect of IS600 on the expression of cse2 in Shigella. Acta Microbiol Sin, 2016, 56(12): 1912-1923 (in Chinese). |
| [52] | 郭向娇. 志贺菌中CRISPR/Cas系统的检测及其与毒力基因的关系[D]. 郑州: 郑州大学, 2015. Guo XJ. Detection of CRISPR/Cas system of Shigella and analysis its relationship with virulence genes[D]. Zhengzhou: Zhengzhou University, 2015 (in Chinese). |
| [53] | Feng PCH, Delannoy S, Lacher DW, et al. Genetic diversity and virulence potential of Shiga toxin-producing Escherichia coli O113:H21 strains isolated from clinical, environmental, and food sources. Appl Environ Microbiol, 2014, 80(15): 4757-4763. DOI:10.1128/AEM.01182-14 |
| [54] | Ison SA, Delannoy S, Bugarel M, et al. Genetic diversity and pathogenic potential of attaching and effacing Escherichia coli O26:H11 strains recovered from bovine feces in the United States. Appl Environ Microbiol, 2015, 81(11): 3671-3678. DOI:10.1128/AEM.00397-15 |
| [55] | Feng PCH, Delannoy S, Lacher DW, et al. Shiga toxin-producing serogroup O91 Escherichia coli strains isolated from food and environmental samples. Appl Environ Microbiol, 2017, 83(18): e01231-17. |
| [56] | Baliga P, Shekar M, Ahamed ST, et al. Antibiotic resistance pattern and its correlation to the presence of tdh gene and CRISPR-Cas system in Vibrio parahaemolyticus strains isolated from seafood. Indian J Fish, 2019, 66(2): 100-108. |
| [57] | Aydin S, Personne Y, Newire E, et al. Presence of Type Ⅰ-F CRISPR/Cas systems is associated with antimicrobial susceptibility in Escherichia coli. J Antimicrob Chemother, 2017, 72(8): 2213-2218. DOI:10.1093/jac/dkx137 |
| [58] | Dang TND, Zhang LX, Z?llner S, et al. Uropathogenic Escherichia coli are less likely than paired fecal E. coli to have CRISPR loci. Infect Genet Evol, 2013, 19: 212-218. DOI:10.1016/j.meegid.2013.07.017 |
| [59] | 李琳, 赵霞, 王磊, 等. 沙门菌CRISPR与耐药性的关系及生物信息学分析. 中国兽医科学, 2018, 48(1): 114-123. Li L, Zhao X, Wang L, et al. Detection of CRISPR and its relationship to drug resistance and bioinformatics methods in Salmonella isolates from chickens. Chin Vet Sci, 2018, 48(1): 114-123 (in Chinese). |
| [60] | Palmer KL, Gilmore MS. Multidrug-resistant enterococci lack CRISPR-cas. mBio, 2010, 1(4): e00227-10. |
