

广东工业大学生物医药学院,广东 广州 510006
收稿日期:2020-09-05;接收日期:2020-11-11;网络出版时间:2021-05-26
基金项目:广州市科技计划项目(No. 201903010025),广东省科技计划项目(No. 2016A010105020) 资助
摘要:紫萍Spirodela polyrrhiza是一种在生物质资源开发利用和水体生物修复中广泛使用的漂浮植物。随着水体富营养化日益严重,紫萍在水面随处可见。有文献报道紫萍对水体中的氮和磷有良好的净化作用。为了深入研究紫萍对水体中无机磷的吸收和转运,以紫萍为材料提取RNA反转录为cDNA,以此为模板,扩增得到1条特异性片段,其开放读码框(ORF) 全长序列为1 620 bp,编码539个氨基酸,命名为SpPHT1;1,在GenBank中登录号为MN720003。生物信息学分析表明,SpPHT1;1没有内含子,编码的蛋白是一种稳定的、疏水的、具有11个跨膜结构域的蛋白,其结构具有主要协助转运蛋白超家族(Major facilitator superfamily,MFS) 保守的结构域,聚类分析显示紫萍SpPHT1;1与玉米ZmPHT2和高粱SbPHT1-8有较近的亲缘关系,推测其属于植物磷转运蛋白家族PHT1成员。正常磷条件,SpPHT1;1在紫萍叶中的表达明显多于根;低磷条件促进该基因的表达,相对表达量在紫萍根和叶生长48 h时均达到峰值;丰磷条件会抑制基因表达,说明SpPHT1;1的表达会受到外界磷浓度的影响。研究结果有助于紫萍磷转运蛋白基因功能的进一步深入研究,为紫萍的进一步开发和利用提供理论依据。
关键词:紫萍SpPHT1;1扩增表达
Cloning and expression pattern of phosphate transporter 1;1 cDNA sequence from Spirodela polyrrhiza
Zhiwei Deng*, Wei Peng*, Ziqing Lu, Minghui Fu


School of Biomedicine Pharmaceutical Sciences, Guangdong University of Technology, Guangzhou 510006, Guangdong, China
Received: September 5, 2020; Accepted: November 11, 2020; Published: May 26, 2021
Supported by: Guangzhou Municipal Science and Technology Project, China (No. 201903010025), Science and Technology Planning Project of Guangdong Province, China (No. 2016A010105020)
Corresponding author: Minghui Fu. Tel/Fax: +86-20-38457043; E-mail: mhfugd@126.com.
*These authors contributed equally to this study.
Abstract: Spirodela polyrrhiza is a floating plant widely used in biomass utilization and eutrophication phytoremediation. It becomes a common aquatic plant everywhere with the increasingly serious eutrophication. It has been reported that S. polyrrhiza has a good effect on the remediation of eutrophication water. In order to study the absorption and transportation of phosphorus in S. polyrrhiza, we extracted RNA from S. polyrrhiza and then reverse transcribed it into cDNA, which was used as a template to amplify a specific fragment. The full-length sequence of the open reading frame (ORF) was 1 620 bp, encoding 539 amino acids, named SpPHT1;1, and the accession number in GenBank was MN720003. Bioinformatical analysis showed that SpPHT1;1 had no intron. The protein it encoded was a stable, hydrophobic protein with 11 transmembrane domains. SpPHT1;1 structure was similar to that of major facilitator superfamily (MFS) superfamily members. The cluster analysis showed that SpPHT1;1 was closely related to ZMPHT2 in maize and SBPHT1-8 in sorghum. So, it might belong to plant PHT1 family. The expression of SpPHT1;1 in leaf was significantly more than that of root under normal phosphorus condition. Low phosphorus condition could promote gene expression, and the relative expression level of SpPHT1;1 arrived at the peak at 48 h both in root and leaf. High phosphorus condition could inhibit gene expression. These results indicated that SpPHT1;1 expression would be affected by external phosphorus concentration. The results of this study are helpful for further research on the function of phosphate transporter. It also can provide theoretical basis for further development and utilization of S. polyrrhiza.
Keywords: Spirodela polyrrhizaSpPHT1;1amplify, expressionexpression
磷是植物生长发育必需的大量营养元素,是核酸、磷脂和ATP等生命大分子的重要组成成分,参与生物体内许多重要的酶促反应及其代谢调节[1-3]。当磷浓度过低时,植物一般会出现成熟周期延长且种子质量不好的现象,例如植株矮小、生长缓慢、叶子颜色暗淡、种子和果实比较小等[4]。当磷浓度过高时,促进植物呼吸作用,使繁殖器官过早发育,抑制植物生长。由于磷和锌及其他元素的拮抗作用,磷过多也会影响植物对镁、铁、锌等其他元素的吸收[5]。植物对磷的吸收一般是通过细胞膜上的磷转运蛋白PHT (Phosphorus transporter) 从外界环境摄取获得,磷转运蛋白基因家族主要可分为四大类:高亲和性的磷转运蛋白PHT1家族;低亲和力磷转运蛋白PHT2家族;新型的磷转运蛋白PHO1家族;最后一类是少量PHT3、PHT4、PHO2等其他基因家族[6-8]。
第一个PHT1基因是Bun-ya等[9]在1991年发现的酿酒酵母的PHO84基因,它能编码无机磷转运蛋白,后来又有很多****从水稻、烟草、拟南芥、马铃薯、番茄、大麦等植物中克隆出来PHT1。根据已克隆的PHT1家族成员结构的研究,发现其一般含有12个跨膜结构域,在第6和第7跨膜结构域之间有一个大的亲水环,亲水N和C末端都位于细胞质[10]。该家族蛋白具有H2PO4–/nH+共运子、糖转运子和MFS通用转运子等特征结构域和一段保守氨基酸序列GGDYPLSATIMSE[11]。这些家族成员基因编码的蛋白主要位于细胞质膜,负责植物根系对无机磷的吸收和体内的转运过程[12]。该家族成员基因的表达会受到外界无机磷浓度的影响,缺磷条件一般可以诱导该基因的表达[13]。
紫萍Spirodela polyrrhiza是浮萍科、浮萍属的一种漂浮植物,适应能力强,一年四季均能在湖泊河流中生存。紫萍的全基因组测序已经完成(Accession No. PRJNA205940)。它有20对染色体,基因组大小约为158 Mbp,预计编码19 263种蛋白质,其数量比拟南芥和水稻分别少28%和50%[14],清晰的遗传背景为紫萍功能基因的深入研究奠定了基础。Ziegler等和艾勇峰等[15-16]的研究发现,紫萍能够吸收大量生活污水中的N、P营养盐,对改善水质、治理水体富营养化有重要意义。蔡树美等[17]研究发现,在稀脉浮萍、少根紫萍和紫萍中,紫萍对无机磷的吸收速率最高,适用于对高磷浓度污水的净化。蔡冬蓉等[18]研究的青萍、少根紫萍和紫萍中,也得出同样结论。但紫萍高效吸收无机磷的机理却未见报道,未见任何有关紫萍磷转运蛋白基因序列克隆和功能研究方面的报道。本研究克隆到一条紫萍磷转运蛋白基因SpPHT1;1,对其进行生物信息学分析,并利用荧光定量PCR研究了该基因在不同磷浓度条件下的表达模式。研究结果有助于进一步深入研究紫萍磷转运蛋白的功能,为紫萍的进一步开发利用提供理论依据。
1 材料与方法1.1 紫萍中磷转运相关蛋白编码序列的分析用拟南芥磷转运蛋白编码序列AtPHT1;2 (NM_123703.3)、AtPHT2;1 (AF515591.1)、AtPHT3;1 (NM_121407.3)、AtPHT4;1 (NM_128519.6) 和AtPHO1 (AF474076.1) 对紫萍全基因组序列(Accession:PRJNA205940) 进行Blast比对,分析紫萍全基因组序列中与磷转运蛋白相关的编码序列,用FGENESH2.6[19]对相关基因组序列进行扫描(搜索物种参数设置为单子叶植物),找到基因组中与磷转运相关蛋白的编码序列。
1.2 材料紫萍由本实验室提供,无菌条件下培养,采用B5培养基加1%蔗糖的混合营养液,28 ℃、2 000 lx光照,明暗周期16 h/8 h,每两周更换1次新的培养液。
RNAiso plu、Prime ScriptTM RT-PCR Kit、Agarose Gel DNA Purification Kit、DL2 000/15 000 DNA marker、LA Taq酶,以上试剂均购自宝生物工程(大连) 有限公司。
1.3 紫萍RNA提取及反转录在液氮中将紫萍充分研磨成粉末,总RNA提取按照试剂盒说明书的方法进行提取。cDNA合成用反转录试剂盒说明书的方法进行,于–20 ℃保存。
1.4 SpPHT1;1基因的克隆利用Primer Premier 5.0设计SpPHT1;1上游引物5′-TGTACACACCGTCCTCT-3′和下游引物5′-TATGGCTTCAAACCTCA-3′。以紫萍cDNA为模板进行PCR扩增反应。扩增体系25 μL,其中:cDNA 2 μL,上下游引物各1 μL,LA Taq酶0.5 μL,dNTPs (10 mmol/L) 4 μL,缓冲液3 μL,灭菌水13.5 μL。扩增程序为:95 ℃变性5 min;95 ℃变性20 s,61.5 ℃退火30 s,72 ℃延伸40 s,进行30次循环;最后72 ℃延伸5 min。PCR扩增产物用1%的琼脂糖凝胶进行电泳验证,与pMD19-T载体连接,导入大肠杆菌,送样测序。
1.5 生物信息学分析采用在线软件ProtParam (https://web.expasy.org/protparam/) 预测SpPHT1;1蛋白序列及性质;采用在线软件PortScale (https://web.expasy.org/cgi-bin/protscale/protscale.pl)预测SpPHT1;1蛋白亲疏水性;采用TMHMM2.0 (http://www.cbs.dtu.dk/services/TMHMM/)和SMART (http://smart.embl-heidelberg.de/)预测SpPHT1;1蛋白的跨膜区;采用在线软件SMART (http://smart.embl-heidelberg.de/)和InterProScan (http://www.ebi.ac.uk/interpro/search/sequence/) 预测SpPHT1;1蛋白的保守结构域;采用软件MEGA 6.0邻接法对SpPHT1;1进行分子进化分析,使用软件默认参数,Bootstrap重复1 000次。
1.6 紫萍SpPHT1;1的表达将紫萍在0.1 mmol/L磷、1 mmol/L磷、2 mmol/L磷3种不同含磷浓度B5培养基中生长(培养基保持阳离子K+不变,用KCl试剂补充)。对3种不同含磷浓度条件下生长的紫萍,分别于培养0 h、24 h、48 h、72 h、96 h、120 h取样其根和叶,液氮速冻后置于–80 ℃保存。总RNA提取按照试剂盒方法进行提取,cDNA合成按照荧光定量PCR试剂盒(Takara公司) 的方法进行qRT-PCR。利用Primer Premier 5.0设计目标基因的上游引物5′-TGGTGATGGTGTGAGCC-3′和下游引物5′-GCAGTGGTGGTGAAGGA-3′。内参基因采用actin,上游引物5′-CAAACGGGAAATCTCTCG-3′和下游引物5′-GGTTCTGTTGGCATAGG C-3′。反应条件为:95 ℃ 40 s;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,40个循环;熔解曲线65–95 ℃,0.5 ℃/5 s;37 ℃ 30 s。取3次样品进行平行实验处理,采用2-ΔΔCT的方法计算,实验结果用平均值±标准差来表示。根据荧光定量PCR检测,研究SpPHT1;1基因在不同磷条件下表达情况。采用SPSS软件进行统计分析,采用单因素方差分析(One-way ANOVA) 中的Duncan法进行数据的多重比较,P < 0.05代表具有显著性差异。
2 结果与分析2.1 紫萍中磷转运相关蛋白编码序列的分析生物信息学分析发现紫萍全基因组序列中与磷转运相关蛋白的编码序列信息汇总如表 1所示,10个磷转运相关蛋白的基因分布在紫萍的7条染色体上。SpPHT1;1和SpPHT2无内含子,其他都有内含子,包含了3–7个外显子,编码序列长度为876–1 809 bp。
表 1 紫萍中磷转运相关蛋白的编码序列信息Table 1 Information of coding sequences related to phosphorus transportation in S. polyrrhiza
| Gene name | Base number | Exon number | Strand (+/–) | Sequence ID | Locus in chromosome |
| SpPHT1;1 | 1 620 | 1 | + | CP019106.1 | Chromosome 14, 1067916-1069535 |
| SpPHT1;2 | 1 623 | 1 | – | CP019095.1 | Chromosome 3, 1479298-1477676 |
| SpPHT2;1 | 1 809 | 3 | + | CP019104.1 | Chromosome 12, 2343549-2346817 |
| SpPHT3;1 | 1 212 | 7 | + | CP019099.1 | Chromosome 7, 1714900-1719902 |
| SpPHT3;2 | 876 | 6 | – | CP019093.1 | Chromosome 1, 9222901-9220711 |
| SpPHT3;3 | 1 083 | 5 | + | CP019098.1 | Chromosome 6, 3707567-3710683 |
| SpPHT3;4 | 906 | 5 | – | CP019106.1 | Chromosome 14, 1863493-1860745 |
| SpPHT4;1 | 1 311 | 6 | – | CP019099.1 | Chromosome 7, 7802292-7799733 |
| SpPHT4;2 | 1 110 | 7 | + | CP019110.1 | Chromosome 18, 448254-454833 |
| SpPHO1 | 1 242 | 7 | – | CP019095.1 | Chromosome 3, 415891-412277 |
表选项
2.2 紫萍SpPHT1;1基因的扩增根据紫萍磷转运蛋白基因序列SpPHT1;1,设计特异性引物。提取紫萍RNA反转录为cDNA,PCR扩增克隆获得紫萍的SpPHT1;1 cDNA序列,1%的琼脂糖凝胶进行电泳得到长度约为1 600 bp的条带(图 1)。连接到pMD19-T载体,转化大肠杆菌DH5α,测序。测序结果表明,SpPHT1;1 cDNA序列长度为1 620 bp,提交NCBI中GenBank数据库,获得登录号MN720003。
 |
| 图 1 SpPHT1;1片段的扩增 Fig. 1 Amplification of SpPHT1;1 fragment. M: DNA 2000 marker; 1: SpPHT1;1. |
| 图选项 |
2.3 紫萍SpPHT1;1c DNA序列分析采用在线软件ProtParam预测SpPHT1;1的序列特性。SpPHT1;1的开放阅读框(ORF)含有539个氨基酸,编码蛋白的分子量59.19 kDa,等电点8.51,分子式C2723H4151N683O740S28,不稳定系数36.14,脂肪系数87.51,总平均亲水性0.294。当不稳定系数小于或等于40时,该蛋白称为稳定蛋白,根据预测结果可知其不稳定系数为36.14,表明紫萍SpPHT1;1蛋白是稳定蛋白。SpPHT1;1c DNA序列与基因组序列中SpPHT1;1基因序列完全相同,说明该基因无内含子,这与前文FGENESH软件预测的结果(表 1) 完全一致。
2.4 紫萍SpPHT1;1蛋白的亲/疏水性采用在线软件PortScale预测SpPHT1;1蛋白的亲疏水性。结果大多数氨基酸的分值为正值,亲疏水性的最大值为2.922,最小值为–3.011,总平均亲水性值是0.294。蛋白亲疏水性是正值,表明紫萍SpPHT1;1蛋白总体为疏水蛋白(图 2)。
 |
| 图 2 SpPHT1;1蛋白的亲水性/疏水性预测 Fig. 2 Hydrophilic/hydrophobic prediction of SpPHT1;1 protein. |
| 图选项 |
2.5 紫萍SpPHT1;1蛋白的跨膜区域采用在线软件TMHMM和SMART预测SpPHT1;1蛋白的跨膜区,结果如图 3A和3B所示。两个软件预测的结果都显示紫萍SpPHT1;1蛋白有11个跨膜区,跨膜区的长度为19或22个氨基酸残基,在第6、7跨膜区之间含有一个亲水性的大环,表明该蛋白是一种跨膜蛋白,结构与之前发现的PHT1家族成员结构类似。
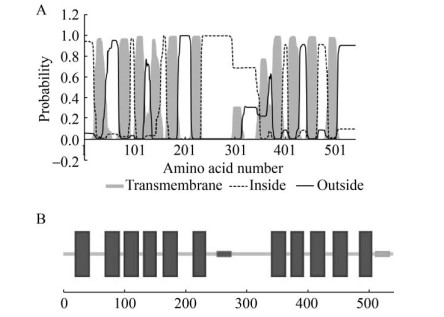 |
| 图 3 SpPHT1;1蛋白的跨膜区预测 Fig. 3 Transmembrane domain prediction of SpPHT1;1 protein. (A) Prediction by TMHMM. (B) Prediction by SMART. |
| 图选项 |
2.6 紫萍SpPHT1;1蛋白保守的功能结构域预测采用在线软件SMART和InterProScan软件对紫萍SpPHT1;1蛋白的结构域进行分析。SMART预测结果显示在SpPHT1;1氨基酸序列的16–457位存在MFS_1超家族保守的结构域(图 4A),在23–517位存在糖转运蛋白结构域(图 4B),InterProScan预测结果显示在SpPHT1;1氨基酸序列的11–517位存在Phos_permease (IPR004738)保守的结构域,在25–515位存在MFS_Sugar_ transport-like (IPR005828) 保守的结构域,根据预测结果推测SpPHT1;1属于植物转运蛋白家族成员。
 |
| 图 4 SpPHT1;1蛋白的结构域分析 Fig. 4 Domain analysis of SpPHT1;1 protein. (A) MFS_1 conserved domain. (B) Sugar_tr conserved domain. |
| 图选项 |
2.7 紫萍SpPHT1;1蛋白的分子进化分析将紫萍SpPHT1;1蛋白对NCBI中蛋白数据库blastp比对,选取相似性80%以上的15种植物磷转运蛋白序列进行分子进化分析(表 2)。发现SpPHT1;1蛋白与12种植物(胡杨PtPHT1-4、白杨PaPHT1、麻疯树JcPHT1-4、橡胶树HbPHT1-4、高粱SbPHT1-8、茶树CsPHT1-4、向日葵HaPHT1-7、枣ZjPHT1-4、川桑MnPHT1-4、阔叶栎QlPHT1-4、杨梅MrPHT1-4、胡桃JrPHT1-4) 有较高相似性,推测紫萍SpPHT1;1属于磷转运蛋白PHT1家族。另外,紫萍SpPHT1;1与玉米ZmPHT2和高粱SbPHT1-8聚到一起,并且分支的可靠性很高(98%),与油茶CoPHT和茶树CsPHT1-4及向日葵HaPHT1-7在不同的分支上,分支的可靠性达100%,SpPHT1;1的分歧与单双子叶分歧一致,说明该基因的进化是伴随着物种的进化而进化的(图 5)。
表 2 植物的PHT氨基酸序列相似性比较Table 2 Similarity analysis of amino acid sequence of plant PHT
| Species name | Accession No. | Identity (%) | Species name | Accession No. | Identity (%) |
| Populus alba | TKS03409.1 | 82.58 | Camellia oleifera | AFU07481.1 | 81.33 |
| Morella rubra | KAB1200467.1 | 82.44 | Juglans regia | XP_018827352.1 | 81.30 |
| Zea mays | NP_001105816.1 | 82.16 | Salix brachista | KAB5557556.1 | 81.27 |
| Sorghum bicolor | XP_002464558.1 | 81.97 | Ziziphus jujuba | XP_015868091.1 | 81.18 |
| Populus trichocarpa | XP_002306845.2 | 81.78 | Jatropha curcas | XP_012085328.1 | 81.16 |
| Camellia sinensis | XP_028077708.1 | 81.70 | Quercus lobata | XP_030934927.1 | 80.59 |
| Helianthus annuus | OTG32872.1 | 81.52 | Morus notabilis | XP_010111916.1 | 80.22 |
| Hevea brasiliensis | XP_021664188.1 | 81.50 |
表选项
 |
| 图 5 SpPHT1;1和其他一些近缘PHT蛋白的系统进化分析 Fig. 5 Phylogenetic analysis of SpPHT1;1 and other some closely related PHT proteins. |
| 图选项 |
2.8 紫萍SpPHT1;1的表达分析研究紫萍SpPHT1;1在不同磷条件下的表达情况,将植株放入3种不同磷浓度条件下培养,于0 h、24 h、48 h、72 h、96 h、120 h取样,利用荧光定量PCR检测紫萍根和叶中SpPHT1;1基因相对表达量。以基因在根中0 h时的表达量为标准,计算出SpPHT1;1基因在不同磷浓度的条件下根和叶中的相对表达量,结果如图 6所示,基因在紫萍叶中表达量明显多于根。0.1 mmol/L磷能够促进SpPHT1;1在紫萍根和叶中的表达,相对表达量都是先上升后下降,且在48 h表达量达到峰值,相对表达量在叶中的变化更为显著。
 |
| 图 6 SpPHT1;1基因在紫萍根和叶中的相对表达量 Fig. 6 Relative expression of SpPHT1;1 gene in root and leaf from S. polyrrhiza. |
| 图选项 |
3 讨论植物中的磷转运蛋白(Phosphate transporter)能够介导植物对磷素的吸收和转运[20]。目前磷转运蛋白中的PHT1家族研究最为广泛,普遍存在于细菌、真菌和植物中。PHT1家族属于高亲和力磷转运蛋白,利用细胞膜内外H+梯度差高效吸收和转运无机磷。自从Muchhal等在拟南芥中克隆得到两个磷酸盐转运体PHT1家族基因[21],越来越多高等植物的PHT1家族基因被克隆得到。水稻中已克隆13个PHT1家族的成员,其中8个成员的功能得以验证[22],拟南芥中至少有9个PHT1家族的成员[23],小麦中已克隆11个PHT1家族的成员[24]。在不同的环境条件下,植物的不同组织中的PHT1家族成员的表达各不相同[25-28]。植物体中各种不同的磷转运蛋白参与植物根土界面无机磷素的吸收、根表皮至根木质部运输、维管组织磷转运至叶片和植物细胞内磷素循环等过程[2-3]。水稻中OsPT1无论在根中还是在叶中都是高表达,而且外界磷浓度不影响水稻中OsPT1的表达;提高水稻中OsPT1的表达,能够提高水稻磷素的获得[27];番茄LePHT1基因在不缺磷或者缺磷条件,在植株根部和地上部均能表达,但低磷胁迫下能促进其表达[28]。不同PHT1基因家族成员表达的差异,对植物适应外界环境条件、合理利用外界无机磷、在体内恰当分配及转运磷元素具有重要意义。
本研究克隆出一条紫萍磷转运蛋白SpPHT1;1 cDNA,生物信息学进行分析发现SpPHT1;1 ORF长度为1 620 bp,编码含有539个氨基酸残基的蛋白质,具有植物转运蛋白保守的结构域,序列还含有磷转运蛋白PHT1家族特征保守结构域GGDYPLSATIMSE。聚类分析可知SpPHT1;1的氨基酸序列与12种植物PHT1家族成员有较高相似性,推测SpPHT1;1编码的蛋白可能为紫萍中的磷转运蛋白。此外,对比钟燕珊[29]克隆的水生植物水葫芦的EcPHT基因其蛋白总平均亲疏水性(0.405) 及其脂肪系数(91.13),发现紫萍SpPHT1;1蛋白的相应特性偏低,说明该蛋白的疏水性偏低,推测蛋白结合膜的能力较弱。
通过研究紫萍SpPHT1;1基因在不同磷浓度下的表达模式,低磷条件会促进基因表达,丰磷条件会抑制基因表达。该结果符合目前大多数克隆得到的植物PHT1家族基因的表达会受低磷胁迫调控的特征[30]。但值得注意的是,在正常磷(1 mmol/L) 条件下,SpPHT1;1基因在植株叶中表达明显多于根,该基因的表达情况与以往报道的PHT1家族成员主要定位在根中有一定差异[12],这可能与紫萍植株结构和生长环境有关。紫萍的根和茎部退化,根部仅剩余少量类似假根,叶子浮在水面上,下表皮紧贴水面,与水体接触的表面积远大于根,叶中存在的转运蛋白使得叶子能够快速吸取满足生长代谢所需营养物质并且运输到其他组织,因此SpPHT1;1在植株叶中表达会明显多于根中。
通过对SpPHT1;1 cDNA的克隆与表达的研究,有助于进一步深入解析SpPHT1;1的磷转运蛋白功能,为揭示紫萍在富营养化水体中快速生长、积累生物量的分子机理奠定基础,为紫萍在生物质资源开发利用上以及在富营养化水体的净化研究中提供理论基础,也能够为高效吸收磷的农作物育种提供优质候选基因资源。
参考文献
| [1] | Poirier Y, Bucher M. Phosphate transport and homeostasis in Arabidopsis. The Arabidopsis book, 2002, 1(1): e0024. |
| [2] | 王萍, 陈爱群, 余玲, 等. 植物磷转运蛋白基因及其表达调控的研究进展. 植物营养与肥料学报, 2006, 12(4): 584-591. Wang P, Chen AQ, Yu L, et al. Advance of plant phosphate transporter genes and their regulated expression. J Plant Nutr Fert, 2006, 12(4): 584-591 (in Chinese). DOI:10.3321/j.issn:1008-505X.2006.04.021 |
| [3] | 孙传范, 肖凯, 韩胜芳, 等. 植物吸收和转运磷素的分子机理研究进展. 中国农业科技导报, 2011, 13(2): 17-24. Sun CF, Xiao K, Han SF, et al. Advances in the molecular mechanism of phosphorus uptake and transportation in plants. J Agr Sci Technol, 2011, 13(2): 17-24 (in Chinese). |
| [4] | Zhang F, Sun Y, Pei W, et al. Involvement of OsPht1;4 in phosphate acquisition and mobilization facilitates embryo development in rice. Plant J, 2015, 82(4): 556-569. DOI:10.1111/tpj.12804 |
| [5] | 李志伟. 氮磷过量对植物生长的影响及有机肥的调控作用[D]. 保定: 河北农业大学, 2010. Li ZW. Effects of nitrogen and phosphorus excess on plant growth and regulation of organic fertilizer[D]. Baoding: Hebei Agricultural University, 2010 (in Chinese). |
| [6] | Catarecha P, Segura MD, Franco-zorrilla JM, et al. A mutant of the Arabidopsis phosphate transporter PHT1;1 displays enhanced arsenic accumulation. Plant Cell, 2007, 19(3): 1123-1133. DOI:10.1105/tpc.106.041871 |
| [7] | Miao J, Sun JH, Liu D, et al. Characterization of the promoter of phosphate transporter TaPHT1.2 differentially expressed in wheat varieties. J Genet Genomics, 2009, 36(8): 455-466. DOI:10.1016/S1673-8527(08)60135-6 |
| [8] | Preuss CP, Huang CY, Gilliham M, et al. Channel-like characteristics of the low-affinity barley phosphate transporter PHT1;6 when expressed in Xenopus oocytes. Plant Physiol, 2010, 152(3): 1431-1441. DOI:10.1104/pp.109.152009 |
| [9] | Bun-ya M, Nishimura M, Harashima S, et al. The PHO84 gene of Saccharomyces cerevisiae encodes an inorganic phosphate transporter. Cell Biol, 1991, 11(2): 3229-3238. |
| [10] | 董旭, 王雪, 石磊, 等. 植物磷转运子PHT1家族研究进展. 植物营养与肥料学报, 2017, 23(3): 799-810. Dong X, Wang X, Shi L, et al. Advances in plant PHT1 phosphate transporter family research. J Plant Nutr Fert, 2017, 23(3): 799-810 (in Chinese). |
| [11] | Karandashov V, Bucher M. Symbiotic phosphate transport in arbuscular mycorrhizas. Trends Plant Sci, 2005, 10(1): 22-29. DOI:10.1016/j.tplants.2004.12.003 |
| [12] | Ticconi CA, Abel S. Short on phosphate: plant surveillance and countermeasures. Trends Plant Sci, 2004, 9(11): 548-555. DOI:10.1016/j.tplants.2004.09.003 |
| [13] | Ye Y, Yuan J, Chang XJ, et al. The phosphate transporter gene OsPht1;4 is involved in phosphate homeostasis in rice. PLoS ONE, 2015, 10(5): e0126186. DOI:10.1371/journal.pone.0126186 |
| [14] | Wang WQ, Wu YR, Messing J. The mitochondrial genome of an aquatic plant, Spirodela polyrhiza. PLoS ONE, 2012, 7(10): 135-139. |
| [15] | Ziegler P, Appenroth KJ, Sree KS. Duckweeds for water remediation and toxicity testing. Toxicol Environ Chem, 2016, 98(10): 1127-1154. DOI:10.1080/02772248.2015.1094701 |
| [16] | 艾勇峰, 梁晓高, 张波, 等. 紫背浮萍对污水中总氮、总磷的去除效果研究. 江苏环境科技, 2008, 21(supp1): 21-28. Ai YF, Liang XG, Zhang B, et al. Purification effect of Spirodela polyhiza on sewage. Jiangsu Environ Sci Technol, 2008, 21(supp1): 21-28 (in Chinese). |
| [17] | 蔡树美, 刘文桃, 张震, 等. 不同品种浮萍磷素吸收动力学特征. 生态与农村环境学报, 2011, 27(2): 48-52. Cai SM, Liu WT, Zhang Z, et al. Phosphorus uptake kinetics of different types of duckweed. J Ecol Rural Environ, 2011, 27(2): 48-52 (in Chinese). DOI:10.3969/j.issn.1673-4831.2011.02.009 |
| [18] | 蔡冬蓉, 徐炎华. 浮萍对富营养化水体中磷的去除规律. 农业工程学报, 2011, 27(supp2): 187-190. Cai DR, Xu YH. Phosphorus removal rules of duckweed plants in eutrophication water. Transact CSAE, 2011, 27(Supp.2): 187-190 (in Chinese). |
| [19] | Solovyev V, Kosarev P, Seledsov I, et al. Automatic annotation of eukaryotic genes, pseudogenes and promoters. Genome Biol, 2006, 7(Supp1): S10. |
| [20] | 郑璐, 包媛媛, 张鑫臻, 等. 植物磷转运蛋白基因的研究进展. 生态环境学报, 2017, 26(2): 342-349. Zheng L, Bao YY, Zhang XZ, et al. Research progress of plant phosphorus transporter gene. J Ecol Environ, 2017, 26(2): 342-349 (in Chinese). |
| [21] | Muchhal US, Pardo JM, Rachothama KG. Phosphate transporters from the higher plant Arabidopsis thaliana. Proc Natl Acad Sci USA, 1996, 93: 10519-10523. DOI:10.1073/pnas.93.19.10519 |
| [22] | Goff SA. A draft sequence of the rice genome (Oryza sativa L. ssp. japonica). Science, 2002, 296(5565): 92-100. DOI:10.1126/science.1068275 |
| [23] | Mudge SR, Rae AL, Diatloff E, et al. Expression analysis suggests novel roles for members of the PHT1 family of phosphate transporters in Arabidopsis. Plant J, 2002, 31(3): 341-353. DOI:10.1046/j.1365-313X.2002.01356.x |
| [24] | Shukla V, Kaur M, Aggarwal S, et al. Tissue specific transcript profiling of wheat phosphate transporter genes and its association with phosphate allocation in grains. Sci Rep, 2016, 6(1): 39293. DOI:10.1038/srep39293 |
| [25] | Mitsukawa N, Okumura S, Shirano Y, et al. Overexpression of an Arabidopsis thaliana high-affinity phosphate transporter gene in tobacco cultured cells enhances cell growth under phosphate-limited conditions. Proc Natl Acad Sci USA, 1997, 94: 7098-7102. DOI:10.1073/pnas.94.13.7098 |
| [26] | Liu JL, Yang L, Luan MD, et al. A vacuolar phosphate transporter essential for phosphate homeostasis in Arabidopsis. Proc Natl Acad Sci USA, 2015, 112(47): 6571-6578. DOI:10.1073/pnas.1514598112 |
| [27] | Seo HM, Jung Y, Song S, et al. Increased expression of OsPT1, a high-affinity phosphate transporter, enhances phosphate acquisition in rice. Biotechnol Lett, 2008, 30(10): 1833-1838. DOI:10.1007/s10529-008-9757-7 |
| [28] | Daram P, Brunner S, Persson BL, et al. Functional analysis and cell-specific expression of a phosphate transporter from tomato. Planta, 1998, 206(2): 225-233. DOI:10.1007/s004250050394 |
| [29] | 钟燕珊. 水葫芦磷转运蛋白基因EcPHT的克隆、功能研究及表达[D]. 广州: 广东工业大学, 2019. Zhong YS. Cloning, functional study and expression analysis of phosphate transporter gene EcPHT from Eichhornia crassipes[D]. Guangzhou: Guangdong University of Technology, 2019 (in Chinese). |
| [30] | Rae AL, Cybinski DH, Jarmey JM, et al. Characterization of two phosphate transporters from barley; evidence for diverse function and kinetic properties amongst members of the Pht1 family. Plant Mol Biol, 2003, 53: 327-361. DOI:10.1023/B:PLAN.0000006942.00464.e3 |
