
 , 辛凤姣
, 辛凤姣

中国农业科学院农产品加工研究所,北京 100193
收稿日期:2020-05-20;接收日期:2020-07-14
基金项目:国家自然科学基金(Nos. 31700701,31801475),中国博士后科学基金(No. 2018M630230),中央级公益性科研院所基本科研业务费专项(No. S2020JBKY-13) 资助
摘要:姜黄素类化合物是植物中一类稀少的二酮类化合物,存在于姜科、天南星科植物的块根或根茎中,是姜黄等植物中主要活性成分,因具有抗氧化、抗癌等诸多药理活性而被广泛应用于食品领域和新药研发中。因其苯环侧链取代基不同,姜黄素类化合物可进一步分为姜黄素、去甲氧基姜黄素、双去甲氧基姜黄素等。目前,姜黄素类化合物主要是通过植物提取法获得,产量远远不能满足市场需求。随着合成生物学和代谢工程技术的发展,采用生物合成法生产姜黄素类化合物开始受到研究人员的广泛关注。近年来,研究人员通过基因挖掘及酶学手段鉴定了姜黄中姜黄素合成途径中的关键酶,并在大肠杆菌Escherichia coli、耶氏解酯酵母Yarrowia lipolytica、恶臭假单胞菌Pseudomonas putida和米曲霉Aspergillus oryzae中重塑其生物合成途径,成功实现了其异源生物合成。文中首先介绍了姜黄素的生物活性及其应用、总结了姜黄中的姜黄素合成途径,并且讨论了姜黄素合成酶的催化机制,进而详尽综述了其生物合成的最新研究进展,特别是代谢工程策略方面,并对其未来发展方向进行了展望。
关键词:姜黄素类化合物生物合成大肠杆菌微生物底盘细胞
Research progresses in the biosynthesis of curcuminoids
Luyao Wang, Xue Han, Fengzhong Wang, Lichao Sun

 , Fengjiao Xin
, Fengjiao Xin

Institute of Food Science and Technology, Chinese Academy of Agricultural Sciences, Beijing 100193, China
Received: May 20, 2020; Accepted: July 14, 2020
Supported by: National Natural Science Foundation of China (Nos. 31700701, 31801475), China Postdoctoral Science Foundation (No. 2018M630230), Central Public-interest Scientific Institution Basal Research Fund (No. S2020JBKY-13)
Corresponding author: Lichao Sun. Tel: +86-10-62893472; E-mail: sun2004go@163.com;
Fengjiao Xin. Tel: +86-10-62815873; E-mail: xinfengjiao@caas.cn.
Abstract: Curcuminoids are rare diketone compounds in plants and can be found in the rhizome of Curcuma longa as well as other Zingiberaceae and Araceae. Curcuminoids have been widely used in food and medical area owing to the yellow colors, as well as the antioxidant and many other pharmacological activities. Curcuminoids are a mixture of compounds containing curcumin, demethoxycurcumin and bisdemethoxycurcumin, which have distinct benzene ring substituents. Currently, curcuminoids are exclusively produced through plant extraction, which do not satisfy the meeting of the market demand. Empowered with new synthetic biology tools and metabolic engineering strategies, there is renewed interest in production of curcuminoids using microorganisms. Heterologous production of curcuminoids has been achieved using Escherichia coli, Yarrowia lipolytica, Pseudomonas putida and Aspergillus oryzae via engineering of curcuminoids biosynthesis pathway. In this review, we first describe the biological activities and various applications of curcuminoids. Next, we summarize the biosynthetic pathway of curcuminoids in Curcuma longa and discuss the catalytic mechanisms of curcumin synthases. Then, we thoroughly explore recent advances in the use of distinct microorganisms for the production of curcuminoids with a special focus on metabolic engineering strategies. Finally, we prospect the microbial production of curcuminoids by highlighting some promising techniques and approaches.
Keywords: curcuminoidsbiosynthesisEscherichia colimicrobial hosts
姜黄Curcuma longa L. 主要产于中国、印度等亚洲国家及非洲、北美洲等地。姜黄中的黄色成分为一种酚酸类物质的混合物,主要包括姜黄素(1, 7-双(4-羟基-3-甲氧基苯基)-1, 6-庚二烯-3, 5-二酮)、去甲氧基姜黄素(1, 5-羟基-7-(3-羟基-4-甲氧基苯基)-1-(3-羟基苯基)-1, 4, 6-庚三烯-3, 5-二酮) 及双去甲氧基姜黄素((1E, 6E)-1, 7-双(4-羟基苯基)庚二烯-1, 5-二酮),这些统称为姜黄素类化合物。其中,姜黄素是由两个芳香族丙烯酸聚合而成的二酮类化合物,分子式为C21H20O6,分子量为368.39。常温下,姜黄素为橙黄色结晶粉末,不溶于水,溶于乙醇和丙二醇,易溶于冰醋酸、甲醇、乙醇、乙酸乙酯和碱液。姜黄素稳定性较差,易与Fe2+、Fe3+螯合形成络合物[1];当pH大于10时,15 h内姜黄素降解率可达到50%以上[2],在室外光照5 d后,降解率高达68.9%[3]。姜黄素在pH大于8的条件下呈红色,在中性和酸性环境下呈黄色,因此化学上常用作酸碱指示剂。
姜黄素着色性较强,常作为食品着色剂。姜黄素因其具有特殊的抗氧化结构,亦可被用于食品的防腐过程。由于香味特殊,姜黄素已被作为食品调味剂广泛应用于咖喱类食品制作中[4]。在药品领域,姜黄素对Ⅱ型糖尿病具有较好的治疗作用[5]。姜黄素还可抑制人类免疫缺陷病毒(Human immunodeficiency virus,HIV) 的病毒活性,具有抗艾滋病功效[6]。姜黄素对治疗癌症、风湿、炎性眼病、肠道疾病以及口腔癌和黏膜白斑病方面均有效果,且安全性高,药效明显,在天然姜黄素类化合物中,姜黄素比去甲氧基姜黄素和双去甲氧基姜黄素在抗氧化及修复DNA等方面具有更高的活性,是发挥药效的最主要成分,也是最具研究潜力的物质[7]。由于具有特殊的多酚结构,姜黄素类化合物还具有诸多其他有益生理活性,如:清除自由基、抗氧化、降低肝肾组织中的磷脂含量[8]、抑制细胞纤维化、减少亚硝酸类物质的生成、增强解毒蛋白、抗炎、抗氧化[9]、抗癌[10]、抗阿尔兹海默综合征[11]、抗帕金森病[12]活性等。此外,在化妆品领域,姜黄素可被用于染发剂和各种彩妆的制造。
目前,姜黄素的生产方法主要为植物提取法,但这种方法获得的姜黄素产量有限,后续分离过程昂贵、耗时。合成生物学作为一门新兴交叉学科,通过深度挖掘高效功能元件、重构代谢网络、优化元件与底盘的适配性,并对代谢网络流量进行精细调控,可以实现生物体的目标化设计、改造乃至重头合成,从而定制化细胞工厂来实现对药物、功能材料或化合物的生物合成及大规模生产。生物合成姜黄素,即在微生物底盘细胞中引入姜黄素生物合成的外源途径,通过将不同来源的酶编码基因整合起来,进而实现姜黄素的异源合成[13]。与植物提取法相比,生物合成姜黄素具有底物价格低廉、生产周期快、易于操作等优势[14-15]。此外,由于微生物中不存在姜黄素的竞争性代谢途径,因此也更容易实现大规模发酵和下游纯化[16]。综上所述,采用微生物底盘细胞生物合成姜黄素具有广阔的市场前景和应用潜力。
1 姜黄中姜黄素合成途径1.1 姜黄素合成途径挖掘姜黄素类化合物属于聚酮化合物,主要通过丙二酰辅酶A的中心碳单元将两个苯丙氨酸单元连接而成[17]。聚酮是一类具有独特生物活性和化学性质的分子,是潜在的药物、燃料和材料前体,可以通过聚酮合成酶(Polyketide synthases,PKS) 将相对简单的辅酶A (Coenzyme A,CoA) 前体添加到聚酮链上,进而环化产生不同类别的聚酮产物[18]。根据所催化的底物种类、中间体结构和分子内环化特点这3个特征,PKS主要可分为3种类型[19]。其中,Ⅰ型PKS为大型多功能蛋白,兼具酰基转移、硫酯化等多个催化结构域;Ⅱ型PKS是酮还原酶、环化酶等多个离散单功能酶的蛋白复合体[20];Ⅲ型PKS (Type Ⅲ polyketide synthase,T3PKS) 则为较小的二聚体蛋白(80–90 kDa)[19],其催化结构域能够同时行使链延伸和链环化的功能。Ramirez-Ahumada等[21]首先从姜黄和生姜的幼叶、茎和根茎中鉴定到了姜黄素合成途径中的相关基因,主要为植物次生代谢苯丙烷途径中的基因:苯丙氨酸解氨酶(Phenylalanine ammonia lyase,PAL) 基因(PAL);羟基肉桂酰辅酶A转移酶(Hydroxycinnamoyl-CoA transferase,HCT) 基因(HCT),包括p-香豆酰莽草酸转移酶(p-coumaroyl shikimate transferase,CST) 基因(CST)、咖啡酰莽草酸转移酶(Caffeoyl shikimate transferase,CaST) 基因(CaST)、阿魏酰莽草酸转移酶(Feruloyl shikimate transferase,FST) 基因(FST) 和p-香豆酰奎宁酸转移酶(p-coumaroyl quinate transferase,CQT) 基因(CQT);咖啡酸O-甲基转移酶(Affeic acid O-methyltransferase,COMT) 基因(COMT);咖啡酰辅酶A-O-甲基转移酶(Caffeoyl-CoA O-methyl-transferase,CCOMT) 基因(CCOMT)。为了进一步鉴定出姜黄素合成酶的关键基因,研究人员提取了姜黄中的蛋白,通过添加不同的底物和延伸中间体,结果发现,姜黄素蛋白提取物能够有效催化丙二酰-CoA与香豆酰-CoA或阿魏酰-CoA生成姜黄素类化合物,这一催化活性与聚酮合成酶的催化功能类似[21]。通过基因挖掘,Katsuyama等[17]从姜黄中鉴定出了两个参与姜黄素合成的T3PKS基因,即二酮-CoA合成酶(Diketide-CoA synthase,DCS) 基因(DCS) 和姜黄素合成酶1 (Curcumin synthase 1,CURS1) 基因(CURS1)。根据Katsuyam[17]和Ramirez- Ahumada[21]的工作,其推测姜黄中姜黄素生物合成途径如下:首先,苯丙氨酸在PAL的催化作用下生成肉桂酸,并由肉桂酸-4-羟化酶(Cinnamate-4- hydroxylase,C4H) 转化为香豆酸。进而,4-香豆酸-CoA连接酶(4-Coumarate-CoA ligase,4CL) 将香豆酸转化为香豆酰-CoA,再由CST和对香豆酰5-O-莽草酸3-羟化酶(p-coumaroyl 5-O-shikimate3'-hydroxylase,CS3'H) 转变生成咖啡酰-CoA,并由CCOMT转化生成阿魏酰-CoA。进一步地,阿魏酰-CoA或者香豆酰-CoA分别与丙二酰-CoA被DCS催化形成阿魏酰二酮-CoA或者香豆酰二酮-CoA,然后在姜黄素合成酶(Curcumin synthase,CURS) 的催化下,与香豆酰-CoA或阿魏酰-CoA缩合形成不同的类姜黄素化合物(图 1)。
 |
| 图 1 姜黄中的类姜黄素生物合成途径(改编自[17], [21]) Fig. 1 Curcuminoid biosynthetic pathway in Curcuma longa (adapted from [17], [21]). |
| 图选项 |
Sun等[22]以叶丛期、根茎膨大期和干物质积累期姜黄的根茎为实验材料,分析了3个发育时期姜黄素类代谢物的转录及代谢变化。研究发现,在植物姜黄素合成过程中,苯丙氨酸合成酶与姜黄素合成酶的表达变化一致,都表现为前2个发育阶段表达水平较低,干物质积累期表达较高,这与姜黄素类代谢物合成水平在干物质积累期最高的结果相一致。该研究为姜黄药效活性成分的形成机制研究提供基础,并为姜黄不同品种中姜黄素生物合成途径的解析以及候选基因的筛选提供了指导和参照。
1.2 姜黄素合成关键酶姜黄素生物合成的关键酶主要包括DCS和CURS。迄今为止,研究人员已从姜黄中鉴定出3种姜黄素合成酶CURSs (CURS1/2/3),其中,CURS1的蛋白结构已经得到解析。目前,普遍认为DCS和CURSs能够通过3步反应生成姜黄素类化合物:1) 起始底物阿魏酰-CoA或香豆酰-CoA能够与丙二酰-CoA在DCS的催化下形成阿魏酰二酮-CoA;2) 阿魏酰二酮-CoA在CURSs的催化下,水解成相应的酮酸;3) 酮酸在CURSs的催化下与另一分子的起始底物香豆酰-CoA或阿魏酰-CoA缩合形成不同的类姜黄素化合物。根据底物和延伸物的不同,类姜黄素化合物主要分为姜黄素,去甲氧基姜黄素和双去甲氧基姜黄素。序列比对结果显示,CURS2和CURS3与CURS1的序列相似度分别为78%和81%。通过比较3种酶的催化活性和底物特异性,研究人员发现,CURS1和CURS2主要催化阿魏酰-CoA合成姜黄素或去甲氧基姜黄素,而CURS3可分别以阿魏酰-CoA和p-香豆酰-CoA作为底物,催化合成姜黄素、去甲氧基姜黄素和双去甲氧基姜黄素[23]。正是由于3种CURSs的底物特异性和表达水平不同,姜黄可以合成产生不同的姜黄素类化合物,且不同化合物的产量也不同。
1.2.1 CURS1Katsuyama等[24]解析了CURS1的结构,发现其与典型的T3PKS结构类似,呈现出αβαβα的二聚体结构,每个单体中的催化三联体(Cys-His-Asn) 均位于各自狭长的CoA结合通道中(图 2)。通过酶动力学和生化分析以及与底物的分子对接研究,作者提出了CURS1的催化机制:1) CURS1将底物阿魏酰-CoA的阿魏酰基转移到催化三联体(Cys164-His303-Asn336) 的Cys-164上,与一分子的丙二酰-CoA形成二酮-CoA;2) 进入CoA结合通道的二酮-CoA被水解,产生的β-酮酸通过其苯基与CoA结合通道中Phe-265周围的疏水空腔结合,进而靠近催化三联体;3) β-酮酸在第二轮延伸中作为延伸底物与起始底物阿魏酰-CoA的阿魏酰基发生脱羧缩合,生成最终产物姜黄素。因此,CURS1并非直接催化酰基-CoA (起始底物) 与丙二酰-CoA (延伸底物) 之间发生脱羧缩合,而是通过催化延伸底物β-酮酸发生脱羧缩合后产生产物。与典型T3PKS的催化机理不同,CURS1并不遵循传统的聚酮头尾组装模型,而是通过催化聚酮链的"头对头"缩合生成产物。
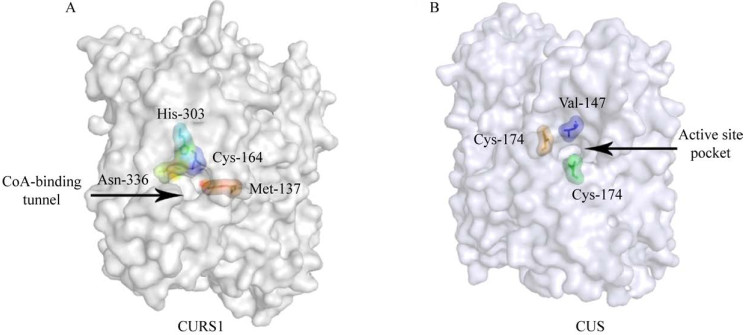 |
| 图 2 CURS1和CUS的整体结构 Fig. 2 Overall structures of CURS1 and CUS. (A) Surface representation for the overall structure of CURS1 (3OV2), the side chains of Cys-164, His-303, Asn-336, and Met-137 are depicted with sticks. (B) Surface representation for the overall structure of CUS (3OIT), with active sites Cys-174 and Val-147 shown with green sticks. Arrows indicate the active site pocket. |
| 图选项 |
1.2.2 CUS在鉴定出姜黄中的姜黄素合酶之前,Katsuyama等[25]通过基因挖掘,成功从水稻中鉴定出一种T3PKS,体外酶学实验表明该酶兼具DCS和CURS的功能,能够催化香豆酰-CoA与丙二酰-CoA向姜黄素的一步转化,于是将该酶命名为姜黄素合酶(Curcuminoid synthase,CUS)。CUS同样具有保守的催化三联体(Cys164-His303- Asn336),但与DCS和CURS1分别只有51%和45%的序列相似度。Miyazono等[26]解析了CUS的结构,发现其与CURS1类似,均呈现αβαβα的二聚体结构(图 2)。Morita等[27]根据CUS的结构提出了具体的一步式催化模型:1) CUS催化位点Cys-174邻近的水分子与Ser351-Asn142-H2O- Tyr207-Glu202形成的氢键网络通过活化水分子断裂CUS与香豆酰-CoA复合体中的硫酯键,从而终止二酮阶段的初始聚酮链延伸,产生香豆酰二酮酸;2) 该β-酮酸的α, β-不饱和羰基被Asn-142和Tyr-207重新固定朝向,酸性羰基与Ser351形成氢键后,被Cys-174夺取质子,从而形成含有羧酸阴离子的二酮中间体;3) 第2个香豆酰-CoA作为起始底物被添加到催化中心的Cys-174活性位点,并被活性中心的His-316激活而重新固定朝向,使得二酮中间体能够被同时容纳在活性中心腔向下拓展的口袋中;4) 被酶结合的香豆酰-CoA会重新调整朝向,实现与香豆酰二酮酸的"尾对尾"排布模式,并在Cys-174的催化下形成最终产物。在该催化机制中,CUS独特的向下拓展的活性口袋能够同时允许两分子香豆酰-CoA和一分子丙酰-CoA的存在,且催化中心附近独特的氢键网络Ser351-Asn142-H2O-Tyr207- Glu202使其能够通过与CURS1不同的催化机制形成C6-C7-C6的二苯基庚烷结构,并采用一种独特的"尾对尾"组装模式将二酮中间体(β-酮酸) 添加到羧酰-CoA上,从而独自完成DCS和CURS的两步催化反应。
2 姜黄素生物合成进展2.1 大肠杆菌中姜黄素生物合成进展组学技术、代谢工程和合成生物学的迅猛发展为姜黄素的生产提供了一种新的策略及技术手段。Katsuyama等[28]将CUS和苯丙烷途径整合在大肠杆菌中,首次成功实现了姜黄素类化合物的生物合成。在该重组途径中,来源于深红酵母的PAL可以催化L-苯丙氨酸形成肉桂酸,来源于紫草的4-香豆酰-CoA连接酶(4-coumarate-CoA ligase from Lithospermum erythrorhizon,Le4CL1) 能够将这些羧酸催化为相应的CoA硫酯,来自水稻的姜黄素合成酶CUS则可以将其进一步转化形成类姜黄素类化合物(图 3)。因PAL同时具有酪氨酸解氨酶(Tyrosine ammonia lyase,TAL) 的功能,该重组菌株也能够直接催化酪氨酸生成姜黄素。虽然,在大肠杆菌中过表达谷氨酸棒杆菌产生的乙酰辅酶A羧化酶(Acetyl-CoA carboxylase,ACC) 能够增加丙二酰-CoA的浓度,但当丙二酰-CoA的浓度远高于中间产物羧酰-CoA时,重组菌株会将羧酸-CoA大量转化生成副产物三酮吡喃酮(209 mg/L),导致双去甲氧基姜黄素的产量降低为53.4 mg/L。因此,研究人员构建了仅含4CL、CUS和ACC三个基因的重组菌株,并通过直接添加外源羧酸底物合成姜黄素类化合物。当分别利用p-香豆酸、肉桂酸和阿魏酸作为底物时,重组菌株能够分别产生91.23 mg/L的双去甲氧基姜黄素、84.15 mg/L的二桂酰基甲烷和113.22 mg/L的姜黄素,明显高于从头合成姜黄素的产量。在另一项研究中,Katsuyama等[29]通过添加不同的非天然羧酸前体,成功合成了17种非天然姜黄素类化合物,最高产量可达到109 mg/L,为新型化合物的生物合成提供了新的思路。
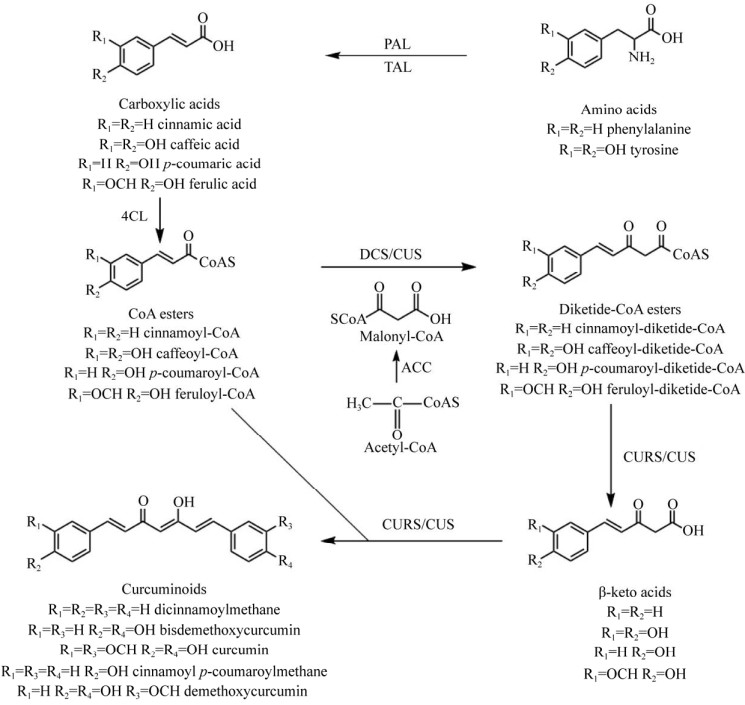 |
| 图 3 重组大肠杆菌中的类姜黄素生物合成途径(改编自[40]) Fig. 3 Biosynthesis of curcuminoids by recombinant Escherichia coli (adapted from [40]), using tyrosine and/or phenylalanine as starter substrates that are converted to carboxylic acids by PAL and TAL. The carboxylic acids, which can also be added directly to the medium, are converted into the corresponding CoA esters by 4CL, which is followed by several reactions catalyzed by DCS, CURSs and CUS. Malonyl-CoA is overproduced by ACC. |
| 图选项 |
Chu等[30]向CUS和欧芹来源的4CL共表达的重组大肠杆菌菌株中引入了ACC复合体(包括酰基-CoA羧化酶、生物素连接酶和乙酰-CoA合成酶),同时通过CRISPRi技术调节三羧酸循环和脂肪酸生物合成途径中相关基因的表达水平来抑制乙酰-CoA和丙二酰-CoA的降解,最终提高了细胞内丙二酰-CoA的水平。当以肉桂酸为底物时,该重组菌株能够在3 L发酵罐中产生41.94 μmol/L (约11.6 mg) 的二桂酰基甲烷。该研究表明CRISPRi技术明显优于传统的基因敲除技术,同时能够有效地降低姜黄素生物合成细胞的代谢负担。
通过对香豆酸合成相关基因进行调控,也能够有效提高姜黄素类化合物的产量。Wang等[31]利用姜黄素产物呈黄色的特点,设计了一种基于微板法的二桂酰基甲烷效价测定方法,并筛选了4种红车轴草来源的PAL (PAL1、PAL2、PAL3和PAL4),当将其分别与拟南芥来源的CoA连接酶At4CL1和水稻来源的CUS在大肠杆菌中共表达时,PAL1的表现最好,重组工程大肠杆菌可产生高达0.36 g/L的二桂酰基甲烷。Kim等[32]在大肠杆菌中分别引入了糖丝菌Saccharothrix espanaensis来源的TAL基因(SeTAL) 和拟南芥Arabidopsis thaliana来源的PAL基因(AtPAL),并与水稻来源的4CL和CUS重组进行姜黄素的合成,结果发现,TAL可以用于去甲氧基姜黄素、二桂酰基甲烷和肉桂酰基对香豆酰甲烷的合成,而PAL则更多地用于二桂酰基甲烷的合成。采用较高拷贝数的质粒过表达4CL和CUS,使其表达量高于TAL或PAL,能够减少底物肉桂酸或香豆酸的积累,从而减弱它们对TAL或PAL的抑制作用。进一步通过敲除tyrR和pheA基因,过表达SeTAL、Os4CL、OsCUS、aroG和tyrA,改造大肠中的莽草酸生物合成途径,作者成功提高了底物酪氨酸或苯丙氨酸的产量,最终含有SeTAL的合成途径的双去甲氧基姜黄素产量从0.32 mg/L提高到4.63 mg/L,而含有AtPAL的合成途径的二桂酰基甲烷产量从1.24 mg/L提高到6.95 mg/L。
虽然水稻来源的CUS具有催化合成姜黄素的功能,但水稻中尚未报道过姜黄素类化合物,猜测其产量可能极低。Rodrigues等[33]采用大肠杆菌比较了DCS、CURS1以及CUS合成姜黄素的能力,结果发现,当以香豆酸或阿魏酸作为前体时,过表达DCS和CURS1的大肠杆菌分别能够产生187.9 μmol/L的姜黄素和5 μmol/L的双去甲氧基姜黄素,而过表达CUS的菌株只能产生6.7 μmol/L的姜黄素和0.9 μmol/L的双去甲氧基姜黄素,这表明虽然CUS能够催化姜黄素的一步化合成,但在催化效率上不如DCS和CURS1;这也是DCS和CURS1成功用于姜黄素异源合成的首次报道;进而,他们采用粘红酵母来源的TAL、一种糖丝菌丝来源的4-香豆酸-3-羟化酶(C3H) 以及紫花苜蓿来源的咖啡酰-CoA-3-O-甲基转移酶(Caffeoyl-CoA 3-O-methyltransferase,CCoAOMT),成功重构了从咖啡酸到姜黄素的合成途径。当分别以咖啡酸、对香豆酸或酪氨酸为前体时,该菌株能够产生3.9 mg/L、0.3 mg/L和0.2 mg/L的姜黄素(表 1)。
表 1 姜黄素生物合成总结Table 1 Summary of curcumin biosynthesis
| Hosts | Strategy | Precursor | Products | Titer | References |
| E. coli | Using exogenous carboxylic acids as precursors | p-coumaric acid Cinnamic acid Ferulic acid. | Bisdemethoxycurcumin Dicinnamoylmethane Curcumin | 91.23 mg/L84.15 mg/L113.22 mg/L | [28] |
| E. coli | Using various analogs of p-coumaric acid as precursors | Analogs of p-coumaric acid | Curcuminoids | 109.8 mg/L | [29] |
| E. coli | Increasing the malonyl-CoA level | Cinnamic acid | Dicinnamoylmethane | 3.87 mg/L | [30] |
| E. coli | Screening efficient PAL for coumaric acid synthesis | Phenylalanine | Dicinnamoylmethane | 360 mg/L | [31] |
| E. coli | Increasing the precursors and reducing the accumulation of carboxylic acids | Glucose | Bisdemethoxycurcumin Dicinnamoylmethane | 4.63 mg/L6.95 mg/L | [32] |
| E. coli | Reconstructing of curcuminoids biosynthetic pathway using caffeic acid as a substrate | Ferulic acidCaffeic acid | Curcumin | 70 mg/L | [33] |
| E. coli | Balancing the metabolic flux | Glucose | Curcumin | (3.9±0.8) mg/L | [34] |
| E. coli | Optimizing the cultivation conditions | Ferulic acid | Curcumin | 353 mg/L | [36] |
| E. coli | Utilizing heat shock promoters to drive pathway genes expression | Ferulic acid | Curcumin | 0.017 mg/L | [37] |
| E. coli | Engineering de novo production of curcuminoids using E. coli co-cultures | Glucose | Bisdemethoxycurcumin | 6.28 mg/L | [38] |
| E. coli | Optimizing the plasmids combination and employing E. coli co-culture strategy | Ferulic acidTyrosineTyrosine | CurcuminTotal curcuminoidsCurcumin | 563.4 mg/L41.5 mg/L15.9 mg/L | [39] |
| Y. lipolytica | Increasing the malonyl-CoA level | p-coumaric acid | Bisdemethoxycurcumin | 0.17 mg/L | [43] |
| P. putida | Leveraging host metabolism | Coumarate | Bisdemethoxycurcumin | 2.5 mg/mL | [45] |
| A. oryzae | Strengthening the malonyl-CoA supply | Feruloyl-N-acetylcysteamine | Curcumin | 404 μg/plate | [49] |
表选项
对于多酶合成途径,代谢流的通量对于产物的最终产量非常重要。为了实现对大肠杆菌中姜黄素生物合成代谢通路的微调,Kang等[34]采用多重自动基因组编辑(Multiplex automatic genome engineering,MAGE) 工具对该途径中6种酶的基因5′-UTR进行了优化,通过调节不同酶的表达比例,最终筛选出8种产量较高的突变菌株,其中,6M08rv突变体中的姜黄素产量最高,比初始菌株提高38.2倍。通过对6M08rv突变体中不同酶的表达比例进行定量,结果发现,DCS和4CL的表达量远低于初始菌株(4CL在6M08rv和初始菌株中的翻译效率分别为0.5×10–3和191.1×10–3,DCS在6M08rv和初始菌株中的翻译效率分别为6.8×10–3和190.7×10–3),这暗示了阿魏酸的积累可能有助于姜黄素产量的提高。Li等[35]以姜黄的两种野生型品种和两种栽培品种为材料,采用RNA-seq技术分析基因转录水平并结合代谢产物产量分析,研究了姜黄素生物合成途径中不同酶的表达水平及其与产物代谢通量之间的关系。结果发现,DCS表达量较低时阿魏酰-CoA更容易发生积累,而CURS1和CURS2表达量较高时则会产生较多的姜黄素和去甲氧基姜黄素。此外,当DCS表达量较高时,姜黄中p-香豆酰-CoA和p-香豆酰二酮-CoA的代谢通量会高于阿魏酰-CoA的通量,从而导致去甲氧基姜黄素的产量高于姜黄素。该方法为提高姜黄素类化合物的产量提供了一种新思路(表 1)。
Couto等[36]评估和优化了姜黄素合成菌株的培养条件,包括菌株类型、异丙基β-D-硫代吡喃半乳糖苷(Isopropyl β-D-thiogalactopyranoside,IPTG) 浓度,蛋白表达诱导时间以及底物类型和浓度,结果发现,最适菌株为蛋白酶Lon和OmpT缺失的大肠杆菌BL21(DE3),当OD达到0.9时,加入0.1 mmol/L的IPTG进行诱导,最终获得的姜黄素产量最高可达959.3 μmol/L (353 mg/L) (LB+M9组合培养基),产率达到了95.9%。类似地,Rodrigues等[37]采用热休克启动子启动大肠杆菌中羟基肉桂酸和姜黄素的合成,通过测试不同的核糖体结合位点(Ribosome binding site,RBS),优化基因表达效率,最终提高了羟基肉桂酸的产量,其中,p-香豆酸产量最高达2.5 mmol/L,咖啡酸的最高产量则分别为370 μmol/L,这表明了热休克启动子在开发新型工艺方面的潜力。但是,该报道中姜黄素的最终产量并不高,仅为17 μmol/L,暗示了合成途径中代谢通量的平衡对于最终产量非常重要(表 1)。
构建重组途径时往往会给细胞产生一定的代谢负担,因此,Fang等[38]构建了大肠杆菌共培养系统,即利用一种大肠杆菌菌株从葡萄糖中合成对香豆酸,再利用另一种大肠杆菌菌株将对香豆酸转化为最终产物双去甲氧基姜黄素;此外,考虑到高水平的丙二酰-CoA对于双去甲氧基姜黄素的合成至关重要,他们还采用了不同策略以提高细胞内丙二酰-CoA的产量。与单培养策略相比,这套共培养系统能够用于葡萄糖快速转化生产姜黄素类化合物,并且获得的双去甲氧基姜黄素产量更高(6.28 mg/L)。这是首次采用共培养方法以葡萄糖为底物生产姜黄素,为姜黄素类高值化产品的制备提供了一种新思路。Rodrigues等[39]采用了类似的共培养策略从头合成姜黄素,即利用一株大肠杆菌从酪氨酸生产阿魏酸,同时采用另一株菌株将前者产生并释放到培养基中的阿魏酸(和/或香豆酸) 用于生产姜黄素(和/或其他姜黄素)。通过质粒组合优化,前者能产生高达257.3 mg/L的阿魏酸。当直接以阿魏酸作为底物时,后者能产生高达563.4 mg/L的姜黄素,这是迄今为止获得的最高的姜黄素产量。此外,与单一培养系统相比,共培养策略使得总的姜黄素类化合物的产量增加6倍(41.5 mg/L)。在接种比为2︰1的条件下,虽然总姜黄素类化合物的产量有所下降,但姜黄素产量达到了15.9 mg/L,比单一培养系统提高了160%。这些结果显示了模块化共培养工程在从头合成姜黄素类化合物中的巨大潜力(表 1)。
2.2 其他微生物底盘细胞中姜黄素生物合成进展2.2.1 耶氏解酯酵母中姜黄素生物合成进展酵母作为底盘细胞,除了遗传操作工具成熟之外,还具有很多独特而优越的特性,包括成熟的翻译后修饰功能,以及较强的抗逆特性和遗传稳定性。不同的酵母还具有特异的代谢通路,如酿酒酵母Saccharomyces cerevisiae能够产生较多的萜类前体物二甲基丙烯基二磷酸(Dimethylallyl pyrophosphate,DMAPP) 和异戊烯焦磷酸(Isopentenyl pyrophosphate,IPP),耶氏解酯酵母Yarrowia lipolytica则能积累大量的脂质。很多酵母因其食品级地位而被批准用于人体营养和制药领域,如酿酒酵母已被用于生产多种聚酮类化合物,如白藜芦醇、柚皮素和松茸等[41-42]。但是,目前姜黄素异源合成工作大部分是在大肠杆菌中完成的,尚未出现酿酒酵母用于姜黄素生物合成的相关报道。近年来,耶氏解酯酵母因其具有丰富的乙酰-CoA和丙二酰-CoA而逐渐受到研究人员的广泛关注。值得一提的是,Palmer等[43]通过在耶氏解酯酵母中表达CUS,同时利用β-氧化介导策略提高乙酰-CoA进而提高丙二酰-CoA的产量,最终通过外源添加4-香豆酸成功合成了双去甲氧基姜黄素。虽然最终产量仅有0.17 mg/L,但这是酵母底盘细胞用于姜黄素生物合成的首次报道。该研究同时测试了耶氏解酯酵母合成柚皮素和白藜芦醇的能力,其中,柚皮素的产量为目前所有微生物底盘细胞中最高值,表明对于4-香豆酰-CoA衍生物如聚酮类产物而言,耶氏解酯酵母是一种非常理想的底盘细胞(表 1)。
2.2.2 恶臭假单胞菌中姜黄素生物合成进展恶臭假单胞菌Pseudomonas putida是一种腐生细菌,具有多样化的碳代谢网络和较强的耐溶剂性,是代谢工程的理想宿主[44]。此外,恶臭假单胞菌基因组编码了多种蛋白和酶类,可以直接用于合成有价值的工业化产品。Incha等[45]通过利用恶臭假单胞菌内源的阿魏酰/香豆酰-CoA合成酶(Feruloyl/coumaroyl-CoA synthetase,Fcs) 合成香豆酰-CoA,同时敲除内源烯酰辅酶A水合裂解酶(Enoyl-CoA hydratase lyase,Ech) 抑制香豆酰-CoA的降解,并在水稻O. sativa来源的CUS的作用下,通过外源添加香豆酸,成功合成了双去甲氧基姜黄素。虽然最终产量只有2.15 mg/mL,远远低于在大肠杆菌菌株中生产姜黄素类化合物的产量,但有研究表明恶臭假单胞菌菌株可以生产高产量的苯丙烷类化合物[46-47],因此未来可以通过共培养策略来生产姜黄素类化合物(表 1)。
2.2.3 米曲霉中姜黄素生物合成进展长期以来,丝状真菌米曲霉Aspergillus oryzae一直用于酱油、清酒和味噌等发酵食品的生产,被美国食品药品监督管理局认证为"公认安全"的微生物[48]。米曲霉背景清晰,其本身很少表达次生代谢产物,是生产植物次生代谢化合物的理想底盘细胞,但目前采用米曲霉生产植物次生代谢物的报道却很少。Kan等[49]通过在米曲霉中过表达CUS,同时提高丙二酰-CoA的水平,最终在含有阿魏酰-N-乙酰半胱胺(Feruloyl-N-acetylcysteamine,Feruloyl-NAC) 的琼脂培养基上成功合成了姜黄素类似物,最高产量达404 μg/皿。为了提高米曲霉中丙二酰-CoA的水平,作者过表达了ACC合成酶,但效果并不明显,而其采用的另外两种策略则对产物的产量贡献明显:1) 敲除米曲霉中与酿酒酵母同源的能够抑制ACC活性的酶基因SnfA,从而提高ACC催化乙酰-CoA生成丙二酰-CoA的能力;2) 敲除米曲霉中甾醇生物合成相关基因SCAP,通过抑制乙酰-CoA下游的竞争性途径从而减少乙酰-CoA的消耗量。这两种策略有效提高了米曲霉中的丙二酰-CoA供应量。该研究也是米曲霉作为底盘细胞用于姜黄素生物合成的首次报道,其snfA SCAP双缺失突变体有望成为其他聚酮类化合物异源生产的有效宿主,为米曲霉异源合成聚酮类化合物提供了较好的基础(表 1)。
2.3 微生物转化法合成姜黄素衍生物微生物转化法即通过微生物细胞中功能各异的酶对目标化合物的结构进行修饰,从而获得各类衍生物。Peng等[50]在姜黄的根部寄生真菌中发现了一种纤维状的腐皮壳菌属Diaporthe sp. ,该菌能够将姜黄素转化为去甲氧基姜黄素及双去甲氧基姜黄素。张维宇[51]发现阿兹威毕赤酵母Pichia kudriavzevii能够将底物姜黄素有效地转化为四氢姜黄素及六氢姜黄素,其转化率可分别达77.43%及47.89%。罗阳春[52]利用红球菌Rhodococcus sp. 对姜黄素进行生物转化,可生成六氢姜黄素及八氢姜黄素,其转化率可分别达59.89%及74.48%。这些研究极大地丰富了目前姜黄素类化合物的种类。
3 姜黄素类化合物生物合成前景与展望姜黄素不仅能够作为食品添加剂用于食品领域,且因具有抗炎和降胆固醇活性而被认为可以用于治疗帕金森病、阿尔茨海默症等疾病,目前正处于临床试验阶段。此外,姜黄素还可以作为一种很有前途的木质素替代单体。Oyarce等[53]通过异源表达姜黄来源的DCS和CURS2,成功将姜黄素生物合成途径导入模式植物拟南芥并合成了二阿魏酰甲烷,从而提高了拟南芥的糖化效率。因此,姜黄素类化合物的生物合成研究可以为生物能源作物的优化提供一种新策略。
随着姜黄素类化合物的市场需求增加,其生物合成逐渐受到了国内外的广泛关注。近十多年来,姜黄中类姜黄素生物合成途径及其在微生物底盘细胞中的异源合成研究取得了一定的进展,但整体产量还处于较低的水平。目前,亟待解决的主要问题包括:产物对微生物底盘细胞的毒性、合成途径代谢流的不平衡、姜黄素合成酶的低活性以及姜黄素产物的混杂性。未来,可以从如下几个层面尝试提高姜黄素类化合物的产量和种类:1) 增加底盘细胞的前体物产量。可以采用高产酪氨酸或者香豆酸等前体物质的菌株作为出发菌株进行后续途径改造。此外,建立姜黄素特异的生物传感器或采用其他T3PK型产物如柚皮素的生物传感器作为输出,对重组菌株的突变体库进行高通量筛选,将有助于获得更加优化的芳香羧酸和丙二酰-CoA代谢通量,相关的有益突变也可以用于进一步的整合改造。2) 平衡姜黄素合成途径代谢流。可以通过系统性调控技术进行代谢流的调控,如基于自主群体感应回路(Autonomous quorum-sensing (QS)-based circuits) 技术进行生物质积累和产物合成的分区调控,或建立合适的生物传感器进行动态调控。也可以通过共培养策略等模块化设计降低细胞代谢负担,间接提高代谢通量。3) 提高姜黄素合成酶的特异性和活性。姜黄素合成酶作为关键限速酶,其催化活性和特
异性直接决定了姜黄素类化合物生物合成的产物效率和种类。通过结构生物学对不同的姜黄素合成酶进行解析,有助于理解其催化机制和底物特异性,从而可以通过理性或半理性设计等蛋白质工程改造方法对酶进行定向改造,减少类似结构的"副产物"积累,实现目的产物的高产。此外,通过"生物勘探"技术从不同物种中挖掘潜在的姜黄素合成酶,或者可以通过组学技术研究不同品种甚至不同物种中的姜黄素产量和种类,有望鉴定出活性更高或特异性更强的姜黄素合成酶。4) 增强底盘细胞对产物的耐受性。研究发现,p-香豆酸和咖啡酸对大肠杆菌具有毒性抑制作用,能够降低细胞的生长率[54]。姜黄素则能够通过铁离子螯合抑制酿酒酵母的生长[55]。因此,克服或降低产物对底盘细胞的毒性将有助于提高最终产量。通过实验室适应进化(Adaptive laboratory evolution,ALE)、高通量筛选、下一代测序以及组学技术的结合,研究相关代谢产物或终产物的毒性作用机制,将有助于构建鲁棒性更高的底盘细胞。此外,深入研究产物的分泌机制,通过设计转运蛋白和产物即时分离技术,亦有助于降低产物对细胞的毒性作用。而采用无细胞体系也是克服产物毒性提高多酶途径反应效率的一种有效方法。5) 扩展姜黄素类化合物的产物多样性。结合不同姜黄素合成酶的底物特异性,向重组菌株提供不同种类的前体化合物,有望获得不同种类甚至非天然的姜黄素化合物。6) 探究和改造合适的微生物底盘细胞。不同的底盘细胞优势不同,非模式生物因具有特定的代谢网络而受到研究人员的广泛关注[56]。随着CRISPR-Cas9等合成生物学工具的快速发展和完善,对不同的底盘细胞甚至非模式生物的基因组编辑已逐渐成为可能。因此,对大肠杆菌之外的其他底盘细胞如解酯酵母这种富含前体物丙二酰-CoA的非模式生物进行深入研究,将有望建立更合适的姜黄素合成底盘细胞,加快姜黄素类化合物的生物合成产业化。综上所述,采用合成生物学策略合成姜黄素类化合物,有望成为植物源姜黄素类化合物生产的有效技术手段。
参考文献
| [1] | 李犇, 李静, 钱海, 等. 姜黄素对金属毒性的抑制作用及机制研究进展. 卫生研究, 2011, 40(3): 399-403. Li B, Li J, Qian H, et al. Progress in the inhibition of curcumin on metal toxicity and its mechanism. J Hyg Res, 2011, 40(3): 399-403 (in Chinese). |
| [2] | Wang YJ, Pan MH, Cheng AL, et al. Stability of curcumin in buffer solutions and characterization of its degradation products. J Pharm Biomed Anal, 1997, 15(12): 1867-1876. DOI:10.1016/S0731-7085(96)02024-9 |
| [3] | 王雪梅, 陈利华, 施文婷. 姜黄素类化合物的光稳定性研究. 安徽大学学报(自然科学版), 2012, 36(3): 73-78. Wang XM, Chen LH, Shi WT. Study on the photo-stability of curcuminoid. J Anhui Univ (Nat Sci), 2012, 36(3): 73-78 (in Chinese). DOI:10.3969/j.issn.1000-2162.2012.03.013 |
| [4] | Sharma RA, Gescher AJ, Steward WP. Curcumin: the story so far. Eur J Cancer, 2005, 41(13): 1955-1968. DOI:10.1016/j.ejca.2005.05.009 |
| [5] | Kumar TP, Antony S, Gireesh G, et al. Curcumin modulates dopaminergic receptor, CREB and phospholipase C gene expression in the cerebral cortex and cerebellum of streptozotocin induced diabetic rats. J Biomed Sci, 2010, 17: 43. DOI:10.1186/1423-0127-17-43 |
| [6] | Gandapu U, Chaitanya RK, Kishore G, et al. Curcumin-loaded apotransferrin nanoparticles provide efficient cellular uptake and effectively inhibit HIV-1 replication in vitro. PLoS ONE, 2011, 6(8): e23388. DOI:10.1371/journal.pone.0023388 |
| [7] | Ahsan H, Parveen N, Khan NU, et al. Pro-oxidant, anti-oxidant and cleavage activities on DNA of curcumin and its derivatives demethoxycurcumin and bisdemethoxycurcumin. Chem Biol Interact, 1999, 121(2): 161-175. DOI:10.1016/S0009-2797(99)00096-4 |
| [8] | Kim M, Kim Y. Hypocholesterolemic effects of curcumin via up-regulation of cholesterol 7a-hydroxylase in rats fed a high fat diet. Nutr Res Pract, 2010, 4(3): 191-195. DOI:10.4162/nrp.2010.4.3.191 |
| [9] | Motterlini R, Foresti R, Bassi R, et al. Curcumin, an antioxidant and anti-inflammatory agent, induces heme oxygenase-1 and protects endothelial cells against oxidative stress. Free Radic Biol Med, 2000, 28(8): 1303-1312. DOI:10.1016/S0891-5849(00)00294-X |
| [10] | Yallapu MM, Ebeling MC, Khan S, et al. Novel curcumin-loaded magnetic nanoparticles for pancreatic cancer treatment. Mol Cancer Ther, 2013, 12(8): 1471-1480. DOI:10.1158/1535-7163.MCT-12-1227 |
| [11] | Ringman JM, Frautschy SA, Cole GM, et al. A potential role of the curry spice curcumin in Alzheimer's disease. Curr Alzheimer Res, 2005, 2(2): 131-136. DOI:10.2174/1567205053585882 |
| [12] | Pan J, Li H, Ma JF, et al. Curcumin inhibition of JNKs prevents dopaminergic neuronal loss in a mouse model of Parkinson's disease through suppressing mitochondria dysfunction. Transl Neurodegener, 2012, 1: 16. DOI:10.1186/2047-9158-1-16 |
| [13] | Jeandet P, Delaunois B, Aziz A, et al. Metabolic engineering of yeast and plants for the production of the biologically active hydroxystilbene, resveratrol. J Biomed Biotechnol, 2012, 2012: 579089. |
| [14] | Horinouchi S. Combinatorial biosynthesis of plant medicinal polyketides by microorganisms. Curr Opin Chem Biol, 2009, 13(2): 197-204. DOI:10.1016/j.cbpa.2009.02.004 |
| [15] | Horinouchi S. Combinatorial biosynthesis of non-bacterial and unnatural flavonoids, stilbenoids and curcuminoids by microorganisms. J Antibiot (Tokyo), 2008, 61(12): 709-728. DOI:10.1038/ja.2008.85 |
| [16] | Chemler JA, Koffas MAG. Metabolic engineering for plant natural product biosynthesis in microbes. Curr Opin Biotechnol, 2008, 19(6): 597-605. DOI:10.1016/j.copbio.2008.10.011 |
| [17] | Katsuyama Y, Kita T, Funa N, et al. Curcuminoid biosynthesis by two type Ⅲ polyketide synthases in the herb Curcuma longa. J Biol Chem, 2009, 284(17): 11160-11170. DOI:10.1074/jbc.M900070200 |
| [18] | Palmer CM, Alper HS. Expanding the chemical palette of industrial microbes: metabolic engineering for type Ⅲ PKS-derived polyketides. Biotechnol J, 2019, 14(1): e1700463. DOI:10.1002/biot.201700463 |
| [19] | Shimizu Y, Ogata H, Goto S. Discriminating the reaction types of plant type Ⅲ polyketide synthases. Bioinformatics, 2017, 33(13): 1937-1943. DOI:10.1093/bioinformatics/btx112 |
| [20] | Grininger M. The role of the iterative modules in polyketide synthase evolution. Proc Natl Acad Sci USA, 2020, 117(16): 8680-8682. DOI:10.1073/pnas.2004190117 |
| [21] | Ramirez-Ahumada MDC, Timmermann BN, Gang DR. Biosynthesis of curcuminoids and gingerols in turmeric (Curcuma longa) and ginger (Zingiber officinale): identification of curcuminoid synthase and hydroxycinnamoyl-CoA thioesterases. Phytochemistry, 2006, 67(18): 2017-2029. DOI:10.1016/j.phytochem.2006.06.028 |
| [22] | Sun JR, Bu JL, Cui GH, et al. Accumulation and biosynthetic of curcuminoids and terpenoids in turmeric rhizome in different development periods. China J Chin Mat Med, 2019, 44(5): 927-934. |
| [23] | Katsuyama Y, Kita T, Horinouchi S. Identification and characterization of multiple curcumin synthases from the herb Curcuma longa. FEBS Lett, 2009, 583(17): 2799-2803. DOI:10.1016/j.febslet.2009.07.029 |
| [24] | Katsuyama Y, Miyazono KI, Tanokura M, et al. Structural and biochemical elucidation of mechanism for decarboxylative condensation of β-keto acid by curcumin synthase. J Biol Chem, 2011, 286(8): 6659-6668. DOI:10.1074/jbc.M110.196279 |
| [25] | Katsuyama Y, Matsuzawa M, Funa N, et al. In vitro synthesis of curcuminoids by type Ⅲ polyketide synthase from Oryza sativa. J Biol Chem, 2007, 282(52): 37702-37709. DOI:10.1074/jbc.M707569200 |
| [26] | Miyazono K, Um J, Imai FL, et al. Crystal structure of curcuminoid synthase CUS from Oryza sativa. Proteins, 2011, 79(2): 669-673. DOI:10.1002/prot.22888 |
| [27] | Morita H, Wanibuchi K, Nii H, et al. Structural basis for the one-pot formation of the diarylheptanoid scaffold by curcuminoid synthase from Oryza sativa. Proc Natl Acad Sci USA, 2010, 107(46): 19778-19783. DOI:10.1073/pnas.1011499107 |
| [28] | Katsuyama Y, Matsuzawa M, Funa N, et al. Production of curcuminoids by Escherichia coli carrying an artificial biosynthesis pathway. Microbiology, 2008, 154(Pt 9): 2620-2628. |
| [29] | Katsuyama Y, Hirose Y, Funa N, et al. Precursor-directed biosynthesis of curcumin analogs in Escherichia coli. Biosci Biotechnol Biochem, 2010, 74(3): 641-645. DOI:10.1271/bbb.90866 |
| [30] | Chu LL, Pandey RP, Dhakal D, et al. Increased production of dicinnamoylmethane via improving cellular malonyl-CoA level by using a CRISPRi in Escherichia coli. Appl Biochem Biotechnol, 2020, 190(1): 325-340. DOI:10.1007/s12010-019-03206-8 |
| [31] | Wang SY, Zhang SW, Zhou T, et al. Design and application of an in vivo reporter assay for phenylalanine ammonia-lyase. Appl Microbiol Biotechnol, 2013, 97(17): 7877-7885. DOI:10.1007/s00253-013-5122-4 |
| [32] | Kim EJ, Cha MN, Kim BG, et al. Production of curcuminoids in engineered Escherichia coli. J Microbiol Biotechnol, 2017, 27(5): 975-982. DOI:10.4014/jmb.1701.01030 |
| [33] | Rodrigues JL, Araujo RG, Prather KLJ, et al. Production of curcuminoids from tyrosine by a metabolically engineered Escherichia coli using caffeic acid as an intermediate. Biotechnol J, 2015, 10(4): 599-609. DOI:10.1002/biot.201400637 |
| [34] | Kang SY, Heo KT, Hong YS. Optimization of artificial curcumin biosynthesis in E. coli by randomized 5'-UTR sequences to control the multienzyme pathway. ACS Synth Biol, 2018, 7(9): 2054-2062. DOI:10.1021/acssynbio.8b00198 |
| [35] | Li DH, Ono N, Sato T, et al. Targeted integration of RNA-Seq and metabolite data to elucidate curcuminoid biosynthesis in four Curcuma Species. Plant Cell Physiol, 2015, 56(5): 843-851. DOI:10.1093/pcp/pcv008 |
| [36] | Couto MR, Rodrigues JL, Rodrigues LR. Optimization of fermentation conditions for the production of curcumin by engineered Escherichia coli. J R Soc Interface, 2017, 14(133): 20170470. DOI:10.1098/rsif.2017.0470 |
| [37] | Rodrigues JL, Couto MR, Araújo RG, et al. Hydroxycinnamic acids and curcumin production in engineered Escherichia coli using heat shock promoters. Biochem Eng J, 2017, 125: 41-49. DOI:10.1016/j.bej.2017.05.015 |
| [38] | Fang Z, Jones JA, Zhou JW, et al. Engineering Escherichia coli co-Cultures for production of curcuminoids from glucose. Biotechnol J, 2018, 13(5): e1700576. DOI:10.1002/biot.201700576 |
| [39] | Rodrigues JL, Gomes D, Rodrigues LR. A combinatorial approach to optimize the production of curcuminoids from tyrosine in Escherichia coli. Front Bioeng Biotechnol, 2020, 8: 59. DOI:10.3389/fbioe.2020.00059 |
| [40] | Rodrigues JL, Prather KLJ, Kluskens LD, et al. Heterologous production of curcuminoids. Microbiol Mol Biol Rev, 2015, 79(1): 39-60. DOI:10.1128/MMBR.00031-14 |
| [41] | Katsuyama Y, Ohnishi Y, Horinouchi S. Production of dehydrogingerdione derivatives in Escherichia coli by exploiting a curcuminoid synthase from Oryza sativa and a β-oxidation pathway from Saccharomyces cerevisiae. ChemBioChem, 2010, 11(14): 2034-2041. DOI:10.1002/cbic.201000379 |
| [42] | Trantas E, Panopoulos N, Ververidis F. Metabolic engineering of the complete pathway leading to heterologous biosynthesis of various flavonoids and stilbenoids in Saccharomyces cerevisiae. Metab Eng, 2009, 11(6): 355-366. DOI:10.1016/j.ymben.2009.07.004 |
| [43] | Palmer CM, Miller KK, Nguyen A, et al. Engineering 4-coumaroyl-CoA derived polyketide production in Yarrowia lipolytica through a β-oxidation mediated strategy. Metab Eng, 2020, 57: 174-181. DOI:10.1016/j.ymben.2019.11.006 |
| [44] | Nikel PI, de Lorenzo V. Pseudomonas putida as a functional chassis for industrial biocatalysis: from native biochemistry to trans-metabolism. Metab Eng, 2018, 50: 142-155. DOI:10.1016/j.ymben.2018.05.005 |
| [45] | Incha MR, Thompson MG, Blake-Hedges JM, et al. Leveraging host metabolism for bisdemethoxycurcumin production in Pseudomonas putida. Metab Eng Commun, 2020, 10: e00119. DOI:10.1016/j.mec.2019.e00119 |
| [46] | Nijkamp K, van Luijk N, de Bont JAM, et al. The solvent-tolerant Pseudomonas putida S12 as host for the production of cinnamic acid from glucose. Appl Microbiol Biotechnol, 2005, 69(2): 170-177. DOI:10.1007/s00253-005-1973-7 |
| [47] | Nijkamp K, Westerhof RGM, Ballerstedt H, et al. Optimization of the solvent-tolerant Pseudomonas putida S12 as host for the production of p-coumarate from glucose. Appl Microbiol Biotechnol, 2007, 74(3): 617-624. DOI:10.1007/s00253-006-0703-0 |
| [48] | Kitamoto K. Molecular biology of the Koji molds. Adv Appl Microbiol, 2002, 51: 129-153. |
| [49] | Kan E, Katsuyama Y, Maruyama JI, et al. Production of the plant polyketide curcumin in Aspergillus oryzae: strengthening malonyl-CoA supply for yield improvement. Biosci Biotechnol Biochem, 2019, 83(7): 1372-1381. DOI:10.1080/09168451.2019.1606699 |
| [50] | 彭清忠, 陈玲, 易浪波, 等. 内生真菌对姜黄素的微生物转化. 生物技术通讯, 2010, 21(2): 196-199. Peng QZ, Chen L, Yi LB, et al. Transformation of curcumin by endophytic fungus Diaporthe sp. isolated from Curcuma Longa. Lett Biotechnol, 2010, 21(2): 196-199 (in Chinese). |
| [51] | 张维宇. 姜黄素微生物转化产物的研究[D]. 杭州: 浙江工业大学, 2013. Zhang WY. Microbial transformation of curcumin to its derivatives[D]. Hangzhou: Zhejiang University of Technology, 2013 (in Chinese). |
| [52] | 罗杨春. 微生物转化法制备姜黄素衍生物的研究[D]. 杭州: 浙江工业大学, 2014. Luo YC. Microbial transformation of curcumin into its derivatives[D]. Hangzhou: Zhejiang University of Technology, 2014 (in Chinese). |
| [53] | Oyarce P, de Meester B, Fonseca F, et al. Introducing curcumin biosynthesis in Arabidopsis enhances lignocellulosic biomass processing. Nat Plants, 2019, 5(2): 225-237. DOI:10.1038/s41477-018-0350-3 |
| [54] | Huang Q, Lin YH, Yan YJ. Caffeic acid production enhancement by engineering a phenylalanine over-producing Escherichia coli strain. Biotechnol Bioeng, 2013, 110(12): 3188-3196. DOI:10.1002/bit.24988 |
| [55] | Minear S, O'Donnell AF, Ballew A, et al. Curcumin inhibits growth of Saccharomyces cerevisiae through iron chelation. Eukaryotic Cell, 2011, 10(11): 1574-1581. DOI:10.1128/EC.05163-11 |
| [56] | Sun LC, Alper HS. Non-conventional hosts for the production of fuels and chemicals. Curr Opin Chem Biol, 2020, 59: 15-22. DOI:10.1016/j.cbpa.2020.03.004 |
