
 , 汤青林1,2, 魏大勇1,2, 王志敏1,2
, 汤青林1,2, 魏大勇1,2, 王志敏1,2

1. 西南大学 园艺园林学院 南方山地园艺学教育部重点实验室,重庆 400715;
2. 重庆市蔬菜学重点实验室,重庆 400715
收稿日期:2019-08-20;接收日期:2019-11-04;网络出版时间:2019-11-27
基金项目:国家自然科学基金(No. 31501756),中央高校基本科研业务费专项(No. XDJK2018B039),重庆市基础研究与前沿探索专项面上项目(No. cstc2019jcyj-msxmX0448)资助
摘要:DNA甲基化是表观遗传学的一种重要修饰形式,也是一种重要的基因表达调控机制。DNA甲基化的异常模式可导致植物生长发育异常。文中从植物DNA甲基化模式入手,对DNA甲基化在调控基因表达和维持基因组稳定性的分子功能、DNA甲基化在植物发育、参与植物对生物和非生物胁迫的反应等方面的相关研究进行回顾和总结,为深入了解DNA甲基化的作用机制并将DNA甲基化应用于植物新品种的培育和遗传改良研究提供一定的参考。
关键词:DNA甲基化分子功能植物生长胁迫反应
Advances in research on the mechanism of DNA methylation in plants
Chao Yuan1,2, Shaowei Zhang1,2, Yi Niu1,2

 , Qinglin Tang1,2, Dayong Wei1,2, Zhimin Wang1,2
, Qinglin Tang1,2, Dayong Wei1,2, Zhimin Wang1,2

1. Key Laboratory of Horticulture Science for Southern Mountainous Regions Ministry of Education, College of Horticulture and Landscape Architecture, Southwest University, Chongqing 400715, China;
2. Chongqing Key Laboratory of Olericulture, Chongqing 400715, China
Received: August 20, 2019; Accepted: November 4, 2019; Published: November 27, 2019
Supported by: National Natural Science Foundation of China (No. 31501756), Special Funds for Basic Scientific Research Business in Central Colleges and Universities (No. XDJK2018B039), Chongqing Foundation Research and Frontier Exploration Project (No. cstc2019jcyj-msxmX0448)
Corresponding author: Yi Niu. Tel/Fax: +86-23-68250731; E-mail: Niuy2001134@163.com;
Zhimin Wang. Tel/Fax: +86-23-68250452; E-mail: minzniwang_555@163.com.
Abstract: DNA methylation is an epigenetic modification that forms an important regulation mechanism of gene expression in organisms across kingdoms. Aberrant patterns of DNA methylation can lead to plant developmental abnormalities. In this article, we briefly discuss DNA methylation in plants and summarize its functions and biological roles in regulating gene expression and maintaining genomic stability, plant development, as well as plant responses to biotic and abiotic stresses. We intended to provide a concise reference for further understanding of the mechanism of DNA methylation and potential applications of epigenetic manipulation for crop improvement.
Keywords: DNA methylationmolecular functionplant growthstress response
DNA甲基化作为一种保守的表观遗传修饰方式,是生物基因组中普遍存在的共价修饰方式,可以在不改变DNA分子一级结构的情况下调节基因组的功能,在基因调控和基因组稳定性中发挥着重要作用。大量研究表明,DNA甲基化是生物体中非常重要的调控模式,其在基因表达、转座子沉默、染色质相互作用、细胞分化以及生长发育过程中起着重要作用。植物DNA甲基化模式的改变不仅可以影响植物的花期、育性、花以及叶的形态等生命活动,而且在植物印记、逆境胁迫和杂种优势等方面也发挥着一定的作用[1]。本文就近些年来DNA甲基化在植物生长中的作用研究进行总结,从而为深入研究DNA甲基化在植物生长发育中的分子调控机制提供一些参考。
1 植物DNA甲基化模式DNA甲基化通常指DNA复制后,在DNA甲基转移酶(DNA methyltransferase,DNMT)的作用下,将S-腺苷甲硫氨酸(SAM)分子上的甲基(CH3-)转移到DNA的CpG两个核苷酸的胞嘧啶上进行DNA甲基化修饰,形成5-甲基胞嘧啶(5-mC)、N6-甲基腺嘌呤(N6-m A)以及7-甲基鸟嘌呤(7-mG)[2]。目前发现的植物甲基化主要有从头甲基化、维持甲基化两种模式。
1.1 从头甲基化从头甲基化(De novo methylation)是指在甲基化转移酶的作用下,不依赖已有的甲基化DNA链而在一个新位点将DNA链中的胞嘧啶C5甲基化。在植物中,从头甲基化是通过RNA指导的DNA甲基化(RdDM)途径介导,涉及到小干扰RNA (siRNA)、支架RNA (scaffold RNA)和一系列蛋白质(图 1)[3]。在RdDM途径中,首先在RNA聚合酶Ⅳ (POL Ⅳ)的参与下富集转座子和重复元件相关区域形成单链RNA (ssRNA),之后RNA聚合酶RDR2以之为模板合成双链RNA (dsRNA),最后被DCL3 (DICER-LIKE PROTEIN 3)切割产生24 nt的siRNA。HEN1 (HUA-ENHANCER 1)是一个RNA甲基转移酶,通过对dsRNA的3′末端的2′-羟基进行甲基化修饰,可防止dsRNA被其他核酸酶降解[4],随后成熟的24 nt siRNA与AGO4 (ARGONAUTE 4)或AGO6结合,并与POLⅤ转录的支架RNA配对并募集结构域重排甲基转移酶DRM2 (DOMAINS REARRANGED METHYLASE 2),从而对siRNAs同源的基因组序列进行甲基化修饰。
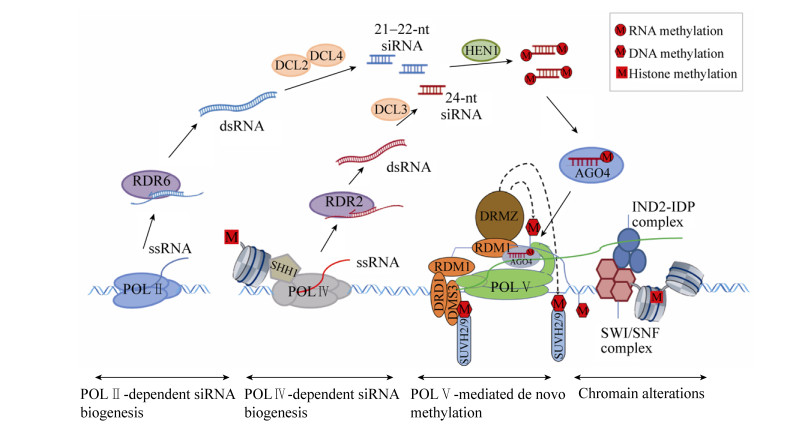 |
| 图 1 植物中RNA介导的DNA甲基化途径的模型 Fig. 1 RNA-directed DNA methylation pathway model in plants. |
| 图选项 |
近期研究发现,除了典型的POL Ⅳ-RDR2- DCL3途径产生24 nt siRNA外,POL Ⅳ的旁系同源物POL Ⅱ也能够生成siRNA触发非典型的RdDM (图 1)。POL Ⅱ介导的转录不仅可以产生24 nt siRNA和支架RNA,并且能够通过募集POL Ⅳ和POLⅤ在RdDM靶基因位点产生siRNA[5]。对于反式激活siRNA基因和一些转录激活转座子区域,RdDM依赖于POL Ⅱ和RDR6而不是POL Ⅳ和RDR2[6-8]。由此可见,RdDM是一个复杂的调控网络。
1.2 维持甲基化维持甲基化是指在甲基化DNA半保留复制产生的新生链的相应位置上进行的甲基化修饰,且新生链仅在与亲本链甲基化位置相同的碱基位置发生甲基化。植物DNA甲基化修饰主要发生在CG、CHG及CHH (H为A、T和G) 3种序列中。
植物DNA甲基化的维持取决于胞嘧啶序列背景,由不同调节机制的DNA甲基转移酶催化。MET1(METHYLTRANSFERASE 1)是最先发现的植物甲基化转移酶,维持基因编码区域中的CG甲基化。CMT为植物特有的甲基转移酶。拟南芥中CHG甲基化的维持主要由DNA甲基转移酶CMT3 (Chromomethylase 3)催化,一定程度上也由CMT2催化[9-10]。此外,当主要负责维持CHG甲基化的CMT3和主要负责H3K9二甲基化作用的SUVH4 (又称KYP,组蛋白甲基转移酶)丢失时会导致DNA甲基化水平显著下降[11]。然而CMT3和SUVH4两种蛋白是如何相互作用来维持CHG位点甲基化的分子机理目前还不清楚。研究发现DRM2和CMT2维持CHH甲基化,DRM2通过RdDM途径维持RdDM靶区域的CHH甲基化,这些区域优先位于在进化上较新的转座子、短转座子以及常染色体臂中的其他重复序列和异染色质中长转座子的边缘[12]。相反,CMT2则催化含有组蛋白H1的异染色质的CHH甲基化。也有研究表明,不对称甲基化的维持也可能受MET1和CMT3的影响,因为MET1维持的甲基化被SUVH2和SUVH9识别后会在RdDM位点募集POL Ⅴ[13],而CMT3维持的CHG甲基化增加H3K9me2水平,促进CMT2催化的非CG甲基化[10]。
1.3 去甲基化DNA甲基化是一种可逆的表观遗传修饰。植物基因组发生去甲基化作用可以激活处于沉默状态的基因。DNA去甲基化分为被动和主动两种去甲基化方式。DNA被动去甲基化依赖于DNA的半保留复制,是当DNA甲基化转移酶的活性受到抑制或浓度偏低时,原有的甲基化胞嘧啶被未甲基化胞嘧啶代替,DNA甲基化水平降低的过程。主动去甲基化是由DNA糖基化酶/裂解酶参与的特殊酶促反应,植物基因组上的5mC可以由DNA糖基化酶/裂解酶ROS1家族蛋白介导切除,之后再由碱基修复机制合成非甲基化胞嘧啶,从而造成基因组的DNA去甲基化[14]。为了鉴定新的DNA去甲基化因子,Nie等[15]通过正向遗传筛选体系鉴定到染色质重塑SWR1复合体中的两个组分蛋白ARP6和PIE1,以及一些已知的DNA去甲基化因子,如ROS1、IDM1和MBD7。研究结果表明,由IDM1建立的乙酰化组蛋白标记可以被含bromo结构域的蛋白NPX1和ATMBD9识别,然后乙酰化的组蛋白标记物将SWR1复合体招募到染色质上去沉积H2A.Z;最后,在这些特定基因组DNA区域中,H2A.Z和ROS1直接相互作用从而招募ROS1开始DNA主动去甲基化过程,同时防止高甲基化和DNA甲基化的传播。该研究确定了由IDM复合物起始的植物DNA主动去甲基的完整调控途径。从应用科学的角度来看,DNA去甲基化机制在保持转基因生物中的转基因活性进而改善生长、耐受环境变化或预防疾病等过程中有十分重要的指导价值。
2 DNA甲基化的生物学功能DNA甲基化作为一种重要的染色质修饰,广泛分布于异染色质区、常染色质区的转座子区和转录不活跃基因的启动子区,参与异染色质结构维持、转座子沉默和基因转录调控等生物学过程。
2.1 调控基因表达DNA甲基化可以通过改变染色质结构、DNA构象、组蛋白修饰及DNA与蛋白质的相互作用方式对基因的表达进行调控[16]。研究发现,在开花诱导通路中DNA甲基化对调控开花基因的表达发挥重要作用,其中去甲基化似乎更能促进开花基因的表达[17]。小麦基因启动子区域DNA甲基化对基因的表达起调控作用,但编码区DNA甲基化对基因表达无明显影响[18]。尽管启动子DNA甲基化通常会抑制基因转录,但在某些情况下也会促进基因转录,如拟南芥的ROS1基因[19]和一些抑制番茄果实成熟的基因[20]。由此可见,DNA甲基化在调控植物生长发育相关基因表达的分子机理有所不同。
组蛋白翻译后共价修饰是表观遗传调控的重要方式之一,通过影响染色质的状态而调控基因表达等过程。组蛋白H3第27位赖氨酸的三甲基化修饰(H3K27me3)通过维持基因的沉默状态,在动植物细胞命运决定以及生长发育中发挥重要的调控作用。研究发现,拟南芥组蛋白H3K27me3去甲基化酶REF6/JMJ12能够通过其自身的锌指结构域特异性地识别拟南芥基因组中CTCTGYTY基序从而去除H3K27me3/me2甲基化修饰,调控基因的时空表达水平[21]。进一步研究发现,并非所有的CTCTGYTY基序都能够被REF6识别,REF6更倾向于结合在开放的染色质区域,而不结合异染色质区域,然而这其中的分子机制尚不清楚。近期研究发现,REF6倾向于结合低甲基化水平的CTCTGYTY基序,并且甲基化的DNA基序在体外系统中降低了REF6锌指结构域与DNA结合的亲和力[22]。该研究揭示了DNA甲基化是调控组蛋白去甲基化酶REF6在基因组中靶向的重要因素,为深入开展组蛋白修饰酶类在染色质上的定位和作用机制开拓了思路。
2.2 维持基因组稳定性DNA甲基化在维持植物基因组稳定性中起重要作用。植物中的DNA甲基化可以抑制转座子和外源DNA的转录,减少由非等位基因易位和重组引起的基因座破坏,从而抵抗外源基因的干扰并维持基因组的稳定。
植物可以通过调节转座子甲基化水平有效抑制转座子活性,从而阻止转座子在基因组中“跳跃”,进而维持基因组的稳定。在玉米基因组中,活性基因和无活性转座子通常被RdDM介导的CHH甲基化岛分开,当CHH甲基化岛丢失时,附近转座子中的CG和CHG位点发生低甲基化,从而导致转录激活,说明玉米中的RdDM是防止沉默的转座子被附近活性基因的常染色质激活所必需的[23]。而在甜菜中沉默的转座子表现出比反转录转座子和基因更高的CHH甲基化水平[24]。在刚刚完成基因组测序的茶树基因组中,研究发现相比古老的转座子的DNA甲基化,近期扩增的转座子呈现高甲基化的状态,表明DNA甲基化对于调控近期活跃的转座子有抑制作用[25]。
为了更好地利用外源DNA以及防止外源DNA的入侵,生物本身不仅能够合成限制性内切酶对外源DNA进行酶切,还可以在自身DNA上相应的限制酶切位点进行甲基化修饰以维持其DNA完整性。此外,植物可以通过特殊的识别机制判断外源DNA并将其甲基化来抑制外来DNA的表达,从而减少外源DNA表达带来的危害[26]。综上所述,DNA甲基化可通过多种方式维持植物基因组稳定性。
3 DNA甲基化对植物生长发育的作用不同时间、空间和组织中DNA甲基化表达的变化是植物正常生长发育的必要条件,且甲基化水平随着植物生长发育的进程和环境条件的改变而发生变化。异常的DNA甲基化状态会直接导致植株生长发育的异常[27-28]。刘琼瑶等发现矮生观赏杉木的木质部DNA甲基化水平(63.52%)显著低于野生型(67.51%),且矮生观赏杉木中参与促分裂原活化蛋白激酶(MAPK)级联途径的蛋白磷酸酶IBR5 (Indole-3-butyric acid response 5)基因启动子区域的甲基化水平上升,推测甲基化程度的降低与杉木矮生变异类型的产生有一定联系[29]。
DNA甲基化的降低还可以促进植物的开花。通过对菊花Chrysanthemum morifolium进行5-azaC处理降低其甲基化水平,发现处理植株的开花时间提前,而其他表型性状未受影响[30]。此外还发现植物能够通过DNA甲基化调控植株育性的表达。在亚棉A花药败育过程中,基因组DNA甲基化程度明显升高,推测花药败育可能受到DNA甲基化的影响[31]。然而在小麦花药发育中DNA甲基化水平呈下降趋势,其中通过化杀剂诱导得到的不育系(1376-CIMS)可以通过外施DNA甲基化抑制剂处理来恢复育性[32]。新的研究证实,DNA甲基化不仅在动物发育过程中被广泛重新编程,而且在开花植物生殖细胞内也经历甲基化重编程,并且甲基化重编程是细胞行使功能的必要条件[33]。关于植物生殖细胞是如何传递和继承DNA甲基化的也是需要进一步研究的重点。
在DNA甲基化对果实发育的研究中发现番茄果实从裂开期到成熟红色期,甲基化水平升高[34]。当控制番茄果实发育基因的Cnr位点(Colorless non-ripening)发生DNA超甲基化时就会抑制番茄果实的成熟[35]。此外,Wang等[36]研究还发现番茄果实成熟过程中的转录因子也受到DNA甲基化的调控,指出DNA甲基化可以通过影响转录因子多拷贝基因调控番茄果实成熟的发育。但是在其他果实尤其是非呼吸跃变型果实中,DNA甲基化是否参与调控果实成熟及其调控机制还不清楚。
4 DNA甲基化与植物胁迫植物基因组DNA甲基化状态会响应生物和非生物胁迫,植物可以调整相关基因的表达状态以应对不良的生长环境,并且由胁迫引起的大多数DNA甲基化变异在世代间可稳定遗传。
4.1 DNA甲基化在非生物胁迫中的作用外界不良的环境条件如干旱、热害、冷害、盐害和重金属等非生物胁迫能够诱导植物基因组DNA甲基化水平发生动态的变化,因此推测DNA甲基化是调控植物防御基因的重要因子之一。
干旱胁迫下,水稻植株DNA甲基化水平升高,且具有一定品种特异性和时空特异性,参与抗旱胁迫性反应的DNA序列在基因编码区和非编码区中CCGG位点发生甲基化的概率基本相等,且非编码区以启动子区甲基化为主[37]。袁溢等[38]通过对干旱胁迫后人工合成甘蓝型油菜进行甲基化敏感多态性分析,发现其甲基化和去甲基化水平均发生了显著的变化,推测植物的甲基化变化有利于提高植物的抗旱能力。但甲基化在植物适应长期干旱胁迫的调控机制尚不清楚。Xu等[39]以苹果抗旱品种“秦冠”和不抗旱品种“蜜脆”为材料,解析了苹果的甲基化组图谱,并从全基因组水平探究了DNA甲基化修饰与基因表达之间的关系,发现启动子未甲基化基因比启动子甲基化基因有更高的表达水平,基因甲基化似乎与基因表达呈负相关。许多基因的甲基化变化与干旱胁迫有关,包括编码转录因子(TF)和转座因子(TE)的基因。研究结果为进一步探索DNA甲基化在苹果以及其他果树上的调控功能提供了基础,并对深入研究果树抗旱的分子机制和分子育种具有重要意义。
研究表明,当棉花花药遭受高温胁迫时,基因组中DNA甲基化发生变化,高温耐受系中具有显著的超CHH甲基化,而高温敏感系的花药在四分体和绒毡层降解阶段出现低CHH甲基化;并且在高温条件下,CHH甲基化在花药中蛋白质编码基因的启动子和下游区域发生显著变化[40]。该研究揭示了在高温胁迫下DNA甲基化调控棉花雄性不育的机制,为使用表观遗传技术创建耐高温新品种开辟了一条新途径。曾子入等[41]发现萝卜肉质根的DNA甲基化在高温胁迫后发生明显变化,且不同材料对热胁迫的响应不同,耐热材料以去甲基化为主,而不耐热材料以超甲基化为主。综上所述,DNA甲基化模式变化有利于植物对高温胁迫的抗性和维持基因组稳定性。
在低温下,植物通过诱导抗逆基因甲基化水平下降或降低甲基化水平激活转座子活性,从而提高抗逆基因的表达以适应低温胁迫。当对西瓜幼苗进行4 ℃低温处理12 h后发现,二倍体和三倍体西瓜的半甲基化率及总甲基化率都有明显变化,其中半甲基化率下降最为明显[42]。在番茄果实中,冷处理下调了DNA去甲基化酶DML2的表达,引起启动子高甲基化,从而沉默了负责风味挥发物生物合成的基因[43]。这也解释了为什么番茄果实在冷藏期间会失去味道。
在高盐条件下,冰叶日中花Mesembryanthemum crystallinum L.卫星DNA的CpHpG位点的甲基化修饰水平增加两倍,光合作用从C3循环变为景天酸代谢以适应外界胁迫[44]。然而在棉花中却发现,盐碱逆境下棉花根和叶的DNA甲基化模式均以去甲基化为主[45]。表明植物DNA甲基化在响应高盐胁迫时有不同的模式变化。也有研究表明,在盐胁迫下,参与植物盐胁迫响应的功能基因MYBS1的表达与其DNA甲基化修饰水平呈负相关,而与组蛋白H3K9ac的修饰呈正相关。由此可见,DNA甲基化可以通过调控相关基因的表达参与植物响应高盐胁迫反应。
不同浓度Cd胁迫下萝卜和拟南芥DNA甲基化水平均明显提高[46-47]。然而,在Pb和Cd胁迫下,中华水韭Isoetes sinensis Palmer在总甲基化水平上没有明显的变化,但表现出较高的半甲基化水平和较低的全甲基化水平[48]。此外,一些研究还表明重金属处理后DNA甲基化水平出现不同的变化,与处理的浓度有关。玉米幼苗的甲基化水平随Zn浓度的增加呈现先上升后降低的趋势[49]。说明重金属胁迫对植物总DNA甲基化水平的影响具有物种特异性,植物应对重金属胁迫有不同的甲基化反应机制[50]。目前,大多数研究主要是通过研究植物DNA甲基化水平的变化来分析重金属胁迫对植物的影响,对特定基因位点发生改变以及基因是如何参与响应重金属胁迫的机制研究较少。
4.2 DNA甲基化在生物胁迫中的作用生物胁迫主要指病毒和病原体对植物生长发育所造成的胁迫。生物胁迫可引起植物相关病菌基因甲基化模式发生变化,之后通过促进抗病相关基因的表达以响应生物胁迫。在被孢囊线虫感染的大豆和拟南芥根中观察到大量的DNA低甲基化[51-52]。蒺藜苜蓿Medicago truncatula的结瘤需要去甲基化酶DME的作用,在结节发育期间,数百个基因组区域包括一小部分结节特异性共生基因被差异甲基化[53-54]。可见,抗病基因甲基化水平的降低,不仅利于其基因的表达,而且有助于加快重组并获得新的抗性基因[1]。
卵菌是一类有别于动植物和绝大部分微生物的独特真核生物。包括多种植物病原菌如疫霉菌、白锈菌、腐霉菌、霜霉菌等,广泛危害粮食作物、蔬菜花卉以及大量林木,严重威胁生态安全和全球粮食安全。研究表明,6mA在疫霉菌中十分常见,并且在卵菌中发现6mA的修饰酶出现基因扩张,6mA修饰位点也通常与低表达基因定位在同一位置,且富集在转座子和基因组变异较快的区域;而且在6mA修饰酶的敲除突变体中发现一些转座子和致病相关基因表现更加活跃,表明6mA修饰对植物卵菌的致病变异有着潜在的重要作用[55]。深入开展疫霉菌遗传变异的分子机制研究,不但可解释农作物抗性丧失的原因,也有望为农作物抗性的精准改良提供依据。
5 基因组印记印记(Imprinting)是通过一种遗传机制保留外遗传信息的一种重要方式。印记基因的表达可以通过DNA甲基化、组蛋白甲基化修饰和非编码RNA (non-coding RNA,ncRNA)及RNA干扰(RNA interference,RNAi)的调节来实现。其中,DNA甲基化是基因印记的基础,许多印记调控区域都包含差异甲基化区域。
印记基因在调节胚乳细胞分裂、生长和营养运输中起重要作用,且胚乳是植物基因组印记发生的主要部位。研究发现,胚乳中的母本基因组比父本基因组的甲基含量低,特别是在CG背景下[56]。亲本特异性甲基化与胚乳中许多基因座上相应的亲本特异性基因表达(基因印记)显著相关[57-58]。母系表达基因(MEGs)的共同特征是母本等位基因是低甲基化的,而父本等位基因发生甲基化并受到抑制。在某些母系表达基因中,例如拟南芥的MEDEA,通过抑制组蛋白修饰H3K27me3沉默父本等位基因从而在胚乳中只有来自母本的MEDEA基因表达[59]。研究也发现母本DME (TRANSCRIPTIONAL ACTIVATOR DEMETER)或父本MET1的功能出现障碍时会破坏母系表达基因的印记[60-61],说明一些母系表达基因可被DNA甲基化的等位基因特异性所抑制。虽然利用全基因组测序技术在基因组整体水平上发现了许多印记基因,但印记基因是一个复杂的调控网络,目前还不清楚印记基因是如何调控胚乳发育。同时,印记基因的功能也有待进一步研究。
6 总结与展望DNA甲基化作为一种重要的非永久性但相对长期可遗传的基因修饰,通过调控基因表达、沉默转座子和染色体相互作用影响植物的多种生命活动,并在植物逆境胁迫、基因组印记等方面起到一定的作用,同时可以通过这种表观遗传修饰能够产生植物的新表型,丰富生物的多样性。尽管近年来对植物DNA甲基化研究已经取得了一些进展,但对DNA甲基化调控植物生长发育的分子机制还不深入,DNA甲基化变化对胁迫响应的机制目前也不清楚,对DNA甲基化修饰所引起的表观遗传现象在细胞世代间遗传的传递机制也知之甚少。在植物细胞中,组蛋白H3K9me2标记和DNA甲基化是富集在异染色质区域的两种表观遗传标记,可以促进转座子及其他重复序列的沉默,并且这两种标记高度相关。这两者之间是否存在相互联系是人们非常感兴趣的一个科学问题。并且,已有的大多数研究多集中在5-mC,近年来对N6-mA在真核生物中的作用功能也相继被报道[62-67],也成为了表观遗传研究的一个新热点领域。随着植物转录组学和甲基化组学的应用,今后的研究应该从分子以及细胞水平上阐明DNA甲基化的动力学和遗传学及其分子调控机制,在基因表达层面上探究植物的生长发育及抗逆性机理,从而为植物优良新品种的培育和遗传改良研究提供更多表观遗传修饰方面的理论依据。
参考文献
| [1] | Ba QS. DNA methylation regulation mechanism of chemically-induced male sterility in wheat (triticum aestivum. L.)[D]. Yangling: Northwest A & F University, 2016 (in Chinese). 巴青松.小麦生理型雄性不育DNA甲基化调控育性机理研究[D].杨凌: 西北农林科技大学, 2014. |
| [2] | Duan LJ. The analysis of DNA methylation in micro RNA promoters and the biological fuction of AtWRKY30 transcription factor in Arabidopsis thaliana[D]. Ji'nan: Shandong Agricultural University, 2012 (in Chinese). 段丽君.拟南芥microRNA启动子甲基化的分析以及AtWRKY30转录因子的生物学功能研究[D].济南: 山东农业大学, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10434-1012487049.htm |
| [3] | Liang WJ. Spatial-temporal expression paltern analasis of DNA methylation regulation genes in Arabidopsis[D]. Yangling: Northwest A & F University, 2016. 梁文洁.拟南芥DNA甲基化调控蛋白的时空表达特征分析[D].杨凌: 西北农林科技大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10712-1016165575.htm |
| [4] | Ji LJ, Chen XM. Regulation of small RNA stability: methylation and beyond. Cell Res, 2012, 22(4): 624-636. DOI:10.1038/cr.2012.36 |
| [5] | Zheng BL, Wang ZM, Li SB, et al. Intergenic transcription by RNA polymerase Ⅱ coordinates Pol Ⅳ and Pol Ⅴ in siRNA-directed transcriptional gene silencing in Arabidopsis. Genes Dev, 2009, 23(24): 2850-2860. DOI:10.1101/gad.1868009 |
| [6] | Wu L, Mao L, Qi YJ. Roles of DICER-LIKE and ARGONAUTE proteins in TAS-derived small interfering RNA-triggered DNA methylation. Plant Physiol, 2012, 160(2): 990-999. DOI:10.1104/pp.112.200279 |
| [7] | Nuthikattu S, Mccue AD, Panda K, et al. The initiation of epigenetic silencing of active transposable elements is triggered by RDR6 and 21-22 nucleotide small interfering RNAs. Plant Physiol, 2013, 162(8): 116-131. |
| [8] | Mccue AD, Panda K, Nuthikattu S, et al. ARGONAUTE 6 bridges transposable element mRNA-derived siRNAs to the establishment of DNA methylation. EMBO J, 2015, 34(1): 20-35. |
| [9] | Lindroth AM, Saarikoski P, Flygh G, et al. Two S-adenosylmethionine synthetase-encoding genes differentially expressed during adventitious root development in Pinus contorta. Plant Mol Biol, 2001, 46(3): 335-346. DOI:10.1023/A:1010637012528 |
| [10] | Stroud H, Do T, Du JM, et al. Non-CG methylation patterns shape the epigenetic landscape in Arabidopsi. Nat Struct Mol Biol, 2013, 21(1): 64-72. |
| [11] | Seymour DK, Becker C. The causes and consequences of DNA methylome variation in plants. Curr Opin Plant Biol, 2017, 36: 56-63. DOI:10.1016/j.pbi.2017.01.005 |
| [12] | Liu ZW, Shao CR, Zhang CJ, et al. The SET domain proteins SUVH2 and SUVH9 are required for Pol Ⅴ occupancy at RNA-Directed DNA methylation Loci. PLoS Genet, 2014, 10(1): e1003948. DOI:10.1371/journal.pgen.1003948 |
| [13] | Johnson LM, Du JM, Hale CJ, et al. SRA- and SET-domain-containing proteins link RNA polymerase Ⅴ occupancy to DNA methylation. Nature, 2014, 507(7490): 124-128. DOI:10.1038/nature12931 |
| [14] | Gong ZZ, Morales-Ruiz T, Ariza RR, et al. ROS1, a repressor of transcriptional gene silencing in Arabidopsis, encodes a DNA glycosylase/lyase. Cell, 2002, 111(6): 803-814. DOI:10.1016/S0092-8674(02)01133-9 |
| [15] | Nie WF, Lei MG, Zhang MX, et al. Histone acetylation recruits the SWR1 complex to regulate active DNA demethylation in Arabidopsis. Proc Natl Acad Sci USA, 2019, 116(33): 16641-16650. DOI:10.1073/pnas.1906023116 |
| [16] | Zhong Y, Xu H, Peng FL. Significance and research progress of DNA methylation in gene expression regulation. China Med Herald, 2019, 16(14): 33-36 (in Chinese). 钟焱, 徐慧, 彭凤兰. DNA甲基化在基因表达调控中的意义及研究进展. 中国医药导报, 2019, 16(14): 33-36. |
| [17] | Zhu QQ, Li ZA, He YX, et al. Research progress on epigenetic and flowering-time regulation. Acta Hortic Sin, 2013, 40(9): 1787-1794 (in Chinese). 朱芹芹, 李忠爱, 何艳霞, 等. 表观遗传与花期调控研究进展. 园艺学报, 2013, 40(9): 1787-1794. |
| [18] | Wang M. The effect of DNA methylation on salinity stress-responsive genes in wheat[D]. Ji'nan: Shandong University, 2015 (in Chinese). 王萌. DNA甲基化对小麦盐胁迫应答基因的影响研究[D].济南: 山东大学, 2015. http://d.wanfangdata.com.cn/Thesis/Y2790779 |
| [19] | Lei MG, Zhang HM, Julian R, et al. Regulatory link between DNA methylation and active demethylation in Arabidopsis. Proc Natl Acad Sci USA, 2015, 112(11): 3553-3557. DOI:10.1073/pnas.1502279112 |
| [20] | Lang ZB, Wang YH, Tang K, et al. Critical roles of DNA demethylation in the activation of ripening-induced genes and inhibition of ripening-repressed genes in tomato fruit. Proc Natl Acad Sci USA, 2017, 114(22): E4511-E4519. DOI:10.1073/pnas.1705233114 |
| [21] | Cui X, Lu FL, Qiu Q, et al. REF6 recognizes a specific DNA sequence to demethylate H3K27me3 and regulate organ boundary formation in Arabidopsis. Nat Genet, 2019, 48(6): 694-699. |
| [22] | Qiu Q, Mei HL, Deng X, et al. DNA methylation repels targeting of Arabidopsis REF6. Nat Commun, 2019, 10: 2063. DOI:10.1038/s41467-019-10026-1 |
| [23] | Li Q, Gent JI, Zynda G, et al. RNA-directed DNA methylation enforces boundaries between heterochromatin and euchromatin in the maize genome. Proc Natl Acad Sci USA, 2015, 112(47): 14728-14733. DOI:10.1073/pnas.1514680112 |
| [24] | Zakrzewski F, Schmidt M, Van Lijsebettens M, et al. DNA methylation of retrotransposons, DNA transposons and genes in sugar beet (Beta vulgaris L.). Plant J, 2017, 90(6): 1156-1175. DOI:10.1111/tpj.13526 |
| [25] | Wang L, Shi Y, Chang XJ, et al. DNA methylome analysis provides evidence that the expansion of the tea genome is linked to TE bursts. Plant Biotechnol J, 2018, 17(4): 826-835. |
| [26] | Wang HH, Wu SJ, Li FF, et al. Transgene silencing caused by 35S promoter methylation in upland cotton (Gossypium hirsutum). Cott Sci, 2008, 20(4): 274-280. |
| [27] | Li PC, Bi ZZ, Liang WJ, et al. DNA methylation involved in regulating drought stress response of potato. Acta Agronom Sin, 2019, 45(10): 1595-1603 (in Chinese). 李鹏程, 毕真真, 梁文君, 等. DNA甲基化参与调控马铃薯干旱胁迫响应. 作物学报, 2019, 45(10): 1595-1603. |
| [28] | Li XN, Cai YL. Analysis of DNA methylation levels and patterns in dwarf rootstock of prunus mahaleb. Acta Bot Boreali-Occid Sin, 2017, 37(5): 864-871 (in Chinese). 李向男, 蔡宇良. 马哈利樱桃矮化砧的DNA甲基化水平及模式分析. 西北植物学报, 2017, 37(5): 864-871. |
| [29] | Liu QY, Huang HH, Feng HP, et al. Analysis of DNA methylation levels and patterns in dwarf ornamental Cunninghamia lanceolata. Acta Hortic Sin, 2015, 42(10): 2015-2022 (in Chinese). 刘琼瑶, 黄华宏, 冯惠平, 等. 矮生观赏杉木DNA甲基化的水平与模式分析. 园艺学报, 2015, 42(10): 2015-2022. |
| [30] | Wang ZC, Nie LJ, He YX. The effect of 5-azacytidine to the DNA methylation and morphogenesis character of chrysanthemum during in vitro growth. Acta Hoticult Sin, 2009, 36(12): 1783-1790 (in Chinese). 王子成, 聂丽娟, 何艳霞. 离体条件下5-氮杂胞嘧啶核苷对菊花DNA甲基化和表型性状的影响. 园艺学报, 2009, 36(12): 1783-1790. DOI:10.3321/j.issn:0513-353X.2009.12.010 |
| [31] | Guo JY. Study on microspore abortion of cytoplasmatic male sterility line Yamian A[D]. Jinzhong: Shanxi Agricultural University, 2013 (in Chinese). 郭俊尧.细胞质雄性不育系亚棉A小孢子败育过程的研究[D].晋中: 山西农业大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10113-1013378745.htm |
| [32] | Ba QS, Zhang GS, Wang JS, et al. Gene expression and DNA methylation alterations in chemically induced male sterility anthers in wheat (Triticum aestivum L.). Acta Physiol Plant, 2014, 36(2): 503-512. DOI:10.1007/s11738-013-1431-6 |
| [33] | Walker J, Gao HB, Zhang JY, et al. Sexual-lineage-specific DNA methylation regulates meiosis in Arabidopsis. Nat Genet, 2018, 50(1): 130-137. DOI:10.1038/s41588-017-0008-5 |
| [34] | Teyssier E, Bernacchia G, Maury S, et al. Tissue dependent variations of DNA methylation and endoreduplication levels during tomato fruit development and ripening. Planta, 2008, 228(3): 391-399. DOI:10.1007/s00425-008-0743-z |
| [35] | Manning K, ToR M, Poole M, et al. A naturally occurring epigenetic mutation in a gene encoding an SBP-box transcription factor inhibits tomato fruit ripening. Nat Genet, 2006, 38(8): 948-952. DOI:10.1038/ng1841 |
| [36] | Wang L, Xie JH, Hu JT, et al. Comparative epigenomics reveals evolution of duplicated genes in potato and tomato. Plant J, 2017, 93(3): 460-471. |
| [37] | Pan YJ, Fu BY, Wang D, et al. Spatial and temporal profiling of DNA methylation induced by drought stress in rice. Sci Agricult Sin, 2009, 42(9): 3009-3018 (in Chinese). 潘雅姣, 傅彬英, 王迪, 等. 水稻干旱胁迫诱导DNA甲基化时空变化特征分析. 中国农业科学, 2009, 42(9): 3009-3018. |
| [38] | Yuan Y, Zhu S, Fang TT, et al. Analysis of drought resistance and DNA methylation level of resynthesized Brassica napus. Acta Agronom Sin, 2019, 45(5): 693-704 (in Chinese). 袁溢, 朱双, 方婷婷, 等. 人工合成甘蓝型油菜抗旱性及DNA甲基化水平分析. 作物学报, 2019, 45(5): 693-704. |
| [39] | Xu JD, Zhou SS, Gong XQ, et al. Single-Base methylome analysis reveals dynamic epigenomic differences associated with water deficit in apple. Plant Biotechnol J, 2017, 16(2): 672-687. |
| [40] | Ma YZ, Min L, Wang MJ, et al. Disrupted genome methylation in response to high temperature has distinct affects on microspore abortion and anther indehiscence. Plant Cell, 2018, 30(5): 1387-1403. |
| [41] | Zeng ZR, He CA, Zhang XK, et al. Analysis of genome methylation mutation in radish (Raphanus sativus L.) induced by heat stress. Mol Plant Breed, 2018, 16(7): 2094-2098 (in Chinese). 曾子入, 贺从安, 张小康, 等. 高温胁迫诱导萝卜基因组甲基化变异分析. 分子植物育种, 2018, 16(7): 2094-2098. |
| [42] | Yang BY, Huo XA, Liu YT, et al. MSAP and differential expression of homologous diploid and triploid watermelon under cold stress. Acta Hortic Sin, 2014, 41(11): 2313-2322 (in Chinese). 杨炳艳, 霍秀爱, 刘云婷, 等. 低温胁迫下西瓜同源二倍体和三倍体甲基化及基因表达的差异分析. 园艺学报, 2014, 41(11): 2313-2322. |
| [43] | Zhang B, Tieman DM, Jiao C, et al. Chilling-induced tomato flavor loss is associated with altered volatile synthesis and transient changes in DNA methylation. Proc Natl Acad Sci USA, 2016, 113(44): 12580-12585. DOI:10.1073/pnas.1613910113 |
| [44] | Dyachenko OV, Zakharchenko NS, Shevchuk TV, et al. Effect of hypermethylation of CCWGG sequences in DNA of Mesembryanthemum crystallinum plants on their adaptation to salt stress. Biochem Moscow, 2006, 71(4): 461-465. DOI:10.1134/S000629790604016X |
| [45] | Cao DH, Gao X, Liu J, et al. Methylation sensitive amplified polymorphism (MSAP) reveals that alkali stress triggers more DNA hypomethylation levels in cotton (Gossypium hirsutum L.) roots than salt stress. Af J Biotechnol, 2011, 10(82): 18971-18980. |
| [46] | Yang JL. Studys on the genotypic difference of cadmium accumulation, physiological response and methylated change in radish (Rapanus Sativus L.) under cadmium stress[D]. Nanjing: Nanjing Agricultural University, 2007 (in Chinese). 杨金兰.萝卜镉累积的基因型差异、生理响应与甲基化变化研究[D].南京: 南京农业大学, 2007. http://d.wanfangdata.com.cn/Thesis/Y1215159 |
| [47] | Ma HX. The effects of two stress treatments on Arabidopsis DNA methylation[D]. Zhengzhou: Henan University, 2009. 马洪霞.两种逆境胁迫对拟南芥DNA甲基化水平的影响[D].郑州: 河南大学, 2009. http://d.wanfangdata.com.cn/Thesis/Y1485107 |
| [48] | Ding GH, Guo DD, Guan Y, et al. Effect of Pb and Cd on DNA methylation of Isoetes sinensis, a rare plant. J Agro-Environ Sci, 2017, 36(2): 246-249 (in Chinese). 丁国华, 郭丹蒂, 关旸, 等. 重金属铅镉对濒危植物中华水韭(Isoetes sinensis) DNA甲基化的影响. 农业环境科学学报, 2017, 36(2): 246-249. |
| [49] | Erturk FA, Agar G, Arslan E, et al. Analysis of genetic and epigenetic effects of maize seeds in response to heavy metal (Zn) stress. Environ Sci Pollut Res, 2015, 22(13): 10291-10297. DOI:10.1007/s11356-014-3886-4 |
| [50] | He YN, Xu ZR, Xiong ZT. DNA methylation patterns of acid invertase gene promoters from Cu-tolerant and non-tolerant populations of Elsholtzia haichowensis under copper stress. Plant Sci J, 2017, 35(4): 574-582 (in Chinese). 何宇宁, 徐仲瑞, 熊治廷. 铜胁迫对不同抗性种群海州香薷酸性转化酶基因启动子甲基化的影响. 植物科学学报, 2017, 35(4): 574-582. |
| [51] | Hewezi T, Lane T, Piya S, et al. Cyst Nematode parasitism induces dynamic changes in the root epigenome. Plant Physiol, 2017, 174(1): 405-420. DOI:10.1104/pp.16.01948 |
| [52] | Rambani A, Rice JH, Liu JY, et al. The methylome of soybean roots during the compatible interaction with the Soybean Cyst Nematode. Plant Physiol, 2015, 168(4): 1364-1377. DOI:10.1104/pp.15.00826 |
| [53] | Nagymihály M, Veluchamy A, Gy?rgypál Z, et al. Ploidy-dependent changes in the epigenome of symbiotic cells correlate with specific patterns of gene expression. Proc Natl Acad Sci USA, 2017, 114(17): 4543-4548. DOI:10.1073/pnas.1704211114 |
| [54] | Satgé C, Moreau S, Sallet E, et al. Reprogramming of DNA methylation is critical for nodule development in Medicago truncatula. Nat Plants, 2016, 2(11): 16166. DOI:10.1038/nplants.2016.166 |
| [55] | Chen H, Shu HD, Wang LY, et al. Phytophthora methylomes are modulated by 6mA methyltransferases and associated with adaptive genome regions. Genome Biol, 2018, 19(1): 181. DOI:10.1186/s13059-018-1564-4 |
| [56] | Gehring M, Bubb KL, Henikoff S. Extensive demethylation of repetitive elements during seed development underlies gene imprinting. Science, 2009, 324(5933): 1447-1451. DOI:10.1126/science.1171609 |
| [57] | Klosinska M, Picard CL, Gehring M. Conserved imprinting associated with unique epigenetic signatures in the Arabidopsis genus. Nat Plants, 2016, 2(10): 16145. DOI:10.1038/nplants.2016.145 |
| [58] | Park K, Kim MY, Vickers M, et al. DNA demethylation is initiated in the central cells of Arabidopsis and rice. Proc Natl Acad Sci USA, 2016, 113(52): 15138-15143. DOI:10.1073/pnas.1619047114 |
| [59] | Jullien PE, Katz A, Oliva M, et al. Polycomb group complexes self-regulate imprinting of the polycomb group gene MEDEA in Arabidopsis. Curr Biol, 2006, 16(5): 486-492. DOI:10.1016/j.cub.2006.01.020 |
| [60] | Hsieh TF, Shin J, Uzawa R, et al. Regulation of imprinted gene expression in Arabidopsis endosperm. Proc Natl Acad Sci USA, 2011, 108(5): 1755-1762. DOI:10.1073/pnas.1019273108 |
| [61] | Vu TM, Nakamura M, Calarco JP, et al. RNA-directed DNA methylation regulates parental genomic imprinting at several loci in Arabidopsis. Development, 2013, 140(14): 2953-2960. DOI:10.1242/dev.092981 |
| [62] | Mondo SJ, Dannebaum RO, Kuo RC, et al. Widespread adenine N6-methylation of active genes in fungi. Nat Genet, 2017, 49(6): 964-968. DOI:10.1038/ng.3859 |
| [63] | Wu TP, Wang T, Seetin MG, et al. DNA methylation on N6-adenine in mammalian embryonic stem cells. Nature, 2016, 532(7599): 329-333. DOI:10.1038/nature17640 |
| [64] | Liu JZ, Zhu YX, Luo GZ, et al. Abundant DNA 6mA methylation during early embryogenesis of zebrafish and pig. Nat Commun, 2016, 7: 13052. DOI:10.1038/ncomms13052 |
| [65] | Greer EL, Blanco MA, Gu L, et al. DNA methylation on N6-adenine in C. elegans. Cell, 2015, 161(4): 868-878. |
| [66] | Zhang GQ, Huang H, Liu D, et al. N6-Methyladenine DNA modification in Drosophila. Cell, 2015, 161(4): 893-906. DOI:10.1016/j.cell.2015.04.018 |
| [67] | Zhou C, Wang CS, Liu HB, et al. Identification and analysis of adenine N6-methylation sites in the rice genome. Nat Plants, 2018, 4(8): 554-563. |
