

1. 商丘师范学院 生物与食品学院,河南 商丘 476000;
2. 中国科学院大连化学物理研究所 生物技术部,辽宁 大连 116023
收稿日期:2019-08-18;接收日期:2019-10-15;网络出版时间:2020-10-25
基金项目:国家自然科学基金(No. 31571997),河南省高等学校重点科研项目(No. 20A180024),商丘师范学院校级科研启动基金(No. 700144)资助
摘要:2-卤代酸脱卤酶(EC 3.8.1.X)催化2-卤代酸脱卤水解形成相应的2-羟基酸。该类酶不仅能够降解环境中的卤代污染物,而且具有宽广底物谱和高效手性拆分特性,因而在环保和手性中间体的绿色合成中具有广阔应用前景。目前已经对多种2-卤代酸脱卤酶进行生化特性表征,并对酶分子三维结构及催化机制进行了深入研究。文中从2-卤代酸脱卤酶的来源、蛋白质结构与催化反应机制、催化特性及应用方面等研究取得的新进展进行综述,并展望了2-卤代酸脱卤酶的进一步研究方向。
关键词:2-卤代酸脱卤酶蛋白结构催化机制应用
Recent progress in 2-haloacid dehalogenases
Yayue Wang1, Song Xue2, Qingfeng Zhou1, Dongli Pei1


1. College of Biology and Food, Shangqiu Normal University, Shangqiu 476000, Henan, China;
2. Laboratory of Biotechnology, Dalian Institute of Chemical Physics, Chinese Academy of Sciences, Dalian 116023, Liaoning, China
Received: August 18, 2019; Accepted: October 15, 2019; Published: October 25, 2020
Supported by: National Natural Science Foundation of China (No. 31571997), Key Scientific Research Projects in Universities of Henan Province (No. 20A180024), Research Project of Shangqiu Normal University (No. 700144)
Corresponding author: Dongli Pei. Tel/Fax: +86-370-3157770; E-mail: peidongli@126.com.
Abstract: 2-Haloacid dehalogenases (EC 3.8.1.X) catalyze the hydrolytic dehalogenation of 2-haloacids, releasing halogen ions and producing corresponding 2-hydroxyacids. The enzymes not only degrade xenobiotic halogenated pollutants, but also show wide substrate profile and astonishing efficiency for enantiomer resolution, making them valuable in environmental protection and the green synthesis of optically pure chiral compounds. A variety of 2-haloacid dehalogenases have been biochemically characterized so far. Further studies have been made in protein crystal structures and catalytic mechanisms. Here, we review the recent progresses of 2-haloacid dehalogenases in their source, protein structures, reaction mechanisms, catalytic properties and application. We also suggest further research directions for 2-haloacid dehalogenase.
Keywords: 2-haloacid dehalogenasesprotein structurecatalytic mechanismapplication
有机卤代物因其优良的热传导性、绝缘性、耐热性、亲油性及生物活性等特点而被广泛应用于工、农、医等生产领域,给社会发展带来巨大的经济效益[1-3]。然而,由于大量生产和不当使用,越来越多的卤代物被排放到环境各处而逐渐恶化生态环境。加之卤代物的化学性能非常稳定,导致其在天然条件下难以被降解,因此在环境中的积累越来越多,进而可通过食物链浓缩、累积于生物体中,呈现“致癌、致畸、致突变”效应,极大地威胁着人类健康,已成为全世界关注的问题[4]。
微生物作为自然界中主要分解者,将复杂的有机物转化为简单的化合物,从而保持生命元素的循环往复。生长在被有机卤代物污染环境中的微生物具有潜在的转化这些化合物的能力,即其细胞内应存在催化脱卤作用的酶,这类酶被称为脱卤酶。其中2-卤代酸脱卤酶是催化2-卤代酸脱卤水解形成相应2-羟基酸的一类水解型脱卤酶(图 1),不仅能够以一种低能耗途径降解环境中的有毒污染物,而且底物谱广,催化效率高,并具有高效手性拆分特性,可用于获得光学纯2-卤代物和2-羟基取代物,因而在环境修复与手性化学品的绿色制造领域具有巨大应用价值。文中将从酶的来源与分类、酶分子结构与催化机制、催化特性及应用进展对2-卤代酸脱卤酶进行详述。
1 2-卤代酸脱卤酶来源与分类2-卤代酸脱卤酶产生菌广泛存在于自然界的各种环境中,早在20世纪80年代后期就已经被发现报道,随后越来越多的酶相继被发掘。目前分离获得的2-卤代酸脱卤微生物已遍布于原核生物和真菌,其中原核生物包括假单胞菌属Pseudomonas[5]、土壤杆菌属Agrobacterium[6]、固氮菌属Azotobacter[7]、根瘤菌属Rhizobium[8]、莫拉克斯氏菌属Moraxella[9]、硫化叶菌属Sulfolobus[10]、黄杆菌属Xanthobacter[11]、副球菌属Paracoccus[12]、产碱杆菌属Alcaligenes[13]、伯克霍尔德菌属Burkholderia[14]、火球菌属Pyrococcus[15]、甲基杆菌属Methylobacterium[2]、冷单胞菌属Psychromonas[16]、红杆菌属Rhodobacteraceae[1]、假交替单胞菌属Pseudoalteromonas[17]、副球孢子菌属Paracoccidioides[18]、克雷白氏杆菌属Klebsiella[19]、杆菌属Ancylobacter[20]、芽孢杆菌属Bacillus[21]与嗜冷单胞菌Psychromonas[22];真菌包括绿僵菌属Metarhizium、镰刀菌Fusarium与白僵菌Beauveria[23],其中假单胞菌属所占比例最高。从分离环境上讲,大多数脱卤微生物分离自陆生环境,仅有少数来自于海洋环境[1, 5, 12, 16, 24-25]。由于海洋环境高压、高盐、低温及寡营养等独特性质,被认为是生命活动的极端环境,因此生活在这一特殊环境中的微生物,在基因组成和生态功能上表现出多样性和特异性[26]。相应地,微生物生理生化特征与胞内酶也表现出多样性和特异性,是发现新酶的重要源泉。
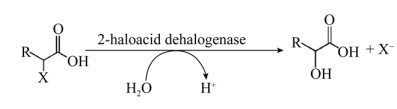 |
| 图 1 2-卤代酸脱卤酶催化反应式 Fig. 1 Dehalogenation catalyzed by 2-haloacid dehalogenase[2]. |
| 图选项 |
2-卤代酸脱卤酶可以依据序列保守性和立体选择性进行分类[27]。根据其底物选择性和产物构型,可分为4种(表 1),包括D-2-卤代酸脱卤酶(D-DEX)、L-2-卤代酸脱卤酶(L-DEX)、构型翻转型DL-2-卤代酸脱卤酶(DL-DEXi)和构型保留型DL-2-卤代酸脱卤酶(DL-DEXr)。根据氨基酸序列同源性,2-卤代酸脱卤酶又可以被分为Group Ⅰ与Group Ⅱ两类酶。Group Ⅰ类酶包括D-DEX与DL-DEX;Group Ⅱ类酶仅包括L-DEX。在本综述中,采用第一种分类方法。
表 1 2-卤代酸脱卤酶的种类Table 1 Types of 2-haloacid dehalogenases
| Types EC number | Reaction |
| L-2-haloacid dehalogenase EC 3.8.1.2 | L-2-haloacid + H2O → D-2-hydroxyacid + halide ion |
| D-2- haloacid dehalogenase EC 3.8.1.9 | D-2-haloacid + H2O → L-2-hydroxyacid + halide ion |
| DL-2- haloacid dehalogenase EC 3.8.1.10 (Conformation inversion) | L-2-haloacid + H2O → D-2-hydroxyacid + halide ion D-2-haloacid + H2O → L-2-hydroxyacid + halide ion |
| DL-2- haloacid dehalogenase EC 3.8.1.11 (Conformation retention) | L-2-haloacid + H2O → L-2-hydroxyacid + halide ion D-2-haloacid + H2O → D-2-hydroxyacid + halide ion |
表选项
2 2-卤代酸脱卤酶结构特征和催化特性2-卤代酸脱卤酶结构的多样性决定了其功能多样性,不同类型2-卤代酸脱卤酶在结构和催化机理上也有所不同。
2.1 L-DEX2.1.1 结构特征和催化机理L-DEX酶类专一性地作用于L-2-卤代酸产生D-2-羟基酸,在自然界中普遍存在,是目前研究最为透彻的一类2-卤代酸脱卤酶[23, 28-29]。至今已解析多种来源的L-DEX及其与底物复合物的三维结构,如来自于假单胞菌Pseudomonas sp. strain YL的L-DEX YL[30],自养黄杆菌Xanthobacter autotrophicus GJ10的DhlB[31],掘越氏火球菌Pyrococcus horikoshii OT3的PH0459[15],洋葱伯克霍尔德菌Burkholderia cepacia MBA4的DehIVa[32],硫化叶菌Sulfolobus tokodaii的DehSft[33]与红杆菌Rhodobacteraceae的DehRhb[1]。L-DEX是α/β类型水解酶,但并不属于α/β水解酶折叠家族。其含有典型的Rossmann-fold-like核心域:6股β-sheets以321456的顺序平行排列,由5股α-helices连接β-sheets形成的3层α/β折叠单元构成三明治结构域[30, 34-35]。不同于α/β水解酶折叠家族典型的结构域,即8股β-strands以12435678的顺序排列,且β2-strands反平行于其他链,从第3股链开始,两条链之间由α-helix连接,形成β/α/β单元,第一个α-helix和最后一个α-helix位于β-sheet的一侧,其余α-helix位于另一侧[36]。其中除了PH0459晶体结构为单体,其他L-DEX晶体结构均为二聚体。除DhlB由一个核心结构域、两个亚结构域组成外,其他L-DEX都是由一个核心结构域与一个亚结构域构成,活性位点位于两个结构域间(图 2)。通过X-射线晶体衍射、O18同位素标记、液质联用、定点突变及量子力学/分子力学(QM/MM)计算等技术研究,发现L-DEX的脱卤反应属于SN2亲核取代反应,其活性中心天冬氨酸残基的羧酸基团为亲核试剂,攻击L-2-卤代酸的C2原子,形成一个酯中间体,中间产物在由His/Glu (DehRhb中)或Asn/Ser (DehIVa中)或Lys (L-DEX YL中)活化的水分子的进攻下水解,离去的卤素离子主要在Arg或Asn或Phe的辅助下稳定(图 3),而卤素离子受体数量愈多,酶分子愈是可以裂解更强的C-X键[37-38]。
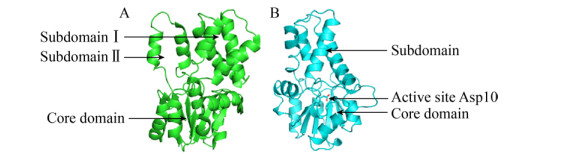 |
| 图 2 L-DEX晶体结构(A:来自Xanthobacter autotrophicus GJ10的DhlB[31]; B:来自Pseudomonas sp. strain YL的L-DEX YL[30]) Fig. 2 Crystal structure of L-DEX. (A) DhlB from Xanthobacter autotrophicus GJ10[31]. (B) L-DEX YL from Pseudomonas sp. strain YL[30]. |
| 图选项 |
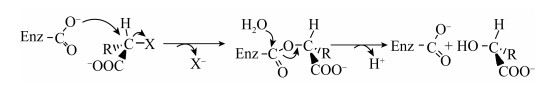 |
| 图 3 L-DEX催化机制[32] Fig. 3 Reaction mechanism of L-DEX[32]. |
| 图选项 |
2.1.2 L-DEX催化特性L-DEX的来源比较广泛,如陆生和海洋环境,不同生境来源的L-DEX在酶学特性上既有相同点又表现出差异。如L-DEX对氯代与溴代底物表现出高的催化活性,对D-2-卤代酸无降解能力,不能催化氟代与C3位置取代卤代酸的脱卤反应;除了具有催化长链卤代酸脱卤特性之外,多数L-DEX仅对于碳链长度为2和3的卤代酸表现出高的催化活性,而对于碳链更长的底物的催化活性很低[5, 11-12, 39]。但在底物特异性上不尽相同,如L-DEX YL对L-2-氯丙酸的催化转化比氯乙酸好,而来源于芽孢杆菌Bacillus strain I37c菌株的L-DEX则更偏好于氯乙酸[39-40]。L-DEX最适反应pH偏碱性范围,在pH 9–11之间,L-DEX亚基分子量为25 000–28 000 Da等。但这类脱卤酶在天然状态下,其分子量不尽相同,分别有单聚体、二聚体和四聚体[5, 11-12, 39]。对来源于不同细菌的脱卤酶,其对温度耐受性也不一致。如陆生来源的恶臭假单胞菌Pseudomonas putida产生的L-DEX最适反应温度为30–45 ℃,55 ℃孵育15 min后,酶活性为原来的50%,热稳定性差;从海冰界面(?10 ℃)分离的嗜冷单胞菌Psychromonas ingrahamii (Pin)菌株具有嗜冷特性,最低生长温度?12 ℃,其产生的L-DEX Pin最适反应温度为45 ℃,熔解温度为85 ℃。L-DEX Pin具有嗜冷酶与嗜热酶特征,与嗜中温酶相比,该酶具有更多疏水表面和更多盐桥[16]。海洋来源的Rhodobacteraceae产生的DehRhb最适反应温度为55 ℃,60 ℃条件下孵育1 h后,活性仍保留在45%左右,表现出适度热稳定性,另外,其催化关键氨基酸为His183和Glu21,不同于陆生来源的L-DEX,可能是以一种新的催化机制进行脱卤[22]。可见,通过利用海洋环境及其他极端环境资源可以开发更多新型天然脱卤酶,为认识其结构、催化机理及催化特性提供新知识,同时为L-DEX及其他酶类的定向改造提供理性指导。
2.2 DL-DEX2.2.1 结构特征和催化机理DL-DEX酶类可同时催化2-卤代酸的两个对映体脱卤反应,水解产生相应的2-羟基酸。根据产物的构型分为构型翻转型(DL-DEXi)与构型保留型(DL-DEXr)。
DL-DEXi酶类催化底物脱卤时,C2原子构型翻转,产物构型与底物构型相反。目前已报道的DL-DEXi包括分离自Pseudomonas sp. 113的DL-DEX 113[41]、Pseudomonas putida PP3的DehI[42]与甲基杆菌Methylobacterium sp. CPA1的DL-DEX Mb[43]。其中后两者晶体结构已知,DL-DEXi为全α-helix类型蛋白,与L-DEX及水解酶中的其他折叠超家族在结构上并没有同源性。如图 4所示,为DehI结构图,其晶体结构为二聚体(图 4A),每个单体的N-端(残基1–130)与C-端(166–296)序列具有16%的同源性,构成了一个pseudo-dimer,活性中心位于每个单体的pseudo-dimer交界面处,可结合D-型与L-型底物[27] (图 4B)。DL-DEXi催化机制也不同于L-DEX,其通过活化水分子直接攻击2-卤代酸的C2原子,断裂C-X键,释放卤素离子,脱卤过程并不涉及共价酯中间体的形成(图 5)[41]。保守的Asp与Asn可能负责水分子的活化,然而并没有相关的实验证据。
 |
| 图 4 来自于Pseudomonas putida PP3的DehI晶体结构[42] (A: DehI二聚体结构; B: DehI单体N-端与C-端重复结构叠合, α-helix在图中标出) Fig. 4 Crystal structure of DehI from Pseudomonas putida PP3[42]. (A) Ribbon representation of DehI dimer. (B) Image of the superposed N- and C-terminal repeats of DehI. The α-helices are labelled. |
| 图选项 |
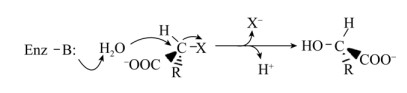 |
| 图 5 L-DEXi脱卤反应机理[41] Fig. 5 Reaction mechanism of DL-DEXi[41]. |
| 图选项 |
DL-DEXr酶类与DL-DEXi催化产物构型变换相反,其催化底物脱卤后,生成的产物构型与底物构型相同。目前可供参考的DL-DEXr是来自于Pseudomonas putida PP3菌株,然而该类酶序列信息仍是未知,反应机理也仍未解析[44]。由于DL-DEXr对N-乙马来酰亚胺与p-对氯汞苯甲酸等巯基破坏试剂高度敏感,因此推测DL-DEXr的巯基基团参与催化反应,在脱卤时可能涉及硫酯中间体的形成。如图 6所示,酶与底物作用,底物C2构型翻转,释放卤素离子,形成酶-底物硫酯中间体,中间体在水分子的进攻下水解,同时C2原子构型发生第二次翻转,生成与底物相同构型的产物,但目前还没有直接的实验证据来支持[44]。
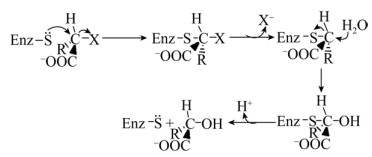 |
| 图 6 DL-DEXr涉及C2-构型保留的可能催化机理[44] Fig. 6 Possible mechanism of DL-DEXr involving the retention of C2-configuration of the substrate[44]. |
| 图选项 |
2.2.2 DL-DEX催化特性目前已分离鉴定的DL-DEX较少,在酶学特性表征方面的研究也较少,目前已报道的DL-DEXi多偏好于L-型底物,如分离自Pseudomonas putida PP3的DehI[42, 45]、分离自根瘤菌Rhizobium sp. RC1的DehE[46]、分离自Pseudomonas sp. 113的DL- DEX 113[45]与来自于木糖氧化产碱杆菌Alcaligenes xylosoxidans ssp. denitrificantsDhIIV的DL-DEX ABIV[47],其中DehI、DehE、DL-DEX 113与DL- DEX 113对L-型底物与D-型底物的催化活性之比分别是1.2、1.6、1.4与1.1DL-DEXi对碳链长度在2–5的卤代酸有降解能力,而且能够降解3-氯乙酸。DL-DEXi包括单体与二聚体,除了DL-DEX YL为单体以外,目前报道酶分子多为同源二聚体,亚基分子量为26 000–36 000 Da[38]。最适反应pH范围为9.5左右,最适反应温度为30–40 ℃[45-46, 48]。催化转化底物和产物构型保持一致的DL-DEXr,对巯基抑制剂比较敏感,对碳链长度为2–4个碳原子的卤代酸有降解能力[44]。
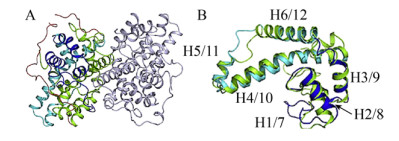
2.3 D-DEX2.3.1 结构特征和催化机理D-DEX酶类是专一性地催化D-2-卤代酸脱卤水解产生L-2-羟基酸的一类酶。目前仅有4种天然来源的D-DEX的一级结构信息可供参考利用,包括来自于根瘤菌Rhizobium sp. RC1的DehD[49]、来自于土壤杆菌Agrobacterium sp. strain NHG3的DehII[50]、来自于恶臭假单胞菌Pseudomonas putida AJ1的HadD AJ1[51]及来自于假单胞菌Pseudomonas sp. ZJU26的DehDIV-R[52],其中HadD AJ1与DehDIV-R间序列D同源性最高为89%,而HadD AJ1与DehD和DehII NHG3间的同源性相对较低,分别为22.2%和32.6%。作者曾对HadD AJ1结构与催化机制进行深入研究,解析晶体结构发现HadD AJ1与DL-DEXi结构相同,都是全α-helix蛋白,不同于L-DEX的α/β折叠结构。HadD AJ1每个单体均由2个氨基酸序列同源性为20%的重复构成(图 7),2个重复折叠分别由N-端α-helix 1–6与C-端α-helix 7–12组成,中间由1个包含33个氨基酸且含有1个310-helix η1的linker连接(图 7B)。每个重复的前3个helices形成了1个three-helix-bundle,后3个helices以three-helix-triangular排布(图 7A)。两个重复折叠通过α6/α12与α4/α10残基间的范德华力、盐键、氢键与疏水相互作用稳定。在空间上,螺旋α4与α10彼此平行排列,α6与α12相互交叉在他们中间的凸起处[53]。这种结构上内在的重复对称折叠在很多蛋白中发现,可能是由蛋白质进化中一些遗传过程如域的融合与裂变及基因复制等改变引起[54]。
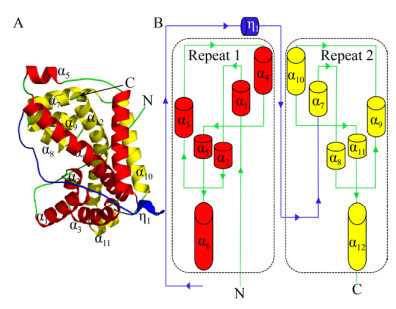 |
| 图 7 HadD AJ1单体结构(A: HadD AJ1单体飘带结构图, 两个重复基序repeat 1 (α1-α6)和repeat 2 (α7-α12)由linker (η1)连接; B: HadD AJ1单体折叠的拓扑结构) Fig. 7 HadD AJ1 monomer. (A) Ribbon representation of monomeric HadD AJ1 composed of repeat 1 (α1-α6), repeat 2 (α7-α12) and a linker (η1). (B) Topological model of HadD AJ1 fold. |
| 图选项 |
如图 8所示,对HadD AJ1催化机理研究表明D-DEX酶类的脱卤反应是由水分子直接介导,反应过程中没有酯中间体的形成。其中,D205为关键催化残基,D205在N131的辅助下共同参与水分子的活化,活化的水分子从正后方进攻底物C2原子,断裂C-X键,卤素离子离去,同时活化的水分子羟基与底物C2原子键合,形成L-型乳酸[53]。
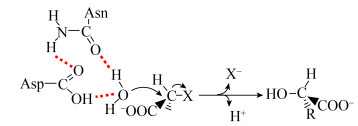 |
| 图 8 D-DEX催化机理 Fig. 8 Reaction mechanism of D-DEX. |
| 图选项 |
2-卤代酸脱卤酶中,D-DEX与DL-DEXi氨基酸序列间具有较高同源性,结构上亦是如此,并且两者催化脱卤都是由水分子直接介导,不同于L-DEX由酶-底物酯中间体介导的脱卤反应,进一步揭示了D-DEX与DL-DEXi在进化上有着密切联系。
2.3.2 D-DEX催化特性目前对D-DEX酶类的研究相对较少,这可能与发现的产D-DEX的细菌种类较少有关。通过对DehD与HadD AJ1生化酶学特性的表征,发现D-DEX酶类偏好于催化碳链长度在2–4个碳原子的D-2-氯代及溴代酸脱卤,对溴代底物的催化活性高于对氯代底物的催化活性[51, 55]。不同来源的酶作用于相同底物的Km值相差较大,如DehD、HadD AJ1与DehDIV-R催化D-2-氯丙酸脱卤的Km值分别为0.06 mmol/L、2.2 mmol/L与2.2 mmol/L。与HadD AJ1和DehDIV-R相比,DehD对卤代底物表现出更高的亲和力,而HadD AJ1与DehDIV-R相同的Km值可能归因于其高度的序列相似性[51-52, 55]。在溶液中,D-DEX的存在状态也有所不同,DehD以同源二聚体形式存在,而HadD AJ1以同源四聚体形式存在。D-DEX酶类最适反应pH在9.0–10.0之间,当pH在8.0–10.0范围之外时,酶活力迅速下降,如HadD AJ1仅表现出低于50%的催化活性。与L-DEX相比,D-DEX酶类呈现嗜中温特性,最适反应温度50–60 ℃,其热稳定性较差,酶分子在30–40 ℃范围内比较稳定,高于40 ℃时,随着温度的升高,酶活性迅速丢失[51-52, 55]。
3 2-卤代酸脱卤酶的应用2-卤代酸脱卤酶可以通过水解脱卤机制脱去有机卤代羧酸中的卤素,在脱卤过程中可以不需另加还原力即可以一条简单的途径进行生物减毒,在有机卤化物污染的环境中起到生物修复的作用;同时,卤代酸脱卤酶还可以高度立体选择性地催化脱卤反应,用于获得小分子的手性羟基酸与卤代酸,这些小分子有机酸通常是农药、医药及化工合成的中间体,因而,该类酶在环境修复及化工合成等领域有着广泛的用途和巨大的经济价值[35, 56-57],主要体现在以下三方面。
1) 环境治理。卤代羧酸如2-氯丙酸和2, 2-二氯丙酸被广泛用作合成农药和医药的中间体,尤其是手性纯2-氯丙酸是合成很多手性药物的前体[58]。但这些卤代酸的大量使用和生产过程中的不当处置使得它们同时成为了常见的有机氯环境污染物。同时卤代酸也是某些卤代物如1, 2-二氯乙烷和杀虫剂六六六降解的中间产物,由于环境中这类污染物的降解而导致了环境中对应卤代酸的污染。这些化合物的积累造成了严重的环境问题并威胁着人类健康。2-卤代酸脱卤酶可以催化2-氯丙酸和2, 2-二氯丙酸脱卤降解为无毒的羟基酸,在环境治理方面具有重要应用价值。
2) 化工合成领域。脱卤酶的手性选择性使其在化工合成领域备受青睐,化学法合成光学纯化合物常常会涉及到刺激性有毒试剂,得到的光学纯度低且产率不高,而利用酶作为生物催化剂不仅反应条件温和,对环境友好且产率高,可获得高度光学纯的手性化合物[16]。如L-2-氯丙酸是合成除草剂和杀虫剂的重要前体,D-DEX可选择性水解外消旋2-氯丙酸,即水解D-2-氯丙酸而不水解L-2-氯丙酸,分离就可以得到高对映体纯度的L-2-氯丙酸[59]。帝国化学工业公司(Imperial Chemical Industries)也早已在工业中应用HadD AJ1拆分消旋2-氯丙酸获得具有光学活性的L-2-氯丙酸,作为农药和消炎药的合成前体,现已成为手性氯丙酸的主要生产方法[60-61]。英国Astrazeneca公司使用D-DEX拆分rac-CPA获得L-2-氯丙酸,对映体纯度高于90%,该方法也适用于生产其他短链手性α-卤代酸,规模可高于1 000 t/年[27]。此外,D-2-氯丙酸也是一种重要的化工原料,可以直接用来合成多种医药中间体,尤其可以用于合成营养药L-丙氨酰-L-谷酰胺、抗结核病药物硫乳霉素,利用L-DEX拆分消旋2-氯丙酸即可获得对映体纯度的D-2-氯丙酸[62]。D-乳酸是农药化工领域中一个重要的手性中间体,可用于合成芳香丙酸类除草剂;同时,D-乳酸在医药方面也有广泛用途,可用于纳米粒子或纳米纤维用作药物载体,以及用于合成重要的手性药物中间体,如D-乳酸甲酯等[63]。解桂秋等曾对来源于嗜热古菌L-DEX催化转化消旋2-氯丙酸生产D-乳酸的反应条件进行研究,通过对底物浓度、缓冲液浓度以及酶浓度等反应条件进行优化,实现以0.5 mol/L 2-氯丙酸为底物制备D-乳酸[64]。
3) 农业生产领域。广谱除草剂如单氯乙酸与2-氯丙酸可以有效地除去多种杂草,但不幸的是,这些除草剂同时可以除去有经济价值的农作物,因此其使用会给农业经济带来大量损失,而构建具有抗除草剂性能的农作物可以避免这些损失。这就需要向农作物中引入编码脱卤酶的基因。如Mohamed等将D-2-卤代酸脱卤酶dehD基因导入烟草中作为一个选择性标签,构建的转基因烟草Nicotiana benthamiana具有抗单氯乙酸的活性[65]。另外,脱卤酶还可用于构建生物感应器,原位检测环境中的有机卤代污染物[66]。如在脱卤酶上融合荧光材料用于细胞成像[67],将融合癌细胞识别肽的脱卤酶连接到多功能的纳米颗粒上用于新的肿瘤诊断和治疗技术的开发等[68]。
4 展望虽然目前已经发现多种2-卤代酸脱卤酶,并且对L-DEX、DL-DEXi与D-DEX三类酶的结构信息和催化机制均有所了解,但对DL-DEXr的研究却仅是停留在酶学性质表征水平,这进一步限制了其应用,因此仍需要对其结构与催化机制进行深入研究。另外,多数2-卤代酸脱卤酶仅对碳链长度小于4个碳原子的卤代酸表现出高催化活性,而对于长链卤代酸仅表现出较弱甚至无催化活性;同时,2-卤代酸脱卤酶对有机溶剂较低的耐受性也限制了其底物谱范围。另外,为了获得对映体纯的手性产物,化学酶促收敛工艺常用于卤代酸脱卤酶的对映体拆分中,然而在下游分离过程中,酶促反应步骤与化学水解所需的高温与极端pH不兼容,因此仍然有必要挖掘新型2-卤代酸脱卤酶。
酶的立体选择性也是一直备受关注的问题,2-卤代酸脱卤酶家族成员表现出典型的立体选择性,但目前对该类酶立体选择性机制的认识也十分有限。Kondo等通过QM/MM与碎片分子轨道计算方法研究L-DEX对映体选择性机制,证实L-DEX与D-型底物结合过程中高的活化能垒阻止了酶作用于D-型底物,然而目前仍不清楚在空间结构上,酶分子是如何调控其对手性底物的选择性的[69]。作者通过对D-DEX的立体选择性机制的研究,发现活性口袋入口处残基Leu288通过空间位阻作用控制L-型底物进入酶活性中心,调控着酶的立体选择性。L288到I的突变使酶催化L-型底物,相比于L288旋转受阻的异丙基,I288侧链乙基可自由旋转,使L-型底物进入酶活性中心[70]。对于DL-DEXr与DL-DEXi如何催化手性底物脱卤这一问题仍不清楚。
生物催化剂的立体选择性使其在光学纯化合物的制备中具有重要的应用价值,是绿色化学研究的重要领域之一。一种理想的工业生物催化剂应该同时具有高催化活性和专一的立体选择性,而探索脱卤酶活性与立体选择性的分子调控机制是人工定制高活性与高立体选择性酶的前提和基础。因此,为了定向调控酶不对称催化反应进行的方向与程度,获得高光学纯度的、具有特殊结构的目标产物,有必要对2-卤代酸脱卤酶的立体选择性催化机制进行深入研究。
参考文献
| [1] | Novak HR, Sayer C, Isupov MN, et al. Marine Rhodobacteraceae L-haloacid dehalogenase contains a novel His/Glu dyad that could activate the catalytic water. FEBS J, 2013, 280(7): 1664-1680. DOI:10.1111/febs.12177 |
| [2] | Kurihara T, Esaki N. Bacterial hydrolytic dehalogenases and related enzymes: Occurrences, reaction mechanisms, and applications. Chem Rec, 2008, 8(2): 67-74. DOI:10.1002/tcr.20141 |
| [3] | Jeschke P. The unique role of halogen substituents in the design of modern agrochemicals. Pest Manag Sci, 2010, 66(1): 10-27. DOI:10.1002/ps.1829 |
| [4] | Rai AR, Singh RP, Srivastava AK, et al. Structure prediction and evolution of a halo-acid dehalogenase of Burkholderia mallei. Bioinformation, 2012, 8(22): 1111-1113. DOI:10.6026/97320630081111 |
| [5] | Zhang JY, Cao XP, Xin YJ, et al. Purification and characterization of a dehalogenase from Pseudomonas stutzeri DEH130 isolated from the marine sponge Hymeniacidon perlevis. World J Microbiol Biotechnol, 2013, 29(10): 1791-1799. DOI:10.1007/s11274-013-1340-2 |
| [6] | K?hler R, Brokamp A, Schwarze R, et al. Characteristics and DNA-sequence of a cryptic haloalkanoic acid dehalogenase from Agrobacterium tumefaciens RS5. Curr Microbiol, 1998, 36(2): 96-101. DOI:10.1007/s002849900286 |
| [7] | Diez A, Prieto MI, Alvarez MJ, et al. Purification and properties of a high-affinity L-2-haloacid dehalogenase from Azotabacter sp. strain RC26. Lett Appl Microbiol, 1996, 23(5): 279-282. DOI:10.1111/j.1472-765X.1996.tb00189.x |
| [8] | Leigh JA, Skinner AJ, Cooper RA. Partial purification, stereospecificity and stoichiometry of three dehalogenases from a Rhizobium species. FEMS Microbiol Lett, 1988, 49(3): 353-356. DOI:10.1111/j.1574-6968.1988.tb02756.x |
| [9] | Kawasaki H, Tone N, Tonomura K. Purification and properties of haloacetate halidohydrolase specified by plasmid from Moraxella sp. strain B. Agric Biol Chem, 1981, 45(1): 35-42. |
| [10] | Xu S, Oin SB, Pan XM. Thermal and conformational stability of Ssh10b protein from archaeon Sulfolobus shibattae. Biochem J, 2004, 382(2): 433-440. DOI:10.1042/BJ20040191 |
| [11] | van der Ploeg J, van Hall G, Janssen DB. Characterization of the haloacid dehalogenase from Xanthobacter autotrophicus GJ10 and sequencing of the dhlB gene. J Bacteriol, 1991, 173(24): 7925-7933. DOI:10.1128/JB.173.24.7925-7933.1991 |
| [12] | Zhang JY, Xin YJ, Cao XP, et al. Purification and characterization of 2-haloacid dehalogenase from marine bacterium Paracoccus sp. DEH99, isolated from marine sponge Hymeniacidon perlevis. J Ocean Univ China, 2014, 13(1): 91-96. DOI:10.1007/s11802-014-2357-3 |
| [13] | Hill KE, Marchesi JR, Weightman AJ, et al. Investigation of two evolutionarily unrelated halocarboxylic acid dehalogenase gene families. J Bacteriol, 1999, 181(8): 2535-2547. DOI:10.1128/JB.181.8.2535-2547.1999 |
| [14] | Tsang JSH, Sam L. Cloning and characterization of a cryptic haloacid dehalogenase from Burkholderia cepacia MBA4. J Bacteriol, 1999, 181(19): 6003-6009. DOI:10.1128/JB.181.19.6003-6009.1999 |
| [15] | Arai R, Kukimoto-Niino M, Kuroishi C, et al. Crystal structure of the probable haloacid dehalogenase PH0459 from Pyrococcus horikoshii OT3. Protein Sci, 2006, 15(2): 373-377. |
| [16] | Novak HR, Sayer C, Panning J, et al. Characterisation of an l-haloacid dehalogenase from the marine Psychrophile psychromonas ingrahamii with potential industrial application. Mar Biotechnol, 2013, 15(6): 695-705. DOI:10.1007/s10126-013-9522-3 |
| [17] | Liao L, Sun X, Zeng YX, et al. A new L-haloacid dehalogenase from the Arctic psychrotrophic Pseudoalteromonas sp. BSW20308. Polar Biol, 2015, 38(8): 1161-1169. DOI:10.1007/s00300-015-1674-3 |
| [18] | Satpathy R, Konkimalla VS, Ratha J. In-silico gene co-expression network analysis in Paracoccidioides brasiliensis with reference to haloacid dehalogenase superfamily hydrolase gene. J Pharm Bioallied Sci, 2015, 7(3): 212-217. DOI:10.4103/0975-7406.160023 |
| [19] | Tahya CY, Ratnaningsih E. Cloning and sequencing of haloacid dehalogenase gene from Klebsiella pneumoniae ITB1. Procedia Chem, 2015, 16: 121-128. DOI:10.1016/j.proche.2015.12.039 |
| [20] | Kumar A, Pillay B, Olaniran AO. L-2-haloacid dehalogenase from Ancylobacter aquaticus UV5: sequence determination and structure prediction. Int J Biol Macromol, 2016, 83: 216-225. DOI:10.1016/j.ijbiomac.2015.11.066 |
| [21] | Ratnaningsih E, Idris I. Cloning and expression of haloacid dehalogenase gene from Bacillus cereus IndB1. Indonesian J Biotech, 2018, 22(2): 55-60. DOI:10.22146/ijbiotech.27338 |
| [22] | Novak H, Littlechild J. 4-Marine enzymes with applications for biosynthesis of fine chemicals// Trincone A, ed. Marine Enzymes for Biocatalysis. Cambridge: Woodhead Publishing, 2013: 89-106. |
| [23] | Satpathy R, Konkimalla VSB, Ratha J. In silico phylogenetic analysis and molecular modelling study of 2-haloalkanoic acid dehalogenase enzymes from bacterial and fungal origin. Adv Bioinformatics, 2016, 2016: 8701201. |
| [24] | Khosrowabadi E, Huyop F. Screening and characterization of several 2, 2-dicholoropropionic acid-degrading bacteria isolated from marine sediment of danga bay and east coast of singapore island. Bioremediat J, 2014, 18(1): 20-27. DOI:10.1080/10889868.2013.834868 |
| [25] | Horisaki T, Yoshida E, Sumiya K, et al. Isolation and characterization of monochloroacetic acid-degrading bacteria. J Gen Appl Microbiol, 2011, 57(5): 277-284. DOI:10.2323/jgam.57.277 |
| [26] | Zhang C, Kim SK. Research and application of marine microbial enzymes: status and prospects. Mar Drugs, 2010, 8(6): 1920-1934. DOI:10.3390/md8061920 |
| [27] | Schober M, Faber K. Inverting hydrolases and their use in enantioconvergent biotransformations. Trends Biotechnol, 2013, 31(8): 468-478. DOI:10.1016/j.tibtech.2013.05.005 |
| [28] | Wang YY, Cao XP, Feng YB, et al. Environment-induced conformational and functional changes of l-2-haloacid dehalogenase. J Biosci Bioeng, 2016, 121(5): 491-496. DOI:10.1016/j.jbiosc.2015.09.008 |
| [29] | Toyoshima C, Nakasako M, Nomura H, et al. Crystal structure of the calcium pump of sarcoplasmic reticulum at 2.6 ? resolution. Nature, 2000, 405(6787): 647-655. DOI:10.1038/35015017 |
| [30] | Hisano T, Hata Y, Fujii T, et al. Crystallization and preliminary X-ray crystallographic studies of L-2-haloacid dehalogenase from Pseudomonas sp. YL. Proteins, 1996, 24(4): 520-522. DOI:10.1002/(SICI)1097-0134(199604)24:4<520::AID-PROT12>3.0.CO;2-N |
| [31] | Ridder IS, Rozeboom HJ, Kalk KH, et al. Three-dimensional structure of L-2-haloacid dehalogenase from Xanthobacter autotrophicus GJ10 complexed with the substrate-analogue formate. J Biol Chem, 1997, 272(52): 33015-33022. DOI:10.1074/jbc.272.52.33015 |
| [32] | Schmidberger JW, Wilce JA, Tsang JSH, et al. Crystal structures of the substrate free-enzyme, and reaction intermediate of the HAD superfamily member, haloacid dehalogenase dehiva from Burkholderia cepacia MBA4. J Mol Biol, 2007, 368(3): 706-717. DOI:10.1016/j.jmb.2007.02.015 |
| [33] | Rye C, Isupov M, Lebedev A, et al. Biochemical and structural studies of a L-haloacid dehalogenase from the thermophilic archaeon Sulfolobus tokodaii. Extremophiles, 2009, 13(1): 179-190. DOI:10.1007/s00792-008-0208-0 |
| [34] | Poelarends GJ, Whitman CP. 8. 04-Mechanistic and structural studies of microbial dehalogenases: how nature cleaves a carbon-halogen bond//Liu HW, Mander L, eds. Comprehensive Natural Products Ⅱ. Oxford: Elsevier, 2010: 89-123. |
| [35] | Zhang CC, Allen KN, Dunaway-Mariano D. Mechanism of substrate recognition and catalysis of the haloalkanoic acid dehalogenase family member α-phosphoglucomutase. Biochemistry, 2018, 57(30): 4504-4517. DOI:10.1021/acs.biochem.8b00396 |
| [36] | Janssen DB. Evolving haloalkane dehalogenases. Curr Opin Chem Biol, 2004, 8(2): 150-159. DOI:10.1016/j.cbpa.2004.02.012 |
| [37] | Kurihara T. A mechanistic analysis of enzymatic degradation of organohalogen compounds. Biosci Biotechnol Biochem, 2011, 75(2): 189-198. DOI:10.1271/bbb.100746 |
| [38] | Kondo H, Nakamura T, Tanaka S. A significant role of Arg41 residue in the enzymatic reaction of haloacid dehalogenase l-DEX YL studied by QM/MM method. J Mol Catal B Enzym, 2014, 110: 23-31. DOI:10.1016/j.molcatb.2014.09.006 |
| [39] | Liu JQ, Kurihara T, Hasan AK, et al. Purification and characterization of thermostable and nonthermostable 2-haloacid dehalogenases with different stereospecificities from Pseudomonas sp. strain YL. Appl Environ Microbiol, 1994, 60(7): 2389-2393. DOI:10.1128/AEM.60.7.2389-2393.1994 |
| [40] | Chiba Y, Yoshida T, Ito N, et al. Isolation of a bacterium possessing a haloacid dehalogenase from a marine sediment core. Microbes Environ, 2009, 24(3): 276-279. DOI:10.1264/jsme2.ME09123 |
| [41] | Nardi-Dei V, Kurihara T, Park C, et al. DL-2-haloacid dehalogenase from Pseudomonas sp. 113 is a new class of dehalogenase catalyzing hydrolytic dehalogenation not involving enzyme-substrate ester intermediate. J Biol Chem, 1999, 274(30): 20977-20981. DOI:10.1074/jbc.274.30.20977 |
| [42] | Schmidberger JW, Wilce JA, Weightman AJ, et al. The crystal structure of DehI reveals a new α-haloacid dehalogenase fold and active-site mechanism. J Mol Biol, 2008, 378(1): 284-294. DOI:10.1016/j.jmb.2008.02.035 |
| [43] | Siwek A, Omi R, Hirotsu K, et al. Binding modes of DL-2-haloacid dehalogenase revealed by crystallography, modeling and isotope effects studies. Arch Biochem Biophysics, 2013, 540(1/2): 26-32. |
| [44] | Weightman AJ, Weightman AL, Slater JH. Stereospecificity of 2-monochloropropionate dehalogenation by the two dehalogenases of Pseudomonas putida PP3: evidence for two different dehalogenation mechanisms. J Gen Microbiol, 1982, 128(8): 1755-1762. |
| [45] | Park C, Kurihara T, Yoshimura T, et al. A new DL-2-haloacid dehalogenase acting on 2-haloacid amides: purification, characterization, and mechanism. J Mol Catal B: Enzym, 2003, 23(2/6): 329-336. |
| [46] | Haziyamin T, Hamid TA, Hamid AAA, et al. A review on non-stereospecific haloalkanoic acid dehalogenases. Afr J Biotechnol, 2013, 10(48): 9725-9736. DOI:10.5897/AJB11.934 |
| [47] | Brokamp A, Happe B, Schmidt FRJ. Cloning and nucleotide sequence of a D, L-haloalkanoic acid dehalogenase encoding gene from Alcaligenes xylosoxidans ssp. denitrificans ABIV. Biodegradation, 1997, 7(5): 383-396. DOI:10.1007/BF00056422 |
| [48] | Leigh JA, Skinner AJ, Cooper RA. Isolation and partial characterisation of dehalogenase-deficient mutants of a Rhizobium sp. FEMS Microbiol Lett, 1986, 36(2/3): 163-166. |
| [49] | Sudi IY, Hamid AAA, Shamsir MS, et al. Insights into the stereospecificity of the D-specific dehalogenase from Rhizobium sp. RC1 toward D-and L-2-chloropropionate. Biotechnol Biotec Equip, 2014, 28(4): 608-615. DOI:10.1080/13102818.2014.937907 |
| [50] | Higgins TP, Hope SJ, Effendi AJ, et al. Biochemical and molecular characterisation of the 2, 3-dichloro-1- propanol dehalogenase and stereospecific haloalkanoic dehalogenases from a versatile Agrobacterium sp. Biodegradation, 2005, 16(5): 485-492. DOI:10.1007/s10532-004-5670-5 |
| [51] | Smith JM, Harrison K, Colby J. Purification and characterization of D-2-haloacid dehalogenase from Pseudomonas putida strain AJ1/23. J Gen Microbiol, 1990, 136(5): 881-886. |
| [52] | Tong CD, Wu JP, Yang LR, et al. Crystal structural analysis of DehDIV-R by X-ray crystallography. China Biotechnol, 2018, 38(8): 19-25 (in Chinese). 童超迪, 吴坚平, 杨立荣, 等. X射线衍射晶体法解析脱卤酶DehDIV-R结构的研究. 中国生物工程杂志, 2018, 38(8): 19-25. |
| [53] | Wang YY, Feng YB, Cao XP, et al. Insights into the molecular mechanism of dehalogenation catalyzed by D-2-haloacid dehalogenase from crystal structures. Sci Rep, 2018, 8: 1454. DOI:10.1038/s41598-017-19050-x |
| [54] | Blaber M, Lee J, Longo LM. Emergence of symmetric protein architecture from a simple peptide motif: evolutionary models. Cell Mol Life Sci, 2012, 69(23): 3999-4006. |
| [55] | Huyop F, Sudi IY. D-specific dehalogenases, a review. Biotechnol Biotec Equip, 2012, 26(2): 2817-2822. |
| [56] | Bommarius AS. Biocatalysis: A status report. Annu Rev Chem Biomol, 2015, 6(1): 319-345. DOI:10.1146/annurev-chembioeng-061114-123415 |
| [57] | Tanokura M, Miyakawa T, Guan LJ, et al. Structural analysis of enzymes used for bioindustry and bioremediation. Biosci Biotechnol Biochem, 2015, 79(9): 1391-1401. |
| [58] | Wen H, Bian QH, Wang M, et al. Synthesis and application of optical purity 2-chloropropionic acid in chiral pesticides. Chin J Pest, 2004, 43(8): 363-366 (in Chinese). 文辉, 边庆花, 王敏, 等. 光学纯2-氯丙酸及其酯的合成与在手性农药中的应用. 农药, 2004, 43(8): 363-366. DOI:10.3969/j.issn.1006-0413.2004.08.010 |
| [59] | Gong YH, Ma SM, Wang YF, et al. Characterization of a novel deep-sea microbial esterase EstC10 and its use in the generation of (R)-methyl2-chloropropionate. J Ocean Limnol, 2018, 36(2): 473-482. DOI:10.1007/s00343-018-6297-4 |
| [60] | Parker K, Colby J. Immobilization of the D-2-haloacid dehalogenase from Pseudomonas putida strain AJ1/23. Biodegradation, 1995, 6(3): 191-201. DOI:10.1007/BF00700457 |
| [61] | Taylor SC. D-2-haloalkanoic acid halidohydrolase: US, 4758518. 1988-07-19. |
| [62] | Breuer M, Ditrich K, Habicher T, et al. Industrial methods for the production of optically active intermediates. Angew Chem Int Edit, 2004, 43(7): 788-824. |
| [63] | Martinez FAC, Balciunas EM, Salgado JM, et al. Lactic acid properties, applications and production: a review. Trends Food Sci Technol, 2013, 30(1): 70-83. |
| [64] | Xie GQ, Pan D, He WL, et al. Application of L-2-haloacid dehalogenase from thermophilic Archaea Sulfolobus tokodaii in the production of D-lactic acid. Chem J Chin Univ, 2015, 36(4): 698-703 (in Chinese). 解桂秋, 潘东, 何文龙, 等. 嗜热古细菌Sulfolobus tokodaii脱卤酶在D-乳酸生产中的应用. 高等学校化学学报, 2015, 36(4): 698-703. |
| [65] | Mohamed E, Rahiman FA, Wahab RA, et al. A plant transformation vector containing the gene dehd for the development of cultivars resistant to monochloroacetic acid. J Anim Plant Sci, 2016, 26(4): 1133-1139. |
| [66] | Bidmanova S, Chaloupkova R, Damborsky J, et al. Development of an enzymatic fiber-optic biosensor for detection of halogenated hydrocarbons. Anal Bioanal Chem, 2010, 398(5): 1891-1898. |
| [67] | Martincová E, Voleman L, Najdrová V, et al. Live imaging of mitosomes and hydrogenosomes by halotag technology. PLoS ONE, 2012, 7(4): e36314. DOI:10.1371/journal.pone.0036314 |
| [68] | Mazzucchelli S, Colombo M, Verderio P, et al. Orientation-controlled conjugation of haloalkane dehalogenase fused homing peptides to multifunctional nanoparticles for the specific recognition of cancer cells. Angew Chem Int Edit, 2013, 52(11): 3121-3125. DOI:10.1002/anie.201209662 |
| [69] | Kondo H, Fujimoto KJ, Tanaka S, et al. Theoretical prediction and experimental verification on enantioselectivity of haloacid dehalogenase l-DEX YL with chloropropionate. Chem Phys Lett, 2015, 623: 101-107. DOI:10.1016/j.cplett.2015.01.053 |
| [70] | Feng YB, Wang YY, Chu HY, et al. Stereoselective catalysis controlled by a native leucine or variant isoleucine wing-gatekeeper in 2-haloacid dehalogenase. FEBS Lett, 2019, 593(3): 308-318. DOI:10.1002/1873-3468.13309 |
