

1. 江南大学 药学院,江苏 无锡 214122;
2. 江南大学 粮食与发酵国家工程实验室,江苏 无锡 214122;
3. 中国科学院天津工业生物技术研究所,天津 300308
收稿日期:2020-03-23;接收日期:2020-05-22;网络出版时间:2020-06-05
基金项目:国家重点研发计划(No. 2018YFA0900300)资助
摘要:氨基酸是一类在食品、医药及化工等领域具有广泛应用的重要化合物。谷氨酸棒杆菌Corynebacterium glutamicum是生物合成氨基酸最重要的微生物菌株,其年产各类氨基酸超过百万吨。谷氨酸棒杆菌高产氨基酸除具有强大的合成代谢能力外,高效的分泌转运能力也是不可忽略的分子基础。文中综述了近年来谷氨酸棒杆菌中氨基酸分泌转运蛋白及其代谢改造的研究进展,并展望了未来发展方向,为进一步改造提升其发酵生产氨基酸的能力提供了可资借鉴的资料。
关键词:谷氨酸棒杆菌氨基酸分泌转运转运蛋白代谢改造
Amino acid exporters and metabolic modification of Corynebacterium glutamicum-a review
Xiaomei Zhang1, Yujie Gao1, Ling Yang1, Yi Yang1, Ping Zheng3, Jibin Sun3, Jinsong Shi1, Zhenghong Xu2


1. School of Pharmaceutics Science, Jiangnan University, Wuxi 214122, Jiangsu, China;
2. National Engineering Laboratory for Cereal Fermentation Technology, Jiangnan University, Wuxi 214122, Jiangsu, China;
3. Key Laboratory of Systems Microbial Biotechnology, Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China
Received: March 23, 2020; Accepted: May 22, 2020; Published: June 5, 2020
Supported by: National Key Research and Development Program of China (No. 2018YFA0900300)
Corresponding author: Zhenghong Xu. Tel/Fax: +86-510-85198206; E-mail: zhenghxu@jiangnan.edu.cn.
Abstract: Amino acids are important compounds with a wide range of applications in the food, medicine and chemical industries. Corynebacterium glutamicum is a powerful workhorse commonly used in industrial amino acid production, with the scale of more than one million tons. In addition to its efficient anabolism, the effective exporters also ensure the high amino acid production by C. glutamicum. In this review, the research progress of amino acid exporter of C. glutamicum is summarized, to provide the foundation for further improving amino acid production by C. glutamicum via metabolic engineering.
Keywords: Corynebacterium glutamicumamino acideffluxexportermetabolic modification
谷氨酸棒杆菌是发酵生产氨基酸最为重要和安全的菌株,通过代谢改造提高它过量合成各种氨基酸的能力一直是研究热点,而对其产物分泌的关注较少。氨基酸的高效分泌可有效降低胞内产物浓度对代谢途径中重要节点的反馈抑制和阻遏作用。已有研究表明氨基酸的分泌主要通过细胞膜上的转运蛋白完成。由于膜蛋白的跨膜结构特性,传统方法难以对其深入研究。近年来,随着谷氨酸棒杆菌全基因组序列的解析,可通过生物信息学的方法预测膜蛋白及其可能的转运底物,这为氨基酸转运蛋白的研究提供了坚实的基础[1-4]。目前,在谷氨酸棒杆菌中共发现了6种氨基酸分泌转运蛋白,它们参与12种氨基酸的分泌转运[5-13] (图 1),仍有约一半重要氨基酸的分泌转运蛋白尚未见研究报道。本文重点对近年来谷氨酸棒杆菌中氨基酸转运蛋白家族、转运蛋白的挖掘、代谢改造、结构特征以及调控机制等方面进行综述,并展望了该领域未来发展趋势。
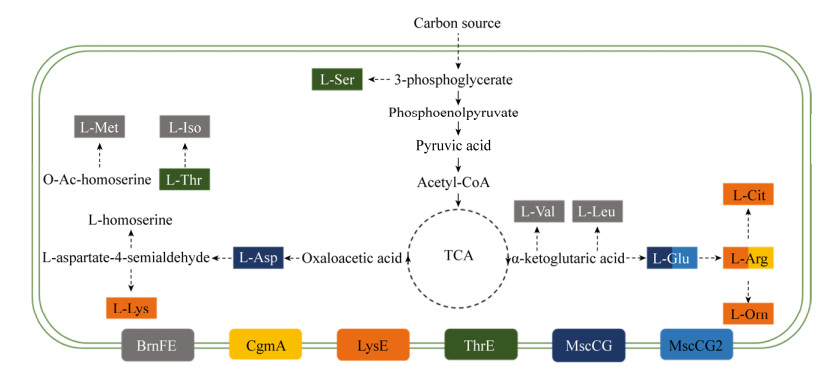 |
| 图 1 谷氨酸棒杆菌氨基酸分泌转运蛋白 Fig. 1 Amino acid exporters of Corynebacterium glutamicum. BrnFE (Gray) belongs to LIV-E family (The Branched Chain Amino Acid Exporter Family), the exporter of L-valine, L-leucine, L-isoleucine, L-methionine; CgmA (Yellow) belongs to MFS family (The Major Facilitator Superfamily), the exporter of L-arginine. LysE (Orange) belongs to LysE Family (The Lysine Exporter Family), the exporter of L-lysine, L-arginine, L-citrulline and L-orithine. ThrE (Green) belongs to ThrE family (Threonine/Serine Exporter Family), the exporter of L-threonine and L-serine. MscCG (Dark blue) and MscCG2 (Blue) belong to the MscS family (Mechanically Sensitive Channel Proteins), the exporter of L-glutamate and aspartate. |
| 图选项 |
1 谷氨酸棒杆菌中氨基酸转运蛋白家族谷氨酸棒杆菌的基因组包含约2 900个蛋白编码基因,预测至少有750个基因编码的蛋白定位于细胞膜上,可能具有转运蛋白(或转运蛋白亚基)的功能。上述蛋白可分为57个不同的转运蛋白家族,其中10个可能参与氨基酸的分泌转运(www.membranetransport.org) (表 1)。
表 1 谷氨酸棒杆菌中的氨基酸分泌转运蛋白家族Table 1 The amino acid exporter families of C. glutamicum
 |
表选项
在谷氨酸棒杆菌中NCgl0565与NCgl2130编码的蛋白属于AAE家族(天冬氨酸/丙氨酸转运蛋白家族),因其与天冬氨酸/丙氨酸转运蛋白AspT序列具有相似性,预测其参与L-天冬氨酸和L-丙氨酸的分泌转运[14]。NCgl1101和NCgl1102编码的蛋白属于ABC家族(ATP结合盒式超家族),因其与大肠杆菌中半胱氨酸分泌转运系统CydDC具有较高的序列相似性,预测具有L-半胱氨酸和谷胱甘肽的分泌转运能力[15]。NCgl0580编码的蛋白属于DMT家族(药物/代谢物转运超家族),论文作者近期研究表明它参与了L-丝氨酸的分泌转运,但其详细信息仍有待研究[16]。NCgl0464编码的蛋白属于HAAAP家族(羟基/芳香族氨基酸透性酶家族),预测其参与芳香族氨基酸分泌转运,而实验证明它具有分泌转运γ-氨基丁酸的功能[17]。NCgl0255和NCgl0254编码的BrnFE属于LIV-E家族(支链氨基酸分泌转运家族),已有研究证明它们具有分泌转运L-甲硫氨酸、L-异亮氨酸、L-亮氨酸和L-缬氨酸的功能[5, 12]。NCgl1214编码的蛋白LysE属于LysE超家族(赖氨酸分泌转运载体家族),具有分泌转运L-赖氨酸、L-精氨酸的功能[7, 18],同时也参与分泌转运L-瓜氨酸和L-鸟氨酸[6, 19];NCgl0143编码的蛋白也属于LysE家族,预测具有L-苏氨酸的分泌转运能力[15]。NCgl1221编码的蛋白MscCG与MscCG2属于MscS家族(机械敏感通道蛋白),具有分泌转运L-谷氨酸的功能;另外MscCG还参与转运L-天冬氨酸,预测它还具备转运L-苯丙氨酸的功能[8, 11, 20]。蛋白CgmA属于MFS家族(主要超易化家族),具有分泌转运L-精氨酸的能力[6]。NCgl2662和NCgl2566编码的蛋白属于RthB家族(耐受高丝氨酸/苏氨酸家族),预测它们参与高丝氨酸和L-亮氨酸分泌转运[15, 21]。NCgl2533编码的蛋白ThrE属于ThrE家族(苏氨酸/丝氨酸分泌转运家族),已证明具有分泌转运L-苏氨酸和L-丝氨酸的功能[9]。
2 氨基酸转运蛋白的挖掘及其代谢改造氨基酸的分泌主要通过细胞膜上的转运蛋白完成,挖掘氨基酸转运蛋白,对其代谢调控机制进行深入研究,为进一步氨基酸菌株高效改造提供更多新的思路。
2.1 谷氨酸棒杆菌分泌转运蛋白的挖掘方法系统挖掘谷氨酸棒杆菌的氨基酸转运蛋白对进一步提高其氨基酸产量至关重要[1-2]。通常采用的转运蛋白挖掘方法有:(1)基于全基因组测序结果,采用生物信息学工具预测获得转运蛋白的编码基因信息并对其进行克隆,构建相应的基因敲除和回补菌株,鉴定其功能,确定其编码蛋白是否具有相应氨基酸的转运能力[22];(2)基于氨基酸序列相似性的挖掘方法,即根据其他物种中已明确功能的转运蛋白的氨基酸一级序列进行类比,预测新的氨基酸转运蛋白,然后验证其功能,如已经鉴定的L-半胱氨酸转运蛋白[23];(3)藉由转录组学测序的转运蛋白挖掘,通过在培养基中添加特定氨基酸多肽或不同发酵阶段菌株转录水平的变化,筛选表达上调的膜蛋白,并进行功能鉴定,转运蛋白BrnFE和SucE就是这样鉴别出来的[5, 24];(4)基于基因组文库的转运蛋白挖掘,采用谷氨酸棒杆菌基因组DNA构建随机片段文库,筛选出对产物积累有显著促进的重组谷氨酸棒杆菌,并进一步从重组DNA片段中鉴别新型转运蛋白编码基因[25];(5)从突变菌株中挖掘转运蛋白,对随机突变菌株中筛选对某种氨基酸或其类似物抗性增加的突变株,分析基因型及表型变化,进而鉴定出与之相关的转运蛋白,如大肠杆菌RhtA以及谷氨酸棒杆菌ThrE的鉴定[9, 26]。
2.2 L-赖氨酸、L-精氨酸、L-瓜氨酸和L-鸟氨酸的分泌转运及其代谢改造L-赖氨酸、L-精氨酸、L-瓜氨酸和L-鸟氨酸广泛应用在食品、医药和化妆品等领域,主要由谷氨酸棒杆菌发酵生产[3-4]。LysE是第一个被鉴定出来的谷氨酸棒杆菌中氨基酸的分泌转运蛋白,属于典型转运载体LysE超家族,具有转运L-赖氨酸与L-精氨酸的功能;LysE的转录受到染色体组中与之相邻的LysG激活,LysG具有自调控转录调控因子的结构特征,属于LysR转录因子家族(LTTR)[18]。L-赖氨酸、L-精氨酸和L-组氨酸都可诱导LysE的表达,但L-组氨酸却不是LysE蛋白的转运底物,LysE对L-赖氨酸和L-精氨酸转运能力相似,相对转运速率均约为0.75 nmol/(min·mg)[7, 18]。值得注意的是,LysE在转运赖氨酸的过程中并不识别其手性,既能转运L-赖氨酸又能转运D-赖氨酸[27];敲除LysE的菌株胞内会过量积累赖氨酸,导致细胞生长缓慢;通过增强LysE表达可提高精氨酸产量至35.9 g/L,提高幅度为13.6%[28]。近期研究表明,LysE还参与了L-瓜氨酸和L-鸟氨酸的转运,通过增强其表达可将L-瓜氨酸和L-鸟氨酸的产量分别提高45%和21.8%;通过在LysE上游添加强启动子也可提高L-鸟氨酸产量,而敲除LysE则会导致L-瓜氨酸产量下降,但LysE作为这两个氨基酸转运蛋白的性质还有待深入研究[6, 19]。在谷氨酸棒杆菌中,加强表达腐胺转运透性酶CgmA可以回补LysE的缺失。此外,在敲除LysE的谷氨酸棒杆菌中表达CgmA可以解除外源添加精氨酸二肽引发的细胞毒性,说明CgmA也参与了L-精氨酸转运。CgmA表达又会受到转录因子CgmR的抑制,敲除CgmR可以提高L-精氨酸的产量[6]。LysE和CgmA双敲除的谷氨酸棒杆菌仍可分泌L-精氨酸,提示菌株中还存在着未知的L-精氨酸分泌转运蛋白[6]。
2.3 L-苏氨酸和L-丝氨酸的分泌转运及其代谢改造L-苏氨酸和L-丝氨酸是重要的含羟基的氨基酸。Simic等对一株谷氨酸棒杆菌进行诱变,分离筛选具有L-苏氨酸抗性的突变株,再通过分析其表型及基因型变化,鉴定出了L-苏氨酸转运蛋白ThrE;由于L-丝氨酸的化学结构与L-苏氨酸相似,Simic等发现敲除ThrE后,重组菌对L-丝氨酸转运速率为0.6 nmol/(min·DCW),而对照菌株中为1.4 nmol/(min·DCW),说明ThrE也具有转运L-丝氨酸的能力[9]。该团队通过在C. glutamicum DR-17中增强表达ThrE,并结合其他代谢改造,使得L-苏氨酸产量由5.8 g/L提高至8.1 g/L[29]。论文作者发现在谷氨酸棒杆菌?SSAAI中增强表达或者敲除ThrE,对L-丝氨酸产量的影响均不显著,说明ThrE可能不是主要的L-丝氨酸转运蛋白。近期研究发现NCgl0580编码蛋白参与了L-丝氨酸的分泌转运,其生理生化特性的研究仍在进行之中[16]。
在谷氨酸棒杆菌中引入异源L-苏氨酸转运蛋白也可以有效促进产物分泌及合成,将大肠杆菌中L-苏氨酸转运蛋白(RhtA、RhtB、RhtC和YeaS)基因克隆至谷氨酸棒杆菌中,结果表明RhtA和RhtC对胞外L-苏氨酸的积累影响显著,说明其在谷氨酸棒杆菌中同样具有转运L-苏氨酸的能力,此外还发现了RhtA具有转运L-高丝氨酸的功能[26]。Diesveld等在C. glutamicum DM368-3中异源表达RhtC,L-苏氨酸产量由0.9 g/L提高至3.7 g/L[30]。
2.4 支链氨基酸和L-甲硫氨酸的分泌转运及其代谢改造支链氨基酸(BCAAs)包括L-亮氨酸、L-异亮氨酸、L-缬氨酸,因其疏水脂质链中具有分支的甲基基团,被称为支链氨基酸[3]。在谷氨酸棒杆菌中,支链氨基酸与L-甲硫氨酸的分泌转运主要由BrnFE完成,该转运蛋白属于LIV-E家族;BrnFE是由NCgl0255 (BrnF)和NCgl0254 (BrnE)两个基因编码的膜蛋白组成的复合体,任何一个基因的缺失或加强表达都会影响氨基酸的分泌转运;BrnFE转运L-异亮氨酸、L-亮氨酸和L-甲硫氨酸的速率相当,而对L-缬氨酸的转运速率则较低[5, 12, 31]。BrnFE的表达受到转录因子Lrp的激活,胞内L-亮氨酸可以诱导BrnFE的表达,而加强表达BrnFE并不能显著提高L-亮氨酸的产量,说明天然的BrnFE活性足以转运过量合成的L-亮氨酸[32],Ma等发现Lrp以及BrnFE的拷贝数的增加有利于L-亮氨酸的合成[33]。当胞内L-甲硫氨酸不超过50 mmol/L时,BrnFE是L-甲硫氨酸的主要转运蛋白;通过加强表达BrnFE可使得L-甲硫氨酸产量增加1.3倍[34]。通过敲除谷氨酸棒杆菌中L-异亮氨酸的吸收转运蛋白BrnQ同时增强表达分泌转运蛋白BrnFE,可积累221 mmol/L L-异亮氨酸,加强表达Lrp和BrnFE的重组菌株可积累205 mmol/L L-异亮氨酸[35-37]。此外,加强表达Lrp及BrnFE能够显著提高谷氨酸棒杆菌L-缬氨酸的产量,可达461.4 mmol/L[38]。
2.5 L-谷氨酸和L-天冬氨酸的分泌转运及其代谢改造L-谷氨酸和L-天冬氨酸是重要的食品添加剂[3-4]。Nakamura等发现L-谷氨酸与其他氨基酸的分泌转运不同,其转运是通过机械敏感通道蛋白MscCG (Mechanosensitive channel)完成。这种组成型表达的转运蛋白在正常生理条件下并不转运L-谷氨酸。当培养基中添加表面活性剂、亚致死浓度的青霉素、抗肺结核(TB)药物乙胺丁醇或者生物素受限时,细胞膜张力发生变化导致分泌转运蛋白变构,启动L-谷氨酸分泌。当敲除MscCG编码基因NCgl1221时,谷氨酸棒杆菌ATCC13869仍可积累少量的L-谷氨酸,说明除了MscCG外,菌株中还存在着其他L-谷氨酸转运蛋白[13]。最近中国科学院天津工业生物技术研究所发现了一种新型的L-谷氨酸分泌转运蛋白MscCG2,MscCG2也具有机械敏感通道典型的结构特征;虽然与MscCG在序列和结构上存在显著的差异,但却有着相似的转录模式;另外MscCG还可参与转运L-天冬氨酸[8]。对谷氨酸棒杆菌中MscCG进行定向突变并加强表达,L-谷氨酸的产量可提高至130 mmol/L[39]。
迄今为止,重要氨基酸分泌转运蛋白的挖掘方兴未艾。通过代谢改造谷氨酸棒杆菌中氨基酸分泌转运蛋白提高各类氨基酸产量的研究结果总结如表 2。
表 2 应用转运蛋白代谢改造谷氨酸棒杆菌生产氨基酸的研究Table 2 Results of modification of amino acid transporters in C. glutamicum
| Products | Exporter and modification | Titer (g/L) | Improvement (%) | References |
| L-lysine | LysE (+) | 6.00 | 21.0 | [40] |
| L-arginine | MscCG (–), LysE (+) | 24.90 | 106.8 | [41] |
| LysE (+) | 35.90 | 13.6 | [28] | |
| L-ornithine | LysE (+) | 25.00 | 63.4 | [19] |
| L-threonine | LysE (–) | 5.52 | 28.0 | [42] |
| ThrE (+) | 8.10 | 49.0 | [29] | |
| RthC (From E. coli) | 3.70 | 157.0 | [30] | |
| L-citrulline | LysE (+) | 2.98 | 45.0 | [6] |
| L-isoleucine | BrnFE (+), BrnQ (–) | 29.00 | 30.0 | [36] |
| BrnFE (+), Lrp (+) | 26.90 | 10.7 | [37] | |
| L-valine | BrnFE (+), Lrp (+) | 54.05 | 115.3 | [38] |
| L-methionine | BrnFE (+) | 6.30 | 30.0 | [34] |
| L-glutamate | MscCG (+) | 19.12 | 381.5 | [39] |
| +: overexpression of the gene; -: knockout of the gene. | ||||
表选项
3 氨基酸转运蛋白的结构特征及调控机制氨基酸的高效分泌至关重要,转运蛋白改造是氨基酸工程菌株代谢改造的关键靶点。解析转运蛋白的结构特征并阐明转运蛋白的调控机制可为进一步高效改造提升氨基酸产量奠定坚实的理论和应用基础[1-2]。
3.1 氨基酸分泌转运蛋白的结构特征及其功能关系氨基酸为极性物质,不能自由通过细胞膜,故其胞外分泌需通过转运蛋白协助完成。解析转运蛋白的结构及其功能,阐明氨基酸转运调控机制十分重要。
在谷氨酸棒杆菌中,氨基酸分泌主要通过二级转运系统起作用,以跨细胞膜电化学离子势为驱动力,一般为H+或Na+电化学势[1]。唯一例外的是谷氨酸的转运,它是藉由通道蛋白进行分泌转运[13]。氨基酸转运蛋白通常是具有多次跨膜结构的整合型膜蛋白,包含一个或多个亚基,各亚基都有跨膜α螺旋。迄今为止,仅有大肠杆菌等极少数微生物中氨基酸转运蛋白的结构被研究鉴定,尚未见谷氨酸棒杆菌中转运蛋白的结构解析报道。通过晶体结构解析发现,大肠杆菌中L-丙氨酸转运蛋白AlaE的45、84位带电荷氨基酸是保持其转运活性的必要基团,而22、28位的氨基酸残基对其转运L-丙氨酸没有影响[43]。杨茂君教授报道了来自腾冲嗜热厌氧菌Thermoanaerobacter tengcongensis中的一种氨基酸转运蛋白ABC与底物结合状态下复合物的晶体结构,发现转运蛋白跨膜区结构域同时存在两个底物结合位点,这些跨膜区底物结合位点关键氨基酸残基的突变对于ArtI-Art(QN)2复合物ATPase的活性有显著影响,为氨基酸转运蛋白的理性改造提供了重要依据[44]。
通过生物信息学分析预测转运蛋白的基本结构,并对转运蛋白相关位点进行突变改造,也可以获得转运蛋白与底物转运之间的关系。大肠杆菌转运蛋白YddG可以转运L-苏氨酸、L-甲硫氨酸、L-赖氨酸和L-谷氨酸等底物,对其进行定点突变发现,当trp101ala和trp163ala发生突变时,YddG对L-苏氨酸的转运能力显著降低;当tyr82ala发生突变时,YddG转运L-甲硫氨酸的能力增强,转运L-苏氨酸的能力下降,说明对转运蛋白进行定点改造不仅可以改变底物的转运效率而且可以改变其底物选择性[45]。已有研究表明,绝大多数氨基酸分泌转运蛋白具有非特异性,如谷氨酸棒杆菌的LysE可分泌转运L-赖氨酸、L-精氨酸等[6]。另外也存在一种氨基酸底物具有多个转运蛋白的现象,如L-谷氨酸可以由MscCG2与MscCG分泌转运[8, 13];当一种氨基酸存在多个转运蛋白时,转运蛋白之间可能是相互协调关系,也可能是竞争关系,明晰多个转运蛋白之间的关系也非常重要。通过放射性同位素标记等方法可以对转运蛋白的转运动力学进行研究,不同的转运蛋白对某一种氨基酸的Km值可能存在几个数量级的差距,某一种转运蛋白对不同的氨基酸亲和力也存在差异;另外转运蛋白在基因转录、蛋白表达水平上也存在复杂的调控机制[2, 46]。因此,转运蛋白的表达调控以及多转运蛋白之间的互相作用关系也是该领域研究的重点。
3.2 谷氨酸棒杆菌中氨基酸转运蛋白的调控机制谷氨酸棒状杆菌中氨基酸转运蛋白的表达受到转录因子及其效应物的严格调控[2, 46],而相较于氨基酸转运蛋白,相关调控因子的鉴定更加困难。到目前为止,仅鉴定了3个在谷氨酸棒杆菌中参与氨基酸分泌转运的调控因子,即与L-赖氨酸和L-精氨酸等分泌转运相关的LysE的调控因子LysG、L-精氨酸分泌转运蛋白CgmA的调控因子CgmR、支链氨基酸分泌转运蛋白BrnFE的调控因子Lrp,这些转运蛋白及其调控因子都位于同一个操纵子中或是染色体中相邻的位置。在鉴定这类转运蛋白时,相应的调控因子即被发现[6-7, 31]。LysE受到上游的LysR家族转录调节因子LysG的激活,当胞内L-赖氨酸、L-精氨酸或L-组氨酸达到一定浓度时都可诱导LysE的表达;但L-组氨酸却不是LysE的运输底物,其分泌转运蛋白尚未被鉴定出来[18]。除LysE外,谷氨酸棒杆菌中还存在一个L-精氨酸分泌转运蛋白CgmA,CgmA还可以分泌转运二胺类物质,其表达受到转录因子CgmR的抑制,但受到二胺类物质的激活。支链氨基酸分泌转运蛋白BrnFE的表达受转录因子Lrp的激活,L-亮氨酸、L-甲硫氨酸、L-异亮氨酸及L-缬氨酸都可以诱导BrnFE的表达,其诱导效果为L-亮氨酸 > L-甲硫氨酸 > L-异亮氨酸 > L-缬氨酸[31]。在谷氨酸棒状杆菌中共表达Lrp和BrnFE,L-异亮氨酸的产量提高63%,在补料分批发酵过程中,L-异亮氨酸的产量提高72%[31, 36]。论文作者近期研究发现,L-丝氨酸的转运蛋白NCgl0580受到其上游序列NCgl0581的调控,其调控机制尚不清晰[16]。转录因子的调控作用随着细胞内效应物的浓度变化而发生改变,而只有当效应物存在或达到一定浓度时,这些基因的转录才被激活。在谷氨酸棒杆菌发酵生产氨基酸的研究过程中,转运蛋白及其调控因子俨然成为代谢改造的关键靶点[1-2]。
4 总结与展望谷氨酸棒杆菌强大的氨基酸代谢和分泌转运能力是其高产的分子基础。氨基酸转运蛋白的鉴定与改造是近年来氨基酸代谢改造的研究热点[1-2]。此外,分泌转运蛋白调控对相应氨基酸衍生物的合成也至关重要。通过失活特定氨基酸的分泌转运蛋白,可使该氨基酸在胞内大量积累,为其衍生物的合成提供更为充足的前体,如敲除谷氨酸棒杆菌赖氨酸分泌转运蛋白LysE,可显著提高赖氨酸衍生物哌啶酸的产量[47]。而到目前为止,仅在谷氨酸棒杆菌中发现了参与12种氨基酸分泌转运的6种蛋白[5-13],说明该领域的研究仍有相当大的空间。转运蛋白通常受到转录因子及其效应物的严格调控,深入理解其调控机制可为氨基酸工程菌的理性改造提供新的理论依据[1-2]。而异源转运蛋白在谷氨酸棒杆菌中功能的挖掘与应用则可为解决现有转运蛋白不足的瓶颈问题开辟新的思路。
今后,该领域的研究可以重点从以下几个方面入手:(1)挖掘并鉴定全新的转运蛋白或已有转运蛋白的新的功能及其调控机制;(2)阐明转运蛋白结构与功能之间的关系,理性设计并改造转运蛋白;(3)精确调控转运蛋白表达,协调胞内产物高效分泌转运与细胞生长、代谢等生理性状之间的关系;(4)引入外源转运系统,研究其在重组菌中氨基酸的转运能力及调控,以及与宿主细胞的生理匹配性。可以预见,随着对氨基酸转运蛋白的研究深入,必将为我国氨基酸工业菌株高效改造以及氨基酸产业的技术升级提供更多新的思路和策略。
参考文献
| [1] | Eggeling L. Exporters for production of amino acids and other small molecules//Yokota A, Ikeda M. Amino Acid Fermentation. Tokyo: Springer, 2016: 199-225. |
| [2] | Pérez-García F, Wendisch VF. Transport and metabolic engineering of the cell factory Corynebacterium glutamicum. FEMS Microbiol Lett, 2018, 365(16): fny166. |
| [3] | D'este M, Alvarado-Morales M, Angelidaki I. Amino acids production focusing on fermentation technologies a review. Biotechnol Adv, 2018, 36(1): 14-25. |
| [4] | Wendisch VF. Metabolic Engineering advances and prospects for amino acid production. Metab Eng, 2020, 58: 17-34. DOI:10.1016/j.ymben.2019.03.008 |
| [5] | Kennerknecht N, Sahm H, Yen MR, et al. Export of L-isoleucine from Corynebacterium glutamicum: A two-gene-encoded member of a new translocator family. J Bacteriol, 2002, 184(14): 3947-3956. DOI:10.1128/JB.184.14.3947-3956.2002 |
| [6] | Lubitz D, Jorge JMP, Pérez-García F, et al. Roles of export genes cgmA and LysE for the production of L-arginine and L-citrulline by Corynebacterium glutamicum. Appl Microbiol Biotechnol, 2016, 100(19): 8465-8474. DOI:10.1007/s00253-016-7695-1 |
| [7] | Vrljic M, Sahm H, Eggeling L. A new type of transporter with a new type of cellular function: L-lysine export from Corynebacterium glutamicum. Mol Microbiol, 1996, 22(5): 815-826. DOI:10.1046/j.1365-2958.1996.01527.x |
| [8] | Wang Y, Cao GQ, Xu DY, et al. A novel Corynebacterium glutamicum L-glutamate exporter. Appl Environ Microbiol, 2018, 84(6): 15. |
| [9] | Simic P, Sahm H, Eggeling L. L-threonine export: Use of peptides to identify a new translocator from Corynebacterium glutamicum. J Bacteriol, 2001, 183(18): 5317-5324. DOI:10.1128/JB.183.18.5317-5324.2001 |
| [10] | Eggeling L, Sahm H. New ubiquitous translocators: Amino acid export by Corynebacterium glutamicum and Escherichia coli. Arch Microbiol, 2003, 180(3): 155-160. |
| [11] | Nakayama Y, Yoshimura K, Iida H. A gain-of-function mutation in gating of Corynebacterium glutamicum NCgl1221 causes constitutive glutamate secretion. Appl Environ Microbiol, 2012, 78(15): 5432-5434. DOI:10.1128/AEM.01310-12 |
| [12] | Tr?tschel C, Deutenberg D, Bathe B, et al. Characterization of methionine export in Corynebacterium glutamicum. J Bacteriol, 2005, 187(11): 3786-3794. DOI:10.1128/JB.187.11.3786-3794.2005 |
| [13] | Nakamura J, Hirano S, Ito H, et al. Mutations of the Corynebactetium glutamicum NCgl1221 gene, encoding a mechanosensitive channel hornolog, induce L-glutarnic acid production. Appl Environ Microbiol, 2007, 73(14): 4491-4498. DOI:10.1128/AEM.02446-06 |
| [14] | Fujiki T, Nanatani K, Nishitani K, et al. Membrane topology of aspartate: Alanine antiporter AspT from Comamonas testosteroni. J Biochem, 2007, 141(1): 85-91. |
| [15] | Marin K, Kr?mer R. Amino acid transport systems in biotechnologically relevant bacteria//Wendisch VF. Amino Acid Biosynthesis-Pathways, Regulation and Metabolic Engineering. Berlin Heidelberg: Springer, 2007, 5: 289-325. |
| [16] | Chen ZW. Investigation of L-serine exporter in Corynebacterium glutamate[D]. Wuxi: Jiangnan University, 2018 (in Chinese). 陈紫薇.谷氨酸棒杆菌中L-丝氨酸转运蛋白的研究[D].无锡: 江南大学, 2018. |
| [17] | Zhao Z, Ding JY, Ma WH, et al. Identification and characterization of γ-aminobutyric acid uptake system GabPcg (NCgl0464) in Corynebacterium glutamicum. Appl Environ Microbiol, 2012, 78(8): 2596-2601. DOI:10.1128/AEM.07406-11 |
| [18] | Bellmann A, Vrlji? M, Pátek M, et al. Expression control and specificity of the basic amino acid exporter LysE of Corynebacterium glutamicum. Microbiology, 2001, 147(7): 1765-1774. DOI:10.1099/00221287-147-7-1765 |
| [19] | Zhang B, Ren LQ, Yu M, et al. Enhanced L-ornithine production by systematic manipulation of L-ornithine metabolism in engineered Corynebacterium glutamicum S9114. Bioresour Technol, 2018, 250: 60-68. DOI:10.1016/j.biortech.2017.11.017 |
| [20] | Nakayama Y, Komazawa K, Bavi N, et al. Evolutionary specialization of MscCG, an MscS-like mechanosensitive channel, in amino acid transport in Corynebacterium glutamicum. Sci Rep, 2018, 8: 12895. DOI:10.1038/s41598-018-31309-5 |
| [21] | Zhao Z. Study on aromatic amino acid-related transporters in Corynebacterium glutamicum[D]. Beijing: Graduate School of Chinese Academy of Sciences, 2011 (in Chinese). 赵智.谷氨酸棒杆菌中芳香族氨基酸相关转运蛋白的研究[D].北京: 中国科学院研究生院, 2011. |
| [22] | Dias O, Gomes D, Vila?a P, et al. Genome-wide semi-automated annotation of transporter systems. IEEE/ACM Trans Comput Biol Bioinformat, 2017, 14(2): 443-456. |
| [23] | Ohtsu I, Kawano Y, Suzuki M, et al. Uptake of L-cystine via an ABC transporter contributes defense of oxidative stress in the L-cystine export-dependent manner in Escherichia coli. PLoS ONE, 2015, 10(4). |
| [24] | Fukui K, Koseki C, Yamamoto Y, et al. Identification of succinate exporter in Corynebacterium glutamicum and its physiological roles under anaerobic conditions. J Biotechnol, 2011, 154(1): 25-34. |
| [25] | Tr?tschel C, Kandirali S, Diaz-Achirica P, et al. Glts, the sodium-coupled L-glutamate uptake system of Corynebacterium glutamicum: Identification of the corresponding gene and impact on L-glutamate production. Appl Microbiol Biotechnol, 2003, 60(6): 738-742. |
| [26] | Livshits VA, Zakataeva NP, Aleshin VV, et al. Identification and characterization of the new gene rhtA involved in threonine and homoserine efflux in Escherichia coli. Res Microbiol, 2003, 154(2): 123-135. |
| [27] | St?bler N, Oikawa T, Bott M, et al. Corynebacterium glutamicum as a host for synthesis and export of D-amino acids. J Bacteriol, 2011, 193(7): 1702-1709. |
| [28] | Xu MJ, Rao ZM, Yang J, et al. The effect of a LysE exporter overexpression on L-arginine production in Corynebacterium crenatum. Curr Microbiol, 2013, 67(3): 271-278. |
| [29] | Simic P, Willuhn J, Sahm H, et al. Identification of glyA (encoding serine hydroxymethyltransferase) and its use together with the exporter ThrE to increase L-threonine accumulation by Corynebacterium glutamicum. Appl Environ Microbiol, 2002, 68(7): 3321-3327. |
| [30] | Diesveld R, Tietze N, Fürst O, et al. Activity of exporters of Escherichia coli in Corynebacterium glutamicum, and their use to increase L-threonine production. J Mol Microbiol Biotechnol, 2009, 16(3/4): 198-207. |
| [31] | Lange C, Mustafi N, Frunzke J, et al. Lrp of Corynebacterium glutamicum controls expression of the brnFE operon encoding the export system for L-methionine and branched-chain amino acids. J Biotechnol, 2012, 158(4): 231-241. |
| [32] | Vogt M, Haas S, Klaffl S, et al. Pushing product formation to its limit: metabolic engineering of Corynebacterium glutamicum for L-leucine overproduction. Metab Eng, 2014, 22: 40-52. |
| [33] | Ma YC, Chen QX, Cui Y, et al. Comparative genomic and genetic functional analysis of industrial L-leucine-and L-valine-producing Corynebacterium glutamicum strains. J Microbiol Biotechnol, 2018, 28(11): 1916-1927. |
| [34] | Qin TY, Hu XQ, Hu JY, et al. Metabolic Engineering of Corynebacterium glutamicum strain ATCC13032 to produce L-methionine. Biotechnol Appl Biochem, 2015, 62(4): 563-573. |
| [35] | Wang XY. Strategy for improving L-isoleucine production efficiency in Corynebacterium glutamicum. Appl Microbiol Biotechnol, 2019, 103(5): 2101-2111. |
| [36] | Xie XX, Xu LL, Shi JM, et al. Effect of transport proteins on L-isoleucine production with the L-isoleucine-producing strain Corynebacterium glutamicum yilw. J Ind Microbiol Biotechnol, 2012, 39(10): 1549-1556. |
| [37] | Yin L, Shi F, Hu X, et al. Increasing L-isoleucine production in Corynebacterium glutamicum by overexpressing global regulator Lrp and two-component export system BrnFE. J Appl Microbiol, 2013, 114(5): 1369-1377. |
| [38] | Zhang HL, Li YY, Wang XY. Metabolic Engineering of L-valine synthesis and secretory pathways in Corynebacterium glutamicum for higher production. Chin J Biotech, 2018, 34(10): 1606-1619 (in Chinese). 张海灵, 李颜颜, 王小元. 代谢工程改造谷氨酸棒状杆菌合成及分泌途径生产L-缬氨酸. 生物工程学报, 2018, 34(10): 1606-1619. |
| [39] | Krumbach K, Sonntag CK, Eggeling L, et al. Crispr/Cas12a mediated genome editing to introduce amino acid substitutions into the mechanosensitive channel MscCG of Corynebacterium glutamicum. ACS Synth Biol, 2019, 8(12): 2726-2734. |
| [40] | Zhou LB, Zeng AP. Engineering a lysine-on riboswitch for metabolic control of lysine production in Corynebacterium glutamicum. ACS Synth Biol, 2015, 4(12): 1335-1340. |
| [41] | Huang MZ, Zhao Y, Li R, et al. Improvement of L-arginine production by in silico genome-scale metabolic network model guided genetic engineering. 3 Biotech, 2020, 10(3): 126. |
| [42] | Dong XY, Zhao Y, Hu JY, et al. Attenuating L-lysine production by deletion of ddh and LysE and their effect on L-threonine and L-isoleucine production in Corynebacterium glutamicum. Enzyme Microb Technol, 2016, 93. |
| [43] | Kim S, Ihara K, Katsube S, et al. Characterization of the L-alanine exporter AlaE of Escherichia coli and its potential role in protecting cells from a toxic-level accumulation of L-alanine and its derivatives. Microbiology Open, 2015, 4(4): 632-643. |
| [44] | Jie Y, Ge JP, Heuveling J, et al. Structural basis for substrate specificity of an amino acid ABC transporter. Proc Nat Acad Sci USA, 2015, 112(16): 5243-5248. DOI:10.1073/pnas.1415037112 |
| [45] | Tsuchiya H, Doki S, Takemoto M, et al. Structural basis for amino acid export by DMT superfamily transporter YddG. Nature, 2016, 534(7607): 417-420. DOI:10.1038/nature17991 |
| [46] | Toyoda K, Inui M. Regulons of global transcription factors in Corynebacterium glutamicum. Appl Microbiol Biotechnol, 2016, 100(1): 45-60. |
| [47] | Pérez-García F, Risse JM, Friehs K, et al. Fermentative production of L-pipecolic acid from glucose and alternative carbon sources. Biotechnol J, 2017, 12(7): 1600646. DOI:10.1002/biot.201600646 |
