

1. 中国科学院天津工业生物技术研究所 中国科学院系统微生物工程重点实验室, 天津 300308;
2. 中国科学院大学, 北京 100049;
3. 天津科技大学 生物工程学院, 天津 300457
收稿日期:2020-03-24;接收日期:2020-04-22
基金项目:国家自然科学基金(No. 31700080),中国博士后科学基金(No. 2017M621082),天津市企业博士后创新项目(No. TJQYBSH2017006)资助
摘要:需钠弧菌Vibrio natriegens作为近几年发展起来的一种新型生长快速底盘细胞,在合成生物学领域展现出良好的应用前景。基因组编辑是合成生物学研究中不可或缺的遗传操作手段。但是,开展需钠弧菌的合成生物学研究仍然有待进一步发展精准、高效的基因组编辑系统。针对这个问题,首先对6株需钠弧菌的生理表型进行检测,选取生长快速、表型稳定的CICC 10908菌株作为基因组编辑研究的宿主细胞。其次,建立并优化需钠弧菌自然转化系统。优化后的系统将筛选标记基因cat-sacB或KanR整合到需钠弧菌染色体上的同源重组效率分别达到4×10–5和4×10–4。再次,在优化的自然转化系统基础上,利用双向选择性筛选方法,建立了精准、高效的需钠弧菌基因组无痕编辑体系。通过测试,基因敲除、回补、插入和替换这4种不同类型基因编辑的阳性率分别为93.8%、100%、95.7%和100%。最后,需钠弧菌可以实现质粒的高效转化和消除。该工作为需钠弧菌合成生物学研究提供精准、高效的基因组无痕编辑手段。
关键词:需钠弧菌底盘细胞自然转化基因组编辑合成生物学
Construction of seamless genome editing system for fast-growing Vibrio natriegens
Fengli Wu1, Yanxia Liang1,2, Yuanyuan Zhang1, Yanan Huo3, Qinhong Wang1


1. CAS Key Laboratory of Systems Microbial Biotechnology, Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China;
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of Biotechnology, Tianjin University of Science & Technology, Tianjin 300457, China
Received: March 24, 2020; Accepted: April 22, 2020
Supported by: National Natural Science Foundation of China (No. 31700080), China Postdoctoral Science Foundation (No. 2017M621082), Enterprise Postdoctoral Innovation Project of Tianjin (No. TJQYBSH2017006)
Corresponding author: Qinhong Wang. Tel: +86-22-84861950; E-mail: wang_qh@tib.cas.cn.
Abstract: Recently, fast-growing Vibrio natriegens, as the great potential chassis, has shown a wide application in synthetic biology. Genome editing is an indispensable tool for genetic modification in synthetic biology. However, genome editing tools with high efficiency and fidelity are still to be developed for V. natriegens synthetic biology. To deal with this problem, the physiological characteristics of 6 V. natriegens strains were evaluated, and CICC 10908 strain with fast and stable growth was selected as the host strain for genome editing study. Then, the natural transformation system of V. natriegens was established and optimized. The efficiencies of optimized natural transformation that integrates antibiotic resistance marker cat-sacB or KanR onto the chromosome of V. natriegens could reach 4×10–5 and 4×10–4, respectively. Based on the optimized natural transformation, a double-selection cassette was used to achieve seamless genome editing with high efficiency and fidelity. The positive rates of four different types of genetic manipulation, including gene deletion, complementation, insertion and substitution, were 93.8%, 100%, 95.7% and 100%, respectively. Finally, transformation and elimination of the recombinant plasmid could be easily achieved in V. natriegens. This work provides a seamless genome editing system with high efficiency and fidelity for V. natriegens synthetic biology.
Keywords: Vibrio natriegenschassisnatural transformationgenome editingsynthetic biology
需钠弧菌Vibrio natriegens是一种具有快速生长特性的革兰氏阴性海洋细菌,其在对数期的倍增时间小于10 min[1-2]。需钠弧菌还具有底物利用多样性、代谢速率快、对人体没有致病性危害、易于遗传操作、易于表达外源蛋白等优点,是近几年发展起来的一种应用于生物技术和合成生物学领域的新型底盘细胞[1, 3-7]。基于需钠弧菌优良的生物学特性,多种天然产物和蛋白质在需钠弧菌中实现了高效生物合成[4, 6-8]。
合成生物学底盘细胞需要具备简便、精准、高效的基因组编辑手段,从而实现靶基因的敲除、插入、替换等遗传操作。虽然传统的基因组编辑方法如Cre/loxP、Flp/FRT等,可以实现基因组的反复多次编辑,但是每次编辑过后会在基因组上遗留loxP、FRT等标签,因此容易导致宿主基因组大片段的重组缺失[9-10]。CRISPR-Cas (Clustered regularly interspaced short palindromic repeats and CRISPR- associated protein)技术具有无痕编辑和效率高的优点,但是其脱靶效应容易导致sgRNA错误结合位点的DNA发生突变,并且有些位点可能缺乏合适的PAM (Protospacer adjacent motif)序列[11-13]。在大肠杆菌Escherichia coli中,利用双向选择性筛选方法如galK、tolC、rpsL、tetA-sacB、cat-sacB等,可以实现精准、高效的基因组无痕编辑[14-18]。这些工作可以为需钠弧菌基因组编辑研究提供借鉴。
据报道,某些弧菌Vibrio spp.在海洋浮游动物的几丁质外壳上附着生长或外界环境中存在几丁质时可以被诱导形成自然感受态[4, 19]。自然感受态细胞能够从外界环境中摄取DNA,通过同源重组的方式整合到自身染色体上[20]。弧菌自然感受态细胞的形成受转录因子TfoX的调控,而tfoX基因的表达受几丁质的诱导[19, 21]。因此,可以通过诱导细胞表达tfoX,使其成为自然感受态,然后摄取外源DNA,从而实现基因组编辑。利用该生物学特性,Dalia等[4]建立了需钠弧菌多基因编辑体系。该系统虽然具有较高的基因组编辑效率,但是需要重组片段的同源臂长达3 kb,不利于重组片段的简单、快速制备,并且会在基因组上留下抗性标记。
本研究将在已报道的需钠弧菌自然转化系统基础上,利用双向选择性筛选标记基因cat-sacB,在需钠弧菌中实现精准、高效的基因组无痕编辑,为其合成生物学研究提供强有力的基因组编辑手段。
1 材料与方法1.1 菌株与质粒需钠弧菌CICC 10908 (同ATCC 14048菌株)和23820菌株购自中国工业微生物菌种保藏管理中心(CICC),CGMCC 1.1594 (同CCUG 16372菌株)、1.5849、1.5850、1.8668菌株购自中国普通微生物菌种保藏管理中心(CGMCC)。大肠杆菌DH5α、ATCC 8739和MG 1655菌株均为本实验室保存菌株。pACYC184[22]、pTrc99A[23]和pTarget[24]均为本实验室保存质粒。
1.2 培养基LB (Luria-Bertani)培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 10。LBv2培养基:LB培养基中添加v2盐(204 mmol/L NaCl,4.2 mmol/L KCl,23.14 mmol/L MgCl2)[3]。LB3培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 30[25]。LBS培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 10,蔗糖100。上述培养基制备固体培养基时添加15 g/L琼脂粉。IOM培养基(Instant ocean medium):28 g/L Instant ocean sea salt (Aquarium Systems Inc.,货号SS15-10)[4]。
1.3 需钠弧菌培养条件需钠弧菌传代培养以及感受态制备用LB3培养基,培养温度30 ℃或37 ℃。传代培养时挑取单菌落于含有5 mL LB3培养基的试管中,30 ℃、250 r/min培养过夜。
需钠弧菌生长速率测试。液体培养:挑取单菌落于含有5 mL LBv2培养基的试管中,于30 ℃、250 r/min摇床中过夜培养作为种子液。接种适量种子液于含有15 mL LB或LBv2培养基的100 mL三角瓶中至初始OD600为0.005,37 ℃、250 r/min培养8 h。计算细胞生长对数期的倍增时间。倍增时间计算参考Eagon [2]报道的方法,计算公式为g=t/n,n=(log10y/x)/log102,其中g代表倍增时间,t代表检测时间间隔(min),n代表繁殖代数,y代表终止点OD600,x代表起始点OD600。固体培养:取种子液分别在LB或LBv2固体培养基上进行平板划线,37 ℃静置培养14 h。大肠杆菌种子培养基为LB,其他同需钠弧菌。
需钠弧菌培养基中氯霉素(Cm)使用浓度为12.5 μg/mL (质粒筛选)或5 μg/mL (基因组整合筛选)[3],卡那霉素(Kan)使用浓度为75 μg/mL[25],壮观霉素(Spec)使用浓度为200 μg/mL[4]。
1.4 需钠弧菌电转化需钠弧菌电转化参考Lee等[25]报道的方法,略有改动。取100 μL种子液接种于50 mL LB3液体培养基中,37 ℃、250 r/min培养至OD600约为0.4。将菌液转移至预冷的50 mL离心管中,冰浴5 min。然后于4 ℃、2 000×g离心5 min,弃上清,菌体沉淀用1 mL预冷的1 mol/L山梨醇缓冲液温柔地重悬,4 ℃、5 000×g离心5 min。菌体沉淀用1 mL预冷的1 mol/L山梨醇缓冲液洗2次(共3次)。菌体沉淀用250 μL预冷的1 mol/L山梨醇缓冲液温柔地重悬,每管50 μL分装,若不立即使用,于–80 ℃冰箱保存。向感受态细胞中加入约100 ng质粒,轻轻混匀,转移至预冷的1 mm电转杯中,静置3 min后进行转化,转化电压0.7–0.9 kV (Bio-Rad,MicroPulserTM)。电转结束后立即加入1 mL预冷的LB3培养基,将菌液转移至2 mL离心管中,37 ℃、250 r/min复苏培养45 min或30 ℃、250 r/min复苏培养1.5 h。将菌液涂布于相应抗性的LB3固体培养基上,30 ℃培养过夜。
1.5 需钠弧菌自然转化载体的构建自然转化载体的构建流程如图 1所示。以需钠弧菌基因组DNA为模板,利用高保真酶PrimeSTAR HS DNA polymerase (TaKaRa,货号R010A)扩增tfoX (基因编号BA890_05980)[3]片段,用NcoⅠ和Hind Ⅲ进行双酶切,将其连接到pTrc99A载体上的NcoⅠ和Hind Ⅲ位点,形成pTrc-tfoX载体。以pTrc-tfoX为模板,扩增lacI-tfoX片段,用NdeⅠ和XhoⅠ进行双酶切,将其连接到pTarget载体上NdeⅠ和XhoⅠ位点,最终形成自然转化载体pMB1-tfoX。将不含有tfoX的pMB1空载体作为阴性对照。
 |
| 图 1 需钠弧菌自然转化载体pMB1-tfoX的构建流程 Fig. 1 The schematic diagram of constructing plasmid pMB1-tfoX for V. natriegens natural transformation. |
| 图选项 |
1.6 需钠弧菌自然转化自然转化参考Dalia等[4]报道的方法,略有改动。挑取含有pMB1-tfoX质粒的需钠弧菌单菌落接种于3 mL LB3+200 μg/mL Spec+100 μmol/L IPTG (Isopropyl-β-D-thiogalactopyranoside)液体培养基中,30 ℃、250 r/min培养过夜,诱导细胞形成自然感受态。向1.5 mL离心管中依次加入3.5 μL自然感受态细胞、350 μL的IOM培养基、100 μmol/L IPTG、100–400 ng转化DNA (tDNA),轻轻混匀。30 ℃静置培养4–6 h后,加入1 mL LB3培养基,于30 ℃、250 r/min摇床中复苏培养1.5 h。将菌液涂布于相应抗性的LB3固体培养基上,30 ℃培养过夜。挑取单菌落进行菌落PCR验证。需钠弧菌自然转化效率计算:具有Cm或Kan抗性的转化子数目与不具有Cm或Kan抗性的活细胞总数的比值。
1.7 需钠弧菌基因组无痕编辑在自然转化的基础上,参考大肠杆菌的双向选择性筛选的基因组编辑方法[18],建立精准、高效的需钠弧菌基因组无痕编辑体系。操作流程如图 2所示。
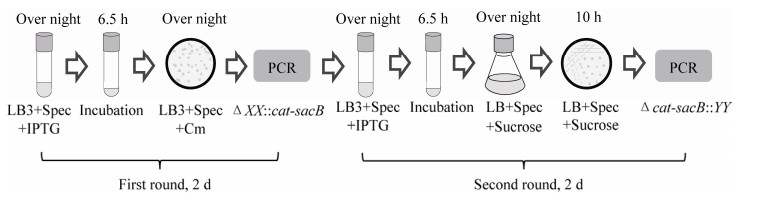 |
| 图 2 需钠弧菌基因组无痕编辑操作流程 Fig. 2 The schematic diagram of seamless genome editing for V. natriegens. |
| 图选项 |
第一轮同源重组:将cat-sacB片段通过插入或替换的方式整合到基因组上的特定位点,挑取在LB3+200 μg/mL Spec+5 μg/mL Cm培养基上正常生长的菌落进行PCR验证,获得cat-sacB正确整合菌株。
第二轮同源重组:sacB编码分泌型果聚糖蔗糖酶,该酶催化蔗糖生成果聚糖,果聚糖在细胞周质大量积累会对细胞产生毒性,造成细胞死亡[17, 26]。第一轮同源重组所得菌株带有反向筛选标记基因sacB,对蔗糖敏感。在反向筛选压力存在条件下,用含有目的序列的同源重组片段替换cat-sacB片段,从而达到基因敲除、插入或替换的目的。与第一轮同源重组不同的是,将复苏培养后的菌液转接到含有LBS+200 μg/mL Spec液体培养基中,30 ℃、250 r/min培养过夜。然后在LBS+200 μg/mL Spec固体培养基上平板划线,30 ℃培养。挑取菌落生长正常、颜色比较透明或较浅的菌落进行PCR和测序验证,最终获得基因组无痕编辑菌株。
1.8 需钠弧菌质粒消除在LB3+200 μg/mL Spec固体培养基上挑取需钠弧菌单菌落于3 mL LB3液体培养基中,37 ℃、250 r/min培养10 h,作为传代培养1次。若传代培养2次,则吸取30 μL传代培养1次的菌液于3 mL LB3液体培养基中,37 ℃、250 r/min再次培养10 h。将传代培养的菌液在LB3固体培养基上进行划线,37 ℃培养8–10 h。挑取单菌落分别在LB3和LB3+200 μg/mL Spec固体培养基上进行划线培养。计算在LB3+200 μg/mL Spec培养基上不能生长的菌落数目与LB3培养基上生长的菌落总数的比值,即为质粒消除效率。
2 结果与分析2.1 需钠弧菌宿主细胞的筛选需钠弧菌具有生长快速的特性,在最适条件下的倍增时间小于10 min[1-2]。为了筛选合适的出发菌株,对6株需钠弧菌的生长表型进行检测。在LBv2固体培养基上,虽然这6株需钠弧菌的生长速率基本相同,但是1.5849、1.5850、1.8668这3个菌株的菌落生长容易发生扩散(图 3A),不利于遗传操作时挑取单克隆。对这6株需钠弧菌的生长曲线进行检测,发现这6株需钠弧菌在LBv2液体培养基中的生长速率基本一致(图 3B)。但是,23820菌株在LB液体培养基中生物量显著低于其他菌株(结果未展示),1.1594菌株基因组中包含潜在的毒力因子基因zot (Zonaoccludens toxin)[3]。综合以上结果,选取需钠弧菌CICC 10908菌株作为后续研究的宿主细胞。
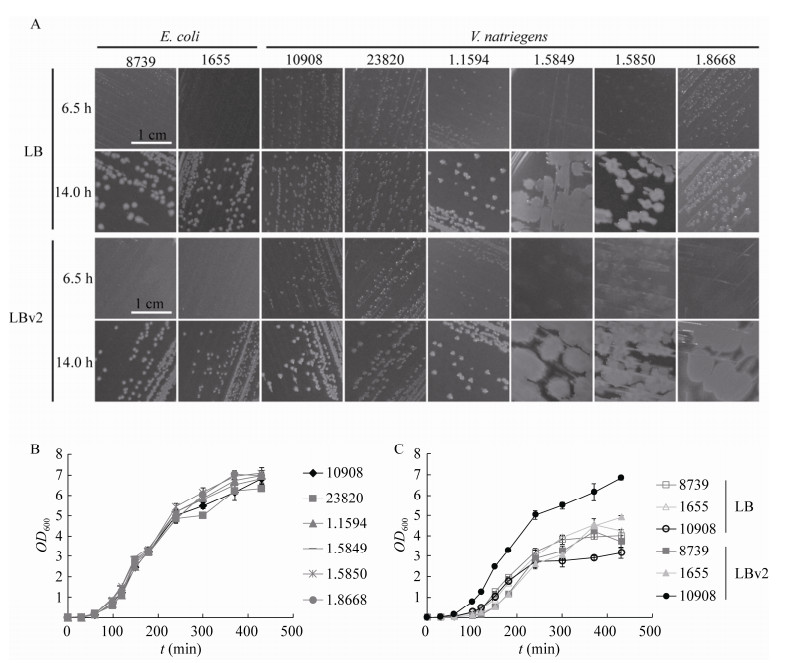 |
| 图 3 不同需钠弧菌菌株的生长表型比较(A:大肠杆菌和需钠弧菌在LB和LBv2固体培养基上的生长表型;B:不同需钠弧菌菌株在LBv2液体培养基中的生长曲线;C:需钠弧菌和大肠杆菌在LB和LBv2液体培养基中的生长曲线.每组实验均设置3个生物学重复) Fig. 3 The comparation of cell growth for different V. natriegens strains. (A) The growth phenotypes of different V. natriegens and E. coli strains on solid LB and LBv2 media. (B) The cell growth curves of different V. natriegens strains in liquid LBv2 medium. (C) The cell growth curves of V. natriegens and E. coli in liquid LB and LBv2 media. All data were derived from three independent biological replicates. |
| 图选项 |
将CICC 10908与代谢工程研究中常用的两株大肠杆菌ATCC 8739和MG1655进行生长速率比较,需钠弧菌在固体和液体培养基中的生长速率均明显快于大肠杆菌(图 3A和3C)。通过计算,需钠弧菌CICC 10908在LBv2液体培养基中的倍增时间为13.89 min,而ATCC 8739和MG1655在LB液体培养基中倍增时间分别为19.41 min和21.08 min,表明需钠弧菌具有比大肠杆菌更快的生长速率。
2.2 需钠弧菌自然转化条件的优化基因组编辑是合成生物学研究过程中必不可少的遗传操作手段。为了实现需钠弧菌基因组编辑,首先尝试将改造后的大肠杆菌CRISPR-cas9系统引入需钠弧菌,但未能成功,可能是因为大肠杆菌同源重组系统相关酶在需钠弧菌中没有重组活性[24, 27]。利用需钠弧菌可以被诱导形成自然感受态的生物学特性,通过IPTG诱导型启动子Ptrc在细胞内诱导表达tfoX,促使需钠弧菌细胞成为自然感受态细胞[4]。
为了测试需钠弧菌的自然转化效率,用两种筛选标记基因cat-sacB和KanR分别替换需钠弧菌基因组上的dns (BA890_12415,编码Deoxyribonuclease I)[3]。在IPTG诱导条件下,含有pMB1-tfoX质粒的需钠弧菌对cat-sacB和KanR片段的转化效率分别达4×10–5和4×10–4,均明显高于IPTG未诱导和空载对照pMB1菌株(图 4A和4B)。当同源重组片段的两条同源臂长度分别为600 bp时,由于cat-sacB重组片段的长度为4 144 bp,明显长于KanR片段的长度2 327 bp,因此转化同样质量的tDNA,cat-sacB片段的转化效率要低于KanR (图 4A和4B)。通过统计分析,cat-sacB和KanR片段的同源重组正确率分别达到97.1%和100% (表 1)。部分cat-sacB和KanR同源重组转化子的琼脂糖凝胶电泳检测结果如图 4E所示,结果显示均正确。
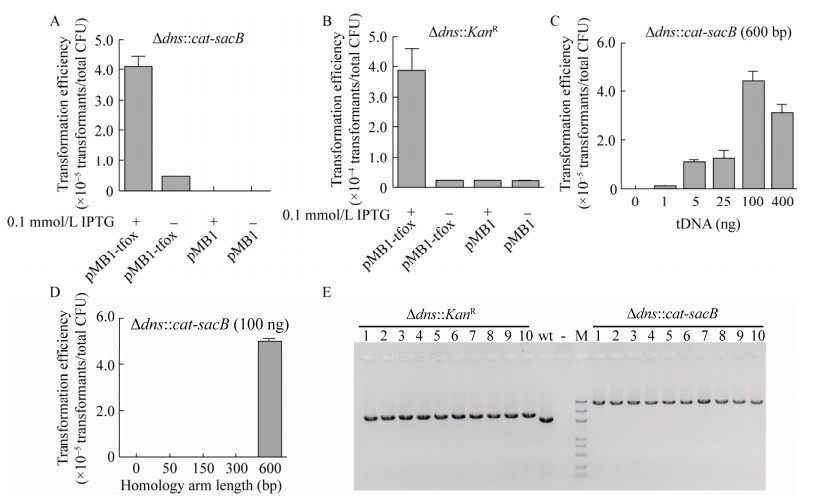 |
| 图 4 需钠弧菌自然转化条件的优化(A和B:自然转化载体pMB1-tofX基因组编辑效率分析,转化含有pMB1和pMB1-tfoX质粒的需钠弧菌所用cat -sacB和Kan R重组片段同源臂长度600 bp,用量100 ng;C:不同转化DNA (tDNA)用量对基因组编辑效率的影响,cat -sacB片段同源臂长度600 bp;D:不同同源臂长度对转化效率的影响,cat -sacB片段用量100 ng.图中所有数值为3个生物学重复的平均值±方差(x±s). E:cat -sacB和Kan R替换dns琼脂糖凝胶电泳检测,wt为野生型,-为阴性对照,DNA marker条带由小到大依次为100、250、500、750、1 000、2 000、3 000、5 000 bp) Fig. 4 The optimization of natural transformation for V. natriegens. (A) and (B) Analysis of natural transformation efficiency for pMB1-tofX. V. natriegens strains containing a pMB1 empty vector or pMB1-tfoX were transformed with 100 ng of a Δdns::cat-sacB or Δdns::KanR tDNA containing 600 bp homology arms. (C) Natural transformation assay of V. natriegens with indicated concentration of Δdns::cat-sacB tDNA containing 600 bp homology arms. (D) Natural transformation assay of V. natriegens with 100 ng of Δdns::cat-sacB tDNA containing indicated lengths of homology arms. All data were derived from three independent biological replicates. (E) Agarose gel electrophoresis analysis of genome editing for Δdns::KanR and Δdns::cat-sacB. -: negative; wt: wild type. DNA marker (M) bands are 100, 250, 500, 750, 1 000, 2 000, 3 000 and 5 000 bp respectively. |
| 图选项 |
表 1 不同类型基因组编辑效率统计Table 1 The efficiency of different types of genome editing
| Genome editing types | Homology arm length (bp/bp) | Positive transformants | Total transformants | Genome editing efficiency (%) |
| Δdns::cat-sacB | 50/50 | 0 | 12 | 0.0 |
| Δdns::cat-sacB | 150/150 | 0 | 20 | 0.0 |
| Δdns::cat-sacB | 300/300 | 10 | 11 | 90.9 |
| Δdns::cat-sacB | 600/600 | 66 | 68 | 97.1 |
| Δdns::KanR | 600/600 | 45 | 45 | 100.0 |
| Δdns | 300/300 | 18 | 18 | 100.0 |
| Δdns | 600/600 | 30 | 32 | 93.8 |
| Δdns::dns | 600/600 | 21 | 21 | 100.0 |
| P29-xylE | 631/638 | 45 | 47 | 95.7 |
| ΔptsI::P29-galP | 629/476 | 55 | 55 | 100.0 |
表选项
以cat-sacB替换dns为例,对同源重组片段的用量进行优化。当重组片段为100 ng时,转化效率最高达4×10–5,用量过高或过低均会导致转化效率降低(图 4C)。当同源臂较长时,会增加同源重组片段的制备难度;较短时,又会导致同源重组效率降低。通过对同源臂的最短有效长度进行测试,当同源臂长度为600 bp时,即可获得较高的转化效率和同源重组效率;当同源臂长度为300 bp时,虽然可以获得同源重组正确的转化子,但转化效率很低;同源臂长度≤150 bp时,很难获得同源重组正确的转化子(图 4D,表 1)。这些结果表明利用自然转化系统可以实现需钠弧菌的高效转化和同源重组,为后续研究提供强有力的基因组编辑手段。
2.3 需钠弧菌基因组无痕编辑效率的测试需钠弧菌自然转化虽然可以高效地实现同源重组,但是需要目的片段与筛选标记基因一起整合至基因组中,无法实现基因组无痕编辑,因此需要建立一种基因组无痕编辑系统。利用双向选择性筛选方法,如tolC、rpsL、tetA-sacB、cat-sacB等,大肠杆菌可以实现精准、高效的基因组无痕编辑[15-18]。因此,本研究采用cat-sacB作为筛选标记,第一轮同源重组将基因组上的XX序列替换为cat-sacB片段,第二轮同源重组将cat-sacB片段替换为目标序列YY,从而实现基因组无痕敲除、插入或替换等遗传操作(图 2)。
利用cat-sacB作为筛选标记基因,对需钠弧菌dns敲除和原位回补效率进行测试。通过两轮筛选,利用600 bp长度的同源臂,dns敲除和原位回补的编辑效率分别达到93.8%和100%。即使将同源臂缩短到300 bp,dns敲除效率仍然达到100% (表 1)。部分dns敲除和原位回补菌株的琼脂糖凝胶电泳检测结果显示DNA条带大小符合编辑后的序列长度,测序结果显示编辑位点同源重组完全正确(图 5A和5B)。
 |
| 图 5 需钠弧菌基因组编辑的琼脂糖凝胶电泳检测(A:dns敲除检测;B:dns敲除后原位回补检测;C:P29启动子插入到xylE位点检测;D:P29-galP替换ptsI检测. -为阴性对照,wt为野生型,cs为cat -sacB,DNA marker条带由小到大依次为100、250、500、750、1 000、1 500 (仅图A)、2 000、3 000、5 000 bp) Fig. 5 Agarose gel electrophoresis analysis of genome editing for V. natriegens. (A) dns delition analysis. (B) dns in situ complementation analysis. (C) Analysis of P29 insertion at xylE site. (D) Analysis of ptsI substitution with P29-galP. -: negative; wt: wild type; cs: cat-sacB. DNA marker (M) bands are 100, 250, 500, 750, 1 000, 1 500 (only in A), 2 000, 3 000 and 5 000 bp, respectively. |
| 图选项 |
利用该基因组无痕编辑系统,选取xylE (BA890_08590,编码D-木糖质子同向转运体)和ptsI (BA890_03825,编码磷酸烯醇丙酮酸蛋白磷酸转移酶)作为靶位点[3],分别在xylE起始密码子ATG前插入P29启动子序列和用P29-galP序列(大肠杆菌ATCC8739 galP,编码半乳糖透性酶)替换ptsI,测试基因插入和替换的效率。通过两轮筛选,P29启动子插入xylE位点的插入效率达到95.7%,而P29-galP替换ptsI的替换效率达到100% (表 1)。部分基因插入和替换菌株的琼脂糖凝胶电泳检测结果显示条带大小符合编辑后的序列长度,测序结果显示编辑位点同源重组完全正确(图 5C和5D)。基因组无痕编辑体系的建立为需钠弧菌合成生物学研究提供强有力的基因组编辑手段。
2.4 需钠弧菌质粒的转化和消除效率分析需钠弧菌作为合成生物学的底盘细胞,还需要具备高效的质粒转化方法。因为不同宿主来源质粒DNA的碱基修饰方式(如甲基化)不同,所以利用来源于宿主本身的质粒进行转化可能具有更高的转化效率[3, 28]。分别将来源于需钠弧菌和大肠杆菌的pACYC184质粒通过电转化和自然转化两种方式转化需钠弧菌。经过统计分析,电转化的转化效率略高于自然转化,并且需钠弧菌本身来源的质粒转化效率明显高于大肠杆菌来源质粒(图 6A)。因此,后续研究如果将需钠弧菌作为基因克隆的宿主细胞,采用其本身来源的质粒进行载体构建等遗传操作可能有利于获得更高的转化效率。
 |
| 图 6 需钠弧菌质粒的转化和消除效率检测(A:pACYC184质粒转化效率分析,Elec为电转化,GENT为自然转化,E. coli和V. natrigens表示质粒的宿主来源,图中数值为3个生物学重复的平均值±方差(x±s),*:P < 0.05;B:pMB1-tfoX质粒消除效率分析) Fig. 6 Analysis of plasmid transformation and elimination efficiencies for V. natriegens. (A) Analysis of pACYC184 transformation efficiency. Elec: electroporation; GENT: natural transformation. E. coli and V. natrigens indicated the sources of pACYC184. All data and standard errors were derived from three independent biological replicates. *: P < 0.05. (B) Analysis of pMB1-tofX elimination efficiency. |
| 图选项 |
质粒作为宿主染色体外的独立存在的遗传物质,当宿主细胞不需要其发挥功能的时候,需要能够方便地从宿主细胞中消除。对携带pMB1-tfoX质粒的需钠弧菌进行无选择压力传代培养。当传代培养一次时,细胞内质粒的消除效率达12.5% (6/48);当传代培养两次时,质粒的消除效率达20.8% (10/48) (图 6B)。这些结果表明需钠弧菌作为宿主细胞可以实现质粒的高效转化和消除,为其合成生物学研究提供有效的遗传操作手段。
3 讨论需钠弧菌具有生长快速、代谢速率快、对人体没有致病性危害、易于遗传操作等许多优点,是近几年发展起来的一种新型底盘细胞,在合成生物学研究领域展现出良好的应用前景[1, 3-4, 6]。但是,需钠弧菌合成生物学相关的研究基础还十分薄弱,有待进一步完善。
生长表型结果表明,不同需钠弧菌菌株虽然均具有生长快速的特性,但是不同菌株之间的生理表型具有一定的差异。因此,选择合适的需钠弧菌初始菌株有利于后续研究工作的顺利开展。
本研究首先尝试通过电转化法,利用构建的CRISPR-Cas9系统,对需钠弧菌dns基因进行敲除,但未能获得阳性转化子。Lee等[25]用阿拉伯糖诱导型启动子ParaBAD带动dCas9的表达,用组成型启动子J23100带动sgRNA的表达,成功实现了对基因组上gfp基因沉默,表明Cas9在需钠弧菌中可以发挥功能。该研究组利用霍乱弧菌V. cholerae的SXT-Beta和SXT-Exo编码基因,连同λ-Gam编码基因,重构需钠弧菌同源重组系统,成功实现了单链和双链DNA的同源重组[27]。这些研究结果表明,本研究CRISPR-Cas9系统没有构建成功,可能是因为大肠杆菌同源重组系统相关酶在需钠弧菌中没有重组活性。
利用需钠弧菌自然转化系统,以cat-sacB或KanR作为筛选标记,当同源臂为600 bp时,转化效率分别达到4×10–5或4×10–4。Dalia等[4]建立的自然转化系统,以KanR作为筛选标记,当同源臂为500 bp时,转化效率约10–4。本研究与文献报道自然转化系统的转化效率基本一致,均能实现靶基因在需钠弧菌基因组上的高效整合。在优化的自然转化系统基础上,利用双向选择性筛选标记基因cat-sacB,建立了需钠弧菌基因组无痕编辑体系。该系统只需大约600 bp长度的同源臂,即可精准、高效地实现基因无痕敲除、插入或替换等遗传操作,并且不会在基因组上遗留任何筛选标记或标签序列。
不同复制子类型的质粒在需钠弧菌细胞中的遗传稳定性不同,其中p15A和部分ColE1复制子类型的质粒在需钠弧菌细胞中具有良好的稳定性。在无抗生素的培养基中传代培养9 h,需钠弧菌细胞中这两种类型质粒保持率均在90%以上[5]。本研究对携带pMB1-tfoX质粒(ColE1 ori)的需钠弧菌进行无选择压力传代培养一次时,质粒消除效率达12.5% (即质粒保持率为87.5%),与文献报道基本一致。
本研究在对需钠弧菌自然转化方法进行优化的基础上,精准、高效地实现了基因的无痕敲除、插入或替换等遗传操作,为其合成生物学研究提供可靠的基因组无痕编辑手段。
参考文献
| [1] | Hoffart E, Grenz S, Lange J, et al. High substrate uptake rates empower Vibrio natriegens as production host for industrial biotechnology. Appl Environ Microbiol, 2017, 83(22): e01614-17. |
| [2] | Eagon RG. Pseudomonas natriegens, a marine bacterium with a generation time of less than 10 minutes. J Bacteriol, 1962, 83: 736-737. DOI:10.1128/JB.83.4.736-737.1962 |
| [3] | Weinstock MT, Hesek ED, Wilson CM, et al. Vibrio natriegens as a fast-growing host for molecular biology. Nat Methods, 2016, 13(10): 849-851. DOI:10.1038/nmeth.3970 |
| [4] | Dalia TN, Hayes CA, Stolyar S, et al. Multiplex genome editing by natural transformation (MuGENT) for synthetic biology in Vibrio natriegens. ACS Synth Biol, 2017, 6(9): 1650-1655. DOI:10.1021/acssynbio.7b00116 |
| [5] | Tschirhart T, Shukla V, Kelly EE, et al. Synthetic biology tools for the fast-growing marine bacterium Vibrio natriegens. ACS Synth Biol, 2019, 8(9): 2069-2079. DOI:10.1021/acssynbio.9b00176 |
| [6] | Ellis GA, Tschirhart T, Spangler J, et al. Exploiting the feedstock flexibility of the emergent synthetic biology chassis Vibrio natriegens for engineered natural product production. Mar Drugs, 2019, 17(12): 679. DOI:10.3390/md17120679 |
| [7] | Schleicher L, Muras V, Claussen B, et al. Vibrio natriegens as host for expression of multisubunit membrane protein complexes. Front Microbiol, 2018, 9: 2537. |
| [8] | Becker W, Wimberger F, Zangger K. Vibrio natriegens: an alternative expression system for the high-yield production of isotopically labeled proteins. Biochemistry, 2019, 58(25): 2799-2803. DOI:10.1021/acs.biochem.9b00403 |
| [9] | Yu BJ, Sung BH, Koob MD, et al. Minimization of the Escherichia coli genome using a Tn5-targeted Cre/loxP excision system. Nat Biotechnol, 2002, 20(10): 1018-1023. DOI:10.1038/nbt740 |
| [10] | Yuan LZ, Rouvière PE, Larossa RA, et al. Chromosomal promoter replacement of the isoprenoid pathway for enhancing carotenoid production in E. coli. Metab Eng, 2006, 8(1): 79-90. |
| [11] | McCarty NS, Graham AE, Studená L, et al. Multiplexed CRISPR technologies for gene editing and transcriptional regulation. Nat Commun, 2020, 11: 1281. DOI:10.1038/s41467-020-15053-x |
| [12] | Schultenk?mper K, Brito LF, Wendisch VF. Impact of CRISPR interference on strain development in biotechnology. Biotechnol Appl Biochem, 2020, 67(1): 7-21. |
| [13] | Yan MY, Yan HQ, Ren GX, et al. CRISPR-Cas12a-assisted recombineering in bacteria. Appl Environ Microbiol, 2017, 83(17): e00947-17. |
| [14] | Warming S, Costantino N, Court DL, et al. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res, 2005, 33(4): e36. DOI:10.1093/nar/gni035 |
| [15] | Gregg CJ, Lajoie MJ, Napolitano MG, et al. Rational optimization of tolC as a powerful dual selectable marker for genome engineering. Nucleic Acids Res, 2014, 42(7): 4779-4790. DOI:10.1093/nar/gkt1374 |
| [16] | Stavropoulos TA, Strathdee CA. Synergy between tetA and rpsL provides high-stringency positive and negative selection in bacterial artificial chromosome vectors. Genomics, 2001, 72(1): 99-104. DOI:10.1006/geno.2000.6481 |
| [17] | Li XT, Thomason LC, Sawitzke JA, et al. Positive and negative selection using the tetA-sacB cassette: recombineering and P1 transduction in Escherichia coli. Nucleic Acids Res, 2013, 41(22): e204. DOI:10.1093/nar/gkt1075 |
| [18] | Liu YL, Yang MH, Chen JJ, et al. PCR-based seamless genome editing with high efficiency and fidelity in Escherichia coli. PLoS ONE, 2016, 11(3): e0149762. DOI:10.1371/journal.pone.0149762 |
| [19] | Meibom KL, Blokesch M, Dolganov NA, et al. Chitin induces natural competence in Vibrio cholerae. Science, 2005, 310(5755): 1824-1827. DOI:10.1126/science.1120096 |
| [20] | Dalia AB, McDonough E, Camilli A. Multiplex genome editing by natural transformation. Proc Natl Acad Sci USA, 2014, 111(24): 8937-8942. DOI:10.1073/pnas.1406478111 |
| [21] | Yamamoto S, Morita M, Izumiya H, et al. Chitin disaccharide (GlcNAc)2 induces natural competence in Vibrio cholerae through transcriptional and translational activation of a positive regulatory gene tfoXVC. Gene, 2010, 457(1/2): 42-49. |
| [22] | Rose RE. The nucleotide sequence of pACYC184. Nucleic Acids Res, 1988, 16(1): 355. DOI:10.1093/nar/16.1.355 |
| [23] | Amann E, Ochs B, Abel KJ. Tightly regulated tac promoter vectors useful for the expression of unfused and fused proteins in Escherichia coli. Gene, 1988, 69(2): 301-315. DOI:10.1016/0378-1119(88)90440-4 |
| [24] | Jiang Y, Chen B, Duan CL, et al. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system. Appl Environ Microbiol, 2015, 81(7): 2506-2514. DOI:10.1128/AEM.04023-14 |
| [25] | Lee HH, Ostrov N, Wong BG, et al. Functional genomics of the rapidly replicating bacterium Vibrio natriegens by CRISPRi. Nat Microbiol, 2019, 4(7): 1105-1113. DOI:10.1038/s41564-019-0423-8 |
| [26] | Gay P, Le Coq D, Steinmetz M, et al. Cloning structural gene sacB, which codes for exoenzyme levansucrase of Bacillus subtilis: expression of the gene in Escherichia coli. J Bacteriol, 1983, 153(3): 1424-1431. DOI:10.1128/JB.153.3.1424-1431.1983 |
| [27] | Lee HH, Ostrov N, Gold MA, et al. Recombineering in Vibrio natriegens. bioRxiv, 2017, doi: https://doi.org/10.1101/130088. |
| [28] | Guss AM, Olson DG, Caiazza NC, et al. Dcm methylation is detrimental to plasmid transformation in Clostridium thermocellum. Biotechnol Biofuels, 2012, 5(1): 30. DOI:10.1186/1754-6834-5-30 |
