

广东药科大学 生命科学与生物制药学院 广东省生物技术候选药物研究重点实验室,广东 广州 510006
收稿日期:2019-02-27;接收日期:2019-05-20;网络出版时间:2019-06-28
基金项目:广东省科技计划项目(No. 2015A01017014)资助,国家自然科学基金(No. 31501895)
摘要:基因工程中,无痕修饰是一种很受欢迎的基因组操作技术。反选择系统的严谨性决定了无痕修饰的效率。近期有文献报道了一种新型的反选择系统kil,该系统与质粒pSim6联用后反选择严谨性较pKD46质粒更高,预示着kil与pSim6质粒联用可以更高效地选择出重组子。据此,对大肠杆菌菌株W3110、MG1655和DH10B中的4个不同的非必需基因位点(lacI、dbpa、ack和glk)进行了分析,将不同长度的外源基因片段对预先插入这些位点的tet/kil进行基因替换。结果表明kil与pSim6质粒联用较其与pKD46质粒联用,重组效率均有显著提高,并且随着外源片段的增长,效果更明显。外源片段为1 000 bp时,kil与pSim6质粒联用是其与pKD46联用的1.2–2倍;外源片段为2 000 bp时,则是pKD46的2.2–5倍。综上,kil与pSim6联用具有更高的重组效率,更有利于大肠杆菌基因组的无痕修饰操作,这为大肠杆菌重组工程提供了更多的选择。
关键词:Red重组系统选择反选择系统无痕修饰大肠杆菌
Combination of novel counter-selection system kil and pSim6 plasmid in recombination engineering
Yujuan Li, Ruyi Chen, Mianfeng Yang, Wei Chen


Guangdong Province Key Laboratory for Biotechnology Drug Candidates, School of Biosciences and Biopharmaceutics, Guangdong Pharmaceutical University, Guangzhou 510006, Guangdong, China
Received: February 27, 2019; Accepted: May 20, 2019; Published: June 28, 2019
Supported by: Science and Technology Program of Guangdong Province (No. 2015A010107014), National Natural Science Foundation of China (No. 31501895)
Corresponding author: Wei Chen. Tel: +86-20-39352201; E-mail:thinker98@163.com.
Abstract: Seamless modification is a popular genomic manipulation technique in genetic engineering. Selection stringency of the counter-selection system determines the efficiency of the seamless modification. Recently, a novel counter-selection system, kil, was constructed. It is reported that the selection selectivity of kil is higher in host bacteria harboring plasmid pSim6 than that harboring pKD46, indicating that recombinants could be selected out more efficiently by combining kil counter-selection system and plasmid pSim6. In order to confirm this speculation, four different loci (lacI, dbpa, ack, glk) in Escherichia coli strains W3110, MG1655 and DH10B were selected for testing: dsDNA fragments of different sizes (500 bp, 1 000 bp, and 2 000 bp) were used to substitute tet/kil. As expected, recombination efficiency was higher in host bacteria harboring plasmid pSim6 than that harboring pKD46, and the results were more obvious with the length of dsDNA increasing. Specifically, recombination efficiency was 1.2 to 2 fold higher in pSim6 harboring bacteria than in pKD46 harboring bacteria when dsDNA fragments were 1 000 bp in length. With the length of dsDNA increasing up to 2 000 bp, the gap increased to 2.2–5 fold. In conclusion, it is easier to perform seamless modification by combining kil counter-selection system and plasmid pSim6 than combining kil and pKD46. An alternative tool in genomic engineering is provided in this study.
Keywords: Red recombination systemselection/counter-selection systemseamless modificationEscherichia coli
同源重组是在基因工程中常用的技术手段,并且在基因打靶和基因克隆方面有着极其重要的作用。运用同源重组技术,可以对巨大、复杂的染色体进行修饰[1]。在代谢工程与功能基因组分析中,对大肠杆菌基因组修饰(点突变、插入、删除、替换)的同源重组,一般用35–50 bp的同源臂,就可以发生有效的重组并且不需要限制性酶切位点。这种重组是由噬菌体重组酶(λ-Red系统)介导的,λ-Red系统是目前研究相对比较丰富的系统,它存在于λ-噬菌体左向操纵子上,其表达受pL启动子和CI857阻遏蛋白控制,由exo、bet和gam这3个基因组成,表达蛋白产物分别为Exo、Beta和Gam[2]。Exo蛋白具有核酸外切酶活性,能以5′–3′方向降解双链DNA末端,从而留下3′单链突出端DNA分子[3-4];Beta蛋白能结合由Exo消化产生的3′单链末端,防止DNA被单链核酸酶消化,同时促进互补单链DNA区域间的退火,其在Red同源重组过程中起着决定性的作用[5];Gam蛋白抑制核酸RecBCD和SbcCD对线性双链DNA分子的降解,协助Exo和Beta共同完成同源重组过程。运用Red重组技术进行染色体修饰主要有3种操作策略:DNA双链介导的重组[6]、单链重组[7]和重叠引物介导的重组[8]。
在Red同源重组操作策略中,两步选择反选择重组法[9]是报道最多且应用最广泛的策略,改造后不会产生多余的基因序列变化,可以达到“无痕修饰”的目的。其策略分为两步:1)通过电击转化将带有同源臂的选择反选择片段转入大肠杆菌感受态细胞中,在Red重组系统的表达下发生同源重组,通过正向选择标记(一般为抗性基因如neo)筛选阳性克隆;2)同样用电转化将带有同源臂的外源片段转入细胞中,在Red重组系统的作用下进行重组,完成基因敲除、替换或插入,通过反向选择标记基因(如sacB、tolC、thyA、ccdB和galk)进行筛选[10-15]。
一般来说,反向选择比正向选择效率低,并且都有很高的背景[16]或者特殊的要求,如宿主基因型需要在使用阴性选择标记基因之前修饰[17],或者依靠最低限度的培养基来实现更好的选择严谨性或需要特殊的选择性条件。2019年,本实验室构建并发表了一个新型的kil反选择标记基因[18]。λ噬菌体的kil基因由温度敏感的cI857阻遏物操控下的pL启动子控制[19],其表达可有效地导致宿主细胞存活力的丧失,因此将其作为反选择标记基因。研究中发现新构建的反选择基因比其他各种负选择基因具有显著的优势:高选择严谨性、低背景、不需要特殊的试剂。我们在之前的研究中,在大肠杆菌MG1655的lacI、ack、dbpa非必需位点分别测试了含有pSim6质粒或含有pKD46质粒与kil反选择系统联用的选择严谨性。发现pKD46质粒与kil联用后选择严谨性为7.6×10–7– 9.3×10–7,pSim6质粒与kil联用后选择严谨性较低至2.3×10–6–5.1×10–6,较pKD46高了2–5倍。因此,我们推断,pSim6质粒与kil反选择基因联用后,双链DNA片段的重组效率会比pKD46质粒与kil反选择基因联用时有所提高。
因此本研究采用含有Red同源系统的pSim6质粒,与新型的kil选择反选择系统联合使用,在不同大肠杆菌菌株(W3110、MG1655和DH10B)中的4个不同位点(lacI、dbpa、ack和glk)进行不同片段长度(500 bp、1 000 bp和2 000 bp)的替换,分析其重组效率,期望可以建立一个高选择效率的重组工程系统,为更好地将重组工程无痕修饰提供了更多的选择。
1 材料与方法1.1 材料菌株和质粒:大肠杆菌菌株Escherichia coli W3110、MG1655和DH10B为均为本室保存,GYpk1-tet (含有tet/kil选择反选择系统)为本室构建,pSim6质粒为美国国立卫生研究院Court DL博士惠赠。
工具酶和试剂:PrimerSTAR?GXL DNA高保真酶购自日本TaKaRa生物公司,PCR mix酶购自民赛生物(广州)科技有限公司;Tet (四环素)、Amp (氨苄霉素)购自生工生物工程(上海)股份有限公司;质粒提取试剂盒和PCR产物回收试剂盒购自Axygen公司;PCR引物为生工生物工程(上海)股份有限公司合成。
PCR扩增及鉴定引物:带同源臂引物序列见表 1,带有下划线的代表同源臂序列,不带下划线的代表PCR扩增引物序列;鉴定引物序列见表 2。
表 1 本研究所用带同源臂引物Table 1 Primers with homology arms used in this study
| Primer name | Primer sequence (5′–3′) |
| lacI-tkF | CCTAATGAGTGAGCTAACTCACATTAATTGCGTTGCGCTCCTAATTTTTGTTGACACTC |
| lacI-tkR | GATAGCGCCCGGAAGAGAGTCAATTCAGGGTGGTGAATTTAGTGAATGCTTTTGCTTG |
| lacI-500F | GATAGCGCCCGGAAGAGAGTCAATTCAGGGTGGTGAATAGCTACCAGATACCATCGTC |
| lacI-500R | CCTAATGAGTGAGCTAACTCACATTAATTGCGTTGCGCGAGTAGCTTGATCAACTCTG |
| lacI-1000R | CCTAATGAGTGAGCTAACTCACATTAATTGCGTTGCGCTAAGAGCGCATGATACGGAC |
| lacI-2000R | CCTAATGAGTGAGCTAACTCACATTAATTGCGTTGCGCGCTGCTGCATAGGTTGCTAC |
| ack-tkF | TTACAAAACAGCACCGCCAGCTGAGCTGGCGGTGTGAAATCCTAATTTTTGTTGACACTC |
| ack-tkR | TGACGTTTTTTTAGCCACGTATCAATTATAGGTACTTCCTTAGTGAATGCTTTTGCTTG |
| ack-500F | TGACGTTTTTTTAGCCACGTATCAATTATAGGTACTTCCAGCTACCAGATACCATCGTC |
| ack-500R | TTACAAAACAGCACCGCCAGCTGAGCTGGCGGTGTGAAAGAGTAGCTTGATCAACTCTG |
| ack-1000R | TTACAAAACAGCACCGCCAGCTGAGCTGGCGGTGTGAAATAAGAGCGCATGATACGGAC |
| ack-2000R | TTACAAAACAGCACCGCCAGCTGAGCTGGCGGTGTGAAAGCTGCTGCATAGGTTGCTAC |
| dbpa-tkF | TCTCATAAAATTGTTGTTCAATGGGTGGCAAAGCATCTGTCCTAATTTTTGTTGACACT |
| dbpa-tkR | CGCCTGCTAACTTTGCGTCGATGACCACGAGAATAGATTTTAGTGAATGCTTTTGCTTG |
| dbpa-500F | CGCCTGCTAACTTTGCGTCGATGACCACGAGAATAGATTAGCTACCAGATACCATCGTC |
| dbpa-500R | TCTCATAAAATTGTTGTTCAATGGGTGGCAAAGCATCTGGAGTAGCTTGATCAACTCTG |
| dbpa-1000R | TCTCATAAAATTGTTGTTCAATGGGTGGCAAAGCATCTGTAAGAGCGCATGATACGGAC |
| dbpa-2000R | TCTCATAAAATTGTTGTTCAATGGGTGGCAAAGCATCTGGCTGCTGCATAGGTTGCTAC |
| glk-tkF | AACAGCGACAATGCGCGGCGGCAATCGGTGCAGCTGTCATCCTAATTTTTGTTGACACT |
| glk-tkR | CAGGGCTTGATTACCCCAGCCTCGAAGCGGTCATTCGCGTTAGTGAATGCTTTTGCTTG |
| glk-500F | CAGGGCTTGATTACCCCAGCCTCGAAGCGGTCATTCGCGAGCTACCAGATACCATCGTC |
| glk-500R | AACAGCGACAATGCGCGGCGGCAATCGGTGCAGCTGTCAGAGTAGCTTGATCAACTCTG |
| glk-1000R | AACAGCGACAATGCGCGGCGGCAATCGGTGCAGCTGTCATAAGAGCGCATGATACGGAC |
| glk-2000R | AACAGCGACAATGCGCGGCGGCAATCGGTGCAGCTGTCAGCTGCTGCATAGGTTGCTAC |
| The single underlined sequences indicate homology arms. | |
表选项
表 2 本研究所用鉴定引物Table 2 Identification primers used in this study
| Primer name | Primer sequence (5′–3′) |
| lacIF | GCATCTTCCGGCGCTACAAC |
| lacIR | CGCTCACAATTCCACACAAC |
| ackF | TGCAGTGCATGATGTTAATC |
| ackR | GGGATCAGCATAATAATACG |
| dbpaF | CACCACCCGATGAAGAACC |
| dbpaR | GAGGATGGCTGATGCAAGC |
| glkF | GATATTTACAGGGAGCCTG |
| glkR | GATTGTCATGGACGATGAG |
| catjdF | TGGAGGAAGGCATTGAGATC |
| TetA-R | TCCTAATTTTTGTTGACACTCTA |
表选项
1.2 方法1.2.1 甘油/甘露醇梯度[20]感受态的制备与电击转化将过夜培养的大肠杆菌W3110等3个菌株按1:100 (500 μL)稀释到50 mL SOB培养基中,30 ℃、180 r/min振荡培养至OD600在0.4–0.6之间。然后冰浴30 min (含有pSim6质粒的菌株在42 ℃诱导15 min后冰浴30 min)。将上述培养液转移至高压灭菌处理的50 mL聚丙烯离心管中,4 ℃、5 500 r/min、7 min离心收集菌体,弃上清,加1 mL预冷的ddH2O重悬,再加15 mL ddH2O混匀,加15 mL甘油/甘露醇溶液伸到管底,缓慢加入,形成分层,4 500 r/min、6 min、4 ℃离心;重复3次后加1 mL甘油/甘露醇溶液重悬于1.5 mL的EP管,6 500 r/min、4 min、4 ℃离心;后弃上清,加甘油/甘露醇溶液使总体积约为200 μL。取50 μL感受态与300 ng的打靶分子DNA片段进行电转(条件为电压1.8 kV,电容25 μF,电阻200 ?),立即用2 mL SOC培养基复苏2 h。
1.2.2 线性双链DNA介导的DNA重组同源臂的设计遵循GC含量分布均匀且含量约为50%的原则,长度在35–50 bp最佳,本实验扩增含同源臂的基因片段由同源臂长度为38 bp以及21 bp的扩增引物组成。使用高保真酶扩增,扩增条件是:98 ℃ 5 min;98 ℃ 10 s,55 ℃ 30 s,68 ℃ 1 kb/min (31个循环);68 ℃ 10 min,4 ℃保温。扩增的线性双链DNA分子经纯化回收后与感受态混合,进行电击转化。用含筛选标记(Amp/Tet)的LB平板或者其他条件(42 ℃)的LB平板筛选发生基因重组后的阳性克隆。
1.2.3 Tet/kil选择反选择方法① 通过tet抗性基因进行正向选择,PCR合成打靶分子(两侧带有同源臂的tet/kil片段),取约300 ng的回收产物与50 μL的感受态细菌混合,电击转化后复苏2 h,涂于含Amp (25 μg/mL)和Tet (10 μg/mL)的LB平板上,30 ℃培养过夜后筛选阳性克隆。②利用kil基因在42 ℃下的致死效应进行第二步反选择筛选,使用catenin基因为模板,通过高保真酶扩增两侧带有同源臂的线性化DNA分子,扩增不同长度的外源片段(500 bp,1 000 bp和2 000 bp)。试剂盒纯化后取300 ng的回收产物与①中鉴定正确的50 μL阳性克隆感受态混合,电击转化后培养2 h,涂于无抗板(200 μL菌液混合物)在42 ℃过夜培养筛选。
1.2.4 重组克隆的鉴定① tet/kil选择反选择系统的鉴定:用各个位点的扩增上游引物(lacIF、ackF、dbpaF和glkF)与tet/kil片段中的下游引物(TetA-R)进行PCR鉴定。②外源片段的鉴定:用外源基因片段中上游引物(catjdF)与各个位点的扩增下游引物(lacIR、ackR、dbpaR和glkR)进行PCR鉴定。用mix聚合酶鉴定,扩增条件是:94 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 kb/min (31个循环);72 ℃ 10 min,4 ℃保温。
1.2.5 重组效率的计算从42 ℃无抗板各挑取23个单克隆与一个阴性克隆(未转外源片段的42 ℃平板)进行PCR鉴定,通过琼脂糖凝胶电泳分析,出现正确条带的数目与总数的比值则为重组效率,重复3次取平均值。
2 结果与分析2.1 ack位点的pSim6与kil联用ack基因(GenBank登录号: BAA16135.1)是大肠杆菌基因组上的非必需基因,其表达乙酸激酶,它的缺失可使乙酸减少,增加谷氨酸的的产量[21]。因此我们在此位点用不同长度的外源片段进行替换。图 1A为重组后tet/kil鉴定图,然后从菌落表型(图 1B、1C)上可以观察到:pSim6与kil联用后与pKD46与kil联用后相比较,空转菌落数量差不多,转cat500的外源片段,pSim6菌落数量相对较多;转cat1k的外源片段,pSim6菌落数量与pKD46差异较为明显;转cat2k的外源片段,pSim6菌落数量与pKD46差异更为明显。因此我们对pSim6质粒与kil联用的重组克隆进行PCR鉴定,每次挑选23个,重复3次,图 1D为琼脂糖凝胶电泳分析。图 1E为从图 1D得到的数据进行重组效率分析图,图中可以观察到500 bp片段很容易被替换,重组率高达90%以上,长度增加至1 000 bp,重组率在71%–88%之间,外源片段为2 000 bp时,同源重组的重组率约为50%–94%。pKD46与kil联用后的重组效率[18]分别为82.61% (500 bp)、47.83% (1 000 bp)和13.04% (2 000 bp)。结果表明pSim6与kil联用后比pKD46与kil联用后的重组效率有明显的提高。
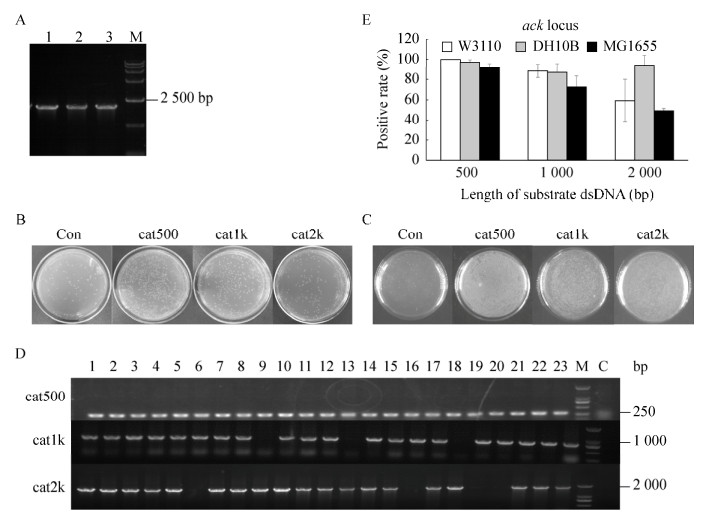 |
| 图 1 ack位点的重组效率分析 Fig. 1 Recombination efficiency analysis of the ack locus. (A) Identification of recombinants harboring tet/kil by PCR. 1–3: PCR identification of recombinants; M: DNA marker DL 15 000. (B) tet/kil substitution with dsDNA in different length (500 bp, 1 000 bp, or 2 000 bp) using plasmid pKD46. Three independent experiments were performed, and a representative replicate is shown. (C) tet/kil substitution with dsDNA in different length (500 bp, 1 000 bp or 2 000 bp) using plasmid pSim6. Three independent experiments were performed, and a representative replicate is shown. (D) PCR detection of the tet/kil substitution using plasmid pSim6. A total of 23 colonies (lane 1 to lane 23) were screened and a 234 bp (cat500), 746 bp (cat1k), or 1 733 bp (cat2k) product indicates successful substitution by donor dsDNA in the ack locus. M: DNA marker DL 2 000; C: control. (E) Positive rates of tet/kil substitution using plasmid pSim6. Results of three independent experiments mentioned above were analyzed and positive rates were calculated. |
| 图选项 |
2.2 lacI位点的pSim6与kil联用通过在ack位点进行不同片段同源重组后,pSim6质粒与kil两者联用,与pKD46质粒与kil联用相比较,重组效率相对提高。对此,我们在其他非必需位点对pSim6质粒和kil反选择系统联用进行多基因位点验证,观察是否有同样的重组效率。挑选了另外3个大肠杆菌非必需位点lacI[22] (GenBank登录号: BAE76127.1)、dbpa[23] (GenBank登录号: BAA14946.1)、glk[24] (GenBank登录号: BAA16258.1)进行分析。首先对lacI基因位点进行分析,结果如图 2所示,用引物lacIF和tetA-R (tet末端序列)鉴定长度为1 931 bp,证明成功得到阳性重组菌(图 2A)。从图 2B中可以看到500 bp片段很容易被替换,重组率为85%–100%。长度增加至1 000 bp,重组率在60%–80%,外源片段为2 000 bp时,同源重组的重组率约为50%–60%。
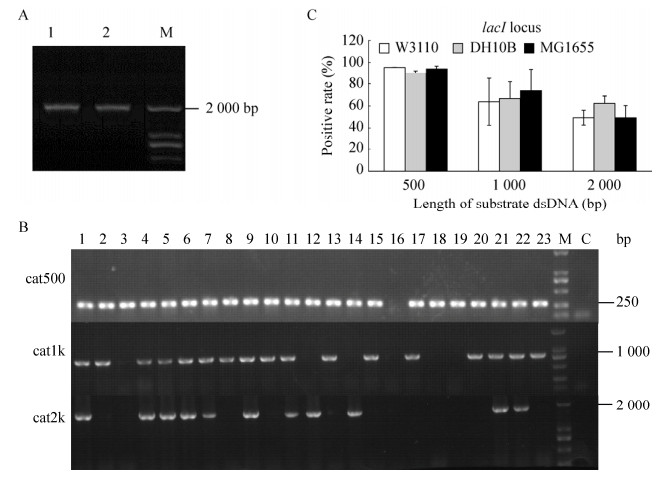 |
| 图 2 lacI位点的重组效率分析 Fig. 2 Recombination efficiency analysis of lacI locus. (A) Identification of recombinants harboring tet/kil by PCR. 1 and 2: PCR identification of recombinants; M: DNA marker DL 2 000. (B) PCR detection of the tet/kil substitution using plasmid pSim6. A total of 23 colonies (lane 1 to lane 23) were screened and a 268 bp (cat500), 780 bp (cat1k), or 1 767 bp (cat2k) product indicates successful substitution by donor dsDNA in the lacI locus. M: DNA marker DL 2 000; C: control. (C) Positive rates of tet/kil substitution using plasmid pSim6. Results of three independent experiments mentioned above were analyzed and positive rates were calculated. |
| 图选项 |
2.3 dbpa位点的pSim6与kil联用接下来对dbpa位点进行分析,用引物dbpaF和tetA-R (tet末端序列)鉴定长度为1 833 bp,证明成功得到阳性重组菌(图 3A)。同样挑选23个单克隆与1个未转外源片段单克隆作对照,用引物catjdF (外源片段上的基因序列)与dbpaR扩增,琼脂糖凝胶电泳并拍照观察正确条带。重复3次取平均值,结果如图 3C所示。从图 3B观察到500 bp片段很容易被替换,重组率高达97%以上,长度增加至1 000 bp,重组率在82%–95%之间,外源片段为2 000 bp时,同源重组的重组率约为75%–82%。
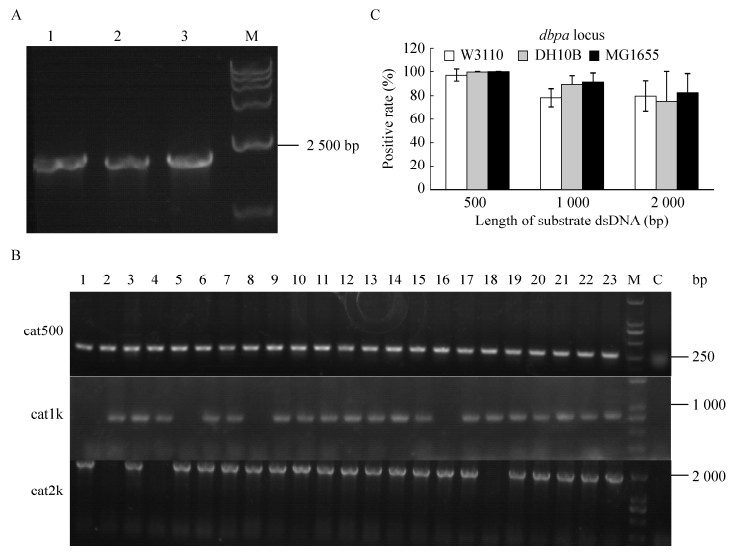 |
| 图 3 dbpa 位点的重组效率分析 Fig. 3 Recombination efficiency analysis of dbpa locus. (A) Identification of recombinants harboring tet/kil by PCR. 1–3: PCR identification of recombinants; M: DNA marker DL 15 000. (B) PCR detection of the tet/kil substitution using plasmid pSim6. A total of 23 colonies (lane 1 to lane 23) were screened and a 234 bp (cat500), 746 bp (cat1k), or 1 733 bp (cat2k) product indicates successful substitution by donor dsDNA in the dbpa locus. M: DNA marker DL 2 000; C: control. (C) Positive rates of tet/kil substitution using plasmid pSim6. Results of three independent experiments mentioned above were analyzed and positive rates were calculated. |
| 图选项 |
2.4 glk位点的pSim6与kil联用最后选择了在非必需基因glk位点进行分析,用引物glkF和tetA-R (tet末端序列)鉴定长度为1 969 bp,证明成功得到阳性重组菌(图 4A)。同样挑选23个单克隆与1个未转外源片段单克隆作对照,用引物catjdF (外源片段上的基因序列)与glkR扩增,琼脂糖凝胶电泳并拍照观察正确条带。重复3次取平均值,结果如图 4B所示。从图 4C观察到500 bp片段很容易被替换,重组率高达85%以上,长度增加至1 000 bp,重组率在63%–100%之间,外源片段为2 000 bp时,同源重组的重组率约为42%–84%。
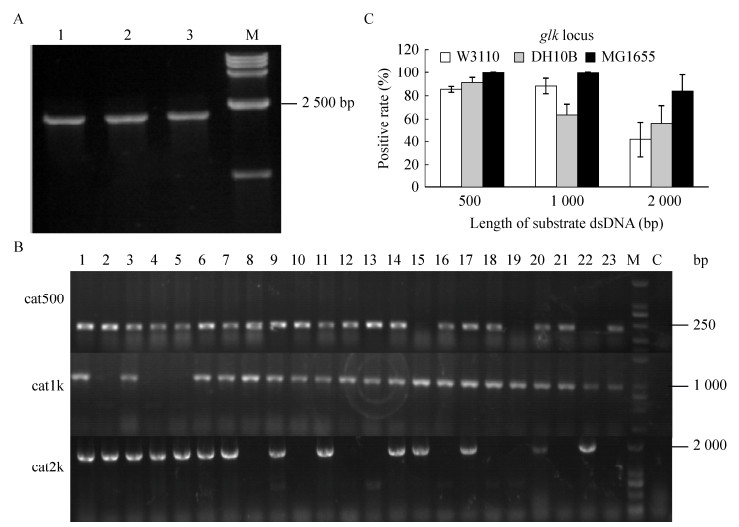 |
| 图 4 glk位点的重组效率分析 Fig. 4 Recombination efficiency analysis of glk locus. (A) Identification of recombinants harboring tet/kil by PCR. 1–3: PCR identification of recombinants; M: DNA marker DL 15 000. (B) PCR detection of the tet/kil substitution using plasmid pSim6. A total of 23 colonies (lane 1 to lane 23) were screened and a 373 bp (cat500), 885 bp (cat1k), or 1 872 bp (cat2k) product indicates successful substitution by donor dsDNA in the glk locus. M: DNA marker DL 2 000; C: control. (C) Positive rates of tet/kil substitution using plasmid pSim6. Results of three independent experiments mentioned above were analyzed and positive rates were calculated. |
| 图选项 |
3 讨论重组工程是一种应用广泛的体内基因工程方法,它使DNA修饰变得简单有效,并对其修饰时不需要限制性内切酶。Datta等[25]构建了一系列的重组质粒,pSim6是里面选择强度较高的质粒。它是从pSim5质粒衍生出来的,来源于严紧型复制质粒pSC101,16个低拷贝数,在质粒中保留了Exo下游的天然tL3转录终止子,这可以防止Exo的过度转录产物进入ori区域。质粒中的Red重组系统在温度敏感(YS)抑制因子CI857控制pL操纵子表达,在30–34 ℃下抑制因子有活性从而阻止pL操纵子表达,遏止Red重组酶的转录。温度短暂升至42 ℃,阻遏物瞬时变性,启动Red系统的表达;再回到低温时,阻遏基因重新复制,结合pL操纵子,重新阻遏Red系统。在多次实验中发现诱导42 ℃、15 min为高效表达Red重组酶的最佳条件[26]。相对比之前所用的pKD46质粒[19],不需要诱导物(L-阿拉伯糖)的诱导,且选择强度高,简捷方便。
为了在基因组工程中为反选基因提供一种替代方法,在本研究中我们使用了一种新型的反选择标记基因kil[27]。kil基因在低温条件下(如30 ℃),pL被cI857抑制,从而抑制kil基因表达。当温度升高至到42 ℃时,因为失活的cI857阻遏蛋白不能阻遏pL启动子,启动了kil基因的转录和表达,最终造成了宿主菌的死亡。在之前的研究[19]中已经对kil基因的选择严谨性在3个大肠杆菌菌株的3个位点进行了分析,发现其选择严谨性相对较高(几乎等同于tet-sacB选择反选择系统[28])。因此我们将pSim6质粒与kil基因联合使用,在3个不同的大肠杆菌菌株W3110、MG1655和DH10B的4个非必需基因位点lacI、ack、dbpa和glk进行重组效率分析。两者联用后,与研究中kil重组效率相比较,从而得出以下结果:外源片段500 bp重组效率两者联用与之前kil均很高,在80%以上;当外源片段增加至1 000 bp时,联用后的重组效率约是之前kil基因的1.2–2倍;当外源片段增加至2 000 bp时,联用后的重组效率约是之前kil基因的2.2–5倍。
两者联用后重组效率增长的原因是未知的。可能来自pSim6的多个cI拷贝在未诱导的情况下赋予对kil基因更高水平的调控,因此在42 ℃条件下kil蛋白可以高效表达进而有效地杀死宿主菌株。这种效率上的差异可能归因于kil基因的更高的选择严谨性。
随着基因工程发展越来越快,基因编辑的方法日益增加。从而,本研究用含有高效的Red重组系统的pSim6质粒通过与新颖的kil反选择系统这一技术融合,降低了反选择标记的高背景,同时提高了对基因无痕修饰重组效率。虽然诱导性毒素系统[10]目前是反选择系统中最好的一种,在一些条件下,kil的选择严谨性比不上诱导型毒素系统的选择严谨性,但是在该技术中不需要延长的孵育、特殊的试剂以及选择性条件,kil基因除了有杀菌作用之外,还有抑菌的作用。这种思路在某些情况下还是有一定的优势,向研究者提供了更多的方向和方法。为代谢工程和功能基因组的分析研究缩短了时间,提高了效率,使大肠杆菌的染色体改造更加便捷,从而推动了微生物科学的前进与发展。
参考文献
| [1] | Sharan SK, Thomason LC, Kuznetsov SG, et al. Recombineering: a homologous recombination-based method of genetic engineering. Nat Protoc, 2009, 4(2): 206-223. DOI:10.1038/nprot.2008.227 |
| [2] | Jeong J, Cho N, Jung D, et al. Genome-scale genetic engineering in Escherichia coli. Biotechnol Adv, 2013, 31(6): 804-810. DOI:10.1016/j.biotechadv.2013.04.003 |
| [3] | Egan M, Ramirez J, Xander C, et al. Lambda Red-mediated recombineering in the attaching and effacing pathogen Escherichia albertii. Biol Proced Online, 2016, 18: 3. DOI:10.1186/s12575-015-0032-8 |
| [4] | Poteete AR. What makes the bacteriophage λ Red system useful for genetic engineering: molecular mechanism and biological function. FEMS Microbiol Lett, 2001, 201(1): 9-14. |
| [5] | Yu DG, Sawitzke JA, Ellis H, et al. Recombineering with overlapping single-stranded DNA oligonucleotides: testing a recombination intermediate. Proc Natl Acad Sci USA, 2003, 100(12): 7207-7212. DOI:10.1073/pnas.1232375100 |
| [6] | Yu DG, Ellis HM, Lee EC, et al. An efficient recombination system for chromosome engineering in Escherichia coli. Proc Natl Acad Sci USA, 2000, 97(11): 5978-5983. DOI:10.1073/pnas.100127597 |
| [7] | Ellis HM, Yu DG, DiTizio T, et al. High efficiency mutagenesis, repair, and engineering of chromosomal DNA using single-stranded oligonucleotides. Proc Natl Acad Sci USA, 2001, 98(12): 6742-6746. DOI:10.1073/pnas.121164898 |
| [8] | Li XT, Costantino N, Lu LY, et al. Identification of factors influencing strand bias in oligonucleotide- mediated recombination in Escherichia coli. Nucleic Acids Res, 2003, 31(22): 6674-6687. DOI:10.1093/nar/gkg844 |
| [9] | Lalioti MD, Heath JK. A new method for generating point mutations in bacterial artificial chromosomes by homologous recombination in Escherichia coli. Nucleic Acids Res, 2001, 29(3): e14. DOI:10.1093/nar/29.3.e14 |
| [10] | Khetrapal V, Mehershahi K, Rafee S, et al. A set of powerful negative selection systems for unmodified enterobacteriaceae. Nucleic Acids Res, 2015, 43(13): e83. DOI:10.1093/nar/gkv248 |
| [11] | Madyagol M, Al-Alami H, Levarski Z, et al. Gene replacement techniques for Escherichia coli genome modification. Folia Microbiol (Praha), 2011, 56(3): 253-263. DOI:10.1007/s12223-011-0035-z |
| [12] | Wong QNY, Ng VCW, Lin MCM, et al. Efficient and seamless DNA recombineering using a thymidylate synthase A selection system in Escherichia coli. Nucleic Acids Res, 2005, 33(6): e59. DOI:10.1093/nar/gni059 |
| [13] | Gregg CJ, Lajoie MJ, Napolitano MG, et al. Rational optimization of tolC as a powerful dual selectable marker for genome engineering. Nucleic Acids Res, 2014, 42(7): 4779-4790. DOI:10.1093/nar/gkt1374 |
| [14] | DeVito JA. Recombineering with tolC as a selectable/counter-selectable marker: remodeling the rRNA operons of Escherichia coli. Nucleic Acids Res, 2008, 36(1): e4. |
| [15] | Stavropoulos TA, Strathdee CA. Synergy between tetA and rpsL Provides high-stringency positive and negative selection in bacterial artificial chromosome vectors. Genomics, 2001, 72(1): 99-104. DOI:10.1006/geno.2000.6481 |
| [16] | Muyrers JPP, Zhang YM, Benes V, et al. Point mutation of bacterial artificial chromosomes by ET recombination. EMBO Rep, 2000, 1(3): 239-243. DOI:10.1093/embo-reports/kvd049 |
| [17] | Wang HL, Bian XY, Xia LQ, et al. Improved seamless mutagenesis by recombineering using ccdB for counterselection. Nucleic Acids Res, 2014, 42(5): e37. DOI:10.1093/nar/gkt1339 |
| [18] | Chen W, Li YJ, Wu GJ, et al. Simple and efficient genome recombineering using kil counter-selection in Escherichia coli. J Biotechnol, 2019, 294: 58-66. DOI:10.1016/j.jbiotec.2019.01.024 |
| [19] | Datsenko KA, Wanner BL. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc Natl Acad Sci USA, 2000, 97(12): 6640-6645. DOI:10.1073/pnas.120163297 |
| [20] | Warren DJ. Preparation of highly efficient electrocompetent Escherichia coli using glycerol/mannitol density step centrifugation. Anal Biochem, 2011, 413(2): 206-207. |
| [21] | Pyne ME, Moo-Young M, Chung DA, et al. Coupling the CRISPR/Cas9 system with lambda Red recombineering enables simplified chromosomal gene replacement in Escherichia coli. Appl Environ Microb, 2015, 81(15): 5103-5114. DOI:10.1128/AEM.01248-15 |
| [22] | Malakar P, Singh VK, Karmakar R, et al. Effect on β-galactosidase synthesis and burden on growth of osmotic stress in Escherichia coli. Springerplus, 2014, 3: 748. DOI:10.1186/2193-1801-3-748 |
| [23] | Liu RT, Wang GR, Liu C, et al. Gene expression profile analysis of dbpA knockdown in colorectal cancer cells. Cell Biol Int, 2016, 40(12): 1280-1293. DOI:10.1002/cbin.10670 |
| [24] | Jiang Y, Chen B, Duan CL, et al. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system. Appl Environ Microbiol, 2015, 81(7): 2506-2514. DOI:10.1128/AEM.04023-14 |
| [25] | Datta S, Costantino N, Court DL. A set of recombineering plasmids for gram-negative bacteria. Gene, 2006, 379: 109-115. DOI:10.1016/j.gene.2006.04.018 |
| [26] | Chen W. The application of Red engineering on Escherichia coli[D]. Xiamen: Xiamen University, 2005 (in Chinese). 陈伟. Red重组工程技术在大肠杆菌中的应用[D].厦门: 厦门大学, 2005. |
| [27] | Haeusser DP, Hoashi M, Weaver A, et al. The kil peptide of bacteriophage lambda blocks Escherichia coli cytokinesis via ZipA-dependent inhibition of FtsZ assembly. PLoS Genet, 2014, 10(3): e1004217. DOI:10.1371/journal.pgen.1004217 |
| [28] | Li XT, Thomason LC, Sawitzke JA, et al. Positive and negative selection using the tetA-sacB cassette: recombineering and P1 transduction in Escherichia coli. Nucleic Acids Res, 2013, 41(22): e204. DOI:10.1093/nar/gkt1075 |
