

1. 江南大学 粮食发酵工艺与技术国家工程实验室,江苏 无锡 214122;
2. 江南大学 生物工程学院,江苏 无锡 214122
收稿日期:2018-12-19;接收日期:2019-02-28;网络出版时间:2019-03-19
基金项目:国家自然科学基金(No. 31670095)资助
摘要:柚皮素是一种天然黄酮类化合物,具有抗炎、抗氧化、抗病毒、预防动脉粥样硬化等多种药理活性,也是其他黄酮类化合物合成的重要前体,具有重要的应用价值。目前,微生物法生产柚皮素等黄酮类化合物由于代谢通路不平衡等原因导致产量较低,在很大程度上限制了其工业应用。文中以一株产柚皮素的酿酒酵母菌株Y-01为研究对象,利用启动子和拷贝数控制柚皮素合成代谢途径关键酶4-香豆酸:CoA连接酶(4CL)、查尔酮合成酶(CHS)和查尔酮异构酶(CHI)编码基因的表达水平,考察这些基因的表达水平对目标产物积累水平的量化影响。结果表明,柚皮素产量与4CL或CHI编码基因的表达量之间关联性较低,而与chs基因的表达量存在显著的正相关性。通过调控chs基因的表达水平,获得一株高产柚皮素的酿酒酵母工程菌株Y-04,产量较出发菌株Y-01提高了4.1倍。研究结果表明,CHS是柚皮素合成过程的关键调控靶点,合理调控CHS表达可以显著促进酿酒酵母积累柚皮素。相关结果为采用代谢工程强化微生物合成柚皮素等重要黄酮类化合物提供了重要的理论参考。
关键词:柚皮素表达量关键基因启动子拷贝数
Quantitative effect of the expression level of key genes in naringenin synthesis on the accumulation level of target products
Tingting Jiao1,2, Jingwen Zhou1,2, Sha Xu1,2


1. National Engineering Laboratory for Cereal Fermentation Technology, Jiangnan University, Wuxi 214122, Jiangsu, China;
2. School of Biotechnology, Jiangnan University, Wuxi 214122, Jiangsu, China
Received: December 19, 2018; Accepted: February 28, 2019; Published: March 19, 2019
Supported by: National Natural Science Foundation of China (No. 31670095)
Corresponding author: Sha Xu. Tel/Fax: +86-510-85914371; E-mail: xus1984@jiangnan.edu.cn.
Abstract: Naringenin is a natural flavonoid compound with anti-inflammatory, anti-oxidation, anti-viral, anti-atherosclerosis and other pharmacological activities. It is also an important precursor of other flavonoid synthesis and with great value of application. At present, the production of flavonoids such as naringenin by microbial methods has a low yield due to imbalance of metabolic pathways, which greatly limits its industrial application. In this study, a naringenin-producing strain of Saccharomyces cerevisiae Y-01 was used in the research object. The expression levels of 4-coumaric acid: CoA ligase (4CL), chalcone synthase (CHS) and chalcone isomerase (CHI) were controlled by promoter and copy numbers to investigate the quantitative effect of key enzyme expression level on the accumulation level of target products. The results showed that the correlation between naringenin production and 4CL or CHI expression was not significant while there was a positive correlation with the expression level of CHS. Strain Y-04 with high yield of naringenin was obtained by regulating the expression level of chs gene, and the yield was increased by 4.1-folds compared with the original strain Y-01. This study indicated that CHS is a key regulatory target of naringenin synthesis. Rational regulation of CHS expression can significantly promote the accumulation of naringenin. The related results provide an important theoretical reference for the use of metabolic engineering to strengthen microbial synthesis of important flavonoids such as naringenin.
Keywords: naringeninexpression levelkey genepromotercopy number
柚皮素(4', 5, 7-三羟基二氢黄酮)是一种重要的二氢黄酮类化合物。柚皮素及其衍生物普遍存在于高等植物中,例如西红柿和柑橘类水果。药理研究表明,柚皮素及其衍生物具有抗炎、抗氧化、抗病毒、预防动脉粥样硬化等各种活性[1]。此外,柚皮素也是大多数黄酮类化合物的合成前体,可以用于合成具有更多生理活性和应用价值的重要黄酮类化合物。目前,国内外报道的柚皮素制备方法主要有植物提取法、化学合成法和微生物法。由于植物提取法存在植物生长周期长、含量低、代谢产物复杂等问题,导致成本较高,严重阻碍了柚皮素的研究和应用[2-3]。化学合成法涉及有毒化学品和极端反应条件,除成本较高外,还无法保证关键手性基团的天然等同性[4]。由于微生物法生产黄酮不受季节限制,具有完全的天然等同性等特点,是目前被认为最有前景的发展方向。
柚皮素的生物合成途径从芳香族氨基酸开始,主要由4种酶参与,即酪氨酸解氨酶(TAL) /苯丙氨酸解氨酶(PAL)、4-香豆酸:CoA连接酶(4CL)、查尔酮合成酶(CHS)和查尔酮异构酶(CHI)。其中,TAL将L-酪氨酸转化为对香豆酸;4CL催化1分子CoA和1分子对香豆酸形成对香豆酰辅酶A;CHS负责将3分子丙二酰辅酶A和1分子对香豆酰辅酶A转化为柚皮素查尔酮;CHI催化柚皮素查尔酮异构化为柚皮素。为了提高目标产物的产量,研究人员首先关注于筛选高活性酶和改善酶的性质并获得了一系列具有较高催化活性的酶[5-6]。大量的代谢工程实例表明,高效的黄酮生物合成途径还需要调节高活性酶的表达水平。目前,基于不同强度启动子和不同拷贝数的模块化优化策略已经用于平衡代谢流,并显著强化了生松素[7]和柚皮素[8]等黄酮类物质的积累。但是,该策略需要筛选具有梯度强度的启动子文库,并且在多基因途径中这些启动子的天文数目组合在实际操作中是一个巨大的挑战。因此,在构建平衡途径的启动子-关键基因组合之前,探究途径中目标物质产量与关键酶编码基因表达量的关系是必要的。为此,文中以一株可以积累柚皮素的酿酒酵母Y-01为研究对象,利用不同强度启动子和整合位点,梯度过量表达酶活较高香芹菜4CL、矮牵牛CHS和紫苜宿CHI[8],结合实时荧光定量PCR (RT-PCR)方法检测它们的表达水平,获得柚皮素产量与这3个关键酶表达量之间的关系。结果表明,CHS是黄酮合成过程的关键调控靶点,合理调控CHS表达有利于酵母中柚皮素的积累。研究结果对后续代谢工程强化微生物合成柚皮素等重要黄酮类化合物具有重要的参考价值。
1 材料与方法1.1 材料1.1.1 菌株大肠杆菌Escherichia coli JM109用于质粒构建和保存。酿酒酵母Saccharomyces cerevisiae Y-01用于整合片段及表达,酿酒酵母S288C用于基因扩增。文中使用的主要菌株如表 1所示。
表 1 本研究所用的主要菌株Table 1 Strains used in this study
| Name | Description | Source |
| Saccharomyces cerevisiae S288C | Navogen | |
| CEN.PK2-1D-Δubi4 | MATa ura3-52; trp1-289; leu2-3, 112; his3Δ 1; MAL28 SUC2 | Navogen |
| Y-01 | CEN.PK2-1D, gal80::Loxp-G418-Loxp, pY26-PGAL10-chi-PGAL1-chs-PGAL7-4cl | This study |
| Y-02 | Y-01, leu2::PGAL10-4cl-Loxp-HIS5-Loxp | This study |
| Y-03 | Y-01, leu2::PTEF-4cl-Loxp-HIS5-Loxp | This study |
| Y-04 | Y-01, leu2::PGAL7-chs-Loxp-HIS5-Loxp | This study |
| Y-05 | Y-01, leu2::PGAL10-chs-Loxp-HIS5-Loxp | This study |
| Y-06 | Y-01, leu2::PGAL10-chi-Loxp-HIS5-Loxp | This study |
| Y-07 | Y-01, leu2::PTEF-chi-Loxp-HIS5-Loxp | This study |
| Y-08 | Y-01, rdna::PTEF-4cl--Loxp-HIS5-Loxp | This study |
| Y-09 | Y-01, rdna::PGAL1-4cl--Loxp-HIS5-Loxp | This study |
| Y-10 | Y-01, rdna::PGAL7-4cl--Loxp-HIS5-Loxp | This study |
| Y-11 | Y-01, rdna::PGAL10-4cl--Loxp-HIS5-Loxp | This study |
| Y-12 | Y-01, rdna::PGAL2-4cl--Loxp-HIS5-Loxp | This study |
| Y-13 | Y-01, rdna::PPGI1-4cl--Loxp-HIS5-Loxp | This study |
| Y-14 | Y-01, rdna::PTEF-chs-Loxp-HIS5-Loxp | This study |
| Y-15 | Y-01, rdna::PGAL1-chs-Loxp-HIS5-Loxp | This study |
| Y-16 | Y-01, rdna::PGAL7-chs-Loxp-HIS5-Loxp | This study |
| Y-17 | Y-01, rdna::PGAL10-chs-Loxp-HIS5-Loxp | This study |
| Y-18 | Y-01, rdna::PGAL2-chs-Loxp-HIS5-Loxp | This study |
| Y-19 | Y-01, rdna::PPGI1-chs-Loxp-HIS5-Loxp | This study |
| Y-20 | Y-01, rdna::PTEF-chi-Loxp-HIS5-Loxp | This study |
| Y-21 | Y-01, rdna::PGAL1-chi-Loxp-HIS5-Loxp | This study |
| Y-22 | Y-01, rdna::PGAL7-chi-Loxp-HIS5-Loxp | This study |
| Y-23 | Y-01, rdna::PGAL10-chi-Loxp-HIS5-Loxp | This study |
| Y-24 | Y-01, rdna::PGAL2-chi-Loxp-HIS5-Loxp | This study |
| Y-25 | Y-01, rdna::PPGI1-chi-Loxp-HIS5-Loxp | This study |
| 4cl from Petroselinum crispum, chs from Petunia X hybrida, chi from Medicago sativa. | ||
表选项
1.1.2 培养基LB (Luria Broth)液体培养基(/L):10 g NaCl,10 g蛋白胨,5 g酵母提取物,pH 7.0。
YNB (Yeast Nitrogen Base)培养基(/L):200 g/L葡萄糖母液100 mL,67.4 g/L YNB (Yeast nitrogen base without amino acids)母液100 mL,并添加所需的缺陷氨基酸(Trp、His、Leu)各10 mL,终浓度均为50 mg/L,加蒸馏水至1 L,过滤除菌,4 ℃冰箱保存。
YPSe培养基(/L):20 g蛋白胨,10 g酵母提取物,10 g蔗糖,10 mL无水乙醇。
1.1.3 主要试剂各种抗生素和氨基酸,包括氨苄青霉素、遗传霉素、色氨酸、组氨酸、亮氨酸,均购自生工生物工程(上海)股份有限公司。柚皮素标样购自Sigma-Aldrich公司。蛋白胨、酵母提取物购自Oxoid公司。乙腈(质谱纯)购自Merck公司。其他本实验中使用的各种分析化学试剂如无特别说明均购自上海国药集团。各种一步克隆酶、连接酶、感受态制备试剂盒、pMD19-T Simple Vector购自大连宝生物(TaKaRa)有限公司。细菌质粒小量提取试剂盒购自于生工生物工程(上海)股份有限公司。胶回收试剂盒购自Fermentas公司。引物合成以及DNA测序服务均由生工生物工程(上海)股份有限公司提供。
1.1.4 摇瓶培养条件从平板上挑取改造后产柚皮素酿酒酵母的单菌落,在含有20 mL YNB培养基(添加Trp和Leu)的250 mL摇瓶中于30 ℃、220 r/min培养18 h。然后以10% (V/V)的接种量转接到20 mL YPSe发酵培养基中,同时加入10 g/L CaCO3及终浓度500 mg/L对香豆酸,于30 ℃、220 r/min培养84 h。
1.2 方法1.2.1 酿酒酵母基因组整合框构建为了使代谢途径基因协同表达,首先确定代谢途径关键基因过量表达对产物柚皮素含量的影响。选择6个不同强度的启动子(PGAL1、PTEF、PGAL7、PGAL10、PGAL2、PPGI1)分别表达4cl、chs、chi基因,并选取了基因组上LEU2和核糖体DNA (rDNA)作为整合位点。启动子PGAL1、PTEF、PGAL7、PGAL10通过实验室原有质粒为模板扩增获得,PGAL2、PPGI1和基因整合的上下游同源臂(上下游同源臂各500-1 000 bp)以S. cerevisiae S288C基因组为模板扩增得到,以HIS5作为营养缺陷标签。先将上下游同源臂、HIS5标签盒通过同源重组连接在T载上,在实验室已有质粒pY26-PGAL10-chs-PGAL1-4cl-PGAL7-chi、pY26-PGAL10-chi-PGAL1-chs- PGAL7-4cl、pY26-PGAL10-chs-PGAL1-chi-PGAL7-4cl上分别扩增PGAL1-4cl-TGAL1、PGAL1-chs-TGAL1和PGAL1- chi-TGAL1片段,通过同源重组的方法得到质粒T- LEU2up-PGAL1-4cl-TGAL1-HIS5-LEU2down、T-LEU2up- PGAL1-chs-TGAL1-HIS5-LEU2down、T-LEU2up-PGAL1- chi-TGAL1-HIS5-LEU2down、T-25Sup-PGAL1-4cl-TGAL1- HIS5-5Sdown、T-25Sup-PGAL1-chs-TGAL1-HIS5- 5Sdown、T-25Sup-PGAL1-chi-TGAL1-HIS5-5Sdown,然后分别将另外5个启动子与这6个质粒中启动子PGAL1通过同源重组的方法替换,得到另外30个质粒。再通过引物Pf_LEU2和Pr_LEU2分别扩增3个基因在LEU2位点上的整合片段,引物Pf_25S和Pr_5S分别扩增3个基因在rDNA区域的整合片段。文中所用主要引物见表 2。
表 2 本研究中所用的主要引物Table 2 Main primers used in this study
| Primer name | Primer sequence (5′-3′) | Product size (bp) | Amplification efficiency (%) |
| Pf_LEU2 | ATGTTCGTTCCAATGTCAAGTTCG | 24 | |
| Pr_LEU2 | ACTTTTTGTGTGGTGCCCTCCTCC | 24 | |
| Pf_25S | GTCGTACTGATAACCGCAGCAGGT | 24 | |
| Pr_5S | CACAACCGAAACCAAAACCAACG | 23 | |
| Pf_4CL | CTGCTGTCCCGTAAAGTTG | 144 | 103 |
| Pr_4CL | CGCCATAGTGCTGATTGC | ||
| Pf_CHS | ATGGTTACGGTGGAAGAATAC | 144 | 101 |
| Pr_CHS | ATGTTCAGAGTTGGTGATACG | ||
| Pf_CHI | GGTCACGGGCAAATCATAC | 162 | 105 |
| Pr_CHI | TCCAGCAGTTCTTCAGAGC | ||
| Pf_Actin | TTATTGATAACGGTTCTGGTATG | 100 | 104 |
| Pr_Actin | CCTTGGTGTCTTGGTCTAC |
表选项
采用醋酸锂转化法将这36个片段分别整合到菌株Y-01的基因组上,用YNB培养基(添加Trp和Leu)筛选,30 ℃恒温培养3-4 d至长出单菌落。挑取单菌落至YNB液体筛选培养基中培养24 h,13 500 r/min离心5 min去上清,收集菌体提取酿酒酵母基因组,进行PCR验证,得到与实际大小相符的特异性条带,则证明菌株构建成功。
1.2.2 基因转录水平的测定取发酵液中培养至对数中期的酿酒酵母细胞,采用热酚法提取总RNA[9]。利用PrimeScriptTM RT reagent Kit with gDNA Eraser试剂盒(TaKaRa)去除总RNA中的基因组DNA后,将总RNA反转录成cDNA。使用TB GreenTM Premix Ex TaqTMⅡ试剂盒处理样品,在LightCycler 480Ⅱ热循环仪系统(Roche,Mannheim,Germany)上进行定量PCR (qPCR)测定。以β-actin基因作为内参基因,用引物Pf_Actin和Pr_Actin进行qPCR值的归一化。4cl、chs和chi分别用引物Pf_4CL/Pr_4CL、Pf_CHS/Pr_CHS和Pf_CHI/Pr_CHI进行扩增。最后采用2-ΔΔCt方法[10]获得4cl、chs和chi的mRNA表达水平来表征它们的相对表达量。
1.2.3 基因整合拷贝数的测定酵母基因组DNA的提取按照Thermo公司GeneJET Genomic DNA Purification Kit中酵母基因组DNA提取说明书进行操作。
以前的研究表明在不同条件下外源基因在基因组上单拷贝克隆具有遗传稳定性[11-12],而很少有研究能够确定多拷贝克隆的遗传稳定性。由于一株菌株中多拷贝克隆位点起源于同一基因座处的多个同源重组事件,外源基因通常在基因组上串联重复可能导致整合基因的不稳定[13]。为了确定基因4cl、chs和chi整合到酵母rDNA区域菌株的遗传稳定性,将以rDNA为整合位点的所有酿酒酵母重组菌株在YNB缺陷型平板上划线,培养3-4 d至长出单菌落,挑取单菌落至20 mL新鲜的液体YNB缺陷型培养基中,30 ℃、220 r/min培养24 h,然后按1%接种量转移到新鲜的20 mL YPD中并在相同条件下温育24 h。该过程再重复5次,总生长时间为144 h,即传代72代。
从原始的非传代菌株和传代72次菌株中提取基因组DNA,通过qPCR检测基因组中关键酶的整合拷贝数。对带有目的基因4cl、chs和chi的T载体分别进行梯度稀释,制备目的基因质粒的标准曲线,取基因组DNA各1 μL为模板,用荧光引物对目的基因进行qPCR,将得到的Ct值代入标准曲线中,求出DNA样品中起始模板的拷贝数。利用相同的方法计算看家基因sga1 (编码葡聚糖1, 4-α-葡糖苷酶,在基因组以单拷贝的形式存在)的起始模板的拷贝数。因为在表达菌株中都导入了质粒pY26-PGAL10-chi-PGAL1-chs-PGAL7-4cl,因此要除去质粒中目的基因的拷贝数。利用上述同样的方法制作该质粒的标准曲线,以质粒中ura3作为目标基因。因此,基因组中关键酶的整合拷贝数计算公式为:N=(N1-N2)/N0+1(N1代表基因组中目的基因的起始模板的拷贝数,N2代表基因组中ura3基因起始模板的拷贝数,N0代表基因组中看家基因sga1起始模板的拷贝数,1代表基因组染色体上ura3原有一个拷贝数)。
1.2.4 柚皮素的提取与检测发酵结束后,收集发酵液并加入同体积的无水乙醇萃取柚皮素,剧烈振荡混匀后,13 500 r/min离心5 min取上清,有机相滤膜过滤后使用Agilent 1260高效液相色谱仪进行产物检测,采用C18色谱柱(4.6 mm×250 mm)进行色谱分离,色谱柱温度控制在25 ℃,进样量为10 μL,流动相为超纯水(0.3%乙酸)和乙腈(0.3%乙酸),流速为1 mL/min。柚皮素通过乙腈/水的梯度洗脱进行分离:0-10 min,色谱条件为10%-40%的乙腈(体积/体积);10-15 min,色谱条件控制为40%-60%的乙腈;15-17 min,色谱条件控制在60%-10%的乙腈[14]。柚皮素的检测波长为290 nm,以标准品柚皮素浓度为横坐标,紫外区吸收峰峰面积为纵坐标,绘制标准曲线。
2 结果与分析2.1 4CL过量表达对柚皮素产量的影响以Y-01为出发菌株,将在不同强度启动子(PGAL1、PTEF、PGAL7、PGAL10、PGAL2、PPGI1)控制下的4cl基因分别整合在基因组LEU2位点和rDNA区域。每株菌在发酵培养基中发酵得到柚皮素的产量,qPCR得到4cl转录水平上的相对表达量,从而获得柚皮素产量与4cl转录水平的关系。
每株菌做3个平行,并重复3次,以确保实验数据的可靠性,从而获得36组数据。结果如图 1所示,柚皮素的产量与4cl的相对表达量几乎没有相关性(R=-0.2,P=1.62×10-3),并且4cl在基因组上单拷贝整合时有利于柚皮素的积累。以LEU2为整合位点,4cl在启动子PGAL10控制下,柚皮素的最高产量为165.9 mg/L (Y-02),比Y-01 (46.1 mg/L)提高了2.6倍;在启动子PTEF控制下,获得最低产量81.1 mg/L (Y-03),比Y-01增加了75.9%。以rDNA区域为整合位点过量表达4cl,柚皮素产量在21.5 mg/L到97.0 mg/L之间变化,最低产量(Y-12)比Y-01减少了一半。结果表明过量表达4cl并不一定能提高柚皮素的产量。
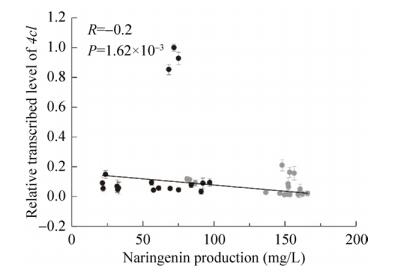 |
| 图 1 柚皮素产量与4cl转录水平的关系 Fig. 1 Relationship between naringenin production and transcription level of 4cl. Gray: single copy clone; black: multicopy clone. |
| 图选项 |
2.2 CHS过量表达对柚皮素产量的影响以上述相同的方法分析CHS过量表达对柚皮素产量的影响。结果如图 2所示,柚皮素的产量与chs转录水平有一定的线性正相关性(R=0.8,P=6.24×10-10),并且chs单拷贝数克隆时更有利于柚皮素的积累。以LEU2为整合位点时,chs在强启动子(PTEF、PGAL1、PGAL7)控制下过量表达,转录水平和柚皮素产量均较高,转录水平在0.8到1.0之间,柚皮素产量在185.3 mg/L到235.9 mg/L之间。当chs受PGAL7启动子控制时,柚皮素最高产量为235.9 mg/L (Y-04),较Y-01提高了4.1倍;当chs受PGAL10启动子控制时,最低产量为114.0 mg/L (Y-05),较Y-01的产量提高了1.5倍。以rDNA为整合位点过量表达chs,柚皮素产量在36.5-174.9 mg/L之间变化,最低产量(Y-14)比Y-01降低了20.8%。结果表明,在一定程度上过量表达chs更有利于柚皮素的大量积累。
 |
| 图 2 柚皮素产量与chs转录水平的关系 Fig. 2 Relationship between naringenin production and transcription level of chs. Gray: single copy clone; black: multicopy clone. |
| 图选项 |
2.3 CHI过量表达对柚皮素产量的影响以上述相同的方法分析CHI过量表达对柚皮素产量的影响。结果如图 3所示,柚皮素的产量与chi转录水平的线性关系较小(R=0.4,P=3.14×10-5),并且CHI在基因组单拷贝克隆时更利于柚皮素的积累。
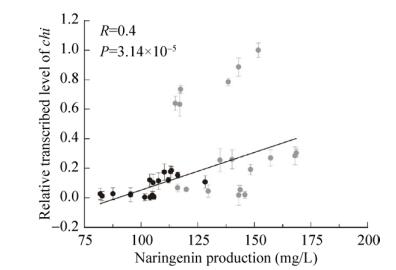 |
| 图 3 柚皮素产量与chi转录水平的关系 Fig. 3 Relationship between naringenin production and transcription level of chi. Gray: single copy clone; black: multicopy clone. |
| 图选项 |
当LEU2作整合位点时,chi在启动子PGAL10控制下,柚皮素最高产量达到168.5 mg/L (Y-06),比Y-01提高了2.7倍;chi在启动子PTEF控制下,柚皮素最低产量为115.0 mg/L (Y-07),比Y-01提高了1.5倍。以rDNA为整合位点过量表达chi,柚皮素的产量在81.9 mg/L到128.2 mg/L之间变化,最低产量(Y-20)比Y-01增加了77.7%。结果表明,过量表达chi提高了柚皮素的产量。
2.4 多拷贝克隆菌株传代稳定性的验证结果如图 4所示,4cl基因组整合重组菌Y-08、Y-09、Y-10、Y-11、Y-12及Y-13未传代与传代72代后拷贝数差异分别为9.0%、5.9%、23.6%、9.5%、10.1%、15.7% (图 4A);chs基因组整合重组菌Y-14、Y-15、Y-16、Y-17、Y-18及Y-19未传代与传代72代后拷贝数差异分别为12.5%、1.3%、0.5%、5.8%、1.7%、2.2% (图 4B);chi基因组整合重组菌Y-20、Y-21、Y-22、Y-23、Y-24及Y-25未传代与传代72代后拷贝数差异分别为6.4%、5.1%、7.9%、11.6%、5.7%、10.6% (图 4C)。上述结果表明,多拷贝克隆菌株传代72代后拷贝数差异基本在15%以下,表明了在本实验条件下多拷贝克隆菌株具有遗传稳定性。
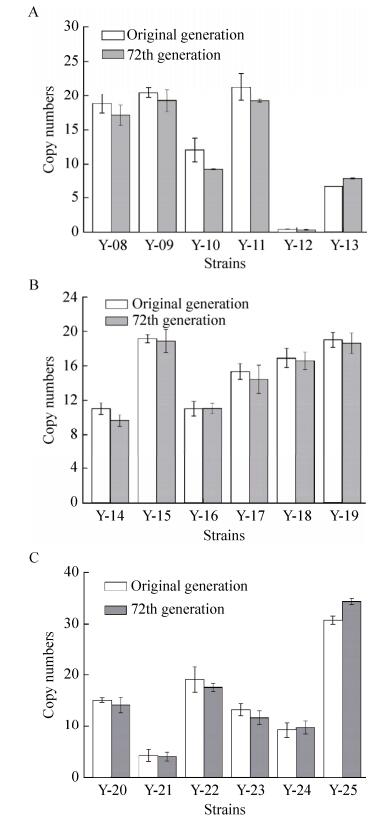 |
| 图 4 关键基因在基因组rDNA上的拷贝数 Fig. 4 Copy number of key genes on genomic rDNA. (A) 4cl. (B) chs. (C) chi. |
| 图选项 |
3 讨论文中以产柚皮素酿酒酵母Y-01为出发菌株,首次考察了关键酶4CL、CHS和CHI分别过量表达对柚皮素产量的影响。当chs在强启动子PGAL7控制下并插入到LEU2位点时,达到最高产量235.9 mg/L,比目前报道的以酿酒酵母为宿主异源合成柚皮素最高产量113.0 mg/L[15]提高了1.1倍。在以前的研究中,因柚皮素合成需要3分子的丙二酰辅酶A,而胞内游离丙二酰辅酶A浓度由于脂肪酸生物合成的内在紧密调节维持在低水平,使丙二酰辅酶A一直被认为是黄酮合成的限制性前体因子。一般采用过量表达乙酰辅酶A羧化酶(Acc1)[16]、敲除丙二酰辅酶A的竞争途径[17]和基于反义RNA策略[18]及CRISPRi系统调控[19]等方法提高胞内丙二酰辅酶A含量,使细胞内丙二酰辅酶A最大程度地流向目标途径以提高目标化合物产量。但是,最新报道显示在酿酒酵母中异源合成柚皮素的前期阶段,丙二酰辅酶A并不是柚皮素合成的限速前体,关键酶之间的协同表达更有利于柚皮素的积累[20]。对于调控途径基因的协同表达,常用的方法是过量表达关键基因的同时调节其表达量,包括调节基因拷贝数、启动子强度及降低蛋白质的降解等方式[15, 21]。但这些方式所调控的范围往往较大,目前还无法对蛋白表达进行精确调控。近年来,越来越多的计算机辅助方法应用于代谢通量平衡分析,如OptForce[22]、OptORF[23]和RobustKnock[24]等。许多基于计算机的途径设计策略已成功应用于所需化合物的生产,例如三乙酸内酯[25]、琥珀酸[26]、脂肪酸[27]等。然而,由于代谢途径涉及众多的前体合成和相关修饰基因导致缺乏准确的酶反应参数,使这些调节策略的使用受到极大的限制。
目前,精确调控途径基因之间的协同表达以平衡代谢通路还是一个巨大的难题。清楚地了解关键酶表达对产物柚皮素的影响有利于后期平衡代谢流的研究。据报道,CHS是燕麦叶中黄酮类化合物的限速酶[28],植物可以通过调控chs表达来调节黄酮类化合物的产生[29-30]。而以微生物为生产宿主异源合成柚皮素途径尚无明确的报道。文中提出了启动子和拷贝数控制代谢工程,并采用qPCR方法检测细胞中靶基因的转录水平,探究了关键酶4CL、CHS和CHI分别过量表达对柚皮素产量的影响。结果显示在一定范围内分别过量表达4CL、CHS和CHI均能提高柚皮素的产量,但是柚皮素产量与4CL或CHI的表达量没有密切的联系,而与CHS的表达量存在线性正相关(R=0.8,P=6.24×10-10),并且柚皮素的产量在过量表达CHS时达到最大值,这表明CHS表达对柚皮素产量的影响最大,并推测CHS是该研究中柚皮素合成途径的限速酶。
此外,实验结果显示,基因单拷贝克隆的表达水平大多高于多拷贝克隆的表达水平,造成的原因可能与酿酒酵母rDNA区域的基因沉默有关[31],外源基因插入该区域,其表达水平往往低于插入到常染色质区域的表达水平。rDNA作为外源基因整合位点,插入其内部的外源基因的遗传稳定性还没有明确的结论,这可能与外源基因的拷贝数和整合形式有关[32]。文中通过qPCR检测未传代和传代后菌株中外源基因拷贝数并比较两者的差异,发现在实验条件下外源基因插入rDNA区域具有遗传稳定性。文中确立了柚皮素产量与关键酶编码基因相对表达量的关系,并揭示了CHS是黄酮化合物合成过程的关键调控位点,为后续深入研究代谢工程强化微生物合成柚皮素等重要黄酮类化合物提供了重要的参考依据。
参考文献
| [1] | Iwashina T. Flavonoid function and activity to plants and other organisms.Biol Sci Space, 2003, 17(1): 24–44.DOI: 10.2187/bss.17.24 |
| [2] | Fowler ZL, Koffas MA. Biosynthesis and biotechnological production of flavanones: Current state and perspectives.Appl Microbiol Biotechnol, 2009, 83(5): 799–808.DOI: 10.1007/s00253-009-2039-z |
| [3] | Wang YC, Chen S, Yu O. Metabolic engineering of flavonoids in plants and microorganisms.Appl Microbiol Biotechnol, 2011, 91(4): 949–956.DOI: 10.1007/s00253-011-3449-2 |
| [4] | Xia YQ, Guo TY, Zhao HL, et al. A novel solid phase for selective separation of flavonoid compounds.J Sep Sci, 2007, 30(9): 1300–1306.DOI: 10.1002/(ISSN)1615-9314 |
| [5] | Guo L, Chen X, Li LN, et al. Transcriptome-enabled discovery and functional characterization of enzymes related to (2S)-pinocembrin biosynthesis from Ornithogalum caudatum and their application for metabolic engineering.Microb Cell Fact, 2016, 15: 27.DOI: 10.1186/s12934-016-0424-8 |
| [6] | Zhou SH, Liu PR, Chen J, et al. Characterization of mutants of a tyrosine ammonia-lyase from Rhodotorula glutinis.Appl Microbiol Biotechnol, 2016, 100(24): 10443–10452.DOI: 10.1007/s00253-016-7672-8 |
| [7] | Wu JJ, Du GC, Zhou JW, et al. Metabolic engineering of Escherichia coli for (2S)-pinocembrin production from glucose by a modular metabolic strategy.Metab Eng, 2013, 16: 48–55.DOI: 10.1016/j.ymben.2012.11.009 |
| [8] | Wu JJ, Zhou TT, Du GC, et al. Modular optimization of heterologous pathways for de novo synthesis of (2S)-naringenin in Escherichia coli.PLoS ONE, 2014, 9(7): e101492.DOI: 10.1371/journal.pone.0101492 |
| [9] | Schmitt ME, Brown TA, Trumpower BL. A rapid and simple method for preparation of RNA from Saccharomyces cerevisiae.Nucleic Acids Res, 1990, 18(10): 3091–3092.DOI: 10.1093/nar/18.10.3091 |
| [10] | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt method.Methods, 2001, 25(4): 402–408.DOI: 10.1006/meth.2001.1262 |
| [11] | Fukunaga T, Cha-Aim K, Hirakawa Y, et al. Designed construction of recombinant DNA at the ura3Δ0 locus in the yeast Saccharomyces cerevisiae.Yeast, 2013, 30(6): 243–253.DOI: 10.1002/yea.v30.6 |
| [12] | Ohi H, Okazaki N, Uno S, et al. Chromosomal DNA patterns and gene stability of Pichia pastoris.Yeast, 1998, 14(10): 895–903.DOI: 10.1002/(ISSN)1097-0061 |
| [13] | Zhu J, Schiestl RH. Human topoisomerase Ⅰ mediates illegitimate recombination leading to DNA insertion into the ribosomal DNA locus in Saccharomyces cerevisiae.Mol Genet Genomics, 2004, 271(3): 347–358.DOI: 10.1007/s00438-004-0987-7 |
| [14] | Wu JJ. Engineering Escherichia coli based on synthetic biology for production of flavonoid scaffolds[D]. Wuxi: Jiangnan University, 2015 (in Chinese). 吴俊俊.合成生物技术改造大肠杆菌生产黄酮骨架物质[D].无锡: 江南大学, 2015. |
| [15] | Koopman F, Beekwilder J, Crimi B, et al. De novo production of the flavonoid naringenin in engineered Saccharomyces cerevisiae.Microb Cell Fact, 2012, 11: 155.DOI: 10.1186/1475-2859-11-155 |
| [16] | Shin SY, Jung SM, Kim MD, et al. Production of resveratrol from tyrosine in metabolically engineered Saccharomyces cerevisiae.Enzyme Microb Technol, 2012, 51(4): 211–216.DOI: 10.1016/j.enzmictec.2012.06.005 |
| [17] | Xu P, Ranganathan S, Fowler ZL, et al. Genome-scale metabolic network modeling results in minimal interventions that cooperatively force carbon flux towards malonyl-CoA.Metab Eng, 2011, 13(5): 578–587.DOI: 10.1016/j.ymben.2011.06.008 |
| [18] | Wu JJ, Yu O, Du GC, et al. Fine-tuning of the fatty acid pathway by synthetic antisense RNA for enhanced (2S)-naringenin production from L-tyrosine in Escherichia coli.Appl Environ Microbiol, 2014, 80(23): 7283–7292.DOI: 10.1128/AEM.02411-14 |
| [19] | Wu JJ, Du GC, Chen J, et al. Enhancing flavonoid production by systematically tuning the central metabolic pathways based on a CRISPR interference system in Escherichia coli.Sci Rep, 2015, 5: 13477.DOI: 10.1038/srep13477 |
| [20] | Lyu XM, Ng KR, Lee JL, et al. Enhancement of naringenin biosynthesis from tyrosine by metabolic engineering of Saccharomyces cerevisiae.J Agric Food Chem, 2017, 65(31): 6638–6646.DOI: 10.1021/acs.jafc.7b02507 |
| [21] | Li MJ, Kildegaard KR, Chen Y, et al. De novo production of resveratrol from glucose or ethanol by engineered Saccharomyces cerevisiae.Metab Eng, 2015, 32: 1–11.DOI: 10.1016/j.ymben.2015.08.007 |
| [22] | Ranganathan S, Suthers PF, Maranas CD. OptForce: An optimization procedure for identifying all genetic manipulations leading to targeted overproductions.PLoS Comput Biol, 2010, 6(4): e1000744.DOI: 10.1371/journal.pcbi.1000744 |
| [23] | Kim J, Reed JL. Optorf: Optimal metabolic and regulatory perturbations for metabolic engineering of microbial strains.BMC Syst Biol, 2010, 4: 53.DOI: 10.1186/1752-0509-4-53 |
| [24] | Tepper N, Shlomi T. Predicting metabolic engineering knockout strategies for chemical production: accounting for competing pathways.Bioinformatics, 2010, 26(4): 536–543.DOI: 10.1093/bioinformatics/btp704 |
| [25] | Chowdhury A, Zomorrodi AR, Maranas CD. k-OptForce: Integrating kinetics with flux balance analysis for strain design.PLoS Comput Biol, 2014, 10(2): e1003487.DOI: 10.1371/journal.pcbi.1003487 |
| [26] | Khodayari A, Chowdhury A, Maranas CD. Succinate overproduction: A case study of computational strain design using a comprehensive Escherichia coli kinetic model.Front Bioeng Biotechnol, 2014, 2: 76. |
| [27] | Ranganathan S, Tee TW, Chowdhury A, et al. An integrated computational and experimental study for overproducing fatty acids in Escherichia coli.Metab Eng, 2012, 14(6): 687–704.DOI: 10.1016/j.ymben.2012.08.008 |
| [28] | Liu WS, Zhang B, Jiang RR. Improving acetyl-CoA biosynthesis in Saccharomyces cerevisiae via the overexpression of pantothenate kinase and PDH bypass.Biotechnol Biofuels, 2017, 10: 41.DOI: 10.1186/s13068-017-0726-z |
| [29] | Eckermann C, Schr?der G, Eckermann S, et al. Stilbenecarboxylate biosynthesis: a new function in the family of chalcone synthase-related proteins.Phytochemistry, 2003, 62(3): 271–286.DOI: 10.1016/S0031-9422(02)00554-X |
| [30] | Sun W, Meng XY, Liang LJ, et al. Molecular and biochemical analysis of chalcone synthase from Freesia hybrid in flavonoid biosynthetic pathway.PLoS ONE, 2015, 10(3): e0119054.DOI: 10.1371/journal.pone.0119054 |
| [31] | Chang JS, Winston F. Spt10 and spt21 are required for transcriptional silencing in Saccharomyces cerevisiae.Eukaryot Cell, 2011, 10(1): 118–129.DOI: 10.1128/EC.00246-10 |
| [32] | Fang C, Wang QH, Selvaraj JN, et al. High copy and stable expression of the xylanase XynHB in Saccharomyces cerevisiae by rDNA-mediated integration.Sci Rep, 2017, 7: 8747.DOI: 10.1038/s41598-017-08647-x |
