
 , 徐平1,3
, 徐平1,3

1. 武汉大学 药学院,湖北 武汉 430000;
2. 武汉大学 基础医学院,湖北 武汉 430000;
3. 军事科学院军事医学研究院生命组学研究所 国家蛋白质科学中心 (北京) 北京蛋白质组研究中心 蛋白质组学国家重点实验室,北京 102206;
4. 华北理工大学 公共卫生学院和附属石家庄市第五医院,河北 唐山 063210;
5. 河北大学 生命科学学院,河北 保定 071000;
6. 中山大学 生命科学学院,广东 广州 510275
收稿日期:2019-03-12;接收日期:2019-06-14
基金项目:国家精准医学重大专项(No. 2017YFC0906600),国家传染病重大专项(No. 2018ZX10302302001003),国家自然科学基金(Nos. 31670834,31870824,91839302),广东省基础及应用基础研究博士科研启动项目(No. 2018A030310257)资助
摘要:Rv2742是本课题组前期基于蛋白质基因组学策略从结核分枝杆菌Mycobacterium tuberculosis H37Rv中发现、鉴定的遗漏注释基因。文中旨在建立结核分枝杆菌H37Rv漏注释蛋白Rv2742的可溶性诱导表达、纯化体系,为进一步探索Rv2742基因参与的生物学功能奠定基础。前期实验发现构建的pGEX-4T- 2-Rv2742、pET-28a-Rv2742、pET-32a-Rv2742及pMAL-c2X-Rv2742原核表达载体均无法实现目的蛋白的诱导表达。但经密码子优化后,仅有pMAL-c2X-Rv2742载体能够实现目的蛋白的可溶性诱导表达。此外,通过比较不同宿主菌、温度及IPTG浓度对目的蛋白表达量的影响,发现目的蛋白在Rosetta(DE3)中,16 ℃及0.5 mmol/L IPTG诱导条件下表达量最高。直链淀粉树脂(Amylose resin)亲和层析柱纯化获得较纯的产物,经LC-MS/MS验证确认是Rv2742融合蛋白肽段序列。成功获得结核分枝杆菌H37Rv新基因Rv2742的重组蛋白,可进一步开展其潜在相互作用及免疫原性研究工作。
关键词:结核分枝杆菌新基因原核表达亲和纯化
Cloning, expression and purification of novel gene Rv2742 in Mycobacterium tuberculosis H37Rv
Jialing Zhao1,3, Shujia Wu2,3, Hong Wang3,4, Qianlin Li4, Jinshuai Sun3,5, Lei Chang3, Erhei Dai4, Junzhu Wu2, Yao Zhang3,6

 , Ping Xu1,3
, Ping Xu1,3

1. School of Pharmaceutical Sciences, Wuhan University, Wuhan 430000, Hubei, China;
2. School of Basic Medical Sciences, Wuhan University, Wuhan 430000, Hubei, China;
3. State Key Laboratory of Proteomics, Beijing Proteome Research Center, National Center for Protein Sciences (Beijing), Beijing Institute of Lifeomics, Beijing 102206, China;
4. School of Public Health and Affiliated Shijiazhuang Fifth Hospital, North China University of Science and Technology, Tangshan 063210, Hebei, China;
5. School of Life Sciences, Hebei University, Baoding 071000, Hebei, China;
6. School of Life Sciences, Sun Yat-sen University, Guangzhou 510275, Guangdong, China
Received: March 12, 2019; Accepted: June 14, 2019
Supported by: Chinese National Basic Research Programs (No. 2017YFC0906600), National Megaprojects for Key Infectious Diseases (No. 2018ZX10302302001003), National Natural Science Foundation of China (Nos. 31670834, 31870824, 91839302), Applied and basic research foundation of Guangdong Province (No. 2018A030310257)
Corresponding author: Yao Zhang. Tel: +86-10-61777113; Fax: +86-10-61777050; E-mail: zhangyaowsw@163.com;
Ping Xu. Tel: +86-10-61777113; Fax: +86-10-61777050; E-mail: xupingghy@gmail.com.
Abstract: Rv2742 is a novel gene identified from Mycobacterium tuberculosis H37Rv by the proteogenomics strategy. The aim of this study was to establish a system of soluble expression and purification of the missing protein Rv2742 in M. tuberculosis H37Rv, to provide reference for further research on the biological function of Rv2742. The soluble protein was not successfully induced by prokaryotic expression vectors pGEX-4T-2-Rv2742, pET-32a-Rv2742, pET-28a-Rv2742 and pMAL-c2X-Rv2742. After the codon of novel gene Rv2742 was optimized according to E. coli codon usage frequency, only the recombinant strain containing plasmid pMAL-c2X-Rv2742 could produce soluble products of Rv2742 encoding gene. In addition, the expression effects of the desired fusion protein were also analyzed under different conditions including hosts, culture temperatures and IPTG concentrations. The optimum expression conditions were as follows: Rosetta(DE3) host, 16 ℃ culture temperature and 0.5 mmol/L IPTG. After being purified by affinity chromatography with amylose resin, the fusion protein sequence was confirmed by LC-MS/MS. These results indicated that the novel gene Rv2742 product could be successfully induced and expressed in a soluble form by the expression system pMAL-c2X with MBP tag. Our findings provide reference for studies on potential interaction and immunogenicity.
Keywords: Mycobacterium tuberculosisnovel geneprokaryotic expressionaffinity purification
结核分枝杆菌(Mycobacterium tuberculosis,MTB)是引起人类结核病的病原菌[1],可入侵多种组织、器官如胸膜、淋巴管、泌尿生殖器和骨关节炎等[2],但以肺结核最为常见。据2018年WHO报道,2017年全球约有130万人死于该病,有1 000万新病例发生。结核病仍然是传染性疾病中的头号杀手。我国依然是全球结核高发病、高耐药、高HIV共感染负担国家之一[3]。因此,有效的早期诊断新方法和及时治疗是控制结核疫情蔓延的重要手段[4]。
目前,临床常用的诊断方法包括涂片显微镜法、细菌培养法、核酸扩增试验(如GeneXpert MTB/RIF)、结核菌素皮肤试验(TST)、γ干扰素释放试验(IGRA)等[5],但存在涂片染色检出率低、培养周期长、结核抗原特异性差和灵敏度不高等问题。其中,结核菌素皮肤试验因与卡介苗(BCG)及非结核分枝杆菌之间存在交叉反应而导致特异性不高[6],而基于ESAT-6与CFP-10研发的T-SPOT试剂盒虽被广泛地用于临床结核菌感染的筛查,但仍存在“阳性不能确证,阴性不能排除”的弊端,不能有效区分活动性结核患者和潜伏感染者[7]。因此,寻找能够有效区分活动性结核患者和潜伏感染者高特异性、灵敏性的有效抗原可解决结核临床诊断的难题。
1998年,Cole等[8-9]基于全基因组鸟枪法和系统测序法,完成了结核分枝杆菌H37Rv的基因组测序和注释工作。2002年,Camus等[10]通过EMBL、TrEMBL和SWISS-PROT三个数据库序列同源性比较,补充了82个新编码基因。传统的基因组注释是利用已知的基因特征和同源性比较实现的,难以发现人类未知的、新基因结构特征的基因,造成这些生物学功能和对菌株鉴别可能重要的基因遗漏,逃离人类研究的视线,形成盲区。国内外研究者基于算法优化[10]、基因预测软件及注释工具开发、转录组学和蛋白质组学[11-15]、比较基因组学[16]等策略,一直在校正和完善H37Rv基因组注释数据库。然而,MTB属于原核生物,受基因组测序质量影响以及缺乏合适的校正评估方法,原核生物基因组注释中尚可能存在注释错误[17] (过度注释;ORF起始、终止位点注释错误;可变剪接;核糖体移位;漏注释等),给准确解析相应物种的生物学机制带来了困扰[18]。
利用蛋白质组学数据,结合基因组数据、转录组数据来研究基因组注释问题,被称为蛋白质基因组学(Proteogenomics)[19]。近10年来,蛋白质组学直接用于基因组的注释已经越来越受到相关领域的关注。本课题组前期基于深度覆盖的精准蛋白质基因组学技术[20, 24]和比较基因组学策略,完成了H37Rv基因组数据库的重注释,发现了22个结核遗漏注释基因,矫正了28个N端注释错误基因(图 1,尚未发表)。此发现有利于结核新型特异性免疫原性分子标志物筛选,为结核病的快速、精准临床检测提供基础理论和原始创新技术支持。
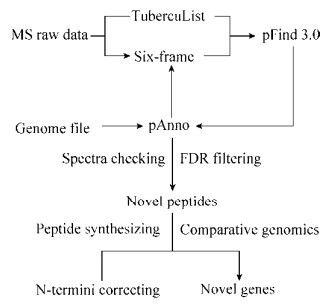 |
| 图 1 结核分枝杆菌H37Rv蛋白质基因组学分析流程图 Fig. 1 Workflow of Mycobacterium tuberculosis H37Rv proteogenomic analysis. |
| 图选项 |
从TubercuList库中下载了H37Rv (http://genolist.pasteur.fr/TubercuList/)基因组注释数据库,共4 031个基因,包含4 018个编码基因和13个假基因。六阅读框翻译数据库由中国科学院计算技术研究所pFind团队与我们课题组合作开发的蛋白质基因组学软件pAnno对NCBI中结核分枝杆菌H37Rv的基因组文件(NC000962.3.fna)按照正链和互补链上连续的3个核苷酸翻译一个氨基酸的规则翻译,使用终止子到终止子的翻译模式,采用MTB特殊的3种起始密码子ATG、GTG和TTG,共翻译得到141 851个开放阅读框(Opening reading frame,ORF)。与已注释的蛋白质序列数据库进行合并,经过严格FDR筛选,结合谱图人工核实、肽段合成验证、转录丰度、基因组多序列比对,获得了一批质量可信的新肽段,完成了H37Rv新编码基因的验证和原先注释错误基因的矫正。
Rv2742基因是我们发现的H37Rv遗漏注释基因之一(根据基因在染色体的坐标位置及结核基因命名法进行命名)。前期深度覆盖质谱数据共鉴定到两条肽段即“PNPWQYIR”和“VHNLDPEL VDEHAR”,化学合成肽段与原先鉴定肽段谱图相似性分别为0.94和0.96,意味着Rv2742基因成功被转录、翻译。T细胞表位预测(https://www.iedb.org/)发现,Rv2742新蛋白中高可信的T细胞表位数有19个(> 0.5分),高可信T细胞表位覆盖到该蛋白66.42%的序列,与已知表位库比较,都是新型的表位,该蛋白具有很好的免疫原性。此外,经NCBI-BLASTP后,数据库中没有比对到相似序列,属于功能未知蛋白。为进一步探索Rv2742新基因功能及其潜在临床应用价值,有必要建立其表达、纯化体系。
本研究比较了4种不同的原核表达载体(pGEX-4T-2、pET-32a、pET-28a和pMAL-c2X)、密码子利用偏性、5种宿主(JM109 (DE3)、BL21、BL21 (DE3)、BL21 (DE3) pLysS和Rosetta (DE3))、不同诱导温度(16 ℃和28 ℃)、不同IPTG浓度(0.5 mmol/L和1.0 mmol/L)等条件下,目的蛋白的表达量。旨在最优实现结核分枝杆菌H37Rv新编码基因Rv2742克隆、可溶性诱导表达,从而能高效纯化Rv2742蛋白,为后续开展该蛋白的潜在相互作用及免疫原性研究奠定基础。后期实验采用的质谱方法是基于“自下而上”(Bottom-up)鸟枪法的策略[21],其基本流程是首先将蛋白质酶解为肽段,再用反相液相色谱进行分离,被洗脱的肽段用串联质谱(一级质谱和二级质谱)检测,最后利用搜库软件对样品中的肽段和蛋白质进行鉴定。
1 材料与方法1.1 材料1.1.1 菌株及质粒M. tuberculosis H37Rv菌株DNA来自河北省石家庄市第五医院。大肠杆菌DH5α、BL21 (DE3)及BL21 (DE3) pLysS感受态细胞均购自北京康为世纪生物科技有限公司。大肠杆菌BL21及Rosetta (DE3)感受态细胞均购自北京全式金生物技术有限公司。JM109 (DE3)感受态细胞购自北京鼎国昌盛生物技术有限责任公司。pGEX-4T-2为本实验室保存质粒;pET-28a质粒由国家蛋白质科学中心田春艳博士馈赠;pET-32a质粒由德阳生物技术(固安)有限责任公司技术总监赵明治博士馈赠;pMAL-c2X质粒由华大青兰生物科技(无锡)有限公司馈赠。引物合成和测序均由北京擎科新业生物技术有限公司完成。
1.1.2 主要试剂DNA marker (DL 1 000、λ-Hind Ⅲ)、pMD18-T载体试剂盒均购自宝生物工程有限公司(大连);2×Taq PCR Master Mix、DNA胶回收试剂盒、质粒DNA提取试剂盒均购自天根生化科技有限公司(北京);限制性内切酶(BamHⅠ、Hind Ⅲ、EcoRⅠ、NotⅠ、XhoⅠ、NdeⅠ及直链淀粉树脂均购自纽英伦生物技术公司(美国);注射用氨苄西林钠购自石药集团中诺药业有限公司;蛋白marker (10 kDa)购自富酶泰斯公司(加拿大);琼脂糖购自Biowest公司(法国);耐自消化高活性乙酰化胰蛋白酶来自北京酶知源生物科技有限公司[22-23]。
1.1.3 主要仪器设备PCR仪、凝胶电泳电源、凝胶成像系统、DNA电泳仪、电泳槽均为BIO-RAD (美国)公司产品;冷冻离心机、摇床、LTQ Orbitrap Velos质谱仪均为Thermo Fisher Scientific (美国)公司产品;紫外分光光度计为岛津公司(日本)产品;纯水仪为Merck Millipore (德国)公司产品;电热恒温水浴锅为北京长风公司产品;电热恒温培养箱为上海一恒科学仪器有限公司产品;超声波细胞粉碎机为宁波新芝生物科技股份有限公司产品。
1.2 方法1.2.1 重组表达质粒的构建以H37Rv DNA为模板,引物如表 1所示,分别扩增获得Rv2742新基因片段。PCR反应体系均为:ddH2O 9.5 μL、2×Taq PCR Master Mix 12.5 μL、DNA 1 μL、上下游引物各1 μL。PCR反应条件:94 ℃预变性3 min;94 ℃变性1 min,60 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃延伸5 min。
表 1 引物序列Table 1 Primer sequence
| Expression vector | Primer name | Primer sequence (5′–3′) | Restriction site | Size (bp) |
| pGEX-4T-2-1 | F-1 | GGATCCATGAGCGACAACGCAATCC | BamHⅠ | 25 |
| pET-32a-1 | R-1 | GCGGCCGCTTAGTCGTTCGGGTCCG | NotⅠ | 25 |
| pET-28a-1 | ||||
| pMAL-c2X-1 | F-2 | CGGAATTCATGAGCGACAACGCAAT | EcoRⅠ | 25 |
| R-2 | AAGCTTTTAGTCGTTCGGGTCCGGC | Hind Ⅲ | 25 | |
| pGEX-4T-2-2 | F-3 | CCGGAATTCATGAGCGATAACGCGA | EcoRⅠ | 25 |
| pET-32a-2 | R-3 | TTGCGGCCGCTCAATCGTTAGGATC | NotⅠ | 25 |
| pET-28a-2 | F-4 | CATATGATGAGCGATAACGCGATTC | NdeⅠ | 25 |
| R-4 | CTCGAGTCAATCGTTAGGATCAGGG | XhoⅠ | 25 | |
| pMAL-c2X-2 | F-5 | GAATTCATGAGCGATAACGCGATTC | EcoRⅠ | 25 |
| R-5 | AAGCTTTCAATCGTTAGGATCAGGG | Hind Ⅲ | 25 | |
| .-1 represented the primer before codon optimization while -2 represented the primer after codon optimization. The single underlined sequences indicate restriction enzyme site. | ||||
表选项
PCR产物利用DNA回收试剂盒回收,与pMD18-T克隆载体于16 ℃过夜连接。连接体系为T4 DNA连接酶缓冲液1 μL、T4 DNA连接酶1 μL、ddH2O 3 μL、pMD18-T克隆载体0.5 μL、目的片段回收产物4.5 μL。将连接产物转化至DH5α感受态细胞。菌落PCR鉴定为阳性的质粒送至北京擎科新业生物技术有限公司进行测序。NCBI在线比对为阳性的质粒命名为pMD18T-Rv2742。
上述测序正确的质粒分别与其相应的载体(pGEX-4T-2、pET-32a、pET-28a或pMAL-c2X)分别利用BamHⅠ/NotⅠ或EcoRⅠ/Hind Ⅲ进行双酶切。双酶切体系为ddH2O 24 μL、质粒20 μL、NE缓冲液3.1 5 μL,BamHⅠ(0.3 μL)/NotⅠ(0.5 μL)或EcoRⅠ/Hind Ⅲ (各0.3 μL) 37 ℃双酶切6 h,DNA回收试剂盒分别回收目的基因及载体片段,经T4 DNA连接酶16 ℃过夜连接。连接体系为T4 DNA连接酶缓冲液1 μL,T4 DNA连接酶1 μL,ddH2O 3 μL,表达载体双酶切产物1 μL,目的片段回收产物4 μL。将连接产物10 μL转化至DH5α感受态细胞,菌落PCR为阳性的质粒命名为pGEX-4T-2-Rv2742-1、pET-32a-Rv2742-1、pET-28a-Rv2742-1及pMAL-c2X-Rv2742-1。
1.2.2 Rv2742基因密码子优化及优化产物重组表达质粒的构建依据大肠杆菌密码子偏好性,将Rv2742基因序列进行密码子优化。对优化后的基因片段进行引物设计(表 1),操作步骤同前。将连接产物转化至DH5α感受态细胞,菌落PCR验证为阳性的质粒分别命名为pGEX-4T-2-Rv2742-2、pET-32a-Rv2742-2、pET-28a-Rv2742-2、pMAL-c2X-Rv2742-2。
1.2.3 重组质粒原核表达及鉴定重组质粒分别转化BL21 (DE3)宿主菌,挑取单菌落于5 mL LB (含100 μg/mL Amp/Kana)液体培养基过夜培养。以起始OD600=0.1接种于50 mL LB (含100 μg/mL Amp/Kana)液体培养基中。37 ℃振荡培养至OD600为0.6–0.8时,加入终浓度为1 mmol/L的IPTG,28 ℃、200 r/min诱导6–8 h。分别收取诱导前后14 OD菌体,离心后用预冷的PBS洗涤两次,加入等体积裂解液(PBS+1 mmol/L PMSF+0.5% Triton X-100)超声破碎,分别取上清和沉淀进行SDS-PAGE,比较IPTG诱导前后目的蛋白的表达情况。
1.2.4 Rv2742新蛋白诱导表达条件的优化将重组质粒pMAL-c2X-Rv2742-2取0.5 μL分别转化至表达宿主菌JM109 (DE3)、BL21、BL21 (DE3)、BL21 (DE3) pLysS及Rosetta (DE3)中,28 ℃、终浓度为1 mmol/L的IPTG诱导表达6–8 h,比较不同宿主菌对目的蛋白表达量的影响。基于表达量较高的宿主进行不同温度(16 ℃和28 ℃)、不同IPTG浓度(终浓度为0.5 mmol/L和1.0 mmol/L)过夜诱导,比较诱导条件对目的蛋白表达量的影响。10% SDS-PAGE检测目的蛋白表达情况。
1.2.5 Rv2742融合蛋白的纯化及LC-MS/MS鉴定将诱导后上清经直链淀粉树脂(Amylose resin)柱(100 μL) 4 ℃孵育1 h,用清洗液(20 mmol/L Tris-HCl (pH 7.4)、200 mmol/L NaCl、1 mmol/L EDTA、1 mmol/L二硫苏糖醇、1 mmol/L苯甲基磺酰氟、0.5% Triton X-100) 500 μL洗3次,用洗脱液(清洗液+10 mmol/L麦芽糖) 100 μL洗脱2次,纯化得到目的融合蛋白,10% SDS-PAGE检测。经考马斯亮蓝R-250染色并脱色后检测目的融合蛋白的纯化情况。
将纯化的目的条带切成1 mm3胶粒,脱色、脱水、蒸干、经乙酰化胰蛋白酶(10 ng/μL) 37 ℃过夜消化、抽提肽段、蒸干后置-80 ℃冻存。
蒸干后的肽段使用上样缓冲液(1% ACN+1% FA+98% ddH2O)进行充分溶解。通过超高压液相色谱进行分离,其中分离柱采用内径75 μm长15 cm的C18反相色谱分析柱,内部C18填料内径3 μm。洗脱组分经纳升级电喷雾离子源接口喷出进入LTQ Orbitrap Velos分析。毛细管离子传输温度为250 ℃,电喷雾电压为1.8 kV。质谱采用一级质谱数据依赖的二级质谱扫描模式(Data dependent MS/MS scan)碰撞诱导裂解(Collision-induced dissociation,CID)模式碎裂一级离子。一级质谱扫描质核比范围为300– 1 600 (m/z),分辨率设置为30 000;自动增益控制(Automatic gain control,AGC)为106。依次选取一级信号强度最高的20个离子进行二级碎裂分析,碰撞归一化能量(Normalized collision energy,NCE)为35%;AGC为104;最大离子注入时间为30 ms;动态排除(Dynamic exclusion)为40 s(40s内不重复扫描已检测母离子)[24]。
1.2.6 数据库搜索pFind3 (https://github.com/pFindStudio/pFind3/issues)对质谱产生的数据文件(.raw)进行蛋白质数据库搜索。数据库由从UniProt (Version:201601)下载目标E. coli完整蛋白质组序列、结核分枝杆菌H37Rv遗漏注释蛋白Rv2742序列、pMAL-c2X载体标签MBP蛋白序列和常见污染构成。搜库参数设置如下:1)胰酶特异性酶切(Trypsin KR_C);2)固定修饰为半胱氨酸的烷基化修饰Carbamidomethyl[C] (+57.021 46 Da);3)可变修饰为甲硫氨酸氧化修饰Oxidation[M] (+15.994 92 Da);4)母离子质量误差20 ppm;5)子离子质量误差0.5 Da;6)允许最大漏切位点数目为2个;7) 7 AA≤肽段长度≤100 AA;8)肽段最大修饰为2种。搜库结果采用目标-诱饵库策略进行过滤,并设定肽段和蛋白质鉴定假阳性率(False discovery rate,FDR)均小于1%[25]。
1.2.7 IPTG诱导表达Rv2742蛋白生长曲线的测定将重组质粒pMAL-c2X-Rv2742-2取0.5 μL转化至BL21 (DE3),挑取单菌落扩大培养于5 mL LB (含100 μg/mL Amp)液体培养基过夜培养。采用比浊法,分光光度计波长为600 nm检测。用LB液体培养基调零,以起始OD600为0.1接种于5 mL LB (含100 μg/mL Amp)液体培养基中,37 ℃、200 r/min振荡培养。待菌体生长至对数期,实验组加入终浓度为1 mmol/L的IPTG诱导,设置3组生物学重复,每隔1.5 h检测OD值。
2 结果与分析2.1 重组表达质粒的构建重组表达质粒构建方法类似,以pMAL-c2X-Rv2742-2为例。以密码子优化后的Rv2742序列为模板扩增,获得Rv2742基因片段约420 bp,与预期的目的片段大小一致(图 2A),表明成功获得目的基因Rv2742。纯化的Rv2742目的片段与pMD 18-T连接转化后进行菌落PCR验证,在目的条带处成功获得特异性条带(图 2B),且测序序列正确。EcoRⅠ和Hind Ⅲ双酶切pMD 18T-Rv2742质粒6 h,酶切片段约为420 bp,与预期大小相符(图 2C)。EcoRⅠ和Hind Ⅲ双酶切质粒pMAL-c2X,酶切片段约为6 478 bp (图 2D ),与预期相符。将双酶切后回收的目的片段与载体片段连接转化进行菌落PCR验证,在目的条带处获得特异性条带(图 2E),说明成功构建重组表达质粒pMAL-c2X-Rv2742-2。
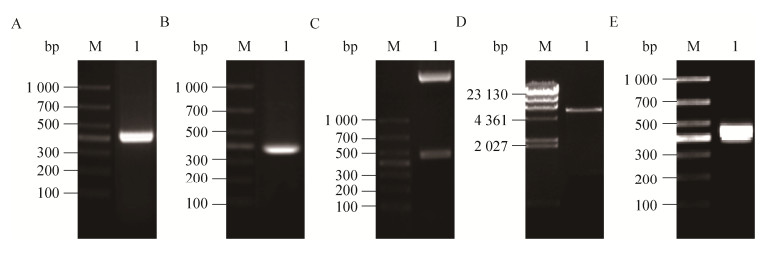 |
| 图 2 重组表达质粒pMAL-c2X-Rv2742-2构建及鉴定 Fig. 2 Construction and identification of recombinant plasmid pMAL-c2X-Rv2742-2. (A) PCR amplification of Rv2742 gene. Lane M: DL 1 000 DNA marker; lane 1: PCR product of Rv2742 gene. (B) Detection of positive bacteria pMD18T- Rv2742 by PCR. Lane M: DL 1 000 DNA marker; lane 1: PCR product of pMD18T-Rv2742. (C) Plasmid pMD18T-Rv2742 digested by EcoRⅠ/Hind Ⅲ. Lane M: DL1 000 DNA marker; lane 1: plasmid pMD18T-Rv2742 digested by EcoRⅠ/Hind Ⅲ. (D) Plasmid pMAL-c2X digested by EcoRⅠ/Hind Ⅲ. Lane M: λ-Hind Ⅲ; lane 1: plasmid pMAL-c2X digested by EcoRⅠ/Hind Ⅲ. (E) Construction of recombinant plasmid pMAL-c2X-Rv2742-2. lane M: DL 1 000 DNA marker; lane 1: Detection of positive bacteria recombinant plasmid pMAL-c2X-Rv2742-2 by PCR. |
| 图选项 |
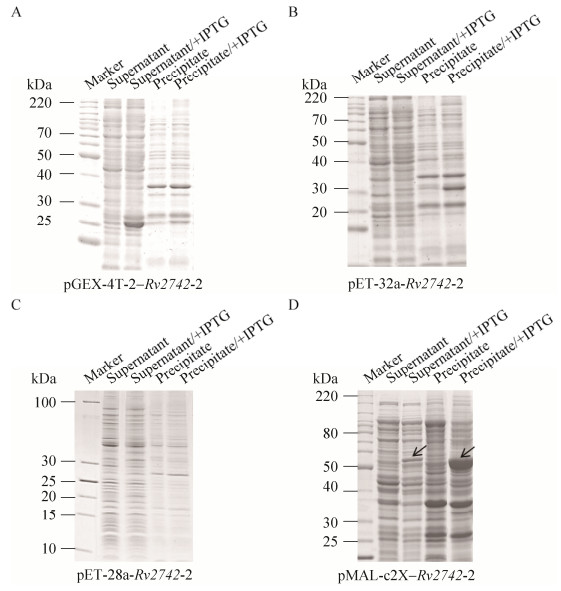
2.2 四种表达载体均未实现Rv2742目的蛋白的诱导表达经Glycine-SDS-PAGE或Tricine-SDS-PAGE[26]电泳结果分析,如图 3A所示,发现在28 ℃ 1 mmol/L IPTG诱导6 h后,pGEX-4T-2系统表达的Rv2742目的融合蛋白(分子量为40.85 kDa)主要分布在沉淀中,但在诱导后上清中并没有明显的新增条带,说明以pGEX-4T-2系统表达的Rv2742目的融合蛋白主要以包涵体形式存在。如图 3B、3C、3D所示,分别以pET-32a、pET-28a、pMAL-c2X系统表达的Rv2742目的融合蛋白(分子量分别为28.16、18.04、56 kDa)在诱导后上清与沉淀中均未出现明显的新增特异性条带,说明以pET-32a、pET-28a以及pMAL-c2X表达的Rv2742目的融合蛋白均未能成功实现诱导表达。综上所述,以H37Rv基因组DNA克隆的Rv2742基因序列为模板所构建的pGEX-4T-2-Rv2742-1、pET-32a-Rv2742-1、pET-28a-Rv2742-1、pMAL- c2X-Rv2742-1原核表达系统均未能实现Rv2742目的蛋白可溶性诱导表达。
 |
| 图 3 分别利用pGEX-4T-2、pET-32a、pET-28a、pMAL-c2X四种表达载体表达Rv2742重组蛋白的Glycine-SDS-PAGE/Tricine-SDS-PAGE图谱(Rv2742密码子优化前) Fig. 3 Glycine-SDS-PAGE/Tricine-SDS-PAGE analysis of expression of Rv2742 recombinant protein by pGEX-4T-2, pET-32a, pET-28a and pMAL-c2X expression vectors before Rv2742 codon optimization. (A) Glycine-SDS-PAGE analysis of expression of Rv2742 recombinant protein by pGEX-4T-2 expression vectors before Rv2742 codon optimization. (B) Glycine-SDS-PAGE analysis of expression of Rv2742 recombinant protein by pET-32a expression vectors before Rv2742 codon optimization. (C) Tricine-SDS-PAGE analysis of expression of Rv2742 recombinant protein by pET-28a expression vectors before Rv2742 codon optimization. (D) Glycine-SDS-PAGE analysis of expression of Rv2742 recombinant protein by pMAL-c2X expression vectors before Rv2742 codon optimization. |
| 图选项 |
2.3 Rv2742基因序列密码子优化前后比较参考大肠杆菌的密码子偏好性,对Rv2742序列进行密码子优化,经DNAMAN序列比对密码子优化前后序列,结果如4A所示。参照GenScript (http://www.genscript.com/)中E. coli同义密码子的相对频率将密码子分为3类:高频密码子、常用密码子及稀有密码子[27],并对优化前后的密码子进行统计分类,结果如4B所示,表明优化前的Rv2742含8.15%的稀有密码子且高频密码子所占比例小(28.15%)。对序列的密码子适应指数(Codon adaptation index,CAI)计算,发现新编码基因Rv2742的CAI从0.73升高到0.92,如图 4C所示,结果显示Rv2742经密码子优化后更适于大肠杆菌的原核表达,这为Rv2742的原核表达提供有利的理论依据。统计结果发现优化前Rv2742的密码子适应度普遍偏低,且最适于E. coli表达的密码子占51% (91–100),而不适于E. coli表达的密码子占9% (0–30),结果如图 4D所示,优化后减少了Rv2742的稀有密码子和适应度低的密码子,不适于E. coli表达的密码子仅占2% (0–30),同时增加了高频密码子的使用,使得最适密码子增加到86% (91–100),有效地提高了其转录和翻译的效率。
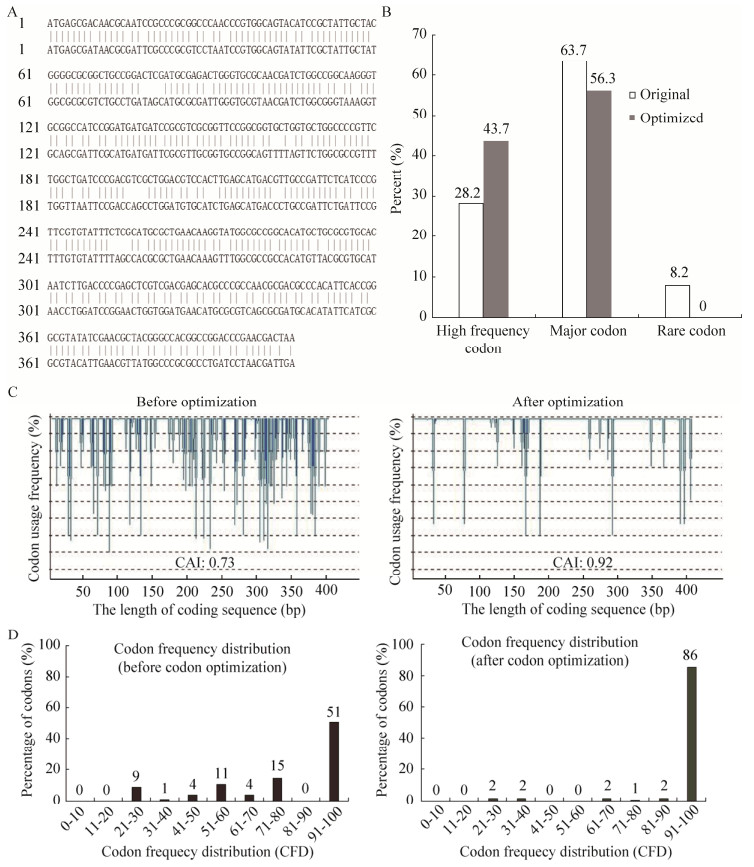 |
| 图 4 Rv2742基因序列密码子优化前后差异的比较 Fig. 4 Comparison of differences from Rv2742 gene sequence before and after codon optimization. (A) Blast analysis of the differences from Rv2742 gene sequence before and after codon optimization. (B) Comparison of the proportion of high frequency codon, major codon and rare codon before and after codon optimization. (C) Comparison of the CAI value before and after codon optimization. (D) Comparison of codon frequency distribution before and after codon optimization. |
| 图选项 |
2.4 Rv2742新基因密码子优化后实现了目的蛋白的诱导表达以Rv2742密码子优化后的序列为模板,将构建成功的重组质粒pGEX-4T-2-Rv2742-2、pET- 32a-Rv2742-2、pET-28a-Rv2742-2分别转化至大肠杆菌BL21 (DE3)中,28 ℃、终浓度为1 mmol/L IPTG诱导6 h后,经Glycine-SDS-PAGE或Tricine- SDS-PAGE分析,如图 5A、5B、5C所示,IPTG诱导后的上清和沉淀中均未出现明显新增条带,表明密码子优化后,pGEX-4T-2、pET-32a、pET-28a原核表达系统均未能实现Rv2742重组蛋白的诱导表达(分子量分别为40.85 kDa、28.16 kDa、18.04 kDa)。而将含重组质粒pMAL-c2X-Rv2742-2的大肠杆菌BL21 (DE3)在28 ℃、终浓度为1 mmol/L IPTG诱导6 h。10% Glycine-SDS-PAGE显示,与未加IPTG诱导前相比,诱导后上清和沉淀均在56 kDa出现特异性新增条带,其大小与目的蛋白预期相符。初步说明目的片段在大肠杆菌pMAL-c2X表达系统中得到了表达,且表达产物能以可溶性蛋白形式出现,但表达量不高(图 5D)。
 |
| 图 5 分别利用pGEX-4T-2、pET-32a、pET-28a、pMAL-c2X四种表达载体表达Rv2742重组蛋白的SDS-PAGE/Tricine-SDS-PAGE图谱(Rv2742密码子优化后) Fig. 5 SDS-PAGE/Tricine-SDS-PAGE analysis of expression of Rv2742 recombinant protein by pGEX-4T-2, pET-32a, pET-28a, pMAL-c2X expression vectors after Rv2742 codon optimization. (A) SDS-PAGE analysis of expression of Rv2742 recombinant protein by pGEX-4T-2 expression vectors after Rv2742 codon optimization. (B) SDS-PAGE analysis of expression of Rv2742 recombinant protein by pET-32a expression vectors after Rv2742 codon optimization. (C) Tricine-SDS-PAGE analysis of expression of Rv2742 recombinant protein by pET-28a expression vectors before Rv2742 codon optimization. (D) SDS-PAGE analysis of expression of Rv2742 recombinant protein by pMAL-c2X expression vectors after Rv2742 codon optimization. |
| 图选项 |
2.5 宿主菌Rosetta (DE3)实现了目的蛋白的高表达及诱导条件的优化将重组质粒pMAL-c2X-Rv2742-2分别转化至宿主菌JM109 (DE3)、BL21、BL21 (DE3)、BL21 (DE3) pLysS及Rosetta (DE3)中,28 ℃、终浓度为1 mmol/L IPTG诱导6–8 h。10% SDS-PAGE结果显示,Rv2742目的蛋白虽在其他4个宿主菌JM109 (DE3)、BL21、BL21 (DE3)、BL21 (DE3) pLysS中均实现了可溶性诱导表达,但在Rosetta (DE3)宿主菌中的表达量尤为显著(图 6A)。在Rosetta(DE3)宿主菌、终浓度为1 mmol/L IPTG诱导条件下,我们比较了28 ℃ (6–8 h)和16 ℃ (12 h)温度下目的蛋白的表达量,结果表明目的蛋白在16 ℃低温诱导时表达量更高(图 6B)。在Rosetta (DE3)宿主菌及16℃过夜诱导条件下,我们比较了终浓度为1 mmol/L和0.5 mmol/L IPTG对目的蛋白表达量的影响,结果表明目的蛋白在终浓度为0.5 mmol/L IPTG诱导条件下表达量最高(图 6B)。综上所述,在宿主菌Rosetta (DE3)、16 ℃及终浓度为0.5 mmol/L IPTG过夜诱导条件下能更有效地实现Rv2742蛋白的融合表达。
 |
| 图 6 Rv2742融合蛋白在五种不同宿主菌中诱导表达及在Rosetta (DE3)宿主菌中诱导条件的优化 Fig. 6 Comparison of Rv2742 expression based on five different bacterial hosts and the optimization of expression conditions in the host Rosetta (DE3). (A) SDS-PAGE analysis of Rv2742 expression by different hosts including JM109 (DE3), BL21, BL21 (DE3), BL21(DE3)pLysS and Rosetta (DE3). (B) The optimization of Rv2742 expression conditions for Rosetta (DE3) including culture temperatures and IPTG concentrations. |
| 图选项 |
2.6 Rv2742目的融合蛋白的纯化及LC-MS/ MS鉴定在Rosetta (DE3)宿主菌中,16 ℃、终浓度为0.5 mmol/L IPTG过夜诱导表达的上清超声裂解后亲和纯化(图 7A),在56 kDa处获得纯度较高的Rv2742目的融合蛋白。经LC-MS/MS鉴定,pFind 3搜库鉴定到4条Rv2742目的蛋白高可信肽段,分别为MSDNAIRP、PNPWQYIR、YGPRPDPND、VHNLDPELVDEHAR (图 7C)。计算得到Rv2742目的蛋白鉴定肽段序列覆盖度达29.63% (图 7B), 而Rv2742融合蛋白鉴定肽段序列覆盖度高达61.46%。说明经过宿主菌、低温、低浓度IPTG诱导条件优化后,纯化得到含量更高的Rv2742融合蛋白,为后续进一步的功能研究提供必要的条件。
 |
| 图 7 Rv2742目的融合蛋白纯化及质谱检测 Fig. 7 Purification and LC-MS/MS identification of Rv2742 fusion protein. (A) Purification of Rv2742 fusion protein. Lane M: protein marker; lane supernatant/+IPTG: supernatant of lysate of BL21(DE3)-pMAL-c2X-Rv2742-2 induced by 1 mmol/L IPTG; lane Flow through: supernatant after induction flow through the amylose resin column; lane Wash1, 2, 3: supernatant after induction washed by Wash Buffer; lane Elution1, 2: supernatant after induction eluted by Elution Buffer. (B) Sequence coverage of Rv2742 fusion protein (61.46%) by LC-MS/MS identification. (The single underlined sequences indicated the peptides were identified by LC-MS/MS). (C) The spectrum of Rv2742 target peptide"VHNLDPELVDEHAR". (D) SDS-PAGE analysis of purification of Rv2742 fusion protein in the host bacteria Rosetta (DE3). |
| 图选项 |
2.7 IPTG诱导表达Rv2742蛋白生长曲线的测定将重组质粒pMAL-c2X-Rv2742-2取0.5 μL转化至大肠杆菌Rosetta (DE3)中。如图 8所示,未加IPTG诱导组(-IPTG)在整个时间段内保持正常生长,经过对数期后达到平台期。而与对照组(-IPTG)相比,加入IPTG诱导(+IPTG)组的生长情况明显受到抑制,诱导后0–1.5 h范围内OD值只有少量增加且诱导3 h后菌体OD值基本维持稳定。由于IPTG是一种极强的诱导剂,不被细菌代谢因而十分稳定,在胞内诱导外源蛋白的表达,会对细胞产生一定的毒性。其次,加入IPTG后,大量外源蛋白的表达造成菌体原有代谢活动的失衡和重新分配。对于菌体内源蛋白来说,是一种对游离氨基酸以及核糖体的竞争和掠夺,这必然会影响内源蛋白的合成速度,导致菌体的生长受到明显抑制,生长速度降低。
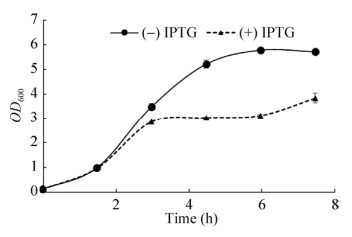 |
| 图 8 IPTG诱导表达Rv2742蛋白生长曲线的测定 Fig. 8 Determination of growth curve of expression of Rv2742 fusion protein induced by 0.5 mmol/L IPTG. |
| 图选项 |
3 讨论大肠杆菌是一种基因组G+C含量低(< 50%)的革兰氏阴性菌,而结核分枝杆菌(M. tuberculosis)是一种基因组G+C含量高(> 65%)的革兰氏阳性放线菌[28],放线菌基因组的典型特征就是基因组G+C含量高。结核分枝杆菌G_和C_结尾末端的密码子具有很强的偏倚性,第3个位点的密码子(G+C)含量高达83%[29]。密码子偏倚性影响翻译过程中氨基酸插入的准确性、多肽折叠和mRNA序列的稳定性等几方面[30]。结核分枝杆菌目的基因在大肠杆菌中不易表达可能主要是由于G+C含量高和独特密码子的使用[31]。生物体中基因所使用的密码子和tRNA的丰度有着强的正相关性。依照物种的偏好性对mRNA的序列进行优化,将目的基因的密码子替换成宿主细胞常用的密码子,能够增加tRNA的结合效率从而提高蛋白质表达水平。
本次实验研究的结核新基因Rv2742编码蛋白分子量本身不大,pET-32a和pET-28a虽然同属pET载体,标签大小对小蛋白的表达有一定程度的影响,所以在实验过程中考虑到使用不同种类和大小标签的pET载体。但仅在密码子优化后,利用pMAL-c2X原核表达系统才在上清和沉淀中都实现了表达,沉淀中表达含量要比可溶性部分高。后续实验需要进一步考虑对以包涵体形式表达的蛋白进行变性和复性。诱导后上清虽然实现了可溶性诱导表达,但表达含量较低,可从温度、IPTG浓度、抗生素浓度、诱导时间、宿主菌等多方面进行优化,从而提高其表达量。本次实验从宿主菌、温度、IPTG浓度等方面进行优化,在Rosetta (DE3)宿主菌中的表达量尤为显著。低温及低浓度诱导剂的条件有利于蛋白的可溶性表达。Rosetta (DE3)是携带氯霉素抗性质粒BL21的衍生菌,补充大肠杆菌缺乏的6种稀有密码子(AUA、AGG、AGA、CUA、CCC、GGA)对应的tRNA,提高外源基因在该系统中的表达水平,为其他结核遗漏注释基因的表达、纯化提供了较好的宿主选择性。
在进行原核表达时,目前多以融合蛋白形式表达,具有融合标签的表达载体可促进蛋白质可溶性表达和正确折叠恢复成天然结构。融合蛋白标签的选择很重要,源于其可能影响天然蛋白质的相互作用、翻译后修饰、重组蛋白的溶解度和细胞定位等[32],其应用取决于对特异性、溶解度、结合和洗脱条件等的要求[33]。添加非肽融合配体具有作为溶解度增强剂作用的优点[34]。最常用的融合标签有:麦芽糖结合蛋白(MBP)、转录终止/抗终止蛋白NusA)、硫氧还蛋白(Trx)、谷胱甘肽巯基转移酶(GST)、ubiquitin、SUMO等[35],但这些融合伴体作为溶解度增强剂起作用的原因尚不清楚。在MBP标签存在情况下,其具有内在的分子伴侣活性促进溶解影响其融合伴侣的折叠[36]。谷胱甘肽巯基转移酶(GST)可以结合到谷胱甘肽树脂上进行纯化,可以保护目的蛋白免受蛋白酶降解,增加蛋白的稳定性。但GST是一个很弱的溶解度增强剂[37],易产生包涵体。而NusA、MBP和Trx显示出较好的溶解度增强特性,但它们的大分子量可能对蛋白质溶解度造成一定的影响[38]。pET-32a含有trxA基因并携带有用于蛋白生产和纯化的双融合伴侣。TrxA是一种存在于大肠杆菌胞质中高度可溶性表达的细胞内热稳定性蛋白,能显著增加重组蛋白的可溶性,减少包涵体形成。但TrxA没有内在的亲和特性,因此在蛋白质纯化时需要附加额外的融合标签,如His6标签[39]。His标签在天然和变性的纯化条件下被用来帮助溶解和折叠[40]。
启动子在控制相关基因的转录起始中起重要作用。本实验研究中运用了4种原核表达系统均能被IPTG诱导,表达载体pGEX-4T-2和pMAL-c2X均带有强大的tac启动子。tac启动子由trp启动子-35区和lac UV5启动子-10区融合而成的杂合启动子,受lac阻抑物调控[41]。在pET系列表达载体中,外源基因在表达时受T7噬菌体RNA聚合酶调控,编码序列在多克隆位点插入,置于天然T7 RNA聚合酶启动子(φ10启动子)或所谓的“T7 lac启动子”的控制之下,后者是带有lac操纵子(lacO)序列的天然T7 RNA聚合酶启动子的衍生体,lac阻抑物的结合能阻断转录起始[42]。
对于特异性抗原的筛选,一直是结核分枝杆菌研究中的重点。基于串联质谱的蛋白质组学被应用于注释基因的鉴定、新基因的发现、个人基因组学和疾病相关研究[43]。本实验通过对结核分枝杆菌H37Rv新基因Rv2742进行克隆、诱导表达和纯化,纯化后的目的蛋白有可能直接作为抗原或制备抗体建立免疫学检测方法,为特异性诊断试剂盒和新型疫苗的研制奠定基础。H37Rv新基因Rv2742在结核三大数据库(UniProt、NCBI、TubercuList)中均没有记录,其功能目前尚未可知,但其表达丰度较高,只是基于成熟的预测模型没有预测出来。这种高表达漏注释基因产物可能在结核分枝杆菌中发挥了某些特殊的生物学功能,值得进一步探索。
目的基因密码子优化、可溶性融合标签筛选及宿主菌、温度、IPTG浓度等表达条件的优化是实现蛋白在大肠杆菌中可溶性诱导表达的有效策略[44]。
参考文献
| [1] | Rijo P, Sim?es MF, Francisco AP, et al. Antimycobacterial metabolites from Plectranthus: royleanone derivatives against Mycobacterium tuberculosis strains. Chem Biodivers, 2010, 7(4): 922-932. DOI:10.1002/cbdv.200900099 |
| [2] | Eddabra P, Benhassou HA. Rapid molecular assays for detection of tuberculosis. Pneumonia, 2018, 10(1): 4. DOI:10.1186/s41479-018-0049-2 |
| [3] | World Health Organization. Global tuberculosis report 2018. Geneva: World Health Organization, 2018. |
| [4] | Gupta S, Kakkar V. Recent technological advancements in tuberculosis diagnostics - a review. Biosens Bioelectron, 2018, 115: 14-29. DOI:10.1016/j.bios.2018.05.017 |
| [5] | Singhania A, Wilkinson WJ, Rodrigue M, et al. The value of transcriptomics in advancing knowledge of the immune response and diagnosis in tuberculosis. Nat Immunol, 2018, 19(11): 1159-1168. DOI:10.1038/s41590-018-0225-9 |
| [6] | Cohn DL, O'Brien RJ, Geiter LJ, et al. Targeted tuberculin testing and treatment of latent tuberculosis infection. MMWR Morb Mortal Wkly Rep, 2000, 49(6): 1-54. |
| [7] | Bosshard V, Roux-Lombard P, Perneger T, et al. Do results of the T-SPOT.TB interferon-γ release assay change after treatment of tuberculosis?. Respir Med, 2009, 103(1): 30-34. DOI:10.1016/j.rmed.2008.09.012 |
| [8] | Cole ST, Barrell BG. Analysis of the genome of Mycobacterium tuberculosis H37Rv. Novartis Found Symp, 1998, 217: 160-172. |
| [9] | Cole ST, Brosch R, Parkhill J, et al. Deciphering the biology of Mycobacterium tuberculosis from the complete genome sequence. Nature, 1998, 393(6685): 537-544. DOI:10.1038/31159 |
| [10] | Camus JC, Pryor MJ, Médigue C, et al. Re-annotation of the genome sequence of Mycobacterium tuberculosis H37Rv. Microbiology, 2002, 148(10): 2967-2973. DOI:10.1099/00221287-148-10-2967 |
| [11] | Brent MR. Steady progress and recent breakthroughs in the accuracy of automated genome annotation. Nat Rev Genet, 2008, 9(1): 62-73. DOI:10.1038/nrg2220 |
| [12] | Goymer P. Genomics: annotating with proteomes. Nat Rev Genet, 2008, 9(6): 418. |
| [13] | de Souza GA, M?len H, S?fteland T, et al. High accuracy mass spectrometry analysis as a tool to verify and improve gene annotation using Mycobacterium tuberculosis as an example. BMC Genomics, 2008, 9: 316. DOI:10.1186/1471-2164-9-316 |
| [14] | Rison SCG, Mattow J, Jungblut PR, et al. Experimental determination of translational starts using peptide mass mapping and tandem mass spectrometry within the proteome of Mycobacterium tuberculosis. Microbiology, 2007, 153(2): 521-528. DOI:10.1099/mic.0.2006/001537-0 |
| [15] | Schubert OT, Mouritsen J, Ludwig C, et al. The Mtb proteome library: a resource of assays to quantify the complete proteome of Mycobacterium tuberculosis. Cell Host Microbe, 2013, 13(5): 602-612. DOI:10.1016/j.chom.2013.04.008 |
| [16] | Castellana N, Bafna V. Proteogenomics to discover the full coding content of genomes: a computational perspective. J Proteomics, 2010, 73(11): 2124-2135. DOI:10.1016/j.jprot.2010.06.007 |
| [17] | Zhang CP, Xu P, Zhu YP. Progress in proteogenomics of prokaryotes. Chin J Biotech, 2014, 30(7): 1026-1035 (in Chinese). 张成普, 徐平, 朱云平. 原核生物蛋白质基因组学研究进展. 生物工程学报, 2014, 30(7): 1026-1035. |
| [18] | Aivaliotis M, Gevaert K, Falb M, et al. Large-scale identification of N-terminal peptides in the halophilic archaea Halobacterium salinarum and Natronomonas pharaonis. J Proteome Res, 2007, 6(6): 2195-2204. DOI:10.1021/pr0700347 |
| [19] | Renuse S, Chaerkady R, Pandey A. Proteogenomics. Proteomics, 2011, 11(4): 620-630. DOI:10.1002/pmic.201000615 |
| [20] | Kelkar DS, Kumar D, Kumar P, et al. Proteogenomic analysis of Mycobacterium tuberculosis by high resolution mass spectrometry. Mol Cell Proteomics, 2011, 10(12): M111.011627. |
| [21] | Domon B, Aebersold R. Mass spectrometry and protein analysis. Science, 2006, 312(5771): 212-217. DOI:10.1126/science.1124619 |
| [22] | Wu FL, Zhao MZ, Zhang Y, et al. Recombinant acetylated trypsin demonstrates superior stability and higher activity than commercial products in quantitative proteomics studies. Rapid Commun Mass Spectrom, 2016, 30(8): 1059-1066. DOI:10.1002/rcm.7535 |
| [23] | Zhao MZ, Wu FL, Xu P. Development of a rapid high-efficiency scalable process for acetylated Sus scrofa cationic trypsin production from Escherichia coli inclusion bodies. Protein Expr Purif, 2015, 116: 120-126. DOI:10.1016/j.pep.2015.08.025 |
| [24] | He CT, Jia CX, Zhang Y, et al. Enrichment-based proteogenomics identifies microproteins, missing proteins, and novel smORFs in Saccharomyces cerevisiae. J Proteome Res, 2018, 17(7): 2335-2344. DOI:10.1021/acs.jproteome.8b00032 |
| [25] | Chi H, Liu C, Yang H, et al. Comprehensive identification of peptides in tandem mass spectra using an efficient open search engine. Nat Biotechnol, 2018, 36(11): 1059-1061. DOI:10.1038/nbt.4236 |
| [26] | Sch?gger S. Tricine-SDS-PAGE. Nat Protac, 2006, 1(1): 16-22. DOI:10.1038/nprot.2006.4 |
| [27] | Luo WJ, Deng C, Li YC, et al. Soluble expression of Tandem Hybrid Ubiquitin-binding Domains (ThUBD) in prokaryotic cytoplasm of Escherichia coli BL21(DE3). Mil Med Sci, 2016, 40(10): 795-800 (in Chinese). 罗维佳, 邓晨, 李衍常, 等. 串联杂种泛素结合结构域蛋白(ThUBD)在大肠杆菌中的可溶性高效表达. 军事医学, 2016, 40(10): 795-800. |
| [28] | Piubelli L, Campa M, Temporini C, et al. Optimizing Escherichia coli as a protein expression platform to produce Mycobacterium tuberculosis immunogenic proteins. Microb Cell Fact, 2013, 12: 115. DOI:10.1186/1475-2859-12-115 |
| [29] | Andersson SGE, Sharp PM. Codon usage in the Mycobacterium tuberculosis complex. Microbiology, 1996, 142(4): 915-925. DOI:10.1099/00221287-142-4-915 |
| [30] | Ayyar BV, Arora S, Ravi SS. Optimizing antibody expression: the nuts and bolts. Methods, 2017, 116: 51-62. DOI:10.1016/j.ymeth.2017.01.009 |
| [31] | Kaur J, Kumar A, Kaur J. Strategies for optimization of heterologous protein expression in E. coli: roadblocks and reinforcements. Int J Biol Macromol, 2018, 106: 803-822. DOI:10.1016/j.ijbiomac.2017.08.080 |
| [32] | Papaneophytou CP, Kontopidis G. Statistical approaches to maximize recombinant protein expression in Escherichia coli: a general review. Protein Expr Purif, 2014, 94: 22-32. DOI:10.1016/j.pep.2013.10.016 |
| [33] | Zhao XY, Li GS, Liang SF. Several affinity tags commonly used in chromatographic purification. J Anal Methods Chem, 2013, 2013: 581093. |
| [34] | Hammarstr?m M, Hellgren N, van den Berg S, et al. Rapid screening for improved solubility of small human proteins produced as fusion proteins in Escherichia coli. Protein Sci, 2002, 11(2): 313-321. |
| [35] | Rosano GL, Ceccarelli EA. Recombinant protein expression in Escherichia coli: advances and challenges. Front Microbiol, 2014, 5: 172. |
| [36] | Kapust RB, Waugh DS. Escherichia coli maltose-binding protein is uncommonly effective at promoting the solubility of polypeptides to which it is fused. Protein Sci, 1999, 8(8): 1668-1674. DOI:10.1110/ps.8.8.1668 |
| [37] | Hammarstr?m M, Woestenenk EA, Hellgren N, et al. Effect of N-terminal solubility enhancing fusion proteins on yield of purified target protein. J Struct Funct Genomics, 2006, 7(1): 1-14. DOI:10.1007/s10969-005-9003-7 |
| [38] | Costa SJ, Almeida A, Castro A, et al. The novel Fh8 and H fusion partners for soluble protein expression in Escherichia coli: a comparison with the traditional gene fusion technology. Applied microbiology and biotechnology, 2013, 97(15): 6779-6791. DOI:10.1007/s00253-012-4559-1 |
| [39] | Kosobokova EN, Skrypnik KA, Kosorukov VS. Overview of fusion tags for recombinant proteins. Biochemistry (Moscow), 2016, 81(3): 187-200. DOI:10.1134/S0006297916030019 |
| [40] | Arnau J, Lauritzen C, Petersen GE, et al. Current strategies for the use of affinity tags and tag removal for the purification of recombinant proteins. Protein Expr Purif, 2006, 48(1): 1-13. DOI:10.1016/j.pep.2005.12.002 |
| [41] | Amann E, Brosius J, Ptashne M. Vectors bearing a hybrid trp-lac promoter useful for regulated expression of cloned genes in Escherichia coli. Gene, 1983, 25(2/3): 167-178. |
| [42] | Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual. 3rd ed. New York: Cold Spring Harbor Laboratory Press, 2001: 1218-1219. |
| [43] | Zhang K, Fu Y, Zeng WF, et al. A note on the false discovery rate of novel peptides in proteogenomics. Bioinformatics, 2015, 31(20): 3249-3253. DOI:10.1093/bioinformatics/btv340 |
| [44] | Itkonen JM, Urtti A, Bird LE, et al. Codon optimization and factorial screening for enhanced soluble expression of human ciliary neurotrophic factor in Escherichia coli. BMC Biotechnol, 2014, 14: 92. DOI:10.1186/s12896-014-0092-x |
