

山东大学 生命科学学院 微生物技术国家重点实验室,山东 济南 250100
收稿日期:2017-12-21;接收日期:2018-02-12; 网络出版时间:2018-05-23 基金项目:国家自然科学基金(No. 31370111),山东省自然科学基金(No. ZR2013CM038),山东大学基本科研业务费专项资金(No. 2015YQ004)资助
摘要:酶分子的高效性和稳定性是工业广泛应用的物质基础。利用分子生物学技术可以将不同酶分子通过串联、插入、翻译后融合等方式构建成符合工业需求的杂合酶,但应用中多结构域杂合酶在表达量与酶活等方面仍存在弊端,而基于特定蛋白质结构域的多功能设计成为新趋势。高通量测序技术的发展,使得生物学家正面临着爆炸式增长的大数据集。近年来“蛋白质功能区”概念的提出,拓宽了人们对蛋白质结构与功能组织层次的认知,功能区残基聚簇的协同演化可导致同一家族不同蛋白质功能的差异。基于海量大数据分析可以快速定位特定功能区以及协同进化的关键位点,再利用合成生物学技术就可实现多种功能残基在同一蛋白质中的精准嫁接,完成天然酶分子的再设计。这将是杂合酶技术发展的新阶段,也会成为生物大数据时代下蛋白质设计的新趋势。
关键词:杂合酶 功能区 生物大数据 合成生物学技术 蛋白质工程
Trend of hybrid enzyme design in the big data era
Qun Zhang, Xiuyun Wu, Xukai Jiang, Yue Zhao, Lushan Wang


The State Key Laboratory of Microbial Technology, College of Life Sciences, Shandong University, Jinan 250100, Shandong, China
Received: December 21, 2017; Accepted: February 12, 2018; Published: May 23, 2018
Supported by: National Natural Science Foundation of China (No. 31370111), Shandong Provincial Natural Science Foundation (No.ZR2013CM038), Shandong University Basic Research Business Special Funds (No. 2015YQ004)
Corresponding author:Lushan Wang. Tel: +86-531-88366202; E-mail: lswang@sdu.edu.cn
Abstract: The high efficiency and stability of enzymes are the basis for industrial application. Hybrid enzyme suitable for industrial applications could be constructed by many molecular biology technologies including tandem fusion, domain insertion and post-translational protein conjugation. However, the low expression and activity of hybrid enzyme limit its application in industrial production, and multifunctional design of a specific protein domain has been becoming a new trend. With the advent of high-throughput sequencing, biologists are starting to wrestling with massive data sets. Besides, the concept of protein sectors and co-evolution provides novel insight into the relationship of protein structure and function. The residues-covariation of a protein sector displays preference, which imparts functional diversity to different enzymes in the same family. The covariation-residues in specific protein sectors can be located based on the analysis of massive data, and then these functional residues can be assembled in a new enzyme variant using the biotechnology of synthetic biology, thus completing the redesign of natural enzymes. This indicates a new stage of designing hybrid enzyme, as well as the new trend of protein design in the era of biological big data.
Key words: hybrid enzyme sectors biological big data synthetic biology protein engineering
酶制剂是工业生物技术快速发展的基石,早在20世纪初期,生物催化在工业生物技术中的应用已初见端倪[1]。由于工业应用过程中酶分子的分离纯化、稳定性等因素影响,行业应用成本较高,因而推广应用范围有限。直至20世纪90年代随着分子生物学技术的快速发展,以生物催化为核心的工业生物技术才取得了革命性的飞跃。然而酶制剂高效性与稳定性仍是行业应用的限制瓶颈,因而设计或改造形成高效稳定的酶制剂仍是工业生物技术时代需要攻克的关键科学问题[2-3]。
1 功能酶分子的筛选与蛋白质工程工业生物技术作为21世纪新技术之一,对解决人类所面临的食品、资源和环境等问题起到越来越重要的作用。获得耐高温、高盐、酸碱的优良酶制剂是工业生物技术的关键,人们常通过极端微生物或宏基因组学筛选获得相关酶类(图 1A)。尽管目前从自然界筛选了大量新型酶分子,但仍不能满足工业生产与应用中的迫切需求[4]。为了克服天然酶分子在工业应用中的固有缺陷,1983年Ulmer提出了蛋白质工程(Protein engineering)的概念,即利用分子生物学技术进行蛋白质的工程化改造[5]。蛋白质工程可设计出符合工业应用的酶分子,在一定程度上加速酶分子的进化历程[6]。自提出以来,该技术先后经过了定向进化、半理性设计以及理性设计3个发展阶段(图 1B-D)。
 |
| 图 1 酶分子的筛选和改造策略[13] Figure 1 Screening and design strategies of enzymes[13]. (A) High-throughput screening. (B) Directed evolution. (C) Semi-rational design. (D) Rational design. |
| 图选项 |
定向进化模拟自然进化过程,无需了解蛋白质的三维结构信息和作用机制,通过随机突变和定向筛选可获得功能改善的酶分子。由于蛋白质序列空间(Sequence space)巨大,因而建立灵敏的高通量筛选方法是决定该策略成功与否的前提[7-8]。半理性设计可将功能变化定位至一个或几个“热点”残基的突变,进而改变酶分子的相应功能[9]。然而从功能保守的蛋白质家族(一般序列一致性大于30%就可以归为一个蛋白质家族)来看,长期的演化过程导致序列间相似性相对较低,但酶分子功能并没有发生明显改变,这说明半理性设计在实际应用中存在低效性问题[10-11]。伴随新一代测序技术,生物大分子序列和结构信息的快速增长,相关数据库也愈发丰富和完善,建立全新的大数据分析与挖掘技术,可指导蛋白质工程走向更加理性的新阶段[12]。
2 杂合酶及其构建的分子生物学技术根据Nixon所述,杂合酶(Hybrid enzyme)是指将两个或多个不同酶分子的结构元件或者功能结构域整合到同一个分子中的新技术,其可以基于工业的需求而设计相应酶类,因而得到工业生物技术行业的高度关注[14]。随着早期分子生物学技术的发展,人们可以利用重组DNA技术或者翻译后修饰方法构建具有应用价值的杂合酶分子,这包括基因水平的杂合与蛋白水平的直接融合等(图 2),早期构建方式主要分为功能结构域的串联融合、插入融合和翻译后融合等形式[15]。
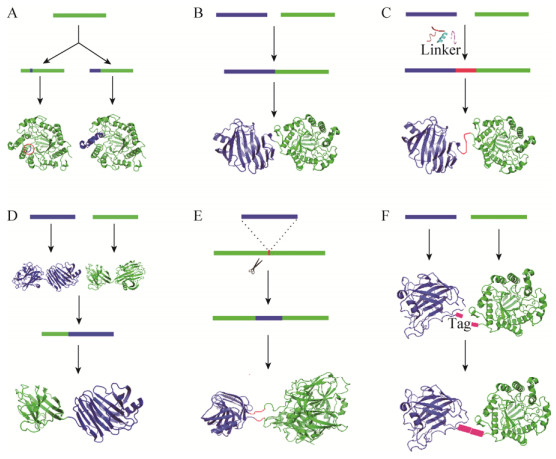 |
| 图 2 构建杂合酶的传统方法[15] Figure 2 Traditional methods of constructing hybrid enzymes[15]. (A) Site and secondary structure mutations. (B) Direct tandem fusion. (C) Indirect tandem fusion. (D) Domain splicing. (E) Insertion fusion. (F) Post-translational fusion. |
| 图选项 |
由于结构域是蛋白质进化的功能单元,多结构域的杂合酶往往比单结构域的酶分子具有更稳定的性质和更多样的功能,因而有利于降低酶制剂的生产成本,简化生产工艺。例如Ye等构建了肝素酶、麦芽糖结合蛋白(MBP)以及荧光蛋白的三重杂合体,基于荧光蛋白的快速追踪以及MBP易于分离的性质,实现了肝素酶的生产、分离以及催化等过程的集成,从而降低了酶制剂的应用成本和低分子量肝素的生产成本[16]。此外,杂合酶可应用于代谢过程中的顺序反应,将不同功能结构域的酶分子杂合在一起,形成临近效应,从而明显提升酶分子的催化效率[17]。
2.1 功能结构域的串联融合杂合酶的串联融合是指将两个或多个不同酶分子连接在一起的融合方式,选择理化性质(最适温度、pH等)兼容的功能结构域可以明显提升杂合酶构建的成功率,而结构域的融合次序也是杂合酶可否构建成功的关键因素[18]。串联融合技术通常可以直接串联融合,即一个酶的N端与另一酶的C端直接融合。Lu等将一个来自解淀粉芽孢杆菌的葡聚糖酶与来自枯草芽孢杆菌的木聚糖酶直接融合,所得杂合酶的葡聚糖酶活提高了3倍,而木聚糖酶活降低了31%[19]。当所融合的N端或C端具有重要功能,或者各结构域距离太近而不能避免空间位阻时,会导致新的杂合酶分子发生错误折叠、表达量偏低或者活性受损等问题[20-21]。解决此类难题的有效方式是引入连接肽(Linker),其可以维持单个结构域的构象延伸性和结构稳定性,改善蛋白质表达水平和生物活性[22]。
如何选择或设计连接肽是一个值得深入研究的领域。研究者已经设计出多种人工连接肽用于杂合酶分子的构建,现将其分成3种类型:柔性型、刚性型和可断裂型,表 1总结了各种类型连接肽的特征与功能。目前已经有很多连接肽实现了应用,如Kim等利用柔性型连接肽(GGGGS)2 实现了纤维素酶Cel5B和木聚糖酶Xyl10g的融合,所得杂合酶可更有效地降解稻草、秸秆等生物质(图 3A)[23]。Chen等利用二硫苏糖醇(DTT)可对二硫键的还原断裂能力,从而设计了一种可断裂连接肽,实现了粒细胞集落刺激因子(G-CSF)与转铁蛋白(Tf)的体内激活(图 3B)[24]。
表 1 各种类型连接肽的汇总Table 1 Summary of all kinds of linkers
| Types | Illustration | Function and characteristics | Examples | References |
| Flexible |  | Maintain flexibility and increase spatial distance; rich in Gly, Ser and other small molecule amino acids | (G)n (GGGGS)n | [23, 25-26] |
| Rigid |  | Maintain distance and functions between domains; α helix/rich in Pro | (EAAAK)n (XP)n | [27-30] |
| Cleavable |  | Release free functional domains in vivo; reducing/ enzymatic hydrolysis | Disulfide bond/ protease target | [24, 31] |
| This table is redrawn from Chen et al[22]. | ||||
表选项
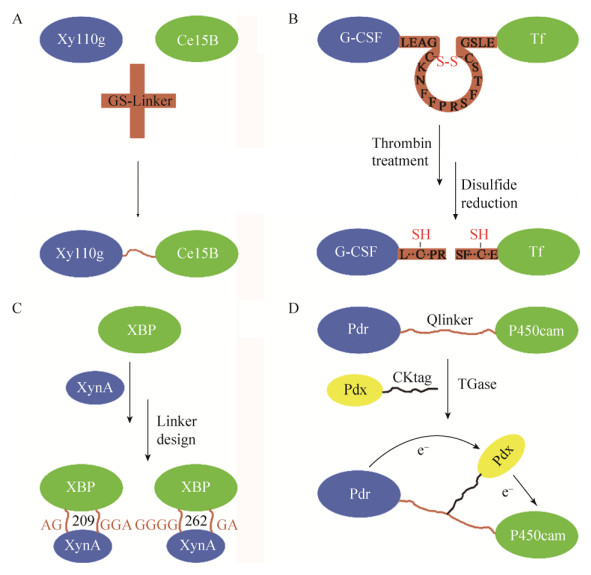 |
| 图 3 构建杂合酶的成功案例[15, 23, 39, 43] Figure 3 Successful cases of constructing hybrid enzymes[15, 23, 39, 43]. (A) The fusion of cellulase Cel5B and xylanase Xyl10g constructed by the (GGGGS)2 linker. (B) An in vivo cleavable linker utilizing the reversible nature of disulfide bond as well as a thrombin-sensitive sequence, which release free functional domains granulocyte-colony stimulating factor (G-CSF) and transferrin (Tf). (C) The xylanase XynA was inserted into xylose binding protein XBP, and by designing the linker, two hybrid enzymes with 20% increase in activity were constructed. (D) A site-specific branched fusion protein of P450 with its electron transfer proteins using enzymatic cross-linking with TGase. |
| 图选项 |
2.2 功能结构域的插入融合多功能酶分子常将不同功能的结构域按一定次序排列在同一多肽链上,然而自然界中仍有9%的多功能酶分子存在插入结构域现象,即一个结构域插入另一个结构域之中[32]。因此,将一个功能结构域插入到宿主结构域来构建杂合酶也是一种可行策略,它通常可形成抗蛋白酶水解的刚性结构[33-34]。
插入融合技术需要两个先决条件。首先,为了降低插入融合对插入结构域柔性构象的破坏,插入结构域最末端两残基的Cα原子距离要在5 ?左右,且大小要小于宿主结构域[35];其次,该类宿主结构域可适应新结构域的插入,即两结构域可互相兼容[36]。PDB数据库中约50%的蛋白质含有临近的末端,满足插入结构域的条件[37]。然而宿主结构域不易寻找到插入位点,这使得该策略要比串联融合操作过程繁琐。人们常选择宿主结构域表面的loop或转角位置作为结构域插入位点,这可消除结构域间位阻冲突,但可能会破坏宿主结构域原有的相互作用网络,使得杂合酶结构稳定性降低[15, 38]。目前,插入融合技术已经有多个成功案例,如Ribeiro等将木聚糖酶XynA插入到大肠杆菌木糖结合蛋白XBP中,筛选到两种活性都提高20%的杂合酶(图 3C)[39]。
2.3 功能结构域的翻译后融合多结构域的杂合酶在异源表达过程中常形成包涵体,导致杂合酶表达量偏低或不能正确折叠,这限制了杂合酶技术的广泛应用。因此人们又开发了翻译后蛋白质水平上的结构域融合技术[40]。翻译后蛋白质水平的融合技术主要包括化学交联法和酶联法。化学交联法可高效构建杂合酶分子,但化学试剂不具有特异性,常常会导致杂合酶分子发生不正确聚集[41]。酶联法是构建杂合酶更温和更常用的方法,该技术主要用转肽酶、谷氨酰胺转胺酶(TGase)以及过氧化物酶等完成相关蛋白质结构域的杂合[42]。Hirakawa等利用TGase构建了一个单加氧酶细胞色素P450cam、假单孢氧还蛋白Pdx以及Pdx还原酶Pdr的三元杂合酶,实现了分子内电子的快速转移,使得电子传递效率与催化效率较3种游离蛋白质混合物有了明显提升(图 3D)[43]。
3 生物大数据时代杂合酶的分子设计3.1 高通量测序技术与生物大数据时代进入新世纪,新一代高通量测序技术快速发展,测序成本急剧降低,使得高通量测序成为通用技术;而Miseq等测序技术测序通量可高达1 Gb/run,这就使得生物序列数据爆炸式增长;截止到2017年9月,NCBI数据库中核酸序列已有约2.45亿条[46-47]。蛋白质序列由基因序列数据翻译而来,去除冗余序列后,Uniprot数据库中蛋白质序列至2017年9月已有9 000万余条,但转录水平证实的只占1.22%,蛋白质水平证实更低,仅有0.15%[48]。同时,近几年冷冻电镜等结构测定技术的快速发展也使得生物大分子的结构信息不断增多,2016年2月23日PDB数据库就已实现10亿原子的存档[49]。因此现代生物学已经进入到大数据时代[50],现在生物工作者所面临的主要科学问题可能不再是序列、结构数据太少,而是如何对生物大数据进行深入分析与挖掘、促进蛋白质功能的准确预测与分析、以及应用于酶分子理性设计与改造等方面[51]。
不同来源的生物大数据已按一定格式与要求存储于相关生物信息学数据库中,由于实验通量限制人们不可能逐条分析验证其生物学功能,因此必须建立相关算法及不同数据间的关联,完成其功能准确有效的注释[52]。早在人类基因组草图完成时,有人就利用同源性方法来预测基因及其功能,提出以蛋白序列一致性30%为标准构建蛋白质家族(Protein family),利用同源模建方法来构建结构并分析预测功能[53]。至2017年3月蛋白质家族的个数为1.6万左右(http://pfam.xfam.org/help)[54-55],PDB数据库中生物大分子结构的数目也已经超过13万个[56],现在每一蛋白质家族基本都含有一个已测定结构的蛋白质。由于同一家族具有相似的空间拓扑结构,而不同蛋白质处于不同的选择压力之下,因而具有不同的进化速率[57-58],所以分析同一家族内特定结构空间的约束与局部功能区的快速演化就可完成其功能的有效预测与分析,这就是“结构生物信息学(Structrual bioinformatics)”的研究策略[59]。
3.2 结构生物信息学大数据的聚类分析与杂合酶的设计由于海量生物大数据的持续增长,人们提出要根据序列相似性程度对蛋白质家族进行进一步分析与更深层次的聚类,以对应酶分子功能分类的不同层次,从而提高序列功能预测的准确度[60]。现在CATH等蛋白质结构分类数据库已经根据不同的序列一致性细化出不同的层次聚类(序列一致性 < 35%为S层,< 60%为O层,< 95%为L层,100%为I层)[61];CAZy数据库也基于序列一致性 > 75%的标准提出了GH5家族新的“亚家族(Subfamily)”分类系统,这为糖苷水解酶等进一步功能分析预测以及功能基元(Motif)的确定奠定了基础[62]。生物大数据的细化分类进一步提升了序列结构分析的效率与准确率[63],特别是基于亚家族等近源序列、结构与功能的分析与其分子动力学模拟,可明显降低序列空间的搜索范围与搜索强度[64-65]。
通过对同一聚类的蛋白质家族或亚家族等成员进行多结构比对分析等,快速定位催化活性中心的活性架构(Active archtechture)[64];再对其进行比较分子动力学模拟(MDs)分析,认识酶分子特定功能簇的分子动态行为,定位功能簇内残基的相互作用网络及其动态学变化,分析如酶蛋白热敏感区等的分子动态性质,从而可确定热稳定性等相关基元并进行精准嫁接[66-67](图 4A)。如Jiang等利用比较分子动力学模拟方法对糖苷水解酶GH12家族5种酶分子的分子动态行为进行系统研究,发现该家族的热敏感区主要位于蛋白质结构的N末端,并且同一家族不同热稳定性的酶分子间存在明显规律性变化,这也使得在GH12家族内不同酶分子之间进行热敏感区的嫁接成为可能[66]。GH12家族结构除N端区域对热敏感外,其催化活性架构的多个loop也是热敏感区,且不同成员对热扰动的抵抗能力也是不同的,嗜热酶的活性架构loop与周围残基倾向于形成更多的相互作用网络[68]。而基于同一蛋白质家族或亚家族成员的比较分析,可以针对特定蛋白质进行蛋白质表面嫁接(Protein surface grafting)等改造,从而完成生物大分子的理性设计。如Kapoor等将里氏木霉的中温纤维素酶(TrCel12A)的活性架构残基嫁接到海洋红嗜热盐菌的嗜热纤维素酶(RmCel12A)的活性架构中,成功地获得了一个适中温活性的热稳定性杂合酶[69]。
 |
| 图 4 生物大数据时代杂合酶的设计策略[86] Figure 4 Hybrid enzyme design scheme in the era of biological big data[86]. (A) Graft of thermo-sensitive sectors and active architecture. (B) Accurate grafting of surface charged residues. (C) The new design strategy for hybrid enzyme in the era of big data. |
| 图选项 |
除热稳定性外,耐盐性和pH耐受性也是限制酶制剂应用的重要因素。通过对蛋白质家族内不同酶分子表面带电残基的系统分析,发现其与耐盐性和pH耐受性密切相关,这不仅涉及分子内残基的相互作用,而且与溶剂分子也存在重要作用[70-71]。已有研究发现耐盐酶结构中α螺旋较少,无规卷曲偏多,而且分子表面分布有较多的负电残基(D/E)和小侧链的疏水残基(如G/A)[72-73]。由于分子表面较多D/E的存在,耐盐酶分子对酸的耐受性明显增强[74]。耐碱酶与耐酸酶明显不同,其分子表面的D/E含量在进化中逐渐降低,取而代之的是较多的正电残基(如R)和中性亲水残基(如N/Q)[75]。人们通过在GH11家族木聚糖酶XynJ的表面引入多个R残基,成功提高了其耐碱性[76]。目前,通过结构生物信息学分析功能残基突变前后的分子表面电势(VEs)变化,可以指导表面带电氨基酸残基的精准嫁接,这已成为构建稳定型杂合酶的全新策略[77](图 4B)。最近研究已有改造成功的案例,如Kiss等利用结构生物信息学方法,将牛碳酸酐酶Ⅱ表面的K、L、R、N、Q、V等残基突变为D、E,通过DNA合成技术构建了4个杂合酶,并成功将其改造为耐盐酶[78]。Woodiey等将植酸酶表面的R、K、Q等残基突变为D,使其在pH 2.8下的稳定性提高了3.8倍[77]。
3.3 蛋白质功能区的精准嫁接与酶分子的再设计蛋白质功能的执行常常是其空间结构的一部分,由功能残基聚簇(Clusters of residues)形成特定的局部结构来行使特定功能。然而蛋白质序列空间巨大,功能残基聚簇的寻找无疑是大海捞针。由于功能基因在天然环境中经亿万年进化筛选,形成了具有功能的天然多样性(Natural diversity)文库,这是经自然选择后的文库,较定向进化产生的文库要小的多[79]。因此基于高通量测序产生的天然多样性文库,研究特定功能残基聚簇的形成途径与策略将会找出“驯化突变(Educated mutations)”,并利用这些突变再赋予天然活性位点以新功能[80]。特别是对多功能的蛋白质家族,就可作为酶分子再设计(Enzyme redesign)的重要起点。通过蛋白质家族序列大数据的深入挖掘与分析,可快速定位相关残基的功能聚簇并认识其演化规律。如Reyndds等2009年提出蛋白质功能区(Sectors)的相关算法[81],该算法通过蛋白质家族内上千条序列的统计分析,定位局部空间聚簇的多个功能区[82-84]。此外,研究发现功能区在整个蛋白质家族中呈现明显分化现象,可导致同一家族成员功能上的显著差异[81]。
在序列大数据快速增长的背景下,Hopf于2012年提出了一种可全面分析单基因突变的统计耦合分析(Statistical coupling analysis,SCA)方法。通过该方法可在同源序列分析相关聚簇氨基酸残基间的协同进化,从而探究功能区内氨基酸之间的关联与其功能的关系。已有研究表明蛋白质中大部分残基几乎都是独立演化的,而约有20%的氨基酸可与协同进化的氨基酸残基形成连续相互作用的功能区[85]。由于序列大数据中包含丰富的蛋白质功能约束及其进化信息,因而可有效分析蛋白质功能区残基间的协同进化,可以帮助确定参与配体结合的功能区、蛋白质复合体形成功能区以及参与构象调控的相关功能残基及其组合,这对蛋白质结构、分子相互作用以及进化动力学等研究都具有重要意义[86-87]。目前已研究证实,以序列/结构大数据为研究驱动,不仅可估计三维结构中联系紧密的残基及其组合[88-90],还可将蛋白质折叠预测提高至合理精确度[91-93]。此外,蛋白质功能区氨基酸协同进化的相关分析平台与网站现已建立并可提供在线服务,如Evfold (http://evfold.org/)等,该网站采用最大熵法在线计算蛋白质家族中协同进化的相关氨基酸残基与组合[94]。
伴随大数据时代序列数据挖掘新技术的不断出现,功能区以及协同进化的相关分析算法将进一步简化,对家族内序列数目的要求也进一步降低,这将使得功能区的设计与其嫁接更加快捷与便利[95]。如SCHEMA等针对蛋白质空间结构优化算法的推出,已成功应用于杂合酶稳定性的设计。基于该算法人们可在不影响蛋白质三维结构稳定的前提下,定位杂合体中可能被破坏的相互作用区域,从而筛选出受稳定性影响最小的杂合酶[96]。这种以结构为基础的重组方案要比基于序列的DNA改组(DNA shuffling)更加高效,可有效避免基因重组过程中的结构域坍塌[97]。此外,DNA合成技术迅速崛起,合成速度、精度、长度的提高以及合成成本的大幅度降低,使其逐渐取代了基因组DNA的提取与克隆等分子生物学技术,这促使蛋白质工程发展到了全新的阶段[7, 98]。
因此,基于序列结构等生物大数据的挖掘与分析,结合动力学模拟分析蛋白质整体与局域分子动态学行为,通过进化分析准确定位家族或亚家族内协同进化的功能残基聚簇及组合,构建“小而精(Small but smart) ”的杂合体文库完成酶分子功能的精准嫁接,这将会明显提高杂合酶分子设计与改造的成功概率(图 4C),从而促进酶分子功能再设计的快速发展[7]。
4 展望工业生物技术的迅猛发展为人类解决能源和环境等问题提供了前所未有的机遇,然而现代化工业生物技术是一个全新的应用环境,所要求的条件明显不同于自然环境,因此对酶制剂提出了更加严苛的要求。尽管近年来人们在构建多结构域的杂合酶中取得了显著成效,但实际应用中多结构域杂合酶常得不到理想的表达量及相关活性,这一定程度上阻碍了杂合酶的工业应用[99-100]。所以为了满足工业生物技术快速发展的迫切需求,就需要对所筛选的酶分子进行系统的再设计与改造。伴随后基因组时代的到来,测序通量迅速增加,结构数据急剧膨胀,如何对生物大数据进行深入挖掘与分析,为酶分子理性设计提供全新思路与平台技术是当下生命科学亟待解决的关键问题。
生物大数据是连接人、机、物的三元纽带,为生命科学在基因和蛋白质层面的研究起到了关键的推进作用[101]。其在为生命科学研究提供新方法论的同时,也不可避免地将我们置身于新的挑战中。如:如何提高生物大数据分析的速度与准确度[102];如何确保软件分析界面的可视化、易用性与普适性,如何有效建立不同大数据之间的关联等[103]。不过,现在生物大数据挖掘的新方法新技术已经开始冲击传统的思维模式,其不断的发展与提升将会促进人们思维模式由“假设驱动”向“数据驱动”转变,因此将会给蛋白质工程带来新技术与新模式。
基于海量生物大数据的系统分析,提出全新的算法与分析平台,提升大数据挖掘的速率、效率,构建友好的人机对话界面,简便快捷地进行生物大分子的序列、结构、动态及功能的系统分析,快速定位结构空间中的相关功能区及其协同进化的功能残基聚簇,指导酶分子的精准嫁接,完成大数据驱动下酶分子的再设计,这将是杂合酶设计的新阶段,也将是蛋白质工程的新趋势。
参考文献
| [1] | Griengl H, Schwab H, Fechter M. The synthesis of chiral cyanohydrins by oxynitrilases.Trends Biotechnol, 2000, 18(6): 252–256.DOI: 10.1016/S0167-7799(00)01452-9 |
| [2] | Finkelstein JM. Enzyme engineering:Just a jump to the left.Nat Chem Biol, 2012, 8(2): 134. |
| [3] | Lorenz P, Eck J. Metagenomics and industrial applications.Nat Rev Microbiol, 2005, 3(6): 510–516.DOI: 10.1038/nrmicro1161 |
| [4] | Lutz S. Reengineering enzymes.Science, 2010, 329(5989): 285–287.DOI: 10.1126/science.1192224 |
| [5] | Ulmer KM. Protein engineering.Science, 1983, 219(4585): 666–671.DOI: 10.1126/science.6572017 |
| [6] | Poglitsch A, Waelkens C, Geis N, et al. The photodetector array camera and spectrometer (PACS) on the Herschel space observatory.Astr Astrophys, 2010, 518: 662–673. |
| [7] | Bornscheuer UT, Huisman GW, Kazlauskas RJ, et al. Engineering the thirdwave of biocatalysis.Nature, 2012, 485(7397): 185–194.DOI: 10.1038/nature11117 |
| [8] | Qu G, Zhao J, Zheng P, et al. Recent advances in directed evolution.Chin J Biotech, 2018, 34(1): 1–11.(in Chinese). 曲戈, 赵晶, 郑平, 等. 定向进化技术的最新进展.生物工程学报, 2018, 34(1): 1-11.DOI:10.25165/j.ijabe.20181101.3210 |
| [9] | Lutz S. Beyond directed evolution-semi-rational protein engineering and design.Curr Opin Biotechnol, 2010, 21(6): 734–743.DOI: 10.1016/j.copbio.2010.08.011 |
| [10] | Gerlt JA, Babbitt PC. Enzyme (re)design:lessons from natural evolution and computation.Curr Opin Chem Biol, 2009, 13(1): 10–18.DOI: 10.1016/j.cbpa.2009.01.014 |
| [11] | Chen RD. Enzyme engineering:rational redesign versus directed evolution.Trends Biotechnol, 2001, 19(1): 13–14.DOI: 10.1016/S0167-7799(00)01522-5 |
| [12] | Lichtarge O, Wilkins A. Evolution:a guide to perturb protein function and networks.Curr Opin Struct Biol, 2010, 20(3): 351–359.DOI: 10.1016/j.sbi.2010.04.002 |
| [13] | Davids T, Schmidt M, B ttcher D, et al. Strategies for the discovery and engineering of enzymes for biocatalysis.Curr Opin Chem Biol, 2013, 17(2): 215–220.DOI: 10.1016/j.cbpa.2013.02.022 |
| [14] | Nixon AE, Ostermeier M, Benkovic SJ. Hybrid enzymes:manipulating enzyme design.Trends Biotechnol, 1998, 16(6): 258–264.DOI: 10.1016/S0167-7799(98)01204-9 |
| [15] | Yu K, Liu CC, Kim BG, et al. Synthetic fusion protein design and applications.Biotechnol Adv, 2015, 33(1): 155–164.DOI: 10.1016/j.biotechadv.2014.11.005 |
| [16] | Ye FC, Zhang C, Togo M, et al. Design of intelligent heparinase by fusion with maltose binding protein and fluorescent proteins.J Biosci Bioeng, 2009, 108(S1): S104. |
| [17] | Good MC, Zalatan JG, Lim WA. Scaffold proteins:hubs for controlling the flow of cellular information.Science, 2011, 332(6030): 680–686.DOI: 10.1126/science.1198701 |
| [18] | Rizk M, Antranikian G, Elleuche S. End-to-end gene fusions and their impact on the production of multifunctional biomass degrading enzymes.Biochem Biophys Res Commun, 2012, 428(1): 1–5.DOI: 10.1016/j.bbrc.2012.09.142 |
| [19] | Lu P, Feng MG, Li WF, et al. Construction and characterization of a bifunctional fusion enzyme of Bacillus-sourced β-glucanase and xylanase expressed in Escherichia coli.FEMS Microbiol Lett, 2006, 261(2): 224–230.DOI: 10.1111/fml.2006.261.issue-2 |
| [20] | Shamriz S, Ofoghi H, Moazami N. Effect of linker length and residues on the structure and stability of a fusion protein with malaria vaccine application.Computers Biol Med, 2016, 76: 24–29.DOI: 10.1016/j.compbiomed.2016.06.015 |
| [21] | Klein JS, Jiang SD, Galimidi RP, et al. Design and characterization of structured protein linkers with differing flexibilities.Prot Eng Des Select, 2014, 27(10): 325–330.DOI: 10.1093/protein/gzu043 |
| [22] | Chen XY, Zaro JL, Shen WC. Fusion protein linkers:property, design and functionality.Adv Drug Del Rev, 2013, 65(10): 1357–1369.DOI: 10.1016/j.addr.2012.09.039 |
| [23] | Kim HM, Jung S, Lee KH, et al. Improving lignocellulose degradation using xylanase-cellulase fusion protein with a glycine-serine linker.Int J Biol Macromol, 2015, 73: 215–221.DOI: 10.1016/j.ijbiomac.2014.11.025 |
| [24] | Chen XY, Bai Y, Zaro JL, et al. Design of an in vivo cleavable disulfide linker in recombinant fusion proteins.BioTechniques, 2010, 49(1): 513–518.DOI: 10.2144/000113450 |
| [25] | De Bold MK, Sheffield WP, Martinuk A, et al. Characterization of a long-acting recombinant human serum albumin-atrial natriuretic factor (ANF) expressed in Pichia pastoris.Regul Pept, 2012, 175(1/3): 7–10. |
| [26] | Rosenblum MG, Cheung LH, Liu Y, et al. Design, expression, purification, and characterization, in vitro and in vivo, of an antimelanoma single-chain Fv antibody fused to the toxin gelonin.Cancer Res, 2003, 63(14): 3995–4002. |
| [27] | Huang ZL, Li G, Zhang C, et al. A study on the effects of linker flexibility on acid phosphatase PhoC-GFP fusion protein using a novel linker library.Enzyme Microb Technol, 2016, 83: 1–6.DOI: 10.1016/j.enzmictec.2015.11.002 |
| [28] | McCormick AL, Thomas MS, Heath AW. Immunization with an interferon-γ-gp120 fusion protein induces enhanced immune responses to human immunodeficiency virus gp120.J Infect Dis, 2001, 184(11): 1423–1430.DOI: 10.1086/jid.2001.184.issue-11 |
| [29] | Huang ZL, Ye FC, Zhang C, et al. Rational design of a tripartite fusion protein of heparinase I enables one-step affinity purification and real-time activity detection.J Biotechnol, 2013, 163(1): 30–37.DOI: 10.1016/j.jbiotec.2012.09.016 |
| [30] | Gramlich PA, Westbroek W, Feldman RA, et al. A peptide-linked recombinant glucocerebrosidase for targeted neuronal delivery:design, production, and assessment.J Biotechnol, 2016, 221: 1–12.DOI: 10.1016/j.jbiotec.2016.01.015 |
| [31] | Zhao HL, Xue C, Du JL, et al. Balancing the pharmacokinetics and pharmacodynamics of interferon-α2b and human serum albumin fusion protein by proteolytic or reductive cleavage increases its in vivo therapeutic efficacy.Mol Pharmaceutics, 2012, 9(3): 664–670.DOI: 10.1021/mp200347q |
| [32] | Pandya C, Brown S, Pieper U, et al. Consequences of domain insertion on sequence-structure divergence in a superfold.Proc Natl Acad Sci USA, 2013, 110(36): 3381–3387.DOI: 10.1073/pnas.1305519110 |
| [33] | Pierre B, Xiong TN, Hayles L, et al. Stability of a guest protein depends on stability of a host protein in insertional fusion.Biotechnol Bioeng, 2011, 108(5): 1011–1020.DOI: 10.1002/bit.23039 |
| [34] | Crasson O, Rhazi N, Jacquin O, et al. Enzymatic functionalization of a nanobody using protein insertion technology.Prot Eng Des Select, 2015, 28(10): 451–460.DOI: 10.1093/protein/gzv020 |
| [35] | Aroul-Selvam R, Hubbard T, Sasidharan R. Domain insertions in protein structures.J Mol Biol, 2004, 338(4): 633–641.DOI: 10.1016/j.jmb.2004.03.039 |
| [36] | Selvam RA, Sasidharan R. DomIns:a web resource for domain insertions in known protein structures.Nucl Acids Res, 2004, 32(S1): D193–D195. |
| [37] | Yudin AK. Macrocycles:lessons from the distant past, recent developments, and future directions.Chem Sci, 2015, 6(1): 30–49.DOI: 10.1039/C4SC03089C |
| [38] | Ribeiro LF, Furtado GP, Lourenzoni MR, et al. Engineering bifunctional laccase-xylanase chimeras for improved catalytic performance.J Biol Chem, 2011, 286(50): 43026–43038.DOI: 10.1074/jbc.M111.253419 |
| [39] | Ribeiro LF, Nicholes N, Tullman J, et al. Insertion of a xylanase in xylose binding protein results in a xylose-stimulated xylanase.Biotechnol Biof, 2015, 8(1): 118.DOI: 10.1186/s13068-015-0293-0 |
| [40] | Schoffelen S, Van Hest JCM. Multi-enzyme systems:bringing enzymes together in vitro.Soft Matt, 2012, 8(6): 1736–1746.DOI: 10.1039/C1SM06452E |
| [41] | Domeradzka NE, Werten MW, De Wolf FA, et al. Protein cross-linking tools for the construction of nanomaterials.Curr Opin Biotechnol, 2016, 39: 61–67. |
| [42] | Heck T, Faccio G, Richter M, et al. Enzyme-catalyzed protein crosslinking.Appl Microbiol Biotechnol, 2013, 97(2): 461–475.DOI: 10.1007/s00253-012-4569-z |
| [43] | Hirakawa H, Kamiya N, Tanaka T, et al. Intramolecular electron transfer in a cytochrome P450cam system with a site-specific branched structure.Prot Eng Des Select, 2007, 20(9): 453–459.DOI: 10.1093/protein/gzm045 |
| [44] | Temperton B, Giovannoni SJ. Metagenomics:microbial diversity through a scratched lens.Curr Opin Microbiol, 2012, 15(5): 605–612.DOI: 10.1016/j.mib.2012.07.001 |
| [45] | Quail MA, Smith M, Coupland P, et al. A tale of three next generation sequencing platforms:comparison of Ion Torrent, Pacific Biosciences and Illumina MiSeq sequencers.BMC Genomics, 2012, 13(1): 341.DOI: 10.1186/1471-2164-13-341 |
| [46] | Dimmer EC, Huntley RP, Alam-Faruque Y, et al. The UniProt-GO annotation database in 2011.Nucleic Acids Res, 2011, 40(D1): D565–D570. |
| [47] | Maia FRNC, Hajdu J. The trickle before the torrent-diffraction data from X-ray lasers.Sci Data, 2016, 3: 160059.DOI: 10.1038/sdata.2016.59 |
| [48] | Tian L, Liu SJ, Wang S, et al. Ligand-binding specificity and promiscuity of the main lignocellulolytic enzyme families as revealed by active-site architecture analysis.Sci Rep, 2016, 6(1): 23605.DOI: 10.1038/srep23605 |
| [49] | Yoo YJ, Feng Y, Kim YH, et al. Fundamentals of Enzyme Engineering.Netherlands:Springer, 2017: 87–100. |
| [50] | Furnham N, de Beer TA, Thornton JM. Current challenges in genome annotation through structural biology and bioinformatics.Curr Opin Struct Biol, 2012, 22(5): 594–601.DOI: 10.1016/j.sbi.2012.07.005 |
| [51] | Baker D, Sali A. Protein structure prediction and structural genomics.Science, 2001, 294(5540): 93–96.DOI: 10.1126/science.1065659 |
| [52] | Finn RD, Bateman A, Clements J, et al. Pfam:the protein families database.Nucleic Acids Res, 2013, 42(D1): D222–D230. |
| [53] | Finn RD, Coggill P, Eberhardt RY, et al. The Pfam protein families database:towards a more sustainable future.Nucleic Acids Res, 2016, 44(D1): D279–D285.DOI: 10.1093/nar/gkv1344 |
| [54] | Prli? A, Kalro T, Bhattacharya R, et al. Integrating genomic information with protein sequence and 3D atomic level structure at the RCSB protein data bank.Bioinformatics, 2016, 32(24): 3833–3835.DOI: 10.1093/bioinformatics/btw547 |
| [55] | Friedberg I. Automated protein function prediction-the genomic challenge.Briefings in Bioinf, 2006, 7(3): 225–242.DOI: 10.1093/bib/bbl004 |
| [56] | Lee D, Redfern O, Orengo C. Predicting protein function from sequence and structure.Nature reviews Mol Cell Biol, 2007, 8(12): 995–1005.DOI: 10.1038/nrm2281 |
| [57] | Gu J, Bourne PE. Structural bioinformatics. 2nd ed. Hoboken, NJ, USA: John Wiley & Sons, 2009: 3-14. |
| [58] | Prakash T, Taylor TD. Functional assignment of metagenomic data:challenges and applications.Brief Bioinf, 2012, 13(6): 711–727.DOI: 10.1093/bib/bbs033 |
| [59] | Sillitoe I, Cuff AL, Dessailly BH, et al. New functional families (FunFams) in CATH to improve the mapping of conserved functional sites to 3D structures.Nucl Acids Res, 2012, 41(D1): D490–D498.DOI: 10.1093/nar/gks1211 |
| [60] | Aspeborg H, Coutinho PM, Wang Y, et al. Evolution, substrate specificity and subfamily classification of glycoside hydrolase family 5 (GH5).BMC Evolut Biol, 2012, 12(1): 186.DOI: 10.1186/1471-2148-12-186 |
| [61] | Niu CT, Zhu LJ, Xu X, et al. Rational design of thermostability in bacterial 1, 3-1, 4-β-glucanases through spatial compartmentalization of mutational hotspots.Appl Microbiol Biotechnol, 2017, 101(3): 1085–1097.DOI: 10.1007/s00253-016-7826-8 |
| [62] | Liu SJ, Shao SJ, Li LL, et al. Substrate-binding specificity of chitinase and chitosanase as revealed by active-site architecture analysis.Carbohyd Res, 2015, 418: 50–56.DOI: 10.1016/j.carres.2015.10.002 |
| [63] | Harms MJ, Thornton JW. Analyzing protein structure and function using ancestral gene reconstruction.Curr Opin Struct Biol, 2010, 20(3): 360–366.DOI: 10.1016/j.sbi.2010.03.005 |
| [64] | Jiang XK, Chen GJ, Wang LS. Structural and dynamic evolution of the amphipathic N-terminus diversifies enzyme thermostability in the glycoside hydrolase family 12.Phys Chem Chem Phys, 2016, 18(31): 21340–21350.DOI: 10.1039/C6CP02998A |
| [65] | Sun JY, Liu MQ, Xu YL, et al. Improvement of the thermostability and catalytic activity of a mesophilic family 11 xylanase by N-terminus replacement.Prot Exp Purificat, 2005, 42(1): 122–130.DOI: 10.1016/j.pep.2005.03.009 |
| [66] | Jiang XK, Li W, Chen GJ, et al. Dynamic perturbation of the active site determines reversible thermal inactivation in glycoside hydrolase family 12.J Chem Informat Model, 2017, 57(2): 288–297.DOI: 10.1021/acs.jcim.6b00692 |
| [67] | Kapoor D, Kumar V, Chandrayan SK, et al. Replacement of the active surface of a thermophile protein by that of a homologous mesophile protein through structure-guided 'protein surface grafting'.Biochim Biophys Acta (BBA)-Prot Proteom, 2008, 1784(11): 1771–1776.DOI: 10.1016/j.bbapap.2008.05.007 |
| [68] | Raghunathan G, Sokalingam S, Soundrarajan N, et al. Modulation of protein stability and aggregation properties by surface charge engineering.Mol BioSyst, 2013, 9(9): 2379–2389.DOI: 10.1039/c3mb70068b |
| [69] | Sokalingam S, Raghunathan G, Soundrarajan N, et al. A study on the effect of surface lysine to arginine mutagenesis on protein stability and structure using green fluorescent protein.PLoS ONE, 2012, 7(7): e40410.DOI: 10.1371/journal.pone.0040410 |
| [70] | Graziano G, Merlino A. Molecular bases of protein halotolerance.Biochim Biophys Acta (BBA)-Prote Proteom, 2014, 1844(4): 850–858.DOI: 10.1016/j.bbapap.2014.02.018 |
| [71] | Kern M, McGeehan JE, Streeter SD, et al. Structural characterization of a unique marine animal family 7 cellobiohydrolase suggests a mechanism of cellulase salt tolerance.Proc Natl Acad Sci USA, 2013, 110(25): 10189–10194.DOI: 10.1073/pnas.1301502110 |
| [72] | Fushinobu S, Ito K, Konno M, et al. Crystallographic and mutational analyses of an extremely acidophilic and acid-stable xylanase:biased distribution of acidic residues and importance of Asp37 for catalysis at low pH.Prot Eng, 1998, 11(12): 1121–1128.DOI: 10.1093/protein/11.12.1121 |
| [73] | Shirai T, Suzuki A, Yamane T, et al. High-resolution crystal structure of M-protease:phylogeny aided analysis of the high-alkaline adaptation mechanism.Prot Eng, 1997, 10(6): 627–634.DOI: 10.1093/protein/10.6.627 |
| [74] | Umemoto H, Ihsanawati, Inami M, et al. Improvement of alkaliphily of Bacillus alkaline xylanase by introducing amino acid substitutions both on catalytic cleft and protein surface.Biosc, Biotechnol, Biochem, 2009, 73(4): 965–967.DOI: 10.1271/bbb.80869 |
| [75] | Shivange AV, Hoeffken HW, Haefner S, et al. Protein consensus based surface engineering (ProCoS):a computer-assisted method for directed protein evolution.BioTechniques, 2016, 61(6): 305–314. |
| [76] | Warden AC, Williams M, Peat TS, et al. Rational engineering of a mesohalophilic carbonic anhydrase to an extreme halotolerant biocatalyst.Nat Commun, 2015, 6: 10278.DOI: 10.1038/ncomms10278 |
| [77] | Woodley JM. Protein engineering of enzymes for process applications.Curr Opin Chem Biol, 2013, 17(2): 310–316.DOI: 10.1016/j.cbpa.2013.03.017 |
| [78] | Kiss G, ?elebi-?l?üM N, Moretti R, et al. Computational enzyme design.Angewandte Chemie Int Edition, 2013, 52(22): 5700–5725.DOI: 10.1002/anie.201204077 |
| [79] | Halabi N, Rivoire O, Leibler S, et al. Protein sectors:evolutionary units of three-dimensional structure.Cell, 2009, 138(4): 774–786.DOI: 10.1016/j.cell.2009.07.038 |
| [80] | Csermely P, Korcsmáros T, Kiss HJ, et al. Structure and dynamics of molecular networks:a novel paradigm of drug discovery:a comprehensive review.Pharmacol Therap, 2013, 138(3): 333–408.DOI: 10.1016/j.pharmthera.2013.01.016 |
| [81] | Reynolds KA, Mclaughlin RN, Ranganathan R. Hot spots for allosteric regulation on protein surfaces.Cell, 2011, 147(7): 1564–1575.DOI: 10.1016/j.cell.2011.10.049 |
| [82] | Csermely P, Palotai R, Nussinov R. Induced fit, conformational selection and independent dynamic segments:an extended view of binding events.Trends Biochem Sci, 2010, 35(10): 539–546.DOI: 10.1016/j.tibs.2010.04.009 |
| [83] | Mclaughlin RN Jr. , Poelwijk FJ, Raman A, et al.The spatial architecture of protein function and adaptation. Nature, 2012, 491(7422): 138–142. |
| [84] | Marks DS, Hopf TA, Sander C. Protein structure prediction from sequence variation.Nat Biotechnol, 2012, 30(11): 1072–1080.DOI: 10.1038/nbt.2419 |
| [85] | Hopf TA, Sch?rfe CP, Rodrigues JP, et al. Sequence co-evolution gives 3D contacts and structures of protein complexes.Elife, 2014, 3: e03430. |
| [86] | Morcos F, Pagnani A, Lunt B, et al. Direct-coupling analysis of residue coevolution captures native contacts across many protein families.Proc Natl Acad Sci USA, 2011, 108(49): 19459–19460. |
| [87] | Jones DT, Buchan DW, Cozzetto D, et al. PSICOV:precise structural contact prediction using sparse inverse covariance estimation on large multiple sequence alignments.Bioinformatics, 2011, 28(2): 184–190. |
| [88] | Weigt M, White RA, Szurmant H, et al. Identification of direct residue contacts in protein-protein interaction by message passing.Proc Natl Acad Sci USA, 2009, 106(1): 67–72.DOI: 10.1073/pnas.0805923106 |
| [89] | Hopf TA, Colwell LJ, Sheridan R, et al. Three-dimensional structures of membrane proteins from genomic sequencing.Cell, 2012, 149(7): 1607–1621.DOI: 10.1016/j.cell.2012.04.012 |
| [90] | Su?kowska JI, Morcos F, Weigt M, et al. Genomics-aided structure prediction.Proc Natl Acad Sci USA, 2012, 109(26): 10340–10345.DOI: 10.1073/pnas.1207864109 |
| [91] | Nugent T, Jones DT. Accurate de novo structure prediction of large transmembrane protein domains using fragment-assembly and correlated mutation analysis.Proc Natl Acad Sci USA, 2012, 109(24): E1540–E1547.DOI: 10.1073/pnas.1120036109 |
| [92] | Marks DS, Colwell LJ, Sheridan R, et al. Protein 3D structure computed from evolutionary sequence variation.PLoS ONE, 2011, 6(12): e28766.DOI: 10.1371/journal.pone.0028766 |
| [93] | Liu JT, Duan XY, Sun JY, et al. Bi-factor analysis based on noise-reduction (BIFANR):a new algorithm for detecting coevolving amino acid sites in proteins.PLoS ONE, 2013, 8(11): e79764.DOI: 10.1371/journal.pone.0079764 |
| [94] | Ho ML, Adler BA, Torre ML, et al. SCHEMA computational design of virus capsid chimeras:calibrating how genome packaging, protection, and transduction correlate with calculated structural disruption.ACS Synth Biol, 2013, 2(12): 724–733.DOI: 10.1021/sb400076r |
| [95] | Kufner K, Lipps G. Construction of a chimeric thermoacidophilic beta-endoglucanase.BMC Biochem, 2013, 14(1): 1–9. |
| [96] | Gibson DG, Glass JI, Lartigue C, et al. Creation of a bacterial cell controlled by a chemically synthesized genome.Science, 2010, 329(5987): 52–56.DOI: 10.1126/science.1190719 |
| [97] | Huang ZL, Zhang C, Wu X, et al. Recent progress in fusion enzyme design and applications.Chin J Biotech, 2012, 28(4): 393–409.(in Chinese). 黄子亮, 张翀, 吴希, 等. 融合酶的设计和应用研究进展.生物工程学报, 2012, 28(4): 393-409. |
| [98] | Elleuche S. Bringing functions together with fusion enzymes-from nature's inventions to biotechnological applications.Appl Microbiol Biotechnol, 2015, 99(4): 1545–1556.DOI: 10.1007/s00253-014-6315-1 |
| [99] | Consortium EP. An integrated encyclopedia of DNA elements in the human genome.Nature, 2012, 489(7414): 57–74.DOI: 10.1038/nature11247 |
| [100] | Krampis K, Booth T, Chapman B, et al. Cloud BioLinux:pre-configured and on-demand bioinformatics computing for the genomics community.BMC Bioinform, 2012, 13(1): 42.DOI: 10.1186/1471-2105-13-42 |
| [101] | Marx V. Biology:The big challenges of big data.Nature, 2013, 498(7453): 255–260.DOI: 10.1038/498255a |
